

1. 山西农业大学 农学院,山西 太谷 030801;
2. 山西农业大学 分子农业与生物能源研究所,山西 太谷 030801;
3. 中国科学院烟台海岸带研究所,山东 烟台 264003
收稿日期:2020-07-27;接收日期:2020-11-26;网络出版时间:2020-12-22
基金项目:国家自然科学基金(No. 31902394),山西省重点研发计划(No. 201803D31063),山西省应用基础研究项目(No. 201801D22125),山西农业大学科技创新基金项目(No. 2018YJ16),国家海洋局海洋生物活性物质与现代分析技术重点实验室项目(No. MBSMAT-2016-03) 资助
*These authors contributed equally to the work.
摘要:雨生红球藻Haematococcus pluvialis在逆境胁迫条件下可大量积累虾青素,被认为自然界最理想的虾青素生产者。高光能有效诱导其虾青素的合成与积累,但藻细胞感知和转导光信号进而调控虾青素积累的机制尚不清楚。文中采用Illumina Hiseq 2000高通量测序技术分别获得正常、高白光及高蓝光处理组4.0 G、3.8 G及3.6 G的原始数据量,经质控与拼接之后获得51 954条长度至少为200 bp的unigenes基因,经过比对分析,共有20 537个unigenes在NR、NT、KO、SwissProt、PfamGO及KOG等数据库中的至少1个数据库中注释成功,达到39.52%。差异表达基因分析显示,高白光vs正常组共获得1 255个DEGs;高蓝光vs正常组共获得1 494个DEGs;高白光与高蓝光vs正常组共同的DEGs有1 008个。KEGG富集分析显示,与对照组相比高白光与高蓝光共同的显著富集通路包括光合作用、类胡萝卜素合成、脂肪酸合成、氧化磷酸化、DNA复制、碳代谢及氮代谢等过程。通过对转录组数据进一步分析,挖掘鉴定了大量光受体及其信号转导通路中的互作蛋白。随机筛选DEGs基因15条,采用荧光定量PCR技术检测其转录水平,结果表明与转录组差异表达数据高度一致。光受体及其信号转导通路中的互作蛋白基因差异表达分析,推测“光信号→光受体→互作蛋白(互作蛋白→转录因子/转录调节子) →功能基因表达→虾青素积累”的信号转导通路可能参与上述调控过程,为深入解析光诱导虾青素合成的转录调控机制奠定了基础。
关键词:雨生红球藻高光虾青素转录组测序光信号通路
Transcriptome analysis of signal transduction pathway involved in light inducing astaxanthin accumulation in Haematococcus pluvialis
Hongli Cui1,2*, Wenxin Xu1,2*, Yulin Cui3, Chunli Ji1,2, Chunhui Zhang1,2, Song Qin3, Runzhi Li1,2


1. College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi, China;
2. Institute of Molecular Agriculture and Bioenergy, Shanxi Agricultural University, Taigu 030801, Shanxi, China;
3. Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003, Shandong, China
Received: July 27, 2020; Accepted: November 26, 2020; Published: December 22, 2020
Supported by: National Natural Science Foundation of China (No. 31902394), Key Research and Development (R & D) Planning Project of Shanxi Province, China (No. 201803D31063), Applying Basic Research Planning Project of Shanxi Province, China (No. 201801D22125), Science and Technology Innovation Planning Project of Shanxi Agricultural University (No. 2018YJ16), Key Lab of Marine Bioactive Substance and Modern Analytical Technique (No. MBSMAT-2016-03).*
Corresponding author: Runzhi Li. Tel: +86-354-6288344; E-mail: rli2001@126.com.
Abstract: The unicellular green alga Haematococcus pluvialis is the best source of natural astaxanthin (AST) in the world due to its high content under stress conditions. Although high light (HL) can effectively induce AST biosynthesis, the specific mechanisms of light signal perception and transduction are unclear. In the current study, we used transcriptomic data of normal (N), high white light (W), and high blue light (B) to study the mechanisms of light inducing AST accumulation from the point of photoreceptors. The original data of 4.0 G, 3.8 G, and 3.6 G for N, W, and B were obtained, respectively, by the Illumina Hi-seq 2000 sequencing technology. Totally, 51 954 unigenes (at least 200 bp in length) were generated, of which, 20 537 unigenes were annotated into at least one database (NR, NT, KO, SwissProt, Pfam, GO, or KOG). There were 1 255 DEGs in the W vs N, 1 494 DEGs in the B vs N, and 1 008 DEGs in the both W vs N and B vs N. KEGG enrichment analysis revealed that photosynthesis, oxidative phosphorylation, carotenoid biosynthesis, fatty acids biosynthesis, DNA replication, nitrogen metabolism, and carbon metabolism were the significantly enriched pathways. Moreover, a large number of genes encoding photoreceptors and predicted interacting proteins were predicted in Haematococcus transcriptome data. These genes showed significant differences at transcriptional expression levels. In addition, 15 related DEGs were selected and tested by qRT-PCR and the results were significantly correlated with the transcriptome data. The above results indicate that the signal transduction pathway of "light signal - photoreceptors - interaction proteins - (interaction proteins - transcription factor/transcriptional regulator) - gene expression - AST accumulation" might play important roles in the regulation process, and provide reference for further understanding the transcriptional regulation mechanisms of AST accumulation under HL stress.
Keywords: Haematococcus pluvialishigh lightastaxanthintranscriptomelight signal pathway
虾青素是一种具有超强抗氧化活性的次生类胡萝卜素,被称为“抗氧化剂之王”,具有良好的着色效果和重要的生物学功能,已被广泛应用于水产养殖、食品工业、医疗卫生、化妆及保健品等领域[1]。雨生红球藻Haematococcus pluvialis是一种淡水单细胞绿藻,是目前已知虾青素含量最高的物种,在胁迫条件下虾青素含量可达细胞干重的4%,是极为理想的天然虾青素来源[2]。然而,藻细胞中虾青素的积累是一个胁迫诱导的过程,即虾青素的合成伴随着大量游动细胞转化为厚壁孢子,此时藻细胞的生长受到严重的抑制,这种矛盾已经成为利用雨生红球藻制备虾青素的限制因子[3-4]。因此,阐明虾青素合成途径、挖掘高效胁迫因子及解析诱导信号通路是解决上述矛盾的关键,也是雨生红球藻制备虾青素产业目前亟需解决的理论与技术瓶颈。
雨生红球藻虾青素的积累受到多种因素的影响,包括光、营养成分、生物节律、活性氧、光合作用、发育过程、表观遗传、自身反馈及某些代谢物等[5],但大多数研究者认为光照是影响雨生红球藻虾青素合成的重要因子[5-8]。前期工作和同行文献表明,在同等光强条件下,与自然光(白光) 相比,蓝光能够更有效地诱导雨生红球藻虾青素的积累,且虾青素合成相关基因在转录水平的表达显著增高[6-9]。蓝光诱导虾青素的积累与相关基因在转录水平的高表达密不可分,但藻细胞感知和转导光信号进而激活基因转录水平的分子机制不清楚。植物依靠光受体,例如蓝光受体向光素PHOT (Phototropin)、隐花色素CRY (Cryptochrome)及紫外光受体UVR8 (UV resistance locus 8) 通过构象改变和磷酸化等感知光信号,并通过多种互作蛋白形成复杂的接收和转导系统,并与体内其他信号转导途径耦合,进而调控基因的表达,最终调控光响应生理过程[10-15]。对真核微藻而言,在已知的光受体中,CRYs以多个等位基因形式广泛分布于真核微藻[16],而PHOT和UVR8则是单个等位基因形式仅存在于真核绿藻[17-18]。突变和RNA干扰实验验证,莱茵衣藻Chlamydomonas reinhardtii中CrePHOT蛋白参与细胞有性生活周期,并能调控捕光复合物、叶绿素和类胡萝卜素的合成等靶基因转录水平的表达[19],这为从光受体角度研究高光诱导雨生红球藻虾青素积累的转录调控机制提供了新思路。
近年来,转录组学技术为揭示生物信号感知与代谢调控机制提供了强有力的工具。例如基于基因组和转录组结合技术对雨生红球藻虾青素合成、积累、调控与转录水平的可变性剪切进行了研究[20]。大量研究者采用转录组测序技术针对胁迫因子诱导雨生红球藻虾青素和脂肪酸积累及相关基因在转录水平的表达进行了广泛的研究,包括不同光质和光强[21]、添加碘化钾[22]、氮缺乏[23]、细胞周期[24-25]、高光诱导下添加醋酸钠和Fe2+[26]、高光诱导下添加茉莉酸甲酯和水杨酸[27]及高CO2浓度等[28]。但上述研究通常重点关注诱导条件对雨生红球藻生长、生理及虾青素积累的影响与其合成途径相关基因的表达模式,对其转录调控机制的研究较少,尤其是未见光受体及其信号通路的相关报道。
针对高光诱导雨生红球藻虾青素积累的转录调控机制不清楚的现状,本文利用转录组测序技术分析了高光处理后的转录差异,对差异表达基因进行了聚类分析,重点分析了光受体及其互作蛋白,研究旨在初步推测存在光受体介导的高光诱导雨生红球藻虾青素积累的信号转导通路,为进一步揭示其转录调控机制提供新视角与奠定基础。
1 材料与方法1.1 雨生红球藻的培养及测序样品制备本实验所用Haematococcus pluvialis Flotow 1844购自藻类和原生生物培养中心(英国CCAP藻种库:https://www.ccap.ac.uk/),由本实验室保存。采用BBM培养基,培养条件为光强25 μmol/(m2·s) (LED白光),温度22±1 ℃,光/暗周期12 h/12 h,静置培养每天摇动2-3次。针对高光诱导培养,取正常培养处于指数生长后期的藻细胞经过黑暗处理24 h后,重新悬浮于BBM培养基中,每组设置3个生物学重复并分别置于正常培养(LED白光,25 μmol/(m2·s))、高白光(390-770 nm,500 μmol/(m2·s))和高蓝光(420-500 nm,500 μmol/(m2·s)) 条件下,于72 h离心收集藻细胞,用PBS缓冲液冲洗两次,样品于液氮中保存。将每组3个生物学重复样品混合测序。参照Plant RNA Purification Reagent试剂盒进行总RNA提取,并利用Nanodrop2000进行浓度和纯度(OD260与OD280比值) 检测,用1%琼脂糖凝胶电泳检测其完整性,最后用Agilent 2100测定其RIN值。cDNA文库构建与测序委托北京诺禾致源生物信息科技有限公司完成。利用带Oligo (dT) 的磁珠进行mRNA的富集,加入裂解液将mRNA随机打断,合成第一条cDNA链,进而合成第二条cDNA链,利用AMPure XP beads纯化cDNA,进而经过末端修复,进行片段大小筛选,最终通过PCR富集得到cDNA文库。采用Illumina Hiseq-2000双端测序,测序读长为PE100。
1.2 雨生红球藻转录组数据预处理及拼接高通量测序获得的原始图像数据文件经CASAVA碱基识别(Base Calling) 分析转化为原始测序序列(Sequenced Reads) 进而经过错误率检查、A/T/G/C含量分布及接头序列去除得到有效测序数据(Clean reads)。采用Trinity软件(版本:v2012-10-05;参数设置:min_kmer_cov为2,其他参数为默认参数) 进行转录本拼接[29],形成一个转录组,以此作为后续分析的参考序列,取每条基因中最长的转录本作为Unigene,并对转录本及Unigene的长度进行统计。
1.3 雨生红球藻转录组基因功能注释、分类及蛋白编码序列(CDS) 预测将获得的Unigene基因序列与Nr、PDB、PIR、PRF、Nt、Pfam、KOG、COG、KEGG及GO等数据库进行比对注释与分类。将Unigene基因比对到蛋白库,进行ORF编码框的提取;或者采用estscan软件预测ORF,从而得到核酸序列和氨基酸序列。
1.4 雨生红球藻转录组基因差异表达及富集分析将Trinity拼接得到的转录组作为参考序列,采用RSEM软件将每个样品的clean reads与参考序列进行匹配[30],对bowtie的比对结果进行统计,进一步得到了每个样品比对到每个基因上的read count数目,并对其进行FPKM转换[31],对于无生物学重复的样品,先采用TMM对read count数据进行标准化处理,再用DEGseq进行差异分析,筛选阈值为qvalue < 0.005且|log2FoldChange| > 1。采用DESeq软件[32]以对其进行筛选,筛选阈值为padj < 0.05。根据实验目的筛选差异基因后,采用GO和KEGG进行富集分析。本文中涉及高光诱导下雨生红球藻转录组原始数据和处理结果都已经提交到国家微生物科学数据中心(NMDCX0000104)。
1.5 雨生红球藻转录组中光受体及其互作蛋白的预测及分析用高等植物和真核微藻中功能验证的光受体、互作蛋白及其调控靶基因为源序列(具体信息见网络版附表S1),采用本地BLAST搜索雨生红球藻转录组中光受体及其互作蛋白同源序列,结合功能注释进一步补充完善,并进行基因差异表达分析。
1.6 重要功能基因qRT-PCR验证随机选择高光诱导下高表达DEGs基因6个(PSY、BCH、BKT、DGAT1-1、DGAT2-2、DGAT2-5)、低表达DEGs基因3个(PEPS、3-KCT、PKS)、光受体基因2个(PHOT、CPF-2) 及其信号转导通路互作蛋白基因4个(WRKY-2、MUB-2、COP1、SPA1),共15个基因。根据预测基因的CDS序列设计荧光定量特异性引物。雨生红球藻β-actin作为内参基因,所有的引物由生工生物工程(上海) 股份有限公司合成,引物序列见表 1。采用宝生物(SYBR Premix Ex Taq Ⅱ) 荧光定量试剂盒进行qRT-PCR检测。程序条件如下:95 ℃ 10 min,95 ℃ 5 s,60 ℃ 30 s,40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 30 s,60 ℃ 15 s。每个样品3个平行,采用相对定量2–ΔΔCT法来验证不同高光诱导72 h条件下选择目的基因的转录水平[33]。
表 1 荧光定量qRT-PCR引物Table 1 Primer sequences of qRT-PCR
| DEGs | Transcriptome data | Gene name | Sequence (5′–3′) | Size |
| Up | comp57514_c0 | PSY (phytoene synthase) | F: CCAAACGCCAATAAGATAACG R: TGCTCTGTCAAGCCAAACTG | 442 |
| Up | comp56899_c0 | BCH (beta-carotene hydroxylase) | F: AATCAGCAAACTGCGGACGAA R: TAGGTGGCAAGGATGGCAAG | 149 |
| Up | comp57659_c2 | BKT (beta-carotene ketolase) | F: CAGTGTTCCTCCACGCCATTT R: GCATACTCTGCCCAAGAAGT | 245 |
| Up | comp59843_c2 | DGAT1-1 (diacylglycerol acyltransferase) | F: TATGTCAGGCAGCACGCCGGT R: CCAATGGTGATGGAGCCAAA | 204 |
| Up | comp54162_c0 | DGAT2-2 (diacylglycerol acyltransferase) | F: CTCCGTGGCGGTGATAGTAG R: GATCTCCAGCGGCTTGTCCT | 432 |
| Up | comp54843_c0 | DGAT2-5 (diacylglycerol acyltransferase) | F: TGTACCTACGCAAGCGCCACG R: ACCCAGCGAACCAGCCAGTC | 137 |
| Down | comp41479_c0 | PEPs (phosphoenolpyruvate synthase) | F: CGCGCCGGGATCGAGGCCCAG R: GAACCCCTGATGCACGCGAT | 274 |
| Down | comp51697_c1 | 3-KCT (3-ketoacyl-CoA thiolase) | F: GTGGCGCAGGCCGCCGACGC R: CTGGATCGCGGCCATGGCCTTC | 258 |
| Down | comp56566_c0 | PKs (pyruvate kinase) | F: TCTTTCCAGGGGCATCAGGACG R: AAGGTCAGCCACTCCACGTC | 234 |
| Up | comp61615_c0 | PHOT (phototropin) | F: TGATGCTACCCTACCTGATATG R: TGACTGCCTTGCCCTCTGTTT | 323 |
| Up | comp59629_c0 | CPF-2 (cryptochrome/photolyase family) | F: GTGGCGGGATTTCTTTCGCTTC R: CGTTCTGCCTGCCTCGGTTG | 209 |
| Up | comp56997_c0 | WRKY-2 (wrky) | F: TCGGAGACCTTGCTGATGACG R: CCCTTTACAAACTTCTGCCCA | 295 |
| Up | comp57039_c0 | MYB-3 (myb) | F: TATTGGGCACTTAACTGTTTC R: TTGCTGGCATTGGGTATCCTT | 215 |
| Up | comp55814_c0 | COP1 (constitutively photomorphogenic1) | F: CGGATACAGGAGCACTTGGAT R: GACGGCAGGTGAGTCTTGG | 310 |
| Down | comp58791_c1 | SPA1 (suppressor of PHYA-105 1) | F: TGCCATCTGATAGCTGGCT R: TTCGGATTGGAATGCTTGCA | 241 |
| Control | comp59437_c0 | ACTIN (actin) | F: AGCGGGAGATAGTGCGGGACA R: ATGCCCACCGCCTCCATGC | 189 |
表选项
2 结果与分析2.1 雨生红球藻转录组测序、拼接、功能注释及蛋白编码序列预测对照(N)、高蓝光(B) 及高白光(W) 处理组分别获得4.0 G、3.8 G及3.6 G的有效数据。通过拼接获得131 385个转录本(transcripts) 和51 954个非重复序列基因(unigenes),其中长度大于2 000 bp的分别达到18 508个和4 284个(表 2)。
表 2 雨生红球藻转录组transcript和Unigene的序列大小Table 2 Sequence size (bp) and number of transcript and Unigene in Haematococcus transcriptome
| Transcript length interval | 200–500 bp | 500–1 000 bp | 1 000–2 000 bp | > 2 000 bp | Total |
| Number of transcripts | 51 222 | 30 279 | 31 376 | 18 508 | 131 385 |
| Number of unigenes | 32 284 | 8 412 | 6 974 | 4 284 | 51 954 |
表选项
基于7个数据库的比对,共有20 537个unigenes在NR、NT、KO、SwissProt、Pfam、GO及KOG等数据库中至少1个数据库中注释成功,注释率达到39.52%。其中在GO、Pfam及NR数据库的注释率较高,分别达到32.28% (16 772条)、30.14% (15 660条) 及28.32% (14 718条)。共有1 424条unigene在所有数据库同时注释,注释率达到2.74% (表 3)。
表 3 雨生红球藻转录组Unigene的功能注释统计结果Table 3 Statistics of annotation results of Unigene functional annotation in Haematococcus transcriptome
| Number of Unigenes | Percentage (%) | |
| Annotated in NR | 14 718 | 28.32 |
| Annotated in NT | 6 582 | 12.66 |
| Annotated in KO | 4 151 | 7.98 |
| Annotated in SwissProt | 11 145 | 21.45 |
| Annotated in Pfam | 15 660 | 30.14 |
| Annotated in GO | 16 772 | 32.28 |
| Annotated in KOG | 7 640 | 14.70 |
| Annotated in all Databases | 1 424 | 2.74 |
| Annotated in at least one Database | 20 537 | 39.52 |
表选项
2.2 雨生红球藻转录组基因差异表达分析通过基因差异表达分析得到,高蓝光vs正常组共获得1 494个DEGs,其中上调基因(Up-regulated genes) 数量为732个,下调基因(Down-regulated genes) 数量为762个;
高白光vs正常组共获得1 255个DEGs,其中上调基因(Up-regulated genes) 数量为664个,下调基因(Down-regulated genes) 数量为591个;高白光与高蓝光vs正常组共同的DEGs有1 008个(图 1A–B)。
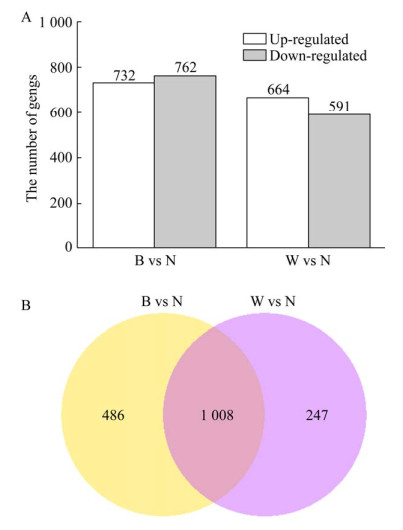 |
| 图 1 雨生红球藻高光诱导下基因差异表达分析 Fig. 1 Analysis of DEGs in Haematococcus transcriptome. |
| 图选项 |
2.3 雨生红球藻转录组差异表达基因的GO注释分析为了研究不同处理组间的基因功能差异,对上述差异表达的基因进行GO功能注释级。结果显示,高蓝光与对照相比有1 025个DEGs注释到GO数据库的生物过程(BP)、细胞组分(CC)及分子功能(MF) 3大类型,其中上调基因511个,下调基因514个。其显著性富集通路有30条,其中生物过程(BP) 8条、细胞组分(CC) 20条及分子功能(MF) 2条。主要涉及具体通路如图 2所示。高白光组与对照组相比有877个DEGs注释到GO数据库3大类型,其中上调基因477个,下调基因400个(图 2)。其显著性富集通路有34条,其中生物过程(BP) 13条、细胞组分(CC) 17条及分子功能(MF) 4条。主要涉及具体通路如图 2A–B所示。有趣的是,在高蓝光和高白光诱导下,共有的显著富集通路涉及光合作用(Photosynthesis)、光合系统(Photosystem)、抗氧化活力(Antioxidant activity) 及代谢过程(Metabolic process)。
 |
| 图 2 雨生红球藻转录组中高光诱导下差异表达基因的GO二级节点注释 Fig. 2 The GO annotation of DEGs in Haematococcus transcriptome. |
| 图选项 |
2.4 雨生红球藻转录组差异表达基因的KEGG富集分析为了明确不同处理组间的差异表达代谢通路,采用KEGG进行差异表达基因代谢通路富集分析。结果显示(表 4),高蓝光组与对照组相比,共有1 328个差异基因注释到KEGG途径的218条具体分支通路,其中上调基因显著富集的5条分支通路包括光合作用(Photosynthesis) 相关基因19个、核糖体(Ribosome) 相关基因32个、氧化磷酸化(Oxidative phosphorylation) 相关基因26个、类胡萝卜素合成(Carotenoid biosynthesis)相关基因6个及不饱和脂肪酸合成(Biosynthesis of unsaturated fatty acids) 相关基因3个;下调基因显著富集的5条分支通路包括DNA复制(DNA replication) 相关基因4个、氮代谢(Nitrogen metabolism) 相关基因4个、碳代谢(Carbon metabolism) 相关基因13个、光合作用中的碳固定(Carbon fixation in photosynthetic organisms) 相关基因7个及嘧啶代谢(Pyrimidine metabolism) 相关基因14个。高白光组与对照组相比,共有1 121个差异基因注释到KEGG途径的215条具体分支通路,其中上调基因显著富集的5条分支通路包括光合作用(Photosynthesis) 相关基因15个、光合作用天线蛋白(Photosynthesis-antenna proteins)相关基因7个、脂肪酸合成(Fatty acid biosynthesis) 相关基因5个、类胡萝卜素合成(Carotenoid biosynthesis) 相关基因5个及氧化磷酸化(Oxidative phosphorylation) 相关基因22个;下调基因显著富集的5条分支通路包括DNA复制(DNA replication) 相关基因13个、嘧啶代谢(Pyrimidine metabolism) 相关基因11个、碳代谢(Carbon metabolism) 相关基因12个、氮代谢(Nitrogen metabolism) 相关基因4个及光转导(Phototransduction) 相关基因2个。具体参与显著富集通路的基因见附表 2 (见网络版)。有趣的是,无论是高白光还是高蓝光,在上调表达基因的KEGG富集通路中共有脂肪酸合成与类胡萝卜素合成通路,暗示高光诱导下雨生红球藻中类胡萝卜素和脂肪酸的积累与这些基因的高表达密不可分。
表 4 雨生红球藻转录组显著KEGG富集通路Table 4 The significant KEGG enrichment pathway in Haematococcus transcriptome
| Group | KEGG ID | Annotation description | P-value | Gene number |
| B vs N Up | ko00195 | Photosynthesis | 1.52E-10 | 19 |
| B vs N Up | ko03010 | Ribosome | 1.38E-15 | 32 |
| B vs N Up | ko00190 | Oxidative phosphorylation | 2.36E-05 | 26 |
| B vs N Up | ko00906 | Carotenoid biosynthesis | 3.25E-10 | 6 |
| B vs N Up | ko01040 | Biosynthesis of unsaturated fatty acids | 2.34E-16 | 3 |
| B vs N Down | ko03030 | DNA replication | 5.64E-23 | 16 |
| B vs N Down | ko00910 | Nitrogen metabolism | 1.87E-15 | 4 |
| B vs N Down | ko01200 | Carbon metabolism | 2.98E-02 | 13 |
| B vs N Down | ko00710 | Carbon fixation in photosynthetic organisms | 3.54E-09 | 7 |
| B vs N Down | ko00240 | Pyrimidine metabolism | 5.64E-36 | 14 |
| W vs N Up | ko00195 | Photosynthesis | 2.39E-24 | 15 |
| W vs N Up | ko00196 | Photosynthesis-antenna proteins | 4.65E-42 | 7 |
| W vs N Up | ko00061 | Fatty acid biosynthesis | 2.98E-19 | 5 |
| W vs N Up | ko00906 | Carotenoid biosynthesis | 5.67E-12 | 5 |
| W vs N Up | ko00190 | Oxidative phosphorylation | 2.54E-11 | 22 |
| W vs N Down | ko03030 | DNA replication | 6.68E-05 | 13 |
| W vs N Down | ko00240 | Pyrimidine metabolism | 8.25E-09 | 11 |
| W vs N Down | ko01200 | Carbon metabolism | 3.25E-54 | 12 |
| W vs N Down | ko00910 | Nitrogen metabolism | 2.48E-08 | 4 |
| W vs N Down | ko04744 | Phototransduction | 1.24E-04 | 2 |
表选项
2.5 雨生红球藻光受体基因挖掘与差异表达分析植物依靠光受体感知光信号,并通过多种互作蛋白形成复杂的接收和转导系统,与体内其他信号转导途径耦合,进而调控基因的表达,最终调控光响应生理过程。我们推测雨生红球藻中存在类似的信号感知与转导通路,所以对其体内的光受体及互作蛋白进行了挖掘与差异表分析。
我们在雨生红球藻转录组数据中共发现了9个光受体同源基因(图 3和表 5),包括1个蓝光受体向光素PHOT (Phototropin)、7个隐花色素与光解酶CPFs (Cryptochrome/photolyase family) 和1个UVR8 (UV resistance locus 8) 成员。其中PHOT和UVR8以单拷贝形式存在,而CPFs以多拷贝形式存在。蓝光受体隐花色素CRY属于隐花色素/光解酶CPF家族,在氨基酸序列水平上与光解酶PHO (Photolyase) 存在极高相似性。通过进一步序列比对分析,我们在7个CPF家族成员中鉴定了2个植物类型CRY和1个DASH类型CRY,其他类型CPF家族成员由于不是编码区全长,因此无法进一步进行分类鉴定。基因差异表达分析表明,PHOT和CPF-2在高光诱导下呈现显著高表达(分别上调2.6–3.2倍和3.7–5.6倍),而CPF-3和CPF-4在高光诱导下低表达(下调0.5–3.2倍)。有趣的是,UVR8和CPF-1仅在高蓝光下高表达,分别达到2.3和3.8倍,而CPF-5和CPF-6仅在高白光下高表达,分别达到3.4和5.1倍,上述结果暗示这些光受体可能以协同作用共同调控雨生红球藻对高光诱导的响应(表 5)。
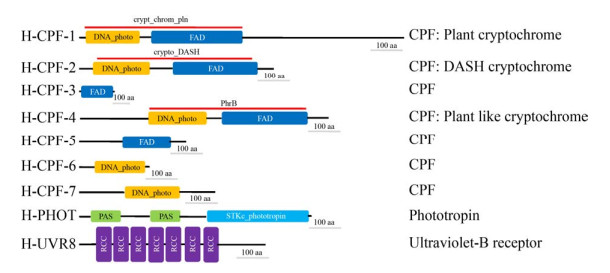 |
| 图 3 雨生红球藻转录组中预测的光受体基因及结构域 Fig. 3 The predicted photoreceptor gene and structure domain in Haematococcus transcriptome. |
| 图选项 |
表 5 雨生红球藻转录组中预测的光受体及互作蛋白信息Table 5 Information for the prediction photoreceptor and interacting protein in Haematococcus transcriptome
| Gene | Transcriptome data | B vs N (log2.Fold) | W vs N (log2.Fold) | Gene | Transcriptome data | B vs N (log2.Fold) | W vs N (log2.Fold) |
| PHOT | comp61615_c0 | 3.2 | 2.6 | CPF-1 | comp59741_c0 | 3.8 | 1.2 |
| CPF-2 | comp59629_c0 | 5.6 | 3.7 | CPF-3 | comp58616_c0 | –3.2 | –2.4 |
| CPF-4 | comp59582_c0 | –0.5 | –1.2 | CPF-5 | comp57508_c1 | –2.5 | 3.4 |
| CPF-6 | comp51104_c0 | –2.3 | 5.1 | CPF-7 | comp59815_c2 | –1.4 | –2.1 |
| UVR8 | comp58392_c7 | 2.3 | 1.5 | COP1 | comp55814_c0 | 2.4 | 1.9 |
| COP8 | comp57867_c1 | 0.5 | 2.4 | COP9 | comp59015_c0 | 0.7 | 2.1 |
| COP13 | comp54442_c0 | 2.8 | 1.9 | SPA1 | comp58791_c1 | –2.1 | –3.5 |
| HY5 | comp59348_c0 | 2.1 | 3.5 | WRKY-1 | comp60211_c0 | –2.7 | –3.4 |
| WRKY-2 | comp56997_c0 | 5.2 | 4.6 | MYB-1 | comp58792_c2 | –2.5 | –1.9 |
| MYB-2 | comp59976_c1 | 2.9 | 3.2 | MYB-3 | comp57039_c0 | 6.5 | 4.8 |
| MYB-4 | comp58936_c1 | –1.3 | –0.9 | MYB-5 | comp59790_c0 | 3.2 | –1.5 |
| BBX21 | comp56830_c0 | 3.5 | 1.8 | CO | comp56830_c0 | 3.5 | 1.8 |
| CSN5A | comp55852_c0 | 3.5 | 1.2 | CUL4-1 | comp57138_c0 | –2.2 | –3.5 |
| CUL4-2 | comp60689_c0 | –2.6 | –3.1 | CUL4-3 | comp60068_c0 | –2.2 | –1.5 |
| DDB1A | comp61166_c1 | –0.3 | –1.2 | DDB1B | comp61620_c0 | –2.3 | –1.5 |
| RUS1-1 | comp57987_c0 | –0.7 | –1.2 | RUS1-2 | comp58436_c6 | –1.5 | –2.3 |
| RUS1-3 | comp61012_c0 | –0.2 | –1.2 | ELI1 | comp47965_c0 | 0.8 | 3.2 |
| LHCSR1 | comp54240_c0 | 4.5 | 3.9 | PIN3 | comp55142_c0 | –2.1 | 1.9 |
| PSBS2-1 | comp62091_c0 | 4.8 | 5.6 | PSBS2-2 | comp53715_c0 | 2.5 | 3.2 |
| PSBS2-3 | comp53715_c1 | 5.2 | 4.6 | PSBS2-4 | comp61375_c0 | 1.4 | 2.5 |
| DET1 | comp55757_c11 | –1.5 | –2.3 | RPT2-1 | comp56679_c0 | 2.7 | –1.2 |
| RPT2-2 | comp58847_c0 | 0.4 | –1.2 | RPT2-3 | comp56653_c0 | 1.8 | 1.3 |
| RPT2-4 | comp56850_c0 | 2.5 | 3.4 | RPT2-5 | comp56882_c0 | 1.9 | 1.6 |
| RPT2-6 | comp60352_c0 | 1.8 | –2.5 | RPT2-7 | comp60015_c2 | –0.5 | –2.1 |
| RPT2-8 | comp59654_c2 | 1.6 | 1.2 | RPT2-9 | comp50090_c0 | –1.2 | –0.9 |
| RPT2-10 | comp60912_c0 | 2.2 | 2.6 | RPT2-11 | comp56654_c1 | –1.9 | –0.5 |
| RPT2-12 | comp52939_c0 | –2.4 | –1.2 | RPT2-13 | comp56654_c2 | –0.6 | –0.9 |
| RPT2-14 | comp462307_c0 | 0.6 | 2.5 | RPT2-15 | comp58999_c5 | –1.3 | –2.4 |
| ABCB19-1 | comp61151_c0 | 1.3 | –2.1 | ABCB19-2 | comp59851_c4 | 1.2 | 0.2 |
| ABCB19-3 | comp57437_c1 | 2.2 | 1.9 | ABCB19-4 | comp60106_c0 | –3.2 | –2.5 |
| ABCB19-5 | comp58093_c0 | –0.1 | –1.2 | ABCB19-6 | comp58498_c4 | –0.8 | –1.5 |
| ABCB19-7 | comp61335_c0 | 1.8 | –2.3 | ABCB19-8 | comp61560_c1 | –0.4 | –1.2 |
| ABCB19-9 | comp62001_c1 | 2.7 | 3.2 | ABCB19-10 | comp61093_c1 | –7.2 | –3.4 |
表选项
通过本地BLAST程序,我们在雨生红球藻转录组数据中发现了大量的已经报道的蓝光受体(PHOT和CRY) 和紫外光受体(UVR8) 信号转导通路的互作蛋白,包括非常保守的信号转导通路核心元件(COP1、SPA1、RPT2、DET1和HY5)、转录因子(MYB和WRKY) 及转录调节子(BBX21、CO、FT、CSN5、CUL4和DDB1) (表 5)。高等植物中已经进行了功能验证的经典的PHOT/CRY/UVR8→COP1/SPA1→HY5光信号转导通路核心元件在雨生红球藻中都存在,暗示这条通路可能也在雨生红球藻中发挥重要的作用,但是具体的作用机制与参与的功能需要进一步验证。基因差异表达分析表明,这些信号转导通路的互作蛋白绝大多数在高光诱导下都呈现明显差异表达,其中COP1/8/9/13和HY5家族成员呈现高表达(上调0.5–2.8倍)、而SPA1家族呈现低表达(下调2.1–3.5倍)、2个MYBs (MYB-2和MYB-3) 和1个WRKYs (WRKY-2) 转录因子成员在高光诱导下转录表达水平显著增强,分别达到2.9–6.5及4.6–5.2倍。在高光诱导下,光受体的互作蛋白LHCSR1、PSBS、FT及ELI1家族成员均呈现一定程度高表达,而CUL4、DDB1和RUS1家族成员普遍低表达。这些结果暗示光受体介导的信号转导通路可能参与雨生红球藻对高光的响应过程,但如何具体参与调控虾青素的积累过程还需要进一步验证。
2.6 荧光定量qRT-PCR验证差异表达基因的表达量为了进一步验证转录组测序及基因差异表达分析结果质量,随机选择高光诱导下高表达DEGs基因6个(PSY、BCH、BKT、DGAT1-1、DGAT2-2、DGAT2-5)、低表达DEGs基因3个(PEPS、3-KCT、PKS)、光受体基因2个(PHOT、CPF-2) 及其信号转导通路互作蛋白基因4个(WRKY-2、MYB-2、COP1、SPA1),共15个基因进行雨生红球藻72 h高光诱导下的qRT-PCR验证。实验结果显示(图 4),高光诱导下转录组中显著高表达的基因表达量提高,而显著低表达的基因表达量显著下降(图 4),上述这些基因的表达趋势与转录组数据的基因差异表达基本一致,说明转录组数据分析合理且可靠。
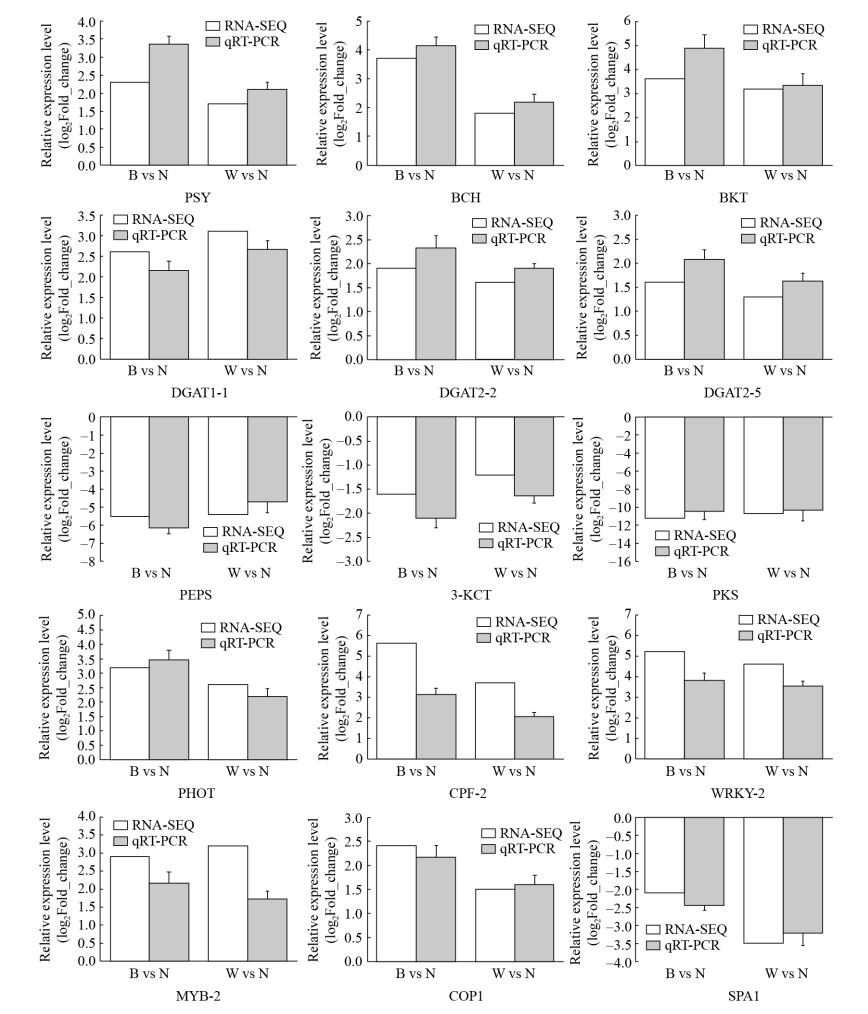 |
| 图 4 差异表达基因qRT-PCR验证 Fig. 4 The qRT-PCR verification of differential expression of genes. |
| 图选项 |
3 讨论高光能有效诱导雨生红球藻积累虾青素,并且大多数合成途径关键酶基因在转录水平高表达,暗示调控机制主要发生在转录水平[6-9],我们前期研究表明高光诱导下雨生红球藻能大量合成与积累虾青素,且虾青素合成相关重要功能基因在转录水平显著高表达[8-9]。这些结果强烈暗示功能基因转录水平高表达与虾青素的合成与积累密切相关,但是光诱导雨生红球藻虾青素积累的转录调控机制还不清楚。转录组测序技术已经成为一种研究生物信号感知与代谢调控机制的强有力工具。近年来,一些****对不同诱导条件下雨生红球藻进行了转录组测序[20-28],集中关注了其生长、生理及虾青素积累的影响与其合成途径相关基因的表达模式,但是对转录调控机制的研究较少,尤其是未见光受体及其信号通路的相关报道。所以本研究选择高光诱导下的转录组数据为基础,重点对雨生红球藻中光受体及信号转导通路中的互作蛋白进行挖掘及基因差异表达分析,以期为从光受体角度揭示高光诱导雨生红球藻虾青素积累的转录调控机制的新视角提供数据支持与功能基因。
本研究中高光诱导下雨生红球藻转录组分别获得对照、高蓝光及高白光处理组4.0 G、3.8 G及3.6 G的有效数据,获得131 385个transcripts和51 954个unigenes (表 2)。共有20 537个unigenes在NR、NT、KO、SwissProt、Pfam、GO及KOG等数据库中的至少1个数据库中注释成功,注释率达到39.52%。其中在GO、Pfam及NR数据库的注释率较高,分别达到32.28%、30.14%及28.32%,共有1 424条unigene在所有数据库同时注释,注释率达到2.74% (表 3)。基因差异表达分析显示,差异表达基因数量达到2 749个DEGs (图 1A–B)。之前报道在红光和蓝光诱导下雨生红球藻转录组获得165 372个transcripts和67 703个unigenes,经过多个数据库比对注释,共有19 591个、15 843个及21 512个unigenes分别成功注释到Phytozome、UniProt及NR数据库。在蓝光诱导下共鉴定出2 245个DEGs,其中上调1 786个,下调459个;红光诱导下共鉴定出171个DEGs,其中上调58个,下调113个[21]。类似的在雨生红球藻中高光和乙酸及Fe2+共同诱导下的转录组测序发现,在高光、高光-醋酸钠双重及高光Fe2+双重诱导下,分别获得7.4 G、8.4 G及8.8 G的数据量,包含156 992个transcripts,平均长度达到1 047 bp。通过比对注释,共有76 774个基因成功注释,注释率达到48.88%,其中共有5 918个基因在7个数据库中同时成功注释,注释率达到3.76%[26]。基因差异表达分析,高光诱导下共发现17 089个DEGs,其中上调8 243个基因,下调8 846个基因[26]。雨生红球藻非游动细胞(Non-motile cell)的转录组数据发现,获得241 869个transcripts和28 407个unigenes,通过比对注释,共有22 990个基因被成功注释,注释率达到了80.93%,进一步分析发现了2 202个DEGs[25]。差异表达基因进一步GO和KEGG富集通路分析发现,本研究高光诱导下,参与光合作用(Photosynthesis)、氧化磷酸化(Oxidative phosphorylation)、核糖体(Ribosome)、类胡萝卜素合成(Carotenoid biosynthesis) 及脂肪酸/不饱和脂肪酸合成(Biosynthesis of unsaturated/saturated fatty acids)等途径大量基因呈现显著高表达;同时参与DNA复制(DNA replication)、碳代谢(Carbon metabolism)及氮代谢(Nitrogen metabolism) 等途径大量基因呈现显著低表达。这与之前的不同光强与光质诱导下雨生红球藻转录组分析结果类似[21, 26],暗示高光诱导下雨生红球藻中类胡萝卜素和脂肪酸的积累与这些基因的高表达密不可分。从转录组数据获得量、注释率及差异基因数量来看,本研究与上述类似研究存在一定差距,主要体现在第一原始有效数据获得量偏小,这个主要原因是本研究过程中将3个生物学重复样品混合为一进行后续测序及分析,这种方式在后续类似的研究中应该避免;第二注释成功率较低,可能的原因是本研究中的转录组数据分析时间是2018年,时间比较早,本身数据库中的雨生红球藻的基因覆盖度较低;第三获得的高光诱导条件下DEGs数量较少,可能与诱导时间有关,本研究中仅仅是选取了高光诱导72 h的时间点取样进行测序,后续的实验过程应该关注不同时间点的影响。除此之外,雨生红球藻的基因组测序工作也有一些进展。例如陈军等发布了雨生红球藻低覆盖度基因组草图[34]和胡章立等发布了雨生红球藻的精细基因组图谱[20],这为转录组数据结合基因组数据分析提供了可能。
对原核蓝藻、真核微藻及高等植物等光合作用的生物而言,光不仅提供能量,而且作为一种重要的环境信号因子,其蕴含的光质、光强及光周期等信息均可调节生物生长、发育及众多生理过程[11]。在漫长的进化过程中光合作用生物获得了一套精细的光信号感知与转导系统。通常来讲,植物依靠光受体感知光信号,并通过复杂的接收和转导系统,进而与体内其他信号转导途径耦联,最终调控光响应生理过程,包括光形态发生、叶绿体运动、下胚轴生长、开花时间、生物钟、生物节律、趋光性及次级代谢产物的合成等[10]。其中光受体是整个信号通路的开关分子,转导通路核心元件是整个信号通路的中枢系统,互作蛋白中转录因子与转录调节子对功能基因的表达起着重要作用[11]。近年来,光信号转导分子机制已成为植物光生物学研究领域的热点。因此,对雨生红球藻中光受体与互作蛋白及信号转导通路的研究具有重要的科学意义。
按照光信号波段的不同,目前发现的光受体主要包括红光受体光敏色素PHY (Phytochrome)、蓝光受体向光素PHOT (Phototrophin)、隐花色素CRY (Cryptochrome) 及AUREO (Aureochrome)与紫外光受体UVR8 (UV resistance locus 8)[10-12]。对真核微藻而言,在已知的光受体中,CRYs以多个等位基因形式广泛分布于几乎所有真核微藻[16],而PHOT和UVR8则是单个等位基因形式仅存在于真核绿藻[17-18],AUREO是异鞭藻门中进行光合作用的藻类特有的蓝光受体,属于LOV光受体蛋白家族[35]。我们在雨生红球藻转录组数据中发现了9个光受体同源基因(图 3和表 5),包括1个蓝光受体向光素PHOT (Phototropin)、7个隐花色素与光解酶CPFs (Cryptochrome/photolyase family) 和1个UVR8 (UV resistance locus 8) 成员。其中PHOT和UVR8以单拷贝形式存在,而CRYs以多拷贝或双拷贝形式存在,这与之前的研究结果一致[16-18]。先前文献表明,在蓝光诱导下的雨生红球藻转录组中,发现了4个可能的隐花色素基因[21],但是我们发现了7个可能的CPFs家族成员。目前真核微藻中光受体的研究集中在基因克隆、表达及功能鉴定方面[10]。例如PHOTs同源基因已经在雨生红球藻H. pluvialis[36]、莱茵衣藻C. reinhardtii[37]、团藻Volvox carteri[38]及金牛鸵球藻Ostreococcus tauri[39]中克隆和功能鉴定。进一步突变和RNA干扰实验验证,CrePHOT参与细胞有性生活周期,并能调控靶基因转录水平的表达,涉及捕光复合物、叶绿素和类胡萝卜素的合成、眼点大小及细胞周期[40]。除此之外,在莱茵衣藻C. reinhardtii[41]、红藻Cyanidioschyzon merolae[42]及三角褐指藻P. tricornutum[43-44]中鉴定了多个不同类型的CRYs。本课题组近期在雨生红球藻中克隆了编码植物类型CRY基因并进行了重组表达及光化学特性鉴定[45]。同时对雨生红球藻编码UVR8的基因进行了克隆及序列分析[46]。基因差异表达分析表明(表 5),PHOT和CPF-2在高光诱导下呈现显著高表达,而CPF-3和CPF-4在高光诱导下低表达。有趣的是,UVR8和CPF-1仅在高蓝光下高表达,而CPF-5和CPF-6仅在高白光下高表达。先前文献表明,HpCPH1基因在蓝光和红光诱导下转录水平表达没有显著差异,但是HpCRY-DASH1基因在上述诱导条件下显著降低[21],造成结果不一致的原因可能是诱导时间的不同或者CPFs家族成员的鉴定不一致。
光受体及其互作蛋白组成的信号转导通路在植物光适应过程中发挥重要的作用[10-15]。我们在雨生红球藻转录组数据中发现了大量的已经报道的各类光受体互作蛋白(图 3和表 5),包括非常保守的信号转导通路核心元件(COP1、SPA1、RPT2、DET1和HY5)、转录因子(MYB和WRKY)及转录调节子(BBX21、CO、FT、CSN5、CUL4和DDB1)。例如,最新研究表明,高等植物中UVR8可分别与WRKY36和MYB30/73/77转录因子互作,进而调控其他相关基因的表达[47-49]。BBXs (B-box proteins) 家族成员是光信号通路中重要的转录调节子,可以与其他转录因子组成复合体(BBX-CO-FT或BBX28/21-COP1-HY5) 共同调控基因的表达[50-52]。CSN1-8家族成员(COP9 signalosome) 也在高等植物光响应过程中发挥重要的作用[53]。CUL4-DDB1-COP1-SPA组成的复合体可以调控转录因子活性进而调控高等植物对UVB的响应过程[54]。RUS家族成员在拟南芥响应低水平UVB诱导过程中发挥重要的作用[55]。RPT2是向光素PHOT感知和转导蓝光信号通路中重要的成员之一[56],我们在雨生红球藻转录组数据中发现了大量的RPT2家族成员(15个),其表达量在不同成员与不同光照条件下呈现多样化。有趣的是,大量的光受体互作蛋白ABCB转运家族成员(10个) 被鉴定,并且部分成员在高光下表达量增强,暗示可能参与虾青素合成过程中的胡萝卜素或虾青素由叶绿体到细胞质的转运过程。
综上所述,本研究挖掘了大量的光受体及其信号转导通路的互作蛋白,高等植物中已经验证的多条经典的光受体介导的信号转导通路的同源基因都在雨生红球藻转录组中存在,暗示类似的光信号感知与转导通路在其体内同样发挥重要的作用。但是上述通路在高等植物调控的多种生理过程在真核微藻雨生红球藻中并不存在,例如光形态发生、叶绿体运动、下胚轴生长及开花时间等。尽管高等植物和绿藻很多光受体起源于共同祖先,在结构方面存在保守性,但作用机制和功能存在物种特异性。因此雨生红球藻中这条通路的行使的具体功能还需要进一步挖掘与验证。更有趣的是,光受体及其信号转导通路的互作蛋白基因表达在雨生红球藻中高光诱导下呈现显著差异。因此,基于高等植物光受体与信号转导通路的作用机制及功能研究进展,结合本研究结果,综上所述,我们推测如下信号转导通路(图 5),“光信号→光受体→互作蛋白(转录因子或者转录调节子→转录因子)→目标靶基因表达→生理过程”的信号转导通路可能参与雨生红球藻对高光的响应过程,进而调控虾青素的积累,但具体的作用机制还需要进一步验证。
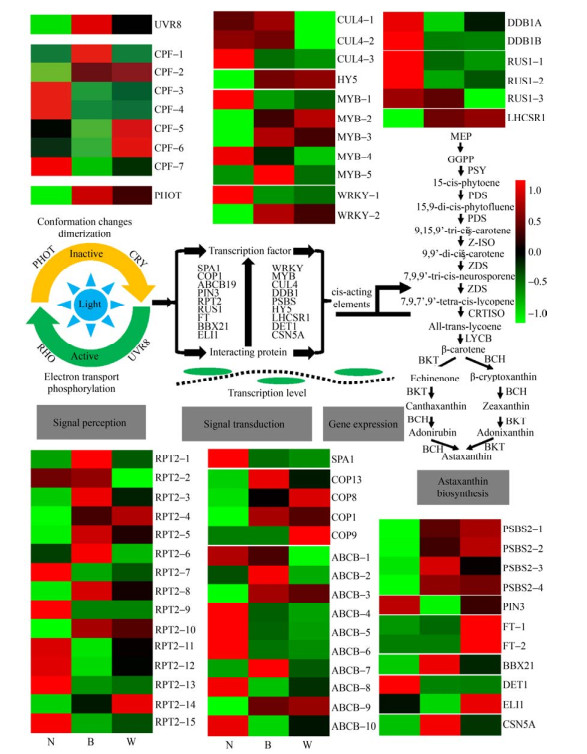 |
| 图 5 雨生红球藻光受体及互作蛋白关键酶基因差异表达(图中–1和1的范围是基于基因的FPKM数值构建heatmap时人为设定的颜色取值范围) Fig. 5 Differential expression of genes encoding photoreceptors and interacting proteins in H. pluvialis. |
| 图选项 |
4 结论基于雨生红球藻高光诱导下的转录组数据,本研究发现高光诱导下参与光合作用、类胡萝卜素合成、脂肪酸合成、氧化磷酸化、DNA复制、碳代谢及氮代谢等过程的功能基因的表达呈现显著差异。通过进一步分析,在雨生红球藻中鉴定了大量光受体及其信号转导通路中的互作蛋白,为下一步这些基因的功能研究提供了候选基因,同时初步推测“光信号→光受体→互作蛋白(转录因子或者转录调节子→转录因子)→目标靶基因表达→生理过程”的信号转导通路可能参与雨生红球藻对高光的响应过程,进而调控虾青素的积累,为从光受体角度解析高光诱导雨生红球藻虾青素积累的转录调控机制奠定了基础,但具体的作用机制还需要进一步验证。
参考文献
| [1] | Shah MMR, Liang YM, Cheng JJ, et al. Astaxanthin-producing green microalga Haematococcus pluvialis: from single cell to high value commercial products. Front Plant Sci, 2016, 7: 531. |
| [2] | Zhang Z, Wang BB, Hu Q, et al. A new paradigm for producing astaxanthin from the unicellular green alga Haematococcus pluvialis. Biotechnol Bioeng, 2016, 113(10): 2088-2099. DOI:10.1002/bit.25976 |
| [3] | Lemoine Y, Schoefs B. Secondary ketocarotenoid astaxanthin biosynthesis in algae: a multifunctional response to stress. Photosynth Res, 2010, 106(1/2): 155-177. DOI:10.1007/s11120-010-9583-3 |
| [4] | 姜思, 佟少明. 雨生红球藻虾青素合成研究进展. 生物工程学报, 2019, 35(6): 988-997. Jiang S, Tong SM. Advances in astaxanthin biosynthesis in Haematococcus pluvialis. Chin J Biotech, 2019, 35(6): 988-997 (in Chinese). |
| [5] | Solovchenko AE. Recent breakthroughs in the biology of astaxanthin accumulation by microalgal cell. Photosynth Res, 2015, 125(3): 437-449. DOI:10.1007/s11120-015-0156-3 |
| [6] | Xi TQ, Kim DG, Roh SW, et al. Enhancement of astaxanthin production using Haematococcus pluvialis with novel led wavelength shift strategy. Appl Microbiol Biotechnol, 2016, 100(14): 6231-6238. DOI:10.1007/s00253-016-7301-6 |
| [7] | Hu CY, Cui DD, Sun X, et al. Transcriptomic analysis unveils survival strategies of autotrophic Haematococcus pluvialis against high light stress. Aquaculture, 2019, 513: 734430. DOI:10.1016/j.aquaculture.2019.734430 |
| [8] | Cui HL, Yu XN, Wang Y, et al. Evolutionary origins, molecular cloning and expression of carotenoid hydroxylases in eukaryotic photosynthetic algae. BMC Genomics, 2013, 14(1): 457. DOI:10.1186/1471-2164-14-457 |
| [9] | Cui HL, Yu XN, Wang Y, et al. Gene cloning and expression profile of a novel carotenoid hydroxylase (CYP97C) from the green alga Haematococcus pluvialis. J Appl Phycol, 2014, 26(1): 91-103. DOI:10.1007/s10811-013-0055-y |
| [10] | Lepetit B, Dietzel L. Light signaling in photosynthetic eukaryotes with 'green' and 'red' chloroplasts. Environ Exp Bot, 2015, 114: 30-47. DOI:10.1016/j.envexpbot.2014.07.007 |
| [11] | 崔红利, 陈军, 侯义龙, 等. 真核微藻蓝光受体及其功能研究进展. 生物技术通报, 2017, 33(4): 51-62. Cui HL, Chen J, Hou YL, et al. Research progress on blue light receptors and their functions in eukaryotic microalgae. Biotech Bull, 2017, 33(4): 51-62 (in Chinese). |
| [12] | Fernaandez MB, Tossi V, Lamattina L, et al. A comprehensive phylogeny reveals functional conservation of the UV-B photoreceptor UVR8 from green algae to higher plants. Front Plant Sci, 2016, 7: 1698. |
| [13] | Han X, Chang X, Zhang ZH, et al. Origin and evolution of core components responsible for monitoring light environment changes during plant terrestrialization. Mol Plant, 2019, 12(6): 847-862. DOI:10.1016/j.molp.2019.04.006 |
| [14] | Okajima K. Molecular mechanism of phototropin light signaling. J Plant Res, 2016, 129(2): 149-157. DOI:10.1007/s10265-016-0783-6 |
| [15] | Wang Q, Lin CT. Mechanisms of cryptochrome- mediated photoresponses in plants. Annu Rev Plant Biol, 2020, 71: 103-129. DOI:10.1146/annurev-arplant-050718-100300 |
| [16] | Oliveri P, Fortunato AE, Petrone L, et al. The cryptochrome/photolyase family in aquatic organisms. Mar Genomics, 2014, 14: 23-37. DOI:10.1016/j.margen.2014.02.001 |
| [17] | Li FW, Rothfels CJ, Melkonian M, et al. The origin and evolution of phototropins. Front Plant Sci, 2015, 6: 637. |
| [18] | Kianianmomeni A, Hallmann A. Algal photoreceptors: in vivo functions and potential applications. Planta, 2014, 239(1): 1-26. DOI:10.1007/s00425-013-1962-5 |
| [19] | Huang K, Merkle T, Beck CF. Isolation and characterization of a Chlamydomonas gene that encodes a putative blue-light photoreceptor of the phototropin family. Physiol Plant, 2012, 115(4): 613-622. |
| [20] | Luo QL, Bian C, Tao M, et al. Genome and transcriptome sequencing of the astaxanthin- producing green microalga, Haematococcus pluvialis. Genome Biol Evol, 2019, 11(1): 166-173. DOI:10.1093/gbe/evy263 |
| [21] | Lee C, Ahn JW, Kim JB, et al. Comparative transcriptome analysis of Haematococcus pluvialis on astaxanthin biosynthesis in response to irradiation with red or blue led wavelength. World J Microbiol Biotechnol, 2018, 34(7): 96. DOI:10.1007/s11274-018-2459-y |
| [22] | Cui DD, Hu CY, Zou ZM, et al. Comparative transcriptome analysis unveils mechanisms underlying the promoting effect of potassium iodide on astaxanthin accumulation in Haematococcus pluvialis under high light stress. Aquaculture, 2020, 525: 735279. DOI:10.1016/j.aquaculture.2020.735279 |
| [23] | Zhao Y, Hou YY, Chai WJ, et al. Transcriptome analysis of Haematococcus pluvialis of multiple defensive systems against nitrogen starvation. Enzyme Microb Technol, 2020, 134: 109487. DOI:10.1016/j.enzmictec.2019.109487 |
| [24] | Li QQ, Zhang LT, Liu JG. Comparative transcriptome analysis at seven time points during Haematococcus pluvialis motile cell growth and astaxanthin accumulation. Aquaculture, 2019, 503: 304-331. DOI:10.1016/j.aquaculture.2018.12.091 |
| [25] | Li QQ, Zhang LT, Liu JG. Examination of carbohydrate and lipid metabolic changes during Haematococcus pluvialis non-motile cell germination using transcriptome analysis. J Appl Phycol, 2019, 31(1): 145-156. DOI:10.1007/s10811-018-1524-0 |
| [26] | He BX, Hou LL, Dong MM, et al. Transcriptome analysis in Haematococcus pluvialis: astaxanthin induction by high light with acetate and Fe2+. Int J Mol Sci, 2018, 19(1): 175. DOI:10.3390/ijms19010175 |
| [27] | Gao ZQ, Li Y, Wu GX, et al. Transcriptome analysis in Haematococcus pluvialis: astaxanthin induction by salicylic acid (SA) and jasmonic acid (JA). PLoS ONE, 2015, 10(10): e0140609. DOI:10.1371/journal.pone.0140609 |
| [28] | Cheng J, Li K, Zhu YX, et al. Transcriptome sequencing and metabolic pathways of astaxanthin accumulated in Haematococcus pluvialis mutant under 15% CO2. Bioresour Technol, 2017, 228: 99-105. DOI:10.1016/j.biortech.2016.12.084 |
| [29] | Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol, 2011, 29(7): 644-652. DOI:10.1038/nbt.1883 |
| [30] | Li B, Dewey CN. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics, 2011, 12: 323. DOI:10.1186/1471-2105-12-323 |
| [31] | Trapnell C, Williams BA, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation. Nat Biotechnol, 2010, 28(5): 511-515. DOI:10.1038/nbt.1621 |
| [32] | Anders S, Huber W. Differential expression analysis for sequence count data. Genome Biol, 2010, 11(21): R106. |
| [33] | Rao XY, Huang XL, Zhou ZC, et al. An improvement of the 2(-delta delta CT) method for quantitative real-time polymerase chain reaction data analysis. Biostat Bioinforma Biomath, 2013, 3(3): 71-85. |
| [34] | 陈军, 郑华军, 刘亚铭, 等. 雨生红球藻低覆盖度基因组草图分析. 中国生物工程杂志, 2018, 38(7): 21-28. Chen J, Zheng HJ, Liu YM, et al. The analysis of the low coverage Haematococcus pluvialis draft genome. China Biotechnol, 2018, 38(7): 21-28 (in Chinese). |
| [35] | Takahashi F, Yamagata D, Ishikawa M, et al. Aureochrome, a photoreceptor required for photomorphogenesis in stramenopiles. Proc Natl Acad Sci USA, 2007, 104(49): 19625-19630. DOI:10.1073/pnas.0707692104 |
| [36] | 崔红利, 陈军, 王林萍, 等. 雨生红球藻PHOT的基因克隆和生物信息学分析. 西南农业学报, 2017, 30(12): 2639-2647. Cui HL, Chen J, Wang LP, et al. Gene cloning and bioinformatics analysis of novel blue photoreceptor phototropin from green alga Haematococcus pluvialis. Southwest China J Agric Sci, 2017, 30(12): 2639-2647 (in Chinese). |
| [37] | Huang KY, Merkle T, Beck CF. Isolation and characterization of a Chlamydomonas gene that encodes a putative blue-light photoreceptor of the phototropin family. Physiol Plant, 2002, 115(4): 613-622. DOI:10.1034/j.1399-3054.2002.1150416.x |
| [38] | Kianianmomeni A, Hallmann A. Transcriptional analysis of Volvox photoreceptors suggests the existence of different cell-type specific light-signaling pathways. Curr Genet, 2015, 61(1): 3-18. DOI:10.1007/s00294-014-0440-3 |
| [39] | Sullivan S, Petersen J, Blackwood L, et al. Functional characterization of Ostreococcus tauri phototropin. New Phytol, 2016, 209(2): 612-623. DOI:10.1111/nph.13640 |
| [40] | Im CS, Eberhard S, Huang KY, et al. Phototropin involvement in the expression of genes encoding chlorophyll and carotenoid biosynthesis enzymes and LHC apoproteins in Chlamydomonas reinhardtii. Plant J, 2006, 48(1): 1-16. DOI:10.1111/j.1365-313X.2006.02852.x |
| [41] | Beel B, Prager K, Spexard M, et al. A flavin binding cryptochrome photoreceptor responds to both blue and red light in Chlamydomonas reinhardtii. Plant Cell, 2012, 24(7): 2992-3008. DOI:10.1105/tpc.112.098947 |
| [42] | Asimgil H, Kavakli IH. Purification and characterization of five members of photolyase/ cryptochrome family from Cyanidioschyzon merolae. Plant Sci, 2012, 185/186: 190-198. DOI:10.1016/j.plantsci.2011.10.005 |
| [43] | Juhas M, Von Zadow A, Spexard M, et al. A novel cryptochrome in the diatom Phaeodactylum tricornutum influences the regulation of light-harvesting protein levels. FEBS J, 2014, 281(9): 2299-2311. DOI:10.1111/febs.12782 |
| [44] | Coesel S, Mangogna M, Ishikawa T, et al. Diatom PtCPF1 is a new cryptochrome/photolyase family member with DNA repair and transcription regulation activity. EMBO Rep, 2009, 10(6): 655-661. DOI:10.1038/embor.2009.59 |
| [45] | Hang W, Gujar A, Zhang HJ, et al. Cloning, expression, and characterization of a novel plant type cryptochrome gene from the green alga Haematococcus pluvialis. Prot Exp Purif, 2020, 172: 105633. DOI:10.1016/j.pep.2020.105633 |
| [46] | 张宏江, 杭伟, 马浩天, 等. 雨生红球藻UVR8的基因克隆和生物信息学分析. 西南农业学报, 2019, 32(9): 2025-2032. Zhang HJ, Hang W, Ma HT, et al. Gene cloning and bioinformatics analysis of novel ultraviolet-B photoreceptor uv resistance locus 8 (UVR8) from Green alga Haematococcus pluvialis. J Southwest Agric, 2019, 32(9): 2025-2032 (in Chinese). |
| [47] | Yang Y, Zhang LB, Chen P, et al. UV-B photoreceptor UVR8 interacts with MYB73/MYB77 to regulate auxin responses and lateral root development. EMBO J, 2020, 39(2): e101928. |
| [48] | Yang Y, Liang T, Zhang LB, et al. UVR8 interacts with WRKY36 to regulate HY5 transcription and hypocotyl elongation in Arabidopsis. Nat Plants, 2018, 4(2): 98-107. DOI:10.1038/s41477-017-0099-0 |
| [49] | Yan Y, Li C, Dong XJ, et al. MYB30 is a key negative regulator of Arabidopsis photomorphogenic development that promotes PIF4 and PIF5 protein accumulation in the light. Plant Cell, 2020, 32(7): 2196-2215. DOI:10.1105/tpc.19.00645 |
| [50] | Song ZQ, Bian YT, Liu JJ, et al. B-box proteins: pivotal players in light-mediated development in plants. J Integr Plant Biol, 2020, 62(9): 1293-1309. DOI:10.1111/jipb.12935 |
| [51] | Lin F, Jiang Y, Li J, et al. B-BOX DOMAIN PROTEIN28 negatively regulates photomorphogenesis by repressing the activity of transcription factor HY5 and undergoes COP1-Mediated degradation. Plant Cell, 2018, 30(9): 2006-2019. DOI:10.1105/tpc.18.00226 |
| [52] | Xu DQ, Jiang Y, Li JG, et al. BBX21, an Arabidopsis B-box protein, directly activates HY5 and is targeted by COP1 for 26S proteasome-mediated degradation. Proc Natl Acad Sci USA, 2016, 113(27): 7655-7660. DOI:10.1073/pnas.1607687113 |
| [53] | Qin NX, Xu DQ, Li JG, et al. COP9 signalosome: Discovery, conservation, activity, and function. J Integr Plant Biol, 2020, 62(1): 90-103. DOI:10.1111/jipb.12903 |
| [54] | Huang X, Ouyang XH, Yang PY, et al. Conversion from CUL4-based COP1-SPA E3 apparatus to UVR8-COP1-SPA complexes underlies a distinct biochemical function of COP1 under UV-B. Proc Natl Acad Sci USA, 2013, 110(41): 16669-16674. DOI:10.1073/pnas.1316622110 |
| [55] | Tong HY, Leasure CD, Hou XW, et al. Role of root UV-B sensing in Arabidopsis early seedling development. Proc Natl Acad Sci USA, 2008, 105(52): 21039-21044. DOI:10.1073/pnas.0809942106 |
| [56] | Inada S, Ohgishi M, Mayama T, et al. RPT2 is a signal transducer involved in phototropic response and stomatal opening by association with phototropin 1 in Arabidopsis thaliana. Plant Cell, 2004, 16(4): 887-896. DOI:10.1105/tpc.019901 |
