

复旦大学 生命科学学院,上海 200438
收稿日期:2020-06-30;接收日期:2020-09-27;网络出版时间:2020-10-13
基金项目:国家重大科技专项“重大新药开发”课题(No. 2018ZX09J18112),国家自然科学基金(Nos. 31671386, 31971377) 资助
摘要:酿脓链球菌(Streptococcus pyogenes Cas9,SpCas9) 已成为强大的基因组编辑工具,但其可识别的前间隔序列临近基序(Protospacer adjacent motifs,PAMs) 范围有限,且存在脱靶效应。为解决这些问题,文中提出一种对SpCas9的定向进化突变体xCas9进行优化的理性方法。首先,使用Rosetta程序进行能量最小化以优化Cas9的三维结构,获得其能量最低的构象;然后,对其定向进化所得氨基酸位点进行组合突变设计;最后,通过自由能排序从设计突变体中筛选出用于实验验证的最优变体。经DNA剪切实验验证,成功地获得了一个多PAM识别和低脱靶的新变体yCas9 (262A/324R/409N/480K/543D/694L/1219T)。该变体可识别NG、GAA和GAT序列,且其由错配sgRNA引导的脱靶DNA剪切活性低,为生物医学领域提供了一个有潜在应用价值的基因编辑工具。同时,文中还对SpCas9、xCas9和yCas9进行了分子动力学模拟,揭示了其PAM识别和脱靶效应的机理,可为进一步的CRISPR/Cas9蛋白优化改造提供理论指导。
关键词:基因编辑CRISPR/Cas9蛋白计算设计分子动力学模拟
Structure-based optimization and design of CRISPR protein xCas9
Dongmei Xue, Haixia Zhu, Wenhao Du, Honghai Tang, Qiang Huang


School of Life Sciences, Fudan University, Shanghai 200438, China
Received: June 30, 2020; Accepted: September 27, 2020; Published: October 13, 2020
Supported by: National Major Scientific and Technological Special Project for 'Significant New Drugs Development' (No. 2018ZX09J18112), National Natural Science Foundation of China (Nos. 31671386, 31971377)
Corresponding author: Qiang Huang. Tel: +86-21-31246589; E-mail: huangqiang@fudan.edu.cn.
Abstract: Streptococcus pyogenes Cas9 (SpCas9) has become a powerful genome editing tool, but has a limited range of recognizable protospacer adjacent motifs (PAMs) and shows off-target effects. To address these issues, we present a rational approach to optimize the xCas9 mutant derived from SpCas9 by directed evolution. Firstly, energy minimization with the Rosetta program was applied to optimize the three-dimensional structure of Cas9 to obtain the lowest energy conformation. Subsequently, combinatorial mutations were designed based on the mutations sites of xCas9 acquired during the directed evolution. Finally, optimal mutants were selected from the designed mutants by free energy ranking and subjected to experimental verification. A new mutant yCas9 (262A/324R/409N/480K/543D/694L/1219T) with multiple PAM recognition ability and low off-target effects was obtained and verified by DNA cleavage experiments. This mutant recognizes the NG, GAA and GAT PAMs and shows low off-target DNA cleavage activity guided by mismatched sgRNA, thus provides a gene editing tool with potential applications in biomedical field. Furthermore, we performed molecular dynamics simulations on the structures of SpCas9, xCas9 and yCas9 to reveal the mechanisms of their PAM recognition and off-target effects. These may provide theoretical guidance for further optimization and modification of CRISPR/Cas9 proteins.
Keywords: gene editingCRISPR/Cas9computational protein designmolecular dynamics simulation
CRISPR是细菌和古菌中抵抗外源核酸入侵的免疫系统。近年来,该系统已被人们开发成一种特异性剪切DNA的基因编辑工具[1]。目前最常用的是Ⅱ型CRISPR系统中的酿脓链球菌Cas9 (Streptococcus pyogenes Cas9,SpCas9)[2]。不过,SpCas9存在前间隔序列临近基序(Protospacer adjacent motifs,PAMs) 识别范围有限和易脱靶的问题,这限制了其广泛应用[3-5]。因此,只有拓展PAM识别范围和降低脱靶效应,才能使SpCas9技术在基因编辑领域发挥更大的作用。
为了达到上述目标,人们开发了多种SpCas9的优化方法。例如,利用细菌选择系统来筛选设计SpCas9,通过该方法获得了PAM识别范围扩大的VQR、EQR和VRER突变体[6];还有,基于SpCas9结构的改造方法,也获得了高保真突变体eSpCas9、SpCas9-HF、HypaCas9和evoCas9[7-8]。此外,David Liu研究组通过PACE定向进化法获得了一个新型SpCas9突变体(xCas9),与野生型相比,该突变体具有更高的特异性,且可以识别包括NGG、NGA、NGT、NGC、GAA和GAT在内的广泛的PAM序列[9]。但是,xCas9在提高特异性和PAM兼容性的同时,其DNA剪切活性有较大损失,对不同PAM的识别能力也存在差异[10-11]。因此,该蛋白还有进一步优化的空间。
然而,上述几种优化SpCas9的方法有一定的盲目性,耗时耗力。在本研究中,我们提出了一种新的优化SpCas9的方法。该方法从能量的角度出发,用Rosetta程序对定向进化突变体xCas9进行优化。Rosetta是一个精确的生物大分子设计程序,可通过突变DNA周围的关键氨基酸来优化蛋白-DNA的相互作用界面,已在蛋白-DNA体系中得到了成功应用[12]。鉴于Cas9剪切DNA属于蛋白-DNA相互作用的一种,我们设想,利用Rosetta来突变对Cas9蛋白特性有重要影响的氨基酸,以优化Cas9-DNA的相互作用界面,获得自由能较低的突变体,这些突变体极有可能具有高PAM兼容性或低脱靶率的属性。
本研究以xCas9为对象,选取其分布在sgRNA-DNA异源双链体两侧的7个定向进化位点[9]作为优化设计的目标位点(图 1),并对这些位点进行20种氨基酸的组合设计,最终获得了一个突变体yCas9。该突变体具有高PAM兼容性,可识别NGG、NGA、NGT、NGC、GAA和GAT序列,且对GAT PAM的识别能力比xCas9高。同时,与野生型SpCas9相比,yCas9的脱靶效应也大幅度降低。同时,我们还通过分子动力学模拟分析了SpCas9的PAM识别和脱靶效应的分子机理,确定了1 219位氨基酸与Cas9的PAM识别之间的关系,以及REC域构象重排与Cas9脱靶效应之间的关系。
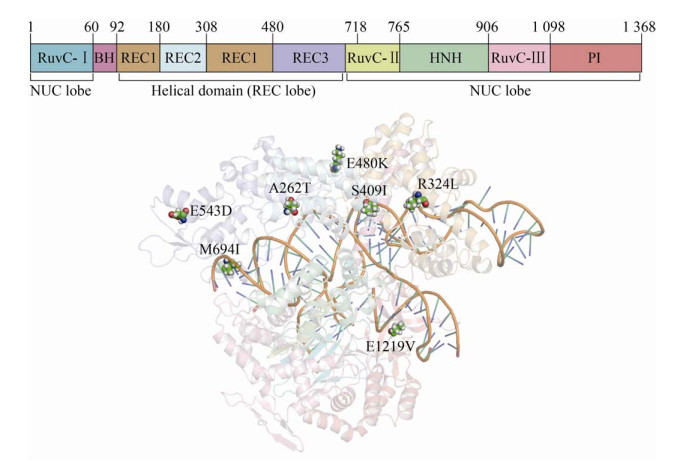 |
| 图 1 xCas9的7个定向进化位点[9]的三维分布图 Fig. 1 3D-distribution of seven directed-evolution sites of xCas9[9]. |
| 图选项 |
1 材料与方法1.1 实验材料本研究使用的pET21-6His-TEV-SpCas9、pET21-6His-TEV-xCas9、psk-sgRNA和psk-DNA质粒由本课题组保存。转化用的大肠杆菌Escherichia coli DH5α (CB101-02) 和Rosetta (DE3) (CB108-02)、质粒提取试剂盒(DP103-02)、点突变试剂盒(KM101) 等购自TIANGEN生化科技(北京) 有限公司;胶回收试剂盒(AP-GX-50) 购自Axygen公司;一步定向克隆试剂盒(NR005-01A) 购自近岸蛋白质科技公司;PrimeSTAR GXL DNA Polymerase (R050Q) 购自TaKaRa公司;RNA转录试剂盒(AM1354) 和RNA纯化试剂盒(AM1908) 购自Thermo Fisher公司。本研究所用引物均采用软件Primer 5设计,满足各试剂盒的引物设计原则,并委托上海杰李生物技术有限公司合成(表 1)。
表 1 本研究所用引物Table 1 Primers used in this study
| Primer name | Sequence (5′–3′) | Usage |
| NKDL-LF | CTGCAGTTGATCCATGATGACTCTCTCAC | Mutations of 409, 480, 543 and 694 |
| NKDL-LR | ATTTCCATTGTCGAAAGTGCGCTGTTTGC | |
| NKDL-SF | GCAAACAGCGCACTTTCGACAATGGAAATATCCCCCACCAGATCACCTGGGC | |
| NKDL-SR | GTGAGAGAGTCATCATGGATCAACTGCAGGAAGTTCCGGTTGGCAAATCCATCG | |
| E1219T-F | CTCGCTAGTGCGGGCACCCTGCAGAAAGGTAACG | Mutation of 1 219 |
| E1219T-R | CGTTACCTTTCTGCAGGGTGCCCGCACTAGCGAG | |
| DNA-F | TAGTCCTGTCGGGTTTCG | Amplification of target DNA (920 bp) |
| DNA-R | TTCCATTCGCCATTCAGG | |
| sgRNA-F | GGAATTCGATATCAAGCTTA | Amplification of sgRNA (97 nt) |
| sgRNA-R | AGCACCGACTCGGTGCCACTT |
表选项
1.2 xCas9的组合设计生物分子结构预测和设计的Rosetta程序包含100多个模块,具有蛋白质结构从头预测、蛋白质结构设计或对接以及蛋白质重设计等功能[13-14]。为了实现这些功能,Rosetta通过其独特的自由能打分函数计算蛋白质能量,包括不同残基间的引力和斥力、蛋白骨架与侧链以及侧链与侧链之间的氢键相互作用、范德华作用等。当使用该打分函数比较同一分子的不同构象时,得分低的构象更稳定,自由能更低,更接近于天然结构。
本文使用了Rosetta程序的两个模块:Relax和Fixed backbone design (Fixbb)。Relax模块通过多次迭代使初始结构的氨基酸侧链重排,并且在每轮迭代中不断地调整分子间作用力,以搜索该结构的最优构象。最终经过Relax模块处理后的结构,一般都处于能量最低的构象。Fixbb模块在固定蛋白骨架的同时,允许氨基酸的侧链类型自由设计,以设计与原蛋白骨架兼容的氨基酸侧链,最后通过自由能排序获得最优突变体。
本文设计Cas9突变体的主要流程是:首先从蛋白质数据库(http://www.rcsb.org/) 下载SpCas9结构(PDB ID:4UN3)。其次,用Relax模块将该结构进行25轮迭代,并根据Rosetta的自由能打分函数选出能量最低的结构作为初始结构。然后,使用Fixbb模块固定Cas9蛋白的主链原子,并对xCas9的7个定向进化位点(262、324、409、480、543、694、1 219) 进行20种氨基酸组合设计。最后,进行10 000次独立设计,并对输出结果进行去重处理和按自由能大小排列筛选。
1.3 蛋白突变体质粒的构造本文分别以质粒pET21-6His-TEV-SpCas9和pET21-6His-TEV-xCas9为模板,通过一步定向克隆和点突变的方式构建突变体质粒。
1.4 蛋白的表达与纯化将Cas9及其突变体质粒(N端带有His-tag) 转入E. coli Rosetta(DE3) 细胞中表达,当菌液的OD600达到0.9时,加入终浓度为0.5 μmol/L的异丙基-β-D-硫代半乳糖苷(Isopropyl-β-D- thiogalactopyranoside,IPTG),16 ℃过夜培养。离心收集菌体,并用裂解液(20 mmol/L HEPES,500 mmol/L KCl,0.1 mmol/L PMSF,pH 7.5) 重悬菌体,超声破碎20 min,12 000 r/min离心1 h。上清液与Ni-NTA孵育90 min后,用缓冲液B (20 mmol/L HEPES,400 mmol/L KCl,500 mmol/L咪唑,1%蔗糖,pH 7.5) 梯度洗脱Cas9蛋白。经过SDS-PAGE鉴定后,收集目的蛋白并将其浓缩至250 μL,保存于蛋白储存液(20 mmol/L HEPES,500 mmol/L KCl,1 mmol/L DTT,pH 7.5) 中。
1.5 底物DNA的获取以psk-DNA质粒为模板,使用引物DNA-F/R对包含PAM序列和20 bp靶序列的DNA进行PCR扩增,然后切胶回收获得920 bp的底物DNA,并将其保存于无核酸酶的水中。DNA包含不同的PAM序列,分别为5′-TGG-3′、5′-TGA-3′、5′-TGC-3′、5′-TGT-3′、5′-GAA-3′和5′-GAT-3′。
1.6 sgRNA的获取首先以psk-sgRNA质粒为模板,使用引物sgRNA-F/R进行PCR扩增。其次,切胶回收获得sgRNA的转录模板,并使用RNA转录试剂盒在体外将模板转录成sgRNA,37 ℃温育6 h。然后,用TURBO DNase消化转录模板。最后,用RNA纯化试剂盒纯化产物,并将其保存于无核酸酶的水中。sgRNA中与底物DNA的靶序列匹配的间隔序列为5′-UACCGCUCCAGUCGUUCAUG-3′。用于脱靶检测的sgRNA构建过程:从PAM远端开始依次将sgRNA间隔序列的两个碱基突变为其互补碱基,共改8次,最终获得9个用于脱靶检测的sgRNA,其序列如表 2所示。
表 2 sgRNA序列Table 2 Sequences of sgRNA
| No. | Sequence (5′–3′) | Site |
| 0 | UACCGCUCCAGUCGUUCAUG | |
| 1 | AUCCGCUCCAGUCGUUCAUG | 01–02 |
| 2 | UUGCGCUCCAGUCGUUCAUG | 02–03 |
| 3 | UAGGGCUCCAGUCGUUCAUG | 03–04 |
| 4 | UACGCCUCCAGUCGUUCAUG | 04–05 |
| 5 | UACCCGUCCAGUCGUUCAUG | 05–06 |
| 6 | UACCGGACCAGUCGUUCAUG | 06–07 |
| 7 | UACCGCAGCAGUCGUUCAUG | 07–08 |
| 8 | UACCGCUGGAGUCGUUCAUG | 08–09 |
| Note: the underline part of the sequence is mutated bases. | ||
表选项
1.7 体外剪切活性检测本文构建的体外剪切活性检测反应体系为20 μL体系,包含200 nmol/L Cas9、200 nmol/L sgRNA、30 nmol/L DNA和反应缓冲液(20 mmol/L HEPES,150 mmol/L KCl,1 mmol/L DTT,10 mmol/L MgCl2,pH 7.5)。首先在反应缓冲液中加入Cas9和sgRNA,混合均匀后将其放置在37 ℃预孵育10 min,然后将DNA加入混合物中,继续孵育,孵育时间随检测目的而定(检测PAM识别时,时间设为2 h;检测脱靶效应时,时间设为3 h)。反应结束后,使用琼脂糖凝胶电泳检测Cas9的底物剪切情况。
1.8 分子动力学模拟本文使用GROMACS对Cas9蛋白进行分子动力学(Molecular dynamics,MD) 模拟,以获得其在溶液环境中的稳定构象[15]。从蛋白质数据库中下载SpCas9结构(PDB ID:4UN3) 和xCas9结构(PDB IDs:6K4P、6AEG)。晶体结构中缺失的氨基酸残基用在线网站SWISS-MODEL (https://swissmodel.expasy.org/) 同源建模补齐,yCas9的3D结构用Pymol (https://pymol.org/2/) 点突变获得。使用Amber ff14SB_Parmbsc1力场和TIP3P水模型对Cas9系统进行建模[16]。在模拟系统中,Cas9的全原子结构被放在一个离盒壁至少1.5×10–9 m的立方体盒中。在该体系中添加一定数量的Na+和Cl–以中和系统的净电荷,并使离子浓度达到0.15 mol/L。模拟过程中先进行能量最小化,使体系达到预平衡状态;接着,依次进行3次NVT平衡,分别为100 K平衡200 ps、200 K平衡200 ps和300 K平衡200 ps,最终使系统温度保持在300 K;然后,进行2 ns的NPT平衡,使体系达到1×10–5 Pa的恒定压力;最后在300 K、1×10–5 Pa的条件下进行200 ns的MD模拟,每40 ps输出一帧,供后续分析使用。
2 结果与分析2.1 突变体的获得本研究使用Rosetta的Fixbb模块对xCas9的7个定向进化位点进行了组合设计,共输出了10 000个独立的组合设计。根据蛋白质折叠的热力学假说,一个蛋白质构象的自由能越低,则该构象在自然界中出现的概率越大,构象越稳定[17-19]。所以,我们对这10 000个设计进行去重处理和排序后,最终获得两个能量最低的突变体(表 3)。如表 3所示,这两个突变体的Rosetta自由能打分函数的值分别为?1 704.054和?1 703.748。因此,在xCas9定向进化位点的组合设计中,设计序号为00329的突变体分值最低,代表该复合物的自由能最低,则理论上活性也高。因此我们将该突变体命名为yCas9,并进行实验验证。
表 3 Rosetta设计结果Table 3 Results of Rosetta design
| ID | Mutation sites | Score | ||||||||||||
| 262 | 324 | 409 | 480 | 543 | 694 | 1 219 | ||||||||
| SpCas9 | A | R | S | E | E | M | E | |||||||
| xCcas9 | T | L | I | K | D | I | V | |||||||
| 00329 | A | R | N | K | D | L | T | ?1 704.054 | ||||||
| 00425 | A | R | N | K | D | M | T | ?1 703.748 | ||||||
表选项
2.2 Cas9剪切活性检测2.2.1 PAM识别检测xCas9可以识别不同的PAM序列,包括NG、GAA和GAT。为了验证Rosetta设计所得的yCas9是否可以识别这些PAM序列,我们对其进行了体外DNA剪切活性检测(图 2A–C)。由图可见,野生型SpCas9主要识别TGG、TGA和TGC,表明其识别的PAM的类型有限(图 2A,lanes 2–4)。与文献报道相一致[9],xCas9可以识别TGN、GAA和GAT序列(图 2B)。与xCas9相比,yCas9不仅对含有这些PAM序列的DNA具有相当的剪切活性(图 2C),而且对GAT的识别能力比xCas9高(图 2C,lane 7)。由此说明,Rosetta设计的yCas9进一步提升了xCas9的PAM识别能力。
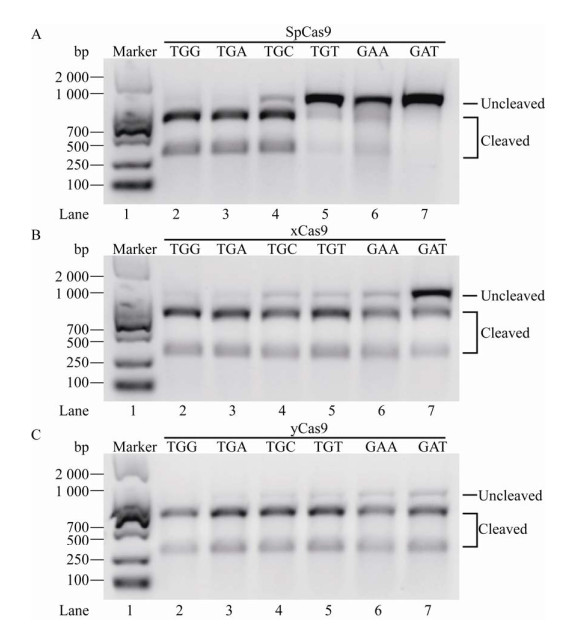 |
| 图 2 SpCas9及其突变体的体外PAM识别检测 Fig. 2 In vitro PAM compatibility cleavage assay of SpCas9 and its mutants. (A–C) Linearized plasmids containing a target sequence adjacent to TGG, TAG, TGC, TGT, GAA and GAT PAMs were incubated with the Cas9-sgRNA complex (200 nmol/L) at 37 ℃ for 2 hours. The reaction products were analyzed using an electrophoresis system. |
| 图选项 |
2.2.2 脱靶效应检测为验证yCas9的特异性,我们通过构建的8个错配sgRNA (表 2,1–8号) 和包含TGG PAM序列的DNA进一步对其进行了体外脱靶检测,结果如图 3所示。由图可见,1–8号sgRNA均可以引导SpCas9剪切底物DNA,其中,1–5号sgRNA引导的剪切活性与0号sgRNA相当(图 3A,lanes 3–8),这表明SpCas9的脱靶效应比较严重。与SpCas9相比,相同反应条件下,错配sgRNA引导xCas9对底物DNA的剪切活性均有不同程度的降低(图 3B,lanes 4–11),说明xCas9的特异性优于SpCas9。由图 3C可见,yCas9的脱靶效应比野生型SpCas9低,但其中3、4、5、7号sgRNA引导的yCas9剪切活性比xCas9高(图 3C,lanes 6、7、8和10)。因此,yCas9的脱靶效应比xCas9稍高,但与野生型SpCas9相比大幅度降低。
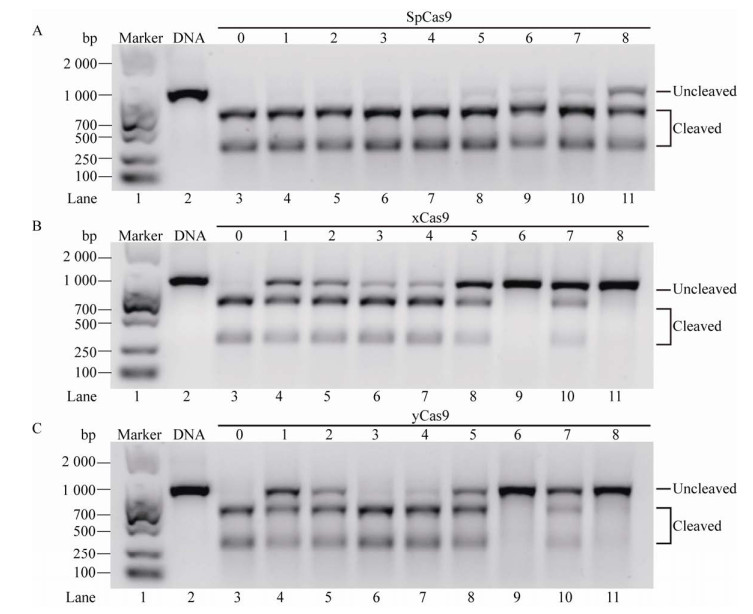 |
| 图 3 SpCas9及其突变体的体外脱靶效应检测 Fig. 3 In vitro off-target activity assay of SpCas9 and its mutants. (A–C) A linearized plasmid target bearing the TGG PAM was incubated with the Cas9-sgRNA complex (200 nmol/L) at 37 ℃ for 3 hours. No. 0 sgRNA was perfectly matched and No. 1–8 were partially mismatched. The reaction products were analyzed using an electrophoresis system. |
| 图选项 |
2.3 Cas9剪切机理分析2.3.1 PAM识别分析全面了解Cas9的PAM识别分子机理,有助于我们更好地认识CRISPR系统。所以,我们将带有不同PAM序列的Cas9结构(TGG:SpCas9,GAT:xCas9和yCas9) 进行了200 ns的分子动力学模拟。模拟结束后,我们首先对最后一帧构象中PAM附近的结构进行单独分析,发现在xCas9和yCas9中,1 219位氨基酸的突变破坏了其与1 335之间的双齿氢键,增强了1 333和1 335位氨基酸的结构柔性,从而削弱了周围氨基酸的相互作用(图 4B和C)。然后,我们对1 219、1 333和1 335位氨基酸的运动轨迹进行了分析。以初始构象为参照,计算其余帧对应构象与初始构象的均方根偏差(Root mean squared deviation, RMSD),作时间依赖性的RMSD图。由于MD模拟的结构在前期没有完全平衡,所以我们选取了185–200 ns的模拟轨迹计算RMSD (图 4D–F)。由图 4D可知,SpCas9的1 333和1 335位氨基酸的RMSD值约为4×10–11 m,低于xCas9和yCas9 (图 4E和F)。这表明1 219位氨基酸突变后增强了1 333位和1 335位氨基酸结构柔性,进而拓展了Cas9蛋白对PAM识别能力。以上结果说明,1 219位氨基酸突变会对Cas9的PAM识别产生影响。
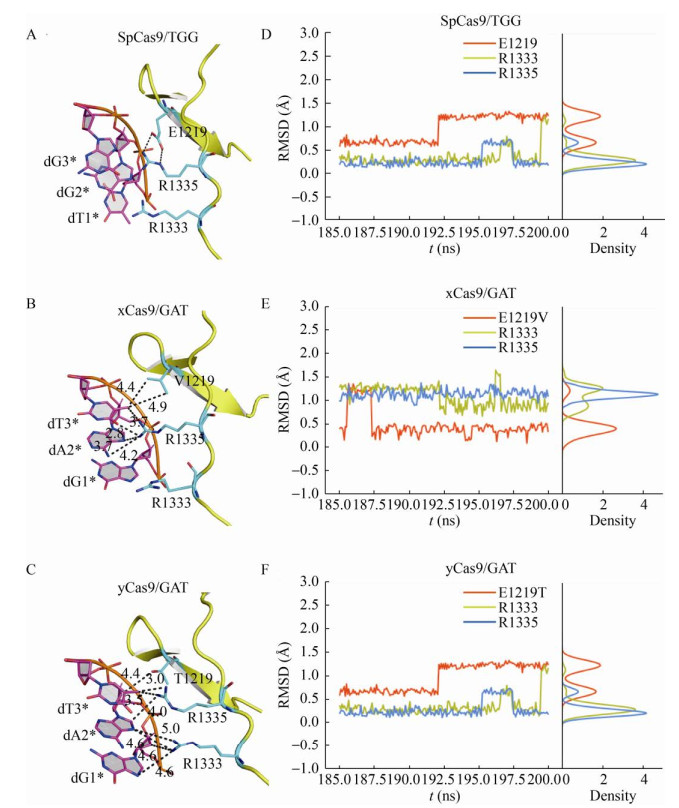 |
| 图 4 SpCas9及其突变体的PAM识别图 Fig. 4 Structural insight into PAM recognition by SpCas9 and its mutants. (A–C) Zoom-in views of PAM recognition by SpCas9 (4UN3), xCas9 (6AEG) and yCas9, respectively. (D–F) RMSDs of 1 219, 1 333 and 1 335 residues in SpCas9 (4UN3), xCas9 (6AEG) and yCas9, respectively. |
| 图选项 |
2.3.2 脱靶效应分析另外,为进一步阐明Cas9的脱靶机理,本研究构建了带TGG PAM序列的xCas9和yCas9的模拟体系。MD结束后,我们取出xCas9和yCas9最后一帧的构象,把它们分别与野生型SpCas9的构象叠合,发现这两个突变体的REC结构域构象有所改变。因此,分别计算了xCas9和SpCas9、yCas9和SpCas9的REC域的RMSD值(图 5A)。结果显示,xCas9移动了4.13×10–11 m,yCas9移动了2.47×10–11 m。最后,我们分析了位于REC域的324和409位的氨基酸(图 5B–D)。由图可知,与野生型SpCas9相比(图 5B),xCas9的R324突变成L324、S409突变成I409,这使得324和409位的氨基酸与周围氨基酸的氢键作用明显减少,只与S320、H328和H167之间存在相互作用(图 5C)。相同地,图 5D表明yCas9的这两个氨基酸与周围氨基酸之间的氢键相互作用也相应减少。所以,xCas9与yCas9的REC结构域构象的变化影响了Cas9脱靶率,且REC域构象改变越大,脱靶率越低。以上结果说明,Cas9的REC域构象重排会对其特异性产生影响。
 |
| 图 5 SpCas9与其突变体的结构比较 Fig. 5 Structural comparison of SpCas9 with its mutants. (A) Superimposition of the REC lobes of SpCas9 (4UN3), xCas9 (6K4P) and yCas9. (B–D) Zoom-in views of 324 and 409 residues in SpCas9, xCas9 and yCas9, respectively. |
| 图选项 |
3 讨论基因编辑是对生物遗传物质进行特异性改变的一种技术,常见的有3种:锌指核酸酶技术[20]、转录激活因子样效应物核酸酶技术[21]和CRISPR系统[22]。目前,CRISPR系统应用最为广泛,主要分为Ⅰ型、Ⅱ型和Ⅲ型[23]。其中,Ⅰ型和Ⅲ型CRISPR系统由多个Cas蛋白组成,需要专门的Cas核酸内切酶处理crRNA。相反,Ⅱ型系统通过不同的方式处理crRNA,Cas蛋白数量明显减少,所以成为最常用的一种类型。
目前,已经有多种天然的Ⅱ型CRISPR核酸酶用于基因编辑。其中,来自酿脓链球菌的Cas9蛋白SpCas9能识别简单的NGG PAM,且其在哺乳动物细胞中的剪切活性高,所以在生物学、生物技术和医学等领域得到了广泛的应用,例如构建细胞模型、动物模型以及治疗疾病等[24-25]。但是,SpCas9仍然存在许多待解决的挑战性问题,比如其PAM识别范围有限[10]、存在脱靶效应等[3]。其中,低兼容性的PAM识别模式限制了SpCas9在基因编辑领域的应用范围,而脱靶效应的存在,则进一步限制了其走向临床应用。为了解决以上问题,人们对SpCas9进行了各种优化改造。
在以往的研究中,人们主要通过序列比对或者定向进化对Cas9蛋白进行改造[26-28]。这些方法有一定的盲目性,往往需要反复试验、耗时耗力。本研究基于Cas9蛋白的三维结构,通过Rosetta程序优化Cas9-DNA相互作用界面,以设计筛选突变体。与以往的SpCas9改造方法相比,本文所用的理性设计方法盲目性低、操作简单、效率高,且具有通用性,可用于其他Cas9蛋白的改造。随着X光晶体衍射技术和冷冻电镜技术的快速发展,越来越多的Cas9蛋白结构被解析[29-31],为该方法的应用提供了坚实的基础。通过分析比较蛋白的电子密度图,找出对Cas9某一功能有重要影响的氨基酸位点,再结合Rosetta进行设计,以获得新型突变体。因此,本研究为人们继续开发多PAM识别和低脱靶的多功能型Cas9蛋白提供了新的途径。
另外,本文通过上述理性设计方法成功获得了一个xCas9新突变体(yCas9)。体外PAM识别检测结果显示,该突变体具有宽泛的PAM识别范围,可识别NGG、NGA、NGT、NGC、GAA和GAT,且对GAT PAM的识别能力高于xCas9。脱靶效应检测结果显示,yCas9的特异性虽然比xCas9稍低,但是与野生型SpCas9相比大幅度提高。因此,yCas9是一个可与xCas9媲美的、有潜在应用价值的基因编辑工具。由于本研究的主要目的是验证基于结构的Cas9蛋白的优化设计方法是否可行,因此,对突变体的活性检测主要在体外进行。当然,为了更准确地描述突变体在动物细胞内的PAM识别能力和脱靶效应,需要进一步开展体内实验:关于PAM活性检测,拟将Cas9蛋白和sgRNA的质粒共转染至含有不同PAM序列的荧光报告基因文库的细胞系中进行编辑,再用流式细胞仪分选出荧光报告基因阳性的细胞,提取其基因组DNA,通过PCR构建二代测序文库,分析并找出产生基因编辑的PAM序列[32];关于脱靶活性检测,拟在HEK293T和U2OS细胞中用GUIDE-seq对SpCas9、xCas9和yCas9进行双链断裂的全基因组无偏差鉴定[25]。
最后,本研究还分析了Cas9的PAM识别和脱靶效应的分子机理,发现了E1219突变为V/T后可以提高R1333和R1335的结构柔性,从而使Cas9可识别较宽泛的PAM序列。另外,我们还发现了R324和S409突变可以促使REC域构象重排,从而使Cas9有高特异性。这些结果为CRISPR/Cas9系统的后续改造提供了重要理论指导和坚实基础。
综上所述,本研究提出了一种用Rosetta优化设计Cas9蛋白的新方法,获得了一个多PAM识别和低脱靶的新突变体yCas9,为生物医学领域提供了有效的基因编辑新工具,同时也为后续优化Cas9蛋白提供了新思路。
参考文献
| [1] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014, 32(4): 347-355. |
| [2] | Lier C, Baticle E, Horvath P, et al. Analysis of the type Ⅱ-A CRISPR-Cas system of Streptococcus agalactiae reveals distinctive features according to genetic lineages. Front Genet, 2015, 6: 214. |
| [3] | Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827-832. |
| [4] | Fu YF, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014, 32(3): 279-284. |
| [5] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. |
| [6] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 2015, 523(7561): 481-485. |
| [7] | Chen JS, Dagdas YS, Kleinstiver BP, et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature, 2017, 550(7676): 407-410. |
| [8] | Kleinstiver BP, Pattanayak V, Prew MS, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016, 529(7587): 490-495. |
| [9] | Hu JH, Miller SM, Geurts MH, et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 2018, 556(7699): 57-63. |
| [10] | Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 2018, 361(6408): 1259-1262. |
| [11] | Legut M, Daniloski Z, Xue XH, et al. High-throughput screens of PAM-flexible Cas9 variants for gene knockout and transcriptional modulation. Cell Rep, 2020, 30(9): 2859-2868.e5. |
| [12] | Kuhlman B, Dantas G, Ireton GC, et al. Design of a novel globular protein fold with atomic-level accuracy. Science, 2003, 302(5649): 1364-1368. |
| [13] | Leaver-Fay A, Tyka M, Lewis SM, et al. ROSETTA3: an object-oriented software suite for the simulation and design of macromolecules. Methods Enzymol, 2011, 487: 545-574. |
| [14] | Broo KS, Brive L, Ahlberg P, et al. Catalysis of hydrolysis and transesterification reactions of p-nitrophenyl esters by a designed helix-loop-helix dimer. J Am Chem Soc, 1997, 119(47): 11362-11372. |
| [15] | Abraham MJ, Murtola T, Schulz R, et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX, 2015, 1-2: 19-25. |
| [16] | Ray A, Di Felice R. Protein-mutation-induced conformational changes of the DNA and nuclease domain in CRISPR/Cas9 systems by molecular dynamics simulations. J Phys Chem B, 2020, 124(11): 2168-2179. |
| [17] | Govindarajan S, Goldstein RA. On the thermodynamic hypothesis of protein folding. Proc Natl Acad Sci USA, 1998, 95(10): 5545-5549. |
| [18] | Anfinsen CB. Principles that govern the folding of protein chains. Science, 1973, 181(4096): 223-230. |
| [19] | Alford RF, Leaver-Fay A, Jeliazkov JR, et al. The Rosetta all-atom energy function for macromolecular modeling and design. J Chem Theory Comput, 2017, 13(6): 3031-3048. |
| [20] | Urnov FD, Rebar EJ, Holmes MC, et al. Genome editing with engineered zinc finger nucleases. Nat Rev Genet, 2010, 11(9): 636-646. |
| [21] | Miller JC, Tan SY, Qiao GJ, et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol, 2011, 29(2): 143-148. |
| [22] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. |
| [23] | Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol, 2011, 9(6): 467-477. |
| [24] | Xue W, Chen SD, Yin H, et al. CRISPR-mediated direct mutation of cancer genes in the mouse liver. Nature, 2014, 514(7522): 380-384. |
| [25] | Matano M, Date S, Shimokawa M, et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nat Med, 2015, 21(3): 256-262. |
| [26] | Casini A, Olivieri M, Petris G, et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nat Biotechnol, 2018, 36(3): 265-271. |
| [27] | Slaymaker IM, Gao LY, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84-88. |
| [28] | Ma DC, Xu ZM, Zhang ZY, et al. Engineer chimeric Cas9 to expand PAM recognition based on evolutionary information. Nat Commun, 2019, 10: 560. |
| [29] | Nishimasu H, Cong L, Yan WX, et al. Crystal Structure of Staphylococcus aureus Cas9. Cell, 2015, 162(5): 1113-1126. |
| [30] | Huai C, Li G, Yao RJ, et al. Structural insights into DNA cleavage activation of CRISPR-Cas9 system. Nat Commun, 2017, 8: 1375. |
| [31] | Yamano T, Nishimasu H, Zetsche B, et al. Crystal structure of Cpf1 in complex with guide RNA and target DNA. Cell, 2016, 165(4): 949-962. |
| [32] | Hu ZY, Wang S, Zhang CD, et al. A compact Cas9 ortholog from Staphylococcus auricularis (SauriCas9) expands the DNA targeting scope. PLoS Biol, 2020, 18(3): e3000686. |
