

江南大学 生物工程学院 糖化学与生物技术教育部重点实验室,江苏 无锡 214122
收稿日期:2019-12-02;接收日期:2020-02-04
基金项目:国家自然科学基金(No. 31800671),中国博士后科学基金(No. 2019M651691)资助
摘要:提高酶的热稳定性是生物催化领域的热点和难点,计算机辅助的理性设计相比于传统的定向进化更加高效,在酶工程领域中的应用越来越广泛和深入。文中以枯草芽孢杆菌脂肪酶A为模式蛋白,首先,利用Rosetta-VIP计算设计对酶的结构空腔进行分析,选择了16个有利于结构空腔填充(ΔΔE < 0)的单点突变,并以突变位点的溶剂可及表面积和进化保守性为二次筛选依据,测定了其热稳定性与酶活性。有6个单点突变体(F17A、V74I、L114P、I135V、M137A、I157L)的热稳定性得到了提高,其中Tm值最大提高3.18 ℃。结果表明,单点突变体满足ΔΔE越低、蛋白溶剂可及表面积减少且符合序列保守性,则得到保留原有酶活力的正向突变的可能性越大。此外,将热稳定性提高的6个单点突变进行迭代组合突变,两点组合突变体的Tm最大提高4.04 ℃,三点组合突变体的Tm最大提高5.13 ℃,四点组合突变体的Tm提高了7.30 ℃,六点组合突变体的Tm提高了7.43 ℃。因此,基于酶的分子结构的空腔分析、溶剂可及表面积及氨基酸序列保守性计算的多重虚拟筛选方法,可有效提高酶的热稳定性。
关键词:热稳定性枯草芽孢杆菌脂肪酶A蛋白质计算设计
Improving the thermal stability of Bacillus subtilis lipase based on multiple computational design strategies
Yu Xiang*, Meng Zhang*, Fei Xu


The Key Laboratory of Carbohydrate Chemistry and Biotechnology, Ministry of Education, School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
Received: December 2, 2019; Accepted: February 4, 2020
Supported by: National Natural Science Foundation of China (No. 31800671), China Postdoctoral Science Foundation (No. 2019M651691)
Corresponding author: Fei Xu. Tel/Fax: +86-510-85197571; E-mail: feixu@jiangnan.edu.cn.
*These authors contributed equally to this study
Abstract: Improving the thermal stability of enzymes is a hot and difficult point in the field of biocatalysis. Compared with the traditional directed evolution, computational assisted rational design is more efficient, and is widely used in enzyme engineering. Using Bacillus subtilis LipA as the model protein, the structure cavity of the enzyme was analyzed by Rosetta-VIP design, the mutation which was beneficial to the filling of the structure cavity (ΔΔE < 0) was selected, followed by the solvent accessible surface area and evolutionary conservation analysis. The thermal stabilities of six out of sixteen designed single-point mutants were improved, with a maximum ΔTm value of 3.18 ℃. These six mutations were further used for iterative combination mutation, the maximum ΔTm of the two-point and three-point combination mutants were 4.04 ℃ and 5.13 ℃, respectively. The Tm of the four-point combination mutant M11 (F17A/L114P/I135V/M137L) was increased by 7.30 ℃. The Tm of the six-point combination mutant M10 (F17A/V74I/L114P/I135V/M137A/I157L) was increased by 7.43 ℃. The thermal stability of mutation with lower energy value, reduced accessible surface area, while conformed to evolutionary conservatism, was more likely to be improved. Therefore, the multiple virtual screening strategy based on the enzyme structure cavity filling, solvent accessible surface area and amino acid sequence conservation analysis can effectively improve the thermal stability of enzyme.
Keywords: thermostabilizationBacillus subtilis LipAcomputational protein design
酶在生物催化和合成生物学中占有十分重要的位置,脂肪酶作为第3大工业用酶,在油脂加工、皮革、造纸、生物柴油等领域有广泛应用[1-2],但是天然酶分子结构大多不稳定,难以满足工业应用过程中对耐高温的要求,因此针对酶的热稳定性改造一直是酶工程领域的长期目标[3]。枯草芽孢杆菌脂肪酶A (Bacillus subtilis LipA,BSL)对碱性pH具有很强的耐受性,能够水解具有sn-1和sn-2甘油酯的长脂肪链,对中等长度(C8)的脂肪酸链具有最高的催化活性,在生产生物柴油和洗涤剂方面具有很大的应用潜力[4],然而野生型枯草芽孢杆菌脂肪酶的热稳定性较低,难以满足工业需求。Rao等通过易错PCR对枯草芽孢杆菌脂肪酶A进行体外定向进化,突变体6B的Tm值提高了22 ℃,T50提高了30 ℃[5-7];Reetz等通过5轮迭代饱和突变获得的突变体,其t1/2提高了900–1 000 min,T50提高了41–45 ℃[8-10]。传统的定向进化虽然可以针对工业应用的特定要求对酶进行筛选,但它通常需要经过多轮的突变和筛选,且对筛选手段的高通量要求很高、耗时耗力[11-12]。而理性设计具有更强的目的性,更加高效、快捷,基于酶的结构,利用计算机辅助的理性设计在酶工程领域中的应用越来越广泛和深入[13-14]。枯草芽孢杆菌脂肪酶A由181个氨基酸残基组成,是目前已知的3个分子量最小的脂肪酶之一,并且其晶体结构已被解析,结构简单,为典型的α/β水解酶折叠域结构,由中心的6条平行β链组成的β片层及两侧5个α螺旋包围构成,不包含大多数脂肪酶具有的盖子结构,是少数在油水界面存在时不显示界面激活效应的脂肪酶之一[4],往往被用作蛋白计算设计的模式蛋白[15-16]。
近年来,基于酶分子结构的计算设计来提高酶热稳定性的策略受到广泛关,如基于能量和序列分析提高酶表达量和稳定性的PROSS server[17]、利用非天然共价键提高酶热稳定性[18]、优化蛋白表面电荷提高酶的热稳定性[19]等。酶的三维结构的核心中,侧链基团的紧密堆积对酶的折叠和稳定性至关重要,在天然酶结构中往往会存在一些空腔,Rosetta-VIP通过将空腔周围的氨基酸突变成侧链基团更大或非极性氨酸,如甘氨酸、丙氨酸、缬氨酸、苯丙氨酸、异亮氨酸、亮氨酸、甲硫氨酸、色氨酸和酪氨酸,可以对蛋白空腔进行填充,提高酶包装的紧密性,从而提高酶的热稳定性[20]。这一方法能很好地将酶的几何结构与空腔填充结合起来,但是无法对空腔结构的体积特征作出分析,并且有些通过Rosetta-VIP筛选的可填充空腔是位于蛋白表面的,难以确定一个空腔或多个空腔突变填充后对酶的整体影响[21-22]。作用在非极性侧链上的疏水效应是驱动蛋白折叠的一个重要因素,而蛋白溶剂可及表面积(Solvent accessible surface area,SASA)反映了氨基酸残基的埋藏程度和表面可及性[23],可以用来评估疏水效应对蛋白折叠自由能影响的大小,有助于分析突变对蛋白稳定性的影响[24]。在自然进化过程中,不利突变往往被自然选择而淘汰,而利用序列比对可以排除不利的突变(共识效应)[17];对共识序列的突变分析可以作为过滤器,协助排除对酶的功能造成损害的不利突变[25]。
因此,本研究采用多重计算设计策略,利用Rosetta-VIP对枯草芽孢杆菌脂肪酶的包装和空腔进行鉴定和分析,结合蛋白可及表面积和进化保守性分析选择合理的突变,从而提高酶的热稳定性。
1 材料与方法1.1 材料1.1.1 菌株与质粒大肠杆菌Escherichia coli BL21 (DE3)菌株及表达载体pET-28a (+)购于Novagen公司,枯草芽孢杆菌脂肪酶A基因bsl由苏州金唯智有限公司合成。
1.1.2 主要试剂PrimeSTAR HS (premix)高保真PCR酶,限制性核酸内切酶Hind Ⅲ、Nco Ⅰ,DNA及蛋白Marker均购于TaKaRa公司;质粒提取、PCR产物回收试剂盒、DNA胶回收试剂盒均购于生工生物工程(上海)股份有限公司;p-nitrophenyl octanoate (pNPC)、p-nitrophenol (pNP)购于Sigma-Aldrich公司,其余试剂均为市售分析纯;BCA蛋白浓度测定试剂盒(增强型)购于碧云天生物技术有限公司。酶及试剂盒的使用方法均按照各公司提供的说明书进行。
1.2 方法1.2.1 枯草芽孢杆菌脂肪酶A的计算设计本研究以枯草芽孢杆菌脂肪酶的结构空腔分析、蛋白溶剂可及表面积和氨基酸序列保守性为依据,计算设计热稳定性提高的突变体(图 1)。首先,使用Rosetta-VIP[20]在线服务器(https://rosie.graylab.jhu.edu/vip)对枯草芽孢杆菌脂肪酶A进行计算设计,其原理是利用RosettaHoles搜索蛋白内部中的疏水空隙,并确定该空隙周围的氨基酸组成,将这些氨基酸突变为疏水氨基酸,并进行结构优化,计算单点突变的ΔΔE值(突变体与野生型能量值的变化,ΔΔE值越小表示设计的突变体比野生型越稳定),并从这些点突变中选择ΔΔE值最小的点,在此突变的基础上进行下一轮迭代,上述过程持续进行直至找不到ΔΔE值更低的突变位点为止,本实验选择最多5轮突变5轮迭代进行运算。其次,利用Accessible Surface Area and Accessibility Calculation for Protein (ver. 1.2)在线服务器(http://cib.cf.ocha.ac.jp/bitool/ASA/)对突变前后的氨基酸残基进行计算,将突变后氨基酸的SASA与野生型蛋白SASA的差值作为ΔSASA。同时,为了探究枯草芽孢杆菌脂肪酶A的序列保守性,将枯草芽孢杆菌脂肪酶A蛋白质序列在NCBI非冗余蛋白质数据库中进行序列相似性的搜索,一共比对了相似度大于50%的500个蛋白氨基酸序列,综合500个序列比对的结果,找出突变位点氨基酸残基多样性,根据它们出现的可能性大小进行排序(以下所有序列保守性结果都是在这500个序列比对结果的基础上进行分析讨论的)。
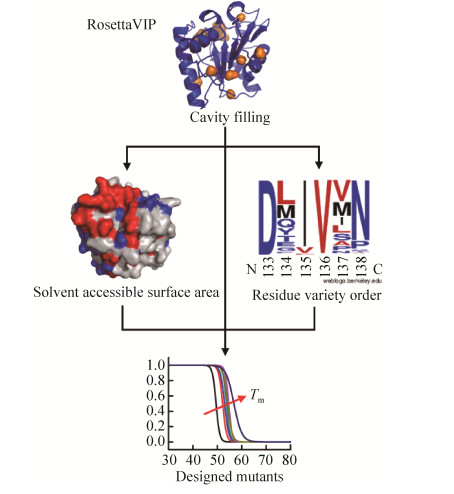 |
| 图 1 计算设计策略示意图 Fig. 1 Schematic diagram of computational design. |
| 图选项 |
1.2.2 定点突变以质粒pET28a-bsl为模板,根据突变体设计PCR引物(表 1)进行全质粒PCR,将PCR产物回收后,用T4多核苷酸激酶进行磷酸化修饰,利用T4 DNA连接酶连接8 h。将处理后的产物用化学转化法导入E. coli BL21 (DE3)感受态中。
表 1 本研究所用引物Table 1 Primers used in this study
| Primer name | Primer sequence (5′–3′) | Size (bp) |
| F17A-F | TGGTGCTTCTGCAAACTTCGCTG | 23 |
| F17A-R | CCGATACCGTGAACCATAACAAC | 23 |
| F19W-F | TCTTTCAACTGGGCTGGTATCAAA | 24 |
| F19W-R | AGCACCACCGATACCGTGAACC | 22 |
| A20I-F | TTTCAACTTCATTGGTATCAAATC | 24 |
| A20I-R | GAAGCACCACCGATACCGTGAAC | 23 |
| W42L-F | GTTGACTTCCTGGACAAAACCG | 22 |
| W42L-R | AGCGTACAGTTTGTCACGAGAC | 22 |
| F58L-F | TCTGTCTCGTCTGGTTCAGAAAG | 23 |
| F58I-F | TCTGTCTCGTATTGTTCAGAAAG | 23 |
| F58L/I-R | ACCGGACCGTTGTTGTAGTTGG | 22 |
| V59I-F | GTCTCGTTTCATTCAGAAAGTTC | 23 |
| V59I-R | AGAACCGGACCGTTGTTGTAGT | 22 |
| V74I-F | GTTGACATCATTGCTCACTCTAT | 23 |
| V74I-R | TTTTTTAGCACCGGTTTCGTCC | 22 |
| M78W-F | TAACACCCTGTACTACATCAAAAA | 24 |
| M78W-R | GCACCACCCCAAGAGTGAGCAAC | 23 |
| L102I-F | CGTTGTTACCATTGGTGGTGCTA | 23 |
| L102I-R | TTAGCAACTTTGTTACCACCGTC | 23 |
| L114P-F | GGTAAAGCTCCGCCGGGTACCGA | 23 |
| L114P-R | GGTGGTCAGACGGTTAGCAC | 20 |
| M134L-F | CTGCTGACCTGATCGTTATGAACT | 24 |
| M134L-R | AAGAGTAGATAGAGGTGTACAG | 22 |
| I135V-F | TGCTGACATGGTTGTTATGAACT | 23 |
| I135V-R | GAAGAGTAGATAGAGGTGTACAG | 23 |
| M137A-F | TGATCGTTGCAAACTACCTGTCT | 23 |
| M137A-R | TGTCAGCAGAAGAGTAGATAGAG | 23 |
| I157L-F | CGGTGTTGGTCACCTGGGTCTGC | 23 |
| I157L-R | TGGATCTGAACGTTACGAGCACC | 23 |
表选项
1.2.3 野生型及突变体蛋白的表达纯化将甘油管保存的E. coli BL21 (DE3)/pET28a-bsl及其突变菌株平板划线于含卡那霉素的固体LB平板,倒置于37 ℃恒温培养箱中过夜培养,挑取单菌落至20 mL LB液体培养基中,置于37 ℃、220 r/min摇床培养9 h,按1%接种量接种至100 mL TB培养基中,置于37 ℃、220 r/min摇床培养至OD600=0.6–0.8,加入终浓度为1 mmol/L的IPTG,放入25 ℃、220 r/min摇床诱导发酵10 h。根据需要,在以上培养基中加入终浓度为100 μg/mL的卡那霉素。
7 000 r/min离心10 min收集发酵菌体,加入30 mL 50 mmol/L Tris-HCl缓冲液(pH 7.5) (含500 mmol/L的NaCl)重悬菌体,在冰上超声破碎15 min (超声2 s,间歇3 s)后,4 ℃、10 000 r/min离心20 min收集破壁上清,经0.45 μm滤膜过滤后,进行Ni2+柱亲和层析纯化。先用A液(20 mmol/L PBS,含1 mol/L的NaCl,20 mmol/L的咪唑)平衡HisTrap HP 1 mL Ni2+亲和层析柱15个柱体积,再将破壁上清以1 mL/min的流速上样,上样结束后,用10%的B液(20 mmol/L PBS,含1 mol/L的NaCl,500 mmol/L的咪唑)进行梯度洗脱除去杂蛋白,再用10%–100%的B液进行线性洗脱获得目的蛋白,用HiTripTM 5 mL Desalting凝胶柱对目的蛋白进行脱盐处理,并更换至的50 mmol/L的磷酸钠缓冲液(pH 8.0)中,最后用超滤管将目的蛋白浓缩至浓度为0.2 mg/mL。对分离纯化后产物,用SDS-PAGE进行检验。
1.2.4 野生型及突变体蛋白的酶活测定酶活定义:40 ℃,pH 8.0,每分钟水解pNPC生成1 μmol/L pNP所需的酶量。
酶活测定:于1.5 mL离心管中先加入450 μL,磷酸钠缓冲液(pH 8.0),再添加25 μL 20%的TritonX-100,振荡混匀后加入25 μL 50 mmol/L的pNPC溶液,充分混匀后超声2 min分装190 μL至96孔板中,先于40 ℃酶标仪中孵育20 min,加入10 μL酶液反应2 min,测定410 nm处的吸光值。测酶活所用的酶液都经过纯化脱盐后稀释至(0.06±0.02) mg/mL。
1.2.5 野生型及突变体的热稳定性测定圆二色谱测定蛋白二级结构及解折叠温度Tm:将纯化脱盐后的样品酶浓度均调节至(0.16±0.03) mg/mL,取200 μL用于圆二色谱仪测定全波长扫描及热变。于190–260 nm进行全波长扫描,取220 nm作为热变吸光值的测定,热变温度范围为30–80 ℃,升温梯度为1 ℃/min。
热半失活温度T50的测定:将纯化脱盐的酶液分别于30–60 ℃ (间隔5 ℃)孵育10 min后取出于冰上放置10 min,测定其残余酶活。
热半失活时间t1/2的测定:将纯化脱盐的酶液于55 ℃分别孵育0–30 min (间隔5 min),取出于冰上放置10 min,测定其残余酶活。
2 结果与分析2.1 枯草芽孢杆菌脂肪酶A的计算设计利用Rosetta-VIP对枯草芽孢杆菌脂肪酶A结构进行分析,共计算设计出16个有利于结构空腔填充(ΔΔE < 0)的单点突变,对其能量变化、溶剂可及表面积和序列保守性分析,如表 2所示。ΔΔE值越小,表示设计的突变体比野生型越稳定,其中突变体F17A、F19W、L114P、I135V和I157L的ΔΔE < -5.0,预测这5个突变体热稳定性较高,其中I157L (ΔΔE=-9.57)是设计的16个突变体中能量降低最多的。同时,这5个突变体的ΔSASA < 0,突变体中对应突变位点的溶剂可及表面积减少,更有利于蛋白的稳定,其中F17A的溶剂可及表面积是16个突变体中减少量最多的(ΔSASA=-90.33 ?2),F19W的溶剂可及表面积变化较小(ΔSASA=-0.21 ?2)。并且F19W没有出现在序列比对的500条同源序列中,不符合蛋白进化保守性,是不利突变的可能性大,其余4个点突变符合蛋白进化保守性。
表 2 Rosetta-VIP计算设计突变位点及其能量、溶剂可及表面积和序列保守性Table 2 Rosetta-VIP designed mutants and their energies, solvent accessible surface areas and residue variety order
| Variant | ΔΔE | ΔSASA (?2) | Residue variety order |
| F17A | -6.20 | -90.33 | Y > F > S > T > A=N > G |
| F19W | -5.88 | -0.21 | F > S=Y |
| L114P | -5.34 | -20.60 | L > P > I > F > Y=N > K=A |
| I135V | -6.57 | -23.20 | I > V > L > N > S |
| I157L | -9.57 | -11.62 | I > L > V > S > M > T=N=H |
| F58I | -1.19 | -18.79 | F > Y > M > A > I > K > L=Q |
| F58L | -1.42 | -4.56 | F > Y > M > A > I > K > L=Q |
| V59I | -2.85 | 0.00 | V > I > A=D > E=F=N |
| V74I | -1.89 | 0.00 | V > I > Y |
| M78W | -2.84 | +62.42 | M > A > L > V > S=H |
| L102I | -2.06 | 0.00 | L > I > V=K > F > T=S |
| M137A | -1.01 | -72.34 | V > M > I > S > L > A > P > N > D > R > Y=F |
| I157A | -1.44 | -43.96 | I > L > V > S > M > T=N=H |
| A20I | -0.90 | +53.92 | A > F > Y > I > T > D > N > M > H > L > E=Y=S > G=R |
| W42L | -0.80 | +12.28 | I > Y > W > P > L > V > A > S > N > Q > T=F > R=M=D > K |
| M134L | -0.58 | +12.68 | L > M > Q > Y > T > > E > G > F > D > C > R=I > S=A |
表选项
单点突变体F58I、F58L、V59I、V74I、M78W、L102I、M137A、I157A的-5.0 < ΔΔE < -1.0,ΔΔE在16个突变体中大小居中,说明这8个突变体的热稳定性有可能提高。其中M137A (ΔSASA= -72.34 ?2)、I157A (ΔSASA=-43.96 ?2)和F58I (ΔSASA=-18.79 ?2)、F58L (ΔSASA=-4.56 ?2)的蛋白溶剂可及表面积减少,说明突变可能有利于蛋白的稳定,V59I、V74I、L102I突变前后的ΔSASA=0 ?2,说明这3个氨基酸位点在蛋白分子内部,与分子内部空腔填充有关,M78W的溶剂可及表面积是所有突变体中增加最大的(ΔSASA= +62.42 ?2),突变有可能影响蛋白质表面排布,从而降低稳定性。F58I、F58L、V59I、V74I、L102I和M137A符合蛋白进化保守性,而M78W和I157A没有出现在序列比对的500条同源序列中,不符合蛋白进化保守性,是不利突变的可能性大。
突变体A20I、W42L、M134L的ΔΔE > -1.0,在16个计算设计的突变体中ΔΔE偏高,预测这3个突变体的热稳定性变化较小。它们的ΔSASA > 0,突变体中对应的突变位点残基蛋白溶剂可及表面积增加,也说明突变可能破坏蛋白稳定性。这3个突变符合蛋白进化保守性。
2.2 野生型脂肪酶及突变体在大肠杆菌中的表达纯化将野生型枯草芽孢杆菌脂肪酶A表达纯化后取20 μL破壁上清样品和纯化后获得的样品,上样至SDS-PAGE进行分析。如图 2所示,SDS-PAGE中目的蛋白条带大小为19.3 kDa与理论值相符,测定野生型枯草芽孢杆菌脂肪酶A的比酶活为(9.24±0.36) U/mg。用表 1中的引物进行全质粒PCR突变,将突变后质粒测序验证正确后,进行蛋白的表达纯化。所有的单点突变均能在大肠杆菌中可溶性表达,且条带对应分子量大小正确,纯度在90%以上(图 2,泳道4–19)。
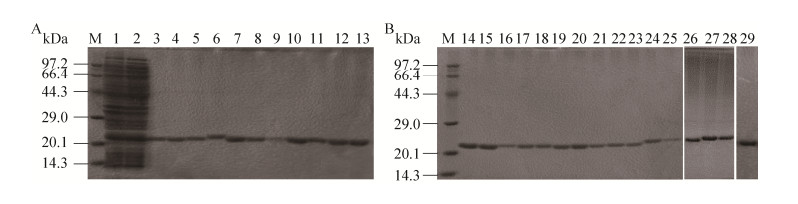 |
| 图 2 野生型枯草芽孢杆菌脂肪酶A及突变体在大肠杆菌中的表达纯化 Fig. 2 Wild-type BSL and mutants expression in E. coli BL21 (DE3). M: premixed protein marker; 1: whole cell protein of wild-type BSL; 2: soluble extract of wild-type BSL; 3: purified wild-type BSL; 4–25: purified protein of mutant F17A, F19W, L114P, I135V, I157L, F58I, F58L, V59I, V74I, M78W, L102I, M137A, I157A, A20I, W42L, M134L, M2, M3, M4, M5, M9, M10 respectively; 26–29: purified protein of mutant M6, M7, M8, M11 respectively. |
| 图选项 |
2.3 突变体酶学性质测定与分析首先用圆二色谱仪对野生型和单点突变蛋白在190 nm–260 nm进行全波长扫描,并且测定了野生型和突变蛋白的Tm。全波长扫描结果如图 3A所示,Tm值测定结果如图 3B所示。根据全波长扫描结果可知,所有的突变蛋白的扫描结果与野生型变化趋势相同,说明突变蛋白全都折叠形成了正确的二级结构。根据Tm测定结果可知,Tm值上升的突变体有F17A、V74I、L114P、I135V、M137A、I157L。
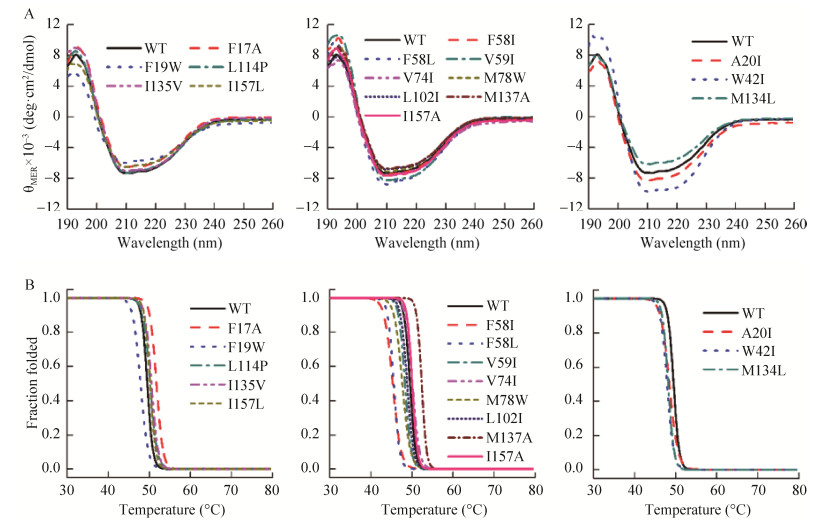 |
| 图 3 单突变位点的圆二色谱分析 Fig. 3 Circular dichroism spectra and melting curves of single-point mutants. (A) CD spectra. (B) Melting curves. |
| 图选项 |
ΔΔE < -5.0的5个点突变中,有4个点突变的热稳定得到提高,分别是F17A (ΔTm=+2.24 ℃)、L114P (ΔTm=+0.78 ℃)、I135V (ΔTm=+1.13 ℃)和I157L (ΔTm=+0.78 ℃),突变体F19W (ΔTm= -1.82 ℃)的溶剂可及表面积变化较小,且不符合蛋白进化保守性,热稳定性降低。ΔΔE值大小排序为I157L < I135V < F17A < L114P,ΔSASA大小排序为I157L < L114P < I135V < F17A,ΔTm大小排序为L114P=I157L < I135V < F17A。-5.0 < ΔΔE < -1.0的8个点突变中,热稳定性提高的有2个,分别是M137A (ΔTm=+3.18 ℃)和V74I (ΔTm=+0.49 ℃),其中M137A是单点突变中Tm上升最大的。ΔΔE > -1.0的3个点突变A20I (ΔTm=-1.08 ℃)、W42L (ΔTm=-1.53 ℃)、M134L (ΔTm=-1.27 ℃)热稳定性均有所降低。ΔΔE < -5.0的突变设计,稳定性提高的比例是80%,-5.0 < ΔΔE < -1.0的突变设计,稳定性提高的比例是25%,ΔΔE > -1.0的突变设计,稳定性提高的比例为0。
进一步测定了热稳定性提高的点突变的T50和t1/2及所有单点突变的酶活,结果如表 3所示。Tm值上升的点突变T50值均得到提高,最低提高了0.8 ℃,最高提高了1.5 ℃。t1/2只有F17A和M137A得到了提高,分别提高了0.4 min和1.2 min。此外,热稳定性提高的6个单点突变体均有酶活,其中L114P的比酶活增加了22%,I135V比酶活降低了26%;A20I和M134L热稳定性有所降低,但酶活分别增加了22.7%和48.8%;M78W和I157A突变位点靠近催化三联体中的S77和H156,且突变不符合进化保守性,导致剩余酶活不足20%。
表 3 单突变位点的ΔTm、T50、t1/2和比酶活Table 3 ΔTm, T50, t1/2 and specific activities of single-point mutants
| Variant | ΔTm (℃) | T50 (℃) | t1/2 (min) | Relative activity (%) |
| WT | 0.00 | 51.2±0.28 | 2.5±0.14 | 100.0 |
| F17A | +2.24 | 52.2±0.22 | 2.9±0.18 | 94.0 |
| F19W | -1.82 | / | / | 93.6 |
| L114P | +0.78 | 52.1±0.23 | 2.5±0.14 | 122.0 |
| I135V | +1.13 | 52.7±0.26 | 2.5±0.21 | 73.8 |
| I157L | +0.78 | 52.5±0.28 | 2.5±0.25 | 105.8 |
| F58I | -4.30 | / | / | 66.3 |
| F58L | -4.00 | / | / | 102.4 |
| V59I | -1.21 | / | / | 76.6 |
| V74I | +0.49 | 52.5±0.14 | 2.5±0.21 | 93.5 |
| M78W | -1.97 | / | / | 11.8 |
| L102I | -0.52 | / | / | 108.9 |
| M137A | +3.18 | 52.0±0.25 | 3.7±0.21 | 102.5 |
| I157A | +0.59 | / | / | 15.4 |
| A20I | -1.08 | / | / | 112.7 |
| W42L | -1.53 | / | / | 85.7 |
| M134L | -1.27 | / | / | 148.8 |
| / indicates that the data has not been determined by experiment. | ||||
表选项
2.4 复合突变体酶学性质的测定通过对枯草芽孢杆菌脂肪酶A三维结构及单点突变位置的分析,如图 4A所示,枯草芽孢杆菌脂肪酶A的催化三联体为S77-D133-H156,与M78、I12一起组成氧阴离子活性位点。热稳定性提高的单点突变体I135V、M137A和I157L的突变位点之间位置相近,且离活性中心较近,距离在8 ?以内(图 4B),而F17A、V74I、L114P的突变位点之间位置较远,且距离活性中心8 ?以外。因此,为了验证点突变之间是否具有叠加效应和协同效应,根据热稳定性提高的6个单点突变与活性中心之间的距离,将单点突变分为两组,分别设计了两点突变体M2 (I135V/I157L)、M3 (M137A/I157L)、M4 (I135V/M137A)、M6 (F17A/ V74I)、M7 (F17A/L114P)、M8 (V74I/L114P),三点突变体M5 (I135V/M137A/I157L)、M9 (F17A/V74I/L114P)以及六点突变体M10 (F17A/V74I/L114P/I135V/ M137A/I157L)共9个复合突变体。所有的复合突变均能在大肠杆菌中可溶性表达,且条带对应分子量大小正确,纯度在90%以上(图 2,泳道20–28)。
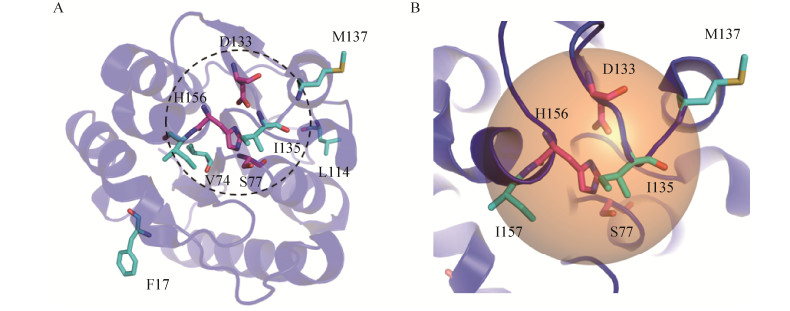 |
| 图 4 枯草芽孢杆菌脂肪酶A的三维结构及突变位点位置分析 Fig. 4 The 3D structure of Bacillus subtilis LipA and the location of mutations. Magenta: catalytic triad; cyans: mutations. (A) 3D structure of Bacillus subtilis LipA. (B) Analysis of mutation sites, orange sphere radius=8 ?. |
| 图选项 |
这9个复合突变体的全波长扫描结果如图 5A所示,热变扫描结果如图 5B所示,9个复合突变体的结构与野生型相比没有明显变化,都能正确折叠,Tm值都得到了提高。其中,离活性中心较近的单点突变I135V、M137A和I157L的两点组合突变体M2、M3、M4的Tm值分别提高了2.65 ℃、3.43 ℃、4.04 ℃,M2热稳定性提高的效果大于单点突变体I135V和I157L叠加的效果,ΔΔTm=+0.74 ℃,M3、M4热稳定提高的效果分别小于相应单点突变叠加的效果,ΔΔTm分别为-0.53 ℃和-0.27 ℃,但ΔΔTm变化幅度不大,说明空间距离较近的单点突变之间进行组合突变时,可产生一定的叠加效果。但同时距离活性中心较近的点突变容易对酶活产生不利影响,3个两点突变的酶活均有所降低,I135V单点突变的相对酶活为野生型的73.8%,突变对酶活的影响较大,含有I135V这一单点突变的M2和M4的酶活损失一半以上。三点组合突变M5的Tm值提高了5.13 ℃,与单点突变Tm叠加相比,ΔΔTm= +0.04 ℃,与两点组合趋势相近,具有一定的叠加效果。
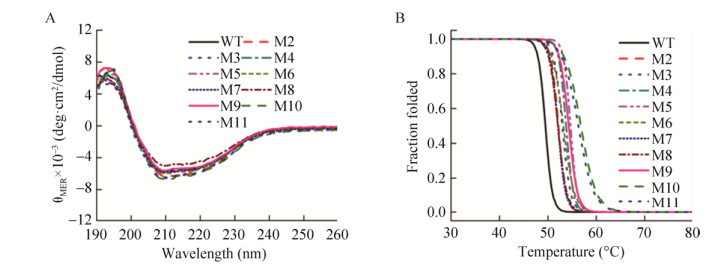 |
| 图 5 组合突变位点的圆二色谱分析 Fig. 5 Circular dichroism spectra and melting curves of combined mutants. (A) CD spectra. (B) Melting curves. |
| 图选项 |
而远离活性中心且突变位点之间位置较远的单点突变F17A、V74I和L114P进行两两组合突变后,M6、M7、M8的Tm值分别提高了4.03 ℃、2.86 ℃、2.86 ℃。M6、M8热稳定性提高的效果分别大于单点突变体F17A和V74I、V74I和L114P叠加的效果,ΔΔTm分别为+1.30 ℃和+1.59 ℃,M7热稳定性提高的效果小于相应单点突变叠加的效果,ΔΔTm=-0.16 ℃,说明空间距离较远且远离活性中心的单点突变之间进行组合突变时,可能通过复杂的别构效应机制产生一定的正协同效果,同时组合突变对酶活产生的不利影响较小,其中M7突变可以保持与野生型相当的酶活力。三点组合突变M9的Tm值提高了4.82 ℃,热稳定性提高的效果大于相应3个单点突变叠加的效果,ΔΔTm为+1.31 ℃,与两点组合趋势相近,具有一定的正协同效果。
进一步将热稳定性提高且同时保持较高酶活力的两点突变M3和M7的突变位点进行组合,经测定四点突变体M11 (F17A/L114P/M137A/ I157L)的Tm值与野生型相比提高了7.30 ℃ (图 5B)。同时,将6个单点突变全部组合,六点组合突变M10的Tm值提高了7.43 ℃,热稳定性提高的效果小于6个单点突变叠加,ΔΔTm值为-1.17 ℃,但M10的Tm值大于2个三点突变M5和M9的Tm。迭代组合突变体的热稳定性都得到了提高,稳定性提高程度:六点突变M10、四点突变M11 > 三点突变M5、M9 > 两点突变M2、M3、M4、M6、M7、M8 > 单点突变,随着突变点的累计,枯草芽孢杆菌脂肪酶A的热稳定性也逐步提高,迭代组合突变设计具有一定的叠加效果。
进一步测定了所有复合突变体的T50、t1/2和酶活,结果如表 4所示。两点突变M2、M3、M4、M6、M7、M8的T50分别提高了1.0 ℃、1.7 ℃、1.9 ℃、3.2 ℃、1.2 ℃和0.8 ℃,t1/2分别提高了0.9 min、3.8 min、7 min、7.3 min、3.2 min和2.7 min,比酶活分别降低了51.3%、12.9%、55.1%、39.3%、4.4%和14%;三点组合突变M5、M9的T50分别提高了2.1 ℃、2.5 ℃,t1/2分别提高了10.3 min和9.5 min,比酶活分别降低了64.5%和43.0%;四点突变M11的T50提高了5.9 ℃,t1/2提高了14.8 min,比酶活是野生型的99.7%;六点组合突变M10的T50提高了8.4 ℃,t1/2提高了18.2 min,比酶活降低了55.2 %。与全部六点的组合突变相比,将热稳定性提高同时酶活力较高的单点突变进行组合,可以维持较高的酶活力,尤其是I135V对酶活的破坏力较大,带有此位点的组合突变酶活降低较明显,说明酶的组合突变易受负突变影响,负突变效应较难通过其他突变弥补。
表 4 组合突变位点的ΔTm、T50、t1/2和比酶活Table 4 Tm, T50, t1/2 and specific activities of combined mutants
| Variant | ΔTm (℃) | T50 (℃) | t1/2 (min) | Relative activity (%) |
| WT | 0.00 | 51.2±0.28 | 2.5±0.14 | 100.0 |
| M2 (I135V/I157L) | +2.65 | 52.2±0.14 | 3.4±0.19 | 48.7 |
| M3 (M137A/I157L) | +3.43 | 52.9±0.28 | 6.3±0.21 | 87.1 |
| M4 (I135V/M137A) | +4.04 | 53.1±0.29 | 9.5±0.15 | 44.8 |
| M5 (I135V/M137A/I157L) | +5.13 | 53.3±0.19 | 12.8±0.14 | 35.5 |
| M6 (F17A/V74I) | +4.03 | 54.4±0.11 | 9.8±0.13 | 60.7 |
| M7 (F17A/L114P) | +2.86 | 53.0±0.15 | 5.7±0.17 | 95.6 |
| M8 (V74I/L114P) | +2.86 | 52.0±0.14 | 5.2±0.15 | 86.0 |
| M9 (F17A/V74I/L114P) | +4.82 | 53.7±0.21 | 12.0±0.19 | 57.0 |
| M10 (F17A/V74I/L114P/I135V/M137A/I157L) | +7.43 | 59.6±0.21 | 20.7±0.22 | 44.8 |
| M11 (F17A/L114P/M137A/I157L) | +7.30 | 57.1±0.04 | 17.3±0.08 | 99.7 |
表选项
3 结论首先利用蛋白空腔填充计算设计了突变位点,以突变能量变化、蛋白溶剂可及表面积分析、蛋白保守进化性分析为依据,确定了3组共16个单点突变,进一步通过实验测定,获得了热稳定性提高的6个单点突变,Tm值最大提高3.18 ℃。
其次,通过迭代组合突变设计,研究了不同组合突变对热稳定性的影响,组合突变的酶热稳定性都得到了提高,随着突变位点积累的越多,热稳定性越高,迭代突变设计具有一定的叠加效果[26],距离较远且远离活性中心的突变位点之间,可能通过别构效应对热稳定性的提高起到一定的正协同效应[27]。
对于大部分酶来说,酶活与稳定性之间往往存在竞争关系,酶的刚性越强、稳定性越高,但往往会对酶的活力产生抑制。本研究中通过计算设计的热稳定性提高的点突变大多可以保持90%以上酶活,尤其是M137A和I157L,虽然距离活性中心较近(< 8 ?),但是都可以保持野生型的酶活力。迭代组合突变设计的六点组合突变体酶活丧失过半,将稳定性提高且酶活较高的突变位点组合获得的四点组合突变体,热稳定性与六点突变体相当,且酶活与野生型相当,说明酶的组合突变易受负突变影响,通过计算设计的正向突变之间迭代组合,可以预期获得热稳定性提高同时酶活丧失少或酶活提高的突变体。
通过对计算设计的突变位点进行深入分析发现以下的筛选原则。首先,利用Rosetta-VIP设计的单点突变的ΔΔE值更多地反映了突变体稳定性提高的可能性,而其数值与Tm没有明显的线性相关,选择ΔΔE值< -5.0的突变设计,正突变的比例在80%,且大部分能保持90%以上的酶活力。其次,利用突变位点的溶剂可及表面积变化可对Rosetta-VIP的计算设计结果进行进一步验证,选择ΔSASA < 0的突变位点,突变体的溶剂可及表面积减少,稳定性提高。最后,运用序列保守性分析,从序列进化的角度,选择符合进化保守性的突变残基,来排除Rosetta-VIP中可能存在的不利突变,从而进一步缩小筛选范围。随着突变位点积累的越多,热稳定性越高,将热稳定性提高明显而酶活丧失少的组合突变进一步叠加,可以在提高热稳定性的同时减少酶活损失。
以枯草芽孢杆菌脂肪酶A为模式蛋白进行的定向进化和计算设计研究比较广泛和深入,如表 5所示,研究者利用多轮定向进化手段,虽然筛选库庞大,但是热稳定性提高的效果明显高于大部分计算设计方法,由于酶稳定性不是单独某种作用力或性质所决定的,计算设计在追求筛选数量低的同时,可能导致稳定性提高幅度有限。本研究中通过计算设计大幅度缩小了筛选文库,筛选到了与Rao和Reetz等[5-10]通过易错PCR、饱和突变等手段获得的突变体中的L114P、I157M突变,及F17、M137位点,与Gohlke等和Peng Zhou等[15-16]的计算设计相比更能筛选到定向进化的关键位点,更加准确、高效。但是稳定性提高的空间还较大,可以结合定点饱和突变和柔性及突变组学分析等手段,进一步提高酶的稳定性。
表 5 提高枯草芽孢杆菌脂肪酶A热稳定性的研究Table 5 Research of the thermostability of Bacillus subtilis LipA
| Mutation sites | Strategy | Clones | ΔT50 (℃) | ΔTm (℃) | Relative activity | Reference |
| 6B (A15S/F17S/A20E/N89Y/G111D/L114P/ A132D/I157M/N166Y/M134E/M137P/S163P) | eq-PCR and site-saturation mutagenesis | 18 000 | ∕ | 22.0 | 12-fold | [7] |
| XI (R33G/K112D/Y139C/I157M/M134D) | Iterative saturation mutagenesis | 8 000 | 45.0 | ∕ | No influence | [9] |
| Single-point mutants V54H, F58I, V96S | Rigidity theory | 12 | 6.7 | ∕ | ∕ | [16] |
| Single-point mutants L55Y, A75V, G80V, M134D, S162P | Mutatomics analysis | 5 | ∕ | 16.7 | ∕ | [15] |
| Single-point mutants F17A, V74I, L114P, I135V, M137A, I157L | Rosetta-VIP, SASA, conservation analysis | 16 | 0.8 | 3.2 | 102.5% | This study |
| M10 (F17A/V74I/L114P/I135V/M137A/I157L) | 10 | 8.4 | 7.4 | 45% | ||
| M11 (F17A/L114P/M137A/I157L) | 10 | 5.9 | 7.3 | 99.7% | ||
| / indicates that the data is not mentioned in the corresponding paper; 6B and XI are the most stable compound mutant of Rao group and Reetz group respectively; F58I and L55Y are the most stable single-point mutant of Gohlke group and Peng Zhou group respectively, ΔTm of L55Y was just predicted; M137A is the most stable single-point mutant of this article; All of the ΔT50 and ΔTm is the most stable mutant of each group. | ||||||
表选项
综上所述,本研究利用了一种通过分析蛋白质空腔填充,在突变能量变化分析的基础上,以蛋白溶剂可及表面积和蛋白进化保守性为依据的多重计算设计策略,可以有效地提高酶的热稳定性。
参考文献
| [1] | Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases. Enzy Microb Technol, 2006, 39(2): 235-251. |
| [2] | Liu L, Cao H, Tao YF, et al. Recent advances in industrial applications of lipases and strategies for improving lipase properties. J Biopr Eng Bioref, 2013, 2(2): 117-124. |
| [3] | Modarres HP, Mofrad MR, Sanati-Nezhad A. Protein thermostability engineering. RSC Adv, 2016, 6(116): 115252-115270. DOI:10.1039/C6RA16992A |
| [4] | van Pouderoyen G, Eggert T, Jaeger KE, et al. The crystal structure of Bacillus subtili lipase: a minimal α/β hydrolase fold enzyme. J Mol Biol, 2001, 309(1): 215-226. |
| [5] | Acharya P, Rao NM. Stability studies on a lipase from Bacillus subtilis in guanidinium chloride. J Prot Chem, 2003, 22(1): 51-60. |
| [6] | Acharya P, Rajakumara E, Sankaranarayanan R, et al. Structural basis of selection and thermostability of laboratory evolved Bacillus subtilis lipase. J Mol Biol, 2004, 341(5): 1271-1281. DOI:10.1016/j.jmb.2004.06.059 |
| [7] | Kamal Z, Ahmad S, Molugu TR, et al. In vitro evolved non-aggregating and thermostable lipase: structural and thermodynamic investigation. J Mol Biol, 2011, 413(3): 726-741. |
| [8] | Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of b factors as a strategy for increasing protein thermostability. Angew Chem Int Ed, 2006, 45(46): 7745-7751. DOI:10.1002/anie.200602795 |
| [9] | Reetz MT, Carballeira JD. Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes. Nat Protocols, 2007, 2(4): 891-903. DOI:10.1038/nprot.2007.72 |
| [10] | Augustyniak W, Brzezinska AA, Pijning T, et al. Biophysical characterization of mutants of Bacillus subtilis lipase evolved for thermostability: factors contributing to increased activity retention. Prot Sci, 2012, 21(4): 487-497. DOI:10.1002/pro.2031 |
| [11] | Lehmann M, Wyss M. Engineering proteins for thermostability: the use of sequence alignments versus rational design and directed evolution. Curr Opin Biotechnol, 2001, 12(4): 371-375. DOI:10.1016/S0958-1669(00)00229-9 |
| [12] | Qu G, Zhao J, Zheng P, et al. Recent advances in directed evolution. Chin J Biotech, 2018, 34(1): 1-11 (in Chinese). 曲戈, 赵晶, 郑平, 等. 定向进化技术的最新进展. 生物工程学报, 2018, 34(1): 1-11. |
| [13] | Goldenzweig A, Fleishman SJ. Principles of protein stability and their application in computational design. Ann Rev Biochem, 2018, 87(1): 105-129. DOI:10.1146/annurev-biochem-062917-012102 |
| [14] | Qu G, Zhu T, Jiang YY, et al. Protein engineering: from directed evolution to computational design. Chin J Biotech, 2019, 35(10): 1843-1856 (in Chinese). 曲戈, 朱彤, 蒋迎迎, 等. 蛋白质工程:从定向进化到计算设计. 生物工程学报, 2019, 35(10): 1843-1856. |
| [15] | Tian FF, Yang C, Wang CC, et al. Mutatomics analysis of the systematic thermostability profile of Bacillus subtilis lipase A. J Mol Model, 2014, 20(6): 2257-2265. DOI:10.1007/s00894-014-2257-x |
| [16] | Rathi PC, Fulton A, Jaeger KE, et al. Application of rigidity theory to the thermostabilization of lipase a from Bacillus subtilis. PLoS Comput Biol, 2016, 12(3): e1004754. DOI:10.1371/journal.pcbi.1004754 |
| [17] | Goldenzweig A, Goldsmith M, Hill SE, et al. Automated structure- and sequence-based design of proteins for high bacterial expression and stability. Mol Cell, 2016, 63(2): 337-346. DOI:10.1016/j.molcel.2016.06.012 |
| [18] | Moore EJ, Zorine D, Hansen WA, et al. Enzyme stabilization via computationally guided protein stapling. Proc Natl Acad Sci USA, 2017, 114(47): 12472-12477. DOI:10.1073/pnas.1708907114 |
| [19] | Gribenko AV, Patel MM, Liu JJ, et al. Rational stabilization of enzymes by computational redesign of surface charge-charge interactions. Proc Natl Acad Sci USA, 2009, 106(8): 2601-2606. DOI:10.1073/pnas.0808220106 |
| [20] | Borgo B, Havranek JJ. Automated selection of stabilizing mutations in designed and natural proteins. Proc Natl Acad Sci USA, 2012, 109(5): 1494-1499. DOI:10.1073/pnas.1115172109 |
| [21] | Sheffler W, Baker D. RosettaHoles2: a volumetric packing measure for protein structure refinement and validation. Prot Sci, 2010, 19(10): 1991-1995. DOI:10.1002/pro.458 |
| [22] | Sheffler W, Baker D. RosettaHoles: rapid assessment of protein core packing for structure prediction, refinement, design, and validation. Prot Sci, 2010, 18(1): 229-239. |
| [23] | Hassan FU, Minhas FUAA. Deep and self-taught learning for protein accessible surface area prediction//2017 International Conference on Frontiers of Information Technology (FIT). IEEE, 2017. |
| [24] | Estrada J, Bernad P, Blackledge M, et al. ProtSA: a web application for calculating sequence specific protein solvent accessibilities in the unfolded ensemble. BMC Bioinformatics, 2009, 10: 104. DOI:10.1186/1471-2105-10-104 |
| [25] | Magliery TJ. Protein stability: computation, sequence statistics, and new experimental methods. Curr Opin Struct Biol, 2015, 33: 161-168. DOI:10.1016/j.sbi.2015.09.002 |
| [26] | Motlagh HN, Wrabl JO, Li J, et al. The ensemble nature of allostery. Nature, 2014, 508(7496): 331-339. DOI:10.1038/nature13001 |
| [27] | Cui Q, Karplus M. Allostery and cooperativity revisited. Prot Sci, 2008, 17(2): 1295-1307. |
