
 , 张明2
, 张明2

1. 江西农业大学 农学院,江西 南昌 330045;
2. 江西生物科技职业学院,江西 南昌 330200
收稿日期:2019-12-03;接收日期:2020-05-14;网络出版时间:2020-06-23
基金项目:国家自然科学基金(No. 31860560),江西农业大学博士启动基金(No. 3181)资助
摘要:细胞自噬是植物逆境应答过程中最常见的保护机制之一。动物中,自噬相关基因抵御镉(Cd)毒害的功能研究较清楚,但植物却知之甚少。文中以芹菜品种‘皇后’为试材,采用外源Cd (终浓度为0、2、4、8 mg/L)添加营养液水培处理,利用转录组测序(RNA-seq)技术筛选细胞自噬相关差异基因并进行qRT-PCR验证。结果表明Cd胁迫对芹菜植株产生了明显的毒害作用,并与浓度间产生了量效关系。在筛选的8个差异表达的自噬相关基因中,ATG8a、ATG8f、ATG13、AMPK-1、AMPK-2基因随Cd浓度升高表达上调,ATG12、VPS30和VPS34则先上调后下降,说明自噬相关基因可能通过表达上调增加了自噬小体结构以抵御Cd毒性作用;而高浓度Cd (8 mg/L)可能超出芹菜的耐受范围,导致多个自噬基因又出现表达下调趋势。以上结果有助于后期自噬相关基因的功能研究,为进一步探讨芹菜对Cd胁迫的耐性机制提供参考依据。
关键词:Cd胁迫芹菜转录组自噬相关基因筛选
Effect of Cd on autophagy-related genes of celery
Xufeng Xiao1, Meng Li1, Shucheng Si1, Shuying Fan1, Caijun Wu1

 , Ming Zhang2
, Ming Zhang2

1. College of Agronomy, Jiangxi Agricultural University, Nanchang 330045, Jiangxi, China;
2. Jiangxi Biotech Vocational College, Nanchang 330200, Jiangxi, China
Received: December 3, 2019; Accepted: May 14, 2020; Published: June 23, 2020
Supported by: National Natural Science Foundation of China (No. 31860560), Doctor Start Fund of Jiangxi Agricultural University (No. 3181)
Corresponding author: Caijun Wu. Tel: +86-791-83813185; E-mail: wucj@126.com;
Ming Zhang. Tel: +86-791-85717558; E-mail: 450461848@qq.com.
Abstract: Autophagy is one of the most common protective mechanisms during plant stress response. We studied the effect of exogenous Cd on autophagy in celery, by using transcriptome sequencing technique to analyze the differentially expressed genes under different Cd concentrations (0, 2, 4 and 8 mg/L). Eight differentially expressed autophagy-related genes were screened and identified by qRT-PCR. Cd had obvious toxic effect on celery, in a dose-dependent manner. Eight differentially expressed autophagy-related genes were screened, among which ATG8a, ATG8f, ATG13, AMPK-1 and AMPK-2 were up-regulated, whereas ATG12, VPS30 and VPS34 were first up-regulated and then decreased. The up-regulated expression of differential genes may resist Cd toxicity by increasing autophagosome structures; however, 8 mg/L Cd exceeded the autophagosome tolerance limit of celery, resulting in decreased expression of multiple autophagy-related genes. The above results can provide help for subsequent functional study of autophagy-related genes, and provide a reference for further exploring the tolerance mechanism of celery to Cd toxicity.
Keywords: Cd stresscelery (Apiumgraveolens L.)transcriptomeautophagy-related genescreen
以近年频频报道的“镉(Cd)米”、“镉麦”事件为例,说明当前我国所面临的土壤污染问题不仅牵涉传统环境法领域环境污染与生态破坏问题,而且更直接、更现实地反映出土壤污染导致食品安全事故造成的公众健康危害。目前,国内****对我国主要城郊蔬菜地的重金属污染现状进行调查研究,结果发现Cd污染的问题尤为突出,其点位超标率达到7.0%,位于无机污染物第一位[1-2]。蔬菜Cd污染是食品污染物中较为严重的一类,Cd在蔬菜中富集累积,通过食物链危害人类健康和生命安全,而叶菜较其他种类蔬菜的污染程度更为严重[2-3]。
细胞自噬(简称“自噬”)是植物逆境应答过程中一种最常见的自我保护机制。植物应对外界环境压力时,如营养不足、缺氧、氧化应激、干旱、高盐、重金属及病原体感染等,自噬通路被激活产生细胞自噬体,用于维持细胞稳态与存活[4-6]。迄今为止,哺乳动物、酵母和植物中已鉴定超过36个进化保守的自噬相关基因(Autophagy-related genes,ATGs),分别参与自噬诱导、小泡成核、延伸、成熟及与液泡融合等过程[7-8]。拟南芥中,ATGs缺失会导致营养缺乏、叶片早衰、寿命缩短、细胞代谢组分改变、先天免疫激活和生物、非生物胁迫耐受性受损等[9-14]。
Cd胁迫属于非生物胁迫。动物中,自噬相关基因抵御Cd毒害的功能研究比较清楚[15-16]。Cd处理哺乳动物细胞后,可引起自噬标志物LC3-Ⅱ (Microtubule-associated protein 1 light chain 3)表达量增加,自噬溶酶体形成及ATGs上调,诱导细胞发生自噬。然而,大量数据证明动物细胞产生自噬具有两面性:一方面维持细胞稳态,以实现细胞存活;另一方面过量持续的自噬会破坏细胞器和重要的蛋白质,导致自噬性细胞死亡,而该过程受到ATGs严密调控[17-20]。目前,植物中是否具有动物类似的调控机制仍不清楚。本研究以重金属Cd超富集蔬菜之一——芹菜(Apiumgraveolens L.)[21]为试材,采用转录组测序(RNA-seq)方法筛选Cd胁迫下芹菜自噬相关基因,并通过后续qRT-PCR实验验证自噬相关基因功能,旨在进一步探讨芹菜对Cd胁迫的耐性机制。
1 材料与方法1.1 材料培养及试验设计供试材料为芹菜品种‘皇后’,购于法国Tezier公司。试验在江西农业大学园艺实验室完成,2017年4月下旬播种于营养钵,待幼苗长至5–6 cm时,挑选长势一致的植株移栽于装有1 L霍格兰氏营养液的塑料桶中,pH 6.0,温度25 ℃,光照强度为200 μmol/(s·m2),每桶6株。预培养2周后,外源添加CdCl2·2.5H2O至Cd2+处理终浓度,参照马兰等[22]报道,分别为2、4、8 mg/L,以不添加重金属为对照组(Control check,CK),重复3次,32 d后收获植株。
1.2 cDNA文库构建、库检及测序分析采用Trizol Plant Extraction Kit (TaKaRa,日本)提取芹菜根系总RNA,1.5%琼脂糖凝胶电泳和核酸蛋白仪检测总RNA纯度、浓度及完整性。
将带有Oligo (dT)的磁珠富集mRNA;加入破碎缓冲液(Fragmentation buffer)将mRNA进行随机打断;以mRNA为模板用六碱基随机引物(Random hexamers)合成第一条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA聚合酶Ⅰ合成第二条cDNA链,利用AMPure XP磁珠纯化cDNA;纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后用AMPure XP磁珠进行片段大小选择;最后通过PCR富集得到cDNA文库[23]。文库检测合格后,采用Illumina HiSeq2500平台进行高通量测序,测序读长为PE100。获得的Raw data经数据过滤,将其中接头序列及低质量序列去除,得到高质量序列数据(Clean data)后,再使用Trinity软件组装[24]。
采用DESeq[25]进行差异表达分析,用FDR (False discovery rate)作为差异表达基因筛选的指标。在筛选过程中,将FDR < 0.01且FC (Fold change) ≥2作为筛选标准。对差异表达基因进行GO (Gene ontology)注释,并用DAVIDv6.8[26]注释工具进一步对差异表达基因分析[23]。同时将差异表达基因在KEGG数据库(Kyoto encyclopedia of gene and genomes)中进行注释,其显著性富集分析的P-value值计算和阈值同GO富集分析相同。
1.3 自噬相关基因筛选及荧光定量PCR (qRT-PCR)验证采用DESeq方法筛选自噬相关基因,qRT-PCR技术采用TransStart Top Green qPCR SuperMix (Transgen生物,中国),内参基因为β-actin。反应所用引物见表 1,由生工生物工程(上海)股份有限公司合成。
表 1 试验引物及序列Table 1 Sequence of primers used in qRT-PCR
| Genotype | Gene ID | Gene symbol | Upstream primer sequence (5′–3′) | Downstream primer sequence (5′–3′) |
| Differential genes | c131646 | ATG8a | CCATCTTCATCCTTCTTCTCGTC | TATCTTGTCCCATCCGATTTGA |
| c140253 | ATG12 | CATCCAATCAACTTGCCACTCC | TTGATGAAAGTTAGCCCCATGC | |
| c143897 | ATG13 | TATAATGCACCAACAAGGTGGC | ACGCCAACTGTTTGGTCTTATG | |
| c144576 | ATG8f | GACCTTACTGTGGGGCAGTTTG | GACATTATCGCTCCTGTGGGTG | |
| c146704 | VPS30 | AATGATTCCTCCATTGCCTTCC | TTCGTTCTGGGATTCAGGGTTC | |
| c149168 | AMPK-1 | GCAGTCCCAACTATGTTGCTCC | CCATTTTGGGATTTGAGCTTCA | |
| c149570 | VPS34 | CCAACATCCCTGACATAGCATC | ACCATTTGAGGAAACAATGCAC | |
| c156499 | AMPK-2 | ATTTACATCTCGCCATTCTCCA | TGAAAGATTAACTGGGCTGCTG | |
| Intrinsic genes | c95536 | β-actin-1 | CTTCTATGTCATTGGCACCGAC | ATTCCGAGAAGGCTGTCATACC |
| c140911 | β-actin-2 | TATTTTGCCTTTGCCTTGCTTC | AGCTTGTCATCTGCCTCCTTGA |
表选项
PCR反应的体系为:2×TransStart Top Green qPCR SuperMix 10 μL,模板0.4 μL,上、下游引物各0.4 μL,加灭菌蒸馏水至200 μL;PCR反应条件为:94 ℃ 2 min;94 ℃ 5 s,58 ℃ 15 s,72 ℃ 10 s,45个循环;72 ℃延伸7 min。
1.4 数据分析在Excel工作表中采用2–ΔΔCt法分析ATGs基因相对表达水平[27]。每个样品设置3个生物学重复,并计算重复数据的标准差。
2 结果与分析2.1 Cd胁迫对芹菜植株生长的影响本研究首先对试验材料进行了表型观察、记录与分析,随Cd浓度升高,芹菜幼苗表现为褪绿萎缩、植株生长缓慢、须根数量减少、产量下降等症状(图 1),说明芹菜植株生长受到明显抑制。此外,2、4、8 mg/L处理组的茎叶鲜重、根鲜重较对照组分别降低了15.29%、42.38%、66.49%和19.42%、50.49%、58.98%,且处理组间差异显著(P < 0.05),说明外源Cd添加产生了明显的负面效应,高浓度Cd加重对植株生长的抑制效果,且与浓度间产生了量效关系(表 2)。
 |
| 图 1 不同浓度Cd胁迫下芹菜植株的形态 Fig. 1 Plant morphology of celery under different Cd concentrations. |
| 图选项 |
表 2 不同浓度Cd胁迫对芹菜幼苗叶茎和根鲜重的影响Table 2 Leaf, stem and root fresh weight of celery under different Cd concentration
| Treatment | Leaf and stem freshweight (g) | Root fresh weight (g) |
| CK | 31.45±2.43a | 4.12±0.25a |
| 2 mg/L | 26.64±1.53b | 3.32±0.12b |
| 4 mg/L | 18.12±1.94b | 2.04±0.13bc |
| 8 mg/L | 10.54±0.41c | 1.69±0.08c |
| Note: the same letters indicate that the difference is not significant at the 5% level and different low-case letters indicate the difference between different treatments at the 5% level. | ||
表选项
2.2 转录本数据质量评估通过对照样品与2、4、8 mg/L处理组样品的转录组测序,共获得83.78 Gb原始数据,各样品的数据量均达到6.21 Gb,GC含量平均值为43.09%,Q30碱基百分比在91.43%以上(表 3),说明转录组测序质量较高。此外,原始读取(Raw reads)的平均数分别为23 905 679、22 971 867、22 306 185、24 666 755。经过Denovo组装后共获得64 914个Unigene,Unigene的N50为2 005个,组装完整性较高,其中长度在1 kb以上的Unigene有24 375个。将各样品过滤后的高质量数据(Clean reads)与组装得到的Unigene库进行序列比对,对照组与2、4、8 mg/L样品中可用于后续分析的比对效率(Mapped ratio)分别占80.26%、80.23%、80.14%和79.32% (表 3)。
表 3 转录组测序数据统计Table 3 RNA-seq data statistics
| ??Cd Concentration | Raw reads | Clean reads | Mapped reads | GC content (%) | Q30 (%) | Mapped ratio (%) |
| ??CK-1 | 25 087 416 | 24 964 488 | 20 215 107 | 43.25 | 91.75 | 80.97 |
| ??CK-2 | 23 631 152 | 23 243 879 | 18 472 467 | 42.93 | 92.53 | 79.47 |
| ??CK-3 | 22 998 469 | 22 888 076 | 18 388 368 | 43.46 | 91.43 | 80.34 |
| ??2 mg/L-1 | 20 766 040 | 20 664 286 | 16 777 406 | 42.75 | 91.82 | 81.19 |
| ??2 mg/L-2 | 24 076 490 | 23 956 110 | 18 841 475 | 43.63 | 92.18 | 78.65 |
| ??2 mg/L-3 | 24 073 071 | 23 957 520 | 19 366 089 | 42.88 | 92.29 | 80.84 |
| ??4 mg/L-1 | 21 164 182 | 21 054 128 | 16 913 491 | 43.03 | 92.49 | 80.33 |
| ??4 mg/L-2 | 21 952 005 | 21 842 751 | 17 330 245 | 43.01 | 92.05 | 79.34 |
| ??4 mg/L-3 | 23 802 368 | 23 707 159 | 19 141 449 | 43.03 | 92.28 | 80.74 |
| ??8 mg/L-1 | 21 577 734 | 21 489 265 | 17 260 274 | 43.09 | 92.17 | 80.32 |
| ??8 mg/L-2 | 28 539 196 | 28 322 893 | 22 027 299 | 43.17 | 92.66 | 77.77 |
| ??8 mg/L-3 | 23 883 336 | 23 766 307 | 18 980 984 | 42.83 | 91.87 | 79.87 |
表选项
对Unigene进行功能注释,包括与NR、Swiss-Prot、GO、COG、KOG、eggNOG及KEGG数据库的比对,共获得40 400条Unigene的注释结果(表 3)。其中,分别比对到NR库的基因数是37 438,注释率为92.67%;Swiss-Prot库的基因数是26 328,注释率为65.17%;GO库的基因数是20 966,注释率为51.9%;COG库的基因数是15 869,注释率为39.28%;KOG库的基因数是25 238,注释率为62.47%;eggNOG库的基因数是36 753,注释率为90.97%;KEGG库的基因数是17 848,注释率为44.18%。使用KOBAS2.0[28]得到Unigene在KEGG中的KEGG Orthology结果,预测完Unigene的氨基酸序列之后使用HMMER软件[29]与Pfam数据库[30]比对,结果表明Pfam库的基因数是31 687,注释率达到78.43% (图 2)。以上结果说明测序序列组装效果较理想,可用于下一步基因筛选。
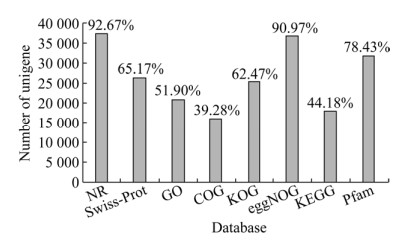 |
| 图 2 Cd胁迫下芹菜序列在7个数据库中的注释情况 Fig. 2 The annotation of Cd stress on celery Unigenes among 7 databases. |
| 图选项 |
2.3 差异表达基因筛选及分析采用DESeq方法筛选,对照组与Cd处理间以及不同浓度Cd处理组两两之间共获得7 253个差异基因,其中上调4 698个,下调2 555个。其中,对照组与Cd处理组间的差异表达基因数量较大,且上调基因数超过下调,说明Cd胁迫对芹菜基因转录调控产生了影响;而处理组间差异基因的数量明显减少,尤其是4 mg/L与8 mg/L组间,推测4 mg/L可能是芹菜抵御Cd毒害的浓度承受拐点(图 3A)。
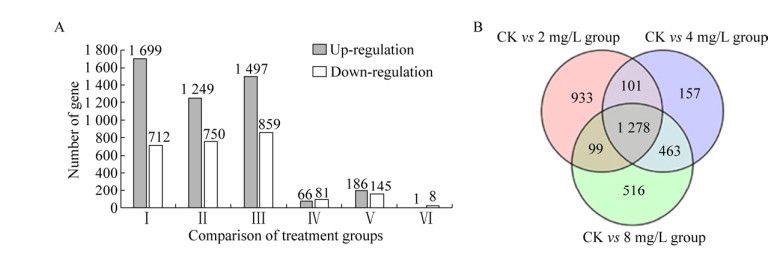 |
| 图 3 对照组与Cd处理组及Cd处理组之间的差异基因数统计 Fig. 3 The number of differential genes between CK and Cd treatment groups, Cd treatment groups. (A) The number of up-regulated and down-regulated genes among treatment groups, Ⅰ, Ⅱ, Ⅲ, Ⅳ and Ⅴ presented CK vs 2 mg/L, CK vs 4 mg/L, CK vs 8 mg/L, 2 mg/L vs 4 mg/L, 2 mg/L vs 8 mg/L and 4 mg/L vs 8 mg/L, respectively. (B) Venn diagram between CK and Cd treatment groups. |
| 图选项 |
从维恩图可以看出,对照组和2 mg/L组间有2 411个基因差异表达,对照组和4 mg/L组间有1 999个,对照组和8 mg/L组间有2 356个基因差异表达(筛选条件|log2.Fold_change.| > 1且q-value < 0.005)。对照组与Cd处理组共同的差异表达基因为1 278个(图 3B)。
应用DAVID注释工具对96个筛选的共同组合差异表达基因进行GO功能聚类分析,共分为3大类19组,其中生物过程占42.1%,以细胞过程及新陈代谢过程注释最多;细胞组分占36.8%,以细胞和细胞成分注释最多;分子功能占21.1%,以结合和催化活性注释最多(图 4)。
 |
| 图 4 差异表达基因分析和GO注释 Fig. 4 Analysis and GO annotation of differentially expressed genes. |
| 图选项 |
通过KEGG富集分析,对照组与2 mg/L处理组间有662个差异Unigene可分类至50个代谢通路;对照组与4 mg/L处理组间有554个差异Unigene可分类至50个代谢通路;对照组与8 mg/L处理组间有683个差异Unigene可分类至50个代谢通路;2 mg/L与4 mg/L处理组间有47个差异Unigene可分类至25个代谢通路;2 mg/L与8 mg/L处理组间有114个差异Unigene可分类至38个代谢通路;4 mg/L与8 mg/L处理组间有11个差异Unigene可分类至5个代谢通路。其中,注释数量从高到低依次为核糖体(Ribosome,136个)、内质网蛋白加工(Protein processing in endoplasmic reticulum,63个)、碳代谢(Carbon metabolism,45个)、植物激素信号转导(Plant hormone signal transduction,42个)、苯丙醇生物合成(Phenylpropanoid biosynthesis,39个)、植物病原相互作用(Plant-pathogen interaction,33个)、氨基酸生物合成(Biosynthesis of amino acids,32个)等。
2.4 自噬相关基因的筛选及qRT-PCR验证为进一步确证转录组测序质量,筛选了8个与自噬形成密切相关的差异基因进行qRT-PCR验证分析。其中,ATG12 (c140253)、VPS30 (c146704)、VPS34 (c149570)先上调后下降,而ATG8a (c131646)、ATG8f (c144576)、ATG13 (c143897)、AMPK-1 (c149168)和AMPK-2(c156499)均为上调基因。从图 5可知,qRT-PCR验证结果与转录组测序结果相一致,说明转录组测序结果可靠,可用于下一步功能分析。
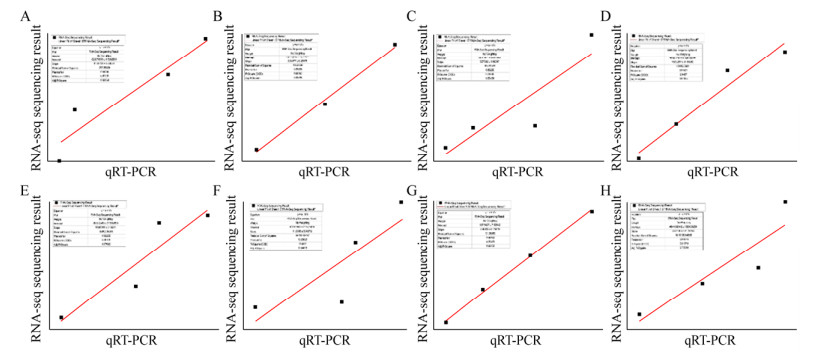 |
| 图 5 自噬相关差异基因的表达量相关性分析 Fig. 5 Analysis on expression abundance of the autophagy-related genes. A, B, C, D, E, F, G and H presented ATG8a, ATG12, ATG13, ATG8f, VPS30, AMPK-1, VPS34 and AMPK-2, respectively. |
| 图选项 |
3 讨论近几年,植物的自噬研究十分活跃,从模式植物拟南芥到藻类、裸子植物和被子植物,包括单子叶植物和双子叶植物[31]。自噬广泛存在于真核细胞中,正常情况下细胞内自噬维持在较低水平,当遭遇逆境胁迫时会被大量诱导,这在酵母、动物和植物中已被证实[2, 32]。哺乳动物中,Cd诱导的自噬可作为一种细胞存活机制保护细胞,也可作为一种死亡机制导致细胞死亡,该过程受自噬相关基因调控[33-34],植物中是否具有类似调控机制,目前尚不清楚。
植物自噬涉及了众多生命活动,早期研究发现植物细胞在营养缺乏条件下能够激活自噬,降解细胞内物质补充给饥饿的细胞,以维持细胞稳态;而后在植物生长发育、免疫应答、叶片衰老及环境胁迫应答等过程中也发现了细胞自噬[35-39]。拟南芥中,已鉴定超过36个进化保守的ATGs分别参与自噬诱导、小泡成核、延伸、成熟及与液泡融合等过程[18],而非生物胁迫是诱导细胞自噬的重要因素之一[2]。本研究中,芹菜植株在Cd胁迫下生长缓慢,须根减少,而且与Cd浓度有明显的量效关系,说明Cd胁迫能阻碍芹菜生长,该结果和赵慧等[40]的研究结果一致。另外,筛选的8个自噬相关基因中,ATG8a、ATG8f、ATG13、AMPK-1和AMPK-2表达一直上调,而ATG12、VPS30、VPS34三个基因则是先上调后下降。其中,ATG13可与ATG1互作共同启动自噬过程,ATG13表达上调恰恰说明了自噬体可能随Cd浓度上升数量不断扩大。陈立德等[41]研究黑暗、高盐、涝害、高温和干旱胁迫处理下,草莓叶片的自噬相关基因表达呈总体上升趋势。Zhou等[42]采用Cu2+、Ni2+、Zn2+、Cd2+和Mn2+处理烟草幼苗,发现18个ATGs对不同重金属处理有共同反应机制,其中11个ATGs表达水平普遍上调。以上结果支持了本研究的观点,但文献未报道有ATGs先上调后下降的现象,这可能是芹菜的特殊性。然而,Cd胁迫下ATG13上调可能引起的自噬体增加究竟对细胞起保护功能还是毒害功能仍然未知,但从4 mg/L与8 mg/L处理组间的差异表达明显减少推测,4 mg/L可能是芹菜抵御Cd毒性的浓度拐点。
另外,ATG8是自噬体形成过程中不可或缺的蛋白,也是自噬的特异性标志蛋白。研究表明,ATG8与磷脂酰乙醇胺形成的复合体(ATG8-PE)伴随着自噬整个过程,决定了自噬体大小。本研究中,ATG8a和ATG8f是芹菜ATG8两个不同的拷贝。随Cd浓度上升,ATG8a和ATG8f上调表达可能增加了ATG8-PE复合体的数量,促进营养物质循环利用,以此改善芹菜应对Cd胁迫的能力来维持植株的正常生长。这与Kaufmann等[43]发现ATG8转录水平在细胞遭遇饥饿胁迫时上调;赵慧等[40]报道在拟南芥中过表达ATG8f基因,转基因植株对Cd胁迫的敏感性增强了,与李微微[44]发现AGT8a可显著提高水稻、拟南芥对低氮胁迫耐性的研究结果十分相似,均是植物应对逆境胁迫时开启高效利用营养物质的一种自我保护机制。另外,AMPK是细胞能量感受器[45],因此AMPK-1和AMPK-2表达水平上调同样也可能是芹菜感受细胞能量和营养状态改变、参与维持细胞能量和营养稳态的表现。
再有,自噬相关蛋白ATG12由ATG12编码,与ATG5结合形成的复合物影响自噬的形成过程[46];而VPS30与VPS34、VPS15等蛋白互作,VPS34又参与自噬起始,VPS34基因一旦缺失将引起细胞自噬的中断[47]。本研究中,ATG12、VPS30和VPS34基因先上调后下降表达,可能与自噬激活有关,也可能是芹菜植株抵御Cd毒性的应激反应所致。8 mg/L Cd2+处理浓度可能超过了芹菜植株耐受范围,致使ATG基因(ATG12、VPS30和VPS34)的mRNA水平降低,这说明ATG在自噬和凋亡之间可能有转换作用,目的是为了确保有效利用有限的能量来维持细胞毒性下的体内平衡,Kang等[33]的研究结果支持了本研究的观点。
4 结论随Cd浓度上升,芹菜出现生长缓慢、叶色褪绿、产量下降等症状,并伴随多个自噬相关基因表达上调。自噬基因在自噬发生过程中发挥重要的调控功能,通过自噬体启动、合成及结构累积等改善芹菜应对Cd胁迫能力。而8 mg/L浓度Cd2+可能超出芹菜耐受极限,致使ATG12、VPS30和VPS34表达呈现下降趋势。
参考文献
| [1] | Ministry of Environmental Protection, Ministry of Land and Resources. Bulletin of the national soil pollution survey. China Environ Prot Ind, 2014, 36(5): 10-11 (in Chinese). 环境保护部, 国土资源部. 全国土壤污染状况调查公报. 中国环保产业, 2014, 36(5): 10-11. |
| [2] | Xiao XF, Li M, Long JM, et al. Relationship of active oxygen, antioxidant enzyme activity and autophagy under Cd stress in pakchoi. J Jiangxi Agric Univ, 2019, 41(5): 873-880 (in Chinese). 肖旭峰, 李猛, 龙俊敏, 等. 镉诱导小白菜活性氧及抗氧化酶活性与自噬关系的分析. 江西农业大学学报, 2019, 41(5): 873-880. |
| [3] | Zhou H, Zhang ZZ. Agricultural pollution situation, source and countermeasures of Chinese vegetable. J Agricul Catastrophol, 2013, 3(5): 27-28, 50 (in Chinese). 周辉, 张志转. 中国蔬菜农业污染现状、污染来源及污染防控. 农业灾害研究, 2013, 3(5): 27-38, 50. |
| [4] | Bassham DC, Laporte M, Marty F, et al. Autophagy in development and stress responses of plants. Autophagy, 2006, 2: 2-11. DOI:10.4161/auto.2092 |
| [5] | Han S, Yu B, Wang Y, et al. Role of plant autophagy in stress response. Protein Cell, 2011, 2: 784-791. DOI:10.1007/s13238-011-1104-4 |
| [6] | Kroemer G, Mari?o G, Levine B. Autophagy and the integrated stress response. Mol Cell, 2010, 40: 280-293. DOI:10.1016/j.molcel.2010.09.023 |
| [7] | Hofius D, Li L, Hafrén A, et al. Autophagy as an emerging arena for plant-pathogen interactions. Curr Opin Plant Biol, 2017, 38: 117-123. |
| [8] | Díaz-Troya S, Pérez-Pérez ME, Pérez-Martín M, et al. Inhibition of protein synthesis by TOR inactivation revealed a conserved regulatory mechanism of the BiP chaperone in Chlamydomonas. Plant Physiol, 2011, 157: 730-741. DOI:10.1104/pp.111.179861 |
| [9] | Thompson AR, Doelling JH, Suttangkakul A, et al. Autophagic nutrient recycling in Arabidopsis directed by the ATG8 and ATG12 conjugation pathways. Plant Physiol, 2005, 138: 2097-2110. DOI:10.1104/pp.105.060673 |
| [10] | Minina EA, Sanchez-Vera V, Moschou PN, et al. Autophagy mediates caloric restriction-induced lifespan extension in Arabidopsis. Aging Cell, 2013, 12: 327-329. DOI:10.1111/acel.12048 |
| [11] | Chung T, Phillips AR, Vierstra RD. ATG8 lipidation and ATG8-mediated autophagy in Arabidopsis requireATG12 expressed from the differentially controlled ATG12A AND ATG12B loci. Plant J, 2010, 62: 483-493. DOI:10.1111/j.1365-313X.2010.04166.x |
| [12] | Yoshimoto K, Hanaoka H, Sato S, et al. Processing of ATG8s ubiquitin-like proteins and their deconjugation by ATG4s are essential for plant autophagy. Plant Cell, 2004, 16: 2967-2983. DOI:10.1105/tpc.104.025395 |
| [13] | Yang Z, Klionsky DJ. Mammalian autophagy: core molecular machinery and signaling regulation. Curr Opin Cell Biol, 2010, 22: 124-131. DOI:10.1016/j.ceb.2009.11.014 |
| [14] | Hanaoka H, Noda T, Shirano Y, et al. Leaf senescence and starvation-induced chlorosis are accelerated by the disruption of an Arabidopsis autophagy gene. Plant Physiol, 2002, 129: 1181-1193. DOI:10.1104/pp.011024 |
| [15] | Lv W, Sui L, Yan X, et al. ROS-dependent Atg4 upregulation mediated autophagy plays an important role in Cd-induced proliferation and invasion in A549 cells. Chem-Biol Interact, 2018, 279: 136-144. DOI:10.1016/j.cbi.2017.11.013 |
| [16] | Comincini S, Manai F, Meazza C, et al. Identification of autophagy-related genes and their regulatory miRNAs associated with celiac disease in children. Int J Mol Sci, 2017, 18(2): pii E391. DOI:10.3390/ijms18020391 |
| [17] | Liu Y, Bassham DC. Autophagy: pathways for selfeating in plant cells. Annu Rev Plant Biol, 2012, 63: 215-237. DOI:10.1146/annurev-arplant-042811-105441 |
| [18] | Maiuri MC, Zalckvar E, Kimchi A, et al. Self-eating and self-killing: crosstalk between autophagy and apoptosis. Nat Rev Mol Cell Biol, 2007, 8(9): 741-752. DOI:10.1038/nrm2239 |
| [19] | Lum JJ, Bauer DE, Kong M, et al. Growth factor regulation of autophagy and cell survival in the absence of apoptosis. Cell, 2005, 120(2): 237-248. DOI:10.1016/j.cell.2004.11.046 |
| [20] | Wang QW. The role and regulation mechanism of autophagy in rat's neurons injured by cadmium[D]. Yangzhou: Yangzhou University, 2014 (in Chinese). 王棋文.自噬在镉致大鼠神经细胞毒性中的作用及调控机制[D].扬州: 扬州大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-11117-1017113760.htm |
| [21] | Zhao YL. Evaluation and enrichment of lead and cadmium pollution in soil vegetables. Vegetables, 2018, 12: 60-64 (in Chinese). 赵亚玲. 土壤蔬菜中铅镉污染评价与富集特征研究. 蔬菜, 2018, 12: 60-64. |
| [22] | Ma L, Zhang BL, Zhang H, et al. Effection of Cd and Zn complex-pollution on nutrient absorption of celery. J Yanshan Univ, 2008, 32(6): 530-534 (in Chinese). 马兰, 张宝莉, 张华, 等. 镉锌复合污染对芹菜养分吸收的影响. 燕山大学学报, 2008, 32(6): 530-534. DOI:10.3969/j.issn.1007-791X.2008.06.013 |
| [23] | Su ZZ, Wang XH, Yang H, et al. Transcriptome analysis of cadmium exposed Jatropha curcas. China Biotechnol, 2016, 36(4): 69-77 (in Chinese). 苏稚喆, 王雪华, 杨华, 等. 镉胁迫下麻疯树转录组测序分析. 中国生物工程杂志, 2016, 36(4): 69-77. |
| [24] | Cui J. Response of Megacoptacribraria to temperature on physiological and biochemical mechanisms[D]. Changchun: Jilin Agricultural University, 2019 (in Chinese). 崔娟.筛豆龟蝽对温度变化的响应及其生理生化机制[D].长春: 吉林农业大学, 2019. http://kns.cnki.net/KCMS/detail/detail.aspx?dbcode=CDFD&filename=1019926028.nh |
| [25] | Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol, 2011, 29(7): 644-652. DOI:10.1038/nbt.1883 |
| [26] | Finn RD, Bateman A, Clements J, et al. Pfam: the protein families database. Nucleic Acids Res, 2014, 27: 222-230. |
| [27] | Yi XJ. Cloning and expression analysis of genesinvolved in Dendrobium flavonoidsbiosynthesis based on RNA-seq[D]. Shanghai: Shanghai Normal University, 2016 (in Chinese). 伊秀娟.基于转录组测序的石斛黄酮生物合成相关基因的克隆与表达分析[D].上海: 上海师范大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10270-1016133668.htm |
| [28] | Xie C, Mao C, Huang J, et al. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases. Nucleic Acids Res, 2011, 39: 316-322. DOI:10.1093/nar/gkr483 |
| [29] | Eddy SR. Profile hidden Markov models. Bioinformatics, 1998, 14(9): 755-763. DOI:10.1093/bioinformatics/14.9.755 |
| [30] | Yang X, Zhu S, Li L, et al. Identification of differentially expressed genes and signaling pathways in ovarian cancer by integrated bioinformatics analysis. Onco Targets Ther, 2018, 11: 1457-1474. DOI:10.2147/OTT.S152238 |
| [31] | Liu Y, Zhang J, Wang QL, et al. Research progress in plant autophagy. Chin Bull Botany, 2018, 53(1): 5-16 (in Chinese). 刘洋, 张静, 王秋玲, 等. 植物细胞自噬研究进展. 植物学报, 2018, 53(1): 5-16. |
| [32] | Zhu J, Tan XR. Reserach advances in ROS and autophagy. Chin Bull Life Sci, 2011, 23(10): 987-991 (in Chinese). 朱京, 谭晓荣. 活性氧与自噬的研究进展. 生命科学, 2011, 23(10): 987-991. |
| [33] | Kang HM, Lee JS, Kim MS, et al. Genome-wide identification of 99 autophagy-related (Atg) genes in the monogonont rotifer Brachionus spp. and transcriptional modulation in response to cadmium. Aquatic Toxicol, 2018, 201: 73-82. DOI:10.1016/j.aquatox.2018.05.021 |
| [34] | Wang Y, Liu YL. Progress in plant autophagy. Chin J Cell Biol, 2010, 32(5): 677-689 (in Chinese). 王燕, 刘玉乐. 植物细胞自噬研究进展. 中国细胞生物学学报, 2010, 32(5): 677-689. |
| [35] | Chen L, Liao B, Qi H, et al. Autophagy contributes to regulation of the hypoxia response during submergence in Arabidopsis thaliana. Autophagy, 2015, 11: 2233-2246. DOI:10.1080/15548627.2015.1112483 |
| [36] | Avin-Wittenberg T, Bajdzienko K, Wittenberg G, et al. Global analysis of the role of autophagy in cellular metabolism and energy homeostasis in Arabidopsis seedlings under carbon starvation. Plant Cell, 2015, 27: 306-322. DOI:10.1105/tpc.114.134205 |
| [37] | Phillips AR, Suttangkakul A, Vierstra RD. The ATG12-conjugating enzyme ATG10 is essential forautophagic vesicle formation in Arabidopsis thaliana. Genetics, 2008, 178(3): 1339-1353. DOI:10.1534/genetics.107.086199 |
| [38] | Wang T. The role and regulation mechanism of autophagy in neuronal senescence induced by cadmium[D]. Yangzhou: Yangzhou University, 2017 (in Chinese). 王涛.自噬在镉致大鼠神经细胞衰老中的作用及调控机制[D].扬州: 扬州大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-11117-1017113760.htm |
| [39] | Li F, Chung T, Vierstra RD. AUTOPHAGY- RELATED11 plays a critical role in general autophagy- and senescence-induced mitophagy in Arabidopsis. Plant Cell, 2014, 26: 788-807. DOI:10.1105/tpc.113.120014 |
| [40] | Zhao H, Zhao YB, Xin CH, et al. Impacts on cadmium stress susceptibility in plants overexpressing autophagy-related gene ATG8f. Molecular Plant Breeding, 2017, 15(8): 3042-3045 (in Chinese). 赵慧, 赵一博, 辛翠花, 等. 过表达细胞自噬基因ATG8f对植物响应镉胁迫的影响. 分子植物育种, 2017, 15(8): 3042-3045. |
| [41] | Chen LD, Li A, Liu ZJ, et al. Effects of different environmental stresses on the number of autophagosomes and expression of autophagy-related genes in 'Ningyu' strawberry leaves. Acta Horticul Sin, 2017, 44(10): 1894-1904 (in Chinese). 陈立德, 李傲, 刘众杰, 等. 逆境胁迫对'宁玉'草莓叶片自噬小体数量及其相关基因表达的影响. 园艺学报, 2017, 44(10): 1894-1904. |
| [42] | Zhou Z, Liu H, Wang C, et al. Long non-coding RNAs as novel expression signatures modulate DNA damage and repair in cadmium toxicology. Sci Rep, 2015, 5: 15293-15305. DOI:10.1038/srep15293 |
| [43] | Kaufmann A, Wollert T. Scaffolding the expansion of autophagosomes. Autophagy, 2014, 10(7): 1343-1345. DOI:10.4161/auto.28980 |
| [44] | Li WW. Funtional analysis of foxtail millet autophagy associated gene SiATG8a in regulated plant response to low nitrogen stress[D]. Harbin: Harbin Normal University, 2017 (in Chinese). 李微微.谷子自噬相关基因SiATG8a调控植物低氮胁迫响应的功能分析[D].哈尔滨: 哈尔滨师范大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10231-1018012148.htm |
| [45] | Hao YH. Research on effects of AMPK/mTOR signal-regulated autophagy on synaptic plasticity damage induced by microwave exposure[D]. Beijing: Academy of Military Science, 2018 (in Chinese). 郝延辉. AMPK/mTOR通路对自噬的调控在微波辐射致突触可塑性损伤中的作用研究[D].北京: 军事科学院, 2018. |
| [46] | Tan DM. Study on the clinical implications and regulation mechanism of autophagy-related protein 12 in cervical lesions[D]. Jinan: Shandong University, 2018 (in Chinese). 谭冬梅.自噬相关蛋白ATG12在宫颈病变中的临床意义及其调控机制研究[D].济南: 山东大学, 2018. http://kns.cnki.net/KCMS/detail/detail.aspx?dbcode=CDFD&dbname=CDFD&filename=1019008633.nh |
| [47] | Liu YL, Li YH, Zhang YN, et al. Construction of VPS34-knockout A549/293T cell line by CRISPR/Cas9 system. Chin J Cell Biol, 2018, 40(10): 1719-1726 (in Chinese). 刘玉兰, 李娅慧, 张宜娜, 等. 利用CRISPR/Cas9技术构建VPS34基因敲除的A549/293T细胞系. 中国细胞生物学学报, 2018, 40(10): 1719-1726. DOI:10.11844/cjcb.2018.10.0101 |
