李豫1,2#, 杨小鹃1#, 张菊梅1, 王涓2, 吴清平1


1. 广东省微生物研究所, 华南应用微生物国家重点实验室, 广东省菌种保藏与应用重点实验室, 广东 广州 510070;
2. 华南农业大学食品学院, 广东 广州 510642
收稿日期:2021-03-26;修回日期:2021-05-11;网络出版日期:2021-10-18
基金项目:国家重点研发专项(2017YFC1601200);广东省“珠江人才计划”本土创新团队(2017BT01S174);广东省科学院实施创新驱动发展能力专项(2018GDASCX-0102)
*通信作者:吴清平, Tel/Fax: +86-20-87688132; E-mail: wuqp203@163.com.
#共同第一作者。
摘要:沙门菌(Salmonella spp.)是公共卫生学上具有重要意义的人畜共患病病原菌。人、畜感染沙门菌后会引起伤寒、副伤寒、胃肠炎、败血症和肠外局灶性感染等疾病。抗生素是治疗沙门菌严重感染的有效手段,随着临床和畜牧业中抗生素的大量使用,使得沙门菌的耐药情况日益严重。整合子是普遍存在于细菌中的一种可移动基因元件,可有效捕获外源基因确保其表达,并复合于转座子、质粒等,使多种耐药基因在细菌种内或者种间进行传播。在过去的二十年中,随着新基因盒和复杂整合子的不断出现,导致整合子系统迅速进化。整合子在沙门菌耐药性传播过程中具有非常重要的作用,因此,本文对整合子系统的分子结构、分类、作用机制,以及沙门菌中存在的Ⅰ、Ⅱ、Ⅲ类整合子介导的耐药性及现有检测方法的研究进展进行综述,以期为沙门菌耐药性研究提供参考。
关键词:沙门菌整合子耐药性耐药基因
Research progress of integron-mediated antimicrobial resistance of Salmonella
Yu Li1,2#, Xiaojuan Yang1#, Jumei Zhang1, Juan Wang2, Qingping Wu1


1. Guangdong Provincial Key Laboratory of Microbial Safety and Health, State Key Laboratory of Applied Microbiology Southern China, Institute of Microbiology, Guangdong Academy of Sciences, Guangzhou 510070, Guangdong Province, China;
2. College of Food Science, South China Agricultural University, Guangzhou 510642, Guangdong Province, China
Received: 26 March 2021; Revised: 11 May 2021; Published online: 18 October 2021
*Corresponding author: Qingping Wu, Tel/Fax: +86-20-87688132; E-mail: wuqp203@163.com.
Foundation item: Supported by the National Key R & D Program of China (2017YFC1601200), by the Local Innovative and Research Teams Project of Guangdong PEARL River Talents Program (2017BT01S174) and by the GDAS' Special Project of Science and Technology Development (2018GDASCX-0102)
#Those authors contributed equally to this work.
Abstract: Salmonella are a group of zoonotic pathogens threatening the public health. The invasion of Salmonella into humans and livestock causes typhoid and paratyphoid fever, gastroenteritis, septicaemia, and extraintestinal focal infections. Antimicrobials are an effective treatment for serious Salmonella infections. However, the extensive use of antimicrobials in clinical practice and animal husbandry has led to increasing antimicrobial resistance among Salmonella. Integrons, mobile genetic elements ubiquitous in bacteria, can efficiently capture exogenous genes and ensure their expression. Moreover, these integrons could be complexed with transposons, plasmids, etc., consequently enabling the intra- and interspecies dissemination of multiple antimicrobial resistance genes in bacteria. Over the past two decades, the emergence of new gene arrangements and complex integrons in gene cassettes has led to the rapid evolution of integron systems. Integrons play a significant role in the spread of antimicrobial resistance in Salmonella. In this paper, we summarized the molecular structure, classification, and action mechanism of integron systems, and reviewed the progress of research on class Ⅰ, Ⅱ, and Ⅲ integron-mediated antimicrobial resistance presented in Salmonella, together with the available detection methods, aiming to provide a reference for the research on antimicrobial resistance of Salmonella.
Keywords: Salmonellaintegronantimicrobial resistanceresistance gene
沙门菌(Salmonella spp.)是一种常见的人畜共患病病原菌,菌型繁多、分布广泛,目前已检出超过2600余种血清型,我国共发现292种。沙门菌可沿食物链等方式传播给人类导致感染,常见临床表现为急性胃肠炎症,其次是败血症和伤寒肠热病,严重者危及生命安全[1]。据统计,全球每年至少有9400万例因沙门菌感染引起的肠胃炎病例,导致15.5万人死亡,其中85%的病例与食品污染有关[2]。沙门菌在食源性致病菌引起食物中毒事件的病因中常居榜首,为此本实验室在2011–2016年间对全国39座城市的零售肉制品、生食蔬菜和零售水产品样品中的沙门菌进行污染性调查,发现生食蔬菜样本中的沙门菌检出率较低为3.4%[3],零售肉制品和零售水产品中沙门菌的检出率较高分别为19.7%和37.5%[4–5],不仅分离出肠炎沙门菌、鼠伤寒沙门菌、德尔卑沙门菌等常见的血清型,还发现了鼠伤寒沙门菌单相变异株,由此可见沙门菌对食品安全构成重大威胁。
抗生素是治疗沙门菌感染的有效手段,然而抗生素的滥用却导致沙门菌耐药性问题日趋严峻,细菌耐药、尤其是多重耐药情况日益增多,多重耐药率已从20%–30%上升到70%,给人类生命健康带来严重威胁[6]。本实验室通过对全国零售食品样本分离出的沙门菌进行抗生素敏感性测试,发现沙门菌的耐药情况非常严重,沙门菌对于一些常见抗生素如萘啶酸、氨苄青霉素、链霉素等具有很高的耐药率[3, 5]。
研究表明,细菌不断进化产生耐药性的主要途径之一是通过水平基因转移的方式获得外源性耐药基因[7]。耐药基因的水平转移主要由整合子、质粒、转座子和噬菌体等可移动遗传元件介导,其中整合子具有整合和表达耐药基因的能力,发挥着重要作用,其介导的耐药基因不仅在细菌种内或种间传播,甚至可以跨越菌属的界限在真菌和细菌之间进行传播[8]。为了系统分析整合子在沙门菌耐药性传播的过程中扮演的角色,本文详细归纳了现有对沙门菌整合子的研究,并提出展望,以期为研究沙门菌耐药基因的产生及传播机制提供思路,进而为寻找有效防控耐药沙门菌措施提供参考。
1 整合子系统结构与分类 1989年,澳大利亚****Strokes首次在革兰氏阴性菌中发现了一种新型的移动基因元件,并将其命名为“DNA整合元件”即整合子(integrons),1991年Hall正式提出整合子系统的概念[8]。整合子系统主要由整合子及携带耐药基因的基因盒两部分构成,其中整合子作为主干,携带耐药基因的基因盒作为支干衔接在后[9]。如图 1所示,整合子基本结构包括两端的保守区(分别称为5′-CS、3′-CS)和一个携带耐药基因盒的可变区(图 1)。5′-CS中包括intI基因、attI位点及一个驱动基因盒的有效转录和表达所必需的、位于attI上游的启动子Pc,3′-CS则因整合子种类不同而有所差别。其中,intI为一类酪氨酸重组酶,通常以拓扑异构酶Ⅰ型反应进行重组。intI不仅能够捕获新外源基因并将其插入可变区域,而且还能够对可变区中已插入的基因盒进行重排或者剔除[10]。intI并非仅能催化整合子和基因盒之间的重组反应,整合子与整合子间也可发生重组。Kotlarska等****研究发现,intI能催化相同甚至不同类型的整合子之间发生重组反应,在重组反应发生后携带的基因盒的数量和顺序都会发生明显的改变[11]。attI一般位于intI的上游,但也有少数attI位于下游的情况出现,attI位点能够被intI特异性识别,并将基因盒整合到该位点。intI对基因盒attC识别的特异性较低,几乎所有的intI都能识别大部分基因盒的attC位点,因此整合子的捕获剪切作用发生频率较高[12]。有研究表明,大部分基因盒不含有启动子,其携带基因的表达依赖Pc启动子[13] (图 1)。
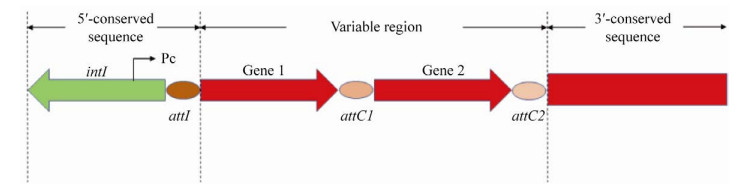 |
| 图 1 整合子基本结构 Figure 1 Integrons basic structure. |
| 图选项 |
整合子有两种分类方法:一是根据整合子的特点和作用分为耐药整合子和超级整合子;另一种是根据其编码的整合酶基因进行分类[14–15]。目前,研究已发现10类不同整合酶基因的整合子系统广泛分布于多种细菌中,但只有5类整合子的基因盒含有抗生素耐药基因[8]。其中Ⅰ、Ⅱ、Ⅲ类整合子与肠杆菌科细菌中多种耐药基因的传播有关,因此现有研究集中于这三类整合子。整合子系统组成复杂,作为一种重组系统,它能够整合、切除以及表达多种耐药基因,实现耐药基因的捕获及传播,因此一直是研究热点。目前发现介导细菌耐药性的基因盒多达130多种,包括含有介导氨基糖苷类抗生素耐药的aadA、aac,介导甲氧苄啶类抗生素耐药的dfr,介导苯丙醇类抗生素耐药的cat、cml和介导四环素类抗生素耐药的tet等。整合子不能进行自我转移,需要将自身与转座子和(或)接合型质粒中存在的插入序列偶联,才能作为种内或种间传播的载体传播耐药基因[16]。整合子可以通过交换和储存外源基因盒,使宿主的适应性得到增强。因此,包括接合型质粒、转座子、插入序列和基因组岛在内的可移动遗传元件都有可能成为整合子的巨大基因库在细菌之间共享[17]。
2 整合子对基因盒的捕获、剪切与表达 整合子具有捕获外源基因盒,使沙门菌获得耐药性的能力。整合子捕获外源基因盒主要依赖自身attI位点与位于基因盒的attC位点。在整合的过程中,intI识别游离的环状基因盒的attC位点使之与自身attI位点在7 bp的核心共有区域GTTRRRY (R为嘌呤,Y为嘧啶)催化重组,成为整合子的一部分(图 2)。这是一个可逆的过程,整合子亦可催化自身attC位点与attI位点发生重组反应,使基因盒被切下恢复环状游离态,进而被其他整合子捕获[18]。一般来说整合活动常见于attI与attC之间,且基因盒严格按照5′→3′、基因靠近intI的方式排列。通过这种方式,基因盒可以从一个整合子传递到其他整合子上。在表达过程中,由于大多数基因盒不含启动子,必须与整合子的启动子Pc形成类似于操纵子结构进行表达。整合子中启动子起始能力的有无、起始能力的强弱和与基因盒的距离等因素共同决定了启动子的转录水平,然而少数自身含有启动子的基因盒,如cmlA、cmlA2、qacF等,其表达不受上游启动子的影响[10]。启动子与intI表达方向相反,即转录区域方向相反。因此,启动子的起始能力会抑制intI的作用,影响intI捕获和剪切基因盒。Escudero等****研究发现基因盒表达水平较高的整合子系统,其捕获和剪切基因盒的能力弱于基因盒表达水平低的系统[19]。
 |
| 图 2 整合子捕获、剪切基因盒示意图 Figure 2 Schematic diagram of integrons capture and shear gene cassette. |
| 图选项 |
3 整合子介导沙门菌耐药 3.1 Ⅰ类整合子介导沙门菌耐药 Ⅰ类整合子最早是在非致病性β-变形菌(Bateproteobacteria)中发现的,也是目前沙门菌中最普遍存在的一类整合子,含有337个氨基酸[20]。Ⅰ类整合子的5′-CS较为相似,均携带intI1;而3′-CS存在个体差异,多数3′-CS有3个开放阅读框架:一个季铵盐化合物及溴乙锭的耐药基因qacEΔ1、磺胺耐药基因sul1和一个功能不明的ORF5[21]。这类整合子的大多数位于接合型质粒上,可以很容易地传递给其他菌株。有****认为Ⅰ类整合子与转座子Tn402相关联,Tn402类转座子通过位点特异性重组的方式捕获Ⅰ类整合子,进而水平转移到多种病原体中,引起Ⅰ类整合子的进化[13],转座子Tn402经常插入到其他大转座子中,例如Tn21/Tn1696[22]。目前对于Ⅰ类整合子固定到病原菌转座子的动力因素尚未确定,研究者推测可能与3′-CS保守端的作用有关[23]。
自1990年以来,多重耐药沙门菌的出现对公共卫生构成了严重的威胁。Ⅰ类整合子在多重耐药沙门菌的检出率高达60%以上,并已发现超过80多种类型基因盒[24]。据报道,这类整合子中的基因盒编码对大多数已知的β-内酰胺类、氨基糖苷类、甲氧苄啶类、利福平类、氯霉素类、喹诺酮类、红霉素类抗生素和季铵盐类化合物的耐药性[25]。目前沙门菌中最常检出的基因盒为dfrA17-aadA5,介导对甲氧苄啶类和氨基糖苷类抗生素耐药,这种现象可能与氨基糖苷类抗生素作为食用动物生长促进剂的大量使用有关[26]。由于抗生素的更新换代,给沙门菌带来的选择性压力也随之改变,沙门菌不断进化产生新的耐药基因以适应环境,而新耐药基因的出现也导致Ⅰ类整合子携带的基因盒不断变化。起初,沙门菌检出的Ⅰ类整合子只携带单个耐药基因(例如dfrA1, aadA1或aadB),后来,2004年在一株库马萨沙门菌和一株布雷登尼沙门菌中分别检测到aadA2-aac(6')-IIc-ereA2[27]和dfrA21-blaOXA-129- aadA1[28]两个基因盒序列。2010年在一株鼠伤寒沙门菌中检测到的Ⅰ类整合子携带超过7个抗生素耐药基因,除了常见的aadA2和sul1外,还包括介导氯霉素耐药的floR、介导四环素耐药的tetG、tetR和介导氨苄青霉素耐药的pse-1等[29]。目前为止,Ⅰ类整合子在各类样本分离的沙门菌中频频检出,并且基因盒种类繁多,大部分携带1–7个数目不等的耐药基因。本实验室对139株肠炎沙门菌的Ⅰ类整合子筛查的结果显示,共有14株Ⅰ类整合子阳性菌株,检出率为10.1%,共检测出3种耐药基因盒,分别为dfrA1-aadA1、dfrA17-aadA5、aadA22,其中dfrA17-aadA5 (8/14)检出率最高。通过接合转移实验验证了其均具有可转移能力(我们将接合成功的菌株称为接合子),根据接合子和受体菌的药敏实验结果对比分析可知,整合子成功将携带的耐药基因转移至受体菌,并使其成功表达。除了典型的Ⅰ类整合子外,非典型的Ⅰ类整合子也在沙门菌中被陆续发现,它们或丧失3′-CS的sul1基因,或由sul3和qacH基因代替qacEΔ1基因等[30–31]。
3.2 Ⅱ类整合子介导沙门菌耐药 Ⅱ类整合子复合于Tn7转座子家族,携带整合位点attI2和启动子Pc,Ⅱ类整合子的intI2的产物约有318个氨基酸,并与Ⅰ类整合子的intI1同源性 < 50%[32]。由于intI2基因被一含有179个氨基酸序列的终止密码子所中断,无法产生有活性的Ⅱ类整合子蛋白,因此限制了其剪切和重组基因盒的能力,故Ⅱ类整合子intI2不活跃。它们的盒式序列相比于Ⅰ类整合子高度保守,功能范围有限[33]。但是有研究报道称,基因突变可以逆转这个终止密码子的作用,使Ⅱ类整合子产生具有功能的intI2基因[34]。
虽然Ⅱ类整合子的检出率较低,但据报道称它们存在于多种沙门菌血清型中,多数携带相同的基因盒dfrA1-sat1-aadA1[35–36],偶有sat2替换 sat1的报道[37]。除此之外,也有一些非典型Ⅱ类整合子的报道。2003年Biskri等在两株肠炎沙门菌中检出了携带ere(A)的Ⅱ类整合子,其完整的基因盒序列为sat1-ere(A)-aadA1[38]。2009年Ahmed在一株肠炎沙门菌中发现了另一种非典型Ⅱ类整合子基因盒estX-sat2-aadA1[39]。2018年在猪源分离的4株沙门菌(包括两株鼠伤寒沙门菌,一株布列德尼沙门菌和一株卡彭巴沙门菌)中检出的Ⅱ类整合子携带2300 bp的基因盒estX-sat2-aadA1[36]。目前为止,在沙门菌Ⅱ类整合子相关研究报道中,以检出携带基因盒dfrA1-sat1-aadA1类型的Ⅱ类整合子为主,携带其他类型基因盒的Ⅱ类整合子的报道较少。综上所述,尽管Ⅱ类整合子比Ⅰ类整合子的检出率低,但也在多种沙门菌血清型中被检出,且不断报道出携带新耐药基因盒,因此,其重要性不容忽视。
3.3 Ⅲ类整合子介导沙门菌耐药 Ⅲ类整合子是1993年从日本的粘质沙雷菌(Serratia marcescens)样本中首次检出,intI3有346个氨基酸,与intI1有61%的同源性。Ⅲ类整合子也是被转座子Tn402捕获的,但与Ⅰ类整合子捕获外源性基因盒的方向相反,Ⅲ类整合子两侧是一对反向重复序列末端,催化环状基因盒的插入与删除[40]。尽管是最不普遍的整合子之一,Ⅲ类整合子却不断在新物种中传播并获得新的耐药基因盒[41–42]。在2017年之前并无沙门菌检出Ⅲ类整合子的报道,2018年在伊朗北部的鸡肉样本分离出的2株多重耐药婴儿沙门菌中检测出了Ⅲ类整合子,其均携带600 bp的intI3,但研究者并未扩增其基因盒[43]。目前为止,对Ⅲ类整合子耐药基因盒的扩增测序结果显示大多数Ⅲ类整合子均包含β-内酰胺类和氨基糖苷类耐药基因盒,如blaIMP–aac(6')-Ib、blaGES-1–blaOXA-10-aac(6')-Ib和blaOXA-2–blaGES-1等,因此Ⅲ类整合子可能在未来对β-内酰胺类和氨基糖苷类抗生素耐药性的传播发挥重要的作用。由于这些抗生素是治疗沙门菌严重感染时使用最多的抗生素,因此Ⅲ类整合子的重要性不言而喻。
4 整合子的检测 整合子种类众多,针对整合子的检测主要是利用分子生物学技术。其中应用最广泛的就是PCR技术,包括普通PCR技术和多重PCR技术等。本实验室采用普通PCR技术,分别在Ⅰ类整合子的5′端保守序列设计上下游引物,检测细菌是否含有Ⅰ类整合子基因。并且分别在Ⅰ类整合子5′和3′端保守序列设计上下游引物,扩增Ⅰ类整合子内可变区及携带的耐药基因盒,结果在14株沙肠炎门菌中检测出Ⅰ类整合子,且均携带耐药基因盒。除了常用的普通PCR技术外,多重PCR技术可提供更加高效的检测,赵永静[44]利用多重PCR技术同时扩增Ⅰ类、Ⅱ类和Ⅲ类整合子,在筛查整合子的同时对整合子进行分类。反向PCR技术可通过扩增Ⅰ类整合子异常3′-CS qnrB2-sul1基因,从而检测Ⅰ类整合子,但这种检测方法特异性较高,不适合大批量筛查[45]。除了PCR技术外,多重LAMP技术也被应用于检测,徐敬潇等[46]根据已知整合子基因序列,通过分析筛选出3种整合子(Ⅰ类、Ⅱ类、Ⅲ类)的特异保守序列,设计LAMP引物组,建立了多重LAMP检测方法。
5 讨论和展望 抗生素的滥用,造成了沙门菌对抗生素的耐药性问题。随着耐药性菌株的逐渐增多、耐药谱越来越广泛,沙门菌对人类的健康威胁日益增加[47]。在耐药性的传播过程中整合子具有非常关键的作用,整合子通过捕获一个或多个耐药基因,使沙门菌表现出对多种抗生素的耐药性。沙门菌中Ⅰ类整合子的检出率比Ⅱ类和Ⅲ类整合子高,且耐药基因盒的多样性和非典型整合子的不断出现,说明整合子系统正在不断地进化。当整合子系统中出现了新的基因盒,特别是新的β-内酰胺类、氟喹诺酮类和三、四代头孢菌素类抗生素耐药基因的出现,对我们使用抗生素治疗沙门菌感染提出了新的挑战。了解整合子在细菌遗传进化过程中的作用将为建立更好的监测系统提供方向,可以在临床和环境中防控这些微生物,以遏制其耐药性在全球范围内的扩散。但是目前存在诸多问题,比如耐药基因盒的来源、整合子在细菌进化中的作用等问题还未解决。
有研究表明,整合子是细菌进化的一个古老的产物,当细菌迫于外界压力的改变,便会通过整合子的重组、剪切外源基因盒的功能进行特异性重组,以适应环境的变化[48]。现在国内外对整合子的研究已从最初的筛选检测深入到了发现整合子的结构和表达耐药基因盒之间的调控机制。虽然研究不断深入,但还有许多方面的问题值得探讨:(1) 耐药基因盒的进化:沙门菌中整合子携带的耐药基因盒从最初的单一耐药基因到现在多样化的发展,已经严重影响了临床中抗生素的治疗效果,未来新基因盒资源能否含有更多编码适应功能的基因以增强细菌生存能力的问题值得探究。2019年本实验室首次在我国即食食品样本中分离的2株副溶血性弧菌VP007和V18J中检测出携带blaVEB-1的Ⅰ类整合子,其基因盒序列分别为dfrA14-blaVEB-1-aadB和blaVEB-1-aadB-arr2-cmlA-blaOXA-10-aadA1[49]。现有研究并无携带blaVEB-1的Ⅰ类整合子在沙门菌中检出的报道,随着耐药基因的传播,未来的某一天在沙门菌很有可能出现。有研究认为耐药整合子上基因盒来源于超级整合子(即第Ⅳ类整合子,由于此类整合子可携带上百个耐药基因盒,因此被称为超级整合子),这一观点将是未来相关研究的方向之一。2018年在伊朗地区分离出的两株婴儿沙门菌中检测出Ⅲ类整合子,在此之前并未出现Ⅲ类整合子在沙门菌中检出的报道,这说明整合子系统不断在新物种中传播,因此有必要对整合子的流行情况进行持续的追踪调查;(2) 整合子水平转移的动力问题:在偏远的山村、北极和濒危物种都发现了整合子和耐药基因的存在[12],这种现象和整合子的水平转移能力是否有关联?能否通过人为干预其转移的动力和方向?虽然整合子为耐药性的控制带来困难,但也为研究细菌耐药性的传播开辟新的思路;(3) 新检测技术的开发:更加快速高效的检测技术的开发,能为临床诊断及治疗提供帮助。例如:可以根据第Ⅰ、Ⅱ、Ⅲ类整合子基因的共同保守序列设计简并引物进行PCR;借助荧光PCR技术可以帮助建立更加快速准确的整合子检测方法;此外,还可利用核酸杂交法,根据整合子两端的保守序列设计探针,对质粒和染色体基因组进行核酸杂交进行检测,该方法也可应用于检测耐药基因盒。
References
| [1] | White AE, Ciampa N, Chen YX, Kirk M, Nesbitt A, Bruce BB, Walter ES. Characteristics of Campylobacter and Salmonella infections and acute gastroenteritis in older adults in Australia, Canada, and the United States. Clinical Infectious Diseases, 2019, 69(9): 1545-1552. DOI:10.1093/cid/ciy1142 |
| [2] | Ricke SC, Kim SA, Shi Z, Park SH. Molecular-based identification and detection of Salmonella in food production systems: current perspectives. Journal of Applied Microbiology, 2018, 125(2): 313-327. DOI:10.1111/jam.13888 |
| [3] | Yang XJ, Wu QP, Huang JH, Wu S, Zhang JM, Chen L, Wei XH, Ye YW, Li Y, Wang J, Lei T, Xue L, Pang R, Zhang YX. Prevalence and characterization of Salmonella isolated from raw vegetables in China. Food Control, 2020, 109: 106915. DOI:10.1016/j.foodcont.2019.106915 |
| [4] | Yang XJ, Wu QP, Zhang JM, Huang JH, Chen L, Wu S, Zeng HY, Wang J, Chen MT, Wu HM, Gu QH, Wei XH. Prevalence, bacterial load, and antimicrobial resistance of Salmonella serovars isolated from retail meat and meat products in China. Frontiers in Microbiology, 2019, 10: 2121. DOI:10.3389/fmicb.2019.02121 |
| [5] | Yang XJ, Huang JH, Zhang YX, Liu SR, Chen L, Xiao C, Zeng HY, Wei XH, Gu QH, Li Y, Wang J, Ding Y, Zhang JM, Wu QP. Prevalence, abundance, serovars and antimicrobial resistance of Salmonella isolated from retail raw poultry meat in China. Science of the Total Environment, 2020, 713: 136385. DOI:10.1016/j.scitotenv.2019.136385 |
| [6] | Zheng L, Zhu LW, Guo XJ, Chen P. Research progress of antimicrobial resistance of major epidemic serotypes of Salmonella. Jiangsu Agricultural Sciences, 2020, 48(6): 8-12. (in Chinese) 郑林, 祝令伟, 郭学军, 陈萍. 沙门氏菌主要流行血清型耐药性的研究进展. 江苏农业科学, 2020, 48(6): 8-12. |
| [7] | 伍好. 肉鸡源性肠杆菌科细菌中耐药基因、整合子及ISCR1元件的研究. 山东大学硕士学位论文, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10422-1016164893.htm |
| [8] | Chen WF, Ye T, Li L, Zhang CH. Research progress of integrons in bacterial resistance. Journal of Jilin Medical University, 2019, 40(3): 216-218. (in Chinese) 陈文凤, 叶彤, 李琳, 张宸豪. 整合子在细菌耐药中的研究进展. 吉林医药学院学报, 2019, 40(3): 216-218. |
| [9] | Estabraghi E, Salehi TZ, Amini K, Jamshidian M. Molecular identification of extended-spectrum β-lactamase and integron genes in Klebsiella pneumonia. JNMA; Journal of the Nepal Medical Association, 2016, 54(202): 72-78. DOI:10.31729/jnma.2822 |
| [10] | Liu YC, Yu BL, Gao F, Li FY, Song SY, Guo Y, Yu LH, Liu MC. New advances in the mechanism of drug resistance transmission in the integron-gene cassette system. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(3): 244-247. (in Chinese) 刘耀川, 于本良, 高锋, 李凤元, 宋淑英, 郭洋, 于立辉, 刘明春. 整合子-基因盒系统耐药性传递机制研究新进展. 中国预防兽医学报, 2017, 39(3): 244-247. |
| [11] | Kotlarska E, ?uczkiewicz A, Pisowacka M, Burzyński A. Antibiotic resistance and prevalence of class 1 and 2 integrons in Escherichia coli isolated from two wastewater treatment plants, and their receiving waters (Gulf of Gdansk, Baltic Sea, Poland). Environmental Science and Pollution Research, 2015, 22(3): 2018-2030. DOI:10.1007/s11356-014-3474-7 |
| [12] | Hosseini SMJ, Naeini NS, Khaledi A, Daymad SF, Esmaeili D. Evaluate the relationship between class 1 integrons and drug resistance genes in clinical isolates of Pseudomonas aeruginosa. The Open Microbiology Journal, 2016, 10: 188-196. DOI:10.2174/1874285801610010188 |
| [13] | Liu M, Li WX, Li SR. Progress in researches on bacterial integrons in antibiotic resistance. Chinese Journal of Microecology, 2020, 32(4): 465-468. (in Chinese) 刘秘, 李万翔, 李世荣. 整合子与细菌耐药性的相关研究进展. 中国微生态学杂志, 2020, 32(4): 465-468. |
| [14] | Stapels DAC, Hill PWS, Westermann AJ, Fisher RA, Thurston TL, Saliba AE, Blommestein I, Vogel J, Helaine S. Salmonella persisters undermine host immune defenses during antibiotic treatment. Science, 2018, 362(6419): 1156-1160. DOI:10.1126/science.aat7148 |
| [15] | Rui YY, Lu WT, Li S, Cheng CC, Sun JJ, Yang Q. Integrons and insertion sequence common region 1(ISCR1) of carbapenem-non-susceptible Gram-negative bacilli in fecal specimens from 5000 patients in Southern China. International Journal of Antimicrobial Agents, 2018, 52(5): 571-576. DOI:10.1016/j.ijantimicag.2018.06.015 |
| [16] | Amos GCA, Ploumakis S, Zhang LH, Hawkey PM, Gaze WH, Wellington EMH. The widespread dissemination of integrons throughout bacterial communities in a riverine system. The ISME Journal, 2018, 12(3): 681-691. DOI:10.1038/s41396-017-0030-8 |
| [17] | Sultan I, Rahman S, Jan AT, Siddiqui MT, Mondal AH, Haq QMR. Antibiotics, resistome and resistance mechanisms: a bacterial perspective. Frontiers in Microbiology, 2018, 9: 2066. DOI:10.3389/fmicb.2018.02066 |
| [18] | Ghaly TM, Geoghegan JL, Tetu SG, Gillings MR. The peril and promise of integrons: beyond antibiotic resistance. Trends in Microbiology, 2020, 28(6): 455-464. DOI:10.1016/j.tim.2019.12.002 |
| [19] | Escudero JA, Loot C, Nivina A, Mazel D. The integron: adaptation on demand. Microbiology Spectrum, 2015, 3(2): MDNA3-MDNA0019-2014. |
| [20] | Wei QH, Jiang XF, Lü Y. Advances in integrons of bacteria. Chinese Journal of Antibiotics, 2008, 33(1): 1-5, 40. (in Chinese) 魏取好, 蒋晓飞, 吕元. 细菌整合子研究进展. 中国抗生素杂志, 2008, 33(1): 1-5, 40. |
| [21] | Ouyang RZ, He YY, Xu H. Advances in integrase regulations of bacteria-A review. Acta Microbiologica Sinica, 2015, 55(10): 1231-1237. (in Chinese) 欧阳润泽, 贺艳艳, 徐海. 细菌整合子中整合酶基因调控的研究进展. 微生物学报, 2015, 55(10): 1231-1237. |
| [22] | Han C, Wang LF. Integrating the research progress of relationship between bacterial drug resistance. World Latest Medicine Information, 2017, 17(99): 61-62. (in Chinese) 韩超, 王凌峰. 整合子与细菌耐药关系的研究进展. 世界最新医学信息文摘, 2017, 17(99): 61-62. |
| [23] | Ghaly TM, Chow L, Asher AJ, Waldron LS, Gillings MR. Evolution of class 1 integrons: Mobilization and dispersal via food-borne bacteria. PLoS ONE, 2017, 12(6): e0179169. DOI:10.1371/journal.pone.0179169 |
| [24] | Noh EB, Kim YB, Jeon HY, Seo KW, Son SH, Lee YJ. Antimicrobial resistance and genetic diversity of Salmonella serotypes recovered from edible pork offal from Korea. Microbial Drug Resistance: Larchmont, N Y, 2019, 25(10): 1514-1520. DOI:10.1089/mdr.2019.0010 |
| [25] | Langata LM, Maingi JM, Musonye HA, Kiiru J, Nyamache AK. Antimicrobial resistance genes in Salmonella and Escherichia coli isolates from chicken droppings in Nairobi, Kenya. BMC Research Notes, 2019, 12(1): 1-6. DOI:10.1186/s13104-018-4038-6 |
| [26] | Meng XF, Zhang ZF, Li KT, Wang Y, Xia XD, Wang X, Xi ML, Meng JH, Cui SH, Yang BW. Antibiotic susceptibility and molecular screening of class I integron in Salmonella isolates recovered from retail raw chicken carcasses in China. Microbial Drug Resistance: Larchmont, N Y, 2017, 23(2): 230-235. DOI:10.1089/mdr.2015.0359 |
| [27] | Gassama-Sow A, A?dara-Kane A, Raked N, Denis F, Ploy MC. Integrons in Salmonella keurmassar, Senegal. Emerging Infectious Diseases, 2004, 10(7): 1339-1341. DOI:10.3201/eid1007.030666 |
| [28] | Michael GB, Cardoso M, Schwarz S. Molecular analysis of multiresistant porcine Salmonella enterica subsp. enterica serovar Bredeney isolates from Southern Brazil: identification of resistance genes, integrons and a group Ⅱ intron. International Journal of Antimicrobial Agents, 2008, 32(2): 120-129. DOI:10.1016/j.ijantimicag.2008.02.024 |
| [29] | Hall RM. Salmonella genomic Islands and antibiotic resistance in Salmonella enterica. Future Microbiology, 2010, 5(10): 1525-1538. DOI:10.2217/fmb.10.122 |
| [30] | Luk-In S, Pulsrikarn C, Bangtrakulnonth A, Chatsuwan T, Kulwichit W. Occurrence of a novel class 1 integron harboring qnrVC4 in Salmonella Rissen. Diagnostic Microbiology and Infectious Disease, 2017, 88(3): 282-286. DOI:10.1016/j.diagmicrobio.2017.03.016 |
| [31] | Wannaprasat W, Padungtod P, Chuanchuen R. Class 1 integrons and virulence genes in Salmonella enterica isolates from pork and humans. International Journal of Antimicrobial Agents, 2011, 37(5): 457-461. DOI:10.1016/j.ijantimicag.2010.12.001 |
| [32] | Sultan I, Rahman S, Jan AT, Siddiqui MT, Mondal AH, Haq QMR. Antibiotics, resistome and resistance mechanisms: a bacterial perspective. Frontiers in Microbiology, 2018, 9: 2066. DOI:10.3389/fmicb.2018.02066 |
| [33] | Mendes Moreira A, Couvé-Deacon E, Bousquet P, Chainier D, Jové T, Ploy MC, Barraud O. Proteae: a reservoir of class 2 integrons?. Journal of Antimicrobial Chemotherapy, 2019, 74(6): 1560-1562. DOI:10.1093/jac/dkz079 |
| [34] | Wei QH, Hu QF, Li SS, Lu HY, Chen GQ, Shen BQ, Zhang P, Zhou YL. A novel functional class 2 integron in clinical Proteus mirabilis isolates. Journal of Antimicrobial Chemotherapy, 2014, 69(4): 973-976. DOI:10.1093/jac/dkt456 |
| [35] | Asgharpour F, Mahmoud S, Marashi A, Moulana Z. Molecular detection of class 1, 2 and 3 integrons and some antimicrobial resistance genes in Salmonella Infantis isolates. Iranian Journal of Microbiology, 2018, 10(2): 104-110. |
| [36] | Argüello H, Guerra B, Rodríguez I, Rubio P, Carvajal A. Characterization of antimicrobial resistance determinants and class 1 and class 2 integrons in Salmonella enterica spp., multidrug-resistant isolates from pigs. Genes, 2018, 9(5): 256. DOI:10.3390/genes9050256 |
| [37] | Doublet B, Praud K, Nguyen-Ho-bao T, Argudin MA, Bertrand S, Butaye P, Cloeckaert A. Extended-spectrum- lactamase- and AmpC -lactamase-producing D-tartrate-positive Salmonella enterica serovar Paratyphi B from broilers and human patients in Belgium, 2008-10. Journal of Antimicrobial Chemotherapy, 2014, 69(5): 1257-1264. DOI:10.1093/jac/dkt504 |
| [38] | Ahmed AM, Nakano H, Shimamoto T. Molecular characterization of integrons in non-typhoid Salmonella serovars isolated in Japan: description of an unusual class 2 integron. Journal of Antimicrobial Chemotherapy, 2005, 55(3): 371-374. DOI:10.1093/jac/dkh534 |
| [39] | Ahmed AM, Ishida Y, Shimamoto T. Molecular characterization of antimicrobial resistance in Salmonella isolated from animals in Japan. Journal of Applied Microbiology, 2009, 106(2): 402-409. DOI:10.1111/j.1365-2672.2008.04009.x |
| [40] | Gillings MR. Integrons: past, present, and future. Microbiology and Molecular Biology Reviews, 2014, 78(2): 257-277. DOI:10.1128/MMBR.00056-13 |
| [41] | Kaushik M, Kumar S, Kapoor RK, Virdi JS, Gulati P. Integrons in Enterobacteriaceae: diversity, distribution and epidemiology. International Journal of Antimicrobial Agents, 2018, 51(2): 167-176. DOI:10.1016/j.ijantimicag.2017.10.004 |
| [42] | Kargar M, Mohammadalipour Z, Doosti A, Lorzadeh S, Japoni-Nejad A. High prevalence of class 1 to 3 integrons among multidrug-resistant diarrheagenic Escherichia coli in southwest of Iran. Osong Public Health and Research Perspectives, 2014, 5(4): 193-198. DOI:10.1016/j.phrp.2014.06.003 |
| [43] | Asgharpour F, Mahmoud S, Marashi A, Moulana Z. Molecular detection of class 1, 2 and 3 integrons and some antimicrobial resistance genes in Salmonella Infantis isolates. Iranian Journal of Microbiology, 2018, 10(2): 104-110. |
| [44] | 赵永静. 多重PCR检测整合子及其在分析金葡菌耐药中的作用. 郑州大学硕士学位论文, 2011. https://d.wanfangdata.com.cn/thesis/Y1931343 |
| [45] | Feng Y, Yuan JL, Wu Y, Qi Y, Chen T. Using reverse PCR to analysis flanking DNA regions of class Ⅰ integrons. International Journal of Laboratory Medicine, 2013, 34(8): 919-920. (in Chinese) 冯银, 袁金玲, 伍勇, 漆涌, 陈体. 反向PCR扩增Ⅰ类整合酶基因侧翼序列. 国际检验医学杂志, 2013, 34(8): 919-920. DOI:10.3969/j.issn.1673-4130.2013.08.003 |
| [46] | Xu JX, Jia XB, Shen HY, Xu M, Dou XH. Study on detection of bacterial integrons by multiple LAMP. China Poultry, 2019, 41(14): 64-66. (in Chinese) 徐敬潇, 贾雪波, 沈海玉, 许明, 窦新红. 多重LAMP检测细菌整合子的研究. 中国家禽, 2019, 41(14): 64-66. |
| [47] | Qin XJ, Sun NY, Li CY, Yang R, Wu YB. Research progress of Salmonella detection methods in food. China Brewing, 2020, 39(9): 18-24. (in Chinese) 覃湘婕, 孙宁与, 李春尧, 杨荣, 吴永宝. 食品中沙门氏菌检测方法研究进展. 中国酿造, 2020, 39(9): 18-24. |
| [48] | Mazel D, Dychinco B, Webb VA, Davies J. A distinctive class of integron in the Vibrio cholerae genome. Science, 1998, 280(5363): 605-608. DOI:10.1126/science.280.5363.605 |
| [49] | Lei T, Zhang JM, Jiang FF, He M, Zeng HY, Chen MT, Pang R, Wu HM, Wu S, Wang J, Ding Y, Wu QP. Characterization of class 1 integrons harboring blaVEB-1 in Vibrio parahaemolyticus isolated from ready-to-eat foods in China. International Journal of Food Microbiology, 2020, 318: 108473. DOI:10.1016/j.ijfoodmicro.2019.108473 |
