翟诗岚1,2, 刘威1, 吴正超1,3, 朱义广1, 张文军1, 张长生1,2, 马亮1


1. 中国科学院南海海洋研究所, 中国科学院热带海洋生物资源与生态重点实验室, 中国科学院海洋微生物研究中心, 广东省海洋药物重点实验室, 广东 广州 510301;
2. 中国科学院大学, 北京 100049;
3. 中国科学院南海海洋研究所, 热带海洋环境国家重点实验室, 广东 广州 510301
收稿日期:2021-02-25;修回日期:2021-04-28;网络出版日期:2021-09-14
基金项目:广东省基础与应用基础研究基金(2021A1515010839);广州市科技计划(202102020513);国家自然科学基金(21907098,41706181);国家重点研发计划(2019YFC0312503);王宽诚教育基金(GJTD-2020-12)
*通信作者:马亮, Tel: +86-20-89108532;Fax: +86-20-84459635;E-mail: maliang@scsio.ac.cn.
摘要:[目的] 从珠江口沉积物来源的菌株SCSIO 40020中分离bafilomycins,并对其生物合成基因簇进行克隆和异源表达研究。[方法] 通过分析菌株SCSIO 40020的16S rRNA基因序列并构建系统发育树以鉴定菌种,以柱层析法和制备色谱法对次级代谢产物进行分离纯化,借助波谱学手段完成单体化合物的结构鉴定,采用生物信息学分析定位bafilomycins的生物合成基因簇,通过筛选菌株SCSIO 40020基因组的细菌人工染色体文库和接合转移将bafilomycins生物合成基因簇导入3种链霉菌进行异源表达,利用高效液相色谱检测异源表达菌株的发酵产物。[结果] 菌株SCSIO 40020被鉴定为链霉菌属菌株,从其发酵产物中分离鉴定了2个单体化合物bafilomycins A1和D。克隆了链霉菌SCSIO 40020中bafilomycins的生物合成基因簇并推导了其生物合成途径,在3种链霉菌中表达产生了bafilomycins。[结论] 从珠江口环境中获得了一株产生bafilomycins的链霉菌SCSIO 40020,成功建立了该菌株次级代谢产物生物合成基因簇的异源表达体系,并首次在链霉菌Streptomyces lividans SBT18、Streptomyces coelicolor M1154和Streptomyces albus J1074中进行了表达,获得了bafilomycins,为后续bafilomycins结构类似物的生产和链霉菌SCSIO 40020中新结构活性化合物的挖掘奠定了基础。
关键词:链霉菌bafilomycins生物合成基因簇异源表达
Identification of bafilomycins and heterologous expression of its biosynthetic gene cluster from the Pearl River Estuary sediment-derived Streptomyces sp. SCSIO 40020
Shilan Zhai1,2, Wei Liu1, Zhengchao Wu1,3, Yiguang Zhu1, Wenjun Zhang1, Changsheng Zhang1,2, Liang Ma1


1. Key Laboratory of Tropical Marine Bio-resources and Ecology, Guangdong Key Laboratory of Marine Materia Medica, RNAM Center for Marine Microbiology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China;
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. State Key Lboratory of Tropical Oceanography, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China
Received: 25 February 2021; Revised: 28 April 2021; Published online: 14 September 2021
*Corresponding author: Liang Ma, Tel: +86-20-89108532;Fax: +86-20-84459635;E-mail: maliang@scsio.ac.cn.
Foundation item: Supported by the Guangdong Basic and Applied Basic Research Foundation (2021A1515010839), by the Science and Technology Planning Project of Guangzhou (202102020513), by the National Natural Science Foundation of China (21907098, 41706181), by the National Key Research and Development Program of China (2019YFC0312503) and by the K.C.Wong Education Foundation (GJTD-2020-12)
Abstract: [Objective] To identify the macrolide antibiotic bafilomycins from the strain SCSIO 40020 isolated from the Pearl River Estuary sediment. To clone and heterologously express the biosynthetic gene cluster (BGC) of bafilomycins. [Methods] The strain SCSIO 40020 was identified based on phylogenetic analysis of its 16S rRNA gene sequence. The generated compounds from SCSIO 40020 were purified via normal phase-column chromatography and semi-preparative chromatography. The chemical structures of the isolated compounds were subsequently elucidated by comprehensive spectroscopic analyses. The bafilomycins BGC was identified by using bioinformatics approach. Through screening the bacterial artificial chromosome (BAC) library of this strain, the BAC containing the bafilomycins BGC was introduced into three Streptomyces hosts by conjugation for expression. Afterwards, the fermentation culture of the recombinant strains were analyzed on high-performance liquid chromatography (HPLC). [Results] The strain SCSIO 40020 was identified as Streptomyces genus and two macrolide compounds bafilomycins A1and D were isolated from the fermentation crude extract. The bafilomycins BGC was cloned and successfully expressed in three heterologous Streptomyces hosts. In addition, the biosynthetic pathway of bafilomycins was proposed. [Conclusion] The bafilomycin-producing strain Streptomyces sp. SCSIO 40020 was obtained from the Pearl River Estuary. The BAC heterologous expression system of this strain was successfully established. Moreover, the bafilomycins BGC was successfully expressed in Streptomyces lividans SBT18, Streptomyces coelicolor M1154 and Streptomyces albus J1074 for the first time, which laid solid foundation for the production of bafilomycin analogues and mining other interesting BGCs from Streptomyces sp. SCSIO 40020 in the further study.
Keywords: Streptomyces sp. SCSIO 40020bafilomycinsbiosynthetic gene clusterheterologous expression
海洋微生物为适应高盐、高压、黑暗、低温(或局部高温)、寡营养等严酷的生存环境而进化出了与之相适应的生理生化特征或特殊的代谢途径,这使得其次级代谢产物往往具有复杂的化学结构或独特的生物学活性,如头孢菌素C、利福霉素和salinosporamide A等[1–2]。珠江口地处亚热带区域,是我国南方最大的河口,其河流生态系统与海洋生态系统的相互作用剧烈而复杂[3]。研究表明,放线菌门的菌株约占该区域细菌的10%[4–6],但相关的放线菌次级代谢产物研究的报道较少,且主要集中于链霉菌属菌株,如中科院南海所周雪峰等从1株珠江口红树林底泥来源的链霉菌Streptomyces iakyrus SCSIO NS104中分离鉴定了一系列新颖的具有抗肾癌活性的piericidin结构类似物,并确认肾癌中低表达的过氧化物酶peroxiredoxin 1 (PRDX1)为piericidin重要的作用靶点[7–8];本课题组从1株珠江口沉积物来源的链霉菌Streptomyces sp. ZJ306中分离获得了抗肿瘤天然产物斑鸠霉素,并揭示了其生物合成过程中独特的还原环化反应机制[9]。这些研究提示珠江口环境中的链霉菌具有产生新结构活性次级代谢产物的潜力。
聚酮化合物是一类重要的天然产物,具有抗菌、抗肿瘤、抗感染等多种活性,其是通过聚酮合酶(polyketide synthase,PKS)催化形成的[10]。根据聚酮合酶的结构可以将其分为三类:Ⅰ型、Ⅱ型和Ⅲ型。其中Ⅰ型PKS是存在最广泛也是研究最透彻的,其产物主要为大环内酯类、多烯类和聚醚类化合物。Ⅰ型PKS是由多个模块(module)构成,每个Module通常由酮基合成酶(ketosynthase,KS)结构域、酰基转移酶(acyltransferase,AT)结构域和酰基载体蛋白(acyl carrier protein,ACP)结构域组成,负责通过Claisen缩合反应引入一个二碳单元。部分Module可能还含有酮基还原酶(ketoreductase,KR)结构域、脱水酶(dehydratase,DH)结构域或烯醇还原酶(enoyl reductase,ER)结构域,负责催化聚酮链上酮基实现不同程度的氧化还原。Ⅱ型PKS也称迭代型PKS,是一类多功能酶复合体,由一套可重复使用的结构域(KSα、KSβ和ACP)组成,主要催化芳香聚酮类化合物的合成。Ⅲ型PKS属于查尔酮(chalcone)合成酶,是一种可重复使用的同源双亚基蛋白,其可不依赖ACP而直接催化酰基辅酶A间的缩合,主要负责单环或双环芳香类聚酮化合物的合成[11–12]。典型的Ⅰ型PKS次级代谢产物生物合成基因簇通常大于50 kb,因此利用传统的cosmid文库很难克隆获得完整的Ⅰ型PKS基因簇,这给其异源表达带来困难。细菌人工染色体(BAC)衍生于大肠杆菌的F因子,为闭合环状DNA分子,其通常可装载120–300 kb的DNA片段,因此被广泛应用于Ⅰ型PKS基因簇的克隆[13–14],如阿维菌素[15]、quinolidomicin[16]、salinomycin[17]、streptoseomycin[18]和lobophorins[19]等都通过BAC载体克隆获得了完整的基因簇并实现在不同链霉菌宿主中的表达。
Bafilomycins是聚酮化合物,属于plecomacrolide家族,其核心结构为十六元环的大环内酯及六元半缩醛环[20]。其最早由Werner等于1983年从链霉菌S. griseus sp. sulfurus Tü 1922中分离得到[21],随后又从不同生境中分离得到,如陆生白丝北里孢菌Kitasatospora setae KM-6054[22]、海洋链霉菌Streptomyces sp. RJA71和RJA635[23]、植物内生链霉菌Streptomyces sp. YIM56209[24]以及亚洲象粪便来源链霉菌S. albolongus YIM101047[25]。研究表明,bafilomycins具有抗菌(革兰氏阳性菌、真菌)、抗肿瘤、抗寄生虫、免疫抑制和抗骨质疏松等活性[20, 26]。鉴于此,本研究采用链霉菌发酵培养、代谢产物分离、基因组测序、BAC文库筛选和接合转移等方法,结合生物信息学分析及文献报道,从1株珠江口沉积物来源的链霉菌Streptomyces sp. SCSIO 40020中分离得到2个单体化合物bafilomycins A1和D,克隆并鉴定了其生物合成基因簇,推导了其生物合成途径,建立了该菌株次级代谢产物生物合成基因簇的异源表达体系,并成功在链霉菌Streptomyces lividans SBT18、Streptomyces coelicolor M1154和Streptomyces albus J1074中表达生产了bafilomycins,为后续bafilomycins的结构改造和菌株SCSIO 40020的基因组挖掘奠定了基础。
1 材料和方法 1.1 材料
1.1.1 菌种: 链霉菌Streptomyces sp. SCSIO 40020 (原始编号LW701)由本实验室刘威博士从珠江口沉积物(东经114.0432°,北纬22.0194°,28 m水深)中分离纯化得到。菌株保存和传代使用固体AM6培养基于28 ℃培养,菌种保藏于中国科学院南海海洋研究所海洋微生物研究中心菌种保藏库。链霉菌S. lividans SBT18[27]、S. coelicolor M1154[28]、S. albus J1074[29]为异源表达宿主菌,由上海交通大学陶美凤教授提供。大肠杆菌Escherichia coli ET12567/pUB307由本实验室收藏。
1.1.2 培养基: 菌株SCSIO 40020固体培养使用AM6固体培养基:可溶性淀粉20 g,葡萄糖10 g,酵母粉5 g,细菌学蛋白胨5 g,CaCO3 5 g,海盐30 g,琼脂20 g,加水定容至1 L,pH 7.0;异源表达野生型及重组菌株固体培养使用改良的SFM培养基:黄豆粉15 g,甘露醇20 g,琼脂20 g,NaCl 2 g,CaCO3 5 g,加水定容至1 L,pH 7.0;菌株SCSIO 40020及异源表达重组菌株液体发酵培养使用ISP3培养基:燕麦粉20 g,CaCO3 5 g,海盐30 g,加水定容至1 L,pH 7.0;接合子培养使用改良的ISP4固体培养基:可溶性淀粉10 g,K2HPO4 10 g,MgSO4·7H2O 1 g,NaCl 1 g,(NH4)2SO4 2 g,CaCO3 2 g,琼脂20 g,微量元素混合液100 μL,加水定容至1 L,pH 7.0;大肠杆菌培养使用LB液体培养基:胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,加水定容至1 L,pH 7.0;种子培养使用TSB液体培养基:胰蛋白胨大豆肉汤培养基30 g,海盐30 g,加水定容至1 L,pH 7.0。
1.1.3 主要试剂和仪器: 所用PCR引物由北京六合华大基因科技有限公司合成;全基因组测序由武汉未来组生物科技有限公司完成;基因组BAC文库由武汉八颗星生物科技有限公司构建;BAC克隆子末端测序由武汉八颗星生物科技有限公司完成;Easytaq DNA聚合酶等酶试剂来自北京全式金生物技术有限公司;抗生素购自Sigma公司。
PCR仪,德国Eppendorf公司;Tanon EPS-300电泳仪,上海天能科技有限公司;凝胶成像系统Gel DocTM XR+,美国Bio-Rad公司;旋转蒸发仪(Laborota4000 efficient),德国Heidolph公司;高效液相色谱仪,美国Agilent公司,C18反相色谱柱(5 μm,250 mm×4.6 mm;Phenomenex Gemini C18);半制备高效液相色谱仪,日本Hitachi公司。
1.2 链霉菌基因组的提取及16S rRNA基因序列的鉴定 采用文献[30]报道的方法提取菌株SCSIO 40020的基因组DNA,由武汉未来组生物科技有限公司完成菌株的全基因组测序。将从基因组序列中获得的16S rRNA基因序列在EzBioCloud 16S database中进行比对,选取相似性较高的序列,利用MEGA7.0软件的Clustal W算法进行多序列比对,并采用邻接法(neighbor-joining)构建系统发育树。
1.3 次级代谢产物生物合成基因簇的分析 将菌株SCSIO 40020的基因组序列上传至antiSMASH网站(https://antismash.secondarymetabolites.org/)进行次级代谢产物生物合成基因簇的分析。利用2ndFind (http://biosyn.nih.go.jp/2ndfind/)软件对目标基因簇所涵盖的序列进行开放阅读框和核糖体结合位点的预测,并结合BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)软件对基因功能进行注释。
1.4 菌株耐盐度实验 将菌株SCSIO 40020的孢子刮到离心管中,加入适当无菌水振荡10 min将孢子充分悬起。用孢子过滤器过滤悬液得到均匀的孢子液,分别在海盐浓度为0%、0.5%、1%、2%、3%、4%、5%、6%、7%、8%、9%、10%的AM6平板上加入等量的孢子液,用竹签划线;在28 ℃培养12 d,观察菌株的生长情况。
1.5 菌株的发酵培养 将活化的菌株SCSIO 40020接入每瓶含有30 mL TSB液体培养基的250 mL锥形培养瓶中,28 ℃、200 r/min培养3 d制得种子液。将种子液以体积分数10%的接种量接入到10 L的ISP3液体培养基中,28 ℃、200 r/min振荡培养7 d,而制得发酵培养物。
1.6 发酵产物的提取和分离 对发酵培养物进行离心得到菌液和菌体两部分,分别用等体积的丁酮和丙酮萃取3次,合并获得粗提物(13.5 g)。将粗提物与硅胶(100–200目)一起进行拌样,经硅胶柱层析,以氯仿/甲醇为洗脱剂,从体积比100/0到0/100梯度洗脱,收集馏分,根据薄层色谱(TLC)和高效液相色谱(HPLC)分析合并得到Fr.1–Fr.3,将馏分Fr.1进行凝胶柱层析(Sephadex LH-20),收集合并得到馏分Fr.1.1–Fr.1.10,然后对馏分Fr.1.6–Fr.1.10进行半制备高效液相柱层析(流动相为体积比85:15的乙腈/水,流速2.5 mL/min),得到化合物1;Fr.2进行C18反相中压液相柱层析(柱型14.5 cm×2.5 cm,流速15 mL/min),得到18个馏分(Fr.2.1–Fr.2.18)。将馏分Fr.2.9–Fr.2.18进行高效液相柱层析纯化,得到化合物2。
1.7 BAC文库的筛选 利用特异性引物Baf-sk-1F (5′-TGTGACTGA GCACGCCAGTG-3′)和Baf-sk-1R (5′-CCGGTGC GTGCATGACCCTC-3′)、Baf-sk-2F (5′-ACCGCAA TGTTTTCGCCATC-3′)和Baf-sk-2R (5′-CTCTTCG TCCGTGGTCTCAG-3′)对菌株SCSIO 40020基因组BAC文库进行PCR筛选。把BAC文库中同一块板上的横向、纵向混合DNA分别作为模板进行PCR检测,再将阳性扩增结果对应的克隆子从BAC文库中挑出,划单克隆进行复筛。PCR体系为:10×Easytaq Buffer 2 μL,dNTPs 1 μL,Easytaq Enzyme 0.2 μL,引物F 0.5 μL,引物R 0.5 μL,DMSO 1.2 μL,模板1 μL,H2O 13.6 μL。PCR反应条件为:95 ℃ 10 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min,32个循环;72 ℃ 5 min。
1.8 异源表达菌株的构建 分别接种活化的E. coli ET12567/pUB307和供体菌(包含阳性BAC载体)于LB液体培养基中,37 ℃、200 r/min培养3–4 h,至OD600约为0.6。收集菌液,并用LB液体培养基洗2次,作为接合转移供体备用;从SFM平板上刮取链霉菌(S. lividans SBT18,S. coelicolor M1154或S. albus J1074)孢子,加入适量TSB液体培养基,50 ℃水浴热激10 min,而后置于摇床(28 ℃,200 r/min)培养萌发2–3 h,作为接合转移受体备用;混合接合转移的供体和受体,涂布于含有MgCl2 (终浓度为20 mmol/L)的ISP4平板上,28 ℃培养18 h;用甲氧苄啶(TMP,终浓度为50 μg/mL)和安普霉素(Apr,终浓度为25 μg/mL)覆盖平板,28 ℃培养5–7 d后,可观察接合子出现。抽提接合子的基因组DNA并利用特异性引物Baf-sk-1F/R和Baf-sk-2F/R进行PCR验证,筛选获得包含目标基因簇的异源表达菌株。
1.9 异源表达菌株的发酵检测 将包含目标基因簇的异源表达菌株接种至SFM平板上,于28 ℃恒温培养7 d。刮取适量孢子接种于ISP3液体培养基中进行发酵培养7 d。取5 mL发酵液加入等体积丁酮,超声后取上层有机相旋干,加入100 μL甲醇溶解后取50 μL进样。HPLC检测条件为:流动相A相为10%乙腈,含0.1%三氟乙酸,流动相B相为90%乙腈,流速1 mL/min,检测波长为254 nm。HPLC走样程序为:0–15 min,5%–100% B相;15–25 min,100% B相;25–26 min,100%–5% B相;26–30 min,5% B相。
2 结果和分析 2.1 菌种鉴定及耐盐度分析 菌株SCSIO 40020由本课题组刘威博士从珠江口沉积物(东经114.0432°,北纬22.0194°,28 m水深)中分离得到。对该菌株进行16S rRNA基因序列鉴定(GenBank编号:MW582618),通过序列比对、相似性分析确定其与Streptomyces halstedii NBRC 12783T的序列相似性最高,为99.86%。进一步构建菌株SCSIO 40020 16S rRNA基因的系统发育树(图 1),结果显示该菌株与链霉菌属的标准菌株聚在同一分支,因此确定菌株SCSIO 40020为链霉菌属菌株,命名为Streptomyces sp. SCSIO 40020。
 |
| 图 1 基于16S rRNA基因序列构建的菌株SCSIO 40020系统发育树 Figure 1 Neighbor-joining phylogenetic tree constructed on the basis of almost-complete 16S rRNA gene sequences showing the relationships between the stain SCSIO 40020 and closely related members of genus Streptomyces. The numbers at branch nodes indicate bootstrap percentages derived from 1000 replications; only values above 50% are shown. Bar, 0.002 substitutions per single nucleotide position. Numbers in parentheses represent the sequences accession number in GenBank. T: Type strain. |
| 图选项 |
将菌株SCSIO 40020在不同海盐浓度的培养基中进行培养,结果发现菌株SCSIO 40020为耐盐菌株,在0%–5%的海盐浓度下生长良好,这与其所处环境的海盐浓度范围[31]相符。
2.2 Bafilomycins A1和D的分离及结构鉴定 对菌株SCSIO 40020进行10 L液体发酵,发酵产物经有机溶剂萃取和各种色谱柱分离,获得化合物1和2。通过ESI-MS、1H NMR、13C NMR等波谱分析及与参考文献相比较,确定化合物1 (bafilomycin A1)和2 (bafilomycin D)的结构(图 2)。
 |
| 图 2 化合物1和2的结构 Figure 2 Chemical structures of bafilomycin A1 (1) and bafilomycin D (2). |
| 图选项 |
化合物1为淡黄色油状物。(ESI-MS) m/z 645.5 [M+Na]+,提示其分子量为622,结合氢谱和碳谱分析,确定其分子式为C35H58O9。1H NMR (700 MHz, CDCl3) δH 6.67 (1H, s, H-3), 6.50 (1H, dd, J=15.0, 10.7 Hz, H-12), 5.81 (1H, d, J=10.6 Hz, H-11), 5.77 (1H, d, J=9.1 Hz, H-5), 5.53 (1H, s, OH-19), 5.16 (1H, dd, J=15.0, 9.4 Hz, H-13), 4.95 (1H, d, J=8.6 Hz, H-15), 4.65 (1H, d, J=3.6 Hz, OH-17), 4.13 (1H, d, J=10.5 Hz, H-17), 3.88 (1H, t, J=9.0 Hz, H-14), 3.63 (3H, s, CH3O-2), 3.48 (1H, dd, J=10.2, 1.9 Hz, H-23), 3.29 (1H, d, J=6.9 Hz, H-7), 3.24 (3H, s, CH3O-14), 2.50 (1H, m, H-6), 2.29 (1H, dd, J=11.9, 4.7 Hz, Ha-20), 1.98 (3H, s, CH3-26), 1.93 (3H, s, CH3-29), 1.32 (1H, td, J=10.0, 6.5 Hz, H-22), 1.15 (1H, t, J=11.1 Hz, Hb-20), 1.06 (3H, d, J=7.0 Hz, CH3-27), 1.04 (3H, d, J=7.2 Hz, CH3-31), 0.93 (3H, dd, J=6.4, 3.7 Hz, CH3-32), 0.90 (1H, d, J=6.9 Hz, H-25), 0.82 (1H, d, J=6.9 Hz, H-30), 0.76 (1H, d, J=6.8 Hz, H-33)。13C NMR (176 MHz, CDCl3) δC 167.5 (C, C-1), 143.2 (C, C-10), 142.9 (CH, C-5), 141.4 (C, C-2), 133.7 (CH, C-3), 133.2 (C, C-4), 133.2 (CH, C-12), 127.3 (CH, C-13), 125.4 (CH, C-11), 99.1 (C, C-19), 82.4 (CH, C-14), 81.3 (CH, C-7), 77.0 (CH, C-15), 76.0 (CH, C-23), 71.1 (CH, C-21), 70.8 (CH, C-17), 60.1 (OCH3, C-2), 55.7 (OCH3, C-14), 43.6 (CH2, C-20), 42.2 (CH, C-18), 41.3 (CH2, C-9), 41.2 (CH, C-22), 40.1 (CH, C-8), 37.3 (CH, C-16), 36.8 (CH, C-6), 28.0 (CH, C-24), 21.8 (CH3, C-28), 21.3 (CH3, C-25), 20.3 (CH3, C-29), 17.4 (CH3, C-27), 14.5 (CH3, C-33), 14.2 (CH3, C-26), 12.3 (CH3, C-32), 10.0 (CH3, C-30), 7.2 (CH3, C-31)。其理化性质和波谱性质与已知化合物bafilomycin A1[32–33]数据一致。
化合物2为淡黄色油状物。(ESI-MS) m/z 627.5 [M + Na]+,提示其分子量为604,结合氢谱和碳谱分析,确定其分子式为C35H56O8。1H NMR (700 MHz, CDCl3) δH 6.90 (1H, d, J=15.8, 8.3 Hz, H-21), 6.64 (1H, s, H-3), 6.48 (s, 1H), 6.28 (1H, d, J=15.8 Hz, H-20), 5.80 (1H, d, J=10.7 Hz, H-11), 5.75 (1H, d, J = 9.1 Hz, H-5), 5.17 (2H, dd, J=15.0, 9.1 Hz, H-13), 5.05 (1H, d, J=8.0 Hz, H-15), 3.80(1H, m, H-14), 3.76 (1H, m, H-17), 3.67 (3H, s, OCH3-2), 3.30 (1H, m, H-7),2.52 (1H, m, H-6), 1.97 (3H, s, H-26), 1.90 (3H, s, CH3-29), 1.19 (3H, d, J= 7.0 Hz, CH3-31), 1.07 (3H, s, CH3-32), 0.93 (3H, d, J=6.7 Hz, CH3-33)。13C NMR (176 MHz, CDCl3) δC 203.3 (C, C-19), 166.6 (C, C-1), 148.7 (CH, C-21), 142.9 (C, C-10), 142.4 (CH, C-5), 141.6 (C, C-2), 133.1 (CH, C-3), 133.0 (C, C-4), 132.9 (CH, C-12), 129.5 (CH, C-20), 127.2 (CH, C-13), 125.5 (CH, C-11), 83.5 (CH, C-14), 81.4 (CH, C-7), 79.9 (CH, C-23), 76.5 (CH, C-15), 72.8 (CH, C-17), 60.3 (OCH3, C-2), 55.8 (OCH3, C-14), 46.4 (CH, C-18), 41.4 (CH2, C-9), 40.1 (CH, C-22), 39.9 (CH, C-8), 38.6 (CH, C-16), 36.9 (CH, C-6), 31.1 (CH, C-24), 22.0 (CH3, C-28), 20.1 (CH3, C-29), 19.8 (CH3, C-25), 17.5 (CH3, C-27), 16.9 (CH3, C-33), 16.7 (CH3, C-32), 14.1 (CH3, C-26), 10.8 (CH3, C-30), 10.4 (CH3, C-31)。其理化性质和波谱性质与已知化合物bafilomycin D[24, 32]数据一致。
2.3 Streptomyces sp. SCSIO 40020中bafilomycins生物合成途径的推导
2.3.1 Bafilomycins生物合成基因簇的定位: 利用antiSMASH 5.0对菌株SCSIO 40020基因组序列进行分析,通过与文献报道的bafilomycins生物合成基因簇进行比对[26, 34–35],发现Region 25可能与bafilomycins合成相关。进一步利用2ndFind和BLAST软件对该区域进行深入分析,确定该区域内一段大小为73549 bp,包含19个开放阅读框的DNA序列(bfl基因簇)负责菌株SCSIO 40020中bafilomycins的合成(图 3)。参照已发表的bafilomycins生物合成基因簇,对bfl基因簇内的基因进行功能注释(表 1),发现其含有6个Ⅰ型PKS编码基因bflA1、bflA2、bflA3、bflA4、bflA5、bflA6以及1个游离的Ⅱ型硫酯酶基因bflG,5个参与甲氧基丙二酰-ACP形成的基因bflB、bflC、bflD、bflE、bfF,5个负责催化bafilomycin A1侧链后修饰的基因bflM、bflL、bflK、bflI、bflJ,2个转录调控基因bflR和bflH。
 |
| 图 3 Streptomyces sp. SCSIO 40020中bafilomycins的生物合成基因簇 Figure 3 The bafilomycins biosynthetic gene cluster from Streptomyces sp. SCSIO 40020. |
| 图选项 |
表 1. Streptomyces sp. SCSIO 40020中bafilomycins生物合成基因簇内各基因的功能注释 Table 1. The deduced gene functions in the bafilomycins biosynthetic gene cluster from Streptomyces sp. SCSIO 40020
| Gene | Size/bp | Proposed function | Protein homologue (GenBank accession No.) identity/similarity | ||
| K. setae KM-6054 | S. griseus DSM 2608 | S. lohii JCM 14114 | |||
| bflM | 1596 | acyl-CoA ligase | BfmM (BAJ33096) 90/93 | BafBI (AGK25217) 92/94 | BafX (ADC79613) 91/94 |
| bflL | 1545 | amide synthetase | BfmL (BAJ33097) 85/89 | BafBII (AGK25192) 89/93 | BafY (ADC79614) 91/93 |
| bflK | 1245 | 5-aminolevulinic acid synthase | BfmK (BAJ33098) 89/94 | BafBIII (AGK25193) 92/95 | BafZ (ADC79615) 94/96 |
| bflA1 | 14424 | polyketide synthase | BfmA1 (BAJ33099) 83/86 | BafSI (AGK25194) 85/87 | BafAI (ADC79616) 88/90 |
| bflA2 | 10038 | polyketide synthase | BfmA2 (BAJ33100) 86/89 | BafSII (AGK25213) 89/91 | BafAII (ADC79617) 91/93 |
| bflA3 | 5187 | polyketide synthase | BfmA2 (BAJ33100) 84/87 | BafSII (AGK25213) 89/92 | BafAII (ADC79617) 89/91 |
| bflA4 | 11877 | polyketide synthase | BfmA3 (BAJ33101) 86/89 | BafSIII (AGK25214) 91/92 | BafAIII (ADC79618) 91/93 |
| bflA5 | 10398 | polyketide synthase | BfmA4 (BAJ33102) 85/89 | BafSIV (AGK25195) 86/88 | BafAIV (ADC79619) 89/90 |
| bflA6 | 6372 | polyketide synthase | BfmA5 (BAJ33103) 83/87 | BafSV (AGK25196) 88/91 | BafAV (ADC79620) 89/91 |
| bflB | 891 | glyceryl-ACP dehydrogenase | BfmB (BAJ33104) 88/91 | BafAI (AGK25191) 92/95 | BafB (ADC79621) 92/94 |
| bflC | 282 | acyl carrier protein (ACP) | BfmC (BAJ33105) 87/90 | BafAII (AGK25197) 90/92 | BafC (ADC79622) 94/95 |
| bflD | 1116 | 3-oxoacyl-ACP dehydrogenase | BfmD (BAJ33106) 92/95 | BafAIII (AGK25198) 94/96 | BafD (ADC79623) 94/96 |
| bflE | 1140 | glyceryl-ACP synthase | BfmE (BAJ33107) 87/90 | BafAIV (AGK25199) 92/96 | BafE (ADC79624) 93/96 |
| bflF | 663 | O-methyltransferase | BfmF (BAJ33108) 91/95 | BafAV (AGK25200) 92/97 | BafF (ADC79625) 93/98 |
| bflR | 1947 | AfsR family transcriptional regulator | BfmR (BAJ33109) 84/88 | BafRI (AGK25201) 90/92 | BafG (ADC79626) 92/94 |
| bflG | 762 | thioesterase II | BfmG (BAJ33110) 82/88 | BafT (AGK25202) 92/93 | BafH (ADC79627) 93/94 |
| bflH | 354 | LuxR family transcriptional regulator | BfmH (BAJ33111) 72/79 | BafRII (AGK25203) 86/90 | Orf1 (ADC79628) 89/91 |
| bflI | 963 | fumaryl transferase | BfmI (BAJ33112) 91/95 | Orf17 (AGK25204) 92/96 | Orf2 (ADC79629) 92/96 |
| bflJ | 1080 | fumarate adenylyltransferase | BfmJ (BAJ33113) 90/92 | Orf18 (AGK25205) 94/96 | Orf3 (ADC79630) 94/96 |
表选项
2.3.2 Bafilomycins生物合成途径的推导: 根据Ⅰ型PKS的装配原理、bfl基因簇内各基因的功能注释及文献报道的bafilomycins生物合成途径[20, 26, 34–39],我们对菌株SCSIO 40020中bafilomycins A1和D的生物合成途径进行了推导(图 4)。
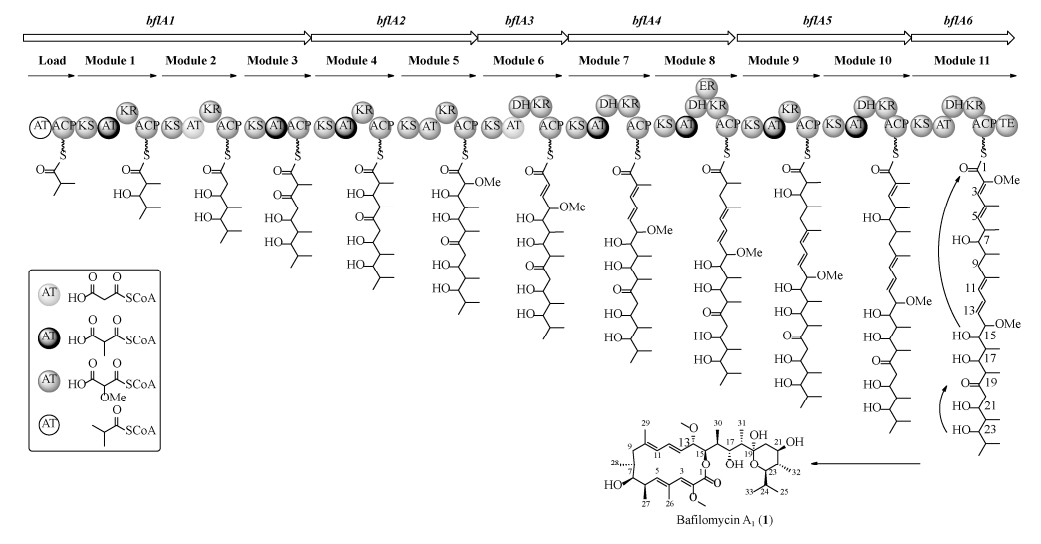 |
| 图 4 Streptomyces sp. SCSIO 40020中bafilomycins可能的生物合成途径 Figure 4 The proposed biosynthetic pathway of bafilomycins from Streptomyces sp. SCSIO 40020. |
| 图选项 |
bfl基因簇内的6个Ⅰ型PKS编码基因共编码了1个起始模块和11个延伸模块。合成途径起始于一个异丁酰辅酶A的装载。AT特异性识别异丁酰辅酶A并将其装载于起始模块的ACP上作为一个四碳单元的起始点进行后续的装配步骤。随后11个延伸模块分别引入7个甲基丙二酰辅酶A、2个丙二酰辅酶A和2个甲氧基丙二酰辅酶A而完成骨架分子的延伸,具体为:模块1中的KS催化缩合一个甲基丙二酰辅酶A,并由KR将C23位的酮基还原为羟基;模块2的KS催化缩合一个丙二酰辅酶A,并由KR将C21位的酮基还原为羟基;模块3的KS催化缩合一个甲基丙二酰辅酶A;模块4的KS催化缩合一个甲基丙二酰辅酶A,并由KR催化C17位的酮基还原为羟基;模块5的KS催化缩合一个甲氧基丙二酰辅酶A,并由KR催化C15位的酮基还原为羟基;模块6的KS催化缩合一个丙二酰辅酶A,并在KR和DH的作用下脱掉C13位的酮基形成C12-C13位的双键;模块7的KS催化缩合一个甲基丙二酰辅酶A,并由KR和DH催化形成C10-C11位的双键;模块8的KS催化缩合一个甲基丙二酰辅酶A,并在KR、DH和ER的作用下将C9位的酮基形成饱和烃结构单元;模块9的KS催化缩合一个甲基丙二酰辅酶A,并由KR催化C7位的酮基还原为羟基;模块10的KS催化缩合一个甲基丙二酰辅酶A,并由KR和DH催化形成C4-C5位的双键;模块11的KS催化缩合一个甲氧基丙二酰辅酶A,并由KR和DH催化形成C2-C3位的双键。最终,装配链末端的硫酯酶(thioesterase,TE)催化C15位的羟基亲核攻击模块11中ACP上的硫酯键而环化释放聚酮链生成bafilomycin A1,其中四氢吡喃环由C23位的羟基攻击C19位的酮基而形成。根据bafilomycin D的结构对其生物合成途径进行推测,一个游离的DH可能参与了模块2的延伸而形成了C20-C21位的双键,进而影响了吡喃环的形成。
2.4 Streptomyces sp. SCSIO 40020中bafilomycins生物合成基因簇的异源表达
2.4.1 菌株SCSIO 40020基因组BAC文库的筛选: 委托武汉八颗星生物科技有限公司构建了菌株SCSIO 40020基因组的BAC表达文库。文库共包含1536个克隆子,平均插入片段约136.7 kb,基因组覆盖率约为25倍。为了克隆到完整的bafilomycins生物合成基因簇,在基因簇上下游分别设计一对特异性引物,利用PCR对所构建的BAC文库进行筛选,从中筛选到21个阳性克隆子。挑取克隆子4-9-H进行末端测序,确认该BAC克隆子所含的插入片段为菌株SCSIO 40020基因组contig 00002中2168509–2279284 nt的DNA序列,包含了完整的bafilomycins生物合成基因簇(2182974–2256522 nt)。
2.4.2 bafilomycins生物合成基因簇的异源表达: 将BAC克隆子4-9-H通过接合转移分别导入到链霉菌S. lividans SBT18、S. coelicolor M1154和S. albus J1074中,筛选得到正确的异源表达菌株S. lividans SBT18/BAC 4-9-H、S. coelicolor M1154/BAC 4-9-H和S. albus J1074/BAC 4-9-H。分别挑取包含BAC 4-9-H的3种异源表达菌株进行发酵检测,同时以携带pSET152空载体的3种链霉菌作为阴性对照。HPLC检测结果显示(图 5),携带BAC 4-9-H的链霉菌S. lividans SBT18和S. coelicolor M1154可产生2个紫外吸收及保留时间与bafilomycin A1和bafilomycin D标准品相同的产物,而携带BAC 4-9-H的S. albus J1074只产生1个紫外吸收及保留时间与bafilomycin A1标准品相同的产物,其中S. coelicolor M1154/BAC 4-9-H异源表达菌株中bafilomycins的产量最高,阴性对照携带空载体的3种链霉菌均没有表达相关产物。由此确认通过构建BAC文库克隆获得了菌株SCSIO 40020中bafilomycins的生物合成基因簇,并在3种链霉菌宿主中异源表达成功。
 |
| 图 5 异源表达菌株发酵产物的HPLC检测 Figure 5 HPLC analysis of metabolic profiles of heterologous expression strains. 1:bafilomycin A1; 2:bafilomycin D. |
| 图选项 |
3 讨论 本文以珠江口沉积物来源的链霉菌Streptomyces sp. SCSIO 40020为研究对象,通过常规发酵分离技术从中获得2个单体化合物bafilomycins A1和D。Bafilomycin A1是bafilomycins类天然产物中的代表化合物,它是一种特异性囊泡型H+-ATP酶(V-ATPase)抑制剂,这种酶是治疗骨质疏松和影响肿瘤转移的重要药物靶点[40–41]。Bafilomycin D最早由Kretschmer等于1985年从S. griseus Tü 2599中分离得到,随后又从多个链霉菌中分离得到,研究表明该化合物具有杀虫活性和抗肿瘤活性[42]。本研究拓展了bafilomycins生产菌的来源,为bafilomycins的工业化发酵生产提供了潜在的菌种资源,同时为研究珠江口环境来源放线菌的次级代谢产物提供了参考。
对菌株SCSIO 40020中bafilomycins的生物合成进行研究,通过生物信息学分析和文献比对确定了bafilomycins的生物合成基因簇(bfl基因簇)并推导了其生物合成途径,进一步利用BAC文库筛选获得了包含完整bfl基因簇的克隆子,通过接合转移实现了bfl基因簇在3种链霉菌S. lividans SBT18、S. coelicolor M1154和S. albus J1074中的异源表达。Ikeda课题组于2013年首次报道了利用BAC载体在链霉菌S. avermitilis SUKA22中异源表达bafilomycins A1和B1[43],但至今未见其他链霉菌宿主成功表达bafilomycins的报道。此外与已报道的bafilomycins生物合成不同的是,bfl基因簇在菌株SCSIO 40020和3种异源表达宿主菌中的主产物均为bafilomycin A1,而无C21位被不同基团取代的结构类似物,推测可能是bfl基因簇中催化bafilomycin A1侧链后修饰的基因bflIJKLM表达量低。后续可通过添加启动子、后修饰基因超量表达或组合生物合成等手段实现bafilomycins的结构多样化,从而获得活性更优的结构类似物。
生物信息学分析揭示微生物基因组中存在多个次级代谢产物生物合成基因簇,且其中许多基因簇在常规条件下表达量极低或不表达,处于隐性状态[44]。因此,研究人员设计、开发了多种基因组挖掘策略(如异源表达、启动子工程、遗传调控等)以试图激活这些隐性基因簇,从而获得新颖结构的天然产物[45–52]。AntiSMASH分析表明菌株SCSIO 40020基因组中共有26个次级代谢产物生物合成基因簇,其中包含12个与已报道生物合成基因簇相似性低于20%的基因簇,提示该菌株具备较大的产生新结构次级代谢产物的潜力。本研究利用BAC文库筛选、接合转移、链霉菌发酵检测等方法实现bfl基因簇在3种链霉菌中的表达,从而成功建立了菌株SCSIO 40020次级代谢产物生物合成基因簇的异源表达体系,为下一步从该菌株中挖掘新颖活性化合物奠定了基础,也为其他大片段Ⅰ型PKS基因簇的异源表达提供了借鉴。
致谢
感谢中国科学院战略性先导科技专项2015年6月珠江口低氧区调查航次提供的沉积物取样帮助,感谢中国科学院南海海洋研究所分析测试中心在化合物鉴定方面提供的帮助以及方春艳博士生和Imran Khan博士生在本文撰写时提供的帮助。
References
| [1] | Choudhary A, Naughton L, Montánchez I, Dobson A, Rai D. Current status and future prospects of marine natural products (MNPs) as antimicrobials. Marine Drugs, 2017, 15(9): 272. DOI:10.3390/md15090272 |
| [2] | Zhang CS, Li WL. Marine Microbiology: new opportunities and new challenges. Microbiology China, 2018, 45(9): 1841-1842. (in Chinese) 张长生, 李文利. 海洋微生物学: 新机遇, 新挑战. 微生物学通报, 2018, 45(9): 1841-1842. |
| [3] | Li JL, Mou XZ, Li WJ. Advances in ecological research of Pearl River Estuarine bacterioplankton. Acta Microbiologica Sinica, 2018, 58(4): 598-607. (in Chinese) 李佳岭, 牟晓真, 李文均. 珠江河口的浮游细菌生态学研究进展. 微生物学报, 2018, 58(4): 598-607. |
| [4] | Li JJ, Jiang X, Jing ZY, Li G, Chen ZZ, Zhou LB, Zhao CY, Liu JX, Tan YH. Spatial and seasonal distributions of bacterioplankton in the Pearl River Estuary: The combined effects of riverine inputs, temperature, and phytoplankton. Marine Pollution Bulletin, 2017, 125(1/2): 199-207. |
| [5] | Sun FL, Wang YS, Wu ML, Wang YT, Li QP. Spatial heterogeneity of bacterial community structure in the sediments of the Pearl River estuary. Biologia, 2011, 66(4): 574-584. DOI:10.2478/s11756-011-0066-6 |
| [6] | Duan L, Li JL, Wang PD, Yin LZ, Li X, Dong L, Xiao M, Li WJ. Comparison study on the isolation of bacterioplankton from surface water of urban nearshore of Pearl River Estuary. Biotic Resources, 2020, 42(5): 522-530. (in Chinese) 段丽, 李佳岭, 王攀登, 尹龄紫, 李鑫, 董雷, 肖敏, 李文均. 珠江口城市段近岸表层水体浮游细菌分离方法比较. 生物资源, 2020, 42(5): 522-530. |
| [7] | Li KL, Liang Z, Chen WH, Luo XW, Fang W, Liao SR, Lin XP, Yang B, Wang JF, Tang L, Liu YH, Zhou XF. Iakyricidins A-D, antiproliferative piericidin analogues bearing a carbonyl group or cyclic skeleton from Streptomyces iakyrus SCSIO NS104. The Journal of Organic Chemistry, 2019, 84(19): 12626-12631. DOI:10.1021/acs.joc.9b01270 |
| [8] | Zhou XF, Liang Z, Li KL, Fang W, Tian YX, Luo XW, Chen YL, Zhan ZK, Zhang T, Liao SR, Liu SW, Liu YH, Fenical W, Tang L. Exploring the natural piericidins as anti-renal cell carcinoma agents targeting peroxiredoxin 1. Journal of Medicinal Chemistry, 2019, 62(15): 7058-7069. DOI:10.1021/acs.jmedchem.9b00598 |
| [9] | Zhang GT, Zhang WJ, Zhang QB, Shi T, Ma L, Zhu YG, Li SM, Zhang HB, Zhao YL, Shi R, Zhang CS. Mechanistic insights into polycycle formation by reductive cyclization in ikarugamycin biosynthesis. Angewandte Chemie International Edition, 2014, 53(19): 4840-4844. DOI:10.1002/anie.201402078 |
| [10] | Staunton J, Weissman KJ. Polyketide biosynthesis: a millennium review. Natural Product Reports, 2001, 18(4): 380-416. DOI:10.1039/a909079g |
| [11] | Fischbach MA, Walsh CT. Assembly-line enzymology for polyketide and nonribosomal peptide antibiotics: logic, machinery, and mechanisms. Chemical Reviews, 2006, 106(8): 3468-3496. DOI:10.1021/cr0503097 |
| [12] | Ma L, Zhang QB, Yang CF, Zhu YG, Zhang LP, Wang LJ, Liu ZW, Zhang GT, Zhang CS. Assembly line and post-PKS modifications in the biosynthesis of marine polyketide natural products. Comprehensive Natural Products Ⅲ.. Amsterdam: Elsevier, 2020: 139-197. |
| [13] | Huang S, Li N, Zhou J, He J. Construction of a new bacterial artificial chromosome (BAC) vector for cloning of large DNA fragments and heterologous expression in Streptomyces. Acta Microbiologica Sinica, 2012, 52(1): 30-37. (in Chinese) 黄胜, 李娜, 周俊, 何璟. 适用于链霉菌大片段基因组DNA克隆和异源表达的细菌人工染色体(BAC)载体的构建及应用. 微生物学报, 2012, 52(1): 30-37. |
| [14] | Zhu MY, He J. Cloning vectors and heterologous hosts for large fragments of actinobacteria genomic DNA. Microbiology China, 2013, 40(10): 1920-1928. (in Chinese) 朱梦奕, 何璟. 放线菌基因组大片段克隆载体及异源表达宿主改造的研究进展. 微生物学通报, 2013, 40(10): 1920-1928. |
| [15] | Deng Q, Zhou L, Luo MZ, Deng ZX, Zhao CM. Heterologous expression of Avermectins biosynthetic gene cluster by construction of a Bacterial Artificial Chromosome library of the producers. Synthetic and Systems Biotechnology, 2017, 2(1): 59-64. DOI:10.1016/j.synbio.2017.03.001 |
| [16] | Hashimoto T, Hashimoto J, Kozone I, Amagai K, Kawahara T, Takahashi S, Ikeda H, Shin-Ya K. Biosynthesis of quinolidomicin, the largest known macrolide of terrestrial origin: identification and heterologous expression of a biosynthetic gene cluster over 200 kb. Organic Letters, 2018, 20(24): 7996-7999. DOI:10.1021/acs.orglett.8b03570 |
| [17] | Yin J, Hoffmann M, Bian XY, Tu Q, Yan F, Xia LQ, Ding XZ, Francis Stewart A, Müller R, Fu J, Zhang YM. Direct cloning and heterologous expression of the salinomycin biosynthetic gene cluster from Streptomyces albus DSM41398 in Streptomyces coelicolor A3(2). Scientific Reports, 2015, 5: 15081. DOI:10.1038/srep15081 |
| [18] | Zhang B, Wang KB, Wang W, Bi SF, Mei YN, Deng XZ, Jiao RH, Tan RX, Ge HM. Discovery, biosynthesis, and heterologous production of streptoseomycin, an anti-microaerophilic bacteria macrodilactone. Organic Letters, 2018, 20(10): 2967-2971. DOI:10.1021/acs.orglett.8b01006 |
| [19] | Tan B, Chen SQ, Zhang QB, Chen YC, Zhu YG, Khan I, Zhang WM, Zhang CS. Heterologous expression leads to discovery of diversified lobophorin analogues and a flexible glycosyltransferase. Organic Letters, 2020, 22(3): 1062-1066. DOI:10.1021/acs.orglett.9b04597 |
| [20] | Li Z, Du L, Zhang W, Zhang XW, Jiang YY, Liu K, Men P, Xu HF, Fortman JL, Sherman DH, Yu B, Gao S, Li SY. Complete elucidation of the late steps of bafilomycin biosynthesis in Streptomyces lohii. Journal of Biological Chemistry, 2017, 292(17): 7095-7104. DOI:10.1074/jbc.M116.751255 |
| [21] | Werner G, Hagenmaier H, Albert K, Kohlshorn H. The structure of the bafilomycins, a new group of macrolide antibiotics. Tetrahedron Letters, 1983, 24(47): 5193-5196. DOI:10.1016/S0040-4039(00)88394-X |
| [22] | Takahashi Y. Genus Kitasatospora, taxonomic features and diversity of secondary metabolites. The Journal of Antibiotics, 2017, 70(5): 506-513. DOI:10.1038/ja.2017.8 |
| [23] | Carr G, Williams DE, Díaz-Marrero AR, Patrick BO, Bottriell H, Balgi AD, Donohue E, Roberge M, Andersen RJ. Bafilomycins produced in culture by Streptomyces spp. isolated from marine habitats are potent inhibitors of autophagy. Journal of Natural Products, 2010, 73(3): 422-427. DOI:10.1021/np900632r |
| [24] | Yu ZG, Zhao LX, Jiang CL, Duan YW, Wong L, Carver KC, Schuler LA, Shen B. Bafilomycins produced by an endophytic actinomycete Streptomyces sp. YIM56209. The Journal of Antibiotics, 2011, 64(1): 159-162. DOI:10.1038/ja.2010.147 |
| [25] | Ding N, Jiang Y, Han L, Chen X, Ma J, Qu XD, Mu Y, Liu J, Li LY, Jiang CL, Huang XS. Bafilomycins and odoriferous sesquiterpenoids from Streptomyces albolongus isolated from Elephas maximus feces. Journal of Natural Products, 2016, 79(4): 799-805. DOI:10.1021/acs.jnatprod.5b00827 |
| [26] | Nara A, Hashimoto T, Komatsu M, Nishiyama M, Kuzuyama T, Ikeda H. Characterization of bafilomycin biosynthesis in Kitasatospora setae KM-6054 and comparative analysis of gene clusters in Actinomycetales microorganisms. The Journal of Antibiotics, 2017, 70(5): 616-624. DOI:10.1038/ja.2017.33 |
| [27] | Zhao ZL, Shi T, Xu M, Brock NL, Zhao YL, Wang YM, Deng ZX, Pang XH, Tao MF. Hybrubins: bipyrrole tetramic acids obtained by crosstalk between a truncated undecylprodigiosin pathway and heterologous tetramic acid biosynthetic genes. Organic Letters, 2016, 18(3): 572-575. DOI:10.1021/acs.orglett.5b03609 |
| [28] | Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology, 2011, 4(2): 207-215. DOI:10.1111/j.1751-7915.2010.00219.x |
| [29] | Zaburannyi N, Rabyk M, Ostash B, Fedorenko V, Luzhetskyy A. Insights into naturally minimised Streptomyces albus J1074 genome. BMC Genomics, 2014, 15: 97. DOI:10.1186/1471-2164-15-97 |
| [30] | Ma L, Zhang WJ, Zhu YG, Wu ZC, Saurav K, Huang H, Zhang CS. Isolation of Actinobacteria with antibiotic activity associated with Soft Coral Nephthea sp.. Acta Microbiologica Sinica, 2013, 53(10): 1063-1071. (in Chinese) 马亮, 张文军, 朱义广, 吴正超, Saurav Kumar, 黄晖, 张长生. 永兴岛白穗软珊瑚共附生放线菌筛选及部分活性次级代谢产物的鉴定. 微生物学报, 2013, 53(10): 1063-1071. |
| [31] | Jia HL, Xie J, Wu SY, He GF. Temporal and spatial variations of salinity in Pearl River estuary in recent years. Transactions of Oceanology and Limnology, 2011(2): 142-146. (in Chinese) 贾后磊, 谢健, 吴桑云, 何桂芳. 近年来珠江口盐度时空变化特征. 海洋湖沼通报, 2011(2): 142-146. DOI:10.3969/j.issn.1003-6482.2011.02.020 |
| [32] | Wei G, Su C, Zhang DJ, Zong ZY, Si CC, Tao LM. Isolation, purification and structure identification of secondary metabolites produced by marine actinomycete Y12-26. Chinese Journal of Antibiotics, 2011, 36(8): 571-575. (in Chinese) 魏刚, 苏超, 张道敬, 宗志友, 斯聪聪, 陶黎明. 海洋放线菌Y12-26代谢产物中bafilomycins分离纯化及结构鉴定. 中国抗生素杂志, 2011, 36(8): 571-575. |
| [33] | Baker GH, Brown PJ, Dorgan RJJ, Everett JR, Ley SV, Slawin AMZ, Williams DJ. A conformational study of bafilomycin A1 by X-ray crystallography and nmr techniques. Tetrahedron Letters, 1987, 28(45): 5565-5568. DOI:10.1016/S0040-4039(00)96783-2 |
| [34] | Hwang JY, Kim HS, Kim SH, Oh HR, Nam DH. Organization and characterization of a biosynthetic gene cluster for bafilomycin from Streptomyces griseus DSM 2608. AMB Express, 2013, 3(1): 24. DOI:10.1186/2191-0855-3-24 |
| [35] | Zhang W, Fortman JL, Carlson JC, Yan JY, Liu Y, Bai FL, Guan WN, Jia JY, Matainaho T, Sherman DH, Li SY. Characterization of the bafilomycin biosynthetic gene cluster from Streptomyces lohii. ChemBioChem, 2013, 14(3): 301-306. DOI:10.1002/cbic.201200743 |
| [36] | Hanh NPK, Hwang JY, Nam DH. Biosynthesis of methoxymalonyl-acyl carrier protein (ACP) as an extender unit for bafilomycin polyketide in Streptomyces griseus DSM 2608. Biotechnology and Bioprocess Engineering, 2018, 23(6): 693-703. DOI:10.1007/s12257-018-0427-x |
| [37] | Hanh NPK, Hwang JY, Oh HR, Kim GJ, Choi H, Nam DH. Biosynthesis of 2-amino-3-hydroxycyclopent-2-enone moiety of bafilomycin in Kitasatospora cheerisanensis KCTC2395. Journal of Microbiology: Seoul, Korea, 2018, 56(8): 571-578. |
| [38] | Li Z, Li S, Du L, Zhang XW, Jiang YY, Liu WH, Zhang W, Li SY. Engineering bafilomycin high-producers by manipulating regulatory and biosynthetic genes in the marine bacterium Streptomyces lohii. Marine Drugs, 2021, 19(1): 29. DOI:10.3390/md19010029 |
| [39] | Molloy EM, Tietz JI, Blair PM, Mitchell DA. Biological characterization of the hygrobafilomycin antibiotic JBIR-100 and bioinformatic insights into the hygrolide family of natural products. Bioorganic & Medicinal Chemistry, 2016, 24(24): 6276-6290. |
| [40] | Bowman EJ, Siebers A, Altendorf K. Bafilomycins: a class of inhibitors of membrane ATPases from microorganisms, animal cells, and plant cells. Procedings of the National Academy of Science of the United States of America, 1988, 85(21): 7972-7976. DOI:10.1073/pnas.85.21.7972 |
| [41] | Dr?se S, Altendorf K. Bafilomycins and concanamycins as inhibitors of V-ATPases and P-ATPases. Journal of Experimental Biology, 1997, 200(1): 1-8. DOI:10.1242/jeb.200.1.1 |
| [42] | Kretschmer A, Dorgerloh M, Deeg M, Hagenmaier H. The structures of novel insecticidal macrolides: bafilomycins D and E, and oxohygrolidin. Agricultural and Biological Chemistry, 1985, 49(8): 2509-2511. |
| [43] | Komatsu M, Komatsu K, Koiwai H, Yamada Y, Kozone I, Izumikawa M, Hashimoto J, Takagi M, Omura S, Shin-Ya K, Cane DE, Ikeda H. Engineered Streptomyces avermitilis host for heterologous expression of biosynthetic gene cluster for secondary metabolites. ACS Synthetic Biology, 2013, 2(7): 384-396. DOI:10.1021/sb3001003 |
| [44] | Hou LK, Li HY, Li WL. Activation of cryptic microbial biosynthetic gene clusters and structure-guided natural product discovery. Acta Microbiologica Sinica, 2017, 57(11): 1722-1734. (in Chinese) 侯路宽, 李花月, 李文利. 隐性次级代谢产物生物合成基因簇的激活及天然产物定向发现. 微生物学报, 2017, 57(11): 1722-1734. |
| [45] | Ziemert N, Alanjary M, Weber T. The evolution of genome mining in microbes - a review. Natural Product Reports, 2016, 33(8): 988-1005. DOI:10.1039/C6NP00025H |
| [46] | Chen RD, Zhang QB, Tan B, Zheng LJ, Li HX, Zhu YG, Zhang CS. Genome mining and activation of a silent PKS/NRPS gene cluster direct the production of totopotensamides. Organic Letters, 2017, 19(20): 5697-5700. DOI:10.1021/acs.orglett.7b02878 |
| [47] | Liu J, Xie XL, Li SM. Guanitrypmycin biosynthetic pathways imply cytochrome P450 mediated regio- and stereospecific guaninyl-transfer reactions. Angewandte Chemie International Edition, 2019, 58(33): 11534-11540. DOI:10.1002/anie.201906891 |
| [48] | Liu J, Xie XL, Li SM. Increasing cytochrome P450 enzyme diversity by identification of two distinct cyclodipeptide dimerases. Chemical Communications: Cambridge, England, 2020, 56(75): 11042-11045. DOI:10.1039/D0CC04772D |
| [49] | Liu W, Zhang WJ, Jin HB, Zhang QB, Chen YC, Jiang XD, Zhang GT, Zhang LP, Zhang WM, She ZG, Zhang CS. Genome mining of marine-derived Streptomyces sp. SCSIO 40010 leads to cytotoxic new polycyclic tetramate macrolactams. Marine Drugs, 2019, 17(12): 663. DOI:10.3390/md17120663 |
| [50] | Saha S, Zhang WJ, Zhang GT, Zhu YG, Chen YC, Liu W, Yuan CS, Zhang QB, Zhang HB, Zhang LP, Zhang WM, Zhang CS. Activation and characterization of a cryptic gene cluster reveals a cyclization cascade for polycyclic tetramate macrolactams. Chemical Science, 2017, 8(2): 1607-1612. DOI:10.1039/C6SC03875A |
| [51] | Yao TT, Liu J, Liu ZZ, Li T, Li HY, Che Q, Zhu TJ, Li DH, Gu QQ, Li WL. Genome mining of cyclodipeptide synthases unravels unusual tRNA-dependent diketopiperazine-terpene biosynthetic machinery. Nature Communications, 2018, 9: 4091. DOI:10.1038/s41467-018-06411-x |
| [52] | Zhang WJ, Zhang GT, Zhang LP, Liu W, Jiang XD, Jin HB, Liu ZW, Zhang HB, Zhou AH, Zhang CS. New polycyclic tetramate macrolactams from marine-derived Streptomyces sp. SCSIO 40060. Tetrahedron, 2018, 74(47): 6839-6845. DOI:10.1016/j.tet.2018.10.007 |
