杨金仙1, 吴成1, 王春晓1

 , 田进1, 许引虎2, 邱树毅1
, 田进1, 许引虎2, 邱树毅1 1. 贵州大学酿酒与食品工程学院, 贵州省发酵工程与生物制药重点实验室, 贵州 贵阳 550025;
2. 国家酵母技术研究推广中心, 湖北 宜昌 443003
收稿日期:2021-01-03;修回日期:2021-03-15;网络出版日期:2021-03-29
基金项目:国家自然科学基金(31801523);贵州省科技计划(黔科合支撑[2019]2263号,黔科合平台人才[2018]5781号);贵州省教育厅青年科技人才成长项目(黔教合KY字[2018]120);贵州省发酵工程与白酒酿造人才基地(黔人领发[2018]3号);贵州大学引进人才科研项目(贵大人基合字[2017]44号)
*通信作者:王春晓, E-mail: cxwang@gzu.edu.cn.
摘要:[目的] 研究贵州紫云县刺葡萄自然发酵过程中野生酿酒酵母的基因型多样性,分析不同基因型酵母在不同发酵时期的动态变化,为优良酿酒酵母资源的开发利用提供理论依据。[方法] 采用Interdelta指纹图谱分析方法和微卫星分子标记法,研究贵州紫云县刺葡萄自然发酵中野生酿酒酵母的基因型多样性,并通过DPS软件分析不同基因型之间的遗传关系。[结果] 贵州紫云县刺葡萄自然发酵中共分离野生酿酒酵母75株,经Interdelta指纹图谱分析方法和微卫星分子标记法鉴定为10个基因型,其中基因型6、9、10、11、14、15、16为野生酿酒酵母独有的7个基因型,7、17和18为野生与商业酿酒酵母共有的3个基因型,此外,本研究所用其他商业酿酒酵母另有独有的9个基因型(1、2、3、4、5、8、12、13和19)。75株野生酿酒酵母中基因型17的占比最高为36%,其次为基因型10占比为13.3%。在自然发酵过程中不同基因型呈现此消彼长的变化,每一种基因型的菌株细胞密度在104-107 CFU/mL之间。[结论] 贵州紫云县刺葡萄自然发酵样品展现了丰富的酿酒酵母菌株基因型多样性,其中基因型10和17为主导基因型,该研究为贵州刺葡萄优良野生酿酒酵母资源的开发奠定了基础。
关键词:Interdelta指纹图谱方法微卫星位点基因型系统发育分析
Genotype diversity analysis of indigenous Saccharomyces cerevisiae during spontaneous fermentations of Vitis davidii F?ex in Ziyun, Guizhou
Jinxian Yang1, Cheng Wu1, Chunxiao Wang1

 , Jin Tian1, Yinhu Xu2, Shuyi Qiu1
, Jin Tian1, Yinhu Xu2, Shuyi Qiu1 1. Province Key Laboratory of Fermentation Engineering and Biopharmacy, School of Liquor and Food Engineering, Guizhou University, Guiyang 550025, Guizhou Province, China;
2. National Center for Yeast Technology Research and Promotion, Yichang 443003, Hubei Province, China
Received: 3 January 2021; Revised: 15 March 2021; Published online: 29 March 2021
*Corresponding author: Chunxiao Wang, E-mail: cxwang@gzu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31801523), by the Guizhou Province Science and Technology Project ([2019]2263, [2018]5781), by the Guizhou Education Bureau Research Program (KY[2018]120), by the Talent Base of Fermentation Engineering and Liquor Making in Guizhou Province ([2018]3) and by the Guizhou University Talent Introduction Research Project ([2017]44)
Abstract: [Objective] The aim is to study the genotype diversity of indigenous Saccharomyces cerevisiae isolated from spontaneous fermentations of Vitis davidii F?ex in Ziyun County of Guizhou Province, to analyze the dynamic changes of different S. cerevisiae genotypes at different fermentation periods, and to provide theoretical references for the industrial application of excellent S. cerevisiae resources. [Methods] We applied interdelta fingerprinting analysis and microsatellite marker analysis to study the genotype diversity of indigenous S. cerevisiae during spontaneous fermentations of Vitis davidii F?ex in Ziyun County of Guizhou Province. DPS software was used to analyze the genetic relationship among different genotypes. [Results] We totally isolated 75 indigenous S. cerevisiae strains, and identified them into 10 genotypes by interdelta fingerprinting analysis and microsatellite marker analysis. Genotypes 6, 9, 10, 11, 14, 15 and 16 are the peculiar seven genotypes owned by indigenous S. cerevisiae strains. Genotypes 7, 17 and 18 are the mutual three genotypes owned by both indigenous and commercial S. cerevisiae strains. Furthermore, other commercial S. cerevisiae strains in the study showed nine other specific genotypes including genotypes 1, 2, 3, 4, 5, 8, 12, 13 and 19. The genotype 17 took the highest proportion in the 75 isolates (36%) followed by the genotype 10 (13.3%). Different genotypes showed waxing and waning ratio changes during fermentations, and the cell density of each genotype varied among 104-107CFU/mL. [Conclusion] Spontaneous fermentation samples of Vitis davidii F?ex in Ziyun County of Guizhou Province showed rich genotype diversity of S. cerevisiae strains. Genotypes 10 and 17 were the predominant S. cerevisiae genotypes. This study laid a basis for the development of excellent indigenous S. cerevisiae resources of Vitis davidii F?exin Guizhou Province.
Keywords: Interdelta fingerprinting analysismicrosatellite sitegenotypephylogenetic analysis
自1845年巴斯德发现了酵母菌后,以酒精发酵为基础的葡萄酒酿造工艺开始作为一门科学被研究和确立。在20世纪50年代,新世界葡萄酒厂开始使用外源酵母进行接种发酵,10年后活性干酵母产品推出并迅速推广到欧洲各葡萄酒酿造国家,现今,全球每年约有40万t商业活性干酵母生产[1]。选用商业活性干酵母接种进行葡萄酒酒精发酵时,所接种菌株一般会形成优势菌群,抑制野生非酿酒酵母在发酵中后期的生长代谢,可以避免由自然发酵引发的发酵缓慢、发酵停滞和风味败坏等风险。因此,世界各葡萄酒产区一般通过接种商业酵母酿造葡萄酒以保证葡萄酒质量的可预测性和稳定性[2]。葡萄酒酿造中主导酵母菌的生长代谢对葡萄酒的化学组成和感官特性有十分重要的影响[3-4],研究指出单一商业酿酒酵母的大范围使用会导致所酿造的葡萄酒风格单一,不利于葡萄酒产区特色和风格的形成[2, 5]。Nikolaou等(2006)提出,优良本土野生酿酒酵母通过筛选后更适合作为该葡萄酒产区的商业发酵剂使用,因为本产区筛选的野生酿酒酵母更能适应葡萄酒产区原本的风土条件,也更容易在发酵中占据主导地位,而且选用本产区筛选的野生酿酒酵母可以确保每个葡萄酒产区酿造葡萄酒的典型风格[6]。Fleet (2008)提出通过合理地管理酵母菌可以创造性地设计葡萄酒的风格,从而解决葡萄酒正面临着的国际竞争,顺应人们更倾向于饮用健康和具有区域特色风格的葡萄酒的消费观念[3]。因此,近10年研究者们聚焦于野生酵母菌的挖掘利用,相关研究表明野生酵母菌相对于商业酵母所酿造的葡萄酒感官特性和地域特征更明显[7-8]。中国地域辽阔,地形、气候和土壤差异形成了复杂的生态环境,因此孕育了丰富的酵母资源,具有地域特色的野生酵母资源的研发对形成中国各产区风格的葡萄酒有着十分重要的意义。
野生酵母资源研发的首要问题是确定它们在种水平甚至是种内水平上的分子身份,以便对后续发酵等应用提供可追踪监测的依据。酿酒酵母(Saccharomyces cerevisiae)种内分子鉴定常用的方法包括RAPD (random amplified polymorphic DNA)标记法、mtDNA-RFLP (restriction fragment length polymorphism of mtDNA)分析、Interdelta指纹图谱分析和微卫星分子标记法等[9],这些方法均为片段长度多态性分析方法,可展现酿酒酵母的基因型多样性。Interdelta指纹图谱分析主要研究酿酒酵母基因组中的重复序列——delta序列的分布特征,delta序列的数目和位置具有丰富的种内差异,是用于区分酿酒酵母菌株的经典方法[10]。由于Interdelta指纹图谱分析操作方法简便且展示的多态性丰富[11-13],在酿酒酵母的种内区分中得到广泛的应用[10, 14-19]。微卫星分子标记法主要依赖微卫星位点的高度多态性和特异性进行分子鉴定,可在种内不同个体间展现差异。相比于其他片段长度多态性分析方法,微卫星分子标记表现出较优的种内分型效果[20-24]。本研究采用经典的Interdelta指纹图谱分析和分型效果优良的微卫星分子标记法对贵州省紫云县刺葡萄自然发酵过程中分离的野生酿酒酵母菌株进行基因型多样性分析,为野生酿酒酵母资源的开发利用提供理论基础。
1 材料和方法 1.1 实验材料
1.1.1 菌株: 实验菌株为93株酿酒酵母,由本实验室保存。其中75株分离自2017年贵州紫云县刺葡萄自然发酵过程中(现保存于贵州大学酿酒与食品工程学院贵州省发酵工程与生物制药重点实验室),18株商业酿酒酵母(71B、F33、CECA、RV171、D254、EC1118、KD、2323、CEC01、VIC、BO213、ADT、RV002、RC212、BV818、F15、AC和X16)作为参考菌株,其中CECA、RV171、CEC01、VIC、ADT、RV002和BV818购自安琪酵母股份有限公司,其他11株商业酿酒酵母购自法国Lallemand集团。
1.1.2 主要试剂: 本实验所用6对PCR扩增引物(具体见方法部分)均由宝日医(TaKaRa)生物技术有限公司合成。MgCl2、Buffer、dNTPmix、Taq DNA聚合酶和DNA marker均购自宝日医(TaKaRa)生物技术有限公司,核酸染料Genegreen购自天根生物科技(北京)有限公司,HyAgarose琼脂糖购自厦门太阳马生物工程有限公司,WL营养琼脂培养基购自上海博威生物科技有限公司,其他常规化学试剂为国产分析纯产品。
1.1.3 主要仪器: YXQ-75SⅡ立式压力蒸汽灭菌器(博讯实业有限公司医疗设备厂,上海);SPX-250B-Z生化培养箱(博讯实业有限公司医疗设备厂,上海);IMS-20制冰机(雪科电器有限公司,常熟);JXFSTPRP-24细胞破碎仪(净信实业发展有限公司,上海);MIT-100恒温混匀仪(米欧仪器有限公司,杭州);Ti-s尼康显微镜(尼康有限股份公司,日本);Allegra X-30R高速冷冻离心机(贝克曼库尔特有限公司(Beckman Coulter),美国);CFX Connect Real-Time System PCR仪(伯乐仪器设备公司(Bio-Rad),美国);核酸电泳仪(伯乐仪器设备公司(Bio-Rad),美国);Bio-Bset140E凝胶成像仪(西盟国际公司(SIM),美国)。
1.2 样品采集与酿酒酵母的初步筛选 2017年8月于贵州省紫云县火花镇(东经105°56'59″,北纬25°38'47″)采摘刺葡萄(2个品种:紫秋和紫秋1号),人工无菌破碎后获取葡萄醪液,调节为两种含糖量水平(Ⅰ为18 °Bx经检测为143 g/L、Ⅱ为24 °Bx经检测为200 g/L)后在28 ℃下进行自然发酵。通过CO2失重法监测发酵进程,在自然发酵期间定期取发酵液在WL鉴定培养基上进行涂布培养,根据稀释涂布系数进行菌落计数。28-30 ℃培养5 d后所有在WL鉴定培养基上表现出圆锥形凸起菌落特征和在显微观察下表现卵圆形细胞形态的酵母均初步认定为酿酒酵母,所分离酵母菌经WL鉴定培养基纯化后保存于-80 ℃冰箱。
1.3 酿酒酵母的种水平鉴定 酿酒酵母的种水平和种内基因型区分所用的DNA获取[26]。首先,将1.2.1保存的酿酒酵母划线接种在YPD固体培养基(10 g/L酵母浸出粉、20 g/L蛋白胨、20 g/L葡萄糖、20 g/L琼脂)于28-30 ℃培养2 d。然后挑取适量菌体于装有200 μL酵母裂解液和0.2 g石英砂的2 mL离心管中,使用细胞破碎仪(振荡频率70 Hz)破碎1 min后,冰浴30 s,并重复破碎和冰浴操作4次。DNA提取后续操作按照周小玲等所述程序进行[26]。
所有初步认定为酿酒酵母的菌落全部通过5.8S-ITS-RFLP技术进行种水平鉴定,首先采用ITS1和ITS4引物进行PCR扩增反应,扩增程序参考Wang等所述程序进行[25],所得PCR产物进一步采用Hae Ⅲ限制性核酸内切酶酶切,酶切体系为20 μL,包含10 μL PCR产物、7 μL双蒸水、2 μL酶反应配套缓冲液和1 μL内切酶。酶切产物经1.5% HyAgarose琼脂糖凝胶电泳进行检测,电泳液为0.5×TBE缓冲液,电泳条件为90 V稳压50 min,使用凝胶成像仪拍照记录电泳图谱。5.8S-ITS-RFLP图谱为290+230+160+130的菌株确认为酿酒酵母,并随机挑选3株代表性菌株进行26S rRNA基因D1/D2序列分析[25]。
1.4 Interdelta指纹图谱分析方法 采用通用DNA引物delta12 (5'-TCAACAAT GGAATCCCAAC-3')和delta21 (5'-CATCTTAACACCGTATATGA-3')作为扩增引物进行酿酒酵母种内区分,该对引物位于delta序列(编号YOLC delta3)的保守区[27]。PCR扩增反应[28]体系总体积为20 μL,包含2.5 mmol/L MgCl2、1×酶反应配套缓冲液、0.2 mmol/L dNTPs、0.5 μmol/L上下游引物、1.6 U Taq DNA聚合酶和1 μL DNA模板。PCR扩增程序为:95 ℃预变性4 min;95 ℃变性30 s,46 ℃退火30 s,72 ℃延伸90 s,35个循环;72 ℃延伸10 min。琼脂糖凝胶电泳条件同1.3所述,使用凝胶成像仪拍照记录电泳图谱。
1.5 微卫星分子标记法 采用微卫星位点ScAAT1、C4和SCYOR267C进行酿酒酵母的种内区分,微卫星位点ScAAT1的上游引物序列为5'-AAAAGCGTAAGCAATGGTGTAGAT-3',ScAAT1的下游引物序列为5'-AGCATGACCTTTACAATTTGATAT-3'[20],微卫星位点C4的上游引物序列为5'-GAGAAAAA TGCTGTTTATTCTGACC-3',C4的下游引物序列为5'-CCTCCGGGACGTGAAATAAC-3'[23],微卫星位点SCYOR267C的上游引物序列为5'-TACTAACGTCAACACTGCTGCCAA-3',SCYOR267C的下游引物序列为5'-GGATCTACTTGCAGTATACGGG-3'[20]。PCR扩增反应[23]体系总体积为15 μL,包含2 mmol/L MgCl2、1×酶反应配套缓冲液、0.2 mmol/L dNTPs、0.2 μmol/L上下游引物(SCYOR267C位点引物使用浓度为0.13 μmol/L)、0.3 U Taq DNA聚合酶和1 μL DNA模板。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸7 min。琼脂糖凝胶电泳条件同1.3所述,使用凝胶成像仪拍照记录电泳图谱。
1.6 系统发育分析 根据凝胶成像仪记录的电泳图谱进行图谱条带的人工读取和准确归类,将每种图谱类型的所有条带按有和无进行1和0转换,使用DPS数据处理系统进行聚类分析,在聚类分析中采用欧氏距离作为聚类距离,采用类平均法(unweighted pair-group method with arithmetic means,UPGMA)作为聚类方法。
1.7 基因型分析和计数方法 根据Interdelta指纹图谱分析和微卫星分子标记的结果,每个酿酒酵母菌株获取4种不同分析方法下的图谱类型,将4种图谱类型进行结合分析,凡有一种图谱类型不同的即归类为不同基因型。所有酿酒酵母都通过种内水平分析确定最终的基因型,每种基因型酵母的计数是根据该基因型菌落所占的比例乘酿酒酵母在该时期的密度值所得。
2 结果和分析 2.1 野生酿酒酵母的初步筛选 从贵州省紫云县刺葡萄自然发酵中共分离鉴定出75株野生酿酒酵母,通过酿酒酵母5.8S rRNA基因的ITS扩增获得810 bp产物,使用Hae Ⅲ限制性内切酶进一步将扩增产物进行酶切,经检测所有的ITS扩增产物都为290+230+160+130 (图 1),说明通过WL培养基分离保存的75株酵母全部为酿酒酵母。随机选择3株代表性菌株进行26S rRNA基因D1/D2区域序列分析(序列登录号为MK581077、MK581099和MK581135),结果表明其与酿酒酵母模式菌株的序列相似性高于99%。在酿酒酵母菌种鉴定的基础上,下一步结合Interdelta指纹图谱分析和微卫星分子标记法进行野生酿酒酵母和商业酿酒酵母的种内区分。
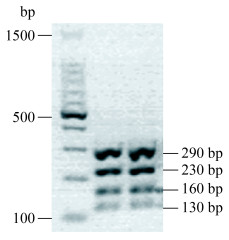 |
| 图 1 酿酒酵母5.8S-ITS-RFLP分析HaeⅢ酶切图谱 Figure 1 The Hae Ⅲ endonuclease digestion profile of S. cerevisiae by 5.8S-ITS-RFLP analysis. |
| 图选项 |
2.2 Interdelta指纹图谱分析 Interdelta指纹图谱分析选用具有丰富扩增多态性的delta12和delta21引物对进行93株酿酒酵母(包含75株野生酿酒酵母和18株商业酿酒酵母)的种内差异分析(图 2),由图中可看出该引物对扩增后形成两种类型的条带:较亮的1000 bp以上的条带和相对较暗且扩增不稳定的1000 bp以下的条带。
 |
| 图 2 酿酒酵母Interdelta指纹图谱分析结果 Figure 2 Interdelta fingerprinting analysis results of S. cerevisiae. A and B: the eight different electrophoresis profiles of selected S. cerevisiae strains by interdelta fingerprinting analysis. M2: 100 bp DNA marker; M3: 250 bp DNA marker; I1-I8: the profiles' name. C: the cluster dendrogram of eight electrophoresis profiles got by interdelta fingerprinting analysis. |
| 图选项 |
根据Interdelta指纹图谱分析,可将全部待测酿酒酵母分为I1-I8共8个图谱类型,8类图谱型菌株的集中电泳分析如图 2所示:I1类图谱型条带为3000+1400+600+450+230+180 (下划线为电泳图谱中的亮条带,下同),有22株菌(包含21株野生酿酒酵母和1株商业酿酒酵母VIC);I2为3000+1400+700+420+220+100,有54株菌(包含48株野生酿酒酵母和6株商业酿酒酵母CEC01、ADT、RV002、BV818、F33和BD213);I3类为640+200,有1株菌(商业酿酒酵母CECA);I4类为3000+2250+1400+870+700+550,有1株菌(野生酿酒酵母);I5类为3000+1400+870+520+470+ 430+250+200,有1株菌(野生酿酒酵母);I6类为3000+2250+1400+680+550+400+300+100,有1株菌(商业酿酒酵母RV171);I7类为3000+2250+1400+900+550+400,有1株菌(商业酿酒酵母EC1118);I8类为3000+1400+700+550+420+320+200+170,有12株菌(包含48株野生酿酒酵母和6株商业酿酒酵母CEC01、ADT、RV002、BV818、F33和BO213)。将8类图谱条带进行聚类分析,如图 2-C所示,I4类与I7类遗传关系最近,而I5类与其他类型的遗传关系最远,如前所述I3、I6、I7类为商业酿酒酵母所特有的图谱类型。
2.3 微卫星分子标记分析 酿酒酵母的16条染色体基因序列上存在丰富的微卫星位点,目前已经报道用于基因型分析的微卫星位点有50余个,本研究通过比对等位基因数目、扩增产物大小和检测获得的基因型数目,采用了具有高度多态性的3个微卫星位点ScAAT1、C4和SCYOR267C对93株酿酒酵母进行种内差异分析,每个微卫星位点展现了不同的区分能力。
根据微卫星位点ScAAT1可将全部待测酿酒酵母分为Sa1-Sa5共5个图谱类型,5类图谱型菌株的集中电泳分析如图 3所示:Sa1类为180(下划线为电泳图谱中的亮条带,下同),有16株菌(包含10株野生酿酒酵母和6株商业酿酒酵母71B、RV002、VIC、ADT、RV171、F33);Sa2类为210,是6株商业酿酒酵母特有的图谱类型(2323、AC、D254、F15、RC212、X16);Sa3类图谱条带为150,有24株菌(全部为野生酿酒酵母);Sa4类为180+250,有46株菌(包含41株野生酿酒酵母和5株商业酿酒酵母CEC01、CECA、BO213、BV818、KD);Sa5类为140+180,是商业酵母EC1118特有的图谱类型。将5类图谱条带进行聚类分析,如图 3-C所示,Sa1类、Sa2类与Sa4类遗传关系最近聚为一类,而Sa3类与其他类型的遗传关系最远。
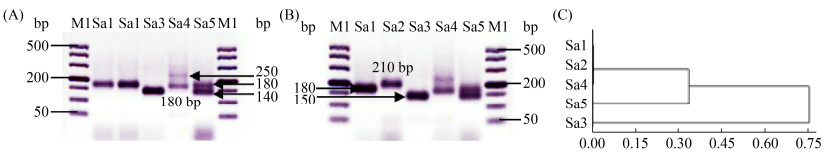 |
| 图 3 酿酒酵母微卫星位点ScAAT1图谱分析结果 Figure 3 Electrophoresis profiles analysis results using microsatellite site ScAAT1 of S. cerevisiae. A and B: the five different electrophoresis profiles of selected S. cerevisiae strains by microsatellite site ScAAT1 analysis. M1: 500DL DNA marker; Sa1-Sa5: the profiles' name. C: the cluster dendrogram of five electrophoresis profiles got by microsatellite site ScAAT1 analysis. |
| 图选项 |
微卫星位点C4将全部待测酿酒酵母分为Cg1-Cg4共4个图谱类型,4类图谱型菌株的集中电泳分析如图 4所示:Cg1类图谱条带为220(下划线为电泳图谱中的亮条带,下同),是6株商业酿酒酵母特有的图谱类型(71B、RV002、VIC、ADT、CEC01、CECA);Cg2类图谱条带为400+300+220,是7株商业酿酒酵母特有的图谱类型(RV171、2323、AC、D254、F15、RC212、X16);Cg3类图谱条带为300,有64株菌(包含59株野生酿酒酵母和5株商业酿酒酵母F33、BO213、BV818、KD、EC1118);Cg4类图谱条带为300+400,有16株菌(全部为野生酿酒酵母)。将4类图谱条带进行聚类分析,如图 4-C所示,Cg2类与Cg4类遗传关系最近,而Cg1类与其他类型的遗传关系最远。
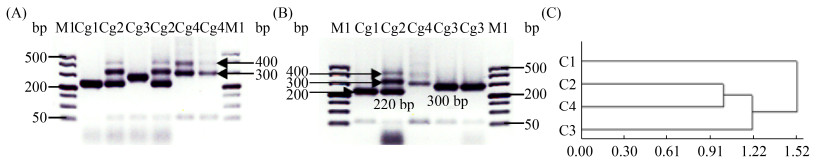 |
| 图 4 酿酒酵母微卫星位点C4图谱分析结果 Figure 4 Electrophoresis profiles analysis results using microsatellite site C4 of S. cerevisiae. A and B: the four different electrophoresis profiles of selected S. cerevisiae strains by microsatellite site C4 analysis. M1: 500DL DNA marker; Cg1-Cg4: the profiles' name. C: the cluster dendrogram of four electrophoresis profiles got by microsatellite site C4 analysis. |
| 图选项 |
微卫星位点SCYOR267C将全部待测酿酒酵母分为Sy1-Sy4共4个图谱类型,4类图谱型菌株的集中电泳分析如图 5所示:Sy1图谱条带为400+350(下划线为电泳图谱中的亮条带,下同),有17株菌(包含16株野生酿酒酵母和1株商业酿酒酵母CEC01);Sy2类为350,有13株菌(包含6株野生酿酒酵母和7株商业酿酒酵母71B、2323、AC、D254、F15、RC212、X16);Sy3类为300,有60株菌(包含53株野生酿酒酵母和7株商业酿酒酵母RV002、RV171、F33、CECA、BO213、BV818、KD);Sy4类图谱条带为350+280,是3株商业酿酒特有的图谱类型(VIC、ADT、EC1118)。将4类微卫星位点图谱条带进行聚类分析,如图 5-C所示,Sy1类与Sy2类遗传关系最近,而Sy4类与其他类型的遗传关系远。
 |
| 图 5 酿酒酵母微卫星位点SCYOR267C图谱分析结果 Figure 5 Electrophoresis profiles analysis results using microsatellite site SCYOR267C of S. cerevisiae. A and B: the four different electrophoresis profiles of selected S. cerevisiae strains by microsatellite site SCYOR267C analysis. M1: 500DL DNA marker; Sy1-Sy4: the profiles' name. C: the cluster dendrogram of eight electrophoresis profiles got by microsatellite site SCYOR267C analysis. |
| 图选项 |
2.4 酿酒酵母基因型多样性分析 将Interdelta指纹图谱分析和微卫星分子标记法获取的全部图谱类型进行结合分析,可将93株酿酒酵母分为19个基因型:75株野生酿酒酵母被鉴定为10个基因型(表 1),包含基因型6 (2株)、7 (8株)、9 (8株)、10 (15株)、11 (1株)、14 (3株)、15 (1株)、16 (6株)、17 (27株)和18 (4株);而商业酿酒酵母F33被鉴定为基因型7,BO213和BV818被鉴定为基因型17,KD被鉴定为基因型18,其余14株商业酿酒酵母可分为9个基因型(71B为基因型1,图谱型为I8-Sa1-Cg1-Sy2;RV002为基因型2,图谱型为I2-Sa1-Cg1-Sy3;VIC为基因型3,图谱型为I1-Sa1-Cg1-Sy4;ADT为基因型4,图谱型为I2-Sa1-Cg1-Sy4;RV171为基因型5,图谱型为I6-Sa1-Cg1-Sy3;2323、AC、D254、F15、RC212和X16为基因型8,图谱型为I8-Sa2-Cg2-Sy2;CEC01为基因型12,图谱型为I2-Sa4-Cg1-Sy1;CECA为基因型13,图谱型为I3-Sa4-Cg1-Sy3;EC1118为基因型19,图谱型为I7-Sa5-Cg3-Sy4)。19个基因型的聚类分析结果(图 6)表明,基因型2与7的遗传关系最近,其中基因型2为国内商业酿酒酵母RV002,基因型7包含8株野生酿酒酵母和1株国外商业酿酒酵母(F33),展现了国内外商业酿酒酵母与野生酿酒酵母相近的系统发育关系。
表 1. 紫秋和紫秋1号刺葡萄自然发酵过程中野生酿酒酵母基因型占比和密度动态变化 Table 1. Dynamic changes in the proportion and concentration of indigenous S. cerevisiae genotypes during spontaneous fermentations of two Vitis davidii F?ex varieties (Ziqiu, and Ziqiu No 1)
| Genotypes (Types of electrophoresis profiles) | The proportion (%)—concentration (CFU/mL) | ||||||
| Ziqiu-Ⅰ-84 h | Ziqiu No 1-Ⅰ-84 h | Ziqiu No 1-Ⅰ-108 h | Ziqiu-Ⅱ-96 h | Ziqiu-Ⅱ-120 h | Ziqiu-Ⅱ-168 h | Ziqiu-Ⅱ-240 h | |
| 6 (I2-Sa1-Cg3-Sy2) | - | - | - | - | 13.33%-4.27×106 | - | - |
| 7 (I2-Sa1-Cg3-Sy3) | - | - | - | - | 6.67%-2.13×106 | 10.53%-5.47×106 | 26.32%-7.30×105 |
| 9 (I2-Sa3-Cg3-Sy3) | - | - | - | - | 6.67%-2.13×106 | 31.58%-1.64×107 | 5.26%-1.46×105 |
| 10 (I1-Sa3-Cg4-Sy1) | 100%-4.76×104 | 100%-1.86×104 | 91.67%-1.56×105 | - | - | - | - |
| 11 (I5-Sa3-Cg4-Sy1) | - | - | 8.33%-1.56×105 | - | - | - | - |
| 14 (I2-Sa4-Cg3-Sy2) | - | - | - | - | 20.00%-6.40×106 | - | - |
| 15 (I4-Sa4-Cg3-Sy2) | - | - | - | - | 6.67%-2.13×106 | - | - |
| 16 (I1-Sa4-Cg3-Sy3) | - | - | - | 16.67%-2.86×104 | 6.67%-2.13×106 | 5.26%-2.74×106 | 15.79%-4.38×105 |
| 17 (I2-Sa4-Cg3-Sy3) | - | - | - | 66.67%-1.14×105 | 40.00%-1.28×107 | 52.63%-2.74×107 | 36.84%-1.02×106 |
| 18 (I8-Sa4-Cg3-Sy3) | - | - | - | 16.67%-2.86×104 | - | - | 15.79%-4.38×105 |
| The different fermentation stages were named with "grape variety-initial sugar content-fermentation time", for example, Ziqiu-Ⅰ-84 h means the 84 h of spontaneous fermentation of Ziqiu variety with initial sugar content of 143 g/L (Ⅰ). There is no samples from Ziqiu No 1-Ⅱ due to no S. cerevisiae isolated from the spontaneous fermentation of Ziqiu No 1 variety with initial sugar content of 200 g/L (Ⅱ). -: no isolation of S. cerevisiae. | |||||||
表选项
 |
| 图 6 酿酒酵母19个基因型的聚类分析图 Figure 6 The cluster dendrogram of 19 S. cerevisiae genotypes |
| 图选项 |
2.5 自然发酵过程中野生酿酒酵母不同基因型的动态变化研究 本实验的前期研究表明,在刺葡萄4种自然发酵样品(2个品种2种含糖量水平)中占据主导地位的酵母菌是日本裂殖酵母(Schizosaccharomyces japonicus),发酵结束时主导菌为日本裂殖酵母而酿酒酵母未得到分离[25]。在自然发酵的整个过程中酿酒酵母为阶段性出现,所检测到的细胞密度与日本裂殖酵母相近,但酿酒酵母在4种自然发酵中出现的时间差别较大(表 1):在刺葡萄2个品种(紫秋和紫秋1号)含糖量I的自然发酵过程中,野生酿酒酵母只在发酵中期被分离到,其密度在104-105 CFU/mL之间,基因型种类较少,其中紫秋样品中分离的野生酿酒酵母全部鉴定为基因型10,紫秋1号样品中分离的野生酿酒酵母以基因型10为主,此外还存在一部分基因型11;在刺葡萄2个品种(紫秋和紫秋1号)含糖量II的自然发酵过程中,在紫秋1号中没有分离出酿酒酵母,野生酿酒酵母只在紫秋品种自然发酵的中后期被分离到,其密度在104-107 CFU/mL之间,且展现了丰富的基因型变化。对比分析发现,两个含糖量水平的自然发酵中未分离到共有基因型。在紫秋含糖量I的自然发酵过程中,基因型6、14和15只在发酵中期(120 h,密度为106 CFU/mL)被分离到,基因型7、9、16、17和18广泛存在于发酵中后期,其中基因型7所占比例随发酵进行呈上升趋势,基因型17占比呈下降趋势,基因型9占比先上升后下降而基因型16和18占比先下降后上升,而且这3种基因型(9、16和18)在发酵中期(96 h/120 h)和发酵末期(240 h)出现时占比相近,展现了可能存在的酿酒酵母种内相互作用及酿酒酵母与以日本裂殖酵母为代表的其他酵母菌种之间相互作用的结果。
3 讨论 作为目前工业应用最广的酵母菌[29-30],酿酒酵母是葡萄酒自然发酵中后期存在的主要野生酵母菌种[3, 28],野生酿酒酵母的基因型多样性研究对其开发利用具有重要意义[11-12]。目前中国具有地域特色的野生酿酒酵母资源及其基因型多样性已得到不断的研发,如分离自沙城产区的龙眼葡萄[11]、宁夏的赤霞珠[31]和陕西的小白玫瑰[28]等的酿酒酵母,为产区特色葡萄酒的酿造提供了必要的菌种基础。本研究从贵州紫云县刺葡萄自然发酵过程中分离鉴定到野生酿酒酵母75株,采用Interdelta指纹图谱分析和微卫星分子标记法对这些野生酿酒酵母进行了基因型分析,并与商业酿酒酵母的基因型进行了比对分析。研究表明,75株野生酿酒酵母被鉴定为10种基因型,其中7种基因型为野生酿酒酵母特有基因型,其余3种基因型与商业酵母为共有基因型,具体来说,8株野生酿酒酵母和F33为基因型7,27株野生酿酒酵母、BO213和BV818为基因型17,4株野生酿酒酵母和KD为基因型18。酿酒酵母基因型在不同的发酵时期展现出了动态变化,该动态变化可能是受到葡萄品种、含糖量和其他主导酵母菌如日本裂殖酵母的影响所致[25]。
Wang和Liu (2013)曾使用Interdelta指纹图谱分析将216株酿酒酵母划分为6个图谱类型[28],本研究中75株野生酿酒酵母具有5个Interdelta指纹图谱类型(I1、I2、I4、I5和I8),并结合微卫星分子标记法划分为10种基因型,表现了贵州紫云县刺葡萄自然发酵过程中丰富的酿酒酵母菌株基因型多样性。刺葡萄发酵前含糖量水平的调节,导致了主导基因型酵母的明显变化,说明发酵条件会影响自然发酵过程中具有不同遗传特点的酿酒酵母基因型之间的竞争。此外,因在刺葡萄的自然发酵中后期,酿酒酵母与日本裂殖酵母共同占据主导地位,发酵末期以日本裂殖酵母为主而酿酒酵母未得到分离[25],推测含糖量水平的变化可能通过影响日本裂殖酵母与酿酒酵母之间的竞争从而间接导致了酿酒酵母主导基因型的明显变化直至消亡。进一步的研究分析需关注酿酒酵母不同基因型及与日本裂殖酵母菌株之间的相互作用。75株野生酿酒酵母中基因型17的占比最高为36%,但该基因型与国外商业酿酒酵母BO213和国内商业酿酒酵母BV818的基因型相同,说明该基因型酵母在世界范围内分布较为广泛。
本研究通过Interdelta指纹图谱分析鉴定了8个图谱类型,其中I2类型的菌株最多,占总数的58.1%。本研究中的Interdelta指纹图谱展现了较亮的1000 bp以上的条带和相对较暗且扩增不稳定的1000 bp以下的条带,明显不同于已报道的分离自沙城产区、新疆、甘肃、内蒙古、陕西和宁夏的酿酒酵母Interdelta指纹图谱[11, 19, 28, 31]。商业酵母EC1118、RC212和71B在本研究中的图谱不同于Legras和Karst (2003)及Feng等(2020)报道的Interdelta指纹图谱[15, 27],证实了实验室PCR扩增和电泳条件及菌种获取来源的不同对Interdelta指纹图谱分析结果的稳定性可能具有一定的影响,因此对中国不同产区酿酒酵母基因型的比较需要选用相同来源(如商业活性干酵母或菌种保藏中心酵母)的参考菌株在相同实验条件下进行比对分析。微卫星分子标记法所采用的3个微卫星位点区分能力类似,但鉴别结果并不完全相同,微卫星位点ScAAT1鉴定了5个图谱类型,Sa4类菌株数量最多占49.5%,微卫星位点C4鉴定了4个图谱类型,其中Cg3类占比最高为68.8%,微卫星位点SCYOR267C鉴定了4个图谱类型,Sy3类占比最高为64.5%,因此多个微卫星位点的结合使用有利于酿酒酵母的种内差异分析。此外,相关研究报道表明微卫星位点ScAAT1具有较多的等位基因数目,因此适用于酿酒酵母的种内区分[11, 13-14],本研究通过琼脂糖电泳分析微卫星扩增图谱时发现ScAAT1位点扩增条带相对暗淡,且部分条带不易扩增,需通过多次PCR获取微卫星图谱,但经过PCR体系的调整,其条带亮度有明显改善。微卫星位点C4和SCYOR267C的扩增表现优于ScAAT1,尤以SCYOR267C的扩增条带亮度最佳且一次性检测通过。总之,Interdelta指纹图谱分析和微卫星分子标记法的结合使用提供了更强的对酿酒酵母基因型多样性分析的能力,也验证了较多遗传标记的使用理论上可以赋予每种酿酒酵母菌株独特的基因型的报道[23, 32]。单核苷酸多态性分析和高通量测序分析可从酿酒酵母DNA序列分析的角度解读酿酒酵母的种内差异,较本研究所用方法更为全面,但这两种分析方法对DNA的完整性要求更高,因此基于全基因组序列分析之上的更多新微卫星位点的发掘使用对酿酒酵母基因型研究具有重大意义[32]。此外,为减少指纹图谱读取的主观性,进一步的研究可引入毛细管电泳技术,以提高条带读取的准确性。值得注意的是,虽然新技术的使用可获得更全面准确的结果,但本研究中使用的Interdelta指纹图谱分析和微卫星分子标记法,在现阶段乃至将来较长时期仍然是酿酒酵母基因型研究的有效补充手段。
Pfliegler和Sipiczki指出酿酒酵母的基因型多样性与菌株的表型多样性之间没有必然的关联[32],因此,进一步的研究将就相关基因型的表型如耐受性、发酵及酿造特性进行分析比对,以证实野生酿酒酵母与商业酿酒酵母之间的表现型关联或差异,并对酿酒酵母与日本裂殖酵母共发酵过程中的消亡关系展开研究,在此基础上筛选优良的野生酿酒酵母为贵州地区特色葡萄酒的酿造提供菌种基础。
References
| [1] | Pérez-Torrado R, Gamero E, Gómez-Pastor R, Garre E, Aranda A, Matallana E. Yeast biomass, an optimised product with myriad applications in the food industry. Trends in Food Science & Technology, 2015, 46(2): 167-175. |
| [2] | Comitini F, Capece A, Ciani M, Romano P. New insights on the use of wine yeasts. Current Opinion in Food Science, 2017, 13: 44-49. DOI:10.1016/j.cofs.2017.02.005 |
| [3] | Fleet GH. Wine yeasts for the future. FEMS Yeast Research, 2008, 8(7): 979-995. DOI:10.1111/j.1567-1364.2008.00427.x |
| [4] | Ma J, Liu YL. Metabolism and gene regulation of important volatile sulfur compounds in wine-A review. Acta Microbiologica Sinica, 2011, 51(1): 14-20. (in Chinese) 马捷, 刘延琳. 葡萄酒中重要挥发性硫化物的代谢及基因调控. 微生物学报, 2011, 51(1): 14-20. |
| [5] | ?uranská H, Vránová D, Omelková J. Isolation, identification and characterization of regional indigenous Saccharomyces cerevisiae strains. Brazilian Journal of Microbiology, 2016, 47(1): 181-190. DOI:10.1016/j.bjm.2015.11.010 |
| [6] | Nikolaou E, Soufleros EH, Bouloumpasi E, Tzanetakis N. Selection of indigenous Saccharomyces cerevisiae strains according to their oenological characteristics and vinification results. Food Microbiology, 2006, 23(2): 205-211. DOI:10.1016/j.fm.2005.03.004 |
| [7] | Parapouli M, Hatziloukas E, Drainas C, Perisynakis A. The effect of Debina grapevine indigenous yeast strains of Metschnikowia and Saccharomyces on wine flavour. Journal of Industrial Microbiology & Biotechnology, 2009, 37(1): 85-93. |
| [8] | Yu H, Guan JX, Yang Y, Huang XY, Cheng G, Xie TL, Xie LJ. Effects of seven wild wine yeast strains on the aroma compounds of 'guipu 3' dry white wines. Food Science, 2019, 40(4): 251-258. (in Chinese) 余欢, 管敬喜, 杨莹, 黄晓云, 成果, 谢太理, 谢林君. 7株野生葡萄酒酵母对'桂葡3号'干白葡萄酒香气成分的影响. 食品科学, 2019, 40(4): 251-258. |
| [9] | Pei YF, Liu YL. Application of DNA molecular markers in genetic diversities of Saccharomyces cerevisiae strains. Scientia Agricultura Sinica, 2010, 43(5): 1023-1030. (in Chinese) 裴颖芳, 刘延琳. DNA分子标记技术在酿酒酵母菌株遗传多样性分析中的应用. 中国农业科学, 2010, 43(5): 1023-1030. DOI:10.3864/j.issn.0578-1752.2010.05.018 |
| [10] | Sun Y, Zhang FF, Chu SX, Li JX, Shao S, Zhang JX. Effects of Saccharomyces cerevisiae strains with different killer activities on yeast diversity during inoculated fermentation of cabernet sauvignon. Food Science, 2020, 41(2): 166-172. (in Chinese) 孙悦, 张方方, 褚遂兴, 李佳幸, 邵帅, 张军翔. 接种不同嗜杀特性的酿酒酵母对赤霞珠发酵中酵母多样性的影响. 食品科学, 2020, 41(2): 166-172. |
| [11] | Zhao JJ, Li Y, Zhang LZ, Zhuang YT. Interdelta PCR fingerprinting for the identification of Saccharomyce cerevisiae from Shacheng area. Food Science, 2010, 31(23): 281-284. (in Chinese) 赵静静, 李艳, 张利中, 庄玉婷. Interdelta PCR指纹图谱法区分鉴定沙城产区酿酒酵母. 食品科学, 2010, 31(23): 281-284. |
| [12] | Liu Y, Wang C, Joseph CML, Bisson LF. Comparison of two PCR-based genetic fingerprinting methods for assessment of genetic diversity in Saccharomyces strains. American Journal of Enology and Viticulture, 2014, 65(1): 109-116. DOI:10.5344/ajev.2013.13056 |
| [13] | Liu N, Qin Y, Song YY, Ye DQ, Yuan W, Pei YF, Xue B, Liu YL. Selection of indigenous Saccharomyces cerevisiae strains in Shanshan County (Xinjiang, China) for winemaking and their aroma-producing characteristics. World Journal of Microbiology and Biotechnology, 2015, 31(11): 1781-1792. DOI:10.1007/s11274-015-1929-8 |
| [14] | Jia J, Wang GQ, Zhu LX, Han PJ, Guo DQ, Chen M. Structure characteristics of Saccharomyces cerevisiae population associated with grape and wine in Xinjiang. Journal of Dalian Polytechnic University, 2018, 37(2): 94-99. (in Chinese) 贾佳, 王冠群, 朱丽霞, 韩培杰, 郭东起, 陈明. 新疆葡萄和葡萄酒相关酿酒酵母种群的结构特征. 大连工业大学学报, 2018, 37(2): 94-99. |
| [15] | Feng L, Wang JM, Ye DQ, Song YY, Qin Y, Liu YL. Yeast population dynamics during spontaneous fermentation of icewine and selection of indigenous Saccharomyces cerevisiae strains for the winemaking in Qilian, China. Journal of the Science of Food and Agriculture, 2020, 100(15): 5385-5394. DOI:10.1002/jsfa.10588 |
| [16] | Agarbati A, Canonico L, Ciani M, Comitini F. Fitness of selected indigenous Saccharomyces cerevisiae strains for white Piceno DOC wines production. Fermentation, 2018, 4(2): 37. DOI:10.3390/fermentation4020037 |
| [17] | de Celis M, Ruiz J, Martín-Santamaría M, Alonso A, Marquina D, Navascués E, Gómez-Flechoso Má, Belda I, Santos A. Diversity of Saccharomyces cerevisiae yeasts associated to spontaneous and inoculated fermenting grapes from Spanish vineyards. Letters in Applied Microbiology, 2019, 68(6): 580-588. DOI:10.1111/lam.13155 |
| [18] | Sun Y, Qin Y, Pei YF, Wang GP, Joseph CML, Bisson LF, Liu YL. Evaluation of Chinese Saccharomyces cerevisiae wine strains from different geographical origins. American Journal of Enology and Viticulture, 2017, 68(1): 73-80. DOI:10.5344/ajev.2016.16059 |
| [19] | Feng L, Jia H, Wang JM, Qin Y, Liu YL, Song YY. Selection of indigenous Saccharomyces cerevisiae strains for winemaking in northwest China. American Journal of Enology and Viticulture, 2019, 70(2): 115-126. DOI:10.5344/ajev.2018.18035 |
| [20] | Franco-Duarte R, Umek L, Zupan B, Schuller D. Computational approaches for the genetic and phenotypic characterization of a Saccharomyces cerevisiae wine yeast collection. Yeast, 2009, 26(12): 675-692. DOI:10.1002/yea.1728 |
| [21] | Xiao Y, Hu Y, Zhang L, Shi GY. Screening of microsatellite markers associated with acetic acid tolerance of Saccharomyces cerevisiae. Microbiology China, 2015, 42(11): 2065-2072. (in Chinese) 肖银, 胡芸, 张梁, 石贵阳. 酿酒酵母乙酸耐受性相关的微卫星分子标记筛选. 微生物学通报, 2015, 42(11): 2065-2072. |
| [22] | Zhang LY, Huang YZ, Liu YL. Genetic diversity analysis of Saccharomyces cerevisiae strains by microsatellite marker technique. Food Science, 2014, 35(1): 130-133. (in Chinese) 张留燕, 黄英子, 刘延琳. 利用微卫星标记分析酿酒酵母的遗传多样性. 食品科学, 2014, 35(1): 130-133. |
| [23] | Tie CY, Hu Y, Zhang L, Shi GY. Genetic analysis of industrial yeast (Saccharomyces cerevisiae) based on sequencing of 26S rRNA gene D1/D2 domain and microsatellite markers. Mycosystema, 2014, 33(4): 894-904. (in Chinese) 铁春燕, 胡芸, 张梁, 石贵阳. 利用26S rDNAD1/D2区序列和微卫星标记分析各种工业酿酒酵母的种内遗传差异. 菌物学报, 2014, 33(4): 894-904. |
| [24] | Feng M, Wang CX, Liu YL. Genetic diversity of Saccharomyces cerevisiae strains revealed by microsatellite sequence polymorphism. Scientia Agricultura Sinica, 2012, 45(12): 2537-2543. (in Chinese) 冯敏, 王春晓, 刘延琳. 利用微卫星多态性揭示酿酒酵母菌株遗传多样性. 中国农业科学, 2012, 45(12): 2537-2543. DOI:10.3864/j.issn.0578-1752.2012.12.024 |
| [25] | Wang CX, Wu C, Qiu SY. Yeast diversity investigation of Vitis davidii F?ex during spontaneous fermentations using culture-dependent and high-throughput sequencing approaches. Food Research International, 2019, 126: 108582. DOI:10.1016/j.foodres.2019.108582 |
| [26] | Zhou XL, Shen W, Rao ZM, Wang ZX, Zhuge J. A rapid method for preparation of fungal chromosome DNA. Microbiology, 2004, 31(4): 89-92. (in Chinese) 周小玲, 沈微, 饶志明, 王正祥, 诸葛健. 一种快速提取真菌染色体DNA的方法. 微生物学通报, 2004, 31(4): 89-92. DOI:10.3969/j.issn.0253-2654.2004.04.021 |
| [27] | Legras JL, Karst F. Optimisation of Interdelta analysis for Saccharomyces cerevisiae strain characterisation. FEMS Microbiology Letters, 2003, 221(2): 249-255. DOI:10.1016/S0378-1097(03)00205-2 |
| [28] | Wang CX, Liu YL. Dynamic study of yeast species and Saccharomyces cerevisiae strains during the spontaneous fermentations of Muscat Blanc in Jingyang, China. Food Microbiology, 2013, 33(2): 172-177. DOI:10.1016/j.fm.2012.09.014 |
| [29] | Jubany S, Tomasco I, Ponce de León I, Medina K, Carrau F, Arrambide N, Naya H, Gaggero C. Toward a global database for the molecular typing of Saccharomyces cerevisiae strains. FEMS Yeast Research, 2008, 8(3): 472-484. DOI:10.1111/j.1567-1364.2008.00361.x |
| [30] | Borneman AR, Pretorius IS. Genomic insights into the Saccharomyces sensu stricto complex. Genetics, 2015, 199(2): 281-291. DOI:10.1534/genetics.114.173633 |
| [31] | Pei YF, Wang GP, Liu YL. Strain typing of Saccharomyces cerevisiae during spontaneous fermentation of cabernet sauvigon wine. Microbiology, 2009, 36(10): 1532-1535. (in Chinese) 裴颖芳, 王国平, 刘延琳. 赤霞珠葡萄酒自然发酵中酿酒酵母的菌株区分. 微生物学通报, 2009, 36(10): 1532-1535. |
| [32] | Pfliegler WP, Sipiczki M. Does fingerprinting truly represent the diversity of wine yeasts? A case study with Interdelta genotyping of Saccharomyces cerevisiae strains. Letters in Applied Microbiology, 2016, 63(6): 406-411. DOI:10.1111/lam.12679 |
