王俊博, 孔令新, 乔永健, 张大杉, 邓子新, 由德林


上海交通大学生命科学技术学院, 微生物代谢国家重点实验室, 上海 200030
收稿日期:2020-11-25;修回日期:2021-01-19;网络出版日期:2021-01-28
基金项目:国家自然科学基金(31630002,31470183)
*通信作者:由德林, Tel:+86-21-62932943; E-mail: dlyou@sjtu.edu.cn.
摘要:[目的] 分析刺孢吸水链霉菌北京变种(农抗120产生菌)基因组和次级代谢产物组分,研究并鉴定农抗120产生菌中未被发现的活性组分。[方法] 利用antiSMASH在线分析农抗120产生菌Streptomyces hygrospinosus var.beijingensis基因组信息,锁定可能的制霉菌素和丰加霉素生物合成基因簇。利用HPLC和LC-MS等分析方法对农抗120产生菌发酵产物进行分析,同时利用制霉菌素和丰加霉素标准品作为对照,以鉴定该菌株代谢组分中的次级代谢产物。此外,通过构建目标基因簇大片段缺失突变株,并对所得突变株发酵产物进行检测,以确定生物合成基因簇与目的代谢产物的对应关系。[结果] 本研究综合利用基因组序列分析、基因缺失突变株构建以及代谢产物检测方法,鉴定了农抗120产生菌中制霉菌素和丰加霉素两种活性成分,并确定了负责这些化合物合成的基因簇。[结论] 本研究所构建的多重基因簇失活突变株为挖掘刺孢吸水链霉菌北京变种更多的天然次级代谢产物奠定了基础。
关键词:农抗120刺孢吸水链霉菌北京变种制霉菌素丰加霉素生物合成
Genome mining guided discovery and identification of nystatin and toyocamycin in Streptomyces hygrospinosus var. beijingensis
Junbo Wang, Lingxin Kong, Yongjian Qiao, Dashan Zhang, Zixin Deng, Delin You


State Key Laboratory of Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200030, China
Received: 25 November 2020; Revised: 19 January 2021; Published online: 28 January 2021
*Corresponding author: Delin You, Tel: +86-21-62932943; E-mail: dlyou@sjtu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31630002, 31470183)
Abstract: [Objective] We aimed to discover and identify the uncharacterized active components of Agricultural Antibiotic 120 by comprehensive analysis of genome sequence and secondary metabolites of Streptomyces hygroscopicus var. beijingensis (Bacteria, which produced Agricultural Antibiotic 120). [Methods] The genome sequence of wild-type strain was analyzed by antiSMASH for the sake of identifying possible biosynthetic gene clusters of nystatin and toyocamycin. HPLC, LC-MS and Q-TOF were used to analyze the secondary metabolites of wild-type strains, using chemical standards as control. Biosynthetic gene cluster (BGC) deletion mutants were constructed, and the fermentation products were analyzed. [Results] Biosynthetic BGCs of nystatin and toyocamycin were identified, and these two compounds were proved to be another two bioactive ingredients of Agricultural Antibiotic 120, besides the known anisomycin and tetramycin. [Conclusion] The multiple BGC deletion mutant deducted the effect of compounds with high abundance and could serve as a good material to mine more natural compounds originally produced by WT strain. Moreover, correspondence analysis of genome sequence and metabolite profile was an effective method to identify compounds encoded by the silent biosynthetic gene clusters hidden in the genome.
Keywords: Agricultural Antibiotic 120Streptomyces hygroscopicus var. beijingensisnystatintyocamycinbiosynthesis
微生物次级代谢产物由于复杂多样的分子结构和优良的生物活性,被广泛应用于农业、畜牧业和生物医药等领域。农抗120是一种由刺孢吸水链霉菌北京变种S. hygrospinosus var. beijingensis产生的多组分新型广谱抗真菌农药。它对白粉病、瓜果和蔬菜炭疽病、西瓜枯萎病以及水稻纹枯病具有显著的防治效果,同时具有促进植物增产的作用[1]。与其他传统的杀菌剂例如代森锰锌[2]、多菌灵[3]、百菌清[4]混配联用,可以有效增强这些杀菌剂的防治效果,从而减少其使用量。作为一种混合型抗菌素,农抗120由水溶性和脂溶性两大组分组成。其中,水溶性组分推测是一种碱性核苷类化合物;脂溶性组分一种具有很强的生物活性,后被证明是茴香霉素[5];另外一种脂溶性活性组分经分离纯化最后鉴定为一种四烯大环内酯类抗生素四霉素B[6]。经过生物活性测试发现,这两种脂溶性组分对清酒酵母均表现出良好的抑制活性[6]。然而,农抗120中是否存在其他活性化合物尚有待于发现和鉴定。
目前,已经在农抗120中发现和鉴定了茴香霉素和四霉素的存在。这两种组分的成功分离鉴定主要采用传统的化学方法,很大程度依赖于这两种化合物在次级代谢产物中的较高丰度。近年来随着测序技术进步,微生物基因组信息被充分地分析和挖掘,与此同时还涌现出了很多预测次级代谢产物生物合成基因簇的软件。因此,通过利用基因组序列分析、以已知化合物标准品为对照等方法,将很容易对那些在分离纯化过程中易被忽视的、产量较低的链霉菌次级代谢产物进行分析和结构鉴定。通过基于基因组信息挖掘的生物合成研究已经成功阐释了茴香霉素(anisomycin,图 1)[7]和四霉素(tetramycin,图 1)[8]的生物合成途径。这些成功案例启示我们更加深入有效地分析基因组信息,对农抗120中其他可能的活性组分进行发现和表征鉴定。
 |
| 图 1 农抗120活性组分的化学结构[7-8] Figure 1 Chemical structures of the active component in Agricultural Antibiotic 120[7-8]. |
| 图选项 |
因此,本研究通过分析农抗120产生菌刺孢吸水链霉菌北京变种的基因组序列,利用antiSMASH[9]预测了分别与制霉菌素和丰加霉素生物合成基因簇相似度较高的基因簇。在此基础上,对这两个可能的生物合成基因簇中基因进行了功能注释。在代谢产物分析上,通过与制霉菌素和丰加霉素标准品进行对比分析,成功证明了农抗120活性成分除四霉素和茴香霉素外,还存在制霉菌素(nystatin,图 1)和丰加霉素(toyocamycin,图 1)。四霉素和制霉菌素同属于大环内酯类抗生素,两者的紫外吸收光谱较为相似,并且在高效液相色谱检测过程中两者保留时间非常接近且无法完全分离,因此本研究构建了四霉素生物合成基因簇敲除突变株,优化了对代谢组分中制霉菌素的检测分析。为了证明生物合成基因簇与代谢组分之间的对应关系,对目标生物合成基因簇进行了大片段基因敲除。在此基础上对所得突变株的代谢产物进行检测,证明了制霉菌素的存在。此外,所构建的四霉素、制霉菌素基因簇双重失活无抗突变株,排除了高丰度组分的干扰,有利于刺孢吸水链霉菌北京变种次级代谢产物中其他活性组分的鉴定,也为挖掘链霉菌基因组中沉默的生物合成基因簇提供了良好的研究材料。
1 材料和方法 1.1 实验材料
1.1.1 菌株与质粒: 刺孢吸水链霉菌北京变种(S. hygrospinosus var. beijingensis)、大肠杆菌DH10B、ET12567/pUZ8002及质粒pIJ773、pJTU1278均由上海交通大学微生物代谢国家重点实验室保藏。pJTU1278衍生质粒:pWJB414-1、pWJB414-2、pWJB453-1、pWJB453-2;四霉素生物合成基因簇大片段缺失突变株WJB-414-1 (阿泊拉霉素抗性)、WJB-414-2 (WJB-414-1衍生菌株、无抗)、制霉菌素生物合成基因簇大片段缺失突变株WJB-453-1 (WJB-414-2衍生菌株,阿泊拉霉素抗性)、WJB-453-2 (WJB-453-1衍生菌株、无抗)为本研究构建。
1.1.2 主要试剂和仪器: PCR反应所用的高保真DNA聚合酶2×Phanta Master Mix和多片段一步克隆试剂盒ClonExpress MultiS为南京诺维赞公司产品,2×Taq PCR Master Mix为生工生物工程(上海)股份有限公司产品。Omega质粒快速提取和琼脂糖凝胶回收试剂盒购自广州飞扬生物工程有限公司,限制性核酸内切酶BamH Ⅰ、EcoR Ⅰ和Hind Ⅲ为Thermofisher赛默飞公司产品,1 kb Plus DNA Ladder为Invitrogen公司产品。丰加霉素标准品购自APExBIO Technology LLC,纯度98%,制霉菌素标准品购自生工生物工程(上海)股份有限公司,纯度98%。有机溶剂:乙酸乙酯、甲醇购自上海泰坦科技股份有限公司,色谱级甲醇、三氟乙酸购自Sigma-Aldrich公司。振荡摇床为上海知楚仪器有限公司产品,高速离心机(Legend Micro 17)为Thermo Scientific公司产品。采用Agilent Technologies 1260 Infinity、Thermo Scientific Dionex Ultimate 3000 Series进行化合物的分析检测,利用Agilent Technologies 6530 Accurate-Mass Q-TOF LC-MS上进行化合物分子量的分析检测。
1.1.3 培养基: (1) 野生型及其衍生突变株的产孢培养基SFM:甘露醇20 g,青岛琼脂15 g,加入灭菌过滤后的20 g黄豆饼粉滤液800 mL,pH调至7.2,去离子水定容至1 L后121 ℃高压灭菌30 min,使用时补加2.5 mol/L MgCl2,4 mL/L。(2) 接合转移培养基ISP4:购自BD-Difco,使用时加入终浓度25 mmol/L MgCl2。(3) 种子培养基TSBY:TSB 30 g/L,酵母提取物5 g/L,蔗糖103 g/L,115 ℃灭菌20 min。(4) 发酵培养基:玉米淀粉10 g/L,可溶性淀粉20 g/L,一同糊化,冷却后加入黄豆饼粉10 g/L,磷酸氢二钾0.2 g/L,氯化钠3 g/L,氯化铵3 g/L,pH调至7.2后加入碳酸钙4 g/L。(5) 孢子预萌发液:1%酵母提取物,1%水解酪蛋白氨基酸。
1.2 制霉菌素和四霉素生物合成基因簇敲除突变株的构建 利用游离型基因敲除载体pJTU1278进行目标基因簇的大片段敲除。首先,利用限制性内切酶BamH Ⅰ、Hind Ⅲ线性化pJTU1278载体,然后利用多片段一步克隆方法,将待敲除的大片段基因两端的3 kb同源臂与线性化载体连接,并在两条同源臂之间通过设计延长PCR扩增引物来引入EcoR Ⅰ酶切位点,以此构建pWJB453-2和pWJB414-2,PCR验证、测序验证质粒的正确性。以pWJB453-2和pWJB414-2为基础构建pWJB453-1和pWJB414-1,首先利用EcoR Ⅰ将两质粒单酶切线性化,利用单片段一步克隆方法将阿泊拉霉素抗性基因片段连接在两条同源臂之间,测序验证其正确性。为获得WJB414-1突变株,将构建好的质粒pWJB414-1转化进入ET12567/PUZ8002,与野生型链霉菌进行双亲本接合转移,30 ℃培养14 h后采用阿泊拉霉素和萘啶酮酸进行覆盖,30 ℃培养5 d,挑取接合子进入含有阿泊拉霉素和萘啶酮酸的30 mL TSBY种子液中振荡培养3 d至菌体粘稠,提取基因组DNA进行PCR验证。为获得WJB414-2突变株,以WJB414-1为原始菌株,利用pWJB414-2,通过双亲本结合转移进行阿泊拉霉素抗性基因敲除,30 ℃培养14 h后采用硫链丝菌素和萘啶酮酸进行覆盖,30 ℃培养5 d,挑取单交换接合子进入无抗TSBY种子液中松弛培养3 d,而后稀释涂布于无抗SFM平板继续松弛培养3 d促进同源双交换,挑取单克隆菌落在无抗和含有阿泊拉霉素的SFM平板上进行影印筛选,重复传代3次以得到无抗突变株WJB414-2。同理,WJB453-1突变株以WJB414-2为原始菌株,对制霉菌素基因簇首先进行抗性替换而得到,然后以WJB453-1为原始菌株,通过影印筛选得到无抗WJB453-2突变株,这里不重复赘述。
1.3 菌株发酵和发酵产物的预处理 发酵方法:取适量保藏在含20%甘油冻存管中的孢子于产孢培养基活化,将活化后的菌株孢子保藏并接种至装有30 mL种子培养基的250 mL底部带弹簧的三角摇瓶中,30 ℃、220 r/min振荡培养36 h至菌丝体粘稠,按照5%的接种量接种至含30 mL发酵培养基的三角瓶中,30 ℃、220 r/min振荡培养6 d。发酵产物的处理方法:将约30 mL发酵液全部转移至50 mL离心管中,4000 r/min离心10 min,将上层清液转移至新的50 mL离心管中,利用等体积乙酸乙酯连续萃取3次,合并乙酸乙酯相,旋蒸并尽量除尽乙酸乙酯,用1 mL甲醇分多次将样品溶解制成粗样。下层固相利用2倍固体体积的丙酮浸泡24 h使细胞裂解,旋蒸干丙酮相,剩余粘稠物加水溶解并用等体积乙酸乙酯连续萃取3次,合并乙酸乙酯相,旋蒸除尽乙酸乙酯,用1 mL甲醇溶解制成粗样。粗样品用有机相针式滤器0.22 μm过滤,13000 r/min离心10 min,取适量体积制成待分析样品。
1.4 发酵产物的检测分析 使用Q-TOF对标准品的精确分子量进行测定。使用HPLC检测分析菌株发酵产物,分析所用的流动相:A相(水,1/1000甲酸),B相(甲醇),流速:0.5 mL/min,检测波长280 nm。洗脱条件:0-5 min:5%甲醇等度洗脱,5-45 min:5%-95%甲醇梯度洗脱,45-50 min:95%甲醇等度洗脱,50-60 min:95%-5%甲醇梯度洗脱。色谱柱:Agilent 5 TC-C18 250×4.6 mm,PN:518925-902,SN:552377。进样体积:20 μL。LC-MS采用相同的洗脱条件,检测波长280 nm。参数设置:正离子模式,H-ESI加热温度:300 ℃,氦气压力:60 psi,辅助气压力:30 psi,喷雾电压:3 kV,离子传输管温度:380 ℃,离子传输管电压:15 V。分子量扫描范围:150-2000 m/z。
1.5 生物信息学分析 利用antiSMASH version 5.1.0对农抗120产生菌S. hygrospinosus var. beijingensis基因组中的生物合成基因簇进行在线分析预测,利用NCBI对基因簇中的功能基因进行PROTEIN-BLAST比对分析。
2 结果和分析 2.1 基于基因组信息对农抗120产生菌次级代谢产物制霉菌素的分析 通过对刺孢吸水链霉菌基因组的contig00005- region3 (75 kb)和contig00016-region1 (43 kb)进行序列分析,并利用次级代谢生物合成基因簇预测软件antiSMASH进行分析预测,前者与Streptomyces noursei ATCC 11455的制霉菌素A1生物合成基因簇(NCBI GenBank:AF263912.1)相似度高达81%,与Streptomyces albulus的制霉菌素生物合成基因簇(NCBI GenBank:MG742725.1)相似度达到74%;后者分别与这两个基因簇的相似度为31%和42%。由于采用第二代基因组测序方法,整个基因簇被分为2个contig,但是拼接后的基因簇长度与制霉菌素生物合成基因簇大小趋于一致(图 2-A),因此初步推测其为制霉菌素生物合成基因簇。对基因簇中的18个功能基因利用BLAST进行序列比对分析,其与上述两株制霉菌素产生菌的功能基因都具有很高的同源性,其中关键的Ⅰ型聚酮合酶orf4、orf5、orf6、orf12、orf13、orf14与制霉菌素产生菌Streptomyces noursei ATCC 11455的Ⅰ型聚酮合酶基因高度同源表 1),这说明该生物合成基因簇可能负责合成制霉菌素。
 |
| 图 2 制霉菌素生物合成基因簇(A)及农抗120产生菌发酵产物制霉菌素的检测(B, C) Figure 2 Organization of the nystatin biosynthetic gene cluster and LC-MS analysis of the fermentation extracts of S. hygrospinosus var. beijingensis. A: Organization of the nystatin biosynthetic gene cluster (BGC). B: HPLC analysis (λ=280 nm) of the nystatin standard (i) and the secondary metabolites of S. hygrospinosus var. beijingensis (ii) with UV spectrum. C: Q-TOF analysis of the nystatin standard (i) and LC-MS analysis of the secondary metabolitesof S. hygrospinosus var. beijingensis (ii). |
| 图选项 |
表 1. 刺孢吸水链霉菌基因组中制霉菌素生物合成基因簇的功能基因分析 Table 1. Deduced functions of ORFs of the nystatin BGC in S. hygrospinosus var. beijingensis
| Gene | aa | Proposed function | Similar protein (acc number) | Identity/% |
| orf1 | 606 | ABC transporter ATP-binding protein | WP_067343761.1 | 89.74 |
| orf2 | 610 | ABC transporter ATP-binding protein | WP_102924881.1 | 88.13 |
| orf3 | 344 | GDP-mannose 4, 6-dehydratase | WP_067343755.1 | 95.35 |
| orf4 | 9500 | Type I polyketide synthase | WP_067343753.1 | 86.20 |
| orf5 | 5371 | Type I polyketide synthase | AAF71767.1 | 85.13 |
| orf6 | 2056 | Type I polyketide synthase | AVX51100.1 | 98.49 |
| orf7 | 394 | Cytochrome P450 | WP_039629369.1 | 93.65 |
| orf8 | 63 | (4Fe-4S)-binding protein | WP_106680950.1 | 69.23 |
| orf9 | 398 | Cytochrome P450 | WP_102924874.1 | 91.96 |
| orf10 | 352 | DegT family aminotransferase | WP_067343741.1 | 91.76 |
| orf11 | 463 | Glycosyltransferase | AAF71773.1 | 93.42 |
| orf12 | 1371 | Type I polyketide synthase | AVX51106.1 | 97.38 |
| orf13 | 3196 | Type I polyketide synthase | AJC53395.1 | 86.60 |
| orf14 | 11082 | Type I polyketide synthase | AJC53394.1 | 79.52 |
| orf15 | 249 | Thioesterase | WP_102924869.1 | 85.94 |
| orf16 | 965 | AAA family ATPase | WP_102924868.1 | 85.91 |
| orf17 | 952 | LuxR family transcriptional regulator | WP_099055646.1 | 80.13 |
| orf18 | 927 | LuxR family transcriptional regulator | WP_102924866.1 | 81.01 |
表选项
通过对农抗120产生菌S. hygrospinosus var. beijingensis次级代谢产物进行HPLC检测分析(图 2-B),发现农抗120产生菌S. hygrospinosus var. beijingensis发酵样品在48.5 min处出现与制霉菌素标准品相同的峰型,且该峰的紫外吸收光谱与标准品相一致。将LC-MS分析的样品分子量与利用Q-TOF精确分析的标准品分子量进行比较,结果表明两者分子量一致(图 2-C)。总之,通过基因组序列信息和代谢产物分析检测,确定了农抗120产生菌S. hygrospinosus var. beijingensis次级代谢产物中含有制霉菌素。
2.2 基于基因组信息对农抗120产生菌次级代谢产物丰加霉素的分析 关于农抗120活性成分中的水溶性组分,以前推断可能是一种碱性核苷类抗生素,但具体是哪一种化合物却一直未得到确定。刺孢吸水链霉菌基因组序列中contig00002-region1含有一个长约9 kb的可能编码核苷类抗生素的生物合成基因簇(图 3-A)。利用antiSMASH对整个基因簇进行分析,发现其与Streptomyces diastatochromogenes中的丰加霉素生物合成基因簇具有较高的同源性(序列相似度高达90%),并且两者在基因排列上也高度相似。对其中关键基因编码的氨基酸序列逐一比对分析(表 2),发现orf1、orf2、orf3、orf4、orf9分别与龟裂链霉菌Streptomyces rimosus中负责丰加霉素生物合成的关键基因toyM、toyL、toyH、toyG、toyB具有较高同源性。这些结果说明农抗120产生菌刺孢吸水链霉菌基因组中这个编码核苷类抗生素的基因簇很可能是丰加霉素生物合成基因簇。
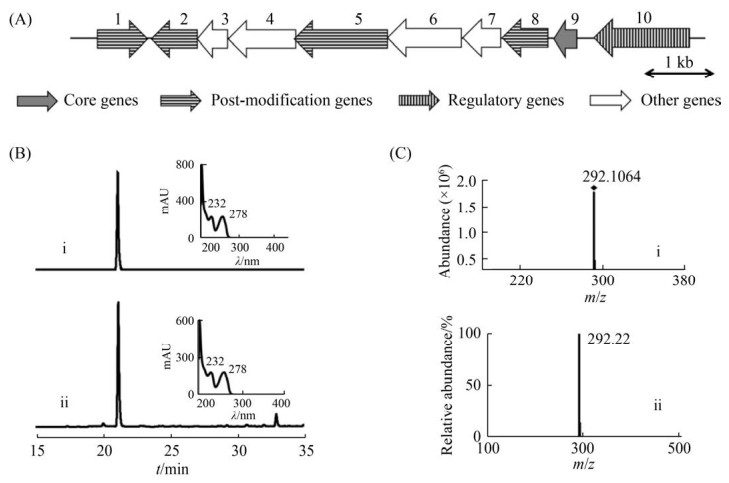 |
| 图 3 丰加霉素生物合成基因簇(A)及农抗120产生菌发酵产物丰加霉素的检测(B, C) Figure 3 Organization of the toyocamycin biosynthetic gene cluster and LC-MS analysis of the fermentation extracts of S. hygrospinosus var. beijingensis. A: Organization of the toyocamycin biosynthetic gene cluster. B: HPLC analysis (λ=280 nm) of the toyocamycin standard (i) and the secondary metabolites of S. hygrospinosus var. beijingensis (ii) with UV spectrum. C: Q-TOF analysis of the toyocamycin standard (i) LC-MS analysis of secondary metabolites of S. hygrospinosus var. beijingensis (ii). |
| 图选项 |
表 2. 刺孢吸水链霉菌基因组中丰加霉素生物合成基因簇的功能基因分析 Table 2. Deduced functions of ORFs of the toyocamycin BGC in S. hygrospinosus var. beijingensis
| Gene | aa | Proposed function | Similar protein (acc number) | Identity/% |
| orf1 | 238 | 7-cyano-7-deazaguanine synthase | ACF06644.1 | 72.27 |
| orf2 | 232 | HAD-superfamily hydrolase | ACF06640.1 | 59.91 |
| orf3 | 173 | Phosphoribosyltransferase | ACF06639.1 | 61.76 |
| orf4 | 351 | Adenylosuccinate synthase | ACF06638.1 | 72.51 |
| orf5 | 461 | Adenylosuccinate synthase | WP_050515133.1 | 66.97 |
| orf6 | 381 | IMP dehydrogenase | WP_050515134.1 | 75.00 |
| orrf7 | 201 | GTP cyclohydrolase I FolE | WP_053803966.1 | 74.59 |
| orf8 | 223 | 7-carboxy-7-deazaguanine synthase | WP_030672084.1 | 76.68 |
| orf9 | 160 | 6-carboxytetrahydropterin synthase | ACF06634.1 | 73.33 |
| orf10 | 432 | SARP family transcriptional regulator | WP_100600129.1 | 82.72 |
表选项
为了进一步确定野生型菌株是否合成丰加霉素,利用LC-MS对野生型菌株的发酵产物进行了分析。与此同时,利用丰加霉素标准品作为对照。经过分析发现,在280 nm紫外波长检测条件下,野生型菌株的发酵产物在21 min出现与丰加霉素标准品具有相同保留时间的峰,且该峰的紫外吸收光谱与丰加霉素标准品相一致(图 3-B)。LC-MS分析样品的分子量与利用Q-TOF精确测定的标准品的分子量一致(图 3-C)。因此,通过以上基因组挖掘和发酵产物的鉴定,证明农抗120产生菌刺孢吸水链霉菌的次级代谢产物含有丰加霉素。
2.3 多重基因簇失活突变株的构建以及代谢产物的检测分析 四霉素是农抗120中的重要活性成分,产量在发酵产物中很高[6]。在进行农抗120产生菌次级代谢产物的高效液相色谱检测过程中,发现制霉菌素和四霉素的保留时间非常接近且无法完全分离。为了排除四霉素的干扰,准确鉴定代谢产物中确实存在制霉菌素,本研究进一步构建了四霉素基因簇无抗敲除突变株,以及在敲除四霉素生物合成基因簇基础上敲除制霉菌素基因簇的突变株来进行验证。
利用pJTU1278衍生载体,将待敲除四霉素生物合成基因大片段进行抗性替换,快速筛选到四霉素生物合成基因缺失突变株WJB414-1。为了方便进一步遗传操作,又将阿泊拉霉素抗性基因进行敲除,构建得到无抗突变株WJB414-2。在此基础上构建了四霉素、制霉菌素基因簇双重敲除无抗突变株WJB453-2 (图 4-A)。分别在左右同源臂设计引物,对突变株WJB414-2和WJB453-2进行PCR验证:突变株可以扩增出1500 bp条带,而野生型菌株由于两条同源臂没有连接,无法扩增出条带(图 4-B,C)。
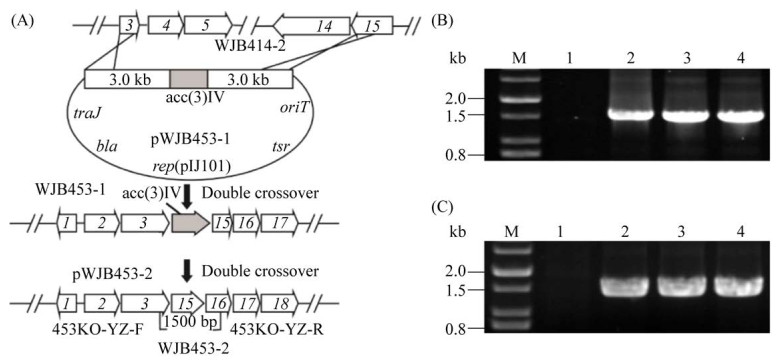 |
| 图 4 制霉菌素和四霉素生物合成基因簇敲除突变株的构建(A)和PCR验证(B) Figure 4 Construction and verification of the nystatin and tetramycin BGC knock-out mutants. A: The schematic construction of nystatin orf4-orf14 deletion strain WJB453-2. The 102 kb fragment of BGC was replaced by an oriT+acc(3)Ⅳ cassette in WJB414-2 through homologous recombination, resulting in the mutant WJB453-1. Then the resistance cassette was removed by the second genetic manipulation, resulting in the mutant WJB453-2. 453KO-YZ-F/R indicate the locations of the primers, which were used to validate the deletion. B: PCR verification of three randomly selected WJB453-2 mutants. Lane 2, 3 and 4, the 1500 bp replicons were amplified using genomic DNA isolated from WJB453-2 strains, while no band was amplified from WT strain (lane 1). C: PCR verification of the tetramycin BGC mutants WJB414-2. Lane 2, 3 and 4, the genomic DNA isolated from WJB414-2 strains was used as template and obtained 1500 bp replicons; lane 1, the WT strain was used as control. |
| 图选项 |
对野生型菌株、WJB414-2和WJB453-2突变株发酵产物以及制霉菌素标准品进行HPLC检测分析,在280 nm紫外波长检测条件下发现WJB414-2突变株不产生四霉素,并可以在48 min观察到明显的与制霉菌素标准品出峰时间相同的峰型(图 5)。WJB453-2突变株缺失制霉菌素生物合成基因簇,在48 min没有任何色谱峰出现,说明该基因簇的缺失导致了制霉菌素不再产生,由此清楚地证明了制霉菌素是农抗120产生菌S. hygrospinosus var. beijingensis的次级代谢产物。
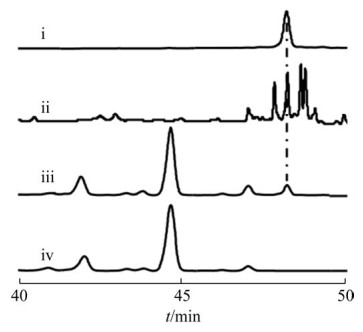 |
| 图 5 农抗120产生菌野生型和突变株中制霉菌素产物的HPLC检测分析 Figure 5 HPLC analysis (λ=280 nm) of the nystatin production in the wild-type and mutant strains of S. hygrospinosus var. beijingensis. (i) Nystatin standard. (ii) Wild-type strain. (iii) WJB414-2. (iv) WJB453-2. |
| 图选项 |
3 讨论 本研究结合基因组序列分析预测和次级代谢产物检测分析方法,发现并鉴定了农抗120活性成分中含有大环内酯类抗生素制霉菌素和核苷类抗生素丰加霉素。根据报道,制霉菌素可以与真菌膜中的甾醇相互作用,从而导致细胞内成分泄漏而死亡[10]。丰加霉素作为一种碱性核苷类抗生素,对病毒[11]、念珠菌[12]和肿瘤细胞[13]具有良好的抑制活性。此外,在生物农业应用方面,研究表明丰加霉素可以拮抗黄瓜立枯丝核菌,防止黄瓜立枯病[14]。这两个化合物鉴定是对之前研究的农抗120活性成分中含有四霉素和茴香霉素的补充和完善。另外,在获得负责丰加霉素和制霉菌素的生物合成基因簇的基础上,开展其生物合成研究,也有助于通过基因工程手段进行农抗120组分的改造。
在分析刺孢吸水链霉菌基因组时,不只一个核苷类抗生素生物合成基因簇被检测,但最后只鉴定到丰加霉素。同样,同属于大环内酯的制霉菌素的产量也较低,这意味着链霉菌在实验室发酵过程中对于一类抗生素的生物合成,可能会优先选择一个生物合成基因簇进行表达,其他孤儿基因簇作为抵抗外界压力或物种竞争中的备选基因,被细胞内更加复杂的调控因子控制而“沉默”[15]。当敲除主产物四霉素生物合成基因簇后,代谢组分中其他化合物例如丰加霉素的产量得以提升,且在四霉素缺失突变株和四霉素、制霉菌素双缺失突变株发酵产物分析中,都观察到相同的差异峰,推测这种现象是由于主产物基因簇的缺失使得前体物质或还原力等流向其他抗生素的生物合成造成的[16]。同时,本研究在野生型菌株中顺序敲除四霉素和制霉菌素生物合成基因簇所得的突变株WJB414-1、WJB414-2、WJB453-1和WJB453-2与野生型相比,在生长速度、产孢方面没有明显差异。这对分析代谢物中其他产量较低的化合物或挖掘基因组中沉默的生物合成基因簇有一定的意义。所以,对已知生物合成基因簇进行定向失活[17],无论是在构建代谢产物背景更加干净的菌株,或对目标化合物的产量进行提升,还是对研究分析隐藏在基因组和代谢组分中的有用信息,都有一定的价值。
References
| [1] | Zhu CX, Tian YL, Jiang XL, Ji JH. Effect of agricultural antibiotic 1204% aqueous solution on main leaf diseases of apple and pear trees. Chinese Journal of Biological Control, 2004, 20(3): 190-192. (in Chinese) 朱昌雄, 田云龙, 蒋细良, 姬军红. 4%增效农抗120对几种主要果树病害的试验效果. 中国生物防治, 2004, 20(3): 190-192. |
| [2] | Han XC, Zhang N, Xiao GR, Hu CF. Study on the synergistic effect of agricultural antibiotic 120 and macozeb against Colletotrichum capsici. Chinese Journal of Pesticides, 2004, 43(11): 522-523. (in Chinese) 韩新才, 张宁, 肖国蓉, 胡长富. 农抗120与代森锰锌对辣椒炭疽病菌的增效作用. 农药, 2004, 43(11): 522-523. DOI:10.3969/j.issn.1006-0413.2004.11.013 |
| [3] | Han XC, Peng KB, Li XS, Shi ZP. Study on the synergism of agricultural antibiotic 120 and carbendazim against Fusarium oxysporum. Cotton Science, 2008, 20(5): 391-393. (in Chinese) 韩新才, 彭坤波, 李喜书, 石章鹏. 农抗120与多菌灵混配对棉花枯萎病菌的增效作用研究. 棉花学报, 2008, 20(5): 391-393. DOI:10.3969/j.issn.1002-7807.2008.05.013 |
| [4] | Han XC, Zhang N, Xiao GR, Hu CF. Studies on the allied toxcity function of agricultural antibiotic 120 and daconil to the Colletotrichum capsici. Journal of Huazhong Agricultural University, 2005, 24(2): 157-160. (in Chinese) 韩新才, 张宁, 肖国蓉, 胡长富. 农抗120和百菌清对辣椒炭疽病菌联合毒力的测定. 华中农业大学学报, 2005, 24(2): 157-160. DOI:10.3321/j.issn:1000-2421.2005.02.013 |
| [5] | Gu HE, Zhang ZY, Wang XS, Tong ZH, Ren QS. Identification of the structure of 120A' of Agricultural antibiotic 120. Chinese Journal of Biological Control, 1994, 10(4): 166-168. (in Chinese) 顾惠儿, 张中岳, 王学士, 仝赞华, 任清水. 农抗120中脂溶性主要组分A'的结构鉴别. 生物防治通报, 1994, 10(4): 166-168. |
| [6] | Yao F, Kang QJ, Lin SJ, Zhu CX, You DL, Deng ZX. Isolation and structural identification of a tetraene macrolide produced by Streptomyces hygrospinosus var. Beijingensis. Journal of Shanghai Jiao Tong University, 2011, 45(1): 88-91. (in Chinese) 姚芬, 康前进, 林双君, 朱昌雄, 由德林, 邓子新. 农抗120活性成分四烯化合物的分离与鉴定. 上海交通大学学报, 2011, 45(1): 88-91. DOI:10.3969/J.ISSN.1671-9964.2011.01.016 |
| [7] | Zheng XQ, Cheng QX, Yao F, Wang XZ, Kong LX, Cao B, Xu M, Lin SJ, Deng ZX, Chooi YH, You DL. Biosynthesis of the pyrrolidine protein synthesis inhibitor anisomycin involves novel gene ensemble and cryptic biosynthetic steps. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(16): 4135-4140. DOI:10.1073/pnas.1701361114 |
| [8] | Cao B, Yao F, Zheng XQ, Cui DB, Shao YC, Zhu CX, Deng ZX, You DL. Genome mining of the biosynthetic gene cluster of the polyene macrolide antibiotic tetramycin and characterization of a P450 monooxygenase involved in the hydroxylation of the tetramycin B polyol segment. Chembiochem, 2012, 13(15): 2234-2242. DOI:10.1002/cbic.201200402 |
| [9] | Blin K, Shaw S, Steinke K, Villebro R, Ziemert N, Lee SY, Medema MH, Weber T. antiSMASH 5.0:updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research, 2019, 47(W1): W81-W87. DOI:10.1093/nar/gkz310 |
| [10] | Fj?rvik E, Zotchev SB. Biosynthesis of the polyene macrolide antibiotic nystatin in Streptomyces noursei. Applied Microbiology and Biotechnology, 2005, 67(4): 436-443. DOI:10.1007/s00253-004-1802-4 |
| [11] | De Clercq E, Bernaerts R, Bergstrom DE, Robins MJ, Montgomery JA, Holy A. Antirhinovirus activity of purine nucleoside analogs. Antimicrobial Agents and Chemotherapy, 1986, 29(3): 482-487. DOI:10.1128/AAC.29.3.482 |
| [12] | Nishimura H, Katagiri K, Sato K, Mayama M, Shimaoka N. Toyocamycin, a new anti-Candida antibiotics. The Journal of Antibiotics, 1956, 9(2): 60-62. |
| [13] | Park SG, Kim SH, Kim KY, Yu SN, Choi HD, Kim YW, Nam HW, Seo YK, Ahn SC. Toyocamycin induces apoptosis via the crosstalk between reactive oxygen species and p38/ERK MAPKs signaling pathway in human prostate cancer PC-3 cells. Pharmacological Reports, 2017, 69(1): 90-96. DOI:10.1016/j.pharep.2016.10.014 |
| [14] | Yu B, Shentu XP, Yu XP. Antifungal activity of toyocamycin on Rhizoctonia solani kühn. Chinese Journal of Biological Control, 2011, 27(3): 373-377. (in Chinese) 于冰, 申屠旭萍, 俞晓平. 丰加霉素对黄瓜立枯丝核菌的拮抗作用. 中国生物防治学报, 2011, 27(3): 373-377. |
| [15] | Amos GCA, Awakawa T, Tuttle RN, Letzel AC, Kim MC, Kudo Y, Fenical W, Moore B, Jensen PR. Comparative transcriptomics as a guide to natural product discovery and biosynthetic gene cluster functionality. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(52): E11121-E11130. DOI:10.1073/pnas.1714381115 |
| [16] | Culp EJ, Yim G, Waglechner N, Wang WL, Pawlowski AC, Wright GD. Hidden antibiotics in actinomycetes can be identified by inactivation of gene clusters for common antibiotics. Nature Biotechnology, 2019, 37(10): 1149-1154. DOI:10.1038/s41587-019-0241-9 |
| [17] | Wang WS, Li SS, Li ZL, Zhang JY, Fan KQ, Tan GY, Ai GM, Lam SM, Shui GH, Yang ZH, Lu HZ, Jin PJ, Li YH, Chen XY, Xia XK, Liu XT, Dannelly HK, Yang C, Yang Y, Zhang SL, Alterovitz G, Xiang WS, Zhang LX. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces. Nature Biotechnology, 2020, 38(1): 76-83. DOI:10.1038/s41587-019-0335-4 |
