赵宇春, 刘向阳, 邓子新, 贺新义, 蒋明


上海交通大学生命科学技术学院, 微生物代谢国家重点实验室, 上海 200030
收稿日期:2020-12-13;修回日期:2021-01-20;网络出版日期:2021-02-23
基金项目:国家自然科学基金(31870026,31300033,21661140002);国家重大研发计划(2018YFA0900402);中科院生命有机化学国家重点实验室开放基金
*通信作者:蒋明, E-mail: jiangming9722@sjtu.edu.cn.
摘要:[目的] 在大肠杆菌中完整重构孢子色素whiE的生物合成途径,分离纯化表达体系中合成的新化合物,并解析whiE的生物合成途径。[方法] 构建whiE-ORFII、whiE-ORFVII和whiE-ORFI的单基因重组质粒,SDS-PAGE检测蛋白表达情况;借助Xba I与Spe I互为同尾酶的特性,实现多基因组合串联;构建好的重组质粒再导入大肠杆菌菌株BAP1中进行异源表达,并用高效液相色谱(HPLC)检测发酵产物;依次使用正相硅胶柱和反向半制备柱分离发酵产物,四级杆飞行时间质谱仪(Q-TOF MS)鉴定发酵产物分子量。[结果] whiE-ORFII、whiE-ORFVII和whiE-ORFI均获得可溶性表达;这3个基因单个串联到菌株BTw95中均未检测到新的产物生成;而whiE-ORFII和whiE-ORFVII、whiE-ORFI和whiE-ORFVII双基因组合以及三基因组合串联到BTw95中可检测得到两种化合物ZYC-1和ZYC-2。在负离子模式下进行Q-TOF MS检测,ZYC-1的[M-H]-为419.0748,推测分子式为C23H16O8;ZYC-2的[M-H]-为465.0743,推测分子式为C24H18O10。[结论] 本研究推进了孢子色素whiE生物合成途径在大肠杆菌中的异源重构,分离鉴定了2个十二酮II型聚酮化合物,并推测了孢子色素whiE的生物合成途径。
关键词:孢子色素whiE大肠杆菌异源生物合成II型聚酮化合物
Heterologous biosynthesis of spore pigment whiE from Streptomyces coelicolor in Escherichia coli
Yuchun Zhao, Xiangyang Liu, Zixin Deng, Xinyi He, Ming Jiang


State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200030, China
Received: 13 December 2020; Revised: 20 January 2021; Published online: 23 February 2021
*Corresponding author: Ming Jiang, E-mail: jiangming9722@sjtu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31870026, 31300033, 21661140002), by the National Major R&D Program (2018YFA0900402) and by the Open Fund of the State Key Laboratory of Life Organic Chemistry, Chinese Academy of Sciences
Abstract: [Objective] Completely reconstruction of the biosynthetic pathway of whiE in Escherichia coli, isolation and purification of new compounds synthesized in the recombinant strain for elucidation of the biosynthetic pathway of whiE. [Methods] Construction of single-gene recombinant plasmids of whiE-ORFII, whiE-ORFVII, whiE-ORFI, and SDS-PAGE analysis of the fusion protein expression; isoschizomers Xba I/ Spe Ⅰ were adopted to assemble multiple genes in a single plasmid construct. The recombinant plasmid was then introduced into Escherichia coli BAP1 for heterologous expression, and the fermentation products was detected by high performance liquid chromatography (HPLC); the normal phase silica gel column and the reverse semi-preparative column were used sequentially to separate the fermentation products, the quadrupole high resolution mass spectrometry (Q-TOF MS) was used to identify the molecular weight of the fermentation products. [Results] whiE-ORFII, whiE-ORFVII and whiE-ORFI were solubly expressed; no new product was detected when these three genes were added to the strain BTw95 individually; two compounds ZYC-1 and ZYC-2 can be detected in the strains coupled with whiE-ORFII & whiE-ORFVII, whiE-ORFI & whiE-ORFVII as well as all the three genes. Q-TOF MS analysis in negative ion mode showed that the[M-H]- of ZYC-1 is 419.0748 with a proposed molecular formula, C23H16O8 and[M-H]- of ZYC-2 is 465.0743 with a proposed molecular formula, C24H18O10. [Conclusion] This study achieved the heterologous reconstruction of the spore pigment whiE biosynthetic pathway in E. coli. Two dodecaketide type II polyketides were identified and isolated. The biosynthetic pathway of the spore pigment whiE was also proposed.
Keywords: spore pigment whiEEscherichia coliheterologous biosynthesistype II polyketide
抗生素和孢子色素是放线菌的次级代谢产物,可由聚酮合酶(PKSs)合成。尽管迄今为止已经研究了大量编码抗生素的基因[1-2],但由于链霉菌中孢子色素可与链霉菌孢子壁结合,导致其不易分离纯化,所以关于孢子色素及其生物合成基因簇的研究报道较少,相应的结构和功能也未得到解析[3-4]。天蓝色链霉菌中的孢子色素whiE是第一个被发现和克隆的孢子色素,该基因簇属于II型聚酮合酶基因簇,为十二酮II型聚酮化合物,由8个开放阅读框组成,whiE-ORFIII (KSα)、whiE-ORFIV (KSβ)和whiE-ORFV (ACP)组成最小聚酮合酶[5]。
Bradley S. Moore等以敲除放线菌紫红素和whiE生物合成基因簇的天蓝色链霉菌作为表达宿主,表达了whiE的生物合成基因簇。当链霉菌中单独表达whiE的最小聚酮合酶时,可以合成30个以上的聚酮化合物,其中8个的结构得到鉴定,8个聚酮化合物的长度不同,说明此时聚酮链长度控制比较松。当whiE最小聚酮合酶与环化酶whiE-ORFVI在链霉菌中共表达,合成了十二酮化合物TW95a和TW95b,表明whiE-ORFVI不仅负责前2个环的环化和芳香化,还与最小聚酮合酶协同控制聚酮链的长度[6]。为了进一步分析每个基因的功能,Bradley S. Moore等将环化酶whiE-ORFII和whiE-ORFVII分别克隆至表达载体中,转入天蓝色链霉菌中异源表达,结果没有检测到新的化合物生成,TW95a和TW95b为主要产物。当将whiE-ORFII和whiE- ORFVII与最小聚酮合酶、环化酶whiE-ORFVI一起克隆至表达载体中,转入天蓝色链霉菌中异源表达,不能再检测到化合物TW95a和TW95b而产生了新的孢子色素,分离得到红色和蓝色的色素,但产量很低也不纯,因此未能进一步鉴定这两种新的色素,但其在结构上应该更加接近野生型的whiE孢子色素[7]。在24碳II型聚酮化合物Pradimicin的生物合成中,当在天蓝色链霉菌中异源表达最小聚酮合酶PdmA、PdmB、PdmC和环化酶PdmD时,也可以合成TW95a和TW95b。当同时串联加入环化酶PdmL、PdmK和氧化酶PdmH合成JX111a和JX111b,其中JX111b由JX111a自发脱羧形成[8]。
几十年来,放线菌一直是发现聚酮类化合物的重要来源和主要的异源表达宿主,大量药物和类似物成功地进入市场,直到今天仍在临床实践中使用[9-10]。但放线菌生长会经历复杂的形态分化,生长缓慢,遗传操作相比大肠杆菌较复杂,在聚酮化合物的生产中,会受到较多限制。而大肠杆菌生长速度快,易于培养,遗传操作简便,是理想的生产菌株。I型聚酮化合物如红霉素[11]、III型聚酮化合物如2, 4-二羟基-6-戊基苯甲酸已成功在大肠杆菌中实现异源合成[12],但II型聚酮化合物在肠杆菌中异源合成的报道较少,主要的原因是II型聚酮化合物的最小聚酮合酶在大肠杆菌中不易实现可溶性表达。2019年,Eriko Takano首次报道了芳香聚酮化合物在大肠杆菌中的异源生物合成,并通过组合生物学证明了她们所建立体系的适用性[13]。本课题组也成功分别实现了醌那霉素和孢子色素whiE的聚酮合酶KSα和KSβ蛋白复合体在大肠杆菌中的可溶性表达,成功建立醌那霉素生物合成中间体Dehydrorabelomycin的大肠杆菌中异源生物合成平台[14]。在实现whiE-ORFIII (KSα)、whiE-ORFIV (KSβ)的可溶性表达之后,构建串联质粒pET28a-whiE-III-IV (pXY-7)和pETM-whiE-V-VI-MCAT (pXY-8),并将其共转入菌株BAP1/pGro7,获得合成TW95c的大肠杆菌生产菌株BTw95,经核磁分析发现TW95c的结构与天蓝色链霉菌异源表达合成的TW95a有很大相似性,推测其由TW95a经过大肠杆菌中的一个氧化酶催化了一个氧化成环反应而生成。
本研究中,我们以BTw95为出发菌株,在大肠杆菌中重构了孢子色素whiE的生物合成途径,分离纯化表达体系中合成的两个可能的新结构化合物,并在此基础上推测了whiE的生物合成途径;以孢子色素whiE的表达体系为平台,建立十二酮II型聚酮化合物大肠杆菌异源生产平台,也为将来十二酮II型聚酮化合物的改造提供一个新的平台。
1 材料和方法 1.1 实验材料
1.1.1 菌株、质粒及引物: 大肠杆菌BAP1、BL21、DH10B和天蓝色链霉菌(Streptomyces coelicolor)均为本实验室保藏。本研究所用质粒见表 1,多基因串联表达载体pXY-7、pXY-8均由本实验室构建保存。引物如表 2所示,由上海擎科生物有限公司合成。
表 1. 本研究中所用到的质粒 Table 1. Strains used in this study
| Plasmids | Relevant Characteristics | Source |
| pCDFduet-1 | CDF ori with PT7; SmR | Novagen |
| pET28a | Neo, T7 lac, pBR322 origin, His6·tag plasmid for target gene expression | Novagen |
| pGro7 | GroES-GroEL ori Cmr | TAKARA |
| pET28a-whiE II | Plasmid for whiE-ORFII expression | This study |
| pET28a-whiE VII | Plasmid for whiE-ORFVII expression | This study |
| pZYC- I | Plasmid for whiE-ORFI expression | This study |
| pXY-7 | pET28a-whiE-III-IV | This lab |
| pXY-8 | pET32a-whiE-VI-MCAT | This lab |
| pXY-7-II | pET28a-whiE-III-IV-II | This study |
| pXY-8-VII | pET32a-whiE-VI-MCAT-VII | This study |
表选项
表 2. 本研究中所用到的引物 Table 2. Primers used in this study
| Primers | Sequences (5′→3′) |
| whiE-ORFI-F | CACATATGACGGTCTCTCCCGT |
| whiE-ORFI-R | GAGGTACCTCAGGAACCGGCCGAA |
| whiE-ORFII-F | GACATATGACAGACCAGCAGGTACG |
| whiE-ORFII-R | CCAAGCTTACTAGTTCATGACACCACCTCGGCC |
| whiE-ORFVII-F | CCCATATGCACCACACACTGATCGT |
| whiE-ORFVII-R | ACAAGCTTACTAGTTCAGCCGGCGGGGGTCCG |
表选项
1.1.2 培养基: 大肠杆菌BL21、BAP1和DH10B的固体培养基为LA,液体培养基为LB。大肠杆菌BAP1的发酵培养基为PM-LBBS复合培养基(酵母提取物5 g,蛋白胨10 g,氯化钠10 g,D-山梨醇185.9 g,甘油15 g,HEPES 23.8 g,甜菜碱0.309 g,蒸馏水定容至1 L,调节pH至7.6),121 ℃、1×105 Pa灭菌30 min。
1.2 主要试剂和仪器 质粒提取试剂盒、胶回收试剂盒,购买于上海柏益思生物技术有限公司;solution I DNA连接试剂盒、Taq酶,购买于TaKaRa公司;高保真酶Phanta Max Super-Fidelity DNA Polymerase,购买于南京诺唯赞生物科技股份有限公司;限制性内切酶,Thermo Fisher Scientific公司;色谱柱(5 μm,4.6×150 mm)、HPLC、Q-TOF MS购买于安捷伦科技有限公司。
1.3 生物信息学分析 利用NCBI对whiE-ORFI、whiE-ORFVII和whiE-ORFII进行序列分析。为了预测环化酶参与成环的顺序,本研究选择了已经研究较为清晰的特曲霉素生物途径中的环化酶TcmN、TcmJ、TcmI和十二酮II型聚酮化合物Benastatin、Pradimicin,孢子色素sch、cur的环化酶以及whiE-ORFI (环化酶结构域)、whiE-ORFVII、whiE-ORFII和whiE-ORFVI进行进化树分析,系统进化树采用MEGA-7绘制,使用的算法是极大似然法[15-18]。
1.4 目的基因的克隆 基因扩增以天蓝色链霉菌(Streptomyces coelicolor)变种M1154基因组DNA为模板,以表 2中所示引物进行PCR扩增。50 μL反应体系:2×Phanta Max Buffer 25 μL,50%二甲亚砜5 μL,dNTP mix (10 mmol/L) 1 μL, Phanta Max Super-Fidelity DNA Polymerase (100 U) 1 μL,上游和下游引物(10 μmol/L)各2 μL,模板(30 ng) 1 μL,ddH2O补足至50 μL。菌落PCR验证体系为20 μL:2×mix 10 μL,上下游引物(10 μmol/L)各0.5 μL,50%二甲亚砜2 μL,挑单菌落作为模板,ddH2O补足20 μL。PCR反应条件:95 ℃预变性3 min;95 ℃变性15 s,根据引物Tm值设置退火温度,根据基因长度设置72 ℃延伸时间,30个循环;最后72 ℃延伸10 min。
1.5 单基因重组质粒的构建 双酶切处理PCR扩增片段,将目的基因分别克隆至表达载体pET28a或pCDFduet-1,将构建成功的重组质粒分别转化至DH10B感受态细胞,菌落PCR和酶切验证成功后送公司测序,测序结果正确即单基因重组质粒构建成功。
1.6 多基因重组质粒的构建 多基因串联系统均在pET系列载体上,所有基因都由统一的T7启动子进行调控。引物设计时在每个基因的3′端添加Spe I酶切位点,再借助Spe I酶切位点与pET系列载体核糖体结合位点前的Xba I酶切位点互为同尾酶的特性,进行多基因串联。多基因组合模式如表 1。
1.7 基因表达鉴定 将构建好的单基因表达质粒转化至BL21/pGro7感受态细胞,37 ℃恒温箱中过夜培养。挑取单克隆于3 mL LB培养基中,加入相应抗生素,37 ℃过夜培养。将过夜培养菌液按照1:100 (1:50)转接到新鲜的LB培养基或发酵发培养基中,37 ℃培养至OD600=0.6时,取出置于冰上降温至10 ℃左右后添加终浓度为0.5 mmol/L的IPTG和终浓度为3 mmol/L的阿拉伯糖诱导目的基因和分子伴侣的表达,放入16 ℃摇床中继续培养18–24 h。4000 r/min、4 ℃离心20 min收集菌体,取预冷的结合缓冲液重悬,超声破碎菌体,12000 r/min离心30 min,分别取全菌和上清进行SDS-PAGE检测,确定蛋白是否可溶性表达。
1.8 菌株发酵及萃取 取10 μL菌种管中的菌液接种到3 mL LB培养基中于37 ℃过夜培养,次日以1:100 (1:50)转接到PM-LBBS复合培养基中,37 ℃培养约6–7 h至OD600达到0.6,置于冰上降温,当温度降至10 ℃以下,添加终浓度为3 mmol/L的L-阿拉伯糖和终浓度为0.5 mmol/L的IPTG分别诱导分子伴侣蛋白和目的蛋白表达,18 ℃培养4–5 d。萃取方法为发酵液4000 r/min离心20 min弃菌体,上清中加1%乙酸,混匀后再添加与发酵液等体积的乙酸乙酯,剧烈摇晃萃取,3500 r/min离心10 min取有机相,旋转蒸发仪旋干后加600 μL甲醇重悬。
1.9 产物HPLC及Q-TOF MS检测 样品重悬后,12000 r/min离心10 min,取10–50 μL于样品瓶内衬管中。HPLC检测时流动相为纯水(A相)和甲醇(B相),均添加千分之一的三氟乙酸,检测方法:流速1 mL/min,初始为25% B相,20 min内B相由25%上升至100%,100% B相保持5 min,最后10 min为25% B相平衡体系。Q-TOF MS检测时,A相添加千分之一的甲酸,B相为纯乙腈,流速为0.3 mL/min,其他条件与HPLC检测相同。
2 结果和分析 2.1 生物信息学分析 通过NCBI网站的BLAST引擎对4个蛋白序列进行比对(图 1)。whiE-ORFII中含cupin结构域,类似于Streptomyces glaucescens TcmJ,是一种参与醌形成的单加氧酶,参与II型和III型聚酮合成酶合成的芳香族聚酮的修饰;whiE-ORFVII是TcmI家族II型聚酮环化酶,类似于淡青链霉菌特曲霉素基因簇中的TcmI,催化特曲霉素F2向特曲霉素F1的转化;whiE-ORFI中含有ABM和SchA/CurD结构域,目前SchA/CurD结构域只在链霉菌属的物种中被确认,参与合成聚酮孢子色素,与参与合成聚酮类抗生素的蛋白同源,该蛋白的功能尚不清楚,但推测其含有NAD(P)结合位点。
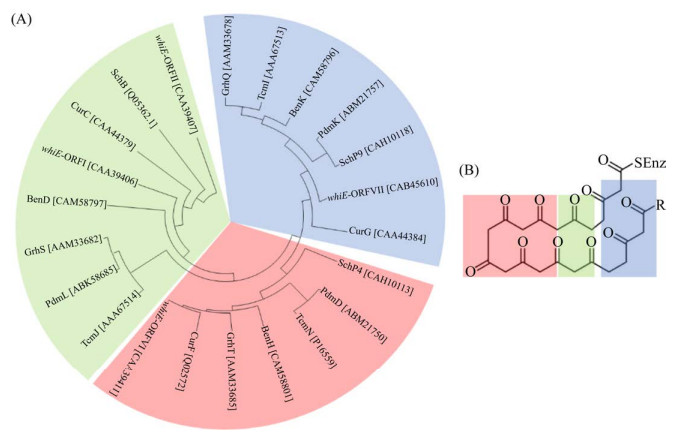 |
| 图 1 MEGA-7软件以极大似然法绘制的whiE -ORFII、whiE -ORFVII、whiE -ORFI (环化酶结构域)与其他II型聚酮化合物合成基因簇中环化酶的系统进化树分析 Figure 1 Phylogenetic tree generated by MEGA-7 with maximum likelihood method for whiE-ORFII, whiE-ORFVII, whiE-ORFI (cyclase domain) with other type II polyketide synthetic proteins. The color code refers to the function of cyclases, red: 1st/2nd ring cyclase; green: 3rd ring cyclase; blue: 4th/5th ring cyclase. A: phylogenetic tree; B: cyclization pattern in aromatic polyketides produced by type II PKS. |
| 图选项 |
系统进化树分析结果显示,whiE-ORFI (环化酶结构域)、whiE-ORFVII、whiE-ORFII和whiE- ORFVI分属3个分支。whiE-ORFVI与特曲霉素和Pradimicin合成途径中的第一个环化酶TcmN和PdmD位于同一个分支,为whiE生物合成途径中的第一个环化酶,推测负责A环和B环的环化;whiE-ORFII、whiE-ORFI (环化酶结构域)与特曲霉素合成途径中的第二个环化酶TcmJ位于同一个分支,为whiE生物合成途径中的第二个环化酶,推测负责C环的环化;whiE-ORFVII与特曲霉素合成途径中的第3个环化酶TcmI位于同一个分支,为whiE生物合成途径中的第3个环化酶,推测负责D环与E环的环化,E环的环化也可能自发形成。
2.2 质粒构建
2.2.1 构建单基因重组质粒: 将PCR扩增片段whiE-ORFII、whiE-ORFVII和whiE-ORFI酶切处理后克隆至载体pET28a、pCDFduet-1。菌落PCR验证正确后,分别用引物设计时引入的酶切位点进行双酶切验证。验证正确后,将构建好的质粒送测序公司进行测序鉴定,测序结果比对显示全部正确。
2.2.2 构建多基因重组质粒: 多基因串联借助Xba I与Spe I互为同尾酶的特性,分别将基因whiE-ORFII、whiE-ORFVII克隆至表达载体pXY-7和pXY-8,构建单基因串联菌株和多基因串联菌株(表 3)。
表 3. 本研究中所用到的菌株 Table 3. Strains used in this study
| Strains | Characteristics | Source |
| BAP1 | F-ompT hsdSB (rB–mB–) gal dcm (DE3) ΔprpRBCD: : T7 prom-sfp, T7 prom-prpE | [11] |
| BTw95 | BAP1/pGro7pXY-7 (whiE-III-IV)/pXY-8 (whiE-V-VI-MCAT) | This lab |
| BTw95-1 | BTw95/pXY-7-II | This study |
| BTw95-2 | BTw95/pXY-8-VII | This study |
| BTw95-3 | BTw95/pZYC-I | This study |
| BTw96-1 | BAP1/pGro7/pXY-7-II/pXY-8-VII | This study |
| BTw96-2 | BAP1/pGro7/pXY-7-II/pXY-8-VII/pZYC-I | This study |
| BTw96-3 | BAP1/pGro7/pXY-7-II/pXY-8/pZYC-I | This study |
| BTw96-4 | BAP1/pGro7/pXY-7/pXY-8-VII/pZYC-I | This study |
表选项
2.3 基因表达结果 将构建好的单基因表达质粒分别转化至BL21/pGro7中,于新鲜LB中分别诱导培养,取全菌和上清样品于SDS-PAGE检测可溶性,结果3个蛋白均可在大肠杆菌中可溶性表达。whiE-ORFI、whiE-ORFII、whiE-ORFVII的大小依次为45.4、19.0、14.7 kDa,结果如图 2所示。
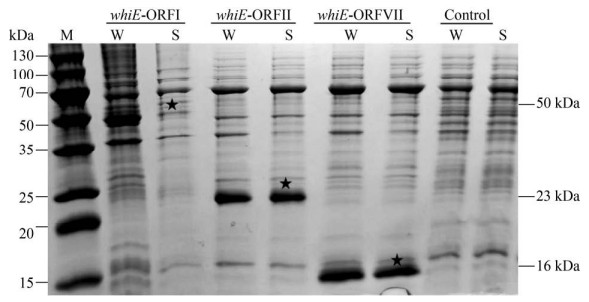 |
| 图 2 融合蛋白表达的SDS-PAGE分析 Figure 2 SDS-PAGE analysis of the fusion protein expression. Lane 1: protein molecular weight marker. W: whole cell lysate of induced cells; S: soluble fraction of induced cells; Control: BL21(DE3) cellular lysate. Over-expressed proteins in the soluble fraction have been denoted with an asterisk. |
| 图选项 |
2.4 HPLC检测发酵萃取液
2.4.1 单个基因串联发酵结果: 将单基因串联菌株BTw95-1、BTw95-2、BTw95-3与对照BTw95菌株在相同条件下发酵,与对照相似,单基因串联菌株的发酵液发酵2 d开始慢慢变为浅红色,随发酵时间延长红色逐渐加深。发酵4 d后进行萃取、HPLC检测,与对照相比,单基因串联菌株发酵液中没有检测到新的化合物产生,在检测波长276 nm下,检测产物以Tw95c为主;在检测波长500 nm下,检测产物以Tw95d为主,检测结果见图 3。
 |
| 图 3 HPLC检测单基因组合串联发酵产物 Figure 3 HPLC analysis of the products for single gene combination. i: BAP1/pGro7(control); ii: BTw95 (control); iii: BTw95-3; iv: BTw95-1; v: BTw95-2; HPLC traces of A and B were recorded at 276 nm and 500 nm, respectively. |
| 图选项 |
2.4.2 基因组合串联发酵结果: 将组合串联菌株BTw96-1、BTw96-2、BTw96-3、BTw96-4与对照BTw95菌株在相同条件下发酵。发酵4 d,BTw96-3发酵液颜色与单基因串联菌株发酵液颜色变化相同,BTw96-4发酵液颜色呈暗红色,BTw96-1、BTw96-2发酵液颜色呈黑色。与对照相比,化合物Tw95c在组合串联发酵菌株中产量都下降,其中BTw96-1和BTw96-2发酵液中未检测到Tw95c。BTw96-1、BTw96-2、BTw96-4的发酵液中均检测到新的化合物,保留时间为17.5 min的3个化合物紫外吸收相同,推测为同一化合物并命名为ZYC-2;BTw96-1发酵液中检测到的保留时间为18.5 min的化合物命名为ZYC-1,检测结果见图 4。ZYC-1与TW95d相比,紫外吸收的最大吸收波长蓝移,如图 5所示。
 |
| 图 4 HPLC检测多基因组合串联发酵产物 Figure 4 HPLC analysis of the products for multi-gene combination. i: BTw95 (control); ii: BTw96-3; iii: BTw96-4; iv: BTw96-1; v: BTw96-2; HPLC traces of A and B were recorded at 276 nm and 500 nm, respectively. |
| 图选项 |
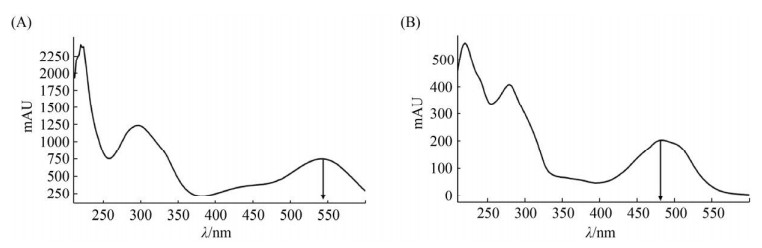 |
| 图 5 化合物Tw95d与ZYC-1的紫外吸收比较 Figure 5 Comparison of UV absorption of compound Tw95d and ZYC-1. A: Tw95d; B: ZYC-1. |
| 图选项 |
2.5 ZYC-1和ZYC-2的分离纯化和结构鉴定 将7L BTw96-1菌株发酵4 d,依次使用正相硅胶柱和反向半制备柱分离发酵产物,制备获得3.5 mg纯品ZYC-1和3 mg纯品ZYC-2。高分辨质谱检测到ZYC-1的[M-H]–为419.0748,推测分子式为C23H16O8,该化合物为紫色固体,溶于二甲亚砜,微溶于甲醇。ZYC-2的[M-H]– 465.0743,推测分子式为C24H18O10,该化合物为红色固体,溶解性能与ZYC-1相同,检测结果如图 6所示。为了进一步解析化合物ZYC-1和ZYC-2的结构,将全部纯品以氘代二甲亚砜作溶剂进行核磁检测时,在检测过程中化合物不稳定,自发转变,最终未能解析其结构,产物的结构还有待于进一步的解析。
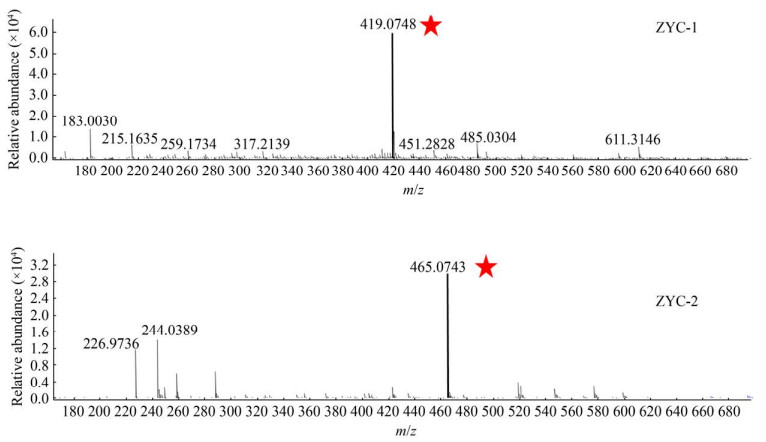 |
| 图 6 Q-TOF MS检测ZYC-1和ZYC-2 Figure 6 Q-TOF MS analysis of ZYC-1 and ZYC-2. |
| 图选项 |
3 讨论 本研究通过多基因串联的方法推进了孢子色素whiE生物合成途径在大肠杆菌中的重构,合成了两种十二酮II型聚酮化合物ZYC-1和ZYC-2,建立了新的十二酮II型聚酮化合物大肠杆菌异源生产平台。在大肠杆菌中构建孢子色素whiE的生物合成途径,涉及到了多个基因的共表达,是一个具有挑战的问题。在本研究中,通过利用Xba I与Spe I互为同尾酶的特性,方便快捷地实现了多个基因的组合串联;并在体系中引入分子伴侣,辅助蛋白折叠,提高蛋白的可溶性。孢子色素whiE的大肠杆菌异源生产平台的建立将有助于十二酮II型聚酮化合物的组合性生物合成,促进更多具有药物活性的天然产物的发现。例如,后期研究工作中,可利用whiE-ORFIII、whiE-ORFIV与其他十二酮类型的II型聚酮化合物生物合成中的后修饰基因偶联,便捷地合成不同的十二酮的II型聚酮化合物。
基于Q-TOF MS检测结果、文献调研和生物信息学分析,我们推测了ZYC-1与ZYC-2的结构,并推测了其合成途径,如图 7所示。ZYC-2与Pradimicin生物合成中间体JX111a结构类似,在20号位多了一个羟基,推测可能由大肠杆菌基因组中的羟化酶氧化形成,ZYC-1由ZYC-2自发脱羧氧化生成。综合发酵结果,当单独加入whiE-ORFII、whiE-ORFVII和whiE-ORFI时,未能检测到新的产物生成;当whiE-ORFVII分别与whiE-ORFII和whiE-ORFI两两组合时检测新的产物生成,说明whiE-ORFVII可能为孢子色素whiE生物合成途径中的第2个环化酶,负责了C环的环化,并且只有当其与环化酶whiE-ORFII或双功能酶whiE-ORFI组合时才能检测到新的产物,因此推测它们之间具有协同作用,负责C环、D环、E环的环化,E环的环化也可能是自发。唐奕课题组报道了第一例环化酶和单脱氧酶协同作用于聚酮链的环化过程。在Pradimicin生物合成中,第三、四、五个环的环化,需要环化酶PdmL、PdmK和氧化酶PdmH协同完成,并提出了一个模型,其中3种酶形成一个多酶复合体,包裹已经环化的B环和未环化的聚酮链部分。氧化酶PdmH靠近B环以促进碳8位的羟基化,而环化酶PdmK和PdmL定位于聚酮链的两端,以完成碳5、碳18和碳4、碳21位的羟醛缩合,而当单个酶对聚酮链顺序结合和修饰时,无法阻止聚酮链的自发环化,而形成多酶复合体可以有效地跨越未环化聚酮化合物的整个长度,从而将骨架锁定在适当位置并抑制异常环化[8]。基于ZYC-1和ZYC-2的预测结构,生物合成过程中B环也经历了碳8位的氧化,推测由双功能酶whiE-ORFI或大肠杆菌基因组中的氧化酶完成。当双功能酶whiE-ORFI与环化酶whiE-ORFVII组合时,同时行使环化酶和氧化酶的功能,这将是一个区别于Pradimicin的生物合成模型。
 |
| 图 7 ZYC-1和ZYC-2的可能的生物合成途径 Figure 7 The proposed biosynthetic pathway for ZYC-1 and ZYC-2. |
| 图选项 |
通过孢子色素whiE生物合成途径在大肠杆菌中的重构,本研究建立了一个十二酮II型聚酮化合物大肠杆菌异源生产体系,为将来改造和修饰十二酮II型聚酮化合物提供了平台,也为聚酮类抗生素的组合生物合成提供了新的参考。目前,化合物ZYC-1和ZYC-2的产量相对较低,为了未来的工业化应用以及后期化合物的分离纯化和结构鉴定,可通过在提高前体丙二酰辅酶A的供给[19]、串联加入硫酯酶促进聚酮链释放[20]以及优化发酵条件等提高目标化合物的产量。
References
| [1] | Hertweck C, Luzhetskyy A, Rebets Y, Bechthold A. Type II polyketide synthases: Gaining a deeper insight into enzymatic teamwork. Natural Product Reports, 2007, 24(1): 162-190. DOI:10.1039/B507395M |
| [2] | Procópio RE, Silva IR, Martins MK, Azevedo JL, Araújo JM. Antibiotics produced by Streptomyces. Brazilian Journal of Infectious Diseases, 2012, 16(5): 466-471. DOI:10.1016/j.bjid.2012.08.014 |
| [3] | Blanco G, Pereda A, Méndez Cal. Cloning and disruption of a fragment of Streptomyces halstedii DNA involved in the biosynthesis of a spore pigment. Gene, 1992, 112(1): 59-65. DOI:10.1016/0378-1119(92)90303-7 |
| [4] | Bergh S, Uhlén M. Analysis of a polyketide synthesis-encoding gene cluster of Streptomyces curacoi. Gene, 1992, 117(1): 131-136. DOI:10.1016/0378-1119(92)90501-F |
| [5] | Yu TW, Hopwood DA. Ectopic expression of the Streptomyces coelicolor whiE genes for polyketide spore pigment synthesis and their interaction with the act genes for actinorhodin biosynthesis. Microbiology: Reading, England, 1995, 141(Pt 11): 2779-2791. |
| [6] | Shen Y, Yoon P, Yu TW, Floss HG, Hopwood D, Moore BS. Ectopic expression of the minimal whiE polyketide synthase generates a library of aromatic polyketides of diverse sizes and shapes. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(7): 3622-3627. DOI:10.1073/pnas.96.7.3622 |
| [7] | Yu TW, Shen YM, McDaniel R, Floss HG, Khosla C, Hopwood DA, Moore BS. Engineered biosynthesis of novel polyketides from streptomyces spore pigment polyketide synthases. Journal of the American Chemical Society, 1998, 120(31): 7749-7759. DOI:10.1021/ja9803658 |
| [8] | Zhan JX, Watanabe K, Tang Y. Synergistic actions of a monooxygenase and cyclases in aromatic polyketide biosynthesis. Chembiochem, 2008, 9(11): 1710-1715. DOI:10.1002/cbic.200800178 |
| [9] | Xu M, Wright GD. Heterologous expression-facilitated natural products' discovery in actinomycetes. Journal of Industrial Microbiology & Biotechnology, 2019, 46(3/4): 415-431. DOI:10.1007/s10295-018-2097-2 |
| [10] | Genilloud O. Actinomycetes: still a source of novel antibiotics. Natural Product Reports, 2017, 34(10): 1203-1232. DOI:10.1039/C7NP00026J |
| [11] | Pfeifer BA, Admiraal SJ, Gramajo H, Cane DE, Khosla C. Biosynthesis of complex polyketides in a metabolically engineered strain of E. coli. Science, 2001, 291(5509): 1790-1792. DOI:10.1126/science.1058092 |
| [12] | Tan ZG, Clomburg JM, Gonzalez R. Synthetic pathway for the production of olivetolic acid in Escherichia coli. ACS Synthetic Biology, 2018, 7(8): 1886-1896. DOI:10.1021/acssynbio.8b00075 |
| [13] | Cummings M, Peters AD, Whitehead GFS, Menon BRK, Micklefield J, Webb SJ, Takano E. Assembling a plug-and-play production line for combinatorial biosynthesis of aromatic polyketides in Escherichia coli. PLoS Biology, 2019, 17(7): e3000347. DOI:10.1371/journal.pbio.3000347 |
| [14] | Liu XY, Hua KM, Liu DX, Wu ZL, Wang Y, Zhang HR, Deng ZX, Pfeifer BA, Jiang M. Heterologous biosynthesis of type II polyketide products using E. coli. ACS Chemical Biology, 2020, 15(5): 1177-1183. DOI:10.1021/acschembio.9b00827 |
| [15] | Lackner G, Schenk A, Xu ZL, Reinhardt K, Yunt ZS, Piel J, Hertweck C. Biosynthesis of pentangular polyphenols: deductions from the benastatin and griseorhodin pathways. Journal of the American Chemical Society, 2007, 129(30): 9306-9312. DOI:10.1021/ja0718624 |
| [16] | Shen B, Summers RG, Wendt-Pienkowski E, Hutchinson CR. The Streptomyces glaucescens tcmKL polyketide synthase and tcmN polyketide cyclase genes govern the size and shape of aromatic polyketides. Journal of the American Chemical Society, 1995, 117(26): 6811-6821. DOI:10.1021/ja00131a002 |
| [17] | Summers RG, Wendt-Pienkowski E, Motamedi H, Hutchinson CR. The tcmVI region of the tetracenomycin C biosynthetic gene cluster of Streptomyces glaucescens encodes the tetracenomycin F1 monooxygenase, tetracenomycin F2 cyclase, and, most likely, a second cyclase. Journal of Bacteriology, 1993, 175(23): 7571-7580. DOI:10.1128/jb.175.23.7571-7580.1993 |
| [18] | Li A, Piel J. A gene cluster from a marine Streptomyces encoding the biosynthesis of the aromatic spiroketal polyketide griseorhodin A. Chemistry and Biology, 2002, 9(9): 1017-1026. DOI:10.1016/S1074-5521(02)00223-5 |
| [19] | Wu J, Zhang X, Xia X, Dong M. A systematic optimization of medium chain fatty acid biosynthesis via the reverse beta-oxidation cycle in Escherichia coli. Metabolic Engineering, 2017, 41: 115-124. DOI:10.1016/j.ymben.2017.03.012 |
| [20] | Hua K, Liu X, Zhao Y, Gao Y, Pan L, Zhang H, Deng Z, Jiang M. Offloading Role of a Discrete Thioesterase in Type II Polyketide Biosynthesis. mBio, 2020, 11(5). |
