王艺璇1,2, 党消消1,2, 董小燕1, 骆永明1,4, 解志红1,3


1. 中国科学院烟台海岸带研究所, 山东 烟台 264003;
2. 中国科学院大学, 北京 100049;
3. 山东农业大学资源与环境学院, 山东 泰安 271018;
4. 中国科学院南京土壤研究所, 江苏 南京 210008
收稿日期:2020-10-13;修回日期:2021-01-06;网络出版日期:2021-03-25
基金项目:NSFC-山东省联合基金重点项目(U1806206);国家重点研发项目(Y91G030602)
*通信作者:解志红, Tel: +86-535-2109183;E-mail: zhihongxie211@163.com.
摘要:[目的] 田菁共生根瘤菌Ensifer alkalisoli YIC4027是从宿主植物田菁的根瘤中分离出来的一株新型高效的固氮菌。本研究对E.alkalisoli的趋化受体基因与其他研究透彻的物种进行比较以及相关蛋白分析。[方法] 利用NCBI的BLAST对E.alkalisoli趋化受体基因进行序列相似性搜索。以Pfam数据库为基础,用HMMR3对甲基化趋化受体蛋白(MCP)进行比较分析。[结果] E.alkalisoli有2个趋化基因簇,共有13个MCP,含有不同的信号传感结构。此外,这些MCPs的胞质结构域除了一个是由40个七肽重复序列组成,其余都是由36个七肽重复序列组成。[结论] 尽管E.alkalisoli的趋化受体与已被广泛研究的物种的趋化受体具有较高的相似性,但仍显示出其特性。通过基因的比对以及相关蛋白的分析,我们能够更好地理解E.alkalisoli是如何通过趋化系统来响应外界变化的。
关键词:E.alkalisoli YIC4027趋化性MCP
Comparison of genes and protein sequence analyses of the chemoreceptors in Ensifer alkalisoli YIC4027
Yixuan Wang1,2, Xiaoxiao Dang1,2, Xiaoyan Dong1, Yongming Luo1,4, Zhihong Xie1,3


1. Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003, Shandong Province, China;
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Resources and Environment, Shandong Agricultural University, Tai'an 271018, Shandong Province, China;
4. Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, Jiangsu Province, China
Received: 13 October 2020; Revised: 6 January 2021; Published online: 25 March 2021
*Corresponding author: Zhihong Xie, Tel: +86-535-2109183; E-mail: zhihongxie211@163.com.
Foundation item: Supported by the NSFC-Shandong Joint Fund Key Project (U1806206) and by the National Key Research and Development Program (Y91G030602)
Abstract: [Objective] Ensifer alkalisoli YIC4027 is a new type of high-efficiency nitrogen-fixing bacteria isolated from the root nodules of the host plant Sesbania cannabina. This study compared the chemoreceptors genes and related proteins of E. alkalisoli with other well-researched species. [Methods] NCBI protein BLAST was used for searching sequence similarity with default parameter values against the genomes of E. alkalisoli. HMMER3, based on Pfam database, was used for comparative analyses of methyl-accepting chemotaxis protein(MCP). [Results] There were two major chemotaxis clusters and 13 MCP homologs containing diverse signal-sensing architectures in E. alkalisoli. In addition, except that one of the cytoplasmic domains of these MCPs was composed of 40 heptad repeats, the rest were composed of 36 heptad repeats. [Conclusion] Despite the high similarity presented between the chemoreceptors of E. alkalisoli and those of well-studied species, E. alkalisoli shows its own unique characteristics. The classification of these chemotactic pathways by gene comparison and related protein analysis enables us to better understand how E. alkalisoli responds to changes in environment via exquisite signal transductions in chemotaxis system.
Keywords: E. alkalisoli YIC4027chemotaxisMCP
滨海耐盐碱植物田菁(Sesbania cannabina)是一种根系发达、土地适应性比较强的豆科植物,具有耐涝抗旱、耐盐碱等优良特点,经常作为改良盐碱地的模范植物[1]。此外,田菁也具有较高的经济价值,例如其秸秆能用于绿肥以及饲料的制作[2],种子产生的田菁胶也可用于食品行业。综上所述,田菁是一种具有较高的经济价值以及生态学意义的作物。田菁根瘤菌是一类革兰氏阴性菌,能与宿主植物田菁共生固氮,形成根瘤或者茎瘤,可以将大气中的氮气还原成氨,由此为宿主提供所需要的氮素营养,然后直接被植物吸收利用,降低了其对土壤中氮的需求[3]。田菁共生根瘤菌Ensifer alkalisoli发现于黄河三角洲地区,其占根瘤菌总数的71%,是最大的类群。本实验室筛选到的YIC4027是一株高效的共生固氮根瘤菌,对滨海盐碱地的改良效果良好[4]。虽然田菁在农业经济以及生态方面具有重要的应用,但是对共生根瘤菌的基因研究却鲜有报道。因此,对田菁共生根瘤菌的研究不仅可以帮助我们发展有效的结瘤菌株,而且有利于提高田菁的抗逆性,对改良滨海盐碱地以及促进生态的可持续发展具有较为深远的影响和意义。
根瘤菌在宿主植物分泌的趋化以及促进生长的化合物(糖、氨基酸、有机酸以及其他次级代谢产物)的影响下迁移到根际处[5],这些化学信号经根瘤菌的传递后与宿主植物进行传导交换。在豆科根瘤菌Rhizobium leguminosarum bv. viciae中,两条趋化性基因簇调节细胞的运动行为和泳动偏向,因此有利于豌豆植物根际内部的共生固氮[6]。趋化性指的是细菌对外界化学刺激产生反应从而定向移动,为根瘤菌提供了优势,也被认为是侵染植物根部的第一步[7]。在共生体系建立的过程中,根瘤菌的趋化作用使其保持竞争性优势,周围多变的环境以及复杂的化学物质也使根瘤菌进化出较为庞大的趋化系统[8]。
趋化基因广泛存在于多种微生物中,大肠杆菌的趋化系统已经被研究透彻,这成为了探索其他微生物趋化信号途径的参照。E. coli整个趋化信号传导通路由感受周围环境中化学物质浓度变化的甲基化趋化受体蛋白MCPs和一个主要的基因簇组成,该基因簇包含了许多与鞭毛基因紧密相连的che基因[9]。MCPs是一个极其敏感的结构,它能感知胞外配体从μmol/L到nmol/L范围内的浓度变化[10-12],存在形式为同源二聚体,具有保守的发卡结构,指导信号传递以及复合体的拼装,包括一个可变的细胞周质感受区来负责与化学物质结合以及一个保守的胞质信号区来提供与CheA和CheW结合的区域,前者主要是蛋白的氨基末端,后者是羧基末端[13]。底部的MCPs结构又参与CheA与CheW的联合,复合体的拼装也是由其决定的[14]。MCPs感应外界物质浓度变化,处于一种“开”和“关”的动态平衡中,这个过程受CheR和CheB对某个特定位点的谷氨酸的甲基化调控,而且仅二聚体受体能进行并完成信号传递过程[15]。E. coli的信号转导途径如图 1所示,MCPs通过调节CheA来介导浓度变化行为,CheA使CheY和CheB磷酸化,分别负责运动控制和感觉适应,支架蛋白CheW与CheA偶联后与受体相互作用[16]。磷酸化的CheY扩散到鞭毛马达处,促使细菌发生由逆时针到顺时针方向的旋转[17]。CheY-P被磷酸酶CheZ水解,进而消除了顺时针旋转信号的活性。甲基转移酶CheR和甲基酯酶CheB介导MCPs的可逆甲基化,从而稳定趋化敏感性的范围[16]。任何一个MCP和che基因的缺失或突变能使细菌失去对诱导剂信号的感知或转达能力,结果使细菌的趋化功能在某种程度上受损或者彻底丧失[18]。
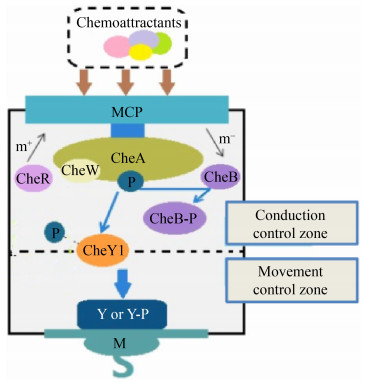 |
| 图 1 大肠杆菌的趋化信号通路 Figure 1 Signal transduction during chemotaxis in E. coli. |
| 图选项 |
迄今为止,对田菁根瘤菌的研究大多局限于菌株分离以及鉴定方面,尽管已经注释了YIC4027的全基因组序列[19],但是对其趋化系统方面的研究甚少。对YIC4027的趋化受体相关基因与其他物种进行序列相似性的比较分析,对我们了解其细胞功能以及研究与宿主田菁的共生作用至关重要。
1 材料和方法 1.1 多序列比对与系统发育分析 使用NCBI的protein BLAST (BLASTp)以及NCBI的position-specific-iterated-BLAST (psi-blast,http://blast.ncbi.nlm.nih.gov)来针对YIC4027 (GenBank:LYBW00000000.1,http://www.ncbi.nlm.nih.gov/nuccore/LYBW00000000.1)趋化受体基因查找其同源序列[20]。使用MAFFT软件(http://mafft.cbrc.jp/alignment/server)进行多序列比对分析,采用MEGA4软件[21]通过基因序列比对进行趋化受体对比分析。
1.2 鉴定趋化相关蛋白和趋化受体 利用MIST2在线数据库(http://mistdb.com/genomes/GCF_001723275.1)和SMART在线数据库(http://smart.embl.de)进行YIC4027趋化受体的查找及分析。利用通过NCBI非冗余数据库搜索与HMMER3[22]相匹配的Pfam MCP信号域来识别化学受体,序列标识利用Weblogo网站[23]在线生成。
1.3 启动子的预测与分析 利用在线软件Berkeley Neural Network Promoter Prediction[24]以及Virtual Footprint[25]进行分析,匹配位于预测ORF上游300 bp内的序列。
2 结果和分析 2.1 YIC4027趋化受体基因的特征 对YIC4027进行了基因组序列分析,发现它有两条趋化基因簇以及有13个MCP基因进行了注释,其中有3个是可溶性趋化受体,分别是CDS_2666、CDS_0566以及CDS_2559,其中CDS_0566和CDS_2559并不典型;其余都是跨膜趋化受体。为揭示YIC4027趋化受体的功能差别,我们把YIC4027与研究较为全面且相似性最高的Ensifer meliloti (原名Sinorhizobium meliloti)的趋化受体进行了比较分析,但Ensifer meliloti只有9个MCP,其中McpY和IcpA是可溶性趋化受体[26]。我们采用最大似然法来进行趋化受体基因序列比对分析(图 2)。结果表明,与CDS_0399具有同源性的McpX的周质结构域能直接感应季铵化合物[27],McpX是第一个已知的季铵类化合物(QACs)的细菌传感器,由于QACs通过使宿主苜蓿种子发芽后以趋化性相关的浓度渗出,因此提出了McpX有助于宿主特异性趋化性[28],由此可推测CDS_0399可能与宿主特异性有关;McpU是表达最强烈的化学受体,可直接感应脯氨酸信号,但不能识别多数的糖类和有机酸,通过感应种子分泌物中的脯氨酸来介导菌株与宿主的识别过程[29];与CDS_2666具有同源性的McpY含有2个PAS结构域,常用来感知光或氧气[26],可溶性趋化受体CDS_2666信号表达可能最为强烈,可能在菌株的趋氧性和调控固氮能力方面具有重要作用。其他趋化受体的功能目前尚不清楚,可能与多种细菌的代谢活动有关,例如运动趋化、病理、调控细胞内部能量代谢以及其他信号转导途径等,可进行后续相关试验进行探究。
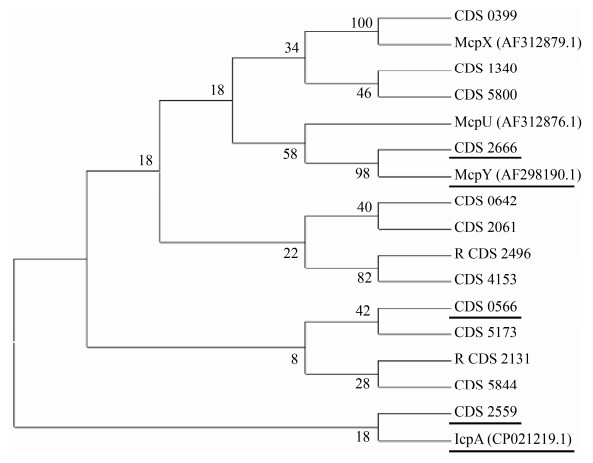 |
| 图 2 YIC4027趋化受体基因序列比较分析 Figure 2 Genetic organization of chemoreceptors of YIC4027 and comparison with E. meliloti. The soluble chemoreceptors are indicated with horizontal lines. |
| 图选项 |
2.2 YIC4027趋化受体的分布与特性 目前已经发现了13个MCP基因分布在YIC4027基因组中。MA结构域由高度保守的氨基酸位点组成,当被甲基转移酶CheR甲基化时,此结构域便会调控生化信号。不同的趋化受体蛋白可以感知外界的大量环境信号。大肠杆菌有5个化学感受器,其结构与2个跨膜螺旋、HAMP以及MA结构域相似[30-31]。我们推测YIC4027的MCPs在结构域组织和拓扑方面会表现出高度多样性,有10个跨膜趋化受体和3个可溶性趋化受体,其中可溶性趋化受体CDS_2666具有PAS结构域,CDS_2131和CDS_5844含有Cache2结构域(图 3-A)。PAS结构域位于受体N端,通过与小分子辅基结合来感受信号变化。Cache2结构域位于胞外,在识别小分子过程中发挥作用。我们预测CDS_2666、CDS_2131和CDS_5844对小分子信号物质的响应会更加强烈。在对MCPs的最初研究中,C端的细胞质结构被认为是由多序列比对支持的超级家族[32]。据Zhulin等的研究发现,微生物化学感受器中绝大多数MCPs的细胞质结构域(MCP-CD)的长度不同,以七肽作为长度单元,可以分为7个种类(24 H,28 H,34 H,36 H,38 H,40 H,44 H)[33]。MCPs细胞内的可溶区域分为3个模块,位于中间的是信号感受区域,两端则是甲基化区域,弹性变化区域把二者间隔开[32]。通过结构分析表明,E. coli的Tsr受体属于36 H[34]。多序列比对表明,尽管有部分MCPs序列缺失,13个MCP中除了CDS_5173属于40 H,其他12个MCP都属于36 H。
 |
| 图 3 E. alkalisoli YIC4027的结构域和保守位点 Figure 3 Conserved subdomains and sites of MCPs in E. alkalisoli. A: Domain architectures of MCPs in E. alkalisoli YIC4027 show diversity. TM1/TM2: transmembrane regions; MH1/MH2: MCP signaling domain and the interaction site of CheW and CheA; HAMP: histidine kinases, adenylyl cyclases, methyl-accepting chemotaxis proteins and phosphatases; PAS: the drosophila period clock protein (PER), the vertebrate aryl hydrocarbon receptor nuclear translocator (ARNT), and the drosophila single-minded protein (SIM); Cache: calcium and chemotaxis receptor domain. B: Class-specific conservation in the signaling subdomain. Eight heptads (N04–C04) in the centre of MCP-CD show extraordinarily high conservation. Each heptad is composed of intradimer (adeg) and interdimer (bcf) interaction sites. |
| 图选项 |
从理论上讲,MCP-CD是由3个子域组成,包括中心区域的一个信号域和两侧的被1个不同弹性束子域分隔开的1个甲基化单元[33]。MCP-CD中心(N04–C04,图 3-B)的8个七肽显示出极高的保守性。所有残基进入外二聚体adeg和内二聚体bcf的相互作用位点,两者均有助于CheW和CheA的连接,但一个是通过自我相互作用,另一个则是通过反向连接来稳定单体[33]。即使在高度保守的位置,也会产生氨基酸位点的差异性,在我们所研究的36 H类化合物中,一个苯丙氨酸残基在N03b位点很明显,在40 H类化合物中,位于N03b位点的则是天冬氨酸残基,Phe残基以及Asp残基在信号转导方面起到了非常重要的作用。
序列-[ASTG]-[ASTG]-x(2)-[EQ]-[EQ]-x(2)- [ASTG]-[ASTG]-具有十分重要的作用,它便于我们确定E. alkalisoli YIC4027的MCPs中所存在的关键甲基化位点。我们发现了同源序列共3段,分别是1个C端和2个N端的同源物,Glx残基在七肽的bc位点,这个基序被认为是甲基酯酶(CheB)和甲基转移酶(CheR)在MCPs中实现可逆甲基化的关键[35]。
2.3 趋化簇启动子的预测分析 启动子作为细菌在面对环境变化时对表达趋化相关蛋白含量调节的作用元件,发挥了不可替代的调控作用。位于细菌体内的RNA聚合酶与功能相关的特异因子σ28、σ54或者σ70结合[36],比如与鞭毛运动和趋化簇转录相关的因子有σ28和σ54。通常情况下,细菌对复杂趋化系统的调控需要σ28与σ54协同进行。在E. coli 中,鞭毛基因簇(flgA-flgN)受到σ28的调控[37]。在R. sphaeroides中,趋化基因簇保持基础的转录水平是靠σ70因子来进行调节,而鞭毛基因簇的合成则是由σ54因子调节[38]。
有研究表明,YIC4027的两条趋化基因簇都与细胞逆转和趋化相关,但存在明显的不同,che1基因簇主要控制趋化与细胞逆转,而che2基因簇对趋化和细胞的逆转影响较小,其参与调控EPS的生成以及竞争性结瘤的过程。在E. alkalisoli YIC4027两条趋化簇的上游,我们没有寻找到典型的σ28启动子结合位点。然后搜索σ54型RNA聚合酶的识别位点,以确定其是否具有σ54规则性,我们在che1簇的ORF上游300 bp以内发现了典型的–24/–12序列(图 4),但在che2簇上并没有发现同源序列,可以确定的是YIC4027的che2簇属于其他细胞功能(ACF)类别,简言之,che2基因簇在EPS生成和趋化中起到的作用提高了对α-变形菌趋化系统功能的认知。
 |
| 图 4 预测的σ54启动子序列 Figure 4 Predicted σ54 promoter sequences. For the E. alkalisoli major che1 cluster, the predicted transcription start site lies within 300 bp upstream of predicted operon GTG start codons. –24/–12 consensus sequences are in italics. The initiation codon (underlined) is also indicated. |
| 图选项 |
2.4 CheR结合部位分析 细菌的趋化感应是通过MCPs内特定的谷氨酰胺残基与谷氨酸的共价修饰进行介导。在E. coli趋化系统中,高峰度的受体Tsr和Tar分别感应环境中的丝氨酸和天冬氨酸,两者通过趋化受体C末端特定的五肽(NWETF)与CheR的β亚基结合[39]。在Thermotoga maritima趋化系统中,虽然位于最后的5个残基位点分别在第3和第5位点的疏水残基上表现出很高的保守性[40],但是CheR与趋化受体蛋白的结合依然与C端序列无关。上述的2个物种分别代表了典型的CheR五肽依赖型和非五肽依赖型。在五肽依赖型中,具有对结合五肽发挥重要作用的结构,有3个甘氨酸残基嵌入在β亚基结构域中,并且此序列十分保守,此外,在CheR的β亚基中还具有长链β环[41]。
为了确定E. alkalisoli YIC4027的CheR结合类型,我们比较了具有差异性的β亚基结构域序列。如图 5所示,β亚基序列不仅具有3个保守的甘氨酸残基,而且在182位的谷氨酰胺残基、187位的精氨酸残基、197位的精氨酸残基处表现出高度的保守性。根据序列比对,可以推测E. alkalisoli YIC4027的CheR对MCPs的甲基化不是五肽依赖型的。
 |
| 图 5 E. alkalisoli YIC4027的CheR蛋白β亚基序列比对 Figure 5 Sequence alignment of the β-subdomain of theCheR of E. alkalisoli YIC4027 and other species. Conserved amino acid residues that are important for CheR-pentapeptide interactions are highlighted. Small (G): green; positively charged (R): yellow; side-chain amine/amide containing residues (Q): blue. Conserved amino acid residues are colored. Hydrophobic (L, V, I, F, Y): red; small (G): green. |
| 图选项 |
3 讨论 田菁根瘤菌全基因组以及比较基因组水平的分析报道很少。迄今为止,大多数的田菁根瘤菌基因组学的研究仍为空白。因此,对田菁共生菌的研究不仅可以填补基因水平方面的空白,而且有助于我们更好地理解植物-菌株的共生体系,进而发展高效的结瘤菌株。
目前,全基因组测序已经完成的菌株正在逐步增多,这些菌株中的大多数都包括趋化基因及运动基因,并且多数土壤细菌含有两套或者更多的趋化系统[42]。我们通过相关生物信息学手段对E. alkalisoli YIC4027中与趋化相关的基因和蛋白进行分析,更好地明确YIC4027的信号传导途径。YIC4027具有两条趋化簇,分别是che1和che2。che1位于鞭毛基因的上游,其功能与共生和趋化有关,由σ54因子参与调控;che2的缺失不影响菌株的趋化和竞争性结瘤,对其成分分析表明che2可能有其他类型细胞功能。趋化受体除了一个是40 H,其余的均为36 H。最保守的核心序列是N4–C4,其中也存在与其他受体不同的氨基酸位点。对CheR进行序列比对后发现,YIC4027的甲基化酶CheR不是五肽依赖型的。
信号转导最基本的单位是MCPs单体形成的同源二聚体,在古菌和细菌的空间构象以及定位都较为相似。MCPs之间的协同工作,可以使细胞对极其微小浓度的趋化引诱剂产生级联放大响应[43]。通常情况下,MCPs的数量在一定程度上反映了细菌生存环境的复杂程度。R. leguminosarum含有6个跨膜受体(McpB-McpG),除McpB能感应棉子糖外,其他受体对常见的氨基酸和有机酸无响应[44];除了McpG,其余均参与结瘤过程[45]。A. brasilense是根际土壤细菌,可定殖于牧草或者谷类作物,其含有48个MCP,包括43个跨膜受体和5个具有PAS结构域的可溶性趋化受体[46],该菌株对能源物质,例如最理想的碳源琥珀酸或苹果酸,具有趋化效应[47]。与YIC4027相似性最高的Ensifer meliloti仅有9个MCP,而YIC4027具有13个MCP,其中一个是还没有被人们关注的40 H型受体以及含有多种配体结构域,可能在EPS的生成以及结瘤过程中发挥重要作用。综上说明YIC4027的趋化系统以及生存环境可能会更加复杂,这彰显出了其作为潜在40 H型受体模式菌株进行趋化研究的意义。目前,田菁根瘤菌的研究还处于起始阶段,这也正显示出了E. alkalisoli YIC4027作为模式菌株进行下一步趋化研究的重要性。
References
| [1] | Li XY, Li Y, Jiang N, Wu HL, Song WP, Xu HL, Xie ZH. Genetic diversity of the Rhizobia and screening of high-efficient growth-promoting strains isolated from Sesbania cannabina in Rudong County. Acta Microbiologica Sinica, 2015, 55(9): 1105-1116. (in Chinese) 李项岳, 李岩, 姜南, 吴海龙, 宋万平, 徐化凌, 解志红. 如东田菁根瘤菌遗传多样性及高效促生菌株筛选. 微生物学报, 2015, 55(9): 1105-1116. |
| [2] | Sun XM, Yu XL, Zhu RY, Wu XY, Tao CL. Study on Sesbania cannabina protein fish feed. Chinese Wild Plants, 1988, 7(3): 1-6. (in Chinese) 孙晓明, 于学玲, 朱荣誉, 吴兴义, 陶长龙. 田菁蛋白鱼饲料的研究. 中国野生植物, 1988, 7(3): 1-6. |
| [3] | Lee KB, De Backer P, Aono T, Liu CT, Suzuki S, Suzuki T, Kaneko T, Yamada M, Tabata S, Kupfer DM, Najar FZ, Wiley GB, Roe B, Binnewies TT, Ussery DW, D'Haeze W, Den Herder J, Gevers D, Vereecke D, Holsters M, Oyaizu H. The genome of the versatile nitrogen fixer Azorhizobium caulinodans ORS571. BMC Genomics, 2008, 9(1): 1-14. DOI:10.1186/1471-2164-9-1 |
| [4] | 李项岳. 黄河三角洲田菁根瘤菌遗传多样性、高效菌株筛选及应用. 中国科学院烟台海岸带研究所硕士学位论文, 2015. |
| [5] | Brencic A, Winans SC. Detection of and response to signals involved in host-microbe interactions by plant-associated bacteria. Microbiology and Molecular Biology Reviews, 2005, 69(1): 155-194. DOI:10.1128/MMBR.69.1.155-194.2005 |
| [6] | Miller LD, Yost CK, Hynes MF, Alexandre G. The major chemotaxis gene cluster of Rhizobium leguminosarum bv. viciae is essential for competitive nodulation. Molecular Microbiology, 2007, 63(2): 348-362. DOI:10.1111/j.1365-2958.2006.05515.x |
| [7] | Li Y, Mu BZ. Progress in chemotaxis of bacteria. Chinese Journal of Applied & Environmental Biology, 2006, 12(1): 135-139. (in Chinese) 李燕, 牟伯中. 细菌趋化性研究进展. 应用与环境生物学报, 2006, 12(1): 135-139. DOI:10.3321/j.issn:1006-687X.2006.01.033 |
| [8] | Fernández M, Morel B, Corral-Lugo A, Krell T. Identification of a chemoreceptor that specifically mediates chemotaxis toward metabolizable purine derivatives. Molecular Microbiology, 2016, 99(1): 34-42. DOI:10.1111/mmi.13215 |
| [9] | Blattner FR, Plunkett G, Bloch CA, Perna NT, Burland V, Riley M, Collado-Vides J, Glasner JD, Rode CK, Mayhew GF, Gregor J, Davis NW, Kirkpatrick HA, Goeden MA, Rose DJ, Mau B, Shao Y. The complete genome sequence of Escherichia coli K-12. Science, 1997, 277(5331): 1453-1462. DOI:10.1126/science.277.5331.1453 |
| [10] | Clarke S, Koshland DE. Membrane receptors for aspartate and serine in bacterial chemotaxis. The Journal of Biological Chemistry, 1979, 254(19): 9695-9702. DOI:10.1016/S0021-9258(19)83572-X |
| [11] | Biemann HP, Koshland DE. Aspartate receptors of Escherichia coli and Salmonella typhimurium bind ligand with negative and half-of-the-sites cooperativity. Biochemistry, 1994, 33(3): 629-634. DOI:10.1021/bi00169a002 |
| [12] | Lin LN, Li JY, Brandts JF, Weis RM. The serine receptor of bacterial chemotaxis exhibits half-site saturation for serine binding. Biochemistry, 1994, 33(21): 6564-6570. DOI:10.1021/bi00187a025 |
| [13] | Zhang WW, Zhang Z. Research progress on mechanism of bacterial chemotaxis. Microbiology China, 1993, 20(3): 175-179. (in Chinese) 张蔚文, 张灼. 细菌化学趋向性机理的研究进展. 微生物学通报, 1993, 20(3): 175-179. |
| [14] | Kim KK, Yokota H, Kim SH. Four-helical-bundle structure of the cytoplasmic domain of a serine chemotaxis receptor. Nature, 1999, 400(6746): 787-792. DOI:10.1038/23512 |
| [15] | Amin DN, Hazelbauer GL. The chemoreceptor dimer is the unit of conformational coupling and transmembrane signaling. Journal of Bacteriology, 2010, 192(5): 1193-1200. DOI:10.1128/JB.01391-09 |
| [16] | Hazelbauer GL, Lai WC. Bacterial chemoreceptors: providing enhanced features to two-component signaling. Current Opinion in Microbiology, 2010, 13(2): 124-132. DOI:10.1016/j.mib.2009.12.014 |
| [17] | Dyer CM, Vartanian AS, Zhou HJ, Dahlquist FW. A molecular mechanism of bacterial flagellar motor switching. Journal of Molecular Biology, 2009, 388(1): 71-84. DOI:10.1016/j.jmb.2009.02.004 |
| [18] | Bi SY, Lai LH. Bacterial chemoreceptors and chemoeffectors. Cellular and Molecular Life Sciences, 2015, 72(4): 691-708. DOI:10.1007/s00018-014-1770-5 |
| [19] | Dang XX, Xie ZH, Liu W, Sun Y, Liu XL, Zhu YQ, Staehelin C. The genome of Ensifer alkalisoli YIC4027 provides insights for host specificity and environmental adaptations. BMC Genomics, 2019, 20(1): 643. DOI:10.1186/s12864-019-6004-7 |
| [20] | Altschul SF, Madden TL, Sch?ffer AA, Zhang JH, Zhang Z, Miller W, Lipman DJ. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [21] | Tamura K, Dudley J, Nei M, Kumar S. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
| [22] | Finn RD, Clements J, Eddy SR. HMMER web server: interactive sequence similarity searching. Nucleic Acids Research, 2011, 39(suppl_2): W29-W37. |
| [23] | Crooks GE. WebLogo: a sequence logo generator. Genome Research, 2004, 14(6): 1188-1190. DOI:10.1101/gr.849004 |
| [24] | Studholme DJ, Buck M, Nixon T. Identification of potential σN-dependent promoters in bacterial genomes. Microbiology, 2000, 146(12): 3021-3023. DOI:10.1099/00221287-146-12-3021 |
| [25] | Münch R, Hiller K, Grote A, Scheer M, Klein J, Schobert M, Jahn D. Virtual Footprint and PRODORIC: an integrative framework for regulon prediction in prokaryotes. Bioinformatics, 2005, 21(22): 4187-4189. DOI:10.1093/bioinformatics/bti635 |
| [26] | Meier VM, Muschler P, Scharf BE. Functional analysis of nine putative chemoreceptor proteins in Sinorhizobium meliloti. Journal of Bacteriology, 2007, 189(5): 1816-1826. DOI:10.1128/JB.00883-06 |
| [27] | Webb BA, Karl Compton K, Casta?eda Salda?a R, Arapov TD, Keith Ray W, Helm RF, Scharf BE. Sinorhizobium meliloti chemotaxis to quaternary ammonium compounds is mediated by the chemoreceptor McpX. Molecular Microbiology, 2017, 103(2): 333-346. DOI:10.1111/mmi.13561 |
| [28] | Shrestha M, Compton KK, Mancl JM, Webb BA, Brown AM, Scharf BE, Schubot FD. Structure of the sensory domain of McpX from Sinorhizobium meliloti, the first known bacterial chemotactic sensor for quaternary ammonium compounds. The Biochemical Journal, 2018, 475(24): 3949-3962. DOI:10.1042/BCJ20180769 |
| [29] | Webb BA, Compton KK, del Campo JSM, Taylor D, Sobrado P, Scharf BE. Sinorhizobium meliloti chemotaxis to multiple amino acids is mediated by the chemoreceptor McpU. Molecular Plant-Microbe Interactions, 2017, 30(10): 770-777. DOI:10.1094/MPMI-04-17-0096-R |
| [30] | Ma QH, Roy F, Herrmann S, Taylor BL, Johnson MS. The aer protein of Escherichia coli forms a homodimer independent of the signaling domain and flavin adenine dinucleotide binding. Journal of Bacteriology, 2004, 186(21): 7456-7459. DOI:10.1128/JB.186.21.7456-7459.2004 |
| [31] | Watts KJ, Ma QH, Johnson MS, Taylor BL. Interactions between the PAS and HAMP domains of the Escherichia coli aerotaxis receptor Aer. Journal of Bacteriology, 2004, 186(21): 7440-7449. DOI:10.1128/JB.186.21.7440-7449.2004 |
| [32] | Le Moual H, Koshland J. Molecular evolution of the C-terminal cytoplasmic domain of a superfamily of bacterial receptors involved in taxis. Journal of Molecular Biology, 1996, 261(4): 568-585. DOI:10.1006/jmbi.1996.0483 |
| [33] | Alexander RP, Zhulin IB. Evolutionary genomics reveals conserved structural determinants of signaling and adaptation in microbial chemoreceptors. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(8): 2885-2890. DOI:10.1073/pnas.0609359104 |
| [34] | Kristich CJ, Ordal GW. Bacillus subtilis CheD is a chemoreceptor modification enzyme required for chemotaxis. The Journal of Biological Chemistry, 2002, 277(28): 25356-25362. DOI:10.1074/jbc.M201334200 |
| [35] | Perez E, West AH, Stock AM, Djordjevic S. Discrimination between different methylation states of chemotaxis receptor Tar by receptor methyltransferase CheR. Biochemistry, 2004, 43(4): 953-961. DOI:10.1021/bi035455q |
| [36] | Shah DS, Porter SL, Martin AC, Hamblin PA, Armitage JP. Fine tuning bacterial chemotaxis: analysis of Rhodobacter sphaeroides behaviour under aerobic and anaerobic conditions by mutation of the major chemotaxis operons and cheY genes. The EMBO Journal, 2000, 19(17): 4601-4613. DOI:10.1093/emboj/19.17.4601 |
| [37] | Chevance FFV, Hughes KT. Coordinating assembly of a bacterial macromolecular machine. Nature Reviews Microbiology, 2008, 6(6): 455-465. DOI:10.1038/nrmicro1887 |
| [38] | Martin AC, Gould M, Byles E, Roberts MAJ, Armitage JP. Two chemosensory operons of Rhodobacter sphaeroides are regulated independently by Sigma 28 and Sigma 54. Journal of Bacteriology, 2006, 188(22): 7932-7940. DOI:10.1128/JB.00964-06 |
| [39] | Wu JR, Li JY, Li GY, Long DG, Weis RM. The receptor binding site for the methyltransferase of bacterial chemotaxis is distinct from the sites of methylation. Biochemistry, 1996, 35(15): 4984-4993. DOI:10.1021/bi9530189 |
| [40] | Perez E, Stock AM. Characterization of the Thermotoga maritima chemotaxis methylation system that lacks pentapeptide-dependent methyltransferase CheR: MCP tethering. Molecular Microbiology, 2007, 63(2): 363-378. DOI:10.1111/j.1365-2958.2006.05518.x |
| [41] | Shiomi D, Zhulin IB, Homma M, Kawagishi I. Dual recognition of the bacterial chemoreceptor by chemotaxis-specific domains of the CheR methyltransferase. The Journal of Biological Chemistry, 2002, 277(44): 42325-42333. DOI:10.1074/jbc.M202001200 |
| [42] | Buchan A, Crombie B, Alexandre GM. Temporal dynamics and genetic diversity of chemotactic-competent microbial populations in the rhizosphere. Environmental Microbiology, 2010, 12(12): 3171-3184. DOI:10.1111/j.1462-2920.2010.02290.x |
| [43] | Tu YH. Quantitative modeling of bacterial chemotaxis: signal amplification and accurate adaptation. Annual Review of Biophysics, 2013, 42: 337-359. DOI:10.1146/annurev-biophys-083012-130358 |
| [44] | Yost CK, del Bel KL, Quandt J, Hynes MF. Rhizobium leguminosarum methyl-accepting chemotaxis protein genes are down-regulated in the pea nodule. Archives of Microbiology, 2004, 182(6): 505-513. DOI:10.1007/s00203-004-0736-7 |
| [45] | Yost CK, Clark KT, del Bel KL, Hynes MF. Characterization of the nodulation plasmid encoded chemoreceptor gene mcpG from Rhizobium leguminosarum. BMC Microbiology, 2003, 3(1): 1-10. |
| [46] | Bible AN, Stephens BB, Ortega DR, Xie ZH, Alexandre G. Function of a chemotaxis-like signal transduction pathway in modulating motility, cell clumping, and cell length in the alphaproteobacterium Azospirillum brasilense. Journal of Bacteriology, 2008, 190(19): 6365-6375. |
| [47] | Alexandre G, Greer SE, Zhulin IB. Energy taxis is the dominant behavior in Azospirillum brasilense. Journal of Bacteriology, 2000, 182(21): 6042-6048. |
