王智慧, 蒋先军


西南大学资源环境学院, 重庆 400715
收稿日期:2020-06-15;修回日期:2020-10-22;网络出版日期:2020-11-10
基金项目:国家自然科学基金(42077035,41671232);重庆市研究生科研创新(CYB19105);王智慧感谢国家留学基金委(CSC)奖学金
*通信作者:蒋先军, Tel: +86-23-68251249;Fax: +86-23-68250444;E-mail: jiangxj@swu.edu.cn.
摘要:[目的] 揭示典型农田旱地紫色土硝化微生物的群落组成及其对pH的响应规律。[方法] 针对同一母质发育但pH差异显著的3种紫色土,利用宏基因组技术深度测序研究土壤中硝化微生物丰度和群落,包括氨氧化古菌(ammonia-oxidizing archaea,AOA),氨氧化细菌(ammonia-oxidizing bacteria,AOB),亚硝酸盐氧化细菌(nitrite-oxidizing bacteria,NOB)和全程氨氧化细菌(complete ammonia oxidizer,Comammox)。[结果] 土壤中硝化微生物的丰度占总微生物的2.130%–6.082%。3种紫色土中AOA、AOB和NOB的相对丰度有显著差异:酸性紫色土中AOA的相对丰度显著大于碱性紫色土,而AOB则相反;NOB的相对丰度在中性紫色土中最高。所有土样中均发现了1种全程氨氧化细菌Candidatus Nitrospira inopinata(Ca.N. inopinata),其在中性紫色土中相对丰度最高,占总微生物的0.203%。3种不同pH紫色土中AOA均以Nitrososphaera为主,NOB均以Nitrospira为主;酸性紫色土中AOB以Nitroscoccus为主,而中性和石灰性紫色土中则以Nitrosospira为主。Pearson相关性分析发现,土壤pH和铵态氮是影响硝化微生物丰度最大的两个因子。[结论] Comammox存在于3种不同pH紫色土中,且偏好中性环境;AOA、AOB和NOB群落结构和相对丰度都存在显著差异,结合相关性分析发现土壤pH和铵态氮是导致差异最重要的两个因子。
关键词:氨氧化细菌氨氧化古菌亚硝酸盐氧化细菌全程氨氧化细菌氮循环
Metagenomics-centric assembly of nitrifying communities in three pH-distinct soils with the same origin of parent material
Zhihui Wang, Xianjun Jiang


College of Resources and Environment, Southwest University, Chongqing 400715, China
Received: 15 June 2020; Revised: 22 October 2020; Published online: 10 November 2020
*Corresponding author: Jiang Xianjun, Tel: +86-23-68251249;Fax: +86-23-68250444;E-mail: jiangxj@swu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (42077035, 41671232) and by the Graduate Research and Innovation Program of Chongqing (CYB19105). Zhihui Wang thanks the China Scholarship Council (CSC) for her fellowship
Abstract: [Objective] Aimed to reveal the community compositions of nitrifiers and its response to pH in typical agricultural upland purple soils. [Methods] Metagenomics was used to investigate the abundances and communities of soil nitrifiers, including ammonia-oxidizing archaea (AOA), ammonia-oxidizing bacteria (AOB), nitrite-oxidizing bacteria (NOB) and complete ammonia oxidizers (Comammox) in three purple soils that developed from the same parent material but varied in pH. [Results] The abundances of nitrifiers accounted for 2.130%-6.082% of the total microorganisms. The relative abundance of AOA in acid soil was significantly higher than that of alkaline soil, whereas AOB was the opposite; the relative abundance of NOB in neutral soil was the highest. A specie of Comammox named Candidatus Nitrospira inopinata (Ca. N. inopinata) was found in all soil samples, and its relative abundance was the highest in neutral soil, accounting for 0.203% of the total microorganisms. The community composition analyses of nitrifier showed that: the Nitrososphaera and Nitrospira were the dominant AOA and NOB in all soil samples; Nitroscoccus was predominant in AOB populations in acid soil, whereas in neutral and alkaline soils the dominant AOB was Nitrosospira. Pearson correlation analysis reveals that the abundances of these nitrifiers were driven by soil pH and NH4+. [Conclusion] Comammox existed in all soil samples and had a preference for a neutral environment; the relative abundances and community compositions of AOA, AOB, and NOB all showed a significant difference, soil pH and NH4+ were the two most influential factors.
Keywords: ammonia-oxidizing bacteriaammonia-oxidizing archaeanitrite-oxidizing bacteriacomammoxnitrogen cycling
微生物驱动的氮素循环过程包括固氮、氨化、硝化和反硝化等[1]。其中,硝化作用,即有氧条件下将氨转化为硝酸盐的过程,对地球生物化学循环以及植物养分高效的农业与环境地球科学研究具有重要意义。同时,微生物硝化过程也被认为是旱地土壤温室气体N2O的主要排放源[2]以及硝酸盐淋失的主要原因[3]。传统的硝化作用包括两步:氨氧化和亚硝酸盐氧化过程。氨氧化过程是硝化作用的第一步,也是限速步骤,一直以来受科学家们关注较多,而亚硝酸盐氧化过程常常被忽视,目前国内外关于亚硝酸盐氧化细菌(NOB)在土壤生态系统中的分布和影响因素还有待深入研究。最近新的研究发现,某些属于NOB硝化螺菌属的细菌可以进行单步硝化,即由一种微生物完成从铵态氮到硝态氮的过程,这类微生物被称为全程氨氧化细菌(Comammox)[4–5],研究证实它们广泛分布于土壤生态系统[6–7],可能在土壤硝化过程中发挥重要作用,但Comammox对全球硝化过程的相对贡献及其在元素生物地球化学循环过程中的作用仍有待深入研究。
硝化微生物占土壤总体微生物的比例较低,特异的标靶基因易导致特定物种的偏好性并产生偏差,在很大程度上限制了对硝化微生物的准确认识,而宏基因组深度测序则有可能克服这一难点。例如,以往的研究中,首先提取土壤微生物总DNA,进一步利用特异性功能引物如amoA基因引物进行扩增,随后开展克隆测序或者扩增子高通量多样性测序,这些分析可能带来较大的物种多样性偏差[8]。同时,如果采用16S rRNA基因通用引物扩增,考虑到16S rRNA基因单一片段的系统发育意义有限,也无法获得土壤微生物组结构全貌的准确认识,如新发现的全程氨氧化微生物,数据库中该类微生物的16S rRNA基因有限,无法进行准确比对。宏基因组深度测序则有可能克服这些难点,其特点是不依赖于扩增,直接从环境样品中提取的DNA进行深度测序,能够更加全面、真实、精确地反映微生物群落的遗传信息,也可为挖掘获取新的微生物或新的功能酶提供可能[9]。
环境要素是影响AOA和AOB地理分异规律的重要驱动力,如pH值、氧气、底物浓度、温度和气候条件等[10–12],其中pH值被认为是影响氨氧化微生物活性、丰度和群落结构的最重要的因素[13]。一般认为AOA在酸性土壤中占主导地位,而AOB则是在中性及碱性土壤中占主导地位[14–15]。pH不仅是土壤中AOA和AOB生态位分化的主要因素,还能显著影响AOA与AOB各种生态型的相对丰度[16–17],说明土壤pH值能形成被特定AOA或AOB生态型类群占据的生态位点。pH对NOB也具有显著的影响[18],NOB在中性或微偏碱(pH 7.6–8.3)的环境中具有更高的活性,在过酸或者过碱的环境下Nitrobacter和Nitrospira的生长均会受到抑制,从而导致亚硝酸氧化作用明显减弱[19–20]。相当一部分NOB为异养微生物,目前似乎尚未有严格嗜酸NOB报道。但值得注意的是,相比于氨氧化微生物,NOB具有更高的丰度及更广泛的种群多样性[21],NOB各类群在环境中分布极其不均,推测在不同pH土壤中NOB也可能存在明显的生态位分化。
紫色土是我国特有的土壤资源,广泛分布于以四川盆地为主的亚热带地区,该区域的紫色土占全国紫色土总面积的90%以上,而且土壤肥力较高,矿质元素丰富,是重要的农业土壤资源[22–23]。土壤常被认为是地球异质性最强的复杂历史自然体,其形成时间可高达上万甚至百万年,土壤发育及微生物形成过程很难模拟,而且由于紫色土的酸碱度与紫色母岩的含钙量有直接关系,随着母岩碳酸钙含量降低,紫色土的酸度会增高。因此,选择同一母质发育形成的不同pH土壤,能够最小化其它环境条件的影响。据此,本研究选取3种发育于同一母质但pH具有显著差异的典型农田旱地紫色土作为研究对象,进行Illumina Hiseq测序,然后通过宏基因组生物信息学分析,对主导硝化作用各个过程的功能微生物群落组成进行全面分析,探究pH对紫色土硝化微生物的群落结构的影响,旨在揭示紫色土中驱动土壤硝化过程的微生物机制,这对于紫色土地区合理利用氮肥,减少环境污染,促进土壤生态系统良性循环具有重要意义。
1 材料和方法 1.1 研究区域概况 供试土壤采集地信息同王智慧的描述[23]。简要说明如下:酸性紫色土和中性紫色土样品分别采集于重庆永川两地(29°11′ N,105°47′ E,海拔582 m;29°23′ N,105°59′ E,海拔387 m)。该研究区域属亚热带季风性湿润气候,年均气温17.7 ℃,极端最高气温42.1 ℃,极端最低气温–2.9 ℃,年均降雨量1015 mm,无霜期317 d。土壤为侏罗系沙溪庙组(J2s)灰棕紫色泥岩、页岩风化物母质上发育的酸性和中性紫色土。石灰性紫色土样品采集于中国科学院盐亭紫色土农业生态试验站(31°16′ N,105°28′ E,海拔400–600 m)。该研究区域属亚热带湿润季风气候区,年均气温16.7 ℃,极端最高气温39.5 ℃,极端最低气温–1.7 ℃,年均降水量863 mm,无霜期294 d。土壤为侏罗系上部蓬莱镇组(J3p)紫色砂泥岩母质上发育的石灰性紫色土。3种紫色土的种植制度均为传统三熟模式——小麦/玉米/红薯间套作,且氮肥(尿素)年施用量也相同,均为150 kg/hm2 (以N计),供试土壤基本理化性质详见王智慧的研究[23]。
1.2 土壤样品采集 土壤样品采集于2014年10月,在上述每个采样地点随机选择3个5 m×5 m的地块,每个地块之间至少相隔10 m,按照梅花形布点方式选取5个采样点,用直径10 cm的土钻取0–20 cm表层耕作土,混合均匀后带回实验室。将采回的土样除去石块和动植物残体等杂物后自然风干,当风干到土壤不黏手时,将土样过2 mm筛,然后放置于4 ℃冰箱保存用于后续提取DNA;另外一小部分土样完全风干后过1 mm筛用于土壤基本理化性质的测定。
1.3 分析测定方法 土壤pH值采用梅特勒LE438复合电极(mettler toledo,中国上海)测定(土水比1:2.5);总氮和土壤有机质分别采用改进后的克氏法及重铬酸钾氧化容量法(K2Cr2O7-H2SO4法)进行测定[24];土壤田间持水量测定方法参照国家农业行业标准测定。NH4+-N、NO3–-N浓度分别采用salicylate法和single reagent法,利用紫外分光光度计比色测定[25–26]。每个土样3次重复。
1.4 土壤总DNA提取及宏基因组测序 采用FastDNA? SPIN Kit for Soil(MP Biomedicals,美国)试剂盒按照说明提取土壤总DNA。对所提取的总DNA进行质量检测:1%琼脂糖凝胶电泳检测DNA完整性;超微量分光光度计NanoDrop 2000 (Thermo Fisher Scientific,美国)检测DNA浓度。后续的宏基因组测序和生物信息学部分由上海美吉生物医药科技有限公司利用高通量测序仪HiSeq? 2000 System (Illumina,美国)完成。每个土样均有3个生物学重复。宏基因组测序原始数据已经提交到NCBI的SRA数据库,登录号SRP241537。
1.5 数据处理 首先用软件Seqprep和Sickle对下机原始序列进行去接头、质量剪切以及去除污染等优化处理,然后使用拼接软件SOAPdenovo和MetaGene分别对优质序列进行拼接组装和基因预测,使用BLASTp (BLAST Version 2.2.28+,http://blast.ncbi.nlm.nih.gov/Blast.cgi)将基因集与NR数据库进行比对(BLAST比对参数设置期望值e-value为1e–5),并通过NR库对应的分类学信息数据库获得硝化微生物物种注释,然后使用物种对应的基因丰度总和计算该物种的相对丰度[23]。采用SPSS 18.0进行统计分析,单因素方差分析(one-way ANOVA)检验处理间均值差异的显著性(差异水平P < 0.05)并用Turkey法进行多重比较,采用Pearson法进行硝化微生物相对丰度与土壤理化性质之间的相关性分析。
2 结果和分析 2.1 宏基因组测序基本数据 经过Hiseq高通量测序,每个土样产生大约11 Gb的数据量,原始序列经过滤,去除低质量序列、模糊序列和嵌合体序列后,酸性、中性和石灰性紫色土分别得到平均73897575、88056082、71098226条clean reads (表 1)。宏基因组测序几乎涵盖了环境样品中的全部微生物,所有样品的覆盖度都在97%以上,说明结果能够比较全面真实地反映土壤微生物群落组成。
表 1. 土壤基因组DNA测序数据统计 Table 1. Statistics of soil genomic DNA sequencing data
| Soil type | Raw data | Clean data | Coverage/% | |||
| Raw reads | Raw bases/bp | Clean reads | Clean bases/bp | |||
| Acidic soil | 71409070 | 10711360500 | 70778280 | 10549196933 | 98.22 | |
| 86581948 | 12987292200 | 85686669 | 12767960951 | 98.41 | ||
| 66190980 | 9928647000 | 65227775 | 9703814331 | 97.94 | ||
| Neutral soil | 90940134 | 13641020100 | 90215067 | 13426340810 | 97.80 | |
| 81093518 | 12164027700 | 80455568 | 11969294554 | 97.09 | ||
| 94489232 | 14173384800 | 93497610 | 13915109045 | 97.71 | ||
| Alkaline soil | 70338366 | 10550754900 | 69437104 | 10325409024 | 97.60 | |
| 73765948 | 11064892200 | 72179735 | 10679436119 | 97.41 | ||
| 72826908 | 10924036200 | 71677839 | 10648925058 | 97.14 | ||
表选项
2.2 不同pH紫色土硝化微生物相对丰度 将clean reads与NR数据库进行比对,挑出属于已知硝化功能微生物的序列并计算其相对丰度。表 2展示了所有涉及硝化作用的功能微生物占总体微生物群落的相对丰度。在3种不同pH紫色土中发现的硝化功能微生物相对丰度在2.130%–6.082%,硝化微生物丰度在中性紫色土中最高,碱性紫色土中最低。AOA的相对丰度在酸性紫色土中最高,显著高于中性和碱性紫色土(P < 0.05),属水平上的三类AOA菌群的相对丰度与AOA呈相同的趋势,都是在酸性紫色土中最高,而在碱性紫色土中最低。与AOA相反,AOB相对丰度在碱性紫色土中丰度最高,显著高于中性和酸性紫色土(P < 0.05)。在属水平上,Nitrosospira和Nitrosomonas属AOB在碱性紫色土中的相对丰度最高,而Nitroscoccus属AOB在酸性紫色土中相对丰度最高。对于NOB,其总丰度在中性紫色土中最高,在酸性紫色土中最低;Nitrobacter、Nitrococcus和Nitrolancea属NOB在石灰性紫色土中的相对丰度最高;Nitrospira属NOB在中性紫色土中相对丰度最高,分别是酸性和石灰性紫色土的5倍和3倍。通过与已知基因数据库进行比对,所有紫色土样品中都发现了1种全程氨氧化细菌,即Ca. N. inopinata,该细菌以前被认为是严格的NOB,只能氧化NO2–,然而Daims等通过富集培养分离纯菌株,并通过测序发现其也含有与氨氧化过程相关的全部基因,能够将氨氮完全氧化为硝酸盐[4]。Ca. N. inopinata在中性土中相对丰度最高,占总体微生物丰度的0.203%,显著高于酸性和石灰性紫色土(P < 0.05)。
表 2. 3种不同pH紫色土硝化功能微生物的相对丰度 Table 2. The relative abundances of nitrification-related functional microorganisms in purple soils with different pH
| Functional groups | Genus level | Acidic soil/% | Neutral soil/% | Alkaline soil/% |
| AOA | Nitrosopumilus | 0.436a | 0.050b | 0.009c |
| Nitrosotalea | 0.060a | 0.011b | 0.002c | |
| Nitrososphaera | 4.343a | 3.670b | 0.234c | |
| Subtotal | 4.839a | 3.731b | 0.245c | |
| AOB | Nitrosospira | 0.062c | 0.185b | 0.345a |
| Nitrosomonas | 0.049c | 0.097b | 0.243a | |
| Nitroscoccus | 0.193a | 0.133c | 0.181b | |
| Subtotal | 0.304c | 0.415b | 0.769a | |
| NOB | Nitrobacter | 0.095b | 0.065c | 0.227a |
| Nitrococcus | 0.060b | 0.033c | 0.099a | |
| Nitrolancea | 0.170b | 0.153c | 0.203a | |
| Nitrospina | 0.027b | 0.057a | 0.058a | |
| Nitrospira | 0.310c | 1.628a | 0.516b | |
| Subtotal | 0.662c | 1.936a | 1.103b | |
| Comammox | Ca. N. inopinata | 0.031b | 0.203a | 0.013c |
| Total | 5.836b | 6.082a | 2.130c | |
| Different letters in the same row indicate significant difference at 0.05 level in three purple soils. | ||||
表选项
2.3 不同pH紫色土氨氧化微生物群落组成分析 各样本中氨氧化古菌群落在属分类水平上的分布比例见图 1。不同pH紫色土中都发现了3个相同的AOA属:Nitrosopumilus、Nitrosotalea和Nitrososphaera。不同pH紫色土中AOA群落都是以Nitrososphaera属AOA为主,其占总AOA的比例达到90%以上。但是同一个属在不同pH的紫色土中占比存在差异,Nitrosopumilus属AOA在酸性紫色土中所占比例(9.0%)显著高于其在中性紫色土中的占比(1.3%);而Nitrososphaera属则相反,其在中性紫色土中占比最大,达到总AOA的98.3%,在酸性紫色土中则最小,为总AOA的90.1%。
 |
| 图 1 3种不同pH紫色土AOA各个属所占比例图 Figure 1 The percentage of each genus in AOA in purple soils with different pH. |
| 图选项 |
各样本中氨氧化细菌群落在属分类水平上的分布比例见图 2。3种不同pH的紫色土中主要包括3个AOB属:Nitrosospira、Nitrosomonas和Nitroscoccus,不同属AOB在不同的pH紫色土中分布具有明显差异(图 2)。在酸性紫色土中的优势菌群为Nitroscoccus属AOB,其占比达到总AOB的63.4%;然而在中性和酸性紫色土中占主导的AOB却是Nitrosospira属,其占比分别为44.6%和44.7%。Nitrosomonas属AOB在酸性紫色土中的占比(16.2%)显著低于其在石灰性紫色土中的占比(31.3%)(P < 0.05),说明Nitrosomonas属AOB更适合高pH营养丰富的环境。
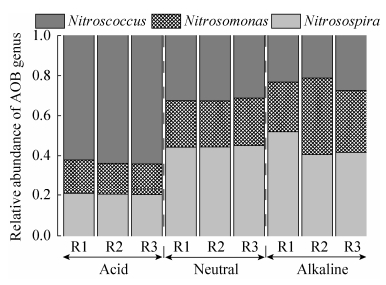 |
| 图 2 三种不同pH紫色土AOB各个属所占比例图 Figure 2 The percentage of each genus in AOB in purple soils with different pH. |
| 图选项 |
2.4 不同pH紫色土亚硝酸盐氧化微生物群落组成分析 各样本中亚硝酸盐氧化细菌群落在属分类水平上的分布比例见图 3。3种不同pH的紫色土中发现了5个NOB属:Nitrospira、Nitrolancea、Nitrospina、Nitrococcus和Nitrobacter,并且不同属在3种土壤NOB群落中占比差异显著(图 3)。在酸性、中性和碱性紫色土中都是Nitrospira属NOB占主导,其占比分别为46.9%、84.1%和46.8%,Nitrospira属NOB在中性紫色土中占比显著大于酸性和石灰性紫色土(P < 0.05);Nitrolancea属NOB在酸性紫色土中占比(25.7%)显著高于其在中性紫色土中的占比(7.9%)(P < 0.05);而Nitrobacter属NOB在石灰性紫色土中占比(20.7%)显著高于其在酸性和中性紫色土中的占比(分别为14.3%和3.4%)(P < 0.05)。
 |
| 图 3 三种不同pH紫色土NOB各个属所占比例图 Figure 3 The percentage of each genus in NOB in purple soils with different pH. |
| 图选项 |
2.5 环境因子对紫色土硝化微生物群落组成的影响 用Pearson相关性系数对土壤硝化微生物的相对丰度和土壤理化性质之间的相关性进行表征,结果见下表(表 3)。
表 3. 土壤基本理化性质与硝化功能微生物的相关性 Table 3. Corrections of nitrification-related functional microorganisms and soil properties
| Functional groups | Genus level | pH | OM | TN | NH4+-N | NH3 | NO3–-N | WHC |
| AOA | Nitrososphaera | 0.780* | –0.792* | –0.777* | –0.963** | 0.200 | 0.351 | –0.854* |
| Nitrosotalea | –0.788* | 0.513 | 0.490 | 0.807* | 0.175 | 0.017 | 0.671 | |
| Nitrosopumilus | –0.808* | 0.818* | 0.804* | 0.972** | –0.244 | –0.393 | 0.867* | |
| AOB | Nitrosospira | 0.934** | –0.930** | –0.936** | –0.949** | 0.534 | 0.661 | –0.919** |
| Nitrosomonas | 0.853* | –0.856* | –0.833* | –0.664 | 0.808* | 0.747* | –0.696 | |
| Nitroscoccus | –0.993** | 0.992** | 0.986** | 0.926** | –0.698 | –0.761* | 0.919** | |
| NOB | Nitrospira | 0.237 | –0.251 | –0.226 | –0.643 | –0.452 | –0.267 | –0.427 |
| Nitrobacter | 0.131 | –0.114 | –0.140 | 0.721* | 0.741* | 0.580 | 0.109 | |
| Nitrolancea | –0.622 | 0.627 | 0.608 | 0.900** | 0.034 | –0.153 | 0.706* | |
| Nitrospina | 0.314 | –0.295 | –0.312 | 0.120 | 0.820* | 0.708* | –0.034 | |
| Nitrococcus | –0.250 | 0.269 | 0.242 | 0.651 | 0.438 | 0.264 | 0.453 | |
| Comammox | Ca. N. inopinata | 0.140 | –0.156 | –0.141 | –0.560 | –0.491 | –0.355 | –0.348 |
| OM: organic matters; TN: total nitrogen; WHC: water holding capability. *: significant difference at 0.05 level; **: significant difference at 0.01 level. | ||||||||
表选项
相关性分析结果表明土壤中AOA和AOB各类群的相对丰度均与pH有显著相关性(P < 0.05),其中Nitrosospira属AOB与土壤pH呈极显著正相关(P < 0.01),Nitroscoccus属AOB与土壤pH呈极显著负相关(P < 0.01);除了土壤pH,AOA和AOB还与有机质、总氮、铵氮以及田间持水量有显著相关关系。与氨氧化微生物相比,土壤中NOB各类群与所列土壤理化性质之间的相关性较弱,Nitrolancea和Nitrobacter属NOB与土壤铵氮呈显著正相关(P < 0.05);Nitrobacter和Nitrospina属NOB与游离态NH3也存在显著相关性(P < 0.05);Nitrolancea属NOB与土壤田间持水量呈显著正相关(P < 0.05);Nitrospina属NOB与土壤硝氮呈显著正相关(P < 0.05)。全程氨氧化细菌Ca. N. inopinata与所有的土壤性质都没有显著相关性。
3 讨论 硝化微生物广泛分布于地球生态系统,根据主导的代谢途径不同被分为三类,包括氨氧化微生物(AOA和AOB),亚硝酸盐氧化微生物(NOB)以及新发现的全程氨氧化微生物(Comammox)。本研究通过宏基因组深度测序发现土壤中硝化微生物占总体微生物的比例在2.130%?6.082%,AOA相对丰度在酸性和中性紫色土中较高,AOB相对丰度在碱性紫色土中最高,NOB和Comammox相对丰度在中性紫色土中最高。宏基因组测序技术不依赖于扩增,直接从环境样品中提取的DNA进行深度测序,是针对环境中微生物群落所有DNA的分析,相较于传统的克隆测序及扩增子测序,宏基因组测序不仅测序范围广,而且测序深度也较高[9],通过某些算法分析甚至可以识别出测序数据中极低丰度的菌株(相对丰度小于0.00001%)[27]。本研究在不同pH紫色土中共检测到了3个AOA属:Nitrosopumilus、Nitrososphaera、Nitrosotalea;3个AOB属:Nitroscoccus、Nitrosospira、Nitrosomonas;5个NOB属:Nitrobacter、Nitrococcus、Nitrolancea、Nitrospira、Nitrospina;以及1种Comammox,而以往的研究通常只能获得一两个属的硝化微生物类群的信息,本研究的结果表明宏基因组测序可以大大提高对典型农田旱地不同pH紫色土中丰度较低的硝化微生物类群的了解。而且随着测序技术的飞速发展和分析流程的不断完善,相信在今后环境微生物组的研究中宏基因组技术将会发挥更大的技术优势,成为研究复杂微生物群落的有力工具。
对硝化微生物进一步的分析发现各个类群在不同pH紫色土中的相对丰度差异显著,AOA、AOB和NOB群落结构也有显著差异,说明特定的微生物类群可能选择了不同土壤pH环境,体现出生态位分化特征。例如,Nitrosopumilus属AOA在酸性紫色土的占比显著高于其在中性及碱性紫色土中的占比(P < 0.05),且相关性分析发现其与土壤pH呈显著负相关,说明Nitrosopumilus属AOA更偏好低pH环境。AOB的群落结构在不同pH的土壤中也有明显差异:在酸性紫色土中AOB以Nitroscoccus为主,占比达到63.4%,而在中性和石灰性紫色土中则以Nitrosospira为主,占比分别为44.6%和44.7%。在中性和石灰性紫色土中Nitrosospira和Nitrosomonas属AOB的占比要显著多于酸性紫色土(P < 0.05),这两个属于β-变形菌纲的类群是陆地生态系统中常见的AOB,而在酸性紫色土中占主导地位的Nitrosococcus属AOB则是普遍存在于海洋生态系统或者高盐环境[28],推测可能是由于Nitrosococcus属AOB在营养贫乏的酸性土壤中比β-变形菌纲的类群更具竞争优势。Pan等在低含盐量(0.6 mmol/L)的草地土壤中也检测出高丰度的Nitrosococcus属AOB,说明Nitrosococcus属AOB能适应的环境可能比以前所认为的要更宽广[29]。NOB被分为4个门下的7个属:属于变形菌门的Nitrobacter,Nitrotoga,Nitrococcus,绿弯菌门的Nitrolancea,硝化螺菌门的Nitrospira,硝化刺菌门的Nitrospina以及新发现的未培养海洋菌“Candidatus Nitromaritima”[21]。在本研究中,共检测到了除Nitrotoga和“Candidatus Nitromaritima”之外的5个属,而以往通过16S rRNA基因或相关功能基因只能针对Nitrobacter和Nitrospira属NOB进行研究。NOB各个属相对丰度及群落结构在3种不同pH的紫色土中也有显著差异:Nitrospira属NOB在中性紫色土中的相对丰度和占比都显著大于酸性和石灰性紫色土(P < 0.05);而Nitrobacter属NOB在石灰性紫色土中的相对丰度和占比都显著高于其在酸性和中性紫色土(P < 0.05),这可能是由于NOB不同类群具有不同的生存策略。与Nitrospira相比,Nitrobacter有着更低的底物亲和力,已有很多研究发现在富营养环境中Nitrobacter占主导地位,而在亚硝酸盐浓度较低环境下,Nitrospira比Nitrobacter更具竞争优势[30–31]。
通过相关性分析发现,土壤AOA和AOB群落的分布是由多因素驱动的,包括土壤pH值、有机质、总氮、铵态氮、硝态氮和田间持水量,这些因素都与氨氧化微生物的群落结构存在显著的相关性(表 3),这与Yao等的研究结果是一致的[32]。其中土壤pH对所有AOA和AOB类群都有显著相关性,其影响最大,说明pH是驱动氨氧化微生物分布最重要的因子。此外,有机质也是影响AOA和AOB群落组成的重要因子。本研究结果发现不同的AOA/AOB类群与有机质的相关性并不一致,已有的相关研究结果也是不一致的:如外源有机质以及植物根系分泌的一些易利用有机碳均能促进AOA的生长[33–34],然而也有研究表明有机质能够抑制AOA的生长[35–36],这可能是由于AOA/AOB的不同类群具有的生理生化差异所导致的,这也解释了为什么以往关于有机质对土壤氨氧化微生物的影响效应存在争议。相关性分析结果并没有发现NOB类群与土壤pH之间存在显著相关性,但是发现铵态氮、游离态氨和田间持水量与NOB某些类群有显著相关性。铵态氮可以被AOA/AOB氧化为亚硝酸盐,间接为NOB提供底物。由于NOB不同类群具有不同的生存策略,与Nitrospira相比,Nitrobacter有着更低的底物亲和力,因此在高氮投入的环境中Nitrobacter占主导地位,而在亚硝酸盐浓度较低环境中则是Nitrospira更具竞争优势[30–31],Han等的研究也发现长期施用氮肥能显著提高土壤Nitrobacter的丰度[18],说明底物浓度可能是NOB生态位分化的一个重要环境因素。
我们前期对紫色土的研究表明,pH能显著影响土壤硝化活性以及氨氧化微生物的丰度[37],通过Miseq测序发现3种不同的紫色土中总微生物和硝化微生物群落结构具有显著差异[38]。并且还发现NOB/AOB的比值在3种不同酸碱性紫色土各处理中最高可达到13,间接表明Comammox可能在紫色土硝化作用中发挥重要作用[38],但当时并没有直接的证据来证明土壤中Comammox的丰度及活性。本研究通过对宏基因组测序数据进行深度挖掘,直接证明了土壤中Comammox的存在,在3种不同pH的紫色土中都发现了1种全程氨氧化微生物(Ca. N. inopinata),其相对丰度在0.013%–0.203%,且在中性土壤中最高(0.203%),说明Ca. N. inopinata这种全程氨氧化微生物更适合在中性土壤中生长。然而这种全程氨氧化微生物是否在土壤硝化作用中发挥作用,以及其对传统硝化作用及传统硝化微生物的影响等还有待进一步的研究。
4 结论 通过Hiseq高通量测序对3种pH差异显著的典型农田旱地紫色土进行宏基因组测序研究,结果发现土壤中硝化微生物的丰度占总微生物的2.130%–6.082%,3种不同pH紫色土中都发现了1种全程氨氧化细菌(Ca. N. inopinata),且其在中性紫色土中相对丰度最高,占总微生物的0.203%,说明其更适宜在中性条件下生长。对硝化微生物丰度和群落分析发现,3种不同pH紫色土中AOA、AOB和NOB的群落结构和相对丰度都有显著差异,土壤pH和铵态氮是导致差异的重要因子。
References
| [1] | Gruber N, Galloway JN. An earth-system perspective of the global nitrogen cycle. Nature, 2008, 451(7176): 293-296. DOI:10.1038/nature06592 |
| [2] | Santoro AE, Buchwald C, McIlvin MR, Casciotti KL. Isotopic signature of N2O produced by marine ammonia-oxidizing archaea. Science, 2011, 333(6047): 1282-1285. DOI:10.1126/science.1208239 |
| [3] | Kowalchuk GA, Stephen JR. Ammonia-oxidizing bacteria: a model for molecular microbial ecology. Annual Review of Microbiology, 2001(55): 485-529. |
| [4] | Daims H, Lebedeva EV, Pjevac P, Han P, Herbold C, Albertsen M, Jehmlich N, Palatinszky M, Vierheilig J, Bulaev A, Kirkegaard RH, von Bergen M, Rattei T, Bendinger B, Nielsen PH, Wagner M. Complete nitrification by Nitrospira bacteria. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [5] | van Kessel MAHJ, Speth DR, Albertsen M, Nielsen PH, den Camp HJMO, Kartal B, Jetten MSM, Lücker S. Complete nitrification by a single microorganism. Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459 |
| [6] | Hu HW, He JZ. Comammox-a newly discovered nitrification process in the terrestrial nitrogen cycle. Journal of Soils and Sediments, 2017, 17(12): 2709-2717. DOI:10.1007/s11368-017-1851-9 |
| [7] | Orellana LH, Chee-Sanford JC, Sanford RA, L?ffler FE, Konstantinidis KT. Year-round shotgun metagenomes reveal stable microbial communities in agricultural soils and novel ammonia oxidizers responding to fertilization. Applied and Environmental Microbiology, 2018, 84(2): e01646-17. |
| [8] | Ye L, Zhang T, Wang TT, Fang ZW. Microbial structures, functions, and metabolic pathways in wastewater treatment bioreactors revealed using high-throughput sequencing. Environmental Science and Technology, 2012, 46(24): 13244-13252. DOI:10.1021/es303454k |
| [9] | Ma HX, Zhang LL, Sun XM, Zhang HQ, He MX, Chen GJ, Wang LS. Understanding microbial communities and their functions by meta-omics approaches.. Microbiology China, 2015, 42(5): 902-912. (in Chinese) 马海霞, 张丽丽, 孙晓萌, 张怀强, 何明雄, 陈冠军, 王禄山. 基于宏组学方法认识微生物群落及其功能. 微生物学通报, 2015, 42(5): 902-912. |
| [10] | He JZ, Hu HW, Zhang LM. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils. Soil Biology and Biochemistry, 2012, 55: 146-154. DOI:10.1016/j.soilbio.2012.06.006 |
| [11] | Erguder TH, Boon N, Wittebolle L, Marzorati M, Verstraete W. Environmental factors shaping the ecological niches of ammonia-oxidizing archaea. FEMS Microbiology Reviews, 2009, 33(5): 855-869. DOI:10.1111/j.1574-6976.2009.00179.x |
| [12] | Zhalnina K, de Quadros PD, Camargo FAO, Triplett EW. Drivers of archaeal ammonia-oxidizing communities in soil. Frontiers in Microbiology, 2012(3): 210. |
| [13] | Hatzenpichler R. Diversity, physiology, and niche differentiation of ammonia-oxidizing archaea. Applied and Environmental Microbiology, 2012, 78(21): 7501-7510. |
| [14] | Leininger S, Urich T, Schloter M, Schwark L, Qi J, Nicol GW, Prosser JI, Schuster SC, Schleper C. Archaea predominate among ammonia-oxidizing prokaryotes in soils. Nature, 2006, 442(7104): 806-809. DOI:10.1038/nature04983 |
| [15] | Hu HW, Zhang LM, Dai Y, Di HJ, He JZ. pH-dependent distribution of soil ammonia oxidizers across a large geographical scale as revealed by high-throughput pyrosequencing. Journal of Soils and Sediments, 2013, 13(8): 1439-1449. DOI:10.1007/s11368-013-0726-y |
| [16] | Nicol GW, Leininger S, Schleper C, Prosser JI. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria. Environmental Microbiology, 2008, 10(11): 2966-2978. DOI:10.1111/j.1462-2920.2008.01701.x |
| [17] | Shen JP, Zhang LM, Di HJ, He JZ. A review of ammonia-oxidizing bacteria and archaea in Chinese soils. Frontiers in Microbiology, 2012, 3: 296. |
| [18] | Han S, Zeng LY, Luo XS, Xiong X, Wen SL, Wang B, Chen WL, Huang QY. Shifts in Nitrobacter-and Nitrospira-like nitrite-oxidizing bacterial communities under long-term fertilization practices. Soil Biology and Biochemistry, 2018, 124: 118-125. DOI:10.1016/j.soilbio.2018.05.033 |
| [19] | Blackburne R, Vadivelu VM, Yuan ZG, Keller J. Kinetic characterisation of an enriched Nitrospira culture with comparison to Nitrobacter. Water Research, 2007, 41(14): 3033-3042. DOI:10.1016/j.watres.2007.01.043 |
| [20] | Grunditz C, Dalhammar G. Development of nitrification inhibition assays using pure cultures of nitrosomonas and nitrobacter. Water Research, 2001, 35(2): 433-440. DOI:10.1016/S0043-1354(00)00312-2 |
| [21] | Daims H, Lücker S, Wagner M. A new perspective on microbes formerly known as nitrite-oxidizing bacteria. Trends in Microbiology, 2016, 24(9): 699-712. DOI:10.1016/j.tim.2016.05.004 |
| [22] | 中国科学院成都分院土壤研究室. 中国紫色土(上篇). 北京: 科学出版社, 1991. |
| [23] | 王智慧. 不同pH紫色土中硝化作用及硝化微生物宏基因组研究. 西南大学硕士学位论文, 2017. |
| [24] | 杨剑虹, 王成林, 代亨林. 土壤农化分析与环境监测. 北京: 中国大地出版社, 2008. |
| [25] | 鲍士旦. 土壤农化分析. 第3版. 北京: 中国农业科技出版社, 2000. |
| [26] | Song G, Sun B, Jiao JY. Comparison between ultraviolet spectrophotometry and other methods in determination of soil nitrate-N. Acta Pedologica Sinica, 2007, 44(2): 288-293. (in Chinese) 宋歌, 孙波, 教剑英. 测定土壤硝态氮的紫外分光光度法与其他方法的比较. 土壤学报, 2007, 44(2): 288-293. DOI:10.3321/j.issn:0564-3929.2007.02.014 |
| [27] | Cleary B, Brito IL, Huang K, Gevers D, Shea T, Young S, Alm EJ. Detection of low-abundance bacterial strains in metagenomic datasets by eigengenome partitioning. Nature Biotechnology, 2015, 33(10): 1053-1060. DOI:10.1038/nbt.3329 |
| [28] | Campbell MA, Chain PSG, Dang HY, Sheikh AFE, Norton JM, Ward NL, Ward BB, Klotz MG. Nitrosococcus watsonii sp. nov., a new species of marine obligate ammonia-oxidizing bacteria that is not omnipresent in the world's oceans: calls to validate the name 'Nitrosococcus halophilus' and 'Nitrosomonas mobilis'. FEMS Microbiology Ecology, 2011, 76(1): 39-48. DOI:10.1111/j.1574-6941.2010.01027.x |
| [29] | Pan H, Liu HY, Liu YW, Zhang QC, Luo Y, Liu XM, Liu YM, Xu JM, Di HJ, Li Y. Understanding the relationships between grazing intensity and the distribution of nitrifying communities in grassland soils. Science of the Total Environment, 2018, 634: 1157-1164. DOI:10.1016/j.scitotenv.2018.04.117 |
| [30] | Attard E, Poly F, Commeaux C, Laurent F, Terada A, Smets BF, Recous S, Le Roux X. Shifts between Nitrospira- and Nitrobacter-like nitrite oxidizers underlie the response of soil potential nitrite oxidation to changes in tillage practice. Environmental Microbiology, 2010, 12(2): 315-326. DOI:10.1111/j.1462-2920.2009.02070.x |
| [31] | Le Roux X, Bouskill NJ, Niboyet A, Barthes L, Dijkstra P, Field CB, Hungate BA, Lerondelle C, Pommier T, Tang JY, Terada A, Tourna M, Poly F. Predicting the responses of soil nitrite-oxidizers to multi-factorial global change: a trait-based approach. Frontiers in Microbiology, 2016, 7: 628. |
| [32] | Yao HY, Campbell CD, Chapman SJ, Freitag TE, Nicol GW, Singh BK. Multi-factorial drivers of ammonia oxidizer communities: evidence from a national soil survey. Environmental Microbiology, 2013, 15(12): 2545-2556. |
| [33] | Wessén E, Hallin S, Philippot L. Differential responses of bacterial and archaeal groups at high taxonomical ranks to soil management. Soil Biology and Biochemistry, 2010, 42(10): 1759-1765. DOI:10.1016/j.soilbio.2010.06.013 |
| [34] | Hartmann AA, Barnard RL, Marhan S, Niklaus PA. Effects of drought and N-fertilization on N cycling in two grassland soils. Oecologia, 2013, 171(3): 705-717. DOI:10.1007/s00442-012-2578-3 |
| [35] | K?nneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA. Isolation of an autotrophic ammonia-oxidizing marine archaeon. Nature, 2005, 437(7058): 543-546. DOI:10.1038/nature03911 |
| [36] | Bates ST, Berg-Lyons D, Caporaso JG, Walters WA, Knight R, Fierer N. Examining the global distribution of dominant archaeal populations in soil. The ISME Journal, 2011, 5(5): 908-917. DOI:10.1038/ismej.2010.171 |
| [37] | Wang ZH, Meng Y, Zhu-Barker X, He XH, Horwath WR, Luo HY, Zhao YP, Jiang XJ. Responses of nitrification and ammonia oxidizers to a range of background and adjusted pH in purple soils. Geoderma, 2019(334): 9-14. |
| [38] | Cao YQ, Yan XJ, Luo HY, Jia ZJ, Jiang XJ. Nitrification activity and microbial community structure in purple soils with different pH. Acta Pedologica Sinica, 2018, 55(1): 194-202. (in Chinese) 曹彦强, 闫小娟, 罗红燕, 贾仲君, 蒋先军. 不同酸碱性紫色土的硝化活性及微生物群落组成. 土壤学报, 2018, 55(1): 194-202. |
