郑义培1,2, 吴丹1,2, 郑璞1,2


1. 江南大学工业生物技术教育部重点实验室, 江苏 无锡 214122;
2. 江南大学生物工程学院, 江苏 无锡 214122
收稿日期:2021-02-04;修回日期:2021-04-18;网络出版日期:2021-04-27
基金项目:国家轻工技术与工程一流学科自主课题(LITE2018-04)
*通信作者:郑璞. Tel: +86-510-85918156;Fax: +86-510-85918165; E-mail: zhengpu@jiangnan.edu.cn.
摘要:[目的] 建立适用于拟无枝酸菌(Amycolatopsis sp.)的CRISPR-Cas9基因编辑系统,敲除其编码香兰素脱氢酶基因(VDH),减少发酵副产物香草酸。[方法] 以VDH为靶标基因,将pKCcas9dO质粒上的tipA、j23119启动子分别替换为pRLE6质粒Kmr启动子、链霉菌中常用的强启动子permE*,同时将sgRNA替换为能识别靶基因香兰素脱氢酶的特异性sgRNA,获得质粒pKCKmCas9VDH。然后将其与靶基因的上下游同源臂连接,获得敲除质粒pLYZYP01。将pLYZYP01质粒电转进Amycolatopsis sp.感受态细胞,筛选获得VDH的敲除突变体菌株。[结果] 利用上述方法,成功获得VDH敲除菌株Amycolatopsis sp.ΔVDH。[结论] 建立了适用于拟无枝酸菌CCTCC M 2011265的基因敲除系统,成功敲除VDH基因,在添加12 g/L底物阿魏酸的情况下,香兰素产量达到9.19 g/L,摩尔转化率由88.6%提高到97.7%。
关键词:拟无枝酸菌CRISPR-Cas9基因敲除香兰素
A vanillin producing Amycolatopsis sp. with the VDH gene knockout by CRISPR-Cas9 technology
Yipei Zheng1,2, Dan Wu1,2, Pu Zheng1,2


1. Key Laboratory of Industrial Biotechnology of Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
2. School of Biological Engineering, Jiangnan University, Wuxi 214122, Jiangsu Province, China
Received: 4 February 2021; Revised: 18 April 2021; Published online: 27 April 2021
*Corresponding author: Zheng Pu. Tel: +86-510-85918156;Fax: +86-510-85918165; E-mail: zhengpu@jiangnan.edu.cn.
Foundation item: Supported by the National First-class Discipline Program of Light Industry Technology and Engineering (LITE2018-04)
Abstract: [Objective] In order to reduce the fermentation by-product vanillic acid, we established a CRISPR-Cas9 gene editing system for Amycolatopsis sp. to knock out the vanillin dehydrogenase gene (VDH). [Methods] Using VDH as the target gene, we replaced the promoters of tipA and j23119 on the pKCcas9dO plasmid with the Kmr promoter of the pRLE6 plasmid and the strong promoter permE* commonly used in Streptomyces, respectively. At the same time, we constructed the plasmid pKCKmCas9VDH by replacing the sgRNA with the specific sgRNA sequence that could recognize the target gene vanillin dehydrogenase, and linked the plasmid with the upstream and downstream homologous arms of the target gene to construct the knockout plasmid pLYZYP01. Then we transformed pLYZYP01 into Amycolatopsis sp. CCTCC M 2011265 and selected the VDH knockout mutant strains. [Results] We successfully obtained a knockout strain Amycolatopsis sp. ΔVDH with higher yield of vanillin. [Conclusion] We established a knockout system for Amycolatopsis sp. CCTCC M 2011265, and successfully knocked out the VDH gene. Under the condition of adding 12 g/L substrate ferulic acid, the yield of vanillin reached 9.19 g/L, and the conversion rate increased from 88.6% to 97.7%.
Keywords: AmycolatopsisCRISPR-Cas9gene knockoutvanillin
香兰素(vanillin)又名香草醛(3-甲氧基-4-羟基苯甲醛),是香草的主要香气成分。香兰素堪称“香料皇后”,被广泛应用于食品、饮料、香料和医药等领域中。由于香荚兰的种植受气候的影响,其产量有限,故从中所提取得到的天然香兰素价格极其昂贵。因此,产率高且成本低的化学合成法一直占据着香兰素生产的主导地位[1]。随着消费者对天然香料需求的不断增长,利用微生物转化天然底物生产香兰素受到重视[2]。目前,所用的天然前体物质有姜黄素、辣椒素、香草胺、香草醇、木质素、阿魏酸(ferulic acid)、藜芦醇、丁香酚、松柏醇、芳香族氨基酸等[3]。其中阿魏酸因其在自然界中分布广泛,且对微生物毒性作用小,而被认为是微生物生产香兰素首选的前体物质[4]。
1996年最先报道了两步法合成香兰素:第一步,先用黑曲霉(Aspergillus niger)将阿魏酸转化为香草酸(vanillic acid);第二步再用朱红密孔菌(Pycnoporus cinnabarinus)将香草酸还原为香兰素,但产香兰素浓度仅1.5 g/L左右[5-6]。其后发现Amycolatopsis和Streptomyces两个属的多个菌株能将底物阿魏酸转化生成浓度大于10 g/L的香兰素[7]。Amycolatopsis sp. ATCC 39116 (以前称为Seteptiices setonii ATCC 39116)是以阿魏酸为底物工业生产香兰素的模式菌株,德国Steinbüchel团队对该菌株进行阿魏酸代谢的蛋白质组学分析,认为底物阿魏酸在阿魏酰辅酶A合成酶、烯酰-CoA醛缩酶的依次作用下生成香兰素,在香兰素脱氢酶的作用下香兰素会进一步降解为香草酸,香草酸分别在香草酸脱羧酶和香草酸脱甲基酶作用下,降解为愈创木酚和原儿茶酸[8]。他们通过删除编码香兰素脱氢酶的VDH基因,使香兰素的降解降低90%以上,进一步对编码阿魏酰-CoA合成酶基因(FCS)和烯酰-CoA醛缩酶基因(ECH)进行增强表达,使工程菌株产香兰素浓度达到22.3 g/L,摩尔产率为94.9%,为目前已报道的最高水平[9]。
本实验室前期筛选到1株拟无枝酸菌CCTCC M 2011265,该菌株能够将30 g/L阿魏酸转化为10 g/L的香兰素[10];采用常压室温等离子体诱变(ARTP),菌株对前体摩尔产率提高到72.1%,但仍有香草酸等其他副产物积累[11]。本文通过在拟无枝酸菌中建立了一种基因编辑方法。利用CRISPR-Cas9系统特异性切割拟无枝酸菌的香兰素脱氢酶基因,以期获得提高香兰素产率、减少副产物的菌株。
1 材料和方法 1.1 菌株和质粒 本研究用到的菌株和质粒如表 1所示。
表 1. 文中所用主要菌株和质粒 Table 1. Main strains and plasmids used in this study
| Strains and plasmids | Characteristics | References or sources |
| Strains | ||
| Amycolatopsis sp. CCTCC M 2011265 | Wild type | Laboratory |
| Amycolatopsis sp. ?VDH | VDH deletion mutant of Amycolatopsis sp. CCTCC M 2011265 | This study |
| Escherichia coli JM109 | For transformation | Laboratory |
| Escherichia coli JM110 | For transformation | Laboratory |
| Plasmids | ||
| pET-28a-VDH | N-His, N-Thrombin, N-T7, VDH, C-His | This study |
| pKCcas9dO | acc(3)Ⅳ, pSG5, tipA-Scocas9, j23119, actⅡ-orf4 guide-RNA, homologous region flanking act-orf4 | Huang et al.[12] |
| pKCKmCas9VDH | acc(3)Ⅳ, pSG5, km-Scocas9, ermE*, VDH guide-RNA | This study |
| pLYZYP01 | acc(3)Ⅳ, pSG5, km-Scocas9, ermE*, VDH guide-RNA, homologous region flanking VDH | This study |
| pUC57-Kmr-promoter | the promoter region of the kanamycin resistance gene of pRLE6 | GENEWIZ |
| pUC57-permE* | ermE* promoter | GENEWIZ |
表选项
1.2 合成序列和引物 本文中所用到的合成序列和引物均由苏州金唯智生物科技有限公司合成,如表 2所示。
表 2. 本研究中所用合成序列和引物 Table 2. Oligonucleotides and primers used and constructed in this study
| Oligonucleotides | Sequences (5′→3′) |
| Kmr promoter | GCAAGCGAACCGGAATTGCCAGCTGGGGCGCCCTCTGGTAAGGTTGGGAAGCCCTGCAAAGTAAACTGGATGGCTTTCTTGCCGCCAAGGATCTGATGGCGCAGGGGATCAAGATCCCCGGGGATCTGATCAAGAGACAGGATGAGGATCGTTTCGC |
| ermE* promoter | CCAGCCCGACCCGAGCACGCGCCGGCACGCCTGGTCGATGTCGGACCGGAGTTCGAGGTACGCGGCTTGCAGGTCCAGGAAGGGGACGTCCATGCGAGTGTCCGTTCGAGTGGCGGCTTGCGCCCGATGCTAGTCGCGGTTGATCGGCGATCGCAGGTGCACGCGGTCGATCTTGACGGCTGGCGAGAGGTGCGGGGAGGATCTGACCGACGCGGTCCACACGTGGCACCGCGATGCTGTTGTGGGCACAATCGTGCCGGTTGGTAGGATC |
| gRNA-F | CGCCTCGCCCCAGGACCTGGCGTTTTAGAGCTAGAAATAGCAAGT |
| gRNA-R | CGACGGCCAGTGCCAAGCTTCTCAAAAAAAGCACCGACTC |
| Kmr-F | TGTCCATATGGCGAAACGATCCTCATCCTGTC |
| Kmr-R | GTCGGGCTGGGCAAGCGAACCGGAATTGC |
| ermE*-F | GTTCGCTTGCCCAGCCCGACCCGAG |
| ermE*-R | CCAGGTCCTGGGGCGAGGCGGATCCTACCAACCGGCACGA |
| VDH-f-F | GAGTCGGTGCTTTTTTTGAGAGCTCGTCCCACGACG |
| VDH-f-R | GGTCGTTGATGTCCACTTCTCGTCGTCGAG |
| VDH-r-F | AGAAGTGGACATCAACGACCAGACGGTCAAC |
| VDH-r-R | CGACGGCCAGTGCCAAGCTTTGCGAGCACCTGGAGAAGG |
| VDH-F | GTGAGCTTTCTCGACGACGAGA |
| VDH-R | TCAGAACGGGTAGCCGGC |
表选项
1.3 主要试剂、仪器及培养基 DNA聚合酶PrimerSTAR Max、DNA Marker购自宝日医生物技术(北京)有限公司。DNA胶回收试剂盒、细菌基因组DNA提取试剂盒购自生工生物工程(上海)股份有限公司。质粒小量制备试剂盒购自上海捷瑞生物工程有限公司。ClonExpress Ⅱ单步克隆试剂盒购自南京诺唯赞生物科技股份有限公司。引物合成和PCR产物测序委托金唯智生物科技有限公司完成。基因组测序委托上海伯豪生物技术有限公司完成。电转仪、PCR仪购自Eppendorf公司。
斜面培养基、种子培养基和发酵培养基参照王兴林的方法[13]。本氏培养基(g/L):葡萄糖1,牛肉浸粉0.1,蛋白胨0.2,酵母浸粉0.1,甘油1,加蒸馏水至1 L,pH 7。YEME培养基(g/L):蛋白胨5,酵母浸粉3,麦芽浸粉3,葡萄糖10,蔗糖220,加蒸馏水至1 L,pH 7。
1.4 香兰素脱氢酶的钓取 以Amycolatopsis sp. CCTCC M 2011265基因组DNA为模板,使用引物VDH-F/VDH-R进行PCR验证。
1.5 质粒构建
1.5.1 pKCKmCas9 VDH的构建: 质粒pKCcas9dO经Nde Ⅰ/Hind Ⅲ双酶切去除原质粒的tipA、j23119启动子、sgRNA和同源臂序列,得到cas9sco线性载体。然后以质粒pKCcas9dO为模板,用引物gRNA-F/gRNA-R扩增获得sgRNA重组片段,再用引物Kmr-F/Kmr-R和ermE*-F/ermE*-R分别以pUC57-Kmr-promoter和pUC57-permE*为模板扩增Kmr启动子和ermE*启动子片段。随后将sgRNA重组片段、ermE*启动子片段进行Overlap (重叠延伸PCR法)重组,获得的片段和Kmr启动子片段再次进行Overlap重组。接着,通过一步克隆,将获得的融合片段和cas9sco载体连接,通过热激转化至E. coli JM109感受态细胞中,30 ℃过夜培养,将长出的单菌落接种到含安普霉素(Apr)的液体LB中,30 ℃培养12 h,然后使用质粒小量制备试剂盒提取质粒,并用引物Kmr-F/gRNA-R进行PCR及测序验证,将验证正确的重组质粒命名为pKCKmCas9VDH (图 1)。
 |
| 图 1 重组质粒pKCKmCas9VDH的构建 Figure 1 Construction of the recombinant plasmid pKCKmCas9VDH. |
| 图选项 |
Overlap重组PCR程序:引物使用上游片段的F引物、下游片段的R引物,延伸时间t为2个片段连接后大片段所需的时间。模板为上下游片段混合物。其余与普通PCR程序相同。
1.5.2 敲除质粒的构建: 根据Amycolatopsis sp. ATCC 39116的VDH基因设计引物,以Amycolatopsis sp. CCTCC M 2011265基因组作为模板,分别用引物VDH-f-F/VDH-f-R和VDH-r-F/ VDH-r-R扩增获得VDH基因上游和下游同源臂。使用Overlap重组法将上下游片段连接,随后通过一步克隆将融合片段和经过Hind Ⅲ酶切处理的pKCKmCas9VDH质粒连接,用引物VDH-f-F/ VDH-r-R进行PCR及测序验证,将验证正确的质粒命名为pLYZYP01 (图 2)。
 |
| 图 2 重组质粒pLYZYP01的构建 Figure 2 Construction of the recombinant plasmid pLYZYP01. |
| 图选项 |
1.6 拟无枝酸菌的培养和转化 将拟无枝酸菌CCTCC M 2011265从甘油管接到YEME培养基,28 ℃、220 r/min培养48 h,然后以5%的接种量接到新鲜的YEME培养基中,28 ℃、220 r/min过夜培养12 h。菌体用冰浴的电击缓冲液(0.5 mol/L蔗糖,15%甘油)洗涤3次,然后用1–2 mL电击缓冲液重悬。
用热激法将质粒pLYZYP01转化进大肠杆菌JM110,使质粒去甲基化。电击转化时,在100 μL感受态细胞中加入1 μg质粒,冰上放置10 min,电击条件为1500 V、5 ms。电击后立刻加入预热的本氏培养基,28 ℃培养约4 h后涂布于添加了安普霉素的本氏培养基,28 ℃培养5–8 d。
1.7 突变菌株的筛选 挑选多株转化子,在含安普霉素的3 mL液态本氏培养基中,28 ℃培养2–3 d。转化子菌液在没有抗性的本氏培养基上划线,37 ℃过夜培养,挑单菌落对照涂布在无抗和含有安普霉素的本氏培养基,28 ℃培养2–3 d。无抗性板上正常生长而有抗性板上不生长的单菌落即为成功消除质粒的阳性菌株。提取基因组,使用引物VDH-f-F/VDH-r-R验证。
突变菌株筛选过程中使用的均为梯度PCR,反应程序为:98 ℃热启动,3 min;98 ℃变性,10 s;退火温度设置为50–62 ℃,15 s;72 ℃延伸,2 min。变性、退火、延伸3个步骤共30个循环。最后,72 ℃延伸10 min,4 ℃放置。
1.8 突变菌的发酵性能分析 将拟无枝酸菌突变菌株与原始菌株分别接种于种子培养基,置于30 ℃、180 r/min恒温摇床中培养36 h。以8%接种量接种生长细胞转化培养基,置于30 ℃、180 r/min恒温摇床中培养,20 h后添加终浓度为12 g/L的底物阿魏酸,同时培养温度上升至35 ℃。12 h后,每隔6 h取样检测。
1.9 分析方法 定量分析(HPLC)[14]:检测波长280 nm,柱温30 ℃,流动相为0.01%的醋酸溶液︰甲醇=7︰3,流速为1 mL/min,进样量20 μL。
摩尔转化率为生成产物的摩尔量除以添加底物的摩尔量。
2 结果和分析 2.1 香兰素脱氢酶的钓取与验证 以本实验室保存的拟无枝酸菌CCTCC M 2011265基因组DNA为模板,使用引物VDH-F/ VDH-R进行PCR。PCR产物条带(图 3)及测序结果表明,CCTCC M 2011265基因组中香兰素脱氢酶(1465 bp)与模式菌株Amycolatopsis sp. ATCC 39116中香兰素脱氢酶基因序列一致。
 |
| 图 3 香兰素脱氢酶基因的PCR产物 Figure 3 PCR product of VDH. M: marker; lane 1: PCR product of genomic DNA with primers VDH-F/VDH-R. |
| 图选项 |
2.2 重组质粒的构建 倪瑶等成功将温敏型质粒pKCcas9dO应用于东方拟无枝酸菌,同时实现基因的敲除与敲入[15],且其是链霉菌CRISPR系统中的常用质粒。而拟无枝酸菌与链霉菌的亲缘关系较近,因此采用质粒pKCcas9dO进行实验。德国Steinbüchel等在构建大肠杆菌与拟无枝酸菌的穿梭质粒时,发现Kmr启动子和ermE*启动子在拟无枝酸菌中表达强度较高[16]。因此将质粒pKCcas9dO中的启动子tipA和j23119更换为Kmr启动子和ermE*启动子。
使用相应引物进行PCR扩增,获得sgRNA序列、Kmr启动子、ermE*启动子片段和VDH基因上下游同源臂。通过Overlap重组法将sgRNA片段、ermE*启动子、Kmr启动子依次连接,融合片段扩增产物如图 4-A所示。然后使用重组克隆试剂盒,将融合片段与线性化的cas9sco载体连接,获得质粒pKCKmCas9VDH。使用引物Kmr-F/gRNA-R对重组质粒进行PCR扩增,如图 4-B所示,产物(565 bp)条带在500 bp左右,随机挑选2个PCR产物测序,结果显示3个片段已成功与质粒相连,重组质粒pKCKmCas9VDH构建成功。接着通过Overlap重组法将VDH基因上下游同源臂连接,融合片段扩增产物如图 4-C所示。然后通过重组克隆试剂盒,将VDH基因上下游同源臂与线性化的质粒pKCKmCas9VDH连接,获得敲除质粒pLYZYP01。使用引物VDH-f-F/VDH-r-R对重组质粒进行PCR扩增,如图 4-D所示,产物(1040 bp)条带在1000 bp左右,测序结果显示VDH上下游同源臂已成功与质粒相连,敲除质粒pLYZYP01构建成功。
 |
| 图 4 重组质粒pKCKmCas9VDH和pLYZYP01构建及验证图谱 Figure 4 The construction of recombinant plasmid pKCKmCas9VDH and pLYZYP01. M: marker; A: lane 1: PCR products of the fusion fragment of sgRNA, permE* and Kmr promoter; B: lane 1–10: PCR product of pKCKmCas9VDH with primers Kmr-F/gRNA-R; C: lane 1: PCR products of the homologous arm of the VDH; D: lane 1–9: PCR product of pLYZYP01 with primers VDH-f-F/VDH-r-R. |
| 图选项 |
2.3 突变菌株的获取 将敲除质粒pLYZYP01电转进拟无枝酸菌CCTCC M 2011265,涂布在含安普霉素的本氏培养基上,28 ℃培养5–8 d后,平板上长出许多清晰可见的单菌落,随机挑选10株进行质粒消除,最终有4株菌成功消除了质粒。将这4株菌接种到YEME培养基中,28 ℃培养24 h后,提取基因组。使用引物VDH-f-F/VDH-r-R,以质粒pKCcas9dO (空白对照)和基因组DNA为模板进行梯度PCR验证,结果4株菌中只有一株菌敲除成功,如图 5所示。其中图 5-A的1–12泳道为质粒pKCcas9dO的空白对照,13–24泳道为敲除成功的突变株;图 5-B为原始菌株。原始菌株基因组DNA扩增的是预期约2500 bp的DNA片段,VDH失活的突变菌株染色体DNA扩增的是约1000 bp的DNA片段(上下游同源臂之间缺失约1500 bp)。将条带大小约为1000 bp的PCR产物进行测序,结果显示上下游同源臂之间缺失了约1500 bp的VDH基因序列(图 5-C),成功获得突变菌株Amycolatopsis sp. ?VDH,整个过程香兰素脱氢酶敲除率为10%。
 |
| 图 5 突变株Amycolatopsis sp. ?VDH的PCR及测序验证 Figure 5 Identification of the mutants by PCR. M: marker. A: lane 1–12 (control): PCR product of pKCcas9dO with primers VDH-f-F/VDH-r-R; lane 13–24: PCR product of genomic DNA of Amycolatopsis sp. ?VDH with primers VDH-f-F/VDH-r-R. B: lane 1–12: PCR product of genomic DNA of WT with primers VDH-f-F/VDH-r-R. C: sequencing results of PCR products of mutant strains. |
| 图选项 |
2.4 突变株转化阿魏酸产香兰素评价 将突变菌和原始菌分别接入摇瓶发酵,培养24 h后添加终浓度为12 g/L的底物进行转化,转化24 h后间隔6 h取样进行HPLC检测,发酵液中香兰素、香草酸和阿魏酸含量的时间曲线见图 6。转化至72 h,阿魏酸基本消耗殆尽,原始菌株和突变菌株Amycolatopsis sp. ?VDH的香兰素产量分别为8.32 g/L和9.02 g/L;继续转化6 h,原始菌株和突变菌株Amycolatopsis sp. ?VDH的香兰素达到最高,分别为8.33 g/L和9.19 g/L,此时,副产物香草酸的浓度分别为170 mg/L和154 mg/L。随着发酵时间的继续增加,Amycolatopsis sp. ?VDH的香兰素浓度基本保持不变,而原始菌株则略有下降,原始菌株和突变菌株的香草酸浓度在转化72 h后迅速增加,84 h时分别增加到222 mg/L和200 mg/L。从图 6可以看出,原始菌株和突变菌株的产物积累、底物消耗趋势基本一致,但突变菌株的产物香兰素浓度提高了10.3%,副产物浓度下降了9.4%,摩尔转化率由88.6%提高到97.7%。
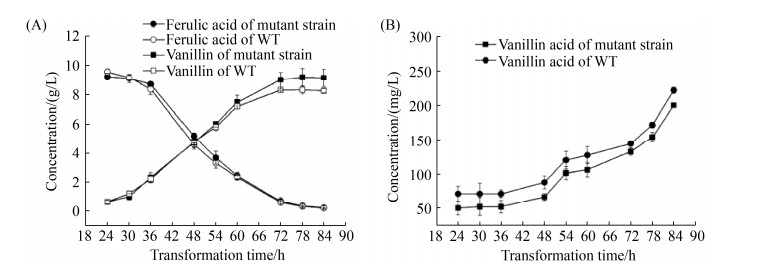 |
| 图 6 原始菌株和突变菌株对阿魏酸的转化 Figure 6 Biotransformation of ferulic acid by the WT and mutant strains. A: the concentration of ferulic acid and vanillin; B: the concentration of vanillic acid. The results are from three independent experiments, and the error bars represent the standard deviations. |
| 图选项 |
3 讨论 Huang等以pKCcas9dO质粒为模板,通过更换靶基因20 nt特异性引导序列和同源臂,实现链霉菌基因的定点突变和单基因、多基因、基因簇的删除[12]。本文运用同样策略,对应用于链霉菌CRISPR遗传操作系统的质粒pKCcas9dO进行了一些改造,将启动子tipA、j23119替换为在拟无枝酸菌中表达强度较高的Kmr启动子和ermE*启动子。同时,更换质粒pKCcas9dO上20 nt靶基因特异性引导序列和同源臂片段。所构建的质粒pLYZYP01成功识别靶序列,并实现了VDH基因敲除,获得目的菌株。突变菌株发酵副产物香草酸浓度明显下降,香兰素浓度得到提升。说明该基因编辑系统可以作为一种拟无枝酸菌CCTCC M 2011265的代谢改造工具。
至今国内外****对微生物法发酵生产香兰素作了大量研究。德国的Steinbüchel等曾试图用原生质体转化法,将大肠杆菌-拟无枝酸菌的穿梭质粒pRLE6导入拟无枝酸菌HR167未果;而用电转化法得到了30/μg DNA的转化子[17]。2013年,他们构建了拟无枝酸菌ACTT 39116的表达载体p6apra[16],随后,通过电转化法将该质粒转化导入拟无枝酸菌ACTT 39116,并通过同源重组法成功敲除香兰素脱氢酶基因[9]。国内周庆礼等通过对链霉菌L1936补加底物使之浓度达到13 g/L,最终产物香兰素浓度达到7.12 g/L,相应的摩尔转化率为69.9%[18]。许平等利用Streptomyces sp. V-1以阿魏酸为底物,借助吸附树脂的作用,经55 h的发酵最终获得19.2 g/L的香兰素,总摩尔得率为54.5%[19]。但国内对产香兰素链霉菌的基因操作鲜有研究报道。同源重组法存在效率较低、操作复杂、实验周期长的缺限。本文在产香兰素的拟无枝酸菌中成功构建了一种CRISPR/Cas系统介导的遗传操作系统,快速有效地敲除香兰素脱氢酶基因(VDH),使香兰素产量达到9.19 g/L,摩尔转化率提高到97.7%。为构建高产香兰素的拟无枝酸菌奠定了基础。
References
| [1] | Luo CH, Yu L, Wei M, Song XY, Chen YK, Chen Y. Research advance on synthesis of vanillin by microbial transformation method. Chemistry & Bioengineering, 2011, 28(12): 11-16, 23. (in Chinese) 罗诚浩, 喻林, 魏敏, 宋旭艳, 陈义坤, 陈勇. 微生物转化法生产香兰素的研究进展. 化学与生物工程, 2011, 28(12): 11-16, 23. DOI:10.3969/j.issn.1672-5425.2011.12.003 |
| [2] | Ma XK, Daugulis AJ. Effect of bioconversion conditions on vanillin production by Amycolatopsis sp(DIAN) ATCC 39116 through an analysis of competing by-product formation. Bioprocess and Biosystems Engineering, 2014, 37(5): 891-899. DOI:10.1007/s00449-013-1060-x |
| [3] | Schrader J, Etschmann MMW, Sell D, Hilmer JM, Rabenhorst J. Applied biocatalysis for the synthesis of natural flavour compounds-current industrial processes and future prospects. Biotechnology Letters, 2004, 26(6): 463-472. DOI:10.1023/B:BILE.0000019576.80594.0e |
| [4] | Priefert H, Rabenhorst J, Steinbüchel A. Biotechnological production of vanillin. Applied Microbiology and Biotechnology, 2001, 56(3/4): 296-314. |
| [5] | Lesage-Meessen L, Delattre M, Haon M, Thibault JF, Ceccaldi BC, Brunerie P, Asther M. A two-step bioconversion process for vanillin production from ferulic acid combining Aspergillus niger and Pycnoporus cinnabarinus. Journal of Biotechnology, 1996, 50(2/3): 107-113. |
| [6] | Sun ZH, Zheng P, Guo XF, Lin GY, Yin HH, Wang J, Bai YB. Method for the producing vanillic acid and vanillin from waste residue of rice bran oil by fermentation and biotransformation. US: 7462470B2, 2008. |
| [7] | Yang WW, Wu QL, Tang HZ, Xu P. Biosynthesis of natural vanillin-the queen of food ingredients. Microbiology China, 2013, 40(6): 1087-1095. (in Chinese) 杨文文, 吴秋林, 唐鸿志, 许平. "香料皇后"——天然香兰素生物合成的研究进展. 微生物学通报, 2013, 40(6): 1087-1095. |
| [8] | Meyer F, Netzer J, Meinert C, Voigt B, Riedel K, Steinbüchel A. A proteomic analysis of ferulic acid metabolism in Amycolatopsis sp(DIAN) ATCC 39116. Applied Microbiology and Biotechnology, 2018, 102(14): 6119-6142. DOI:10.1007/s00253-018-9061-y |
| [9] | Fleige C, Meyer F, Steinbüchel A. Metabolic engineering of the actinomycete Amycolatopsis sp(DIAN) strain ATCC 39116 towards enhanced production of natural vanillin. Applied and Environmental Microbiology, 2016, 82(11): 3410-3419. DOI:10.1128/AEM.00802-16 |
| [10] | Zheng P, Li H, Wang XL, Zhou HJ, Zhu SB. An Amycolatopsis sp. Strain and Methods of Using the Same for Vanillin Production. US: 9115377B2, 2017. |
| [11] | Liu Q, Zheng P. Induced mutation breeding of high vanillin-production Amycolatopsis sp(DIAN) by atmospheric and room temperature plasmas. Food and Fermentation Industries, 2013, 39(10): 40-44. (in Chinese) 刘琼, 郑璞. 常压室温等离子体诱变选育高产香兰素拟无枝酸菌(Amycolatopsis sp.). 食品与发酵工业, 2013, 39(10): 40-44. |
| [12] | Huang H, Zheng GS, Jiang WH, Hu HF, Lu YH. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces. Acta Biochimica Et Biophysica Sinica, 2015, 47(4): 231-243. DOI:10.1093/abbs/gmv007 |
| [13] | Wang XL, Zheng P, Liu Q. Biotransformation of ferulic acid to vanillin by Amycolatopsis sp... Industrial Microbiology, 2013, 43(5): 34-38. (in Chinese) 王兴林, 郑璞, 刘琼. Amycolatopsis sp.转化阿魏酸生产香草醛. 工业微生物, 2013, 43(5): 34-38. |
| [14] | Li YH, Wang R, Sun ZH. Simultaneous determination of vanillin and isoeugenol in bioconversion broth by RP-HPLC. Journal of Henan University of Technology: Natural Science Edition, 2007, 28(5): 50-52. (in Chinese) 李永红, 王瑞, 孙志浩. RP-HPLC方法同时测定生物转化液中的香草醛和异丁香酚含量. 河南工业大学学报: 自然科学版, 2007, 28(5): 50-52. |
| [15] | 倪瑶, 魏维, 钱秀萍, 夏兴, 戈梅. 一种基因编辑方法在东方拟无枝酸菌中的应用. CN: 109609537A, 2019. |
| [16] | Fleige C, Steinbüchel A. Construction of expression vectors for metabolic engineering of the vanillin-producing actinomycete Amycolatopsis sp(DIAN) ATCC 39116. Applied Microbiology and Biotechnology, 2014, 98(14): 6387-6395. DOI:10.1007/s00253-014-5724-5 |
| [17] | Priefert H, Achterholt S, Steinbüchel A. Transformation of the Pseudonocardiaceae Amycolatopsis sp(DIAN) strain HR167 is highly dependent on the physiological state of the cells. Applied Microbiology and Biotechnology, 2002, 58(4): 454-460. |
| [18] | Zhou QL, Huang YF, Han YS, Wang CL. Bioconversion for vanillin from ferulic acid. Food and Fermentation Industries, 2004, 30(3): 18-20. (in Chinese) 周庆礼, 黄艳凤, 韩英素, 王昌禄. 微生物转化法生产香兰素. 食品与发酵工业, 2004, 30(3): 18-20. |
| [19] | Hua DL, Ma CQ, Song LF, Lin S, Zhang ZB, Deng ZX, Xu P. Enhanced vanillin production from ferulic acid using adsorbent resin. Applied Microbiology and Biotechnology, 2007, 74(4): 783-790. |
