李伟勋, 芦晶, 张书文, 逄晓阳

 , 吕加平
, 吕加平

中国农业科学院农产品加工研究所, 北京 100193
收稿日期:2020-11-25;修回日期:2021-03-06;网络出版日期:2021-03-22
基金项目:国家自然科学基金面上项目(31871833);国家重点研发计划课题(2017YFC1600903);中国农业科学院科技创新工程项目(CAAS-ASTIP-2020-IFST-04);中央级公益性科研院所基本科研业务费专项(S2020JBKY-18)
*通信作者:逄晓阳, Tel: +86-10-62815542, E-mail: pangxiaoyang@163.com;
吕加平, Tel: +86-10-62819421, E-mail: lvjiapingcaas@126.com.
摘要:乳酸菌是一类重要的食品工业微生物,目前对其功能基因鉴定和挖掘优良功能基因主要依赖于传统的基因同源重组技术,该技术尽管有较高的可靠性,但是存在操作繁琐、效率低下等不足,严重制约了乳酸菌优良菌株的遗传选育。CRISPR/Cas基因编辑技术极大提升了对多物种基因组的编辑效率,这为乳酸菌功能基因的快速鉴定及遗传改良提供了可能,但是现有的CRISPR/Cas基因编辑技术在乳酸菌的应用还存在诸多限制。本文综述了CRISPR/Cas基因编辑技术在乳酸菌基因组上的应用现状及亟待解决的问题,并展望了乳酸菌基因组编辑技术的未来发展趋势,为乳酸菌功能基因鉴定及遗传改良提供参考。
关键词:CRISPR/Cas基因组编辑乳酸菌遗传改良
Perspectives and applications of CRISPR/Cas-mediated genome editing in lactic acid bacteria
Weixun Li, Jing Lu, Shuwen Zhang, Xiaoyang Pang

 , Jiaping Lv
, Jiaping Lv

Institute of Food Science and Technology, Chinese Academy of Agricultural Sciences, Beijing 100193, China
Received: 25 November 2020; Revised: 6 March 2021; Published online: 22 March 2021
*Corresponding author: Xiaoyang Pang, Tel: +86-10-62815542, E-mail: pangxiaoyang@163.com;
Jiaping Lv, Tel: +86-10-62819421, E-mail: lvjiapingcaas@126.com.
Foundation item: Supported by the National Natural Science Foundation of China (31871833), by the National Key R & D Program of China (2017YFC1600903), by the Science and Technology Innovation Project of Chinese Academy of Agricultural Sciences (CAAS-ASTIP-2020-IFST-04) and by the Special Funds for Fundamental Scientific Research Operation of Central Public Welfare Scientific Research Institutes (S2020JBKY-18)
Abstract: Lactic acid bacteria are an important class of food industry microorganisms. At present, the identification and excavation of functional genes of lactic acid bacteria mainly rely on traditional gene homologous recombination technology. Although this technology has high reliability, there are complicated operations and low efficiency, which severely restrict the genetic selection of lactic acid bacteria. CRISPR/Cas gene editing technology has greatly improved the editing efficiency of multi-species genomes, which provides opportunities for rapid functional gene identification and genetic improvement of lactic acid bacteria. However, the application of existing CRISPR/Cas gene editing technology in lactic acid bacteria still has many limitations. This article summarizes the application status of CRISPR/Cas gene editing technology in the genome of lactic acid bacteria and the urgent problems to be solved, and prospects the future development of the lactic acid bacteria genome editing technology, providing references for the identification and genetic improvement of lactic acid bacteria.
Keywords: CRISPR/Casgenome editinglactic acid bacteriagenetic improvement
基因组编辑技术,是指对基因组进行靶向修饰的过程,包括对基因的敲除、插入或单碱基的删除、插入和替换等操作。对生命科学研究来说,简便、高效的基因操作技术,可使研究人员能够快速分析特定基因和调控元件的功能,同时多重基因编辑将大规模地构建核酸互作,核酸-蛋白质互作网络研究成为可能[1]。传统的细菌基因组编辑技术多是基于细菌内源DNA同源重组机制,同源重组介导的靶向DNA修饰精度较高,但重组效率非常低(10-6-10-9 cells)[2],同时存在极性突变效应[3]等缺陷。为了解决这些问题,目前已经开发了多种细菌基因组编辑技术应用于不同细菌的基因组编辑,如基于外源噬菌体重组酶系统的Red/RecET同源重组技术[4-6],基于Cre-LoxP位点特异性的重组酶技术[7],以及工程化的内切核酸酶介导的基因组编辑技术[8-10]。
近几年快速发展的CRISPR (clustered regularly interspaced short palindromic repeats)/Cas (CRISPR associated)基因组编辑技术实现了对多种生物进行快速基因编辑的目标[1]。CRISPR/Cas技术,由具有特异性的小RNA引导核酸内切酶Cas (或多个Cas蛋白的复合体)至基因组靶位点,介导靶基因座产生DNA双链断裂(double strand break,DSB)[11]。细菌主要通过同源重组(homologous recombination repair,HR) DNA修复机制对DSB进行修复[12],少数细菌可利用非同源末端连接(non-homologous end joining,NHEJ) DNA修复机制来进行修复[13]。通过修复导致基因组产生基因缺失或者引入新的基因,从而达到基因编辑的目的。
乳酸菌是一类通过发酵碳水化合物产生乳酸的革兰氏阳性兼性厌氧菌的总称,乳酸菌被分为43个属,超过373种[14]。最近的研究表明,乳酸菌对恶劣环境具有良好的抗逆性,预示了乳酸菌作为细胞工厂进行生物精制和生产生物活性物质的潜力[15]。有些乳酸菌已被证明具有很好的益生菌功能,如抑制病原菌的繁殖,增强益生菌的活性,缓解炎症反应[16],它们被广泛用于治疗抗生素相关性腹泻、肠易激综合征和炎症性肠病[17]。此外,乳杆菌广泛地参与宿主的发育、健康维持和疾病发生,甚至是行为[18-19]。由此揭示了乳酸菌在促进人类健康方面的重要作用。
在目前高通量基因测序技术飞速发展并产生海量基因组数据的背景下,高效的基因组编辑技术将有助于我们快速高效鉴定未知基因功能、挖掘优良功能基因、遗传改良工业用乳酸菌株,比如获得某些噬菌体抗性菌株,提升发酵产品风味,获得抗逆性更强的乳酸菌株。乳酸菌的高效基因操作技术将可以强化菌株已有益生特性,消除菌株的不良基因,或根据需要增加新的益生功能,这将拓展它们在食品开发、疾病治疗和营养保健方面的应用。本文综述了CRISPR/Cas基因编辑技术的原理,对该技术在乳酸菌基因组上的应用现状进行了总结,分析了目前存在的问题,并展望了乳酸菌基因组编辑技术的未来发展趋势,为乳酸菌基因组编辑研究者提供参考。
1 乳酸菌中的CRISPR/Cas系统 CRISPR/Cas系统是细菌的获得性免疫系统,是在研究细菌和古细菌多样性的过程中被发现的。目前在40%以上的细菌和90%以上古细菌基因组上均发现了CRISPR/Cas系统,CRISPR/Cas基因座通常由一组Cas基因和具有菌株特异性的CRISPR阵列基因组成,CRISPR阵列基因由高度保守的直接重复序列(direct repeat sequence,DR)和序列可变的间隔区基因(spacer)组成,DR序列被Spacer序列依次隔开[20]。虽然CRISPR/Cas系统目前被分为两大类、六个小类和几十个亚型[21],还有其他假定类型,但整个系统的工作原理基本类似,总体上可分为3个阶段,分别是适应阶段、表达阶段[22-23]和效应阶段[24]。适应阶段是指CRISPR的高度可变间隔区的获得,其实质是噬菌体或质粒等外源遗传元件在一系列Cas蛋白的辅助下,被整合到宿主菌基因组的过程;表达阶段是指CRISPR基因座转录及转录后加工的过程,CRISPR基因座首先被转录成前体crRNA (Pre-crispr RNA),在单一或多种Cas蛋白和必要的辅助元件辅助下,pre-crRNA被加工成成熟离散的crRNA (crispr RNA)单元;效应阶段是指CRISPR/Cas系统介导的,对外源遗传元件进行靶向破坏的过程。
在1262个乳杆菌基因组中,60%乳杆菌基因组可检测到CRISPR阵列基因,约40%的乳杆菌具有完整的CRISPR/Cas系统。在具有完整CRISPR/Cas系统的乳杆菌中,多数乳杆菌仅有一个CRISPR/Cas系统被表征,少数乳杆菌菌株包含多个CRISPR/Cas系统,发酵乳杆菌(Lactobacillus fermentum,L. fermentumstrains) NB-22和MTCC 8711同时含有I、II、III型CRISPR/Cas系统,不同CRISPR/Cas系统可能在crRNA合成中独立发挥作用[25]。乳杆菌中完整的CRISPR/Cas系统的分布分析表明,II型系统丰度最高,其次是I型系统,III型系统丰度最低。I-E亚型和II-A亚型分别为乳杆菌中表征的I型和II型系统中最具优势的亚型,未检测到IV、V和VI型系统;但一些乳杆菌似乎含有假定的V-U亚型CRISPR/Cas系统[26]。乳杆菌属中还存在一类特殊的CRISPR/Cas系统,称之为孤立(orphan) CRISPR/Cas系统,这类系统仅具有CRISPR序列或Cas基因[27]。例如克鲁氏乳杆菌(Lactobacillus crustorum,L. crustorum)、巴氏乳杆菌(Lactobacillus backii,L. backii)和嗜酸乳杆菌(Lactobacillus acidophilus,L. acidophilus),仅含有CRISPR序列但不含Cas基因;副柯林氏乳杆菌(Lactobacillus paracollinoides,L. paracollinoides)中,仅含有Cas基因而不含CRISPR序列[26],表明它们可能通过其他机制来实现抗病毒感染。本实验室对副干酪乳杆菌(Lactobacillus paracasei,L. paracasei)基因组上的CRISPR/Cas系统进行了较为全面的表征。对L. paracasei中完整的CRISPR/Cas系统的分布分析显示,CRISPR/Cas系统在L. paracasei中表征率约为43%,被表征的系统大多为II-A亚型。对L. paracasei的CRISPR序列分析得知,同一亚型的重复序列长度以及转录成RNA后形成的二级机构非常保守。同一亚型不同菌株的间隔序列数目及碱基排列顺序非常多样,反映了不同L. paracasei菌株在不同环境下“免疫接种”的多样性。原间隔序列(protospacer)同源性分析表明,间隔区靶向的噬菌体基因多为噬菌体结构蛋白编码基因,包括衣壳蛋白基因、头蛋白基因、尾蛋白基因等,L. paracasei中的CRISPR/Cas系统通过靶向干扰这些基因,增强其抗病毒能力[28-29]。
2 建立乳酸菌CRISPR/Cas基因组编辑系统 2.1 CRISPR/Cas系统在细菌基因组编辑中的工作原理 CRISPR/Cas系统高度序列特异性的DNA切割特性已被利用和重新编程来断裂基因,形成的DNA断裂能在体内触发修复,从而实现对染色体的遗传修饰[30]。因其载体构建相对简单,成本低廉,适用范围广,现已成为最受欢迎的基因组工程方法。工程化的CRISPR/Cas系统包含2个组件:sgRNA (single guide RNA)和Cas效应蛋白(Cas effector protein,CEP)。I类CRISPR系统CEP常常以复合体的形式存在,多个Cas蛋白组合在一起进而对靶标DNA进行切割,干扰机制较为复杂[23],因此多利用外源II类CRISPR/Cas系统对细菌基因组进行编辑。sgRNA是一种短的合成RNA,由CEP结合所需的scaffold序列和自定的20 nt的guide RNA (gRNA)组成[31],scaffold序列可以通过自身互补配对形成类似repeat-tracrRNA二聚体的结构,对CEP起到支撑作用。在编辑过程中,CEP首先结合sgRNA形成具有一定空间结构的核酸蛋白复合体,称之为预靶向态(pre-target state),该复合体识别靶基因座附近的PAM (protospacer adjacent motif)序列,同时guide RNA与靶基因互补配对,CEP的核酸内切酶活性被激活,然后在靶基因处介导DSB,如图 1所示[32]。通过改变guide RNA的序列可以容易地改变CEP靶向的位置。
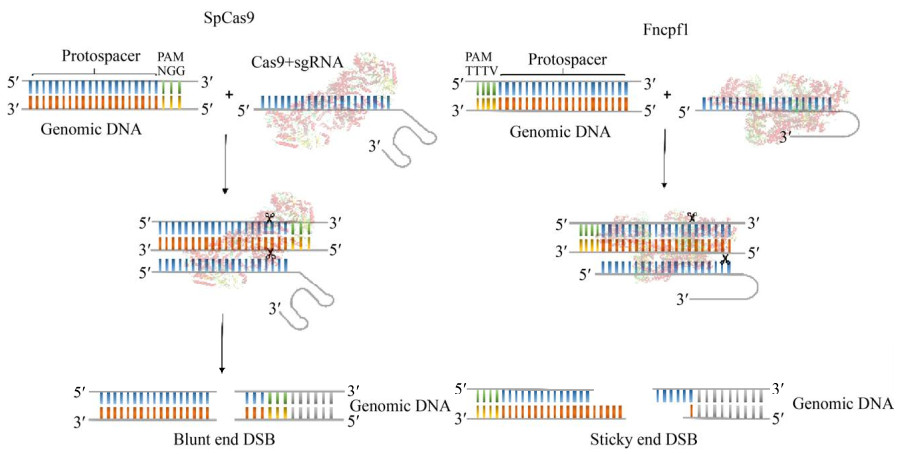 |
| 图 1 II类CRISPR/Cas系统基因编辑原理 Figure 1 Gene editing principle of Class 2 CRISPR/Cas system. CEP and sgRNA firstly form a nucleic acid-protein complex. When CEP recognizes the PAM sequence on the target sequence, and the target sequence is paired with guide RNA, its endonuclease activity is activated, and then CEP performs targeted cleavage to form DSB. Different types of CEP cleavage produce different DSBs. |
| 图选项 |
虽然CRISPR/Cas基因组编辑技术已经在多物种中得到了应用,但遗憾的是,其在乳酸菌中的应用相对较少,研究人员难以对乳酸菌进行有效的遗传操作,特别是乳杆菌,目前仅有10余种乳杆菌可进行遗传操作。最初多数工作都基于乳酸菌本身的CRISPR系统对乳酸菌进行精确分型,这是一种能够对菌株进行指纹识别的方法,能够区分同种不同菌株间的差异[29]。近年来,研究者们正不断尝试利用CRISPR/Cas基因编辑技术对乳酸菌基因组进行修饰。
2.2 利用外源CRISPR/Cas系统进行乳酸菌基因组编辑 利用外源CRISPR/Cas系统进行基因编辑的编辑质粒主要有以下关键组件:靶向靶标基因的sgRNA、介导DSB的CEP和同源修复模板(图 2)。利用该编辑质粒实现基因敲入或是敲除由同源修复模板决定。CEP的表达由组成型启动子P23驱动,该启动子是在乳酸菌中普遍使用的具有高转录活性的广泛宿主启动子。通过调节相关的启动子来调节sgRNA的转录强度,可以提高编辑效率。启动子可以从乳酸菌必需基因的启动子中进行选择,如rRNA的启动子。值得注意的是异源Cas9可能具有密码子偏好性,在待编辑菌株中翻译水平较低,因此有必要进行密码子优化。目前应用较为广泛的异源CEP有Cas9、Cas12a,它们都能介导DSB,Cas9介导的双链DNA断裂为平末端,而Cas12a介导的DNA断裂为粘性末端[33]。如果某种乳酸菌对CEP介导的DSB较为敏感,可以尝试利用Cas9切口酶(Cas9nickase,Cas9n),切口酶是基于CRISPR/Cas9基因编辑系统的补充,可用于缺乏DSB修复乳酸菌的基因组编辑。切口酶介导的DNA单链断裂(single strand break,SSB)可由同源序列指导修复(homology- directed repair,HDR)[34]。设计靶向质粒后,选择处于对数生长期的宿主菌,利用合适的转化方式将编辑质粒转化进宿主菌进而发挥作用。合适的转化方式可能比质粒设计本身更具挑战性,目前多使用电转化方式将编辑载体递送至乳酸菌中。
 |
| 图 2 编辑/辅助质粒构建[35] Figure 2 The construct of editing/assisted plasmid[35]. For host bacteria with low homologous recombination repair capacity, additional assisted plasmids need to be constructed to assist the host in homologous recombination repair. Depending on the type of exogenous recombinase selected, the donor sequence is also different. For RecE/T, ssDNA donors are preferred. The elements in the plasmid are as follows. P: promoter; T: terminator; Marker: screening marker, sometimes resistance genes; minicrRNA: in the middle of the mini-CRISPR sequence is the guide RNA, which is similar to spacers, and the flanks are repeat sequences. Its gene structure is similar to the crRNA sequence unit, supporting CEP and targeting. |
| 图选项 |
2.3 利用内源CRISPR/Cas系统进行基因组编辑 本身具有CRISPR/Cas系统的乳酸菌给我们对其进行基因组编辑提供了另外一种思路,可以充分利用乳酸菌已有的内源CRISPR/Cas相关组件,通过设计合适的gRNA进行自体靶向基因编辑。利用内源性CRISPR/Cas系统进行基因组编辑有非常明显的优势:由于不需要表达外源CEP,一个质粒可以携带多个gRNA,很容易地靶向多个基因,质粒可以以合适的大小顺利转化进细菌[36];在尚未对CRISPR/Cas系统的机制完整阐明前,利用内源性CRISPR/Cas系统进行自体靶向的过程更为接近天然机制,相较于外源系统,内源系统的编辑效率可能更高[37]。
首先,为了充分利用内源性CRISPR/Cas系统进行乳酸菌基因组编辑,必须充分表征该种乳酸菌的CRISPR/Cas系统,包括检测CRISPR/Cas系统的活性以及鉴定该系统所识别的PAM序列。对目标乳酸菌全基因组进行分析可获知该菌是否有CRISPR系统,以及是何种亚型的系统。根据预测出的CRISPR系统,进行小RNA测序或设计特异性的探针,进行Northern blotting实验均可以检测CRISPR/Cas系统的表达水平。
以II-A型系统为例,利用CRISPRTarget网页工具进行PAM的识别(http://crispr.otago.ac.nz/CRISPRTarget/crispr_analysis.html)[38]。利用Weblogo对每个位点的碱基偏好性进行可视化(https://weblogo.berkeley.edu/logo.cgi)[39]。本实验室对多株干酪乳杆菌的分析结果显示,干酪乳杆菌源Cas9识别的PAM序列为TGAAA[40]。得到生物信息分析结果后,还需在其基础上进行PAM序列的实验验证至最终确定准确的PAM序列[41]。
利用内源性CRISPR/Cas系统进行乳酸菌基因组编辑时,编辑质粒应当具有以下几个重要元件:(1) mini-CRISPR序列,mini-CRISPR序列中间部分为靶向靶标序列的guide RNA,侧翼均为repeat序列,其基因结构类似crRNA序列单元,起到支撑CEP和靶向作用;(2) 标记基因;(3)同源修复模板。设计靶向质粒后,选择处于对数生长期的宿主菌,利用合适的转化方式如电转化或化学转化将靶向质粒转化进宿主菌进而发挥作用。利用细菌内源性CRISPR系统已成功靶向多种菌的基因组,降低了多重基因编辑的门槛,部分菌株的编辑效率甚至能达到100%[36-37, 42-43]。
3 CRISPR/Cas系统在乳酸菌基因组编辑中的应用及改进 3.1 CRISPR/Cas系统在乳酸菌基因组编辑中的应用 目前CRISPR/Cas基因编辑技术在乳酸菌基因组中的应用集中在点突变、基因敲除、基因敲入等,实现大片段缺失(> 1 kb)、等位基因替换仍然具有挑战性。Oh等开发并优化了CRISPR/Cas9技术,对L. reuri ATCC PTA 6475中3个靶标基因lacL、srtA和sdp6进行基因敲除,其突变率可以达到90%-100%,首次实现了CRISPR/Cas9技术在乳酸菌中的应用,并证实了CRISPR/SpCas9结合ssDNA重组是一种切实可行的高效编辑方法[44]。利用相似的策略,在植物乳杆菌(Lactiplantibacillus plantarum,L. plantarum WCFS1)中,Leenay等成功地将点突变D480Y整合到rpoB基因中[45]。上述两种基于CRISPR/Cas的基因工程方法实质上是利用CRISPR/Cas系统作为一类非抗生素依赖的筛选标记。其原理是利用外源RecE/T介导的ssDNA重组的编辑系统将寡核苷酸整合到复制叉中,以实现多重编辑和诱变,而CRISPR/Cas9靶向未修饰的染色体DNA以消除野生型细胞,从而促进突变体筛选。利用CRISPR/Cas9基因编辑系统和乳酸菌内源性同源重组修复系统,研究者在实验室环境下敲除了Bififidobacterium animals subsp. lactis基因组上的四环素抗性基因tet-W,对解决日益严重的益生菌耐药性问题提出了新的方法[46]。通过向Bififidobacterium longum subsp. longum基因组上敲入新型寡糖水解酶基因β-Gal,使其能够利用宿主难以消化吸收的复杂多糖如β-低聚半乳糖和果寡糖,赋予其定殖宿主肠道的竞争性优势[47]。
除了外源Cas9工具,乳酸菌内源性CRISPR/Cas系统的编辑工具也具有其独特的优势。Barrangou等利用卷曲乳杆菌(Lactobacillus crispatus,L. crispatus)中内源I-E型CRIPSR/Cas系统,实现了对L. crispatus自身基因组的编辑,敲除了p-gtf基因(643 bp,100%效率)和原噬菌体DNA包装基因Nu1 (308 bp,20%效率),插入了绿色荧光白蛋白报告基因(730 bp,23%效率)。在p-gtf基因内实现了包括终止密码子插入(36%效率)和单核苷酸替换(19%效率)等在内的多种突变,这是首次成功利用内源性CRISPR/Cas系统在乳杆菌中实现基因编辑[43]。内源性基因编辑方法还能够在一些难以遗传操作的其他乳酸菌中得到复用,Selle等利用嗜热链球菌(Streptococcus thermophilus,S. thermophilus)内源II-A型CRISPR/Cas9系统靶向删除自身基因组上非必需基因的基因岛,结果表明内源性的自体靶向CRISPR/Cas系统可以指导细菌基因组在种群水平上的显著进化,影响基因组的稳态和重塑[48]。同样地,基于S. thermophilus的内源性CRISPR/Cas系统,Alexander等实现了对CRISPR序列的重编程,使之获得了对特定噬菌体的免疫[49]。在不提供修复模板的前提下,由于大部分细菌没有非同源末端连接的修复系统,CEP造成的DSB对细菌来说是致死性的。研究人员利用致病菌内源CRISPR/Cas系统同时靶向自体基因组上多个位点而不提供修复模板,靶标序列包括多个必需基因及非必需基因,成功从乳酸菌群落中特异性地清除了致病菌如沙门氏菌及大肠杆菌[46, 50]。
利用CRISPR/Cas技术也可以用来研究细菌中转录调控因子对于基因功能的转录调控。SpCas9的RuvC和HNH核酸酶结构域都可以通过点突变(SpCas9中的D10A和H840A)转变为无切割活性但有靶标结合活性的dCas9 (dead Cas9),dCas9基于sgRNA靶向序列与靶DNA结合[31]。Xiong等通过sgRNA将dCas9募集到启动子区或者开放阅读框(open reading frame,ORF)区,从而阻止RNA转录起始或延伸来抑制L. lactis中的基因转录[51]。利用该系统实现了对upp、sod和LLNZ_07335三个基因的转录抑制,抑制效率可达30%-90%。Ale? Berlec等也通过此策略实现了L. lactis中的基因转录抑制,与Xiong等的研究不同,该研究仅用了一个工作质粒pNZCRISPRi就实现了转录抑制,但由于sgRNA也受到诱导型启动子PnisA的调控,其表达水平处于较低水平,对转录抑制效率有一定影响[52]。
3.2 CRISPR/Cas技术在乳酸菌基因组编辑中的改进 成功的基因编辑包含两个层面,一是CEP高效精准地引起靶位点断裂,二是利用合适的修复机制使得靶位点DNA断裂能够得到期望中的修复。研究者或通过改变CEP本身的特性,使之靶向断裂的效率更高,脱靶率更低,或通过开发辅助DSB修复系统,使得目的突变能高效整合至细菌基因组。
在Cas9核酸酶的固定表达水平下,sgRNA的转录量与Cas9-sgRNA复合物在染色体上的切割效率呈正相关[53]。因此,换用强启动子增加sgRNA的转录量能提高编辑效率。基于乳杆菌核糖体RNA启动子的变体P11是一个广宿主强启动子,其转录特性已被充分表征,转录强度和种间转录活性也已经在植物乳杆菌、清酒乳杆菌和布氏乳杆菌在内的数种乳杆菌[54-55]中得到验证。研究表明,相比于启动子PLp_0537,换用强启动子P11能够增加sgRNA的转录量,从而提高了L. plantarum WCFS1编辑效率[56]。
避开DSB也是一个可替代的乳酸菌基因组编辑策略。Song等使用CRISPR/Cas9D10A在干酪乳杆菌中建立了一种快速且准确的基因编辑方法。该研究利用Cas9D10A对干酪乳杆菌基因组进行编辑。Cas9D10A是Cas9的突变体,称为Cas9切口酶(nickase),其RuvC核酸酶结构域活性丧失,仅保留一个HNH核酸酶结构域,能够靶向靶标位置形成单链DNA断裂(single strand break,SSB)。利用该方法研究人员敲除了4个非必需基因,插入了1个绿色荧光蛋白报告基因,基因敲除和敲入的效率为26%-62%。在实验过程中,研究人员观测到混合菌落的产生,即某一菌落同时含有突变株和野生株,因此这种基因编辑方法虽然简单易用,但编辑效率可能低于预期。一个可能的原因是,与Cas9介导的染色体双链断裂相比,切口酶诱导的SSB更容易借助高保真的单链修复机制修复,从而导致更多的野生型细胞逃逸[53]。
乳杆菌中的内源同源重组修复能力有很大不同,有些乳杆菌内源同源重组系统活性较弱,即使提供了修复模板或利用Cas9切口酶进行编辑,也无法观察到成功编辑的菌落。Yang等借助RecE/T介导的重组,通过CRISPR/Cas9与辅助修复途径偶联来改善基因组编辑系统[56]。但限制此方法应用的主要因素是,该RecE/T辅助重组系统不能在多宿主中适用,因此需开发与宿主相匹配的噬菌体来获得RecE/T活性组分。RecE/T辅助重组酶的表达需要诱导型启动子驱动(图 2),持续性表达RecE/T可能对后续转化进来的编辑质粒在乳酸菌中复制产生影响,一个可能的机制是编辑质粒通过滚环复制来进行扩增,在此过程中会产生ssDNA中间体,RecT可能结合ssDNA并干扰复制[44]。
利用CRISPR/Cas9进行基因编辑时的特异性部分取决于gRNA与基因组非靶标位点结合的特异性。理想情况下,gRNA与靶标DNA严格互补配对结合,而与基因组中的其他位置均无结合。实际上,给定的gRNA有可能与基因组非靶标区结合,这些非靶标区称为脱靶位点,在实验设计gRNA时需要考虑脱靶位点对实验的影响。研究发现,gRNA中一段长度为10 bp的seed region区决定了Cas9靶向基因组的特异性,如图 3所示,而gRNA中远PAM端序列对碱基错配的容忍度很高,越高的碱基错配容忍度代表该sgRNA越易脱靶[32, 57]。因此,研究者通过截短gRNA远PAM端序列2-3 nt,增强了Cas9的靶向特异性[58]。
 |
| 图 3 SgRNA二级结构[32] Figure 3 The secondary structure of SgRNA[32]. Normal SgRNA has three stem loops, which combine with Cas9 to form a nucleic acid-protein complex, ready to pair with the target sequence. A 10 nt seed region sequence determines the specificity of editing. |
| 图选项 |
2016年,研究人员使用定点突变技术开发了高保真Cas9核酸酶eSpCas9。eSpCas9相较于野生型SpCas9,其包含了4个氨基酸突变位点,分别为N497A、R661A、Q695A和Q926A。eSpCas9可削弱HNH/RuvC凹槽与非靶标DNA链之间的相互作用,从而防止链分离和在脱靶位点切割[59]。2018年,研究者通过基于酵母的筛选系统发现了进化的Cas9 (evoCas9),其在Rec3结构域中具有4个突变。与eSpCas9相比,evoCas9的脱靶活性更低[60]。
在靶向范围方面,通过PACE (phage-assisted continuous evolution)蛋白进化技术,利用噬菌体介导的普遍性转导,将突变质粒上的序列整合到受体菌内的受体质粒上随机突变、定向筛选。研究者们开发出一种xCas9,xCas9是一种具有高特异性和PAM兼容性的SpCas9变体,xCas9可针对广泛的PAM序列,例如NG、GAA和GAT,极大地加大了Cas9可以靶向的范围,同时还可以显著降低脱靶风险,SpCas9-NG是xCas9的其中一种识别NG PAM的变体,相对于其他Cas9核酸酶,其体外切割活性增加[61]。目前开发的各种Cas9变体所识别的PAM如表 1所示[62]。
表 1. Cas9变体PAM识别范围 Table 1. PAM recognition scope of Cas9 variants
| Cas9 variants | PAM scope | Example |
| SpCas9 | NGG | AGG/GGG/CGG/TGG |
| xCas9 | NG | AG/GG/CG/TG |
| SpCas9-NG | NG+NANG | NAAG/NAGG/NACG/NATG |
| SpCas9-VRER | NGG+NGCG | AGCG/GGCG/CGCG/TGCG |
| SpCas9-VQR/VPQR | NGR+NGCG | NGA/NGG |
| SpCas9-NRRH | NGG+NRRH | NAAA/NAAT/NAAC |
| SpCas9-NRTH | NGG+NRTH | NATA/NATT/NATC |
| SpCas9-NRCH | NGG+NRCH | NACA/NACT/NACC |
| SaCas9-KKH | NNNRRT | NNNAAT/NNNGGT/NNNAGT/NNNGAT |
| Degenerate base: R: A/G, H: A/T/C. | ||
表选项
4 CRISPR/Cas技术在乳酸菌基因组编辑中的应用展望 基于CRISPR/Cas的基因编辑技术已经在原核生物中得到广泛应用,包括单基因[63]、多基因编辑[10],单碱基编辑[64]和转录调控[65],但在乳酸菌中的应用相对较少。数十年的功能基因组学研究已经部分揭示了食品发酵和宿主-益生菌相互作用的遗传基础。利用不断发展的CRISRP/Cas基因编辑工具箱对乳酸菌进行基因修饰,以增强其发酵性能、定殖宿主肠道以调节宿主的免疫反应将具有广泛的应用前景。得益于某些乳酸菌对人体肠道的独特适应性和安全性,有研究表明几种乳杆菌已被用作粘膜递送疫苗和生物抗菌剂的底盘生物[15, 66]。
基于CRISPR/Cas基因编辑技术改善乳酸菌发酵性能,通过使用内源CRISPR/Cas系统将单核苷酸突变C266T引入S. thermophilus的eps基因簇中,可以改善乳酸菌的发酵性能,增加胞外多糖(EPS)的合成。EPS水平升高可以改善乳制品特别是酸奶和奶酪的流变性能,提高其感官性能[67]。利用CRISPR/Cas基因编辑技术更换L. gaseri中bshA基因的启动子,增加bshA的转录,从而增加L. gaser的胆汁盐水解能力,促进其宿主肠道定殖能力[68]。利用DSB对大部分无NHEJ修复机制细菌都是致命的这一特点,可以制成基于CRIPSR/Cas系统的抗菌剂。在实验室环境中,研究者们甚至成功去除了不同菌株混合培养物中的特定菌株[50]。很多细菌的抗药性获得是基于质粒间的基因水平转移[69],基于相同策略,也可以从细菌群落中特异性去除耐药质粒,但如何在非实验室环境如工业发酵中原位递送载体可能是个问题。利用溶原噬菌体递送载体可能是备选策略之一,工程改造噬菌体以编码Cas9核酸酶、tracrRNA和一系列靶向质粒或靶向基因组的crRNA,crRNA被设计为靶向细菌染色体或自身质粒的序列,将噬菌体DNA注入细菌的混合种群中,可以去除目标菌株或质粒,而不会影响其余种群[70]。
在健康促进方面,CRISPR/Cas基因编辑技术介导的重组乳酸菌菌株也具有非凡的潜力。嗜酸乳杆菌(Lactobacillus acidophilus,L. acidophilus) NCFM是一种食用了将近40年的安全乳酸菌菌株,由于其对胆汁和酸的耐受性,可以通过胃肠道存活,因此已被证明是将抗原递送至肠粘膜的主要候选菌株。可以通过CRISPR/Cas基因编辑技术进一步增强其治疗潜力,将编码致病菌的表面抗原基因插入基因组改变其细胞表面组成,利用该策略已成功地在L. acidophilus NCFM中表达致病菌抗原如肉毒梭菌和炭疽芽孢杆菌的表面抗原,使得这些重组菌株成为开发针对肉毒梭菌和炭疽芽孢杆菌的粘膜疫苗的有希望候选者[71]。
Addgene (https://www.addgene.org/crispr/bacteria/)上提供了数目繁多用于细菌基因组编辑的工具,不难发现,在大多数菌株中,利用外源CRISPR/Cas系统进行单基因编辑已不是难题,但多基因编辑仍然面临挑战。在其他细菌中已有多基因编辑的相关应用,但在乳酸菌中则罕有报道,主要是乳酸菌特别是乳杆菌属的同源重组能力较弱,很难同时完成多处靶向基因编辑,因为乳酸菌在多处产生DSB后,极易死亡,因此要保证多个sgRNA不能同时靶向基因组或者借助足够活力的外源重组修复系统对DSB进行精准高效的修复。即使绕过多处同时产生DSB,如何保证转录顺序靠后的sgRNA有足够的表达量是一个非常重要的问题,但也有研究人员通过采用sgRNA表达不受排列顺序影响的Cas12a解决了这一问题[72]。
利用乳酸菌内源性同源重组系统进行乳酸菌基因组编辑,到现在利用CRISPR/Cas技术高效、快速对乳酸菌基因组进行修饰,技术手段在不断地升级,我们正在向编辑效率越来越高、脱靶率越来越低的目标靠近,但必须看到CRISPR/Cas技术的局限性,如SpCas9对部分乳酸菌的细胞毒性[35],很多CRISPR/Cas系统在模式菌株中表现良好,在非模式菌株中表现得却不尽如人意。乳杆菌是乳酸菌家族中最大的属,其成员既相似又在遗传和进化背景上表现出非凡的多样性[26],很难找到一类通用基因编辑工具适用于所有的乳杆菌属,因此开发多元化的基因编辑工具,编辑策略彼此互补,并基于此建立适合不同种的乳酸菌基因编辑系统仍然任重道远。当然,基因编辑工具的不断探索也意味着我们对细菌生物学系统的理解不断加深。可以预见的是,当未来多基因编辑技术和合成生物学相结合时,利用安全的乳酸菌作为底盘生物按照需要去设计优良的发酵性能,出色的宿主定殖能力以及定制健康和治疗功能,将为整个社会生产生活方式产生巨大的影响[73-74]。
References
| [1] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [2] | Capecchi MR. Altering the genome by homologous recombination. Science, 1989, 244(4910): 1288-1292. DOI:10.1126/science.2660260 |
| [3] | Burby PE, Simmons LA. CRISPR/Cas9 editing of the Bacillus subtilis genome. Bio-protocol, 2017, 7(8): e2272. |
| [4] | Zhang YM, Buchholz F, Muyrers JPP, Stewart AF. A new logic for DNA engineering using recombination in Escherichia coli. Nature Genetics, 1998, 20(2): 123-128. DOI:10.1038/2417 |
| [5] | Muyrers JPP, Zhang YM, Testa G, Stewart AF. Rapid modification of bacterial artificial chromosomes by et-recombination. Nucleic Acids Research, 1999, 27(6): 1555-1557. DOI:10.1093/nar/27.6.1555 |
| [6] | Zhang YM, Muyrers JPP, Rientjes J, Stewart F. Phage annealing proteins promote oligonucleotide-directed mutagenesis in Escherichia coli and mouse ES cells. BMC Molecular Biology, 2003, 4(1): 1. DOI:10.1186/1471-2199-4-1 |
| [7] | Lambert JM, Bongers RS, Kjeerebezem M. Cre-lox-based system for multiple gene deletions and selectable-marker removal in Lactobacillus plantarum. Applied and environmental microbiology, 2007, 73(4): 1126-1135. DOI:10.1128/AEM.01473-06 |
| [8] | Niu QH, Zheng HY, Zhang L, Qin FJ, Facemire L, Zhang G, Cao F, Zhang KQ, Huang XW, Yang JW, He L, Liu CJ. Knockout of the adp gene related with colonization in Bacillus nematocida B16 using customized transcription activator-like effectors nucleases. Microbial Biotechnology, 2015, 8(4): 681-692. DOI:10.1111/1751-7915.12282 |
| [9] | Jiang Y, Qian F, Yang J, Liu Y, Dong F, Xu C, Sun B, Chen B, Xu X, Li Y, Wang R, Yang S. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nature communications, 2017, 8: 15179. DOI:10.1038/ncomms15179 |
| [10] | Jiang Y, Chen B, Duan C, Sun B, Yang J, Yang S. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied and environmental microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [11] | Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 2013, 8(11): 2281-2308. DOI:10.1038/nprot.2013.143 |
| [12] | Cromie GA. Phylogenetic ubiquity and shuffling of the bacterial RecBCD and AddAB recombination complexes. Journal of bacteriology, 2009, 191(16): 5076-5084. DOI:10.1128/JB.00254-09 |
| [13] | Shuman S, Glickman MS. Bacterial DNA repair by non-homologous end joining. Nature reviews Microbiology, 2007, 5(11): 852-861. DOI:10.1038/nrmicro1768 |
| [14] | Song YQ, Sun ZH, Zhang HP. Research progress on the microevolution of lactic acid bacteria. Acta Microbiologica Sinica, 2015, 55(11): 1371-1377. (in Chinese) 宋宇琴, 孙志宏, 张和平. 乳酸菌微进化的研究进展. 微生物学报, 2015, 55(11): 1371-1377. |
| [15] | Van Pijkeren JP, Barrangou R. Genome editing of food-grade Lactobacilli to develop therapeutic probiotics. Microbiology spectrum, 2017, 5(5). |
| [16] | Rieder R, Wisniewski PJ, Alderman BL, Campbell SC. Microbes and mental health: A review. Brain, Behavior, and Immunity, 2017, 66: 9-17. DOI:10.1016/j.bbi.2017.01.016 |
| [17] | Tremaroli V, B?ckhed F. Functional interactions between the gut microbiota and host metabolism. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [18] | Bravo D, Landete JM. Genetic engineering as a powerful tool to improve probiotic strains. Biotechnology and Genetic Engineering Reviews, 2017, 33(2): 173-189. DOI:10.1080/02648725.2017.1408257 |
| [19] | Zitvogel L, Daillère R, Roberti MP, Routy B, Kroemer G. Anticancer effects of the microbiome and its products. Nature reviews Microbiology, 2017, 15(8): 465-478. DOI:10.1038/nrmicro.2017.44 |
| [20] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709. DOI:10.1126/science.1138140 |
| [21] | Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [22] | Makarova KS, Zhang F, Koonin EV. Snapshot: Class 2 CRISPR-Cas systems. Cell, 2017, 168(1-2): 328-328. e321. DOI:10.1016/j.cell.2016.12.038 |
| [23] | Makarova KS, Zhang F, Koonin EV. Snapshot: Class 1 CRISPR-Cas systems. Cell, 2017, 168(5): 946-946. e1. DOI:10.1016/j.cell.2017.02.018 |
| [24] | Shmakov S, Abudayyeh OO, Makarova KS, Wolf YI, Gootenberg JS, Semenova E, Minakhin L, Joung J, Konermann S, Severinov K, Zhang F, Koonin EV. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell, 2015, 60(3): 385-397. DOI:10.1016/j.molcel.2015.10.008 |
| [25] | Carte J, Christopher RT, Smith JT, Olson S, Barrangou R, Moineau S, Glover CV 3rd, Graveley BR, Terns RM, Terns MP. The three major types of CRISPR-Cas systems function independently in CRISPR RNA biogenesis in Streptococcus thermophilus. Molecular microbiology, 2014, 93(1): 98-112. DOI:10.1111/mmi.12644 |
| [26] | Crawley AB, Henriksen ED, Stout E, Brandt K, Barrangou R. Characterizing the activity of abundant, diverse and active CRISPR-Cas systems in Lactobacilli. Scientific Reports, 2018, 8(1): 11544. DOI:10.1038/s41598-018-29746-3 |
| [27] | Zhang Q, Ye Y. Not all predicted CRISPR-Cas systems are equal: Isolated Cas genes and classes of CRISPR like elements. BMC Bioinformatics, 2017, 18(1): 92-92. DOI:10.1186/s12859-017-1512-4 |
| [28] | 杨兰. Lactobacillus paracasei CRISPR-Cas系统多样性分析及PAM验证. 中国农业科学院硕士学位论文, 2020. |
| [29] | Yang L, Li W, Ujiroghene OJ, Yang Y, Lu J, Zhang S, Pang X, Lv J. Occurrence and diversity of CRISPR loci in Lactobacillus casei group. Frontiers in Microbiology, 2020, 11(624). |
| [30] | Jiang W, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [31] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [32] | Jiang F, Zhou K, Ma L, Gressel S, Doudna JA. A Cas9-guide RNA complex preorganized for target DNA recognition. Science, 2015, 348(6242): 1477. DOI:10.1126/science.aab1452 |
| [33] | Bin Moon S, Lee JM, Kang JG, Lee N-E, Ha D-I, Kim DY, Kim SH, Yoo K, Kim D, Ko J-H, Kim Y-S. Highly efficient genome editing by CRISPR-Cpf1 using CRISPR RNA with a uridinylate-rich 3'-overhang. Nature communications, 2018, 9(1): 3651. DOI:10.1038/s41467-018-06129-w |
| [34] | Davis L, Maizels N. Homology-directed repair of DNA nicks via pathways distinct from canonical double-strand break repair. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(10): E924-932. DOI:10.1073/pnas.1400236111 |
| [35] | Li Q, Wu MX, Guo QH, Shao YR, Yang JJ, Jiang Y, Yang S. Advances in bacterial genome editing technology. Life Science, 2019, 31(5): 473-492. (in Chinese) 李琦, 武美贤, 郭清华, 邵悠然, 杨俊杰, 蒋宇, 杨晟. 细菌基因组编辑技术进展. 生命科学, 2019, 31(5): 473-492. |
| [36] | Cheng F, Gong L, Zhao D, Yang H, Zhou J, Li M, Xiang H. Harnessing the native type i-b CRISPR-Cas for genome editing in a polyploid archaeon. Journal of genetics and genomics=Yi chuan xue bao, 2017, 44(11): 541-548. DOI:10.1016/j.jgg.2017.09.010 |
| [37] | Zhang J, Zong W, Hong W, Zhang Z-T, Wang Y. Exploiting endogenous CRISPR-Cas system for multiplex genome editing in Clostridium tyrobutyricum and engineer the strain for high-level butanol production. Metabolic Engineering, 2018, 47: 49-59. DOI:10.1016/j.ymben.2018.03.007 |
| [38] | Biswas A, Gagnon JN, Brouns SJ, Fineran PC, Brown CM. CRISPRtarget: Bioinformatic prediction and analysis of crRNA targets. RNA Biology, 2013, 10(5): 817-827. DOI:10.4161/rna.24046 |
| [39] | Crooks GE, Hon G, Chandonia JM, Brenner SE. Weblogo: A sequence logo generator. Genome Research, 2004, 14(6): 1188-1190. DOI:10.1101/gr.849004 |
| [40] | Yang L, Yang Y, Li WX, JOY O, Pang XY, Lu JP. CRISPR locus analysis of Lactobacillus casei. Chinese Agricultural Sciences, 2019, 52(3): 521-529. (in Chinese) 杨兰, 杨洋, 李伟勋, JOY O, 逄晓阳, 吕加平. 干酪乳杆菌CRISPR基因座分析. 中国农业科学, 2019, 52(3): 521-529. |
| [41] | Xie H, Tang L, He X, Liu X, Zhou C, Liu J, Ge X, Li J, Liu C, Zhao J, Qu J, Song Z, Gu F. SaCas9 requires 5'-nngrrt-3' pam for sufficient cleavage and possesses higher cleavage activity than SpCas9 or FnCpf1 in human cells. Biotechnology journal, 2018, 13(4): e1700561. DOI:10.1002/biot.201700561 |
| [42] | Xu Z, Li M, Li Y, Cao H, Miao L, Xu Z, Higuchi Y, Yamasaki S, Nishino K, Woo PCY, Xiang H, Yan A. Native CRISPR-Cas-mediated genome editing enables dissecting and sensitizing clinical multidrug-resistant P. aeruginosa. Cell reports, 2019, 29(6): 1707-1717.e1703. DOI:10.1016/j.celrep.2019.10.006 |
| [43] | Hidalgo-Cantabrana C, Goh YJ, Pan M, Sanozky-Dawes R, Barrangou R. Genome editing using the endogenous type I CRISPR-Cas system in Lactobacillus crispatus. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(32): 15774. DOI:10.1073/pnas.1905421116 |
| [44] | Oh JH, Van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri. Nucleic Acids Research, 2014, 42(17): e131. DOI:10.1093/nar/gku623 |
| [45] | Leenay RT, Vento JM, Shah M, Martino ME, Leulier F, Beisel CL. Genome editing with CRISPR-Cas9 in Lactobacillus plantarum revealed that editing outcomes can vary across strains and between methods. Biotechnology Journal, 2019, 14(3): e1700583. DOI:10.1002/biot.201700583 |
| [46] | Hidalgo-Cantabrana C, O'Flaherty S, Barrangou R. CRISPR-based engineering of next-generation lactic acid bacteria. Current Opinion in Microbiology, 2017, 37: 79-87. DOI:10.1016/j.mib.2017.05.015 |
| [47] | Goh YJ, Klaenhammer TR. Genetic mechanisms of prebiotic oligosaccharide metabolism in probiotic microbes. Annual Review of Food Science and Technology, 2015, 6(1): 137-156. DOI:10.1146/annurev-food-022814-015706 |
| [48] | Selle K, Klaenhammer TR, Barrangou R. CRISPR-based screening of genomic island excision events in bacteria. Proceedings of the National Academy of Sciences, 2015, 112(26): 8076. DOI:10.1073/pnas.1508525112 |
| [49] | Hynes AP, Labrie SJ, Moineau S. Programming native CRISPR arrays for the generation of targeted immunity. mBio, 2016, 7(3): e00202-00216. |
| [50] | Gomaa AA, Klumpe HE, Luo ML, Selle K, Barrangou R, Beisel CL. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems. mBio, 2014, 5(1): e00928-00913. |
| [51] | Xiong ZQ, Wei YY, Kong LH, Song X, Yi HX, Ai LZ. Short communication: An inducible CRISPR/dCas9 gene repression system in Lactococcus lactis. Journal of Dairy Science, 2020, 103(1): 161-165. DOI:10.3168/jds.2019-17346 |
| [52] | Berlec A, Skrlec K, Kocjan J, Olenic M, Strukelj B. Single plasmid systems for inducible dual protein expression and for CRISPR-Cas9/CRISPRi gene regulation in lactic acid bacterium Lactococcus lactis. Scientific Reports, 2018, 8(1): 1009. DOI:10.1038/s41598-018-19402-1 |
| [53] | Song X, Huang H, Xiong Z, Ai L, Yang S. CRISPR-Cas9(d10A) nickase-assisted genome editing in Lactobacillus casei. Applied and environmental microbiology, 2017, 83(22): e01259-01217. |
| [54] | Rud I, Jensen PR, Naterstad K, Axelsson L. A synthetic promoter library for constitutive gene expression in Lactobacillus plantarum. Microbiology, 2006, 152(4): 1011-1019. DOI:10.1099/mic.0.28599-0 |
| [55] | Tauer C, Heinl S, Egger E, Heiss S, Grabherr R. Tuning constitutive recombinant gene expression in Lactobacillus plantarum. Microbial cell factories, 2014, 13: 150. DOI:10.1186/s12934-014-0150-z |
| [56] | Huang H, Song X, Yang S. Development of a RecE/T-assisted CRISPR-Cas9 toolbox for lactobacillus. Biotechnology journal, 2019, 14(7): e1800690. DOI:10.1002/biot.201800690 |
| [57] | Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li Y, Fine EJ, Wu X, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology, 2013, 31(9): 827-832. DOI:10.1038/nbt.2647 |
| [58] | Fu Y, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using truncated guide rnas. Nature Biotechnology, 2014, 32(3): 279-284. DOI:10.1038/nbt.2808 |
| [59] | Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016, 529(7587): 490-495. DOI:10.1038/nature16526 |
| [60] | Casini A, Olivieri M, Petris G, Montagna C, Reginato G, Maule G, Lorenzin F, Prandi D, Romanel A, Demichelis F, Inga A, Cereseto A. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology, 2018, 36(3): 265-271. DOI:10.1038/nbt.4066 |
| [61] | Hu JH, Miller SM, Geurts MH, Tang W, Chen L, Sun N, Zeina CM, Gao X, Rees HA, Lin Z, Liu DR. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 2018, 556(7699): 57-63. DOI:10.1038/nature26155 |
| [62] | Miller SM, Wang T, Randolph PB, Arbab M, Shen MW, Huang TP, Matuszek Z, Newby GA, Rees HA, Liu DR. Continuous evolution of SpCas9 variants compatible with non-G PAMs. Nature Biotechnology, 2020, 38(4): 471-481. DOI:10.1038/s41587-020-0412-8 |
| [63] | Li Y, Lin Z, Huang C, Zhang Y, Wang Z, Tang YJ, Chen T, Zhao X. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing. Metabolic Engineering, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006 |
| [64] | Chen W, Zhang Y, Zhang Y, Pi Y, Gu T, Song L, Wang Y, Ji Q. CRISPR/Cas9-based genome editing in Pseudomonas aeruginosa and cytidine deaminase-mediated base editing in pseudomonas species. iScience, 2018, 6: 222-231. DOI:10.1016/j.isci.2018.07.024 |
| [65] | Bikard D, Jiang W, Samai P, Hochschild A, Zhang F, Marraffini LA. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. Nucleic Acids Research, 2013, 41(15): 7429-7437. DOI:10.1093/nar/gkt520 |
| [66] | LeCureux JS, Dean GA. Lactobacillus mucosal vaccine vectors: Immune responses against bacterial and viral antigens. mSphere, 2018, 3(3): e00061-18. |
| [67] | Hidalgo-Cantabrana C, Sánchez B, álvarez-Martín P, López P, Martínez-álvarez N, Delley M, Martí M, Varela E, Suárez A, Antolín M, Guarner F, Berger B, Ruas-Madiedo P, Margolles A. A single mutation in the gene responsible for the mucoid phenotype of Bifidobacterium animalis subsp. Lactis confers surface and functional characteristics. Applied and environmental microbiology, 2015, 81(23): 7960-7968. DOI:10.1128/AEM.02095-15 |
| [68] | Ruiz L, Margolles A, Sánchez B. Bile resistance mechanisms in Lactobacillus and Bifidobacterium. Frontiers in Microbiology, 2013, 4: 396. |
| [69] | Zhang WJ, Wang XM, Dai L, Hua X, Dong Z, Schwarz S, Liu S. Novel conjugative plasmid from Escherichia coli of swine origin that coharbors the multiresistance gene cfr and the extended-spectrum-β-lactamase gene blaCTX-M-14b. Antimicrobial Agents & Chemotherapy, 2015, 59(2): 1337-1340. |
| [70] | Beisel CL, Gomaa AA, Barrangou R. A CRISPR design for next-generation antimicrobials. Genome Biology, 2014, 15(11): 516. DOI:10.1186/s13059-014-0516-x |
| [71] | O'Flaherty S, Klaenhammer TR. Multivalent chromosomal expression of the clostridium botulinum serotype a neurotoxin heavy-chain antigen and the bacillus anthracis protective antigen in lactobacillus acidophilus. Applied and Environmental Microbiology, 2016, 82(20): 6091-6101. DOI:10.1128/AEM.01533-16 |
| [72] | Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [73] | Du RX, Guo MZ, Xie ZX, He XY, Huang KL, Xu WT. Application and prospects of synthetic biology in improving intestinal health. Biotechnology Bulletin, 2018, 34(1): 49-59. (in Chinese) 杜若曦, 郭明璋, 谢子鑫, 贺晓云, 黄昆仑, 许文涛. 合成生物学在改善肠道健康状态中的应用与展望. 生物技术通报, 2018, 34(1): 49-59. |
| [74] | Liu D, Du J, Zhao GR, Yuan YJ. Application of synthetic biology in medicine and energy. CIESC Journal, 2011, 62(9): 2391-2397. (in Chinese) 刘夺, 杜瑾, 赵广荣, 元英进. 合成生物学在医药及能源领域的应用. 化工学报, 2011, 62(9): 2391-2397. DOI:10.3969/j.issn.0438-1157.2011.09.002 |
