李毅, 张利环, 冯焱, 李宏, 张锋, 白芸, 丁云云, 康美琳, 王雨竹, 雷嘉轩, 申少斐


山西农业大学生命科学学院, 山西 太谷 030801
收稿日期:2021-03-24;修回日期:2021-05-17;网络出版日期:2021-06-03
基金项目:山西省优秀博士来晋工作奖励项目(SXYBKY2019025);山西农业大学科技创新基金项目(2020BQ39);国家自然科学基金(31700749)
*通信作者:申少斐, Tel: +86-354-6286908;E-mail: shenshaofei@nwafu.edu.cn.
摘要:[目的] 南极洲不同地区环境极端多样,且受人类活动影响不一。本研究旨在探究南极不同纬度地区土壤抗生素抗性基因(ARGs)的分布特征与迁移机制。[方法] 下载南极不同纬度地区及加拿大阿尔伯特地区养殖场附近土壤宏基因组数据集,利用MetaWRAP进行组装,使用CARD、PlasFlow和ICEberg数据库对ARGs与可移动遗传元件(MGEs)进行注释。[结果] 在南极不同纬度地区土壤中,优势菌门为变形菌门、放线菌门、拟杆菌门和厚壁菌门。共注释出25类406种ARGs,以多重耐药类、四环素类及氨基糖苷类抗生素抗性基因为主。NMDS分析结果表明,南极不同纬度地区与养殖场附近土壤中ARGs的分布特征显著不同(ANISOM,P=0.001)。南极高纬度地区ARGs占总基因数的比例为0.28%,显著低于低纬度地区(1.93%,P < 0.01)。不同抗生素类型的ARGs呈现不同的区域分布模式,其中硝基咪唑类、氨基糖苷类、糖肽类与大环内酯类ARGs主要分布在南极高纬度地区,四环素类与磺胺类ARGs主要分布在南极低纬度地区(P < 0.05)。南极土壤中ARGs的迁移研究表明,质粒携带的ARGs占检测到的ARGs的17%。同时,共发现163个整合与接合元件(ICEs)可携带多抗耐药类、肽类和四环素类等14类ARGs。这些携带ARGs的ICEs主要分布于α-、β-与γ-变形菌纲中。[结论] 南极高纬度与南极低纬度地区土壤中ARGs的分布存在差异性,质粒与ICEs共同介导ARGs的迁移。本研究为进一步了解抗生素时代之前的原始抗性组提供数据基础。
关键词:抗生素抗性基因宏基因组学微生物多样性可移动遗传元件南极土壤
Deciphering distribution and migration of soil antibiotic resistance genes in different latitude regions of Antarctica by metagenomics
Yi Li, Lihuan Zhang, Yan Feng, Hong Li, Feng Zhang, Yun Bai, Yunyun Ding, Meilin Kang, Yuzhu Wang, Jiaxuan Lei, Shaofei Shen


College of Life Sciences, Shanxi Agricultural University, Taigu 030801, Shanxi Province, China
Received: 24 March 2021; Revised: 17 May 2021; Published online: 3 June 2021
*Corresponding author: Shaofei Shen, Tel: +86-354-6286908;E-mail: shenshaofei@nwafu.edu.cn.
Foundation item: Supported by the Excellent Talents Come to Shanxi to Reward Scientific Research Projects (SXYBKY2019025), by the Scientific and Technological Innovation Programs of Shanxi Agricultural University (2020BQ39) and by the National Natural Science Foundation of China (31700749)
Abstract: [Objective] Antarctic environments are extremely diverse and affected by human activities in varying degrees. The aim of this study is to explore distribution and migration of soil antibiotic resistant genes (ARGs) in different latitude regions of Antarctica. [Methods] We selected soil metagenomic data from Antarctic high (HLG) and low latitude regions (LLG) and surrounding regions of feedlot. Metagenomic reads was assembled using software MetaWRAP. ARGs, plasmids and integrative and conjugative elements (ICEs) were annotated using CARD (The Comprehensive Antibiotic Resistance Database), PlasFlow and ICEberg, respectively. [Results] In the Antarctic soils, Proteobacteria, Actinomycetes, Bacteroidetes and Firmicutes were the dominant phyla. Belonging to 25 ARG types, 406 ARGs were found, among which multidrug, tetracycline and aminoglycoside resistance genes dominated. The NMDS analysis showed that the characteristics of ARGs in Antarctic soils were significantly different from those in agricultural soils nearby feedlot (ANOSIM, P=0.001). The proportion of ARGs in all ORFs in the Antarctic HLG was 0.28%, lower than that in the LLG (1.93%, P < 0.01). Different ARGs present different latitude-region resistance patterns. Therein, nitroimidazole, aminoglycoside, glycopeptide and macrolide resistance genes were mainly distributed in the HLG, while tetracycline and sulfonamide resistance genes were mainly distributed in the LLG (P < 0.05). The migration analysis of ARGs in Antarctic soils showed that 17% of the detected ARGs were carried by plasmids. In addition, 163 ICEs carrying ARGs, belonging to 14 ARG types (e.g., multidrug, peptide and tetracycline resistance genes), were found. These ICEs carrying ARGs were extensively distributed among α-, β- and γ-proteobacteria. [Conclusion] The differences in microbial communities and ARGs between the Antarctic high and low latitude areas occurred, plasmids and ICEs facilitated migration of nature ARGs. This study provided a certain data basis for understanding the natural antibiotic resistance from the pre-antibiotic era.
Keywords: antibiotic resistance genesmetagenomicsmicrobial diversitymobile genetic elementsAntarctic soils
作为一种新型“环境污染物”[1],抗生素抗性基因(ARGs)引起的微生物耐药性(AMR)是21世纪人类面临最紧迫的公共卫生问题之一,严重阻碍现代医学的发展[2]。早于抗生素时代的微生物群落对抗生素非常敏感。然而,人类在医疗、畜牧业和水产养殖中对抗生素的滥用,为抗生素抗性菌的富集和进化创造了理想环境[3–4]。微生物对抗生素或其他抗菌药物的敏感性越来越低,各种“超级病菌”相继产生,人类有必要引起重视并果断采取应对措施。
研究结果表明抗生素的生物合成已发展了数百万年[5]。在抗生素被人类使用之前,自然界中的抗生素广泛用于微生物之间的生存竞争,同时长期对环境微生物进行驯化[6]。细菌对抗生素的耐药性也是一种自然而古老的现象[7],环境中ARGs可以通过质粒等可移动遗传元件(MGEs)或噬菌体进行传播与扩散[8]。然而,抗生素的滥用加剧了抗生素耐药性的传播,使抗生素抗性细菌(ARBs)在环境中不断累积。目前,人们对微生物ARGs的研究多聚焦于抗生素污染严重的城市污水处理厂[9]、养殖场[10]、医院[11]等环境中,却很少关注受人为活动影响较小的偏远原始地区,比如南极洲。
南极洲位于地球最南端,环境极端、复杂多样,具有多种生态位环境(例如南极湖泊、海水、海冰、冻土和干谷等)[12]。因远离人类大陆,土著微生物群落受人为影响较小。之前的研究报道了南极低纬度地区如南极半岛海水[13]及研究站附近[14]抗生素抗性组,但低纬度地区受人类活动影响巨大,例如可引入非本地微生物和相关遗传因素[15],导致低纬度地区原始环境中ARGs的多样性发生变化。而南极高纬度地区(如联合冰川)因其环境温度极低,辐射强度极高,人为活动较少,因此该地区土壤可代表一些不受人类活动干扰的生态位[16]。目前,南极不同纬度地区原始土壤中ARGs的分布特征与迁移机制尚不完全清楚。本研究利用宏基因组学方法对南极联合冰川、麦克默多干旱谷和南极半岛地区土壤中微生物抗生素抗性基因的多样性进行分析,探究ARGs的分布和迁移机制,为全面了解抗生素时代之前的原始抗性组提供数据基础。
1 材料和方法 1.1 样本采集 本研究从NCBI网站上的Sequence Read Archive (SRA)数据库下载16个宏基因组数据集。这些土壤样品分别来自南极联合冰川、麦克默多干旱谷、南极半岛及加拿大阿尔伯特地区养殖场附近农田。具体的采样地点信息、样品编号和核酸序列号见表 1。根据纬度差异将南极土壤样品分为两组:高纬度区域组(HLG)和低纬度区域组(LLG)。高纬度区域组由联合冰川与麦克默多干旱谷样品组成,该地区环境极端,几乎不受人类活动影响,代表原始土壤;低纬度区域组由南极半岛样品组成,由于研究站较多,受人类活动影响相对较大,代表相对原始土壤。加拿大阿尔伯特地区养殖场附近农田土壤代表受人类活动影响较大的土壤,用于与南极土壤中ARGs的分布特征进行比较。
表 1. 本研究中宏基因土壤样品信息 Table 1. The information of soil metagenomic datasets used in this study
| Habitat types | Habitats | Samples | Sampling sites | Coordiantes | Accession numbers |
| Antarctic high latitude regions (HLG) | Union Glacier | CP | Charles Peak | 79.731° S 83.199° W | SRR13094682 |
| EH | Elephant Head | 79.822° S 83.430° W | SRR7741809 | ||
| McMurdo Dry Valleys | AH | Miers Valley | 78.100° S 164.000° E | SRR3471615 | |
| Antarctic low latitude regions (LLG) | Antarctic Peninsula | NI | Nelson Island | 62.265° S 58.879° W | SRR7440170 |
| PH | Penguin habitat | 62.217° S 58.962° W | SRR7440171 | ||
| CGWS | Near Chinese Great Wall Station | 62.220° S 58.964° W | SRR7440174 | ||
| AI_1 | Ardley Island | 62.211° S 58.928° W | SRR7440168 | ||
| AI_2 | Ardley Island | 62.211° S 58.925° W | SRR7440169 | ||
| AI_3 | Ardley Island | 62.211° S 58.925° W | SRR7440167 | ||
| AI_4 | Ardley Island | 62.214° S 58.931° W | SRR7440165 | ||
| TWS | The west seacoast | 62.203° S 58.996° W | SRR7440163 | ||
| TSS | The south seacoast | 62.229° S 58.948° W | SRR7440166 | ||
| The surrounding regions of feedlot (SRF) | Alberta, Canada | AS1 | Agricultural soil with no fertilization | 53.93° N 116.58° W | SRR9030504 |
| Alberta, Canada | AS2 | Agricultural soil with fertilization for 6 months | 53.93° N 116.58° W | SRR9030505 | |
| Alberta, Canada | AS3 | Agricultural soil with fertilization for 1 year | 53.93° N 116.58° W | SRR9030506 | |
| Alberta, Canada | AS4 | Agricultural soil with fertilization for 2 years | 53.93° N 116.58° W | SRR9030507 |
表选项
1.2 宏基因数据组装与基因预测 本研究利用MetaWRAP v1.3.2软件[17]的Read_QC子程序对原始reads进行质量过滤(删除长度小于50 bp、平均碱基质量小于20、含有模糊碱基“N”的reads)并去除污染,使用MEGAHIT v1.2.9软件对获得的clean reads进行组装,kmer从21到141。利用FastQC评估拼接后的contigs,基于contigs数目、N50/N80值以及组装序列总长度,选择kmer为141的contigs (> 1000 bp)用于下一步分析。使用Prodigal v2.6.3软件预测不同样品contigs上的基因,利用CD-HIT-EST[18]以95%的相似性构建非冗余基因集,利用Salmon v0.9.1软件[19]将保留的clean reads与基因集进行比对,以计算各个样品中不同基因的丰度(TPM,transcripts per kilobase of exonmodel per million mapped reads)。
计算公式:TPMi=(Ni/Li)×1000000/sum (Ni/Li+……..+ Nm/Lm);其中,Ni:映射到基因i上的read数;Li:基因i的外显子长度的总和。
1.3 物种、ARGs及MGEs注释 本研究利用kraken v2.1.1软件[20]对南极土壤样品中的物种进行分类注释。使用Blastp将基因集与CARD数据库进行比对以鉴定ARGs (e值≤10–5)。筛选南极样品中含有ARG的contigs (抗性contigs),利用PlasFlow v1.1软件分析ARG在染色体以及质粒中的分布特征。进一步将获得的抗性contigs与ICEberg数据库进行比对,以e值为10–5作为比对标准,确定整合与接合元件(ICEs),并从描述的标题中提取物种分类信息。
1.4 统计分析 本研究采用Microsoft Excel 2019软件计算不同抗生素类型的ARGs丰度,利用R语言与Origin 9.1完成所有的图像绘制。基于标准化的ARGs丰度表,使用Primer 6软件进行NMDS分析与ANOSIM分析,以探究南极不同纬度地区与养殖场附近区域中ARGs的分布差异。采用Wilcoxon秩和检验对南极不同分组中微生物丰度与ARGs丰度等指标进行显著性差异分析。
2 结果和分析 2.1 序列组装分析 对16个样品的reads进行过滤处理,共获得674526370条clean reads,每个样品的reads数目范围为10579044–158811693。对contigs进行评估发现,contigs长度超过1000 bp的平均数目、平均N50及平均L50分别为61887、2339 bp与12541 bp,表明组装结果较好,序列长度可用。
2.2 南极土壤微生物群落 在contigs层面上对12个南极样品中土壤微生物进行物种注释,共获得3998个物种。其中,细菌占据主导地位(99.15%),其次是古菌(0.58%)与病毒(0.04%),0.23%的contigs没有分类学注释信息。在细菌类群中,优势门类为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),占比分别是50.58%、25.45%、6.58%和1.80%。作为变形菌门中丰度最高的类群,γ-变形菌纲主要分布在南极半岛附近,尤其是在西海岸地区,占变形菌门类群的80.25%;β-变形菌纲的分布趋势与γ-变形菌纲一致;而α-变形菌纲主要分布在南极高纬度地区(图 1)。其余每个细菌门的相对丰度均小于1%。
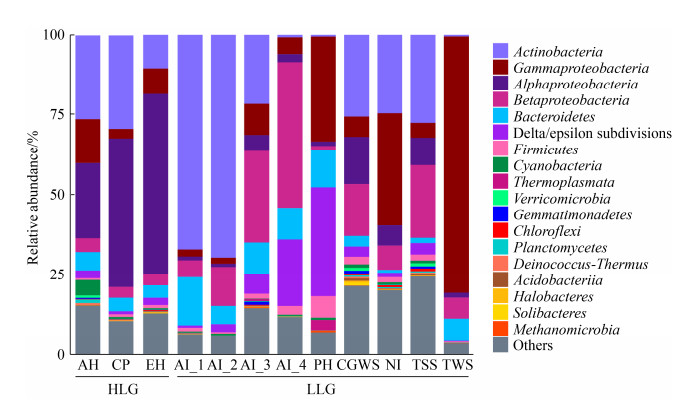 |
| 图 1 南极各站点土壤微生物在门或纲水平的组成分布 Figure 1 Microbial composition distributions at the phylum or class level across all soil samples in Antarctica. |
| 图选项 |
在古菌类群中,广古菌门(Euryarchaeota)丰度最高,占比为97.01%。其中,嗜盐杆菌(Halobacteria)占绝对优势(71.06%),其次是甲烷微菌(Methanomicrobia)、热球菌(Thermococci)、古丸菌(Archaeoglobi)、甲烷球菌(Methanococci) 与甲烷杆菌(Methanobacteria)。此外,奇古菌门(Thaumarchaeota,2.38%)与泉古菌门(Crenarchaeota,0.58%)类群也属于优势古菌门类。在病毒组中,98.42%的病毒(以双链DNA病毒、单链DNA病毒与逆转录病毒为主)分布在南极高纬度地区。其中,优势类群是微病毒(Microviridae)、有尾病毒(Caudovirales)、藻病毒(Algavirales)与疱疹病毒(Herpesvirales),分别占比79.02%、18.75%、1.99%和0.19%。
2.3 南极土壤ARGs的多样性 通过对16个样品组装的contigs进行基因预测,共发现2827824个非冗余开放阅读框(ORFs)。在南极土壤中,4662个ORFs预测为ARGs,ARGs占总基因数的比例范围为0.21%–2.97% (图 2)。其中,南极高纬度地区ARGs占比为0.28%,显著低于低纬度地区(1.93%) (P < 0.01)。在养殖场附近土壤中,23886个ORFs预测为ARGs,占总基因数的7.84%,显著高于南极地区(P < 0.01)。进一步研究发现,南极土壤中ARGs分布在4261个contigs上,每个样品中抗性contigs数目范围为93–1705个,而养殖场附近土壤中共发现20743个contigs携带ARGs,这些抗性contigs的占比是南极地区的24倍(平均占比:13.38% VS 0.56%) (P < 0.01)。
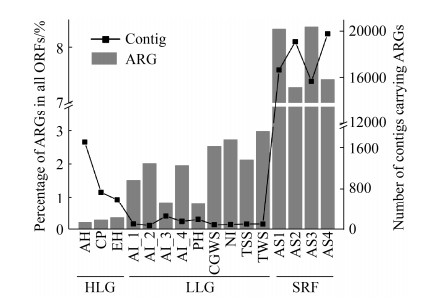 |
| 图 2 南极及养殖场附近土壤样品中ORFs预测为ARGs的百分比及抗性contigs数目 Figure 2 Percent of ORFs predicted as ARG and number of resistant contigs for each soil sample in Antarctica and the surrounding regions of feedlot. |
| 图选项 |
在养殖场附近土壤中,共检测到807种(23886个) ARGs,而南极土壤中检测到406种(4662个) ARGs。其中,231种ARGs在HLG、LLG以及SRF中都有发现,448种ARGs仅在SRF中被检测到,9种ARGs仅在南极土壤样品中检测到,分别为arr-1、AAC(3)-VIIIa、AAC(2')-Ic、LRA-1、MSI-OXA、CTX-M-121、APH(3'')-Ia、qacE与MCR-5.1 (图 3-A)。通过NMDS分析发现,南极土壤与养殖场附近土壤中ARGs呈现明显的分布差异(图 3-B)。进一步分析表明,在50%相似性水平下,16个样本分为3个大组,分别是南极高纬度地区、低纬度地区与养殖场附近农田区。ANOSIM分析表明,这三大组之间存在显著差异(R=0.975,P=0.001)。
 |
| 图 3 南极及养殖场附近土壤样品中ARGs的维恩分析(A)与NMDS分析(B) Figure 3 Venn analysis (A) and NMDS analysis (B) of ARGs in Antarctica and the surrounding regions of feedlot. |
| 图选项 |
根据抗生素抗性类型对南极地区ARGs进行分类,共发现25类ARGs (图 4)。最常见的ARGs是多重耐药类抗性基因,占所有ARGs丰度的50.70%,其次是四环素、氨基糖苷、氨基香豆素、大环内酯、硝基咪唑和利福平类抗性基因。这些抗性基因代表3种经典的耐药机制:(1) 通过RND型(resistance-nodulation-cell division)、ABC型(ATP-binding cassette)和MFS型(major facilitator superfamily)等外排泵排出多重耐药、氨基糖苷和大环内酯类抗生素;(2) 产生灭活酶(例如β-内酰胺酶、乙酰转移酶、磷酸转移酶),引起氨基糖苷、利福平和链阳霉素等抗生素失活;(3) 改变或保护抗生素靶位蛋白(例如抗四环素核糖体保护蛋白)。此外,一些ARGs可通过降低外膜通透性减少或阻止多抗耐药类、碳青霉烯类抗生素进入细胞内(图 4)。
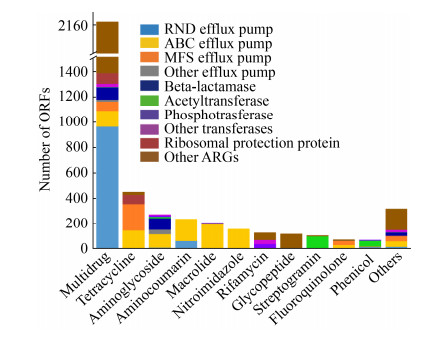 |
| 图 4 南极土壤样品中分配在主要抗生素耐药性机制的ORF数目 Figure 4 Number of ORFs assigned to major mechanisms for antibiotic resistance for all soil samples in Antarctica. |
| 图选项 |
在南极土壤中,检测到的406种ARGs隶属于95种抗生素耐药性(AMR)基因家族。其中,RND与rpoB多抗耐药基因是最常见的基因家族,其丰度占AMR基因家族总数的44.7%。而β-内酰胺酶类抗性基因是AMR基因家族中成员种类最多的一类(33种),其次是氨基糖苷修饰酶(如乙酰基转移酶或磷酸转移酶)抗性基因与多抗耐药类抗性基因(分别都是13种) (图 5-A)。在高纬度地区,共检测到373种ARGs,高于低纬度地区(273种ARGs) (图 3-A)。在检测到的25类ARGs中,18类ARGs在高纬度和低纬度地区都有发现(图 5-B)。其中,88%的ARGs类型在所有高纬度地区样本中存在,72%的ARGs类型在所有低纬度区域被发现,这表明在南极联合冰川和干谷地区抗生素耐药机制的组成更为复杂。34种ARGs (属于碳青霉烯类与头孢菌素类抗生素抗性基因)仅在高纬度地区样品中被检测到,而tet37、dfrE、msrE与qacE这4种ARGs仅在低纬度南极半岛土壤中存在(图 3-A)。此外,南极高纬度地区总的ARGs与硝基咪唑类、氨基糖苷类、糖肽类以及大环内酯类ARGs相对丰度显著高于南极半岛地区(P < 0.05),而四环素类与磺胺类ARGs相对丰度显著低于南极半岛地区(P < 0.05) (图 5-B)。
 |
| 图 5 南极不同纬度地区土壤中AMR基因家族(A)与不同抗生素类型抗性基因(B)的多样性 Figure 5 Diversity of antimicrobial resistance (AMR) gene families (A) and ARGs for different antibiotic classes (B) in Antarctic high and low latitude regions. |
| 图选项 |
2.4 MGEs介导ARGs的迁移 通过对南极土壤中抗性contigs进行可移动遗传元件分析,发现ARGs在染色体中比在质粒中更普遍(个数:1903 VS 741;频率:0.45 VS 0.17),例如多抗耐药类、四环素类、氨基糖苷类、肽类及大环内酯类抗性基因多分布于染色体上(图 6)。然而,磺胺类抗生素抗性基因多分布于质粒中。另外,通过与ICEberg数据库比对,共发现163个ICEs可携带14类ARGs,包括多抗耐药类、肽类、四环素类、氨基糖苷类和大环内酯类抗性基因等。这些携带ARGs的ICEs广泛分布于α-、β-与γ-变形菌纲中(图 7)。其中,假单胞菌(Pseudomonas)中ICE携带的ARGs种类最多(10种),其次是中慢生根瘤菌(Mesorhizobium)、鲍特氏杆菌(Bordetella)、布克氏菌(Burkholderia)和黄单胞杆菌(Xanthomonas),分别携带9、4、3和3种抗生素抗性基因。76%的细菌可携带两种以上抗性基因,属于多抗耐药菌(图 7)。
 |
| 图 6 南极土壤中按抗生素类型区分的染色体或质粒中ARGs的频率 Figure 6 The frequency of ARGs in chromosomes or plasmids summarized by antibiotic class in Antarctic soils. |
| 图选项 |
 |
| 图 7 南极土壤样本中ICEs宿主及其携带ARGs的丰度 Figure 7 Abundance of ICE hosts and their ARGs across all Antarctica soil samples. |
| 图选项 |
3 讨论 本研究利用宏基因组技术分析南极不同纬度地区土壤微生物群落的组成结构差异。相比于南极半岛等低纬度地区,南极高纬度地区(例如联合冰川、麦克默多干谷)靠近南极点,环境极端多样,微生物的多样性与生物量较低[21–22]。本研究发现,南极土壤生境中细菌类群占主导地位,这与南极其他地区报道的结果一致[12]。以鞘脂单胞菌目(Sphingomonadales)、红杆菌目(Rhodobacterales)和根瘤菌目(Rhizobiales)为主的α-变形菌纲主要分布在高纬度的联合冰川与麦克默多干旱谷地区(42.06%),其丰度显著高于低纬度的南极半岛地区(4.69%) (P < 0.01)。在高纬度地区,慢生根瘤菌(Bradyrhizobium)等类群可以耦合碳固定、进行氮固定以及硫矿化反应,为其他异养微生物提供碳、氮、硫源及能量[22–23],以弥补高纬度地区极端环境中的营养不足。红杆菌(Rhodobacter)可作为光自养细菌,通过光合作用供能[24]。鞘脂单胞菌(Sphingomonas)可参与该地区芳香化合物的生物降解与胞外高聚物的生物合成[25]。本研究中,嗜盐杆菌(Halobacteria)丰度为71.06%,广泛分布于各个采样点(除企鹅栖息地外),可产生菌视紫红质,促进ATP合成[26],为其他类群提供能量并参与南极生态系统稳定。本研究发现南极地区病毒类群种类多样,几乎所有的病毒(98.42%)都分布在高纬度地区,说明南极联合冰川与麦克默多干谷原始土壤可作为一种潜在的病毒资源库。
对原始生态环境中ARGs的研究有助于了解抗生素使用对细菌抗生素抗性组的影响。之前研究发现,在原始环境(例如深海、藏族地区)中ARGs的分布特征不同于人为影响较大的环境[27–28]。与此结果相一致,本研究NMDS分析表明,养殖场附近收集的土壤样品与南极土壤样品之间的ARGs分布存在显著差异(ANOSIM,P=0.001) (图 3-B)。南极土壤中ARGs的分布可以反映当地微生物群落的天然抗生素抗性组的主要特征[29]。但人类足迹(例如科考、旅游)的不断增加正在改变南极环境中的ARGs分布,尤其是在研究站附近[15, 30]。正如本研究报道,人类活动较多的南极半岛地区ARG占总基因数目的比例显著高于高纬度极端地区(P < 0.01) (图 2),且与人类临床抗生素(例如硝基咪唑、四环素类与大环内酯类)有关的ARGs也更多分布于南极半岛地区(图 5-B)。许多人为引入的非本地微生物可以在南极的恶劣条件下生存,并可能以新的抗性特征改变本地微生物群落结构[15, 31–32]。β-内酰胺类抗性基因是温带土壤抗性组的常见组成部分,但最近在未受干扰的寒冷土壤生态系统(例如北极多年冻土[33]和冰川冰芯[34])中发现了内酰胺酶基因。在本研究中,也观察到南极联合冰川和麦克默多干谷土壤中β-内酰胺类抗性基因的高度多样性(图 5-A)。
在406种南极土壤抗性基因中,最丰富的前10种ARGs (rpoB2、tetA(58)、APH(3')-Ia、mtrA、golS、novA、soxR、oleC、rsmA和msbA)占总ARGs的43%以上,它们与利福平、多抗耐药、四环素、氨基糖苷类、氨基香豆素、大环内酯类和硝基咪唑等抗生素的抗性有关。rpoB2的产物是抗利福平的RNA聚合酶亚基,通过改变利福平对其结合位点的亲和力而产生抗性,其宿主通常是诺卡氏菌[35]。APH(3')-Ia可编码氨基糖苷磷酸转移酶,造成氨基糖苷类抗生素失活,在许多重要病原体(例如大肠杆菌、鲍氏不动杆菌、肺炎杆菌、肠道沙门氏菌、粘质沙雷氏菌和棒状杆菌)的基因组、质粒以及转座子上普遍存在[36]。tetA(58)可编码四环素外排泵的关键组成部分,其宿主以类芽孢杆菌为主[37]。此外,novA、soxR、oleC和msbA属于ATP型运输家族,golS、novA和rsmA属于RND型运输家族,均可从细菌细胞中排出多种抗生素。
在外部环境的胁迫下,ARGs可通过ICEs、转座子和质粒等MGEs在不同基因之间或细菌宿主之间移动和迁移[8, 38]。南极大陆(尤其是高纬度极端地区)相对封闭,土著细菌群落受人为因素的影响相对较小,在该区域中MGEs的分布特征很好地反映了抗生素时代之前ARGs的迁移性[31]。本研究发现,除磺胺类抗生素抗性基因以外,其他所有检测到的ARGs主要由染色体携带,这与受抗生素污染严重的环境中的ARGs主要由质粒携带的结果不一致[9, 39]。本研究中,ICEs携带14类ARGs,占所有ARGs类型的56%。其中,位于ICEs上的多抗耐药类抗性基因种类最多,占ICEs携带总ARGs类型的68%,其次是四环素类,这与质粒主要携带的ARGs种类相一致(图 6)。因此,ICEs可能在ARGs的传播中扮演重要角色,特别是对于南极土壤中多抗耐药类和四环素类抗性基因。据报道,由ICEs携带的ARGs可促进多种革兰氏阳性和革兰氏阴性病原菌多重耐药性的产生[40–41]。
在南极不同纬度土壤样品中,携带ICEs的ARGs宿主分布在25种细菌中,其中24种细菌属于变形菌门类群(图 7),这反映了该类群在南极土壤生态位中的生存优势。之前研究表明,变形菌门成员是许多临床病原菌的主要类群[42]。本研究中,属于γ-变形菌纲的条件致病菌铜绿假单胞菌(Pseudomonas aeruginosa)含有8个ICEs家族,可携带多抗耐药类、氨基糖苷类和头孢菌素类ARGs。这些带有多种ARGs的ICEs家族存在于多种细菌中,并有可能驱动非自动转移元件的迁移[43–44]。肠道沙门氏菌(Salmonella enterica)的ICEs携带ompK37与mdtM,可通过降低外膜通透性阻止β-内酰胺类抗生素进入细胞[45]或通过外排泵将多抗耐药类等抗生素排出细胞[46]。奇异变形杆菌(Proteus mirabilis)的ICEPmiCHN905携带tetC,占ICEs携带所有tet的44%。因此,奇异变形杆菌可能作为四环素抗性基因迁移的贮藏库。R391 ICEs参与雷氏普罗威登斯菌(Providencia rettgeri)中APH(3')-Ia的携带,可使氨基糖苷类抗生素失活。令人意外的是,作为临床相关的致病菌,肠道沙门氏菌、奇异变形杆菌及雷氏普罗威登斯菌都只在高纬度联合冰川及麦克默多地区发现。我们推测,在抗生素时代之前,这些抗性菌可能已存在于原始自然环境中[8]。
综上所述,宏基因组学分析揭示了南极不同纬度地区土壤中微生物群落及其抗生素抗性基因的分布差异,这可能与该地区受不同程度人类活动的影响有关。后续可以对原始南极土壤的宏基因组数据进行宏基因组分箱(binning)分析,进一步确定这些天然ARG的宿主及其抗性与迁移机制。
致谢
感谢中山大学海洋科学学院陈保卫、加拿大莱斯布里奇研究和发展中心Tim A McAllister、南非比勒陀利亚大学微生物生态和基因组学中心(CMEG) Don A Cowan提供的宏基因组数据。
References
| [1] | Pruden A, Pei R, Storteboom H, Carlson KH. Antibiotic resistance genes as emerging contaminants: ? studies in northern Colorado. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [2] | Organization WH. Antibiotic resistance: global report on surveillance. World Health Organization, Geneva, Switzerland, 2014. |
| [3] | Livermore DM. Has the era of untreatable infections arrived?. Journal of Antimicrobial Chemotherapy, 2009, 64(suppl_1): i29-i36. |
| [4] | Sommer MOA, Dantas G, Church GM. Functional characterization of the antibiotic resistance reservoir in the human microflora. Science, 2009, 325(5944): 1128-1131. DOI:10.1126/science.1176950 |
| [5] | Baltz RH. Renaissance in antibacterial discovery from actinomycetes. Current Opinion in Pharmacology, 2008, 8(5): 557-563. DOI:10.1016/j.coph.2008.04.008 |
| [6] | Newman DJ, Cragg GM. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. Journal of Natural Products, 2020, 83(3): 770-803. DOI:10.1021/acs.jnatprod.9b01285 |
| [7] | D'Costa VM, King CE, Kalan L, Morar M, Sung WWL, Schwarz C, Froese D, Zazula G, Calmels F, Debruyne R, Golding GB, Poinar HN, Wright GD. Antibiotic resistance is ancient. Nature, 2011, 477(7365): 457-461. DOI:10.1038/nature10388 |
| [8] | Calero-Cáceres W, Ye M, Balcázar JL. Bacteriophages as environmental seservoirs of antibiotic resistance. Trends in Microbiology, 2019, 27(7): 570-577. DOI:10.1016/j.tim.2019.02.008 |
| [9] | Fresia P, Antelo V, Salazar C, Giménez M, D'Alessandro B, Afshinnekoo E, Mason C, Gonnet GH, Iraola G. Urban metagenomics uncover antibiotic resistance reservoirs in coastal beach and sewage waters. Microbiome, 2019, 7(1): 35. DOI:10.1186/s40168-019-0648-z |
| [10] | Peng S, Feng Y, Wang Y, Guo X, Chu H, Lin X. Prevalence of antibiotic resistance genes in soils after continually applied with different manure for 30 years. Journal of Hazardous Materials, 2017, 340: 16-25. DOI:10.1016/j.jhazmat.2017.06.059 |
| [11] | Cave R, Misra R, Chen J, Wang S, Mkrtchyan HV. Whole genome sequencing revealed new molecular characteristics in multidrug resistant staphylococci recovered from high frequency touched surfaces in London. Scientific Reports, 2019, 9(1): 9637. DOI:10.1038/s41598-019-45886-6 |
| [12] | Boetius A, Anesio AM, Deming JW, Mikucki JA, Rapp JZ. Microbial ecology of the cryosphere: sea ice and glacial habitats. Nature Reviews Microbiology, 2015, 13(11): 677-690. DOI:10.1038/nrmicro3522 |
| [13] | De Souza M-J, Nair S, Loka Bharathi PA, Chandramohan D. Metal and antibiotic-resistance in psychrotrophic bacteria from Antarctic marine waters. Ecotoxicology, 2006, 15(4): 379-384. DOI:10.1007/s10646-006-0068-2 |
| [14] | Wang F, Stedtfeld RD, Kim O-S, Chai B, Yang L, Stedtfeld TM, Hong SG, Kim D, Lim HS, Hashsham SA, Tiedje JM, Sul WJ. Influence of soil characteristics and proximity to Antarctic research stations on abundance of antibiotic resistance genes in soils. Environmental Science & Technology, 2016, 50(23): 12621-12629. |
| [15] | Stark JS, Corbett PA, Dunshea G, Johnstone G, King C, Mondon JA, Power ML, Samuel A, Snape I, Riddle M. The environmental impact of sewage and wastewater outfalls in Antarctica: An example from Davis station, East Antarctica. Water Research, 2016, 105: 602-614. DOI:10.1016/j.watres.2016.09.026 |
| [16] | Van Goethem MW, Pierneef R, Bezuidt OKI, Van De Peer Y, Cowan DA, Makhalanyane TP. A reservoir of 'historical' antibiotic resistance genes in remote pristine Antarctic soils. Microbiome, 2018, 6(1): 40. DOI:10.1186/s40168-018-0424-5 |
| [17] | Uritskiy GV, DiRuggiero J, Taylor J. MetaWRAP-a flexible pipeline for genome-resolved metagenomic data analysis. Microbiome, 2018, 6(1): 158. DOI:10.1186/s40168-018-0541-1 |
| [18] | Li W, Godzik A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics, 2006, 22(13): 1658-1659. DOI:10.1093/bioinformatics/btl158 |
| [19] | Patro R, Duggal G, Love MI, Irizarry RA, Kingsford C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods, 2017, 14(4): 417-419. DOI:10.1038/nmeth.4197 |
| [20] | Wood DE, Salzberg SL. Kraken: ultrafast metagenomic sequence classification using exact alignments. Genome Biology, 2014, 15(3): R46. DOI:10.1186/gb-2014-15-3-r46 |
| [21] | Cordero RR, Damiani A, Ferrer J, Jorquera J, Tobar M, Labbe F, Carrasco J, Laroze D. UV irradiance and albedo at Union Glacier Camp (Antarctica): A case study. PLoS ONE, 2014, 9(3): e90705. DOI:10.1371/journal.pone.0090705 |
| [22] | Li Y, Cha QQ, Dang YR, Chen XL, Wang M, McMinn A, Espina G, Zhang YZ, Blamey JM, Qin QL. Reconstruction of the functional ecosystem in the high light, low temperature Union Glacier region, Antarctica. Frontiers in Microbiology, 2019, 10: 2408. DOI:10.3389/fmicb.2019.02408 |
| [23] | VanInsberghe D, Maas KR, Cardenas E, Strachan CR, Hallam SJ, Mohn WW. Non-symbiotic Bradyrhizobium ecotypes dominate North American forest soils. The ISME Journal, 2015, 9(11): 2435-2441. DOI:10.1038/ismej.2015.54 |
| [24] | Imam S, Noguera DR, Donohue TJ. Global insights into energetic and metabolic networks in Rhodobacter sphaeroides. BMC Systems Biology, 2013, 7: 89. DOI:10.1186/1752-0509-7-89 |
| [25] | Zhou L, Li H, Zhang Y, Han S, Xu H. Sphingomonas from petroleum-contaminated soils in Shenfu, China and their PAHs degradation abilities. Brazilian Journal of Microbiology, 2016, 47(2): 271-278. DOI:10.1016/j.bjm.2016.01.001 |
| [26] | Oren A. The microbiology of red brines. Advances in Applied Microbiology, 2020, 113: 57-110. |
| [27] | Chen B, Yang Y, Liang X, Yu K, Zhang T, Li X. Metagenomic profiles of antibiotic resistance genes (ARGs) between human impacted estuary and deep ocean sediments. Environmental Science & Technology, 2013, 47(22): 12753-12760. |
| [28] | Chen B, Yuan K, Chen X, Yang Y, Zhang T, Wang Y, Luan T, Zou S, Li X. Metagenomic analysis revealing antibiotic resistance genes (ARGs) and their genetic compartments in the Tibetan environment. Environmental Science & Technology, 2016, 50(13): 6670-6679. |
| [29] | Yuan K, Yu K, Yang R, Zhang Q, Yang Y, Chen E, Lin L, Luan T, Chen W, Chen B. Metagenomic characterization of antibiotic resistance genes in Antarctic soils. Ecotoxicology and Environmental Safety, 2019, 176: 300-308. DOI:10.1016/j.ecoenv.2019.03.099 |
| [30] | Miller RV, Gammon K, Day MJ. Antibiotic resistance among bacteria isolated from seawater and penguin fecal samples collected near Palmer Station, Antarctica. Canadian Journal of Microbiology, 2009, 55(1): 37-45. DOI:10.1139/W08-119 |
| [31] | Hughes KA, Cowan DA, Wilmotte A. Protection of Antarctic microbial communities-'out of sight, out of mind'. Frontiers in Microbiology, 2015, 6: 151. |
| [32] | Brat K, Sedlacek I, Sevcikova A, Merta Z, Laska K, Sevcik P. Imported anthropogenic bacteria may survive the Antarctic winter and introduce new genes into local bacterial communities. Polish Polar Research, 2016, 37(1): 89-104. DOI:10.1515/popore-2016-0001 |
| [33] | Perron GG, Whyte L, Turnbaugh PJ, Goordial J, Hanage WP, Dantas G, Desai MM. Functional characterization of bacteria isolated from ancient Arctic soil exposes diverse resistance mechanisms to modern antibiotics. PLoS ONE, 2015, 10(3): e0069533. DOI:10.1371/journal.pone.0069533 |
| [34] | Segawa T, Takeuchi N, Rivera A, Yamada A, Yoshimura Y, Barcaza G, Shinbori K, Motoyama H, Kohshima S, Ushida K. Distribution of antibiotic resistance genes in glacier environments. Environmental Microbiology Reports, 2013, 5(1): 127-134. DOI:10.1111/1758-2229.12011 |
| [35] | Ishikawa J, Chiba K, Kurita H, Satoh H. Contribution of rpoB2 RNA polymerase beta subunit gene to rifampin resistance in Nocardia species. Antimicrob Agents Chemother, 2006, 50(4): 1342-1346. DOI:10.1128/AAC.50.4.1342-1346.2006 |
| [36] | Tauch A, Krieft S, Kalinowski J, Pühler A. The 51, 409-bp R-plasmid pTP10 from the multiresistant clinical isolate Corynebacterium striatum M82B is composed of DNA segments initially identified in soil bacteria and in plant, animal, and human pathogens. Molecular & General Genetics, 2000, 263(1): 1-11. |
| [37] | Pawlowski AC, Wang W, Koteva K, Barton HA, McArthur AG, Wright GD. A diverse intrinsic antibiotic resistome from a cave bacterium. Nature Communications, 2016, 7: 13803. DOI:10.1038/ncomms13803 |
| [38] | Bellanger X, Guilloteau H, Bonot S, Merlin C. Demonstrating plasmid-based horizontal gene transfer in complex environmental matrices: A practical approach for a critical review. Science of The Total Environment, 2014, 493: 872-882. DOI:10.1016/j.scitotenv.2014.06.070 |
| [39] | Che Y, Xia Y, Liu L, Li A-D, Yang Y, Zhang T. Mobile antibiotic resistome in wastewater treatment plants revealed by Nanopore metagenomic sequencing. Microbiome, 2019, 7(1): 44. DOI:10.1186/s40168-019-0663-0 |
| [40] | Mingoia M, Morici E, Morroni G, Giovanetti E, Del Grosso M, Pantosti A, Varaldo PE. Tn5253 family integrative and conjugative elements carrying mef(I) and catQ determinants in Streptococcus pneumoniae and Streptococcus pyogenes. Antimicrobial Agents and Chemotherapy, 2014, 58(10): 5886-5893. DOI:10.1128/AAC.03638-14 |
| [41] | Wang Y, Wang GR, Shelby A, Shoemaker NB, Salyers AA. A newly discovered Bacteroides conjugative transposon, CTnGERM1, contains genes also found in gram-positive bacteria. Applied and Environmental Microbiology, 2003, 69(8): 4595-4603. DOI:10.1128/AEM.69.8.4595-4603.2003 |
| [42] | Boucher HW, Talbot GH, Bradley JS, Edwards JE, Gilbert D, Rice LB, Scheld M, Spellberg B, Bartlett J. Bad bugs, no drugs: no ESKAPE! An update from the Infectious Diseases Society of America. Clinical Infectious Diseases, 2009, 48(1): 1-12. DOI:10.1086/595011 |
| [43] | Xiao KQ, Li B, Ma L, Bao P, Zhou X, Zhang T, Zhu YG. Metagenomic profiles of antibiotic resistance genes in paddy soils from South China. FEMS Microbiology Ecology, 2016, 92(3): fiw023. DOI:10.1093/femsec/fiw023 |
| [44] | Fierer N, Leff JW, Adams BJ, Nielsen UN, Bates ST, Lauber CL, Owens S, Gilbert JA, Wall DH, Caporaso JG. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes. Proceedings of the National Academy of Sciences, 2012, 109(52): 21390-21395. DOI:10.1073/pnas.1215210110 |
| [45] | Doménech-Sánchez A, Hernández-Allés S, Martínez-Martínez L, Benedí VJ, Albertí S. Identification and characterization of a new porin gene of Klebsiella pneumoniae: its role in beta-lactam antibiotic resistance. Journal of Bacteriology, 1999, 181(9): 2726-2732. DOI:10.1128/JB.181.9.2726-2732.1999 |
| [46] | Holdsworth SR, Law CJ. The major facilitator superfamily transporter MdtM contributes to the intrinsic resistance of Escherichia coli to quaternary ammonium compounds. The Journal of Antimicrobial Chemotherapy, 2013, 68(4): 831-839. DOI:10.1093/jac/dks491 |
