唐昊#, 郑莉#, 雷露, 王明珺, 李沅秋, 罗朝兵


乐山师范学院生命科学学院, 四川 乐山 614000
收稿日期:2020-08-29;修回日期:2020-10-23;网络出版日期:2020-11-10
基金项目:四川省科技计划(2019YFG0139)
*通信作者:罗朝兵, E-mail: 13366181512@163.com.
#共同第一作者。
摘要:[目的] 烟曲霉Aspergillus fumigatus作为一类具有纤维素降解能力的真菌,对其基因组的研究,将有利于从A.fumigatus中挖掘和开发利用与纤维素降解相关的酶资源。[方法] 利用CMC选择培养基和刚果红染色法从长足大竹象肠道中分离和筛选出纤维素降解菌A.fumigatus HZ1,同时采用Illumina PE150平台进行基因组测序,随后进行了相关的生物信息学分析,此外还利用了DNS法测定了其纤维素酶活。[结果] 纤维素降解菌A.fumigatus HZ1基因组大小为27.45 Mb,GC含量为49.43%;通过NR、KOG、GO、Swissprot、eggNOG、KEGG和Pfam数据库注释结果表明基因组包含9473个基因;同时碳水化合物活性酶(CAZyme)注释结果表明基因组含有534个CAZyme基因,并与其他4种A.fumigatus基因组CAZyme分布无显著差异;本研究还鉴定出多种与木质纤维素降解相关的纤维素酶基因、半纤维素酶基因和木质素酶基因;此外纤维素酶活结果表明,在CMC培养基中其酶活呈上升趋势且具有较高活性。[结论] 本研究首次对A.fumigatus HZ1基因组进行了测序和分析,探讨了其纤维素降解的遗传基础,并通过酶活验证了其纤维素降解潜力,为该菌的实际应用提供了理论基础。
关键词:烟曲霉HZ1长足大竹象基因组测序生物信息学分析纤维素酶纤维素降解真菌
Genome sequencing and bioinformatics analysis of cellulolytic fungus Aspergillus fumigatus HZ1
Hao Tang#, Li Zheng#, Lu Lei, Mingjun Wang, Yuanqiu Li, Chaobing Luo


College of Life Sciences, Leshan Normal University, Leshan 614000, Sichuan Province, China
Received: 29 August 2020; Revised: 23 October 2020; Published online: 10 November 2020
*Corresponding author: Chaobing Luo, E-mail: 13366181512@163.com.
Foundation item: Supported by the Sichuan Science and Technology Program (2019YFG0139)
# These authors contributed equally.
Abstract: [Objective] Aspergillus fumigatus is a cellulolytic fungus. The research of its genome will facilitate the mining and development of enzyme resources associated with cellulose degradation. [Methods] A. fumigatus HZ1 was isolated from the gut of Cyrtotrachelus buqueti by CMC selective medium and Congo red staining method. Illumina PE50 platform was used to conduct genome sequence and the relative bioinformatics analysis was performed subsequently. Furthermore, the cellulase activities of this strain were determined by DNS method. [Results] The genome size of A. fumigatus HZ1 was 27.45 Mb with a GC content of 49.43%. The results from NR, KOG, GO, Swissprot, eggNOG, KEGG and Pfam database annotations reveal that the genome contained 9473 genes, and the carbohydrate active enzyme (CAZyme) annotation shows that the genome contained 534 CAZyme genes and coincided with the distribution of CAZyme in the other four A. fumigatus genomes. A variety of cellulase genes, hemicellulase genes and ligninase genes related to lignocellulose degradation were identified. In addition, cellulase activity showed an increasing trend and high activity in CMC medium. [Conclusion] Our findings provide reference for the application of A. fumigatus.
Keywords: Aspergillus fumigatus HZ1Cyrtotrachelus buquetigenome sequencebioinformatics analysiscellulasecellulolytic fungus
纤维素由β-1, 4-糖苷键将葡萄糖结合在一起,其分布广、储量丰富,提供了可再生、可持续的绿色有机资源[1],但其具有难降解及不溶于水等特性,阻碍了对纤维素的利用。目前,利用微生物降解纤维素以解决能源短缺和环境污染已成为热门课题,而分离纤维素降解菌株是研究这一课题的基础。真菌能够通过产生纤维素酶并将这些酶运输到外部环境中降解纤维素[2],曲霉(Aspergillus)因其能分泌与木质纤维素降解有关的多种酶而备受关注。Aspergillus niger NS-2以麦麸为底物时能够产生可观的木质纤维素酶产量[3];从堆肥中分离出Aspergillus fumigatus Z5,经初步研究表明,在固态发酵条件下能分泌耐热纤维素酶[4];Aspergillus fumigatus可以高效糖化碱预处理的水稻秸秆[5];Aspergillus fumigatus NS可产生大量具有热稳定性的纤维素酶[6]。
植食性的昆虫肠道中往往存在能够降解纤维素的菌群。长足大竹象(Cyrtotrachelus buqueti)作为一种专食性害虫,严重危害竹产业的发展。Luo等[7-8]通过转录组测序等手段揭示了长足大竹象可降解慈竹木质纤维素,进一步通过微生物测序等手段发现长足大竹象肠道内存在大量的共生菌,并参与木质纤维素的降解,包括纤维素降解[9]。并且一些纤维素降解菌已被成功分离鉴定并表现出纤维素降解能力,如贝莱斯芽胞杆菌(Bacillus velezensis LC1)[10]。本研究从长足大竹象肠道内筛选出一株纤维素降解真菌,通过基因组测序及分析揭示其降解纤维素的遗传基础,并且通过纤维素酶活的测定确定其纤维素降解能力。
1 材料和方法 1.1 材料
1.1.1 底物缓冲液: 1% CMC缓冲溶液:200 mL蒸馏水中加入2 g羧甲基纤维素钠,用纱布过滤水浴加热后的溶液。取100 mL滤液、40 mL蒸馏水和20 mL的0.2 mol/L醋酸缓冲液(pH 4.8)振荡混匀,4 ℃低温保存;1%水杨苷溶液:取1 g D-水杨苷和适量0.2 mol/L的醋酸-醋酸钠缓冲液(pH 4.8)于烧杯加热溶解,定容至100 mL,4 ℃低温保存;1%微晶纤维素:取1 g微晶纤维素和适量0.2 mol/L的醋酸-醋酸钠缓冲液(pH 4.8)于烧杯中加热溶解,定容至100 mL,4 ℃低温保存。
1.1.2 培养基: CMC培养基(g/L):羧甲基纤维素钠5,NaNO3 3,KH2PO4 1,MgSO4 0.5,KCl 0.5,FeSO4 0.01,琼脂15,pH调至7.0;PDA培养基(g/L):马铃薯200,葡萄糖20,琼脂粉17;TSA培养基(g/L):胰蛋白胨17,葡萄糖2.5,NaCl 5,K2HPO3 2.5,KH2PO3 2.35,大豆蛋白胨3,琼脂15。
1.2 菌株的分离筛选 长足大竹象样品从四川乐山市沐川县(东经103°98′,北纬28°96′)采集。将生理盐水、蒸馏水、研钵、镊子、解剖针、剪刀、试管等以121 ℃灭菌20 min,将挑选出的成虫用75%的酒精擦拭后,紫外杀菌2 h。无菌条件下,解剖长足大竹象,挑出其完整肠道,于装有少量无菌水的灭菌研钵中研磨均匀。将肠道研磨液按浓度梯度稀释为10–1至10–9,分别取0.1 mL的10–7至10–9的稀释液涂布于CMC培养基,30 ℃恒温培养。CMC培养基用0.2%刚果红染液染色后用于筛选纤维素降解菌。
1.3 真菌的分子生物学鉴定 以真菌鉴定通用引物ITS1 (5′-TCCGTAGG TGAACCTGCGG-3′)和ITS4 (5′-TCCTCCGCTTA TTGATATGC-3′)扩增序列。PCR反应条件如下:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 7 min。
扩增产物检验使用1%琼脂糖凝胶电泳,序列测序由上海欧易生物医学科技有限公司完成。通过MEGA5软件邻接法构建NCBI GenBank中检索到的20条序列和参考基因之间的系统发育树。
1.4 纤维素酶活的测定 将目的菌株接种于PDA培养基上,28 ℃培养7 d至产生大量孢子后,收集孢子并制备孢子悬液,1:50接种于CMC培养基,28 ℃、220 r/min振荡培养6 d。分别收集培养第1、第3和第6天培养物,使用冷冻离心机4 ℃、8000 r/min离心20 min收集上清液制成粗酶液,用于纤维素酶活测定。
分别在0.3 mL的1% CMC、1%水杨苷和1%微晶纤维素溶液中加入0.1 mL粗酶液,50 ℃恒温反应60 min后加入0.6 mL DNS显色剂,经过5 min沸水浴使反应终止。酶标板中加入0.2 mL反应液,OD值于540 nm波长下测定。每组实验设置5个重复,空白对照为高温灭活的粗酶液。
1.5 基因组DNA提取与测序 将目的菌株接种于PDA培养基上,28 ℃培养7 d至产生大量孢子后,收集孢子并制备孢子悬液,接种于TSA培养基中,28 ℃、220 r/min振荡培养2 d,使用冷冻离心机4 ℃、8000 r/min离心10 min收集菌体。菌株基因组DNA提取采用CTAB法[11],纯化用中国南京诺唯赞生物科技有限公司生产的Wizard基因组DNA纯化试剂盒,–20 ℃备用。
将提取的真菌基因组DNA委托上海欧易生物医药科技有限公司进行基因组二代测序。测序平台为Illumina PE150平台,双端测序,测序深度≥100X,插入片段为350 bp,读长150 bp,数据量为6 G。
1.6 基因组组装及生物信息学分析 通过Illumina平台,得到了大量的样本双端测序数据。鉴于数据错误率对结果的影响,采用Trimmomati[12]软件对原始数据进行质量预处理,并对整个质控过程中的reads数进行统计汇总。使用SOAPdenovo[13]软件对质控后的reads进行序列拼接,拼接成scaffolds。使用软件GeneMark-ES (v4.33)[14]来进行编码基因预测,使用tRNAscan-SE (v1.3.1)[15]软件预测tRNA,RNAmmer (v1.2)[16]软件预测rRNA,Rfam (v10.0)[17]软件预测sRNA,RepeatMasker (v4.0.7)[18]软件进行重复序列预测。
常见数据库注释包括NR注释、KOG功能注释[19]、GO分类[20]、Swissprot[21]、eggNOG[22]、KEGG[23]和Pfam[24]。对于NR、KOG、GO、Swissprot、eggNOG和KEGG数据库注释,采用Diamond[25]软件进行比对,取E-value < 1E-5的注释,筛选具有最高序列相似性的蛋白,从而得到功能注释信息。Pfam数据库注释采用HMMER[26]软件和蛋白家族模型比对,取E-value < 1E-5,从而筛选出得分最高的家族。
专门功能数据库包括耐药基因CARD、碳水化合物活性酶CAZy、真菌致病菌毒力因子DFVF。CARD数据库注释,采用blast[27]与数据库比对,取E-value < 1E-10的注释;CAZy数据库,利用HMMER[26]软件与数据库比对,取E-value < 1E-10的注释;DFVF注释利用Diamond[25]软件与数据库比对,取E-value < 1E-10的注释。
2 结果和分析 2.1 纤维素降解菌Aspergillus fumigatus HZ1的鉴定及酶活测定 从长足大竹象肠道中筛选到一株纤维素降解真菌,Sanger测序获得其ITS序列(登录号:NMDCN0000104),其长度为500 bp。将ITS序列提交NCBI进行blast,并将所得序列用于构建最大似然系统进化树。结果表明A. fumigatus HZ1与菌株A. fumigatus HNC-92Y-3 (MN588060)相似性最高,表明A. fumigatus HZ1与A. fumigatus HNC-92Y-3亲缘性最近(图 1-A)。
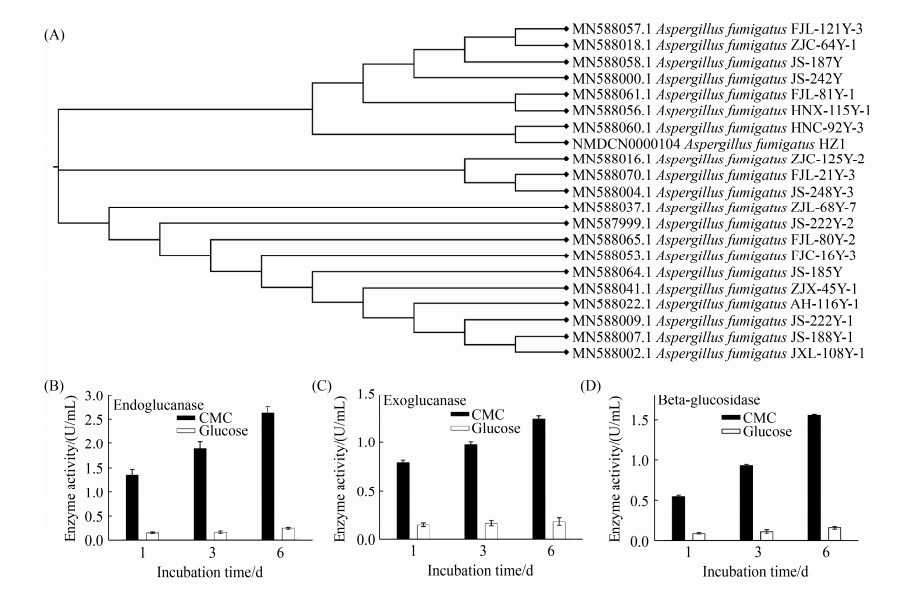 |
| 图 1 A. fumigatus HZ1分子鉴定(A)及纤维素酶活(B–D) Figure 1 Molecular identification (A) and cellulolytic activities (B–D) of A. fumigatus HZ1. Descriptive data were expressed as mean±standard error of mean (SEM, n=5). SEM describes the dispersion degree of mean sampling distribution and measures the size of mean sampling error of the enzyme activities (n=5). |
| 图选项 |
为了探究A. fumigatus HZ1纤维素降解能力,测定了该菌株在羧甲基纤维素(CMC)培养中的纤维素酶活,包括内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶。结果表明3种纤维素酶活随处理时间呈显著上升(P < 0.05);处理第1天A. fumigatus HZ1内切葡聚糖酶酶活为1.358±0.11 U/mL,然后处理第6天上升到2.634±0.13 U/mL (图 1-B);
外切葡聚糖酶酶活从0.797±0.024 U/mL上升到1.247±0.033 U/mL (图 1-C);而β-葡萄糖苷酶酶活从0.548±0.017 U/mL上升到1.556±0.12 U/mL (图 1-D)。
2.2 基因组测序与组装 本研究利用二代Illumina Novaseq平台对Aspergillus fumigatus HZ1基因组测序(表 1)。一共得到5232.64 Mb原始数据量,对原始数据过滤后得到4956.16 Mb的数据量,其中Q30含量为97.73%,GC含量为48.89%,K-mer预估其基因组大小为30.10 Mb (K-mer值为17,深度为31),修正后为29.70 Mb。将过滤后的数据组装成Scaffold后,统计数据(表 1)。结果表明,该组装基因组一共有1587个Scaffolds,总长度(即基因组大小)为27.45 Mb,其中大于500 bp的有266个,大于1 kb的有218个,最长Scaffold长度为2537.63 kb,基因组GC含量为49.43%,N50值为547959 bp。组装基因组包含9473个基因,总长度为13, 834.71 kb,GC含量为54.12%,包含29个rRNA、1个sRNA和1个tRNA。重复序列的总长度为232558 bp,占基因组长度的0.81%。在重复元件中,串联重复序列占0.74%,散在重复序列0.08%。基因组数据已上传国家微生物科学数据中心(登录号:NMDC40001003)。
表 1. A. fumigatus HZ1基因组测序和组装 Table 1. Genome sequence and assemble of A. fumigatus HZ1
| Assemble | Genome |
| Raw bases/Mb | 5232.64 |
| Clean bases/Mb | 4956.16 |
| Genome size of K-mer/Mb | 30.10 |
| Revised Genome size of K-mer/Mb | 29.70 |
| Scaffolds number (≥0 bp) | 1587 |
| Scaffolds number (≥500 bp) | 266 |
| Scaffolds number (≥1 kb) | 218 |
| Longest scaffold/kb | 2537.63 |
| Total length/Mb | 27.45 |
| GC/% | 49.43 |
| N50/bp | 547959 |
| Gene number | 9473 |
| Total length/kb | 13834.71 |
| GC% (gene region) | 54.12 |
| rRNA | 29 |
| tRNA | 1 |
| sRNA | 1 |
| Tandem repeat | 213192 |
| Interspersed repeat | 19366 |
表选项
2.3 基因组注释 通过NR、KOG、GO、Swissprot、eggNOG、KEGG和Pfam数据库注释。结果表明A. fumigatus HZ1基因组9473个基因中,分别有9425 (99.49%)、6674 (70.45%)、3487 (36.81%)、5033 (53.13%)、7363 (77.73%)、6540 (69.04%)、1 (0.01%)个基因注释到NR、Swissprot、KEGG、KOG、eggNOG、GO和Pfam数据库(图 2)。这些注释基因中,2877个基因同时注释到6个数据库,2283个基因同时注释到5个数据库,1240个基因只注释到NR数据库(图 2)。
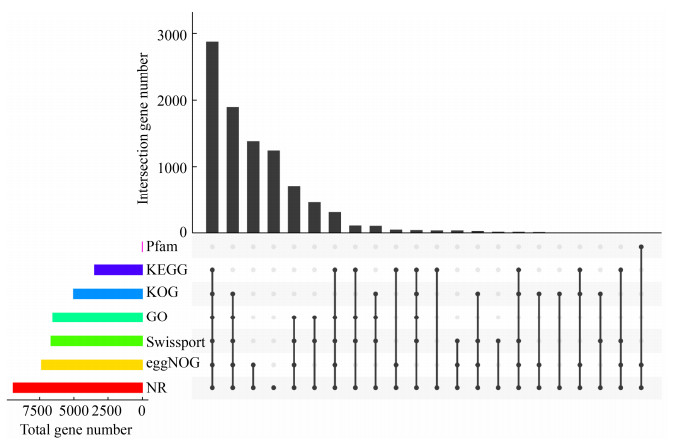 |
| 图 2 基因功能注释韦恩图 Figure 2 Venn plot of gene annotation. |
| 图选项 |
2.3.1 KOG注释: 共有6674个基因被注释到2989个KOGs中,其中富集基因最多的4个KOGs分别是主要易化超家族载体(predicted transporter major facilitator superfamily,KOG0254,124个基因)、主要易化超家族突触囊泡转运蛋白和相关转运蛋白(synaptic vesicle transporter SVOP and related transporters major facilitator superfamily,KOG0255,64个基因)、易化超家族透性酶(permease of the major facilitator superfamily,KOG2533,52个基因)和氨基酸转运载体(amino acid transporters,KOG1286,51个基因) (图 3)。为了从遗传水平阐明A. fumigatus HZ1纤维素降解功能,本研究分析了参与碳水化合物代谢的KOGs。共有300个基因被注释到碳水化合物代谢中,包括297个KOGs,以及α-淀粉酶(KOG0471)、β-葡萄糖苷酶(KOG0496)和几丁质酶(KOG2806)等。
 |
| 图 3 A. fumigatus HZ1的KOG分析 Figure 3 Gene distribution based on KOG classification of A. fumigatus HZ1. |
| 图选项 |
2.3.2 GO注释: GO注释表明,分子功能包含最多的基因数(5612),其次是生物过程(基因数:5509)和细胞成分(基因数:5488) (图 4)。此外,确定了与碳水化合物代谢相关的GO,包括GO0004553 (水解O-糖基化合物的水解酶活性)、GO0005975 (碳水化合物代谢过程)和GO0016787 (水解酶活性)。
 |
| 图 4 A. fumigatus HZ1的GO分析 Figure 4 Gene distribution based on GO classification of A. fumigatus HZ1. |
| 图选项 |
2.3.3 KEGG注释: KEGG途径的6个分类中,基因数量最多的是与代谢相关的,其次是遗传信息处理(图 5)。在KEGG注释的代谢中,最主要的途径是分别包括385和292个基因的碳水化合物代谢以及氨基酸代谢。蔗糖和淀粉代谢(starch and sucrose metabolism,ko00500,89个基因)、糖酵解/糖异生(glycolysis/gluconeogenesis,ko00010,51个基因)、氨基糖和核苷酸糖代谢(amino sugar and nucleotide sugar metabolism,ko00520,74个基因)等途径在上述代谢途径中占主导地位。蔗糖和淀粉代谢途径中的内切葡聚糖酶(EC.3.2.1.4)参与纤维素降解,表明A. fumigatus HZ1能将纤维素水解成纤维二糖,最终变为β-D-葡萄糖。
 |
| 图 5 A. fumigatus HZ1的KEGG分类 Figure 5 Gene distribution based on KEGG classification of A. fumigatus HZ1. |
| 图选项 |
2.4 碳水化合物活性酶(CAZyme)注释 基因组碳水化合物活性酶注释结果表明,A. fumigatus HZ1基因组注释到534个CAZyme基因,占全部编码基因的5.64%,其中糖苷水解酶(GH)对碳水化合物的降解具有关键作用,基因组中注释到270个GH基因(图 6)。此外,还鉴定了96个糖基转移酶(GT)基因,80个糖酯酶(CE)基因,15个多糖裂解酶(PL)基因,66个具有辅助活性的酶(AA)基因和7个碳水化合物结合模块(CBM)基因(图 6)。一共有270个基因注释到62个GH家族,其中数量最多的为GH43、GH3、GH13、GH18、GH16和GH5,分别有19、18、16、16、14和14个成员;80个CE基因分配到11个CE家族,包括,CE1、CE2、CE3、CE4等,其中CE10成员数量最多,达到40个;AA中共有11个家族被注释,其中成员数量最多的是AA7和AA3;6个PL家族被注释,包括PL1、PL20、PL26、PL3、PL4和PL9。
 |
| 图 6 A. fumigatus HZ1的碳水化合物活性酶及结合模块注释 Figure 6 Gene distribution based on CAZyme and carbohydrate binding module annotation A. fumigatus HZ1. GH: glycoside hydrolases; CBM: carbohydrate binding module; CE: carbohydrate esterase; GT: glycosyltransferases; PL: polysaccharide lyase; AA: auxiliary activities. |
| 图选项 |
本研究进一步通过比较基因组学研究了A. fumigatus HZ1与其他A. fumigatus 基因组中CAZyme基因。结果表明A. fumigatus HZ1与其他4种A. fumigatus的CAZyme分布无显著差异(表 2),GH成员数量最多,其次是GT。A. fumigatus HZ1基因组中CAZyme基因数量最多,其中AA和CE基因数目最多,GH和GT数目最少。
表 2. 烟曲霉基因组中CAZyme基因数目 Table 2. CAZyme gene numbers in A. fumigatus genomes
| Species | CAZyme proteins | GH | GT | CE | PL | AA | CBM |
| A. fumigatus Z5 | 529 | 272 | 98 | 69 | 14 | 55 | 21 |
| A. fumigatus AF293 | 528 | 269 | 109 | 75 | 15 | 57 | 3 |
| A. fumigatus var. RP-2014 | 498 | 257 | 99 | 68 | 15 | 50 | 9 |
| A. fumigatus A1163 | 532 | 270 | 106 | 73 | 15 | 58 | 10 |
| A. fumigatus HZ1 | 534 | 270 | 96 | 80 | 15 | 66 | 7 |
| Enzymes: GH: glycoside hydrolase; GT: glycosyltransferase; CE: carbohydrate esterase; PL: polysaccharide lyase; CBM: carbohydrate-binding module; AA: auxiliary activities. Genomes: A. fumigatus Z5 (GenBank: GCA_001029325.1); A. fumigatus AF293 (GenBank: AAHF00000000.1); A. fumigatus var. RP-2014 (GenBank: JHOI00000000.1); A. fumigatus A1163 (GenBank: ABDB00000000.1). | |||||||
表选项
2.5 木质纤维素酶基因
2.5.1 纤维素酶: A. fumigatus HZ1基因组中共注释到55个纤维素酶基因,其中包括25个内切葡聚糖酶基因、7个外切葡聚糖酶基因和23个β-葡萄糖苷酶基因(表 3)。内切葡聚糖酶基因编码的蛋白质属于GH12、GH5、GH51、GH6、GH7和GH74家族;外切葡聚糖酶基因编码的蛋白质属于GH55家族;β-葡萄糖苷酶基因编码的蛋白质属于GH1和GH3 (表 2)。丰富的纤维素酶基因表明A. fumigatus HZ1具有较强的纤维素降解潜力。
表 3. A. fumigatus HZ1基因组中纤维素酶基因 Table 3. Cellulase genes in the genome of A. fumigatus HZ1
| Classification | Gene ID | CAZy modules | E-value | Classification | Gene ID | CAZy modules | E-value | |
| Endoglucanase | 1709_t | GH12 | 4.90E-51 | β-glucosidase | 370_t | GH1 | 5.40E-100 | |
| 2927_t | GH12 | 9.20E-27 | 5677_t | GH1 | 1.80E-159 | |||
| 3476_t | GH12 | 2.10E-45 | 5823_t | GH1 | 6.10E-165 | |||
| 8667_t | GH12 | 6.50E-25 | 7227_t | GH1 | 2.00E-155 | |||
| 8722_t | GH5 | 6.70E-219 | 8889_t | GH1 | 5.80E-154 | |||
| 8272_t | GH5 | 5.50E-150 | 1708_t | GH3 | 1.70E-61 | |||
| 1164_t | GH5 | 5.60E-159 | 1847_t | GH3 | 2.10E-62 | |||
| 1657_t | GH5 | 2.60E-131 | 1974_t | GH3 | 2.50E-64 | |||
| 6881_t | GH5 | 2.20E-27 | 1983_t | GH3 | 1.50E-59 | |||
| 4314_t | GH5 | 6.40E-135 | 3016_t | GH3 | 3.20E-67 | |||
| 4194_t | GH5 | 2.70E-68 | 33_t | GH3 | 8.20E-63 | |||
| 5332_t | GH5 | 1.80E-113 | 3575_t | GH3 | 3.70E-56 | |||
| 6578_t | GH5 | 2.30E-116 | 5056_t | GH3 | 3.00E-62 | |||
| 8258_t | GH5 | 6.90E-116 | 5570_t | GH3 | 4.10E-61 | |||
| 376_t | GH5 | 1.20E-88 | 5621_t | GH3 | 9.60E-52 | |||
| 6830_t | GH5 | 3.70E-87 | 591_t | GH3 | 1.10E-61 | |||
| 3375_t | GH5 | 3.10E-112 | 602_t | GH3 | 1.60E-66 | |||
| 5108_t | GH5 | 5.10E-108 | 6911_t | GH3 | 3.30E-65 | |||
| 6294_t | GH51 | 2.70E-129 | 7041_t | GH3 | 4.50E-46 | |||
| 2998_t | GH6 | 2.30E-97 | 7148_t | GH3 | 6.60E-61 | |||
| 2180_t | GH7 | 1.30E-145 | 7750_t | GH3 | 5.00E-57 | |||
| 5291_t | GH7 | 4.20E-198 | 7969_t | GH3 | 3.00E-53 | |||
| 6577_t | GH7 | 2.10E-202 | 8902_t | GH3 | 5.70E-58 | |||
| 6782_t | GH7 | 8.10E-146 | Exoglucanase | 137_t | GH55 | 2.80E-233 | ||
| 4919_t | GH74 | 2.00E-25 | 1980_t | GH55 | 5.70E-278 | |||
| 2102_t | GH55 | 5.10E-284 | ||||||
| 5466_t | GH55 | 2.30E-105 | ||||||
| 5849_t | GH55 | 4.90E-183 | ||||||
| 8039_t | GH55 | 3.30E-218 | ||||||
| 8851_t | GH55 | 2.00E-223 |
表选项
2.5.2 半纤维素酶: A. fumigatus HZ1基因组中共有39个基因编码半纤维素酶,包括19个β-木糖苷酶基因、8个木聚糖酶基因及12个其他酶基因(甘露糖苷酶、α-L-阿拉伯呋喃糖苷酶和聚阿拉伯糖外切酶基因) (表 4)。β-木糖苷酶主要属于GH43家族;木聚糖酶属于GH10、GH11和GH30家族;甘露糖苷酶,α-L-阿拉伯呋喃糖苷酶和聚阿拉伯糖外切酶属于GH127、GH134、GH62、GH54、GH47、GH38和GH125家族(表 3)。
表 4. A. fumigatus HZ1基因组中半纤维素酶基因 Table 4. Hemicellulase genes in the genome of A. fumigatus HZ1
| Classification | Gene ID | CAZy modules | E-value | Classification | Gene ID | CAZy modules | E-value | |
| Beta-xylosidase | 7906_t | GH43 | 9.50E-163 | Xylanase | 1232_t | GH10 | 9.90E-87 | |
| 1705_t | GH43 | 3.10E-156 | 1965_t | GH10 | 2.30E-35 | |||
| 237_t | GH43 | 1.90E-125 | 2135_t | GH10 | 6.00E-103 | |||
| 2765_t | GH43 | 6.70E-133 | 4814_t | GH10 | 1.90E-107 | |||
| 8894_t | GH43 | 8.10E-129 | 11_t | GH11 | 1.70E-74 | |||
| 8060_t | GH43 | 1.30E-96 | 2002_t | GH11 | 2.00E-75 | |||
| 1197_t | GH43 | 1.90E-67 | 25_t | GH11 | 3.00E-78 | |||
| 2715_t | GH43 | 3.60E-87 | 384_t | GH30 | 9.70E-125 | |||
| 2978_t | GH43 | 5.10E-85 | Other enzymes | 6386_t | GH127 | 9.6E-170 | ||
| 5197_t | GH43 | 9.00E-12 | 264_t | GH134 | 5.4E-87 | |||
| 2424_t | GH43 | 5.60E-98 | 2542_t | GH62 | 7.8E-120 | |||
| 735_t | GH43 | 1.10E-96 | 8086_t | GH62 | 6E-118 | |||
| 7423_t | GH43 | 9.20E-85 | 6660_t | GH54 | 5.2E-162 | |||
| 8087_t | GH43 | 1.50E-134 | 2017_t | GH47 | 3.2E-176 | |||
| 1219_t | GH43 | 6.00E-127 | 4763_t | GH47 | 2E-175 | |||
| 1284_t | GH43 | 1.70E-129 | 5264_t | GH47 | 1E-166 | |||
| 1456_t | GH43 | 4.80E-116 | 5839_t | GH47 | 7.3E-136 | |||
| 5580_t | GH43 | 9.70E-107 | 6709_t | GH47 | 4.4E-144 | |||
| 8887_t | GH43 | 7.20E-129 | 8782_t | GH38 | 1.5E-86 | |||
| 2236_t | GH125 | 5.30E-164 |
表选项
2.5.3 木质素酶: A. fumigatus HZ1基因组中共有18个基因编码木质素酶,包括4个漆酶基因、3个锰过氧化物酶基因和11个纤维素二糖脱氢酶基因,其中漆酶基因编码的蛋白质属于AA1家族,锰过氧化物酶基因编码的蛋白质属于AA2家族,纤维素二糖脱氢酶基因编码的蛋白质属于AA3家族(表 5)。该结果表明A. fumigatus HZ1同时具有木质素降解能力。
表 5. A. fumigatus HZ1基因组中半木质素酶基因 Table 5. Ligninase genes in the genome of A. fumigatus HZ1
| Classification | Gene ID | CAZy modules | E-value |
| Laccase | 5161_t | AA1 | 1.1E-44 |
| 5162_t | AA1 | 4.1E-120 | |
| 5731_t | AA1 | 1.6E-38 | |
| 4662_t | AA1 | 7.1E-119 | |
| Manganese peroxidase | 2131_t | AA2 | 7.7E-60 |
| 2845_t | AA2 | 2.8E-13 | |
| 7286_t | AA2 | 4.4E-65 | |
| Cellobiose dehydrogenase | 5600_t | AA3 | 3.5E-52 |
| 2396_t | AA3 | 4.7E-179 | |
| 2415_t | AA3 | 2.8E-126 | |
| 2970_t | AA3 | 1.3E-156 | |
| 3830_t | AA3 | 4.9E-155 | |
| 4306_t | AA3 | 1E-96 | |
| 5515_t | AA3 | 3.1E-179 | |
| 6155_t | AA3 | 1.3E-170 | |
| 7966_t | AA3 | 1E-140 | |
| 8795_t | AA3 | 1.2E-175 | |
| 6411_t | AA3 | 6.3E-178 |
表选项
2.6 其他功能注释 耐药基因(CARD)数据库注释结果表明,A. fumigatus HZ1基因组中共有2个耐药基因(5567_t和5860_t) (表 6);真菌致病菌毒力因子(DFVF)注释结果表明,A. fumigatus HZ1基因组中共有1082个真菌致病菌毒力因子,其中动物宿主311个,草本宿主313个,植物宿主177个,脊椎动物宿主245个,木本宿主34个(表 6)。
表 6. A. fumigatus HZ1其他功能注释 Table 6. Other functional annotations of A. fumigatus HZ1 genome
| Functional annotation | Number |
| Resistance Type | |
| abcA | 2 |
| Disease-Hostkey | |
| Animals | 311 |
| Herb | 313 |
| Plants | 177 |
| Vertebrata | 245 |
| Xyloid | 34 |
表选项
3 讨论 微生物降解纤维素是一种绿色、经济的方式。通过基因组测序及分析,可以阐明微生物降解纤维素的潜力。Aspergillus fumigatus作为一类具有纤维素降解能力的菌,其基因组具有丰富的CAZymes基因,可分泌多种多样的木质纤维降解酶[28]。对A. fumigatus菌株基因组进行深入研究,将有利于A. fumigatus降解纤维素相关酶资源的挖掘与开发利用。
本研究从长足大竹象肠道内筛选出一株纤维素降解真菌Aspergillus fumigatus HZ1,其纤维素酶活在CMC培养基中培养6 d过程中呈上升趋势,在第6天时内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶酶活分别达到2.634±0.13 U/mL、1.247±0.033 U/mL和1.556±0.12 U/mL,与已报道的烟曲霉菌株酶活相似[4-6]。A. fumigatus HZ1基因组大小为28788216 bp,GC含量为49.43%,与A. fumigatus Z5基因组相似[28]。
A. fumigatus HZ1基因组注释到534个CAZyme基因,包括270个GH基因,96个GT基因,80个CE基因,15个PL基因,66个AA基因和7个CBM基因。在103个可公开获得的真菌基因组中,Aspergillus属在CAZyme基因数量上居于领先地位[29]。Miao等[28]通过比较基因组学对4株A. fumigatus CAZyme进行比较,包括A. fumigatus Z5、A. fumigatus AF293、A. fumigatus var. RP-2014和A. fumigatus A1163,揭示CAZyme基因在A. fumigatus菌株中的共性。研究表明,CAZyme六大分类中,A. fumigatus基因组内GH基因数量和比例最大[28],与本研究结果一致。
纤维素降解需要内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶。Miao等[28]挖掘了A. fumigatus Z5基因组中纤维素酶基因,包括25个内切葡聚糖酶基因、3个外切葡聚糖酶基因和至少15个β-葡萄糖苷酶基因。本研究中,A. fumigatus HZ1基因组中也包含丰富的纤维素酶基因,包括25个内切葡聚糖酶基因、7个外切葡聚糖酶基因和23个β-葡萄糖苷酶基因,表明其巨大的纤维素降解潜力。
本研究首次从长足大竹象肠道内筛选出烟曲霉,解析了A. fumigatus HZ1基因组,探讨了其纤维素降解的遗传基础,并通过酶活验证了其纤维素降解潜力,为该菌的实际应用提供了理论基础和新的微生物资源。
References
| [1] | Wang XD, Yao CH, Wang F, Li ZD. Cellulose-based nanomaterials for energy applications. Small, 2017, 13(42): 1702240. DOI:10.1002/smll.201702240 |
| [2] | Aro N, Pakula T, Penttil? M. Transcriptional regulation of plant cell wall degradation by filamentous fungi. FEMS Microbiology Reviews, 2005, 29(4): 719-739. DOI:10.1016/j.femsre.2004.11.006 |
| [3] | Bansal N, Tewari R, Soni R, Soni SK. Production of cellulases from Aspergillus niger NS-2 in solid state fermentation on agricultural and kitchen waste residues. Waste Management, 2012, 32(7): 1341-1346. DOI:10.1016/j.wasman.2012.03.006 |
| [4] | Liu DY, Zhang RF, Yang XM, Wu HS, Xu DB, Tang Z, Shen QR. Thermostable cellulase production of Aspergillus fumigatus Z5 under solid-state fermentation and its application in degradation of agricultural wastes. International Biodeterioration and Biodegradation, 2011, 65(5): 717-725. DOI:10.1016/j.ibiod.2011.04.005 |
| [5] | Sharma M, Soni R, Nazir A, Oberoi HS, Chadha BS. Evaluation of glycosyl hydrolases in the secretome of Aspergillus fumigatus and saccharification of alkali-treated rice straw. Applied Biochemistry and Biotechnology, 2011, 163(5): 577-591. DOI:10.1007/s12010-010-9064-3 |
| [6] | Srivastava N, Rawat R, Sharma R, Oberoi HS, Srivastava M, Singh J. Effect of nickel-cobaltite nanoparticles on production and thermostability of cellulases from newly isolated thermotolerant Aspergillus fumigatus NS (Class: Eurotiomycetes). Applied Biochemistry and Biotechnology, 2014, 174(3): 1092-1103. DOI:10.1007/s12010-014-0940-0 |
| [7] | Luo CB, Li YQ, Liao H, Yang YJ. De novo transcriptome assembly of the bamboo snout beetle Cyrtotrachelus buqueti reveals ability to degrade lignocellulose of bamboo feedstock. Biotechnology for Biofuels, 2018, 11: 292. DOI:10.1186/s13068-018-1291-9 |
| [8] | Luo CB, Li YQ, Chen Y, Fu C, Nong X, Yang YJ. Degradation of bamboo lignocellulose by bamboo snout beetle Cyrtotrachelus buqueti in vivo and vitro: efficiency and mechanism. Biotechnology for Biofuels, 2019, 12: 75. DOI:10.1186/s13068-019-1406-y |
| [9] | Luo CB, Li YQ, Chen Y, Fu C, Long WC, Xiao XM, Liao H, Yang YJ. Bamboo lignocellulose degradation by gut symbiotic microbiota of the bamboo snout beetle Cyrtotrachelus buqueti. Biotechnology for Biofuels, 2019, 12: 70. DOI:10.1186/s13068-019-1411-1 |
| [10] | Li YQ, Lei L, Zheng L, Xiao XM, Tang H, Luo CB. Genome sequencing of gut symbiotic Bacillus velezensis LC1 for bioethanol production from bamboo shoots. Biotechnology for Biofuels, 2020, 13: 34. DOI:10.1186/s13068-020-1671-9 |
| [11] | Lin T, Zhu GT, Zhang JH, Xu XY, Yu QH, Zheng Z, Zhang ZH, Lun YY, Li S, Wang XX, Huang ZJ, Li JM, Zhang CZ, Wang TT, Zhang YY, Wang AX, Zhang YC, Lin K, Li CY, Xiong GS, Xue YB, Mazzucato A, Causse M, Fei ZJ, Giovannoni JJ, Chetelat RT, Zamir D, St?dler T, Li JF, Ye ZB, Du YC, Huang SW. Genomic analyses provide insights into the history of tomato breeding. Nature Genetics, 2014, 46(11): 1220-1226. DOI:10.1038/ng.3117 |
| [12] | Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [13] | Li RQ, Zhu HM, Ruan J, Qian WB, Fang XD, Shi ZB, Li YR, Li ST, Shan G, Kristiansen K, Li SG, Yang HM, Wang J, Wang J. De novo assembly of human genomes with massively parallel short read sequencing. Genome Research, 2010, 20(2): 265-272. DOI:10.1101/gr.097261.109 |
| [14] | Lomsadze A, Ter-Hovhannisyan V, Chernoff YO, Borodovsky M. Gene identification in novel eukaryotic genomes by self-training algorithm. Nucleic Acids Research, 2005, 33(20): 6494-6506. DOI:10.1093/nar/gki937 |
| [15] | Lowe TM, Eddy SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Research, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [16] | Lagesen K, Hallin P, R?dland EA, St?rfeldt HH, Rognes T, Ussery DW. RNAmmer: consistent and rapid annotation of ribosomal RNA genes. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [17] | Griffiths-Jones S, Bateman A, Marshall M, Khanna A, Eddy SR. Rfam: an RNA family database. Nucleic Acids Research, 2003, 31(1): 439-441. DOI:10.1093/nar/gkg006 |
| [18] | Tarailo-Graovac M, Chen NS. Using RepeatMasker to identify repetitive elements in genomic sequences. Current Protocols in Bioinformatics, 2009, 25(1): 4.10.1-4.10.14. |
| [19] | Tatusov RL, Natale DA, Garkavtsev IV, Tatusova TA, Shankavaram UT, Rao BS, Kiryutin B, Galperin MY, Fedorova ND, Koonin EV. The COG database: new developments in phylogenetic classification of proteins from complete genomes. Nucleic Acids Research, 2001, 29(1): 22-28. DOI:10.1093/nar/29.1.22 |
| [20] | Conesa A, G?tz S, García-Gómez JM, Terol J, Talón M, Robles M. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics, 2005, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
| [21] | Boeckmann B, Bairoch A, Apweiler R, Blatter MC, Estreicher A, Gasteiger E, Martin MJ, Michoud K, O'Donovan C, Phan I, Pilbout S, Schneider M. The SWISS-PROT protein knowledgebase and its supplement TrEMBL in 2003. Nucleic Acids Research, 2003, 31(1): 365-370. DOI:10.1093/nar/gkg095 |
| [22] | Huerta-Cepas J, Szklarczyk D, Forslund K, Cook H, Heller D, Walter MC, Rattei T, Mende DR, Sunagawa S, Kuhn M, Jensen LJ, von Mering C, Bork P. eggNOG 4.5:a hierarchical orthology framework with improved functional annotations for eukaryotic, prokaryotic and viral sequences. Nucleic Acids Research, 2016, 44(D1): D286-D293. DOI:10.1093/nar/gkv1248 |
| [23] | Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and genomes. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27 |
| [24] | Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer EL, Tate J, Punta M. Pfam: the protein families database. Nucleic Acids Research, 2014, 42(D1): D222-D230. DOI:10.1093/nar/gkt1223 |
| [25] | Buchfink B, Xie C, Huson DH. Fast and sensitive protein alignment using DIAMOND. Nature Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [26] | Eddy SR. A new generation of homology search tools based on probabilistic inference. Genome Informatics, 2009, 23(1): 205-211. |
| [27] | Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [28] | Miao YZ, Liu DY, Li GQ, Li P, Xu YC, Shen QR, Zhang RF. Genome-wide transcriptomic analysis of a superior biomass-degrading strain of A. fumigatus revealed active lignocellulose-degrading genes. BMC Genomics, 2016, 16(1): 459. |
| [29] | Zhao ZT, Liu HQ, Wang CF, Xu JR. Comparative analysis of fungal genomes reveals different plant cell wall degrading capacity in fungi. BMC Genomics, 2013, 14: 274. DOI:10.1186/1471-2164-14-274 |
