刘宇1#, 李永霞2#, 童胜2, 王俊尧2, 朱生安2, 张乐源1, 范艳华2


1. 家蚕基因组生物学国家重点实验室, 农业农村部蚕桑生物学与遗传育种重点实验室, 西南大学蚕桑纺织与生物质科学学院, 重庆 400715;
2. 西南大学生物技术中心, 重庆 400715
收稿日期:2020-09-23;修回日期:2021-01-07;网络出版日期:2021-01-13
基金项目:重庆市自然科学基金(cstc2019jcyj-msxmX0388);国家级大学生创新创业训练计划(202010635054)
*通信作者:范艳华, Tel: +86-23-68250042;Fax: +86-23-68251883;E-mail: fyh@swu.edu.cn.
#并列第一作者。
摘要:[目的] 探究参与硝酸盐同化的一个特异转录因子NirA1对球孢白僵菌(Beauveria bassiana)生长、抗逆及毒力的影响。[方法] 利用RT-PCR检测球孢白僵菌NirA1基因在不同培养基中的表达特征。通过构建敲除突变株ΔNirA1、超量表达OENirA1及回复互补菌株ComNirA1,解析NirA1在球孢白僵菌生长发育、逆境胁迫反应和致病昆虫中的功能。[结果] NirA1基因在营养较贫瘠的CZB培养基中表达量高于丰富培养基PDB和SDB。敲除NirA1导致了菌株在人工培养基上的生长缓慢,对NaNO2和Urea的利用效率降低。RT-PCR显示,ΔNirA1中硝酸盐转运蛋白基因NrtA的表达较野生型显著下调。在CZA、PDA和1/4 SDAY培养基中,其分生孢子产量分别较野生型降低了21.6%、16.2%和25.6%。逆境胁迫分析发现,在高温(32℃),高渗(1 mol/L NaCl)和H2O2处理后,ΔNirA1菌落生长抑制率较野生型分别降低29.0%、25.2%及49.0%;但在添加SDS和刚果红(CR)的处理中,突变体菌落的生长抑制率分别较野生型升高34.1%和96.2%。因此,缺失NirA后,菌株对SDS和CR更加敏感,但对高温、H2O2和NaCl的耐受性提高。以3龄的大蜡螟为试虫的毒力测定表明,敲除NirA1基因导致菌株的毒力提高,半致死时间较野生型提前17.4%。[结论] NirA1在球孢白僵菌生长过程中参与了氮源的利用,并在球孢白僵菌的菌落生长、分生孢子生产、胁迫反应和致病宿主过程中发挥了重要作用。
关键词:NirA1球孢白僵菌表达特征抗逆性毒力
Effects of NirA1 gene on growth, stress resistance and virulence of Beauveria bassiana
Yu Liu1#, Yongxia Li2#, Sheng Tong2, Junyao Wang2, Shengan Zhu2, Leyuan Zhang1, Yanhua Fan2


1. State Key Laboratory of Silkworm Genome Biology, Key Laboratory of Sericultural Biology and Genetic Breeding, Ministry of Agriculture and Rural Affairs, College of Sericulture, Textile and Biomass Sciences, Southwest University, Chongqing 400715, China;
2. Biotechnology Research Center, Southwest University, Chongqing 400715, China
Received: 23 September 2020; Revised: 7 January 2021; Published online: 13 January 2021
*Corresponding author: Yanhua Fan, Tel: +86-23-68250042; Fax: +86-23-68251883; E-mail: fyh@swu.edu.cn.
Foundation item: Supported by the Natural Science Foundation of Chongqing, China (cstc2019jcyj-msxmX0388) and by the National Undergraduate Innovation and Entrepreneurship Training Program (202010635054)
#Those authors contributed equally to this work.
Abstract: [Objective] We investigated the effects of the nitrate-specific activator NirA1 on the growth and stress resistance of Beauveria bassiana. [Methods] The expression of NirA1 was detected by RT-PCR in different culture mediums. Several NirA1 strains, including gene knockout (ΔNirA1), complementary (ComNirA1), and over-expression (OENirA1), were constructed and the functions of NirA1 involved in fungal developmental differentiation, stress reactions and virulence were analyzed. [Results] Expression level of NirA1 was higher in CZB medium than that in PDB or SDB. The growth of ΔNirA1 in mediums with different nitrogen sources was significantly slower compared to wild type. The expression of nitrate transporter NrtA was down-regulated in NirA1 deletion mutant. Compared to wild type, conidial yield in CZA, PDA and 1/4 DAY mediums decreased by 21.6%, 16.2% and 25.6% respectively. The growth inhibition rate of ΔNirA1 in 32 ℃, 1 mol/L NaCl and H2O2mediums was reduced by 29.0%, 25.2% and 49.0%, but increased by 34.1% and 96.2% in SDS and Congo red mediums respectively. Bioassay of fungal strains were performed using 3rd-instar larvae of G. mellonella. Compared to wild type, deletion of NirA1 increased fungal virulence, decreasing mean lethal time (LT50) by 17.4%. [Conclusion] These results indicated NirA1 is involved in the utilization of nitrogen sources and play crucial roles in colony growth, conidiation, stress responses, and pathogenesis of B. bassiana.
Keywords: NirA1Beauveria bassianaexpression profilestress resistancevirulence
氮是几乎所有生物大分子的重要组成成分。对真菌而言,碳、氮吸收对其生长、发育或致病是必需的。真菌能利用不同成分作为氮源,这要求在需要的时候表达不同途径的代谢酶[1-2]。目前,氮代谢和调节已在酿酒酵母、构巢曲霉及粗糙脉孢菌等真菌中被广泛研究[3]。当存在初级氮源(氨、谷氨酰胺、谷氨酸)时,真菌利用次级氮源相关基因的表达就会被抑制;但在缺少初级氮源时,真菌可以表达次级氮源相应基因,利用其他含氮化合物,如硝酸盐或亚硝酸盐等。研究发现,在曲霉中硝酸盐代谢基因的激活受到转录因子AreA和NirA的调控[3]。AreA是解除氮代谢物抑制的系统正向调节基因,NirA为硝酸盐代谢途径特异的转录因子,能被NO3– (或NO2–)诱导活化[4]。NirA属于GAL4转录因子家族,含有一个Cys6/Zn2型的双核锌簇[5]。这类锌指蛋白在真菌中常参与调控生长发育及响应外界胁迫应答[6]。当真菌处于氮饥饿状态时,转录因子AreA与硝酸盐同化基因簇(亚硝酸还原酶niiA基因和硝酸还原酶niaD基因)双向启动子结合,促进NirA的DNA结合位点也被定位在该双向启动子序列中[7]。AreA和NirA共同启动硝酸盐代谢途径特异酶的表达,调控硝酸还原酶和亚硝酸还原酶的转录[8]。在构巢曲霉中缺失NirA或者NirA发生突变时,硝酸还原酶基因在铵态氮和硝态氮条件下不能正常表达,并导致菌株生长受到影响,同时可能出现非致病表型[9-10]。
球孢白僵菌(Beauveria bassiana)是一种广谱性的昆虫病原真菌。因为其致病力强且环境友好的特点,已经被广泛用于农、林及卫生害虫的生物防治,也常被用作研究昆虫与病原菌间相互关系的模式真菌[11-12]。除此之外,球孢白僵菌还能作为植物内生菌,拮抗植物病原菌的生长[13]。因此,对应不同的生活方式和环境条件,球孢白僵菌需要激活相应转录因子,调控相关基因表达,使其更好地充分利用环境中的营养成分(如碳、氮源)完成生长发育、致病昆虫及拮抗环境微生物的生长。Luo等研究了球孢白僵菌中的碳代谢抑制转录因子BbcreA的功能。敲除该基因,导致了菌株在丰富培养基及基本盐上生长缓慢(26 ℃),而且在32 ℃条件下进一步恶化。与野生型相比,BbcreA缺失菌株还出现产孢量下降、细胞裂解和毒力降低等表型[14]。但目前对球孢白僵菌氮代谢利用及调控基因的研究还较少。
在本研究中,我们对球孢白僵菌中的1个NirA同源基因进行了研究。通过构建敲除突变株ΔNirA1、超量表达OENirA1及回复互补菌株ComNirA1,比较了NirA1基因突变菌株与野生菌株在球孢白僵菌生长发育、分生孢子抗逆境胁迫及昆虫致病中的差异,为进一步研究NirA基因对昆虫病原真菌抵御外界逆境胁迫的分子机制提供一定的理论依据。
1 材料和方法 1.1 菌株和质粒 球孢白僵菌(B. bassiana) Bb0062 (CGMCC 7.34),由本实验室分离和保存,用于遗传转化的真菌亲本菌株;大肠杆菌(Escherichia coli) DH5α,购于TaKaRa公司。根癌农杆菌(Agrobacterium tumefaciens) AGL-1用于真菌遗传转化。PUC-sur-gpdA为真菌超量表达载体,pK2-PtrpC-sur-TtrpC为真菌敲除载体并保存于本实验室,PUC-bar为真菌回复载体。真菌毒力分析使用的试虫为大蜡螟(Galleria mellonella),购于河南科云生物。
1.2 球孢白僵菌NirA1基因与蛋白序列分析 采用MEGA 6软件进行NirA1蛋白与多种真菌的同源蛋白的系统进化分析。
1.3 超量表达载体、同源敲除载体及回复载体的构建
1.3.1 同源敲除载体的构建: 利用同源重组的方法,以氯嘧磺隆的抗性基因Sur表达元件替换NirA1基因中的部分序列。以野生型基因组为模板,分别扩增NirA1基因的上下游区域(LB/RB)。LB正向引物为5′-CCAAGCTTGGGTTGCATCAT GTATGTCTTC-3′ (P1-F),反向引物为5′-GCTCTA GAGCAATATCACCAACGGCAGTGTCAA-3′ (P1-R),扩增片段含EcoR I和Spe I酶切位点;RB正向引物为5′-GGACTAGTCCCGACTTTTGC TCTCCCTTTC-3′ (P2-F),反向引物为5′-CGGAAT TCCGTTGTCACTAGATGGCTGGCA-3′ (P2-R),扩增片段含Xba I和Hind III位点。测序验证正确后片段用T4 DNA连接酶连接到pK2-PtrpC-sur-TtrpC载体上,得到pK2-LB-PtrpC-sur-TtrpC-RB。
1.3.2 回复载体的构建: 以基因组为模板,利用正向引物5′-GTACTGAGAGTGCACCATATGAAGCGCTCAAGTCTCTCTGC-3′ (P3-F)和反向引物5′-CGGTATTTCACACCGCATATGAACCCATCTGCGCTTAAGAA-3′ (P3-R),扩增获得包括启动子(885 bp)、NirA1编码区(2411 bp)和终止子(84 bp)片段,连接到pUC-bar载体(Xba I线性化)。经大肠杆菌(DH5α)转化,筛选出符合目标大小的转化子,测序验证。
1.3.3 NirA1组成型表达载体的构建: 为构建NirA1组成型表达载体,利用正向引物5′-TTTAATCAATAACAATCTAGAATGTCTCCAGCTCGA GGCTC-3′ (P4-F)和反向引物5′-GAGGTAATCCTTCTTTCTAGATTAGTGAATATCAACATTGCC-3′(P4-R)从cDNA模板中扩增出NirA1片段,连接到pUC-sur-gpdA (Xba I线性化)上,经大肠杆菌转化及PCR扩增验证(引物pgpdA-F 5′-CCCACGACTACAAAACCATT-3′和NirA1-R 5′-TATCACCAACGGCAGTGTCA-3′)获得正确的质粒,并测序验证。将含有pUC-sur-pgpdA-NirA1质粒转入球孢白僵菌野生型中,在具有Sur的察氏液体培养基(Czapek-Dox Broth,CZB)上进行筛选。
1.4 NirA1表达模式分析 Real-time RT-PCR检测NirA1表达:分别接种球孢白僵菌野生型菌株于察氏液体培养基(Czapek-Dox Broth,CZB)、马铃薯葡萄糖液体培养基(potato dextrose broth,PDB)及沙氏葡萄糖液体培养基(sabouraud dextrose broth,SDB)中,利用RT-PCR检测基因的表达情况。提取经摇床培养(200 r/min,26 ℃) 3 d的菌丝总RNA,反转录获得cDNA,对不同转化子中的目标基因表达量进行定量分析。以球孢白僵菌actin为内参基因(正向引物5′-TCTCCCTCTACGCCTCTG-3′,反向引物5′-GATCTTCATCAGGTAGTCGGTA-3′),利用系统自带的Bio-Rad CFX manager 3.0软件进行相对表达量计算(ΔΔcq)。
1.5 NirA1对球孢白僵菌生长发育的影响 为了探究球孢白僵菌NirA1在氮源代谢中功能,将突变菌株接种到察氏固体培养基(Czapek-Dox agar,CZA)、不含NaNO3的CZA培养基(加入Tryptone、NaNO2、(NH4)2SO4、NH4NO3及Urea等氮源代替NaNO3)、营养丰富的1/2萨氏培养基(sabouraud dextrose agar with yeast extrac,SDAY)和马铃薯葡萄糖固体培养基(potato dextrose agar,PDA)中,26 ℃培养7 d。以野生型球孢白僵菌为对照,观察各突变株在不同氮源中的生长发育,分析同源敲除突变株(ΔNirA1)在基础培养基CZA中下游硝酸盐还原酶及转运蛋白基因的表达。同时观察不同菌株在不同胁迫条件下的生长差异及生长抑制率,并分析了各突变株中H2O2抗氧化胁迫相关基因(Bbcyb、Bbgas、Bbmsn、Bbpacc和Bbslt2)及刚果红胁迫相关基因(SOD和CAT)的表达。不同胁迫条件包含:高渗(0.8 mol/L NaCl和0.02% SDS)、高温(32 ℃)、25 μg/mL刚果红和3 mmol/L氧化剂H2O2。
1.6 NirA1对球孢白僵菌分生孢子的影响
1.6.1 球孢白僵菌分生孢子表位碳源的染色观察: 为分析NirA1敲除对分生孢子表位碳源的影响,试验中使用了以下几种凝集素:植物凝集素PNA (结合末端β-半乳糖)、刀豆素A ConA (结合碳源α-吡喃甘露糖和α-吡喃葡萄糖残基)、麦胚素WGA (结合碳源N-乙酰氨基葡萄糖和N-乙酰神经氨糖酸残基)。染色参照Wanchoo等的方法[15],孢子为菌株在固体平板PDA上生长14 d后所得。
1.6.2 球孢白僵菌分生孢子产量及孢子萌发测定: 为分析NirA1对球孢白僵菌分生孢子产量的影响,测定了CZA、PDA和1/4SDAY培养基中各菌株分生孢子的产量。利用0.05% Tween-80制备真菌孢悬液(1×107 conidia/mL)备用。准备3种不同的固体培养基CZA、PDA、1/4 SDAY (50 mL),在高温灭菌后温度降至50 ℃左右时,加入100 μL上述孢悬液,混匀后倒入90 mm的培养皿中(各3个)。将平板在26 ℃培养14 d后,用打孔器(直径1 cm)在每个平板取3个样,在3 mL Tween-80 (0.05%)中充分涡旋,统计并计算分生孢子产量。另外,为分析NirA1敲除对孢子的影响,将孢悬液稀释至3×107 conidia/mL,取100 μL孢悬液涂布于CZA培养基中,26 ℃培养8 h。用手术刀每隔1 h切取1 cm左右的培养基,滴加乳酸酚棉蓝染色液进行染色后,在显微镜下观察孢子萌发。
1.7 NirA1对球孢白僵菌毒力的影响 以大蜡螟为试虫分析球孢白僵菌的毒力。以0.05% (V/V)的Tween-80收集在PDA培养基上培养14 d后的孢子,并将浓度调整为1×107 conidia/mL。将大蜡螟(35只/每组)在20 mL孢悬液中浸泡15 s后取出,置于150 mm培养皿中,加棉球保持湿度。0.05% Tween-80处理的大蜡螟为对照,每个菌株设置3组生物学重复。接种后的大蜡螟置于26 ℃的黑暗条件下,每天统计死亡数量。
2 结果和分析 2.1 NirA1基因与蛋白序列分析 NirA1开放阅读框全长2013 bp,编码1个含670个氨基酸的蛋白质(EJP68720.1)。在NCBI中,NirA1蛋白的功能注释为氮源吸收转录因子,在其N端有一Cys6/Zn2结构域(图 1-A)。通过NirA1蛋白与多种真菌同源蛋白的进化树分析,发现NirA1与虫草属同源蛋白的亲缘关系较近,而与尖孢镰刀菌亲缘关系较远(图 1-B)。
 |
| 图 1 NirA1蛋白序列分析及与其他真菌同源蛋白的系统进化树分析 Figure 1 Domain structures and phylogenetic analysis of NirA1. A: conserved domains in NirA1. B: a phylogenetic tree was constructed by MEGA 6. NirA1 was indicated by red square. |
| 图选项 |
2.2 同源重组、回复互补和超量表达突变株的筛选及分子验证 同源重组示意图如图 2-A。通过根癌农杆菌介导的遗传方法,获得了NirA1敲除突变体。由于在敲除突变体中,NirA1基因中的440 bp片段被3600 bp的Sur表达元件所替换,利用左右臂上的上下游引物扩增,在不同菌株中得到不同大小的片段。野生型能扩增获到997 bp大小的片段,突变体中片段大小为4600 bp,回复互补菌株中能同时获得两个片段(图 2-B)。Real-time PCR (RT-PCR)结果进一步表明突变体中的NirA1基因已被敲除(图 2-C)。以pgpdA-F/NirA1-R为引物,利用PCR验证筛选NirA1超量转化子。RT-PCR分析超量表达菌株中的NirA1表达水平,选择表达量最高的4号转化子,进行后续实验(图 2-D)。
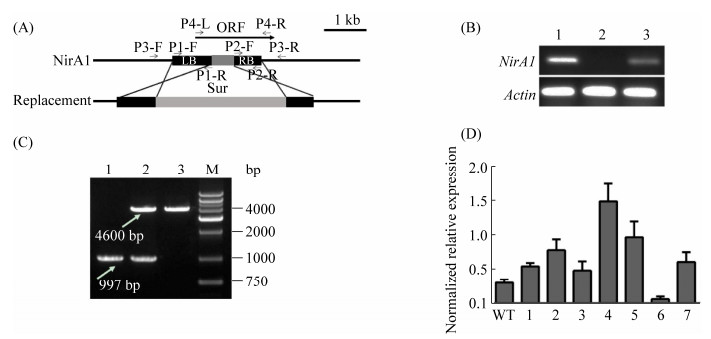 |
| 图 2 载体构建和突变体的筛选 Figure 2 Vector construction and mutant screening. A: gene replacement strategy of NirA1 in B. bassiana. Chlorimuron-methyl resistance gene Sur was used to replace the deletion region of NirA1 gene. B: NirA1 transformants screening. Lanes 1, 2 and 3 indicate B. bassiana wild type (WT), ComNirA1 and ΔNirA1, respectively. M is DNA marker 2000. C: RT-PCR analysis of NirA1 strains. Lanes 1, 2, and 3 indicate wild type, ΔNirA1 and ComNirA1 strains, respectively. Actin is the reference gene of B. bassiana. D: RT-PCR validation of OENirA1. WT is wild type B. bassiana. 1–7: seven NirA1 overexpression strains. Error bars: SD of the mean from three replicate assays. |
| 图选项 |
2.3 NirA1表达模式分析 表达结果如图 3所示。NirA1基因在CZB、PDB及SDB培养基中均有不同水平的表达。其中,表达水平最高的为营养较贫瘠的CZB,而在营养较丰富的PDB和SDB中表达量较低。
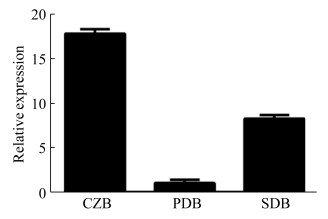 |
| 图 3 NirA1表达模式分析 Figure 3 Expression analysis of NirA1. RT-PCR analysis of the expression of the NirA1 gene in different mediums. B. bassiana wild type was inoculated into CZB, PDB and SDB liquid media and cultured at 26 ℃ for 3 d. Total RNA was extracted and RT-PCR was performed as described in materials and methods. Error bars: SD of the mean from three replicate assays. |
| 图选项 |
2.4 NirA1对球孢白僵菌生长发育的影响
2.4.1 NirA1影响球孢白僵菌在不同氮源上的生长及氮源利用相关基因的表达: 如图 4-A所示,ΔNirA1菌株在单一氮源NaNO2和Urea培养基中的生长明显较野生型缓慢,OENirA1和ComNirA1的菌落表型与野生型相比没有明显差异。基因表达分析发现,敲除NirA1明显下调了硝酸盐转运蛋白基因NrtA的表达量,但对亚硝酸还原酶(NiiA)和硝酸还原酶(NiaD)基因的影响较低(图 4-B)。
 |
| 图 4 NirA1对球孢白僵菌在不同氮源上生长的影响及氮源利用相关基因表达 Figure 4 Effects of NirA1 on B. bassiana growth on mediums contained different nitrogen sources and the expression of nitrate assimilation genes. A: plates with fungi were cultured at 26 ℃ for 7 days. B: RT-PCR analyses of NiiA, NiaD and NrtA. Error bars: SD of the mean from three replicate assays. |
| 图选项 |
2.4.2 NirA1对球孢白僵菌逆境胁迫反应的影响: 为了探究NirA1是否参与到球孢白僵菌对外界环境胁迫应答反应,分析了ΔNirA1在不同胁迫条件下的生长情况。在NaCl、SDS和刚果红(CR)的胁迫条件下,ΔNirA1株菌落生长均较野生型缓慢。其中,在刚果红存在时,ΔNirA1菌落生长受到明显抑制,菌落边缘菌丝较野生型致密。在32 ℃、H2O2和NaCl处理条件下,ΔNirA1生长抑制率较野生型分别降低29.0%、25.2%和49.0% (图 5-A);但在SDS和CR培养基中,ΔNirA1菌落的生长抑制率分别较野生型升高34.1%和96.2% (图 5-B)。超量表达菌株OENirA1和互补菌株ComNirA1在不同胁迫条件下菌落生长与野生型没有明显的差异。以上结果表明,球孢白僵菌中NirA1的缺失导致了菌株对SDS和CR的更加敏感,但同时提高了对高温、H2O2和NaCl的耐受性。
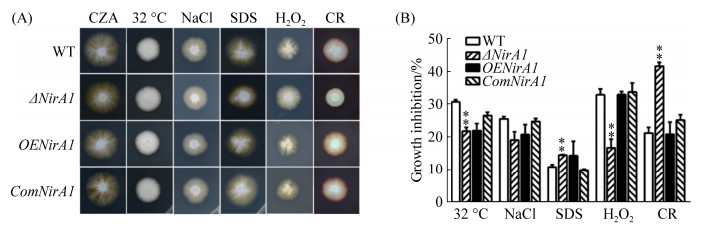 |
| 图 5 NirA1缺失对菌株抗逆能力的影响 Figure 5 Effects of NirA1 deletion on fungal stress tolerance. A: colony growth of B. bassiana on CZA mediums contained NaCl (0.8 mol/L), SDS (0.02%), H2O2 (3 mmol/L), Congo Red (CR, 25 μg/mL), respectively. Plates were incubated at 26 ℃ for 7 days. B: calculated growth inhibition rates of ΔNirA1. Error bars: SD of the mean from three replicate assays. Two asterisks indicates 0.01 < P < 0.05. |
| 图选项 |
2.4.3 逆境胁迫相关响应基因表达分析: 为应对外界的氧化胁迫,机体会产生抗氧化酶清除活性氧(ROS)。为分析NirA1对球孢白僵菌抗氧化胁迫能力的影响,检测了突变株中相关超氧化物歧化酶SOD及过氧化氢酶CAT相关基因的表达。未用H2O2处理时,ΔNirA1中除sod1、sod2和catC外,其余表达量均高于野生型。H2O2胁迫后,SOD家族大部分基因在野生型和ΔNirA1中均发生上调,其中sod3基因的变化最明显(在ΔNirA1中是未处理的5.4倍)。野生型和ΔNirA1中,catA和catP表达上调,但是NirA1缺失突变体中上调幅度明显低于野生型(图 6-A)。
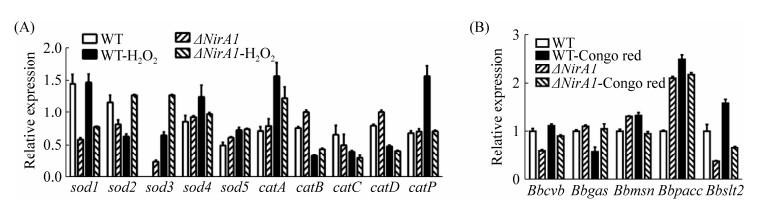 |
| 图 6 逆境胁迫相关响应基因表达分析 Figure 6 Expression analysis of stress response genes. A: effects of NirA1 deletion on expression of SOD and CAT family genes. Each strain was inoculated into 1/4 SDB medium with or without H2O2 (3 mmol/L) and incubated at 26 ℃ for 3 days. B: expression analysis of genes involved in cell wall integrity in the ΔNirA1 mutant strain. All strains were inoculated into 1/4 SDB medium with or without Congo red (25 μg/mL) and incubated at 26 ℃ for 3 days. Total RNA was extracted and used for real-time PCR analysis. Error bars: SD of the mean from three replicate assays. |
| 图选项 |
在刚果红胁迫时,敲除NirA1影响了菌株的生长。利用RT-PCR检测了NirA1敲除对逆境相应相关基因的表达。未添加刚果红时,ΔNirA1中Bbgas、Bbmsn和Bbpacc表达量均高于野生型,其中Bbpacc表达量是野生型的2.1倍。在刚果红胁迫时,野生型Bbgas表达量下降了42%,Bbpacc表达量是未处理的2.5倍。ΔNirA1与未处理差异不大,但是Bbmsn表达量的下调与野生型表达量上升却正好相反。无论刚果红存在与否,Bbcyb和Bbslt2在ΔNirA1中的表达量均低于野生型;但用刚果红处理后,ΔNirA1中Bbcyb和Bbslt2的表达量较未处理分别上升了1.5倍和1.8倍,明显高于野生型的上升幅度(图 6-B)。
2.5 NirA1对球孢白僵菌胞壁成分、产孢量及孢子萌发的影响 分生孢子表位碳源染色结果显示(图 7-A)。与野生型相比,ΔNirA1菌株分生孢子上的植物凝集素(PNA)和麦胚凝集素(WGA)荧光信号较弱,推测可能是NirA1缺失影响了分生孢子的表位碳源,如β-半乳糖、N-乙酰氨基葡萄糖和N-乙酰神经氨糖酸残基。分生孢子产量分析显示,在CZA、PDA和1/4 SDAY培养基中,ΔNirA1的分生孢子产量分别较野生型降低21.6%、16.2%和25.6%。而OENirA1和ComNirA1的分生孢子产量与野生型相比没有明显的差异(图 7-B)。此外,敲除NirA导致了孢子萌发时间较野生型提前了1 h (10.3 h VS 11.2 h) (图 7-C)。
 |
| 图 7 NirA1对球孢白僵菌分生孢子胞壁成分、孢子产量及孢子萌发率的影响 Figure 7 Effecst of NirA1 on cell wall compositions of conidia, conidial yield and conidial germination rate. A: lectin binding profiles in B. bassiana conidia. B: analysis of conidial yield. C: conidial germination rate of ΔNirA1 on CZA. Error bars: SD of the mean from three replicate assays. |
| 图选项 |
2.6 菌株毒力分析 利用大蜡螟幼虫进行了真菌菌株的毒力分析(体表接种)。与野生型相比,敲除或超量表达NirA1均引起了菌株毒力提高,其中ΔNirA1菌株导致试虫的半致死时间较野生型提前了17.4% (P < 0.01) (图 8)。ComNirA1菌株毒力与野生型相比无明显变化。
 |
| 图 8 菌株毒力测定 Figure 8 Virulence analysis of fungal strains against G. mellonella larvae. Error bars=SD of the mean from three replicate assays. ** indicate 0.01 < P < 0.05. *** indicate P < 0.01. |
| 图选项 |
3 讨论 动植物病原真菌在腐生或侵染过程中,有效吸收和利用环境中的氮源至关重要。氮源的利用受到一系列转录调控因子的协同调控。在构巢曲霉中,硝酸盐的利用调控机制已被广泛研究[4-6]。这些硝酸盐代谢基因在优先氮源存在时被抑制,但当缺乏氮源时,在外源NO3– (或NO2–)的诱导下,通路特异性调控基因NirA同另一转录因子基因AreA一起,共同启动硝酸盐代谢途径特异酶的表达[7-8]。构巢曲霉已分离出大量NirA突变等位基因,大多数突变株被认为不能在硝酸盐或亚硝酸盐作为唯一氮源上进行生长,并被归类为不可诱导且失去功能的突变,但极少的突变却反映出即使在诱导剂选择性缺失时,也能合成硝酸盐和亚硝酸盐还原酶[16-17]。同时,NirA的一些突变导致氮代谢的抑制,即使在氨盐存在的情况下也会合成硝酸盐同化酶[4]。而NirA缺失或突变菌株则显示NO3–转运蛋白功能降低,硝酸还原酶活性降低,往往造成真菌在以硝酸盐或亚硝酸盐为单一氮源的培养基上生长缓慢,甚至导致非致病表型[9-10]。我们的研究结果发现,球孢白僵菌的NirA1在营养贫瘠的条件下高水平表达,敲除该基因导致菌株在不同的氮源培养基中的生长较野生型缓慢,对NaNO2和Urea的利用效率降低。RT-PCR分析发现,NirA1突变体中硝酸盐转运蛋白基因NrtA的表达下调,亚硝酸还原酶基因(NiiA)和硝酸还原酶基因(NiaD)的表达却没有下调。这与构巢曲霉中的NirA基因存在不同,构巢曲霉中的NirA缺失显著下调了NiiA和NiaD基因表达[7]。我们推测球孢白僵菌中的NirA1可能存在功能冗余现象,即可能存在另外调节NiiA、NiaD及NrtA表达的转录因子。所以尽管球孢白僵菌NirA1与模式真菌中的NirA存在同源性,且都影响氮源的利用,但二者可能在功能上存在差异。
真菌对逆境胁迫的耐受在其环境适应性中具有重要作用,在昆虫病原真菌中还对菌株的侵染致病能力有影响[18]。如同其他病原真菌一样,球孢白僵菌在不同的生态环境及侵染昆虫过程中需要利用不同的碳氮源、面临各种胁迫及宿主的免疫反应等。我们的研究发现,球孢白僵菌中NirA1缺失增加了真菌对H2O2的耐受性和对刚果红的敏感性。抗氧化酶(SOD和CAT)在真菌对氧化胁迫的响应中发挥重要作用[19]。RT-PCR分析发现,在氧化胁迫时,ΔNirA1中的sod2和sod3基因表达较野生型显著上调,这可能是导致NirA1突变体对双氧水耐受的原因。ΔNirA1在刚果红胁迫时,较野生型相比,逆境胁迫相关响应基因Bbgas表达量显著上调,Bbmsn和Bbpacc表达量显著下调则可能是增加球孢白僵菌敏感性的因素。此外,NirA1也影响了孢子发育。敲除NirA1后,孢子细胞壁上的N-乙酰氨基葡萄糖和N-乙酰神经氨糖酸成分(lectin染色结果所示)减少、孢子萌发速度加快,但突变体的产孢量却较野生型降低。推测由于NirA1的缺失导致真菌对营养成分的利用降低(硝酸盐转运蛋白表达量下降所致),因此影响了真菌孢子的发育,导致了产孢量下降及孢子细胞壁成分的改变。
其他真菌的毒力实验大都显示敲除NirA1基因后毒力出现降低的现象。如植物病原炭疽菌中敲除NirA1基因(NirA的同源基因),突变体在含以硝酸盐或亚硝酸盐为单一氮源的培养基上不能生长,在草莓叶片上的毒力显著降低[20]。但我们以大蜡螟为试虫进行的毒力分析发现,敲除NirA1却导致菌株毒力的提高,推测可能是由于NirA1的敲除代偿性的促使了球孢白僵菌中其他营养代谢基因或毒力相关基因的表达增强。这也表明球孢白僵菌中NirA1的功能可能与其它真菌中的类似蛋白存在不同之处。关于超量表达引起菌株毒力的提高,我们推测球孢白僵菌中的NirA1可能存在多效性,过多的NirA1影响了侵染过程中致病相关基因的表达。但是相关的机制还不清楚,需要进一步探索其它相关氮源利用调节因子。
本研究通过表达特性分析与基因敲除技术证实NirA1参与调控球孢白僵菌氮源的利用,影响球孢白僵菌生长发育和逆境胁迫应答反应。研究结果为进一步明确NirA1在球孢白僵菌发育分化、逆境胁迫反应和毒力的生理功能奠定了基础。
References
| [1] | Wang CS, Wang SB. Insect pathogenic fungi: genomics, molecular interactions, and genetic improvements. Annual Review of Entomology, 2017, 62: 73-90. DOI:10.1146/annurev-ento-031616-035509 |
| [2] | Chen YX, Cen K, Lu YZ, Zhang SW, Shang YF, Wang CS. Nitrogen-starvation triggers cellular accumulation of triacylglycerol in Metarhizium robertsii. Fungal Biology, 2018, 122(6): 410-419. DOI:10.1016/j.funbio.2017.07.001 |
| [3] | Marzluf GA. Genetic regulation of nitrogen metabolism in the fungi. Microbiology and Molecular Biology Reviews, 1997, 61(1): 17-32. |
| [4] | Burger G, Tilburn J, Scazzocchio C. Molecular cloning and functional characterization of the pathway-specific regulatory gene nirA, which controls nitrate assimilation in Aspergillus nidulans. Molecular and Cellular Biology, 1991, 11(2): 795-802. |
| [5] | Burger G, Strauss J, Scazzocchio C, Lang BF. nirA, the pathway-specific regulatory gene of nitrate assimilation in Aspergillus nidulans, encodes a putative GAL4-type zinc finger protein and contains four introns in highly conserved regions. Molecular and Cellular Biology, 1991, 11(11): 5746-5755. |
| [6] | Imbeault M, Helleboid PY, Trono D. KRAB zinc-finger proteins contribute to the evolution of gene regulatory networks. Nature, 2017, 543(7646): 550-554. DOI:10.1038/nature21683 |
| [7] | Punt PJ, Strauss J, Smit R, Kinghorn JR, van den Hondel CA, Scazzocchio C. The intergenic region between the divergently transcribed niiA and niaD genes of Aspergillus nidulans contains multiple NirA binding sites which act bidirectionally. Molecular And Cellular Biology, 1995, 15(10): 5688-5699. DOI:10.1128/MCB.15.10.5688 |
| [8] | Berger H, Pachlinger R, Morozov I, Goller S, Narendja F, Caddick M, Strauss J. The GATA factor AreA regulates localization and in vivo binding site occupancy of the nitrate activator NirA. Molecular Microbiology, 2006, 59(2): 433-446. DOI:10.1111/j.1365-2958.2005.04957.x |
| [9] | Bernreiter A, Ramon A, Fernández-Martínez J, Berger H, Araújo-Bazan L, Espeso EA, Pachlinger R, Gallmetzer A, Anderl I, Scazzocchio C, Strauss J. Nuclear export of the transcription factor NirA is a regulatory checkpoint for nitrate induction in Aspergillus nidulans. Molecular and Cellular Biology, 2007, 27(3): 791-802. DOI:10.1128/MCB.00761-06 |
| [10] | Schinko T, Berger H, Lee W, Gallmetzer A, Pirker K, Pachlinger R, Buchner I, Reichenauer T, Güldener U, Strauss J. Transcriptome analysis of nitrate assimilation in Aspergillus nidulans reveals connections to nitric oxide metabolism. Molecular Microbiology, 2010, 78(3): 720-738. DOI:10.1111/j.1365-2958.2010.07363.x |
| [11] | Hajek AE, Delalibera I. Fungal pathogens as classical biological control agents against arthropods. BioControl, 2010, 55(1): 147-158. DOI:10.1007/s10526-009-9253-6 |
| [12] | Jackson MA, Dunlap CA, Jaronski ST. Ecological considerations in producing and formulating fungal entomopathogens for use in insect biocontrol. BioControl, 2010, 55(1): 129-145. DOI:10.1007/s10526-009-9240-y |
| [13] | Lane BS, Trinci APJ, Gillespie AT. Endogenous reserves and survival of blastospores of Beauveria hassiana harvested from carbon- and nitrogen-limited batch cultures. Mycological Research, 1991, 95(7): 821-828. DOI:10.1016/S0953-7562(09)80045-2 |
| [14] | Luo ZB, Qin YQ, Pei Y, Keyhani NO. Ablation of the creA regulator results in amino acid toxicity, temperature sensitivity, pleiotropic effects on cellular development and loss of virulence in the filamentous fungus Beauveria bassiana. Environmental Microbiology, 2014, 16(4): 1122-1136. DOI:10.1111/1462-2920.12352 |
| [15] | Wanchoo A, Lewis MW, Keyhani NO. Lectin mapping reveals stage-specific display of surface carbohydrates in vitro and haemolymph-derived cells of the entomopathogenic fungus Beauveria bassiana. Microbiology, 2009, 155(9): 3121-3133. DOI:10.1099/mic.0.029157-0 |
| [16] | Pateman JA, Cove DJ. Regulation of nitrate reduction in Aspergillus nidulans. Nature, 1967, 215(5107): 1234-1237. DOI:10.1038/2151234a0 |
| [17] | Berger H, Basheer A, B?ck S, Reyes-Dominguez Y, Dalik T, Altmann F, Strauss J. Dissecting individual steps of nitrogen transcription factor cooperation in the Aspergillus nidulans nitrate cluster. Molecular Microbiology, 2008, 69(6): 1385-1398. DOI:10.1111/j.1365-2958.2008.06359.x |
| [18] | Xie XQ, Li F, Ying SH, Feng MG. Additive contributions of two manganese-cored superoxide dismutases (MnSODs) to antioxidation, UV tolerance and virulence of Beauveria bassiana. PLoS One, 2012, 7(1): e30298. DOI:10.1371/journal.pone.0030298 |
| [19] | Morano KA, Grant CM, Moye-Rowley WS. The Response to heat shock and oxidative stress in Saccharomyces cerevisiae. Genetics, 2012, 190(4): 1157-1195. DOI:10.1534/genetics.111.128033 |
| [20] | Horowitz S, Freeman S, Zveibil A, Yarden O. A defect in nir1, a nirA-like transcription factor, confers morphological abnormalities and loss of pathogenicity in Colletotrichum acutatum. Molecular Plant Pathology, 2006, 7(5): 341-354. DOI:10.1111/j.1364-3703.2006.00341.x |
