杨丽娟2, 赖芬菊2, 谢宁清2, 戴益民1


1. 江西农业大学动物科学技术学院, 江西 南昌 330045;
2. 江西农业大学生物科学与工程学院, 江西 南昌 330045
收稿日期:2019-06-19;修回日期:2019-08-04;网络出版日期:2019-12-05
基金项目:国家自然科学基金(31360617,31802213);江西省自然科学基金(20161BAB204183);大学生创新创业训练计划(201810410018)
*通信作者:戴益民, Tel:+86-791-83813503;E-mail:daiym1125@jxau.edu.cn.
摘要:[目的] 为了给外源蛋白在酿酒酵母细胞中的定位提供参考,构建酿酒酵母荧光定位报告菌株。[方法] 运用染色体同源重组的方法,将突变的、已进行酵母表达优化的红色荧光蛋白RedStar分别整合到12个酵母细胞器标记蛋白的C端,与之进行融合表达,用特异性引物对每一个酵母荧光定位报告菌株进行PCR扩增和测序验证,用激光共聚焦显微镜进行荧光检测,对线粒体和细胞核进行特异性染料染色,用EGFP标记沙门氏菌已知定位蛋白SipA,与构建的相应荧光定位报告菌株进行共定位。[结果] 构建的酿酒酵母荧光定位报告菌株可分别标示酵母细胞的肌动蛋白、晚期胞内体、细胞核、核周质、纺锤体、线粒体、过氧化物酶体、脂滴、初级内吞体、次级内吞体、高尔基体顺面及高尔基体反面。PCR扩增及测序验证、荧光检测、染料与相应报告菌株的共定位、已知定位蛋白SipA与相应报告菌株的共定位均提示报告菌株构建成功。[结论] 这些报告菌株的构建,为日后在酵母中观察细胞器动态变化,以及未知蛋白在酵母中的定位提供了基础性工具。
关键词:蛋白定位RedStar酵母报告菌株共定位
Development of fluorescent localization reporter strains of the budding yeast
Yang Lijuan2, Lai Fenju2, Xie Ningqing2, Dai Yimin1


1. School of Animal Science and Technology, Jiangxi Agricultural University, Nanchang 330045, Jiangxi Province, China;
2. School of Biology Science and Engineering, Jiangxi Agricultural University, Nanchang 330045, Jiangxi Province, China
Received: 19 June 2019; Revised: 4 August 2019; Published online: 5 December 2019
*Corresponding author: Yimin Dai, Tel:+86-791-83813503;E-mail:daiym1125@jxau.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31360617, 31802213), by the Natural Science Foundation of Jiangxi Province (20161BAB204183) and by the National Undergraduate Innovation and Entrepreneurship Training Program (201810410018)
Abstract: [Objective] To facilitate the localization of proteins in budding yeast, we developed yeast reporter strains expressing fusion proteins that localize to specific subcellular structures. [Methods] The yeast expression vectors expressed organelle-specific proteins tagged with much brighter variant red fluorescent protein (RedStar) at their carboxy terminal were constructed and transferred into yeast cells to generate the reporter strain collection. To this end, the yeast codon that optimized coding sequence of RedStar were in-frame inserted immediately preceding the stop codon of each ORF that controlled by their endogenous promoters. Each strain was verified by PCR and sequencing, and then analyzed by fluorescence microscopy. Co-localization of DNA-binding dyes (DAPI), Mitotracker (mitochondria) and SipA from Salmonella enteritidis in the corresponding reporter strain were also performed. [Results] The yeast strain collection containing indicators for the actin, endoplasmic reticulum, nuclei, mitochondria, peroxisomes, lipids, endosomes, and the Golgi apparatus were constructed successfully. [Conclusion] Our studies provide a fundamental tool for observing the dynamic changes of organelles and identifying the localization of unknown proteins in yeast.
Keywords: subcellular localizationRedStaryeast reporter strainsco-localization
酿酒酵母(Saccharomyces cerevisiae)是第一个完成基因组测序的单细胞真核生物,遗传背景非常清楚[1-3]。酿酒酵母基因组数据库(Saccharomyces Genome Database,SGD)含有酵母基因组的详细注释,是研究真核细胞遗传学和生理学的重要工具。酿酒酵母具有生长迅速、易于遗传操作、含有稳定的单倍体和二倍体、易于突变、高效转化和同源重组率高等特点。酿酒酵母的基因结构、转录及转录后加工、翻译及修饰、细胞周期调控、细胞信号传导、环境胁迫反应、能量代谢等生物学进程,都比其他高等真核生物保守[4-5]。因此,酿酒酵母是研究真核生物细胞生化活动进程的经典模式生物,在人类癌症、衰老、凋亡、抗癌药物、抗菌药物、抗衰老药物的研发中都有广泛的应用。
酿酒酵母细胞内具有复杂的膜相结构,可将细胞区分为不同的隔室,执行特定的生物学功能,从而对细胞内各种途径进行严格的时空调控。解析外源目的基因在酿酒酵母中的定位、互作蛋白、靶标及生物学功能是一种常用的研究方法[6-8]。蛋白质必须到达特定的部位才能发挥其生物学功能,因此确定目的蛋白在细胞中的定位,对于揭示其功能、互作蛋白和解析其参与的生物学过程至关重要。
蛋白胞内定位研究的首要任务是给目的蛋白标记上一个便于检测的信号。目前,通过3种不同的标记方法:免疫荧光标记、小分子有机染料染色或荧光蛋白对目的蛋白进行标记。免疫荧光标记是将已知的抗原或抗体标记上荧光素,制成荧光标记物,再用荧光抗体作为分子探针检查酵母细胞内的目的蛋白,其局限在于它的作用对象只能是已经被固定的死细胞。小分子有机染料可以发出荧光,并且能与特定蛋白质或某种细胞器结合,进行活细胞特异性荧光染色。比较常用的如DAPI (4, 6-二脒基-2-苯基吲哚)可以标记DNA,Mito traker标记线粒体。利用荧光蛋白能稳定遗传且根据启动子特异性地表达这一优点,选择荧光蛋白对目的蛋白进行标记,通常是将目标蛋白和荧光蛋白(绿色荧光蛋白GFP及其衍生物、红色荧光蛋白RFP及其衍生物)标签融合[9-12],然后与各酵母荧光定位报告菌株进行共定位来间接确定目的蛋白的定位。因此,构建酿酒酵母定位报告菌株是进行外源蛋白定位的前提条件。
本试验利用染色体同源重组的方法,构建了12个组成型表达突变红色荧光RedStar的酵母细胞器荧光定位报告菌株[9],用特异性引物PCR扩增并测序进行逐一验证,并用激光共聚焦显微镜进行了各亚细胞器形态特征检测。另外,还对线粒体和细胞核进行了特异性染料染色,用EGFP标记沙门氏菌已知定位蛋白SipA,与相应报告定位菌株进行共定位,进一步证明了这些报告菌株构建成功,为日后在酵母中观察亚细胞水平细胞器动态变化,以及未知目标蛋白在酵母中的定位提供了基础性工具。
1 材料和方法 1.1 菌株和质粒 本实验所用菌株和质粒见表 1。
表 1. 本研究所用菌株及质粒 Table 1. Strains and plasmids used in this study
| Strains/Plasmids | Genotype/ Description | Source |
| W303-1A | MATa ade2-1 ura3-1 his3-11, 15 trp1-1 leu2-3, 112 can1-100 | Lab stock |
| Salmonella enterica | subsp. enterica serovar Agona str. SL483 | Gift from Dr. Jinghua Zhang, School of Animal Science and Technology |
| DH5α | F– Φ80lacZ?M15 ?(lacZYA-argF) U169 recA1 endA1 hsdR17 (rK–, mK+) phoA supE44 λ– thi-1 gyrA96 relA1 | Lab stock |
| pYM38 | RedStar, kanMX4 | Clontech |
| pEGFP | CMV, neomycin, kanamycin, EGFP | Clontech |
| pYES2-NTA | 2μ origin, GAL1 promoter, URA3, ampicillin | Invitrogen |
表选项
1.2 试剂 PrimeSTAR? HS DNA聚合酶、dNTP、限制性内切酶BamHⅠ、EcoRⅠ、XhoⅠ、T4 DNA连接酶、DNA分子量标准DL5000均购自TaKaRa公司;质粒DNA小提试剂盒、胶回收试剂盒购自北京全式金生物技术有限公司;Yeast DNA Kit购于广州飞扬生物工程有限公司;遗传霉素G418购自生工生物工程(上海)股份有限公司;抗荧光淬灭PVP封片液购于碧云天生物技术;DAPI (4′, 6-Diamidino-2-Phenylindole,Dilactate)、Mito TrakerTM Green FM购于赛默飞世尔科技有限公司。
1.3 酿酒酵母荧光标记同源重组引物的设计 本实验的12个细胞器荧光定位报告菌株引物见表 2。参照Janke等[13]方法,以带有标记基因(荧光标记:RedStar;抗生素标记:kanMX4)的质粒pYM38为PCR模板,设计与特定细胞器定位基因终止密码子上下游含有同源臂标记基因扩增引物。设计原则为上游引物5′带有目的基因终止密码子TGA、TAA或TAG (不含终止密码子)上游同源序列(44 bp),3′端为质粒pYM38上RedStar和kanMX4模块上游的部分序列(15 bp);下游引物5′端由目的基因终止密码子开始向后45 bp的反向互补序列(含终止密码子),3′端与模板pYM38上RedStar和kanMX4模块下游的部分序列反向互补(15 bp)。上下游5′端44 bp用于与特定基因编码框进行同源重组,3′端15 bp序列用于扩增RedStar和kanMX4模块。
表 2. 本研究所用引物 Table 2. Primers used in this study
| Primers | Sequences (5′→3′) | ||
| Sac6-F | GATTAATTATTACTTTTATCGCTTCGTTAATGACTTTGAACAAACGTACGCTGCAGGTC | ||
| Sac6-R | AAAGCTGAGTAGAAAACAGGTTACGAAAGTTGTTTGTTGGCTCAATCGATGAATTCGAG | ||
| Cop1-F Cop1-R | TTTCTAAGATCGGTGCACCTGCATCCGGATTAAGAATACGTGTACGTACGCTGCAGGTC GCAGAATACAATCCCGAGAAAATCAATAGGCAAAATAGCATTTAATCGATGAATTCGAG | ||
| Snf7-F Snf7-R | AAGATGAAAAAGCATTAAGAGAACTACAAGCAGAAATGGGGCTTCGTACGCTGCAGGTC CACCTTTTTTTTTTCTTTCATCTAAACCGCATAGAACACGTTCAATCGATGAATTCGAG | ||
| Sec13-F Sec13-R | AAAATCTTGAGGGTAAATGGGAACCCGCTGGTGAAGTTCATCAGCGTACGCTGCAGGTC TTTTTCTTTTGAGATGTTTCATTTTAAATTCTTGATACTCTTCAATCGATGAATTCGAG | ||
| Anp1-F Anp1-R | AACATCCTAAAGAAGTTCCATTAGACTTCGACCCTGATAGAAACCGTACGCTGCAGGTC ATGAATTCGTACTTCATATGTAGGTCACTAAAAAACCGAGCCTAATCGATGAATTCGAG | ||
| Chc1-F | TGATGCTGATGAACAGCGCGATGAACGTTCAACCCACAGGATTTCGTACGCTGCAGGTC | ||
| Chc1-R | TACACGATGGGGTACAGCAAACGAATTATTTTATCCACGTCTTAATCGATGAATTCGAG | ||
| Erg6-F Erg6-R | AAAACGCCGAAACCCCCTCCCAAACTTCCCAAGAAGCAACTCAACGTACGCTGCAGGTC TATATCGTGCGCTTTATTTGAATCTTATTGATCTAGTGAATTTAATCGATGAATTCGAG | ||
| Sik1-F Sik1-R | AAGATAAAAAGGAAAAGAAGGATAAAAAGAAGAAAAGTAAGGATCGTACGCTGCAGGTC CAAAAAGATGGGATATACTTTATTTCGATTCATTGTTCCTTTTAATCGATGAATTCGAG | ||
| Nic96-F Nic96-R | CAAGGGAAACGTACAGCACTTTAATTAATATAGACGTCTCTCTACGTACGCTGCAGGTC GATATATAGATATAAACAAAAATATACAATATTTAAAAAAATCAATCGATGAATTCGAG | ||
| Pex3-F Pex3-R | TATACAGCAACTTTGGCGTCTCCAGCTCGTTTTCCTTCAAGCCTCGTACGCTGCAGGTC ATATATATATTCTGGTGTGAGTGTCAGTACTTATTCAGAGATTAATCGATGAATTCGAG | ||
| Spc42-F Spc42-R | ATAATATGTCAGAAACATTCGCAACTCCCACTCCCAATAATCGACGTACGCTGCAGGTC CTTTAAGAATGCGCCATACTCCTTAACTGCTTTTTAAATCATCAATCGATGAATTCGAG | ||
| Cox4-F Cox4-R | ACAAACTAAACCCTGTTGGTGTTCCAAATGATGACCACCATCACCGTACGCTGCAGGTC AAAAGTAAAAGAGAAACAGAAGGGCAACTTGAATGATAAGATTAATCGATGAATTCGAG | ||
| EGFP-F | CCGGAATTCTGGCAGGTGCTGGTGCTGGTGCTGGAGCAATCCTGGTGAGCAAGGGCGA AGAGAGAGAIL*(EcoRⅠ) | ||
| EGFP-R | CCGCTCGAGCTTACTTGTACAGCTCGTC(XhoⅠ) | ||
| SipA-F | AAGGTACCTAGGATCCAGGTTACAAGTGTAAGGACT(BamHⅠ) | ||
| SipA-R | AGCACCTGCCAGAATTCCACGCTGCATGTGCAAGCC(EcoRⅠ) | ||
| D-Sac6-F | ACGTCGGGTTGGTTG | D-Cop1-F | ATCTGCAGAAGTCAGGT |
| D-Snf7-F | ATGTGGTCATCACTT | D-Sec13-F | CACGTTGACTGGCCA |
| D-Anp1-F | AATCAGAAGGGGGTC | D-Chc1-F | AAGTTTCTAACTTAGAAAT |
| D-Erg6-F | AGCAAGAGAGATTGC | D-Sik1-F | ATGGCTCCTATTGAATACC |
| D-Nic96-F | AGTCGGGAAACAGTACCT | D-Pex3-F | ATCGTGGTGGAGCG |
| D-Spc42-F | CCTACAGATAAAAATAAGCG | D-Cox4-F | ATGCTTTCACTACGT |
| D-R | ATCGATGAATTCGAG | ||
| *The coding region of the EGFP was cloned into the multiple cloning site of pYES2-NTA plasmid preceded by a spacer sequence that codes for the peptide ‘AGAGAGAGAIL’. This spacer peptide can facilitate the correct folding of the fluorescent proteins when coupled to the protein of interest. The restriction endonuclease sites are underlined. | |||
表选项
1.4 含同源臂的RedStar和kanMX4模块的扩增 以质粒pYM38为模板(0.1 ng),用细胞器荧光标记引物进行PCR扩增。PCR扩增产物用1.5%琼脂糖凝胶进行分离,EB染色,凝胶成像仪进行检测。
1.5 酿酒酵母细胞的转化 采用醋酸锂化学转化方法转化质粒或PCR产物至酵母细胞中。接种新鲜W303-1A酵母单菌落到3 mL YPD培养基中,220 r/min、30 ℃过夜培养。用新鲜培养基稀释至5×106细胞/mL(OD600≈0.5),摇床培养3–4 h,5000 r/min离心2 min收集细胞,无菌ddH2O洗涤,离心重复洗涤1次;然后分装大约108个细胞进行一个转化,离心弃上清后加入200 μL 0.1 mol/L的醋酸锂重悬细胞,30 ℃水浴15 min;5000 r/min离心2 min,弃上清,加入240 μL 50% PEG3350 (W/V)涡旋混匀后,加入110 μL转化混合物(36 μL 1 mol/mL醋酸锂;24 μL 10×TE,pH=7.5;5 μL 10 mg/mL ssDNA;5 μL PCR产物或100 ng质粒DNA;用无菌ddH2O补齐至110 μL)重悬细胞沉淀;30 ℃温浴30 min;加入40 μL DMSO,充分混匀后42 ℃热激20 min。12000 r/min离心30 s,移去上清,加入100–200 μL无菌ddH2O重悬细胞,涂布在含200 mg/L G418的YPD平板上,待菌落长出后挑取单克隆,划线在新的含有200 mg/L G418的YPD平板上,接种单菌落进行PCR鉴定。
1.6 酿酒酵母定位菌株的PCR验证 使用引物设计软件Primer Premier 5.0设计定位菌株PCR验证引物。原则为上游引物位于定位基因的编码框中,下游引物位于质粒pYM38的RedStar和kanMX4模块中(共用),引物长度为15–20 bp。提取G418平板上筛选得到的阳性克隆基因组DNA,以基因组DNA作为模板进行PCR扩增,PCR产物用1.5%琼脂糖凝胶进行分离,EB染色,凝胶成像仪检测。扩增产物符合预设的菌株可用作下一步荧光观察。
1.7 酿酒酵母定位菌株的荧光显微镜观察确认 取适量对数生长期的酵母细胞(不多于107细胞/mL),用500 μL无菌ddH2O重悬洗涤,离心10000 r/min 10 s,再用1 mL PBS洗涤2次,离心,去上清后加入1 mL 70%乙醇室温固定20 min,离心12000 r/min 20 s,去上清,用1 mL PBS洗涤2次,重悬于100 μL PBS中。取10 μL细胞悬液滴加到载玻片,加入5 μL抗荧光淬灭剂,用微量移液器吹打均匀,盖上盖玻片,晾干后使用激光共聚焦显微镜,通过油镜(放大倍数×100倍)观察其荧光定位情况并拍照记录。
1.8 酿酒酵母的活细胞染色 运用荧光染料DAPI标记酵母细胞核,用线粒体绿色荧光探针Mito tracker Green标记酵母线粒体,然后在激光共聚焦显微镜下观察相应酵母定位菌株与荧光染料的共定位情况。细胞收集和处理步骤与1.7相同,在重悬于100 μL PBS后,加入1 mg/μL DAPI或1 mg/μL Mito tracker Green,室温暗处孵育15 min,离心去上清后用1 mL PBS洗涤3次,余50 μL PBS重悬细胞,取10 μL细胞悬液滴加到载玻片,加入5 μL抗荧光淬灭剂用微量移液器吹打均匀,盖上盖玻片,稍微静置晾干后使用激光共聚焦显微镜,通过油镜(放大倍数×100倍)观察红色荧光(Excited:570 nm,Emission:595 nm)、蓝色荧光(Excited:358 nm,Emission:461 nm)、绿色荧光(Excited:488 nm,Emission:509 nm)及在可见光下的细胞形态,拍照记录经成像软件生成DAPI标记的细胞核和RedStar标记的细胞器或Mito tracker Green标记的线粒体和RedStar标记的细胞器共定位图片。
1.9 沙门氏菌SipA-EGFP质粒的构建 采用两步克隆的方法构建沙门氏菌SipA-EGFP质粒。以质粒pEGFP为模板,设计EGFP特异性扩增引物,进行PCR扩增。用EcoRⅠ和XhoⅠ对EGFP扩增产物和pYES2-NTA质粒分别进行双酶切,将回收的扩增产物和质粒酶切产物用T4连接酶连接并转化至DH5α感受态细胞。用扩增引物对所得到的转化子进行菌落PCR,1%琼脂糖凝胶检测,并将阳性转化子送上海生工进行测序,保存得到的pYES2-NTA-EGFP质粒。
以肠炎沙门氏菌基因组DNA为模板,设计SipA特异性基因扩增引物,进行PCR扩增。用BamHⅠ和EcoRⅠ对SipA扩增产物和pYES2- NTA-EGFP质粒分别进行双酶切,将回收的扩增产物与质粒酶切产物用T4连接酶连接并转化至DH5α感受态细胞。用扩增引物对所得到的转化子进行菌落PCR,1%琼脂糖凝胶检测,并将阳性转化子送上海生工进行测序,保存得到的pYES2-NTA-SipA-EGFP质粒。
1.10 沙门氏菌SipA在酿酒酵母细胞内的定位研究 将含有沙门氏菌SipA-EGFP融合质粒的酵母菌株接种于SCM-Ura (2%葡萄糖)液体中,30 ℃过夜培养。用新鲜培养基将培养液稀释至5×106细胞/mL (OD600≈0.5),摇床培养3–4 h,5000 r/min离心2 min收集细胞,无菌ddH2O洗涤,离心重复洗涤1次。将细胞重悬于SCM-Ura (2%半乳糖)液体中,诱导表达6 h。离心收集细胞,用ddH2O洗涤1次,无菌ddH2O重悬调整至合适的浓度,取10 μL酵母细胞滴加于载玻片上,加入5 μL抗荧光淬灭剂,用微量移液器吹打均匀,盖上盖玻片,于暗处稍微静置晾干使用激光共聚焦显微镜通过油镜镜检观察其绿色荧光、红色荧光及在可见光下的细胞形态,拍照记录相应的荧光及可见光图像,经成像软件叠加绿色荧光、红色荧光及可见光的图片,生成EGFP标记的SipA和RedStar标记的细胞器的共定位图片,分析SipA在酵母细胞中的定位情况。
2 结果和分析 2.1 酿酒酵母定位报告基因的选择 本实验参照相关文献,选择了12个定位于不同细胞器的基因作为报告基因来进行构建[14],基因简要描述见表 3。
表 3. 本研究所选择基因的标准名、定位及其简要描述 Table 3. The standard name, localization and description of selected gene
| Standard name | Localization | Description |
| Sac6 | Actin cytoskeleton | Fimbrin, actin-bundling protein; cooperates with Scp1p in organization and maintenance of the actin cytoskeleton; modulate actin filament binding affinity and actin cable dynamics |
| Cop1 | Early Golgi/COPI | Alpha subunit of COPI vesicle coatomer complex; complex surrounds transport vesicles in the early secretory pathway |
| Snf7 | Endosome | Endosomal sorting complex required for transport III, involved in the sorting of transmembrane proteins into the multivesicular body (MVB) pathway; recruited from the cytoplasm to endosomal membranes |
| Sec13 | Endoplasmic reticulum to Golgi | Subunit of the Nup84p nuclear pore subcomplex that contributes to nucleocytoplasmic transport and NPC biogenesis; subunit of the COPII vesicle coat required for ER-to-Golgi transport |
| Anp1 | Golgi apparatus | Type II membrane protein; a role in retention of glycosyltransferases in the Golgi |
| Chc1 | Late Golgi/clathrin | Subunit of the major coat protein involved in intracellular protein transport and endocytosis; the light chain (CLC1) is thought to regulate function |
| Erg6 | Lipid particle | Localized to lipid particles, the plasma membrane-associated endoplasmic reticulum, and the mitochondrial outer membrane |
| Sik1 | Nucleolus | Essential evolutionarily-conserved nucleolar protein; overexpression causes spindle orientation defects |
| Nic96 | Nuclear periphery | Linker nucleoporin component of the nuclear pore complex (NPC); contributes to nucleocytoplasmic transport and NPC biogenesis |
| Pex3 | Peroxisome | Peroxisomal membrane protein (PMP); required for proper localization and stability of PMPs; anchors peroxisome retention factor Inp1p at the peroxisomal membrane |
| Spc42 | Spindle pole | Central plaque component of spindle pole body (SPB); involved in SPB duplication, may facilitate attachment of the SPB to the nuclear membrane |
| Cox4 | Mitochondria | Subunit IV of cytochrome c oxidase; the terminal member of the mitochondrial inner membrane electron transport chain; precursor N-terminal 25 residues are cleaved during mitochondrial import |
表选项
2.2 酿酒酵母定位报告菌株构建原理 利用酿酒酵母同源重组率高的特点,定位菌株的构建原理如图 1,将红色荧光蛋白RedStar和抗生素kanMX4的序列,通过同源重组的方式分别整合到12个报告基因C端,该定位基因表达时,与红色荧光蛋白RedStar融合表达。在适当的激发光激发下产生的红色荧光可指示该特定基因的定位,这种菌株构建的方法使得荧光蛋白在酵母胞内的表达受标记基因的调控模式所调控,其表达水平和表达模式与野生型相似,干扰最小。
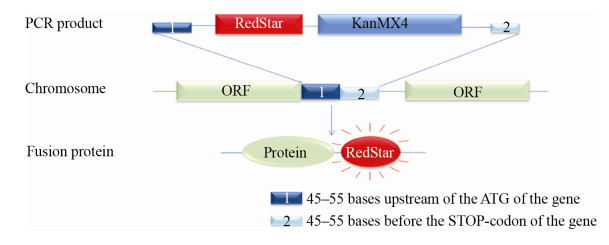 |
| 图 1 菌株构建策略 Figure 1 Strategy for strains construction |
| 图选项 |
2.3 检测含同源臂的RedStar和kanMX4模块的扩增产物 将含同源臂的RedStar和kanMX4模块的扩增产物用1.5%琼脂糖凝胶检测(图 2)。从图中可以看出,产物扩增片段长度为2500 bp,与预计大小相符。
 |
| 图 2 含有RedStar和kanMX4模块的PCR框的扩增 Figure 2 Amplification of PCR-cassettes containing RedStar and kanMX4 Modules. M: DL5000 DNA marker; 1: Negative control; 2: Sac6; 3: Cop1; 4: Snf7; 5: Sec13; 6: Anp1; 7: Chc1; 8: Erg6; 9: Sik1; 10: Nic96; 11: Pex3; 12: Spc42; 13: Cox4 |
| 图选项 |
2.4 酿酒酵母阳性转化株的鉴定 将含同源臂的RedStar和kanMX4模块的扩增产物转化酿酒酵母菌株W303-1A,在YPD+G418选择性平板上培养,挑取转化株,划线至新的YPD+G418板上,挑取单克隆菌株提取基因组,用验证引物(前引物为报告基因特异,后引物位于RedStar和kanMX4模块内,公用)进行PCR扩增,以鉴定筛选阳性转化株。如图 3所示,PCR扩增产物与预计扩增产物大小一致,将扩增产物送至生工生物工程(上海)股份有限公司进行测序,结果与预期相符,可对酵母转化株进行下一步的胞内蛋白定位的荧光显微镜检测。
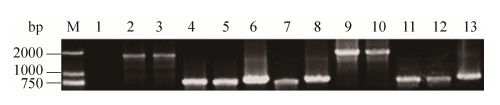 |
| 图 3 12株荧光定位报告菌株的特异性PCR检测 Figure 3 Special PCR detection of 12 fluorescent localization report strains. M: DL2000 DNA marker; 1: Negative control; 2: Sac6 (1700 bp); 3: Cop1 (1700 bp); 4: Snf7 (750 bp); 5: Sec13 (750 bp); 6: Anp1 (750 bp); 7: Chc1 (750 bp); 8: Erg6 (750 bp); 9: Sik1 (1700 bp); 10: Nic96 (1700 bp); 11: Pex3 (750 bp); 12: Spc42 (750 bp); 13: Cox4 (750 bp) |
| 图选项 |
2.5 酿酒酵母荧光标记的荧光观察 在激光共聚焦显微镜下检验和评估各个酵母报告菌株的荧光定位和信号强弱。对12株酵母荧光定位报告菌株的荧光显微镜观察的结果如图 4和图 5所示,12株菌株均观察到红色信号,表明菌株RedStar表达正常,各菌株之间RedStar分布模式存在定位差异,各亚细胞器形态特征与已有参考文献相符[14],可用作本实验研究的酵母荧光定位报告菌株。
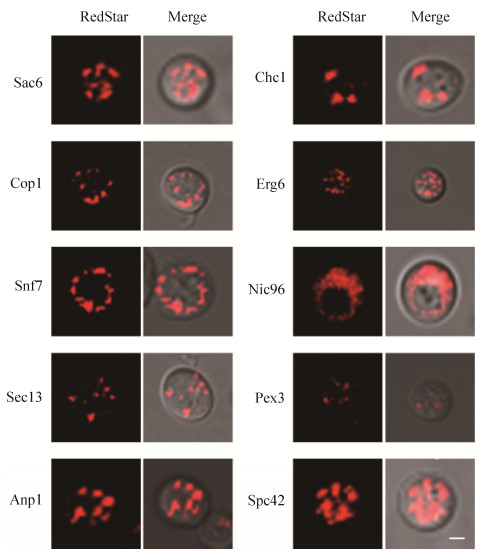 |
| 图 4 10株不同亚细胞红色荧光蛋白定位酿酒酵母菌株荧光检测 Figure 4 Fluorescence detection of 10 specific sub-cellular Redstar-tagged S. cerevisiae strains. The cells were collected in logarithmic growth and fixed with paraformaldehyde. The cells were analysed by fluorescence microscopy. Bars: 2 μm |
| 图选项 |
 |
| 图 5 Sik1和Cox4标记的报告菌株与小分子染料特异染色的共定位分析 Figure 5 Co-localization analysis of reported strains labeled with Sik1 and Cox4 and specific staining with small molecular dyes. The cells were collected in logarithmic growth and fixed with paraformaldehyde. The cells were analysed by fluorescence microscopy. Bars: 2 μm |
| 图选项 |
2.6 酿酒酵母荧光标记与小分子染料特异染色的共定位分析 为了进一步验证构建的荧光定位报告菌株,利用构建的荧光定位报告菌株与小分子染料特异染色进行共定位分析。Sik1是小分子核仁核糖核蛋白(snoRNP)复合物成分,定位于核仁。在构建的酵母定位报告菌株Sik1-Redstar中,使用DAPI标记细胞核,在激发光下会发出蓝色荧光,与Sik1-Redstar的红色荧光共定位后重合,产生紫色荧光(图 5)。
Cox4是线粒体中细胞色素C氧化酶(线粒体内膜电子传递链中心酶复合物)的第4亚基。使用线粒体特异性染料Mito tracker对构建的酵母定位报告菌株Cox4-Redstar进行染色后发现,绝大部份的Cox4的红色荧光与染料的绿色荧光共定位后重合,产生黄色荧光(图 5)。这一结果验证了构建的菌株的正确性。
2.7 肺炎沙门氏菌SipA-EGFP融合表达质粒的构建 为了进一步验证构建的酿母荧光定位报告菌株可以用于目的蛋白的共定位研究,选择已知定位的肺炎沙门氏菌效应蛋白SipA作为目的蛋白。本实验用两步克隆方法构建了沙门氏菌SipA-EGFP融合表达质粒,中间过程中提取的质粒、扩增产物和酶切产物均用1%琼脂糖凝胶进行电泳,凝胶成像仪进行检测(图 6)。选送相应的目标克隆质粒进行测序后发现,序列与NCBI上传(序列号:AF458099.1)氨基酸序列一致,质粒构建成功,可进行下一步实验。本实验用两步克隆方法构建了沙门氏菌SipA-EGFP融合表达质粒(图 6-B)。
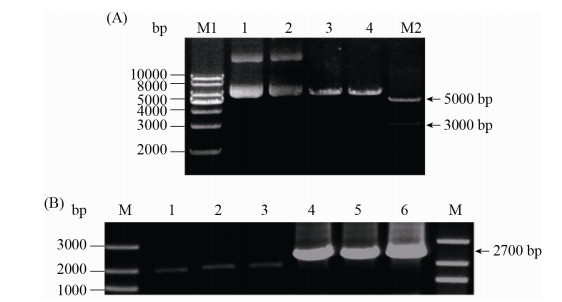 |
| 图 6 SipA-EGFP质粒构建 Figure 6 Construction of SipA-EGFP plasmid. A: M1: DL1 kb DNA marker; M2: DL5000 DNA marker; lane: 1–2: pYES2-NTA plasmid; lane:3–4: pYES2-NTA plasmid digested by EcoR I and Xho I. B: M: DL5000 DNA marker; lane:1–3: SipA PCR-product; lane: 4–6: Colony PCR-product of pYES2-NTA-SipA-EGFP |
| 图选项 |
2.8 肺炎沙门氏菌SipA与肌动蛋白标记的酿酒酵母的共定位分析 已有文献表明,SipA在酿酒酵母中表达时,会与丝状肌动蛋白结合,降低肌动蛋白聚合所需的肌动蛋白浓度,与肌动蛋白骨架共定位[15]。与野生型相比,诱导表达SipA会使得酵母的丝状肌动蛋白束变厚,体外表达的SipA会抑制肌动蛋白解聚[16]。
Sac6是一种丝束蛋白,能够横向连接相邻微丝,形成排列紧密的微丝束。因此,Sac6的定位可以间接反映细胞中聚合的纤维状肌动蛋白(微丝)的形态和动态变化。
为了鉴定SipA、Sac6这两种肌动蛋白结合蛋白在酿酒酵母中是否存在共定位,在构建的Sac6-RedStar菌株中转入pYES2-NTA-SipA-EGFP质粒,并进行SipA-EGFP融合蛋白的诱导表达。
利用激光共聚焦显微镜,通过绿、红、可见光三通道对pYES2-NTA-SipA-EGFP转化株进行观察,拍照记录各通道的图像,并对其进行叠加生成EGFP标记的效应蛋白和RedStar标记的丝束蛋白的共定位图片。从图 7中可以看出,融合蛋白SipA-EGFP和Sac6-RedStar吻合叠加产生黄色,表明沙门氏菌的效应蛋白SipA与丝束蛋白Sac6存在共定位。
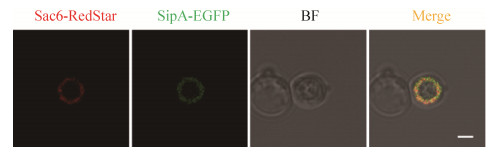 |
| 图 7 肺炎沙门氏菌SipA与Sac6标记的报告菌株的共定位分析 Figure 7 Co-localization analysis of reported strains labeled with SipA and Sac6 of Salmonella pneumoniae. The cells were collected in logarithmic growth and fixed with paraformaldehyde. The cells were analysed by fluorescence microscopy. Bars: 2 μm |
| 图选项 |
3 讨论 本试验利用染色体同源重组的方法构建了12个组成型表达红色荧光的酵母细胞器荧光定位报告菌株,用特异性引物进行PCR扩增和验证,用激光共聚焦显微镜进行荧光检测,各亚细胞器形态特征与已有参考文献相符[14]。对线粒体和细胞核进行了特异性染料染色,用EGFP标记沙门氏菌已知定位蛋白SipA,与相应报告定位菌株进行共定位,进一步证明了这些报告菌株构建成功,为日后在酵母中观察亚细胞水平细胞器动态变化,以及未知目标蛋白在酵母中的定位提供了基础性工具。
2003年,Huh课题组首次尝试在真核模式生物——酿酒酵母细胞中全面确定每种蛋白质的亚细胞定位[14]。Huh等利用染色体同源重组技术将绿色荧光蛋白(GFP)和选择标记(G418)分别融合到97%已命名的酵母开放阅读框的C端[14]。这种方法利用定位基因自身的启动子,不会造成过度表达影响定位。该课题组又用同样方法,将红色荧光蛋白(mRFP)融合到已知定位的酵母细胞器标记基因C端,构建了11个共定位参考菌株[14]。本实验参考了这11个定位标记基因,添加线粒体标记基因Cox4,构成一套酿酒定位报告菌株。本试验用红色荧光蛋白的突变体RedStar,替代Huh课题组所用的mRFP,作为融合的荧光蛋白。RedStar寡聚化程度低、成熟速率快。pYM38中编码的RedStar序列,根据酿酒酵母的密码子偏爱性进行了同义突变,适合在酵母中表达[17]。
国内谢志平课题组构建两套含有酵母细胞器标记蛋白和GFP/mCherry荧光蛋白标签相融合的组成型表达质粒[18],利用Western blotting及密度梯度离心法进行检测标记基因。其选用的定位基因与我们选择的基因有部分重合,分别得到两套定位参考质粒和一套定位参考菌株,可视自身需要,选用任意一种构建下一步的共定位实验。
Huh课题组和谢志平组均运用荧光显微镜对参考菌株进行了荧光观察[14, 17]。本实验运用激光共聚焦显微镜来检测和跟踪荧光。酵母细胞较小,直径只有3–5 μm左右(哺乳动物细胞可达到10–100 μm),一般的荧光显微镜难以产生好的荧光图片,而共聚焦显微镜的精度已提高到了0.4 μm,足以全面多角度观察酵母细胞的内部结构。因此,本实验中各细胞器定位基因的亚细胞分布及形态特征更清晰明确。
在使用激光共聚焦显微镜观察野生型菌株中Sac6的定位,发现Sac6在细胞中呈点状或斑状,显示其可能与微丝中的皮质斑共定位,但当有SipA-EGFP诱导表达时,Sac6在细胞中呈环状分布。已有研究文献中都是用荧光素标记的鬼笔环肽对细胞中的肌动蛋白细胞骨架进行染色,并未得到相似结果[15]。因此,用Sac6和鬼笔环肽对微丝进行定位的区别还有待进一步解析,SipA与Sac6是否存在互作,SipA是否会影响Sac6与肌动蛋白的结合,还有待进一步实验来确认。
References
| [1] | Duina AA, Miller ME, Keeney JB. Budding yeast for budding geneticists: a primer on the Saccharomyces cerevisiae model system. Genetics, 2014, 197(1): 33-48. DOI:10.1534/genetics.114.163188 |
| [2] | Siggers KA, Lesser CF. The yeast Saccharomyces cerevisiae: a versatile model system for the identification and characterization of bacterial virulence proteins. Cell Host Microbe, 2008, 4(1): 8-15. DOI:10.1016/j.chom.2008.06.004 |
| [3] | Popa C, Coll NS, Valls M, Sessa G. Yeast as a heterologous model system to uncover type Ⅲ effector function. PLoS Pathogens, 2016, 12(2): e1005360. DOI:10.1371/journal.ppat.1005360 |
| [4] | Kwak GH, Kim JR, Kim HY. Expression, subcellular localization, and antioxidant role of mammalian methionine sulfoxide reductases in Saccharomyces cerevisiae. BMB Reports, 2009, 42(2): 113-118. DOI:10.5483/BMBRep.2009.42.2.113 |
| [5] | Matsushika A, Watanabe S, Kodaki T, Makino K, Inoue H, Murakami K, Takimura O, Sawayama S. Expression of protein engineered NADP+-dependent xylitol dehydrogenase increases ethanol production from xylose in recombinant Saccharomyces cerevisiae. Applied Microbiology and Biotechnology, 2008, 81(2): 243-255. DOI:10.1007/s00253-008-1649-1 |
| [6] | Curak J, Rohde J, Stagljar I. Yeast as a tool to study bacterial effectors. Current Opinion in Microbiology, 2009, 12(1): 18-23. DOI:10.1016/j.mib.2008.11.004 |
| [7] | Suter B, Auerbach D, Stagljar I. Yeast-based functional genomics and proteomics technologies: the first 15 years and beyond. Biotechniques, 2006, 40(5): 625-644. |
| [8] | Chong YT, Koh JL, Friesen H, Duffy K, Cox MJ, Moses A, Moffat J, Boone C, Andrews BJ. Yeast proteome dynamics from single cell imaging and automated analysis. Cell, 2015, 161(6): 1413-1424. DOI:10.1016/j.cell.2015.04.051 |
| [9] | Shaner NC, Steinbach PA, Tsien RY. A guide to choosing fluorescent proteins. Nature Methods, 2005, 2(12): 905-909. DOI:10.1038/nmeth819 |
| [10] | Pédelacq JD, Cabantous S, Tran T, Terwilliger TC, Waldo GS. Engineering and characterization of a superfolder green fluorescent protein. Nature Biotechnology, 2006, 24(1): 79-88. DOI:10.1038/nbt1172 |
| [11] | Gerdes HH, Kaether C. Green fluorescent protein: applications in cell biology. FEBS Letters, 1996, 389(1): 44-47. DOI:10.1016/0014-5793(96)00586-8 |
| [12] | Chudakov DM, Lukyanov S, Lukyanov KA. Using photoactivatable fluorescent protein Dendra2 to track protein movement. Biotechniques, 2007, 42(5): 553-563. DOI:10.2144/000112470 |
| [13] | Janke C, Magiera MM, Rathfelder N, Taxis C, Reber S, Maekawa H, Moreno-Borchart A, Doenges G, Schwob E, Schiebel E, Knop M. A versatile toolbox for PCR-based tagging of yeast genes: new fluorescent proteins, more markers and promoter substitution cassettes. Yeast, 2004, 21(11): 947-962. DOI:10.1002/yea.1142 |
| [14] | Huh WK, Falvo JV, Gerke LC, Carroll AS, Howson RW, Weissman JS, O'Shea EK. Global analysis of protein localization in budding yeast. Nature, 2003, 425(6959): 686-691. DOI:10.1038/nature02026 |
| [15] | Lesser CF, Miller SI. Expression of microbial virulence proteins in Saccharomyces cerevisiae models mammalian infection. The EMBO Journal, 2001, 20(8): 1840-1849. DOI:10.1093/emboj/20.8.1840 |
| [16] | Zhou DG, Mooseker MS, Galán JE. Role of the S. typhimurium actin-Binding protein SipA in bacterial internalization. Science, 1999, 283(5410): 2092-2095. DOI:10.1126/science.283.5410.2092 |
| [17] | Knop M, Barr F, Riedel CG, Heckel T, Reichel C. Improved version of the red fluorescent protein (drFP583/DsRed/RFP). Biotechniques, 2002, 33(3): 592, 594, 596–568. |
| [18] | 赵柏淞.酵母细胞器荧光标记系统的开发及鉴定.上海交通大学博士后学位论文, 2017. |
