檀玲1, 郭林霞1, 陈博文1, 张昭寰1,2, 王敬敬1,3,4, 刘海泉1,3,4, 潘迎捷1,3,4, 赵勇1,3,4


1. 上海海洋大学食品学院, 上海 201306;
2. 上海海洋大学水产与生命学院, 上海 201306;
3. 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
4. 农业农村部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
收稿日期:2019-06-26;修回日期:2019-08-20;网络出版日期:2019-09-20
基金项目:国家自然科学基金面上项目(31571917,31671779);国家重点研发计划(2018YFC1602205);上海市科技兴农项目(沪农科推字2017第4-4号);上海市教育委员会科研创新计划(2017-01-07-00-10-E00056)
*通信作者:赵勇, Tel:+86-21-61900503;E-mail:yzhao@shou.edu.cn.
摘要:[目的] 副溶血性弧菌是水产品中常见的食源性致病菌,生物被膜的形成对副溶血性弧菌的环境生存和传播至关重要。这项工作的目的是评估临床和环境中分离出的44株副溶血性弧菌菌株形成的生物被膜的结构多样性。[方法] 该研究基于共聚焦激光扫描显微镜的高通量方法,使用与高分辨率成像兼容的96孔微量滴定板,结合结构分析软件ISA-2来研究生物被膜形成和结构,分析22株食品与22株临床来源的副溶血性弧菌菌株形成的生物被膜结构参数(生物体积、平均厚度、粗糙系数)。[结果] CLSM图像显示,44株副溶血性弧菌菌株在培养48 h后能够形成3D结构,进一步比较分析了临床来源菌株与环境来源菌株形成的生物被膜结构异同,发现临床菌株生物被膜的变异系数比环境菌株生物被膜的变异系数小,且同时携带tdh和trh两种毒力因子的菌株生物被膜变异性最小。凝聚层次聚类分析结果显示,副溶血性弧菌生物被膜可以分为致密且表面光滑(39%)、斑驳且表面粗糙(27%)、疏松且表面坑洼(34%),临床菌株易形成致密且表面光滑和斑驳且表面粗糙的生物被膜,而环境菌株易形成致密且表面光滑和疏松且表面坑洼的生物被膜。[结论] 该研究深入了解了副溶血性弧菌生物被膜结构差异性,为今后对临床和环境来源的副溶血性弧菌生物被膜采取不同的防控和清除措施提供了理论支撑。
关键词:副溶血性弧菌生物被膜共聚焦激光扫描显微镜结构参数
Quantitative analysis of Vibrio parahaemolyticus biofilm structure based on high-throughput confocal laser scanning microscopy
Ling Tan1, Linxia Guo1, Bowen Chen1, Zhaohuan Zhang1,2, Jingjing Wang1,3,4, Haiquan Liu1,3,4, Yingjie Pan1,3,4, Yong Zhao1,3,4


1. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China;
2. College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306, China;
3. Shanghai Engineering Research Center of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
4. Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation(Shanghai), Ministry of Agriculture and Rural Affairs, Shanghai 201306, China
Received: 26 June 2019; Revised: 20 August 2019; Published online: 20 September 2019
*Corresponding author: Zhao Yong, Tel:+86-21-61900503; E-mail:yzhao@shou.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31571917, 31671779), by the National Key R & D Program of China (2018YFC1602205), by the Shanghai Agriculture Applied Technology Development Program (T20170404) and by the Innovation Program of Shanghai Municipal Education Commission (2017-01-07-00-10-E00056)
Abstract: [Objective] Vibrio parahaemolyticus is a common foodborne pathogen in aquatic products. The formation of biofilm is essential for the survival and spread of Vibrio parahaemolyticus. The aim of this study was to evaluate the structural diversity of biofilms formed by 44 strains of Vibrio parahaemolyticus isolated under clinical and environmental conditions. [Methods] This study proposed a high-throughput method based on confocal laser scanning microscopy (CLSM), using 96-well microtiter plates compatible with high-resolution imaging, combined with structural analysis software ISA-2 to study biofilm formation and structure, the biofilm structure parameters (Biovolume, Average thickness, Biofilmroughness) formed by 22 clinical strains and 22 environmental strains were analyzed. [Results] CLSM images showed that 44 strains of Vibrio parahaemolyticus could form 3D structure after 48 h incubation, and further analyzed the similarities and differences of biofilm formation between clinically derived strains and environmentally derived strains, showed that the coefficient of variation in the clinical strains were smaller than that of the environmental strains, and the strains BF with both tdh and trh virulence factors had the smallest variability. Agglomerative hierarchical clustering (AHC) showed that biofilm can be divided into dense and smooth surface (39%), mottled and rough surface (27%) and loose and uneven surface (34%), the clinical strain formed a biofilm which is easy to form dense and smooth surface, mottled and rough surface, whereas the environmental strain formed a biofilm which is easy to dense and smooth surface and loose and uneven surface. [Conclusion] This study provides an in-depth understanding of the biofilm structure of Vibrio parahaemolyticus, it provides theoretical support for different prevention and control measures for Vibrio parahaemolyticus biofilms from clinical and environmental sources.
Keywords: Vibrio parahaemolyticusbiofilmconfocal laser scanning microscopystructure parameters
副溶血性弧菌(Vibrio parahaemolyticus,VP)是革兰氏阴性细菌,栖息于海洋和河口环境中,是全球大多数人类海鲜相关肠胃炎和虾急性肝胰脏坏死综合征的主要病原体,摄入被细菌污染的未煮熟或生海鲜导致急性肠胃炎,其特征为腹泻、恶心、呕吐、轻度发烧和腹部绞痛[1-2]。细菌致病性通常与毒力因子的存在有关,副溶血性弧菌的致病性主要归因于两种主要毒力因子的产生:由tdh基因编码的热稳定的直接溶血素(Thermostable direct hemolysin,TDH)和由trh基因编码的TDH相关的溶血素(Thermolabile hemolysin,TRH)[2-4]。除了TDH、TRH等致病因子的作用外,副溶血性弧菌菌株的致病性也与生物被膜的形成有关,在大多数天然或食品加工环境中,副溶血性弧菌通过形成生物被膜而存活[2, 5]。
生物被膜是在表面密集附着的微生物群落,嵌入在胞外多糖、蛋白质、脂质和eDNA的基质中,俗称胞外聚合物(EPS),它们形成支架以提供结构稳定性,对微生物起到保护屏障的作用[6-9]。生物被膜结构决定了其持久性和抗性,对各种抗微生物剂(如消毒剂和抗生素等)的耐受性增强,例如,抗微生物剂的扩散受到生物被膜的形状和厚度的限制,因此限制了它们对生物被膜消除的功效[6-7]。虽然与生物被膜相关的大多数表型与其空间组织有关,但大多数公布的数据对副溶血性弧菌分离株形成的生物被膜的比较是基于定量结晶紫测定,其不能获得结构信息。目前主要使用扫描电子显微镜(SEM)[10]、透射电子显微镜(TEM)[11]和共聚焦激光扫描显微镜(CLSM)[12]来研究生物被膜的三维结构。CLSM与图像分析软件一起可用于定量评估生物被膜的结构特征,然而,SEM与TEM费用昂贵,并且不能定量生物被膜。生物被膜内的3D空间结构可以通过与CSLM结合的荧光染色然后结合图像分析软件计算来获得,并且对结构参数(如厚度、生物体积、粗糙度等)进行量化[13]。
目前已经开发了用于图像分析的软件工具,包括COMSTAT[14]、ISA[15]、ISA 3D[16]和ISA-2[17],PHLIP[18]和BIOFILMDIVER[13]已经用于与生物被膜相关的研究目的。COMSTAT是一种用于快速、轻松地分析大量的三维空间的生物被膜图像数据计算机程序[14]。ISA自动量化来自生物被膜图像的参数,并且可以执行批处理,这在图像数量很大时很重要[15],但是COMSTAT和ISA软件只能处理得出二维的参数,而ISA3D涵盖了其他软件可以计算的大多数参数,并在三个维度上引入了新参数[16]。ISA-2包含了用于二维参数的ISA和三维参数的ISA3D[17]。PHLIP通过多通道CLSM软件展示了生物被膜图像分析的强大功能,并展示了PHLIP作为自动图像处理的灵活和可扩展图像分析平台对科学界的重要性[18]。BIOFILMDIVER是用来开发用于执行动态分析并平行处理多个菌株[13]。
这些图像分析软件主要应用于表征动态单核细胞增生李斯特菌生物被膜结构的研究。Rieu等[19]使用CLSM和PHLIP在批次和流动条件下比较了单核细胞增生李斯特菌生物被膜的生物体积和平均厚度。Bridier等[20]使用PHLIP量化了10株单核细胞增生李斯特菌菌株在24 h形成的生物被膜的最大厚度、生物体积和粗糙度。Mukherjee等[21]开发了一种非破坏性微流体膜流动池,结合CLSM和定量图像分析,以量化生物污损参数,如表面覆盖、生物体积和生物被膜厚度。
国内基于高通量共聚焦成像方法结合结构分析软件来分析生物被膜的研究尚未报道,关于副溶血性弧菌形成的生物被膜结构的种内多样性的数据有限。基于CLSM的高通量方法结合使用96孔微量滴定板应用于本文的研究,选择了副溶血性弧菌菌株的生物被膜生长的静态培养条件,并探究了从不同来源(临床、环境)收集的44个副溶血性弧菌菌株形成的生物被膜结构的多样性,这项工作目的是为了降低临床和食品安全风险以及防控安全事件提供理论支撑。
1 材料和方法 1.1 材料
1.1.1 实验菌株: 本文使用实验室已保存的44株不同来源的副溶血性弧菌,其基本信息为:22株临床来源副溶血性弧菌菌株是从2014年1月至2015年12月在上海医院收集的1900份粪便标本中分离的,另外22株环境来源副溶血性弧菌菌株来源于上海市售水产品(表 1)。
表 1. 菌株信息表 Table 1. Strain information
| Isolate | tdh | trh | Origin |
| VPC1 | + | – | Clinical sample from hospital patient |
| VPC2 | + | – | Clinical sample from hospital patient |
| VPC15 | + | – | Clinical sample from hospital patient |
| VPC16 | + | – | Clinical sample from hospital patient |
| VPC17 | + | – | Clinical sample from hospital patient |
| VPC18 | – | + | Clinical sample from hospital patient |
| VPC19 | + | – | Clinical sample from hospital patient |
| VPC20 | + | – | Clinical sample from hospital patient |
| VPC21 | + | – | Clinical sample from hospital patient |
| VPC22 | + | – | Clinical sample from hospital patient |
| VPC26 | + | – | Clinical sample from hospital patient |
| VPC33 | + | – | Clinical sample from hospital patient |
| VPC34 | + | – | Clinical sample from hospital patient |
| VPC35 | + | – | Clinical sample from hospital patient |
| VPC36 | – | + | Clinical sample from hospital patient |
| VPC43 | + | + | Clinical sample from hospital patient |
| VPC44 | – | – | Clinical sample from hospital patient |
| VPC48 | + | – | Clinical sample from hospital patient |
| VPC54 | + | + | Clinical sample from hospital patient |
| VPC85 | – | + | Clinical sample from hospital patient |
| VPC89 | + | – | Clinical sample from hospital patient |
| VPC94 | – | + | Clinical sample from hospital patient |
| VPD14 | + | – | Macrobrachium nipponense |
| VPD18 | + | – | Macrobrachium nipponense |
| VPD33 | + | – | Penaeus monodon |
| VPD34 | + | – | Crassostrea gigas |
| VPD57 | + | – | Macrobrachium nipponense |
| VPD58 | + | – | Penaeus vannamei |
| VPD61 | + | – | Penaeus vannamei |
| VPR102 | – | + | Penaeus vannamei |
| VPR103 | – | + | Penaeus vannamei |
| VPR104 | – | + | Penaeus vannamei |
| VPR105 | – | + | Penaeus vannamei |
| VPR108 | – | + | Penaeus vannamei |
| VPR110 | – | + | Penaeus vannamei |
| VPS16 | – | – | Macrobrachium nipponense |
| VPS17 | – | – | Macrobrachium nipponense |
| VPS18 | – | – | Macrobrachium nipponense |
| VPS19 | – | – | Macrobrachium nipponense |
| VPS20 | – | – | Macrobrachium nipponense |
| VPS21 | – | – | Macrobrachium nipponense |
| VPS33 | – | – | Macrobrachium nipponense |
| VPS34 | – | – | Macrobrachium nipponense |
| VPS36 | – | – | Macrobrachium nipponense |
表选项
1.1.2 实验试剂与仪器设备: 无水乙醇(AR),国药集团化学试剂(上海)有限公司;20×磷酸缓冲液(PBS)、50%戊二醛和SYBR Green I染料购于生工生物工程(上海)股份有限公司;胰酶大豆肉汤培养基(TSB)和硫代硫酸盐柠檬酸盐胆盐蔗糖琼脂培养(TCBS)购于北京陆桥技术有限责任公司;96孔聚苯乙烯微量滴定板购于德国葛莱娜公司;激光共聚焦扫描显微镜Leica TCS SP8购于德国徕卡公司。
1.2 菌株的活化与生物被膜的培养 将44株VP菌株在TCBS固体培养基上划线,37 ℃过夜培养。挑取单菌落到加3% NaCl的TSB液体培养基中,放入摇床(180 r/min)中培养10–12 h,然后调活菌数为OD600=0.4。生物被膜的培养根据文献方法稍作改进[22-23],将这些悬浮液(250 μL)用于接种96孔聚苯乙烯微量滴定板的孔中,将悬浮液与新鲜的3% NaCl的TSB分别以50 μL︰200 μL加入到96孔聚苯乙烯微量滴定板的每个孔中,将微量培养板在25 ℃静态条件下孵育。在25 ℃下粘附1 h后,除去含有非粘附细胞的上清液,并用250 μL培养基重新填充孔,未接菌液的纯培养基作空白对照。在25 ℃静态温育48 h后形成生物被膜,每个菌株设3个平行试验。
1.3 激光共聚焦显微镜观察 将1.2培养48 h之后的生物被膜,吸取浮游菌之后,用200 μL的0.1 mol/L PBS清洗残留的浮游菌,然后用4% (V/V)戊二醛固定30 min,用PBS洗去多余的戊二醛。之后用现配现用的SYBR Green I染料染色,置于避光处染色30 min,然后用PBS洗掉未结合的染料。最后在63倍的488 nm氩激光器的油浸物镜观察3D结构,每张获得Z轴为0.7 μm的水平面图像。每个VP菌株样品随机选取3个不同的位置进行观察,进行3次平行实验。通过ISA-2软件(美国Montana州立大学Haluk Beyenal教授提供)分析CLSM图像以确定生物被膜结构参数,例如生物体积(Biovolume,BV)、平均厚度(Average thickness,AT)和粗糙系数(Biofilmroughness,BR)[24]。
1.4 统计分析 使用SPSS 19统计软件对实验数据进行皮尔方差分析,检验水平为P < 0.05时具有显著性差异。使用统计软件R分析所有菌株的BV、AT和BR进行凝聚层次聚类分析(Agglomerative hierarchical clustering,AHC),并以热图的形式呈现。数据用平均值±标准偏差来表示,用Origin 8.0软件作图。
2 结果和分析 2.1 激光共聚焦扫描显微镜观察成熟副溶血性弧菌生物被膜三维结构
2.1.1 三维结构图像分析: CLSM图像采集显示,所有菌株在培养48 h后能够形成3D结构(图 1),包括22株临床菌株(图 1-A)和22株环境菌株(图 1-B)形成的生物被膜。从图 1可以看出,生物被膜结构从平坦的均匀细胞层到蜂窝状结构。相对于22株环境菌株形成的生物被膜的三维结构,22株临床菌株形成的生物被膜的3D结构比较均一,而环境菌株的3D结构差异较明显。
 |
| 图 1 由44株副溶血性弧菌菌株形成的生物被膜的3D CLSM图像 Figure 1 Representative 3D CLSM images of the biofilms formed by the 44 Vibrio parahaemolyticus strains. A: Clinical isolates; B: Environmental isolates. The scale bar represents 50 μm. |
| 图选项 |
2.1.2 结构参数分析: CLSM图像能够直观地观察到44株副溶血性弧菌生物被膜的3D结构以外,也可以利用ISA软件分析其3D结构参数,3D结构参数(BV、AT、BR)的分析提供了关于菌株之间差异的进一步信息。BV对应于细菌占据的体积,MT表示生物被膜的空间尺寸,BR提供了生物被膜厚度变化的量度,并且是生物被膜异质性的指标[16]。从图 2-A可以得出临床菌株中VPC34表现出最大的BV,环境菌株中VPR110表现出最大的BV,图 2-B和2-C中VPC85表现出最高的MT和最小的BR,VPS18表现出最高的MT和最小的BR。
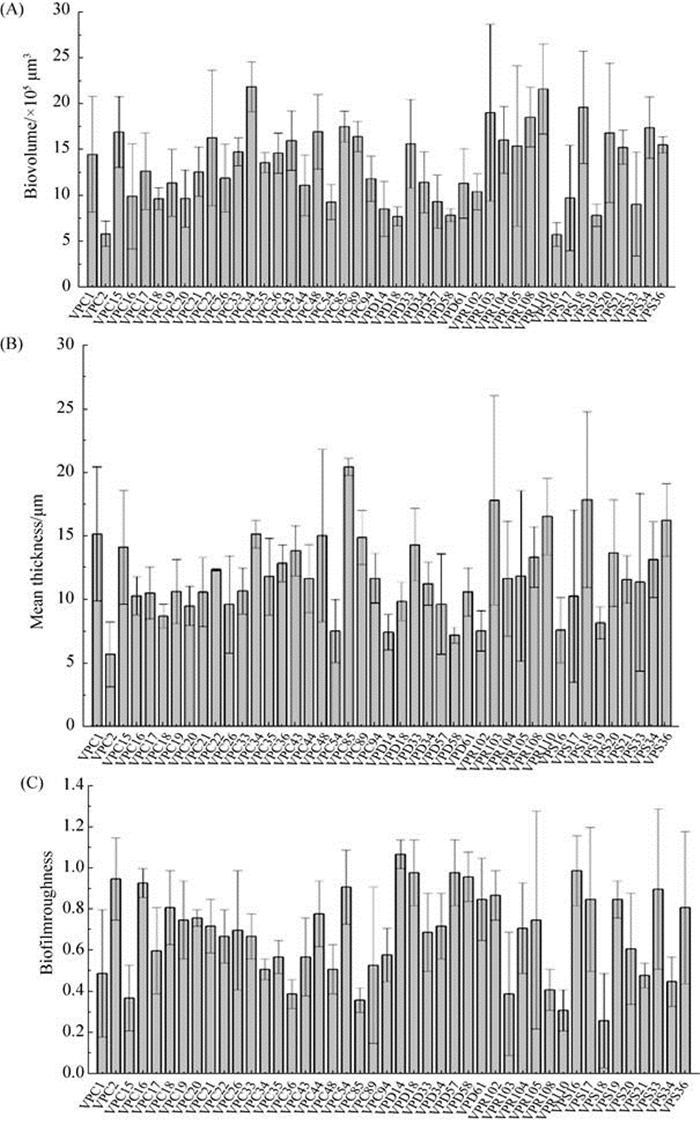 |
| 图 2 44株副溶血性弧菌菌株形成的生物被膜的结构参数 Figure 2 Structural parameters of biofilms formed by 44 Vibrio parahaemolyticus strains. A: Biovolume; B: Mean thickness; C: Biofilmroughness. All CLSM measurements were averaged from three values for three independent replicates. |
| 图选项 |
2.2 副溶血性弧菌生物被膜之间结构参数的比较分析 CLSM图像能够直观地观察到44株副溶血性弧菌生物被膜的3D结构以外,也可以利用ISA软件分析其3D结构参数,3D结构参数(BV、AT、BR)的分析提供了关于菌株之间差异的进一步信息。从表 2中我们可以看出不同来源的副溶血性弧菌形成的生物被膜结构参数有差异性,使用菌株的变异系数(Coefficient variation,CV=标准差/平均值100%)来量化菌株变异性。临床菌株生物被膜结构参数的变异系数比环境菌株生物被膜结构参数的变异系数小,这反映了临床菌株形成的生物被膜结构之间差异性较小。同时为了比较携带不同毒力基因tdh和trh基因的副溶血性弧菌生物被膜3D结构,分析了tdh+/trh+、tdh+/trh–、tdh–/trh+和tdh–/trh–之间的3D结构参数之间的变异系数,发现携带tdh+/trh+两个毒力基因的副溶血性弧菌生物被膜结构参数变异性最小,而只携带trh毒力基因的生物被膜的粗糙系数BR最大,说明只携带trh毒力基因的生物被膜表面不光滑,形成不平整的表面结构。
表 2. 44株副溶血性弧菌菌株形成的生物被膜的变异系数 Table 2. Coefficient variation of biofilms formed by 44 Vibrio parahaemolyticus strains
| Biofilm parameters | BV/(×105μm3) | AT/μm | BR |
| Clinical strains | 13.39±4.81 | 12.05±4.03 | 0.64±0.24 |
| CV | 36.46% | 33.41% | 36.69% |
| Environmental strains | 13.44±6.00 | 11.67±4.83 | 0.71±0.30 |
| CV | 44.65% | 41.35% | 42.92% |
| tdh+/trh+ | 13.63±4.34 | 11.58±3.78 | 0.70±0.25 |
| CV | 31.86% | 32.65% | 35.27% |
| tdh+/trh– | 12.44±5.07 | 4.95±1.08 | 1.07±0.05 |
| CV | 40.77% | 33.98% | 35.56% |
| tdh–/trh+ | 15.72±5.94 | 5.72±0.78 | 1.05±0.05 |
| CV | 37.79% | 40.92% | 51.27% |
| tdh–/trh– | 12.66±5.40 | 5.01±1.08 | 1.04±0.04 |
| CV | 42.63% | 39.03% | 40.26% |
表选项
同时我们将全部的数据统计分析显示,AT与其BV之间呈正相关(P < 0.05),这两个参数均与BR呈负相关(P < 0.05)(图 3)。
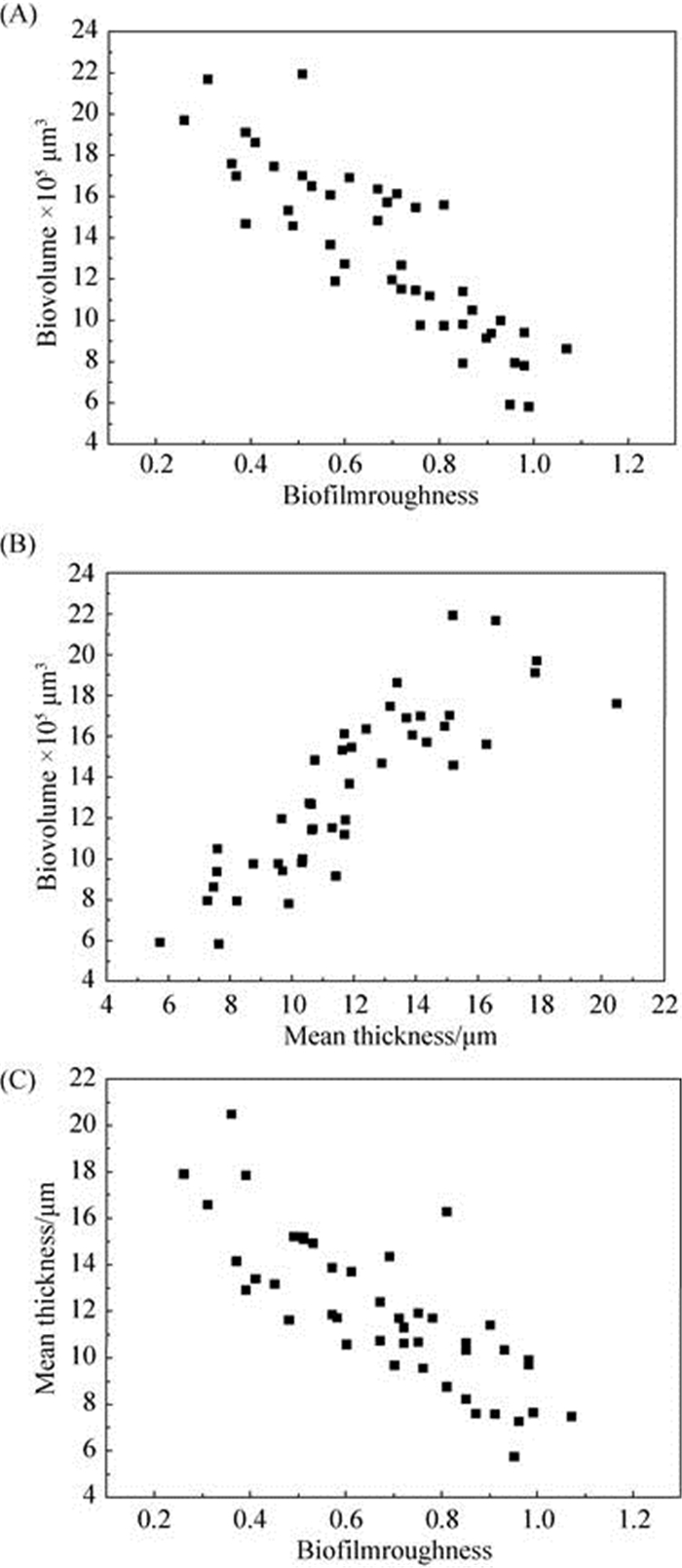 |
| 图 3 44株副溶血性弧菌生物被膜结构参数之间的相关性 Figure 3 Correlation between the biofilm structural parameters of the 44 Vibrio parahaemolyticus isolates. A: Biovolume and Biofilmroughness; B: Biovolume and Mean thickness; C: Mean thickness and Biofilmroughness. All CLSM measurements were averaged from three values for three independent replicates. |
| 图选项 |
2.3 副溶血性弧菌生物被膜之间结构参数的聚类分析 AHC用于分析副溶血性弧菌菌株形成的生物被膜的BV、AT和BR之间的关系。根据ISA-2测量,基于AHC树状图鉴定了三种生物被膜结构类型:致密且表面光滑,斑驳且表面粗糙,疏松且表面坑洼(表 3),分别对应生物被膜代表性CLSM图像(图 4-A),第一种生物被膜结构是形成致密且表面光滑的生物被膜结构:BV > 14× 105μm3,AT > 10,0.2 < BR < 0.9(图 4-A);第二种生物被膜结构是形成斑驳且表面粗糙的生物被膜结构:BV > 10×105μm3,AT < 12,BR < 0.8(图 4-Aii);第三种是形成疏松且表面坑洼的生物被膜结构:BV < 12×105μm3,AT < 12,BR > 0.8(图 4-Aiii)。
表 3. 本研究中描述的副溶血性弧菌生物被膜结构类型的定义 Table 3. Definitions of the Vibrio parahaemolyticus biofilm structure patterns described in this study
| Biofilm structure type | BV(×105μm3) | AT (μm) | BR |
| Dense and smooth surface | > 14 | > 10 | 0.2–0.9 |
| Mottled and rough surface | > 10 | < 12 | < 0.8 |
| Loose and uneven surface | < 12 | < 12 | > 0.8 |
表选项
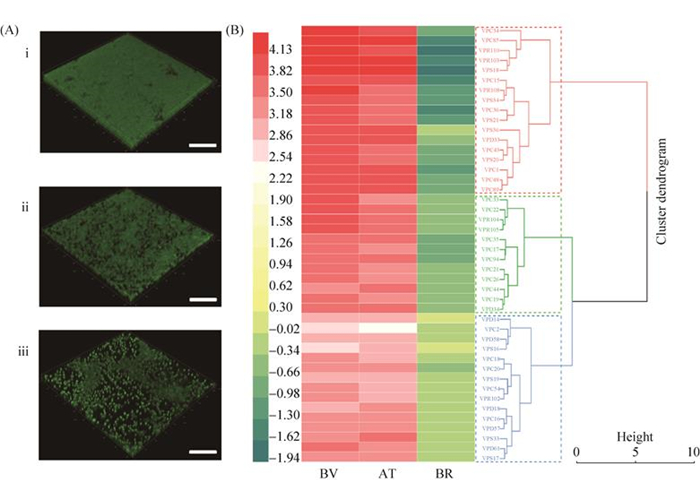 |
| 图 4 副溶血性弧菌生物被膜的三种模式菌株CLSM代表性图像和凝聚层次聚类(AHC)用生物被膜形态类型标记的树状图 Figure 4 Representative 3D CLSM images obtained by Vibrio parahaemolyticus biofilm from three patterns isolates and agglomerative hierarchical clustering (AHC) dendrogram labelled with biofilm morphotypes. A: Example of CLSM images comprising orthogonal view of 3D image. i: Dense and smooth surface; ii: Mottled and rough surface; iii: Loose and uneven surface. The scale bar represents 50 μm. B: Vibrio parahaemolyticus biofilm structure patterns defined according to CLSM measurements. Biovolume, average thickness and roughness coefficient), the height represents the dissimilarity of clusters. |
| 图选项 |
AHC树状图是使用来自44株副溶血性弧菌菌株形成的生物被膜的BV、AT和BR测量值的多变量聚类分析,并且基于结构度量将聚类分离物聚集在一起(图 4-B)。其中致密且表面光滑的生物被膜结构类型中(39%):临床菌株占47%、环境菌株53%;斑驳且表面粗糙的生物被膜结构类型中(27%):临床菌株75%、环境菌株25%;疏松且表面坑洼的生物被膜结构类型中(34%):临床菌株33%、环境菌株67%(表 4)。这些数据结果可以说明临床来源的副溶血性弧菌菌株更倾向于形成致密且表面光滑和斑驳且表面粗糙的生物被膜结构类型,而环境来源的副溶血性弧菌菌株更倾向于形成致密且表面光滑和疏松且表面坑洼的生物被膜结构类型。
表 4. 临床菌株和环境菌株形成的副溶血性弧菌生物被膜结构类型比例 Table 4. Proportion of biofilm structure types of Vibrio parahaemolyticus formed by clinical strains and environmental strains
| Biofilm structure type | Strains origin/% | |
| Clinical strains | Environmental strains | |
| Dense and smooth surface (39%) | 47 | 53 |
| Mottled and rough surface (27%) | 75 | 25 |
| Loose and uneven surface (34%) | 33 | 67 |
表选项
3 讨论 目前已经开发了几种高分辨率成像技术(例如CLSM和SEM),并用于探索生物被膜形成的特征。CLSM是目前最广泛用于研究生物被膜结构的工具之一,它能够使用特定的荧光标记直接原位和非破坏性地研究生物被膜结构并且能够结合图像分析软件定量表征生物被膜。图像分析软件实现了直接从共焦图像堆栈获得关于生物被膜的详细定量结构参数。因此,除了关于生物被膜结构的定性信息之外,还可以使用数值数据进行详细表征,并且能够进行统计分析。然而,通常用于生物被膜的共聚焦研究的装置,例如微流体膜流动池或玻璃盖玻片,可能相对昂贵或不适用于大规模研究。因此,本研究选用了基于高通量96孔微量滴定板的测定法并结合ISA-2图像分析软件来定量副溶血性弧菌生物被膜结构参数。
已有研究显示,副溶血弧菌生物被膜的形成受各种环境条件的影响,包括培养温度、接触表面以及菌株的毒力因子[5, 25]。Song等[5]利用结晶紫染色研究了在15、25和37 ℃下聚苯乙烯微量滴定板上致病性和非致病性副溶血性弧菌生物被膜发育过程,结果表明,生物被膜的形成和菌株致病性之间的相关性受温度的影响,以及副溶血性弧菌在生物被膜形成过程中存在广泛的变异,并且菌株变异性随温度升高而增加。此外,Kadam等[26]研究表明,来自血清型1/2b和1/2a的单核细胞增生李斯特氏菌菌株比血清型4b菌株形成更多的生物被膜。并且Diez-Garcia等[27]也证实沙门氏菌致病性和非致病性菌株血清型之间生物被膜形成的差异。本研究中,图 1的CLSM图像观察得出副溶血性弧菌均能在培养48 h后形成3D结构,相比于环境菌株,来源于临床的副溶血性弧菌形成的生物被膜结构之间差异性小,其次,致病性菌株比非致病菌株生物被膜结构变异系数小,说明致病菌株形成的生物被膜结构之间差异性较小、较稳定(表 2)。
Guilbaud等基于高通量CLSM成像方法分析了从各种环境中分离出的96株单核细胞增生李斯特菌形成的生物被膜结构多样性,结果显示,分离株的遗传谱系与生物被膜的结构特性之间仅有微弱的相关性[28]。然而,它们的几何描述符(生物体积,平均厚度和粗糙度)的梯度,从平面多层到复杂的蜂窝状结构,并且统计分析显示,生物被膜厚度与其生物体积之间存在正相关关系(P < 0.05),这两种结构参数均与粗糙度呈负相关(P < 0.05),这个结论与本文结果一致(图 3)。Harris等[29]研究了临床样品中表皮葡萄球菌分离株的遗传多样性,并证实了这些细菌粘附和形成的广泛能力,同时CLSM基于其厚度、生物体积和粗糙度将生物被膜分类成5种结构形态类型。不同于Harris等研究,本文同样基于其厚度、生物体积和粗糙度将临床和环境来源的VP菌株生物被膜分为三种结构类型,结果显示,临床菌株易形致密且表面光滑和斑驳且表面粗糙的生物被膜,而环境菌株易形成致密且表面光滑和疏松且表面坑洼的生物被膜。
副溶血性弧菌是引起海产品污染的主要食源性病原体之一。本研究介绍的方法可以快速提供大量副溶血性弧菌生物被膜的定量结构数据,全自动系统的实现(微量滴定板扫描和图像处理)将从根本上放大可用于解读生物被膜结构特征,通过对临床和环境来源的副溶血性弧菌生物被膜进行结构表征,从而研究44株副溶血性弧菌生物被膜结构的多样性。该研究提供了对生物被膜结构的深入了解,这为今后防控和清除副溶血性弧菌生物被膜提供了理论基础。
References
| [1] | Osei-Adjei G, Huang XX, Zhang YQ. The extracellular proteases produced by Vibrio parahaemolyticus. World Journal of Microbiology and Biotechnology, 2018, 34(5): 68. |
| [2] | Pang R, Xie TF, Wu QP, Li YP, Lei T, Zhang JM, Ding Y, Wang J, Xue L, Chen MT, Wei XH, Zhang YX, Zhang SH, Yang XJ. Comparative genomic analysis reveals the potential risk of Vibrio parahaemolyticus isolated from ready-to-eat foods in China. Frontiers in Microbiology, 2019, 10: 186. |
| [3] | Letchumanan V, Chan KG, Lee LH. Vibrio parahaemolyticus:a review on the pathogenesis, prevalence, and advance molecular identification techniques. Frontiers in Microbiology, 2014, 5: 705. |
| [4] | Li L, Wong HC, Nong WY, Cheung MK, Law PTW, Kam KM, Kwan HS. Comparative genomic analysis of clinical and environmental strains provides insight into the pathogenicity and evolution of Vibrio parahaemolyticus. BMC Genomics, 2014, 15(1): 1135. |
| [5] | Song XY, Ma YJ, Fu JJ, Zhao AJ, Guo ZR, Malakar PK, Pan YJ, Zhao Y. Effect of temperature on pathogenic and non-pathogenic Vibrio parahaemolyticus biofilm formation. Food Control, 2017, 73: 485-491. |
| [6] | Flemming HC, Wingender J. The biofilm matrix. Nature Reviews Microbiology, 2010, 8(9): 623-633. |
| [7] | Flemming HC, Wingender J, Szewzyk U, Steinberg P, Rice SA, Kjelleberg S. Biofilms:an emergent form of bacterial life. Nature Reviews Microbiology, 2016, 14(9): 563-575. |
| [8] | Nadell CD, Bassler BL. A fitness trade-off between local competition and dispersal in Vibrio cholerae biofilms. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(34): 14181-14185. |
| [9] | Nadell CD, Drescher K, Foster KR. Spatial structure, cooperation and competition in biofilms. Nature Reviews Microbiology, 2016, 14(9): 589-600. |
| [10] | Hannig C, Follo M, Hellwig E, Al-Ahmad A. Visualization of adherent micro-organisms using different techniques. Journal of Medical Microbiology, 2010, 59(1): 1-7. |
| [11] | Azeredo J, Azevedo NF, Briandet R, Cerca N, Coenye T, Costa AR, Desvaux M, Di Bonaventura G, Hébraud M, Jaglic Z, Ka?ániová M, Kn chel S, Louren o A, Mergulh o F, Meyer RL, Nychas G, Sim es M, Tresse O, Sternberg C. Critical review on biofilm methods. Critical Reviews in Microbiology, 2017, 43(3): 313-351. |
| [12] | Pamp SJ, Sternberg C, Tolker-Nielsen T. Insight into the microbial multicellular lifestyle via flow-cell technology and confocal microscopy. Cytometry Part A, 2009, 75(2): 90-103. |
| [13] | Mosquera-Fernández M, Sanchez-Vizuete P, Briandet R, Cabo ML, Balsa-Canto E. Quantitative image analysis to characterize the dynamics of Listeria monocytogenes biofilms. International Journal of Food Microbiology, 2016, 236: 130-137. |
| [14] | Heydorn A, Nielsen AT, Hentzer M, Sternberg C, Givskov M, Ersboll BK, Molin S. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology, 2000, 146(10): 2395-2407. |
| [15] | Yang XM, Beyenal H, Harkin G, Lewandowski Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods, 2000, 39(2): 109-119. |
| [16] | Beyenal H, Donovan C, Lewandowski Z, Harkin G. Three-dimensional biofilm structure quantification. Journal of Microbiological Methods, 2004, 59(3): 395-413. |
| [17] | Yun MA, Yeon KM, Park JS, Lee CH, Chun J, Lim DJ. Characterization of biofilm structure and its effect on membrane permeability in MBR for dye wastewater treatment. Water Research, 2006, 40(1): 45-52. |
| [18] | Mueller LN, de Brouwer JF, Almeida JS, Stal LJ, Xavier JB. Analysis of a marine phototrophic biofilm by confocal laser scanning microscopy using the new image quantification software PHLIP. BMC Ecology, 2006, 6: 1. |
| [19] | Rieu A, Briandet R, Habimana O, Garmyn D, Guzzo J, Piveteau P. Listeria monocytogenes EGD-e biofilms:No mushrooms but a network of knitted chains. Applied and Environmental Microbiology, 2008, 74(14): 4491-4497. |
| [20] | Bridier A, Dubois-Brissonnet F, Boubetra A, Thomas V, Briandet R. The biofilm architecture of sixty opportunistic pathogens deciphered using a high throughput CLSM method. Journal of Microbiological Methods, 2010, 82(1): 64-70. |
| [21] | Mukherjee M, Menon NV, Liu X, Kang YJ, Gao B. Confocal laser scanning microscopy-compatible microfluidic membrane flow cell as a nondestructive tool for studying biofouling dynamics on forward osmosis membranes. Environmental Science & Technology Letters, 2016, 3(8): 303-309. |
| [22] | Han Q, Song XY, Zhang ZH, Fu JJ, Wang X, Malakar PK, Liu HQ, Pan YJ, Zhao Y. Removal of foodborne pathogen biofilms by acidic electrolyzed water. Frontiers in Microbiology, 2017, 8: 988. |
| [23] | Balsa-Canto E, Vilas C, López-Nú ez A, Mosquera-Fernández M, Briandet R, Cabo ML, Vázquez C. Modeling reveals the role of aging and glucose uptake impairment in L1A1 Listeria monocytogenes biofilm life cycle. Frontiers in Microbiology, 2017, 8: 2118. |
| [24] | Resat H, Renslow RS, Beyenal H. Reconstruction of biofilm images:combining local and global structural parameters. Biofouling, 2014, 30(9): 1141-1154. |
| [25] | Zhao AJ, Fu JJ, Song XY, Sun XH, Pan YJ, Zhao Y. Analysis of biofilm formation by pathogenic and no-pathogenic Vibrio parahaemolyticus at various temperatures and contact surfaces. Journal of Food Science and Biotechnology, 2018, 37(1): 7-14. (in Chinese) 赵爱静, 付娇娇, 宋雪迎, 孙晓红, 潘迎捷, 赵勇. 致病性与非致病性副溶血性弧菌在不同温度和接触材料表面生物被膜形成情况分析. 食品与生物技术学报, 2018, 37(1): 7-14. |
| [26] | Kadam SR, den Besten HMW, van der Veen S, Zwietering MH, Moezelaar R, Abee T. Diversity assessment of Listeria monocytogenes biofilm formation:impact of growth condition, serotype and strain origin. International Journal of Food Microbiology, 2013, 165(3): 259-264. |
| [27] | Díez-García M, Capita R, Alonso-Calleja C. Influence of serotype on the growth kinetics and the ability to form biofilms of Salmonella isolates from poultry. Food Microbiology, 2012, 31(2): 173-180. |
| [28] | Guilbaud M, Piveteau P, Desvaux M, Brisse S, Briandet R. Exploring the diversity of Listeria monocytogenes biofilm architecture by high-throughput confocal laser scanning microscopy and the predominance of the honeycomb-like morphotype. Applied and Environmental Microbiology, 2015, 81(5): 1813-1819. |
| [29] | Harris LG, Murray S, Pascoe B, Bray J, Meric G, Magerios L, Wilkinson TS, Jeeves R, Rohde H, Schwarz S, De Lencastre H, Miragaia M, Rolo J, Bowden R, Jolley KA, Maiden MCJ, Mack D, Sheppard SK. Biofilm morphotypes and population structure among Staphylococcus epidermidis from commensal and clinical samples. PLoS One, 2016, 11(3): e0151240. |
