陈垚1,2

 , 程启洪1, 郑爽1, 余雪花1, 任萍萍1
, 程启洪1, 郑爽1, 余雪花1, 任萍萍1 1. 重庆交通大学河海学院, 重庆 400074;
2. 重庆交通大学环境水利工程重庆市工程实验室, 重庆 400074
收稿日期:2019-05-22;修回日期:2019-08-07;网络出版日期:2019-12-05
基金项目:国家自然科学基金(51709024);重庆市研究生科研创新项目(CYS18219);重庆市基础科学与前沿技术研究项目(cstc2017jcyjAX0292);重庆市留创计划(cx2017065);重庆市教育委员会科学技术研究项目(KJ170540)
*通信作者:陈垚, Tel:+86-23-62652714;Fax:+86-23-62650204;E-mail:chenyao@cqjtu.edu.cn.
摘要:[目的] 为探究生物滞留系统干湿交替下环境因子对氮素功能微生物群落的影响。[方法] 应用高通量测序技术(Illumina MiSeq PE300),并以amoA和nirS功能基因为分子标记,对无植物型和植物型生物滞留系统在干湿交替下不同土壤空间位置(种植层、淹没层)的硝化和反硝化细菌的多样性和群落结构进行研究,并对微生物群落与环境因子的相互关系进行相关性分析。[结果] 微生物种群的功能基因存在显著的空间差异,相比淹没层,种植层的功能细菌更丰富。种植层的OTUs高于淹没层,而进水再湿润促使两种功能基因在种植层和淹没层的OTUs占比差异性增大。群落组成分析表明,amoA型硝化细菌和nirS型反硝化细菌的优势细菌门均为变形菌门(Proteobacteria)。虽然植物根系对氮素功能微生物的多样性指数影响不显著,但在属水平上,植物系统种植层的反硝化菌群种类高于淹没层,而无植物系统则刚好相反。CCA/RDA分析表明,土壤空间位置是影响硝化和反硝化菌群结构的最重要环境因子。[结论] 本研究证实干湿交替运行下生物滞留系统中的氮素功能微生物群落受土壤空间位置、水分含量和植物根系的共同调控,其机制有待进一步研究。
关键词:生物滞留系统干湿交替amoAnirS细菌多样性群落结构
Effects of drying-rewetting alternating on nitrogen-related microbial functional communities in bioretention systems
Yao Chen1,2

 , Qihong Cheng1, Shuang Zheng1, Xuehua Yu1, Pingping Ren1
, Qihong Cheng1, Shuang Zheng1, Xuehua Yu1, Pingping Ren1 1. School of River and Ocean Engineering, Chongqing Jiaotong University, Chongqing 400074, China;
2. Key Laboratory of Hydraulic & Waterway Engineering of the Ministry of Education, Chongqing Jiaotong University, Chongqing 400074, China
Received: 22 May 2019; Revised: 7 August 2019; Published online: 5 December 2019
*Corresponding author: Yao Chen, Tel: +86-23-62652714; Fax: +86-23-62650204; E-mail:chenyao@cqjtu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (51709024), by the Chongqing Scientific Research Innovation Program for Graduate Students (CYS18219), by the Science and Technology Research Program of Chongqing Science and Technology Commission (cstc2017jcyjAX0292), by the Venture & Innovation Support Program for Chongqing Overseas Returnees (cx2017065) and by the Science and Technology Research Program of Chongqing Municipal Education Commission (KJ170540)
Abstract: [Objective] This study aims to explore the effect of environmental factors on nitrogen-related microbial functional community structure of bioretention systems under the condition of drying-rewetting alternation. [Methods] Soil samples were collected from spatial distribution (planting layer, submerged layer) in vegetated and un-vegetated bioretention systems after drying and rewetting. The amoA and nirS functional genes were used as molecular marker. The diversity and community structure of nitrifying and denitrifying bacteria in different soil samples were studied by high-throughput sequencing technology named Illumina MiSeq PE300. Canonical correlation analysis (CCA) and redundancy analysis (RDA) were performed to assess the relationship between nitrogen-related microbial functional community structure and environmental factors was analyzed. [Results] The functional genes of microbial populations showed significant spatial differences. Nitrogen-related functional bacteria in the planting layer were more abundant and diverse than which in the submerged layer. However, the differences in proportion of operational taxonomic units (OTUs) between the two functional genes in planting layer and submerged layer were increased by rewetting. The results from the community composition analysis of nitrogen-related functional microbes showed that the dominant phylum of amoA-nitrifying and nirS-denitrifying bacteria were the Proteobacteria in all the soil samples. The root system of plants had no significant effect on the diversity index of nitrogen-related functional microbes, but in the plant system layer, the denitrifying bacteria species (at the genus level) were more than that in the submerged layer, and the opposite trend were showed in the plant-free system. The analysis of CCA and RDA showed that soil spatial distribution was the most important environmental factor on the distribution of nitrifying and denitrifying microbial communities. [Conclusion] The nitrogen-related functional microbial community in bioretention system under drying and rewetting alternation was controlled by the soil spatial distribution, water content and plant roots. However, the underlying reasons still await further investigation.
Keywords: bioretention systemdrying-rewetting alternatingamoAnirSbacterial diversitycommunity structure
雨水生物滞留系统由植物、土壤、微生物等多介质组成,降雨径流中的氮素进入系统后,经过一系列与多介质相关的生物地球化学过程而逐渐存储和衰减。在氮素的生物地球化学循环过程中,生物转化比非生物转化更重要,微生物是氮循环的驱动引擎,一方面使氮循环不被中断,另一方面维持生态系统的氮素平衡[1]。生物过程涉及同化、氨化、硝化、反硝化、厌氧氨氧化、硝酸盐异化还原成铵(DNRA)和固氮等作用[2-3],而固氮、氨化、硝化和反硝化作用是氮循环的4个主要过程,均由微生物所驱动。其中,硝化作用是连接固氮作用与反硝化作用的中间环节,不仅决定着植物对氮素的利用程度,并可直接造成硝酸盐淋洗和温室气体一氧化二氮(N2O)释放等一系列生态环境问题[4]。通过为反硝化作用提供底物,硝化和反硝化作用通常耦合发生,二者作用构成土壤氮素损失的最主要途径[5]。由此可见,硝化和反硝化作用共同决定着雨水生物滞留系统中氮素的平衡和归趋。生物滞留系统会因气候变化和降雨特性呈现出随机的干湿交替过程,而这种高度变异的土壤水分波动可能会对土壤微生物造成不规则和极端性的水分胁迫作用[6]。干湿交替形成的水分动力学过程可增强土壤有机质的矿化作用,促进土壤在长时间内维持较高的有机质[7],从而提高微生物活性及其多样性[8-10],还可通过影响土壤中的氧含量直接影响土壤微生物活性,进而影响硝化和反硝化过程[11-12]。干湿交替还会驱动植物根系构型特征发生演替,通过根系诱导作用改变土壤中的土壤氧动力学并使其与非根际土壤在空间上产生异质性,影响根际功能微生物的空间分布特性,调控氮素的微生物转化过程[13]。因此,干湿交替过程形成的土壤水分波动可直接或间接影响微生物的空间分布特性,调控功能菌群与氮素过程的耦合。
虽然在同样具有干湿交替的湿地、消落带等土壤系统研究中指出,土壤水分可调控土壤中固氮功能基因(nifH)、硝化功能基因(amoA、amoB)和反硝化功能基因(nirK、nirS、nosZ)等氮素功能基因丰度[14],如适当延长干旱持续时间可显著增加硝化和反硝化功能基因丰度[15]。但也有研究认为,土壤水分的增加才能提高反硝化功能基因丰度[16]。显然,即使在同样具有干湿交替且广泛研究的土壤系统中,土壤水分对氮素功能微生物的影响也未形成统一结论。而生物滞留系统固有的特殊性还会改变氮素发生迁移转化的时刻和区位,使氮素代谢功能菌在土壤中的存在形态及群落结构更加复杂[13]。这一特殊性使生物滞留系统不可完全照搬和借用其他土壤系统在微生物方面的研究结果与结论,需对干湿交替过程中氮素行为的微生态机制进行系统研究。但目前缺乏对生物滞留系统中氮素功能微生物群落特征的解析研究,尤其尚未掌握干湿交替对功能微生物群落的影响,仅有少量研究关注湿润期氮素功能微生物群落的时空分布特性[17]。
鉴于此,利用无植物和有植物的生物滞留系统,在干湿交替条件下收集不同空间的土壤样品,采用Illumina MiSeq PE300测序技术研究amoA和nirS功能基因菌群结构和多样性,对比分析干湿交替下硝化和反硝化功能菌群的时空演替情况,探析植物对功能菌群在时空分布特性上的调控作用。研究有助于进一步探明生物滞留系统干湿交替过程对氮素功能微生物多样性和菌群结构的影响。
1 材料和方法 1.1 样品采集 土样分别采自无植物生物滞留系统和栽种风车草的植物生物滞留系统(分别简称为无植物系统和植物系统,试验装置如图 1所示)在进水湿润后和排水落干2 d后种植层和淹没层土壤,土壤类型分别为沙壤土和细沙。在本研究采用的干湿交替周期下,种植层水分含量呈现波动状态,而位于储水层的淹没层,其水分含量基本处于饱和状态。采样时,在不干扰土壤层结构的基础上,利用PVC塑焊枪沿滤柱上的贯穿缝将焊缝热熔并小心剖开滤柱。每一土样采用五点采样法采集0-30 cm种植层或30-50 cm淹没层混合样,充分混匀后装袋并对土样编号(表 1)备用。
 |
| 图 1 装置示意图 Figure 1 Schematic diagram of devices. A: un-vegetated system; B: vegetated system. |
| 图选项 |
表 1. 土样采集编号一览表 Table 1. Soil sample collection number list
| Sample | Sample plot |
| s0_1 | Planting layer of un-vegetated system after rewetting |
| s0_2 | Submerged layer of un-vegetated system after rewetting |
| s1_1 | Planting layer of vegetated system after rewetting |
| s1_2 | Submerged layer of vegetated system after rewetting |
| g0_1 | Planting layer of un-vegetated system after drying |
| g0_2 | Submerged layer of un-vegetated system after drying |
| g1_1 | Planting layer of vegetated system after drying |
| g1_2 | Submerged layer of vegetated system after drying |
表选项
1.2 主要试剂和仪器 FastDNA Spin Kit for Soil土壤DNA提取试剂盒,MP Biomedicals公司;ABI GeneAmp? 9700型PCR仪,ABI公司;TransStart Fastpfu DNA Polymerase聚合酶,TransGen公司;NanoDrop 2000分光光度计,Thermo Scientific公司;高速冷冻离心机(J-251型),Beckman公司;ChemiDocTM XRS凝胶成像仪,Bio-Rad公司。
1.3 土壤微生物基因组DNA提取 DNA的提取采用土壤DNA提取试剂盒,按照说明书的提取步骤进行。将提取到的土壤微生物总DNA在冰上融化后,充分混匀并离心,取适量样品利用NanoDrop2000分光光度计检测DNA纯度和相对浓度,并利用1%琼脂糖凝胶电泳(电压5 V/cm,时间20 min)检测DNA的完整性。
1.4 土壤细菌功能基因PCR扩增 以稀释后的基因组DNA为模板,分别选用功能引物bamoA1F (5′-GGGGTTTCTACTGGTG GT-3′)和bamoA2R (5′-CCCCTCKGSAAAGCCT TCTTC-3′)[18],nirS4F (5′-TTCRTCAAGACSCAYC CGAA-3′)和nirS6R (5′-CGTTGAACTTRCCGGT- 3′)[19],DNA Polymerase聚合酶进行amoA基因和nirS基因的扩增。PCR反应体系为20 μL包括:5×FastPfu Buffer (4 μL)、2.5 mmol/L dNTPs (2 μL)、Forward Primer (5 μmol/L) (0.8 μL)、Reverse Primer (5 μmol/L) (0.8 μL)、FastPfu Polymerase (0.4 μL)、BSA (0.2 μL)、Template DNA (10 ng),并用ddH2O补至20 μL。反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min。
1.5 土壤基因测序 将扩增后的土壤基因组DNA送至上海美吉生物医药科技有限公司进行Illumina MiSeq PE300高通量测序。
1.6 数据分析 利用Usearch 7.0软件按照97%相似水平对非重复序列进行OTUs (operational taxonomic units)聚类,通过QIIME平台上RDP Classifier贝叶斯算法对OTUs代表序列进行生物信息统计分析,所用比对数据库为来源于GenBank整理的FRG/amoA-bacteria和FRG/nirS功能基因数据库(置信度阈值为0.7)[20]。利用Mothur软件包以97%为阈值对amoA和nirS基因序列划分分类单元,并计算序列随机抽样下各OTUs的Alpha多样性指数,并以抽到的序列数与其对应的对应性指数构建稀释曲线。其中,Sobs、Chao和ACE指数反映群落丰富度,其值越高表明群落物种的丰富度越高;Shannon和Simpson指数反映群落多样性,Shannon指数越高、Simpson指数越低表明群落物种的多样性越高;而Coverage指数反映群落覆盖度,其值越高表明微生物类群覆盖度越高。
2 结果和分析 2.1 测序结果质量分析 通过对土样的amoA和nirS基因测序,7个样品(其中编号为s1_2的土壤因DNA扩增产物目的条带太弱无法进行后续测序而舍弃)的amoA基因经优化筛选后有效序列的总数为103149,8个样品的nirS基因经优化筛选后有效序列的总数为115553。分析时选用相似度为97%的OTUs,按最小样本序列数抽平,amoA和nirS基因的样品分别产生97和854个OTUs。稀释曲线反映了样品的取样深度,可用以评价测序量是否足以覆盖所有类群[21]。从各样品基因的Shannon-Wiener稀释曲线(图 2)可知,曲线趋向平坦,说明测序数据量足够大。同时Coverage指数均达到99%以上,表明测序量能基本覆盖所有类群,可较好反映土壤样本细菌群落的真实情况。
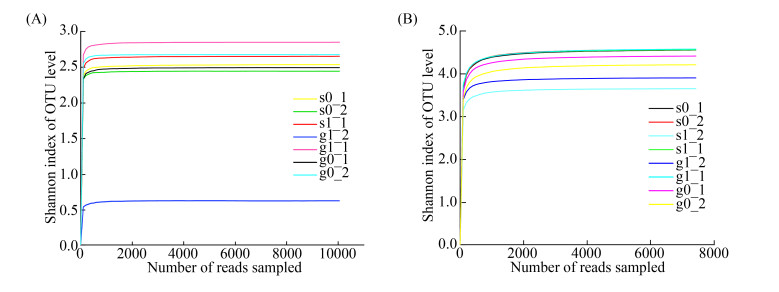 |
| 图 2 各土壤样品功能基因稀释曲线(相似度0.97) Figure 2 Dilution curves of genes of each soil sample (similarity 0.97). A: amoA gene; B: nirS gene. |
| 图选项 |
2.2 氮素功能细菌多样性及相关性分析
2.2.1 硝化功能菌群(amoA基因): 各样品amoA基因的Alpha多样性指数见表 2。综合Sobs、Chao和ACE指数可知,硝化功能菌群的丰富度受土壤空间的显著影响(P < 0.05),且种植层的丰富度远高于淹没层。而植物根系和土壤水分短暂变化对其丰富度有一定影响但均不显著。其中,植物系统和进水湿润会略微增加硝化功能微生物的丰富度。Shannon和Simpson指数表明,干湿交替下土壤空间位置、水分变化和植物根系对硝化功能菌群多样性指数的影响并不显著。
表 2. 不同土壤样品硝化功能菌群多样性指数 Table 2. Diversity indices of nitrifying functional bacteria in different soil samples
| Sample plot | Sobs index | Shannon index | Simpson index | ACE index | Chao index |
| Planting layer | 48.500±1.26a | 2.633±0.08a | 0.114±0.01a | 51.202±1.84a | 50.688±1.99a |
| Submerged layer | 25.333±1.76b | 1.917±0.65a | 0.329±0.22a | 27.993±3.54b | 25.667±2.03b |
| Un-vegetated system | 36±6.481a | 2.538±0.05a | 0.117±0.008a | 39.297±6.585a | 37.625±7.267a |
| Vegetated system | 42±8.083a | 2.044±0.708a | 0.325±0.226a | 43.868±8.974a | 43.083±8.761a |
| After drying | 37±5.802a | 2.163±0.515a | 0.269±0.169a | 39.479±5.442a | 37.438±5.781a |
| After rewetting | 40.667±9.404a | 2.544±0.060a | 0.122±0.005a | 43.625±10.445a | 43.333±10.682a |
| Results from ANOVA analysis and the different lowercase letters in the same column indicate significant difference at the 0.05 level. | |||||
表选项
在0.97相似度下,可获得各土壤样品的OTUs,并绘制分类水平上的Venn图,该图可显示多样品相同和各自特有的OTUs,以直观获得样品间OTUs的重叠情况,如图 3所示。对于无植物系统而言,排水落干后的种植层和淹没层共有22个相同OTUs,分别占据总OTUs的74.2%和45.2%;而进水湿润后的种植层和淹没层共有18个相同OTUs,分别占据总OTUs的77.7%和35.5%。在排水落干后的植物系统中,种植层和淹没层共有3个相同OTUs,分别占据总OTU数的58.5%和31.7%。相关性分析结果表明,种植层OTUs高于淹没层的OTUs,且进水湿润后会使硝化功能菌群在种植层和淹没层的OTUs差值增大。
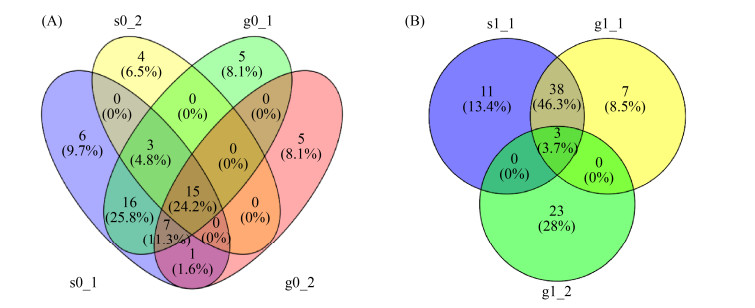 |
| 图 3 土壤样品amoA基因在OTUs分类水平上的Venn图 Figure 3 Venn diagram of amoA gene in soil samples at OTUs classification level. A: un-vegetated system; B: vegetated system. |
| 图选项 |
2.2.2 反硝化功能菌群(nirS基因): 各样品nirS基因的Alpha多样性指数见表 3。综合Sobs、Chao和ACE指数可知,反硝化功能菌群的群落丰富度受空间位置的显著影响(P < 0.05),且种植层反硝化功能菌群的丰富度远高于淹没层。而植物根系和土壤水分短暂变化对其丰富度有一定影响但并不显著。其中,排水落干过程可略微增加反硝化功能菌群的群落丰富度,无植物系统的反硝化功能菌群丰富度略高于植物系统。Shannon和Simpson指数表明,土壤空间位置、植物根系和水分变化对于反硝化功能菌群多样性指数的影响并不显著。
表 3. 不同模式下反硝化功能菌群多样性指数 Table 3. Diversity indices of denitrifying functional bacteria under different modes
| Sample plot | Sobs index | Shannon index | Simpson index | ACE index | Chao index |
| Planting layer | 400.750±9.578a | 4.525±0.037a | 0.028±0.003a | 489.220±20.574a | 484.783±17.994a |
| Submerged layer | 281.500±44.356b | 4.085±0.198a | 0.041±0.006a | 312.529±43.296b | 307.623±43.655b |
| Un-vegetated system | 375.000±13.241a | 4.437±0.083a | 0.035±0.005a | 419.558±26.265a | 417.850±80.643a |
| Vegetated system | 307.250 ±59.067a | 4.173±0.233a | 0.034±0.007a | 382.191±81.108a | 396.218±39.979a |
| After drying | 343.000±40.708a | 4.277±0.144a | 0.036±0.005a | 413.012±49.307a | 406.693±50.752a |
| After rewetting | 339.250±52.628a | 4.333±0.227a | 0.034±0.006a | 388.737±70.510a | 385.743±69.352a |
| Results from ANOVA analysis and the different lowercase letters in the same column indicate significant difference at the 0.05 level. | |||||
表选项
根据反硝化功能菌群在OTUs分类水平上的Venn图(图 4)可知,对于无植物系统而言,排水落干后的种植层和淹没层共有155个相同OTUs,分别占据总OTUs的51.2%和49.3%;而进水湿润后的种植层和淹没层共有161个相同OTUs,分别占据总OTUs的56.7%和48.8%。对于植物系统而言,排水落干后的种植层和淹没层共有94个相同OTUs,分别占总OTUs的64.0%和34.9%;而进水湿润后的种植层和淹没层共有73个相同OTUs,分别占据总OTUs的62.4%和28.8%。相关性分析结果表明,种植层OTUs高于淹没层的OTUs,植物根系可调控反硝化功能菌群的空间分布特性,造成种植层和淹没层的OTUs存在较大差异;而无植物系统中反硝化功能菌群的空间分布主要受水分变化的调控,特别是进水湿润可促使种植层和淹没层的OTUs差异性增大。
 |
| 图 4 土壤样品nirS基因在OTUs分类水平上的Venn图 Figure 4 Venn diagram of nirS genes in soil samples at OTUs classification level. A: un-vegetated system; B: vegetated system. |
| 图选项 |
2.3 干湿交替对氮素功能细菌群落结构的影响
2.3.1 硝化功能菌群结构: 7个土壤样品中共检测到硝化细菌门4个和属12个,如图 5所示。在所有样本中相对丰度占比均小于1%的物种归为others。在门级上,硝化菌群丰度最高的是变形菌门(Proteobacteria),相对丰度均超过60%。在属级上,相对丰度大于1%的有亚硝化螺菌属(Nitrosospira)、亚硝酸弧菌属(Nitrosovibrio)、变形菌β亚纲亚硝化细菌(Betaproteobacteria),以及亚硝化单胞菌目(Nitrosomonadales)亚硝化单胞菌科(Nitrosomonadaceae)下的亚硝化单胞菌属(Nitrosomonas)等,这些细菌均属于氨氧化细菌(ammonia oxidizing bacteria,AOB),参与硝化作用的第一步氧化过程。排水落干后,植物系统淹没层的优势硝化功能菌为亚硝化单胞菌属(unclassfied_o_Nitrosomonadales),其相对丰度高达87.9%。
 |
| 图 5 amoA基因微生物结构组成 Figure 5 Microorganism composition of amoA gene. A: phylum level; B: genus level. |
| 图选项 |
通过CCA分析土壤空间位置、水分含量和植物根系等环境因子与amoA型硝化菌群落之间的相互关系(图 6)。CCA分析表明,土壤空间位置(以土层中心距离地面高度表示)(r2=0.852,P=0.029)和植物根系(以根系长度表示,无植物系统根系长度视为0)(r2=0.830,P=0.049)与微生物群落结构具有显著相关性,而水分含量(r2=0.632,P=0.115)对微生物群落结构有一定影响但不显著。这是由于种植层氧浓度明显高于淹没层,更有利于硝化功能微生物的生长繁殖,从而造成不同空间位置对硝化菌群结构影响显著。干湿交替过程中硝化功能菌结构演替结果表明,土壤水分含量的增加在一定程度上可提高系统硝化功能菌的种群丰富度,这与相关研究结果一致[22-23]。分析认为,土壤水分含量的升高可通过降低水分胁迫压力[24],从而使硝化菌丰度及其优势菌群发生变化。但也有研究认为,土壤水分含量的进一步增加反而会降低氧气在土壤中的扩散能力,从而降低硝化菌群的丰度[25],导致丰度随含水量的增加而降低[26]。从研究结果来看,AOB丰度并未随土壤水分含量的进一步增加而显著降低,这主要是由于生物滞留系统的土壤含水率并未达到限制硝化功能微生物丰度的阈值。排水落干期间,光照引起的植物蒸腾作用会改变植物体内与大气的压强梯度,促使氧气通过植物组织到达根系,增强根系泌氧能力[27]。根系泌氧作用也会激发某类好氧微生物的活性,导致种植层硝化菌群种类更丰富,而淹没层则较为单一。同时,排水落干造成的水流势能也会将根系周围的氧气不断运移,从种植层移至淹没层,营造出适合硝化细菌生长的氧环境,从而表现为排水落干2 d后植物系统淹没层仍存在大量的亚硝化单胞菌属(unclassified_o_Nitrosomonadales)。上述结果充分说明,土壤空间位置、植物根系和水分含量等环境因子共同影响雨水生物滞留系统中amoA型硝化菌群结构。
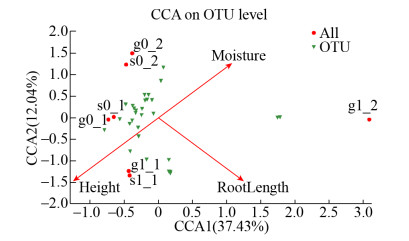 |
| 图 6 amoA基因的CCA相关性分析 Figure 6 CCA correlation analysis of amoA gene. |
| 图选项 |
2.3.2 反硝化功能菌群结构: 8个土壤样品中共检测到反硝化细菌门3个和属24个,如图 7所示。在门级上,反硝化菌群结构较为简单,丰度最高的为变形菌门(Proteobacteria),所有样品的相对丰度均达60%以上。在属级上,反硝化细菌的优势菌属有脱氯单胞菌属(Dechloromonas)、变形杆菌门下的属类(Proteobacteria)、β亚纲变形杆菌纲下的属类(Betaproteobacteria)、红环菌科下的属类(Rhodcyclaceae)、固氮弧菌属(Azoarcus)、红杆菌属(Rhodobacter)、产黄杆菌属(Rhodanobacter)和趋磁螺菌属(Magnetospirillum)。
 |
| 图 7 nirS基因微生物结构组成 Figure 7 Microorganism composition of nirS gene. A: phylum level; B: genus level. |
| 图选项 |
土壤空间位置、植物根系和水分含量均会影响反硝化菌群结构。从门级看,淹没层的变形菌门(Proteobacteria)的丰度总高于种植层。从属级看,由于低氧浓度更适合趋磁螺菌属(Magnetospirillum)的生长,所以其仅存于淹没层;植物系统的脱氯单胞菌属(Dechloromonas)和红环菌属(Rhodcyclaceae)的丰度均高于无植物系统;植物系统种植层的反硝化菌的种群丰富度高于淹没层,而无植物系统则刚好相反。为进一步研究环境因子与nirS型反硝化菌群间的相互关系,对其进行RDA分析(图 8)。RDA分析表明,第一、二排序轴解释率分别为50.38%和13.54%,说明排序轴能较好地反映出反硝化菌群与环境因子之间的相互关系。土壤空间位置(r2=0.966,P=0.032)和水分含量(r2=0.753,P=0.036)与微生物群落结构具有显著相关性,而植物根系(r2=0.907,P=0.053)对微生物群落结构的影响不显著。分析认为,淹没层氧浓度明显低于种植层,更有利于反硝化功能微生物的生存,从而导致土壤空间位置对反硝化菌群结构影响显著。植物系统可通过根系诱导作用改变土壤中的氧动力学和水分运移过程造成土壤特性的空间异质性,影响根际功能微生物的空间分布特性[28],形成反硝化菌群生长微环境,从而表现为种植层的菌群种类高于淹没层。而无植物时,由于淹没层水分含量远高于种植层,且排水落干形成的水分运移作用将硝化产物运移储存于淹没层,从而增加了该空间的反硝化菌群种类。可见,在无植物系统中,nirS型反硝化功能微生物群落结构主要受土壤水分含量影响。相关研究也表明,水分含量对以nirS基因为代表的反硝化菌群结构影响显著[29-30]。
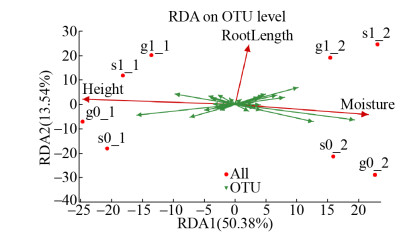 |
| 图 8 nirS基因的RDA相关性分析 Figure 8 RDA correlation analysis of nirS gene. |
| 图选项 |
3 结论 研究表明,干湿交替运行下生物滞留系统中的氮素功能微生物群落受土壤空间位置、水分含量和植物根系等环境因子的共同调控,但相关机制需进一步研究。其中,环境因子会影响硝化和反硝化功能微生物的群落丰富度及多样性,种植层的功能细菌比淹没层更丰富;不同环境因子与氮素功能微生物群落结构的相关性有所不同,土壤空间位置是影响硝化和反硝化菌群结构的最主要环境因子,而反硝化菌群结构还与水分含量显著相关。
References
| [1] | Zeng J, Yang LY, Xiao L, Yin DQ, Qin BQ. Biogeochemical cycling of nitrogen in lakes and the role of microorganisms in conversion of nitrogen compounds. Journal of Lake Sciences, 2007, 19(4): 382-389. (in Chinese) 曾巾, 杨柳燕, 肖琳, 尹大强, 秦伯强. 湖泊氮素生物地球化学循环及微生物的作用. 湖泊科学, 2007, 19(4): 382-389. DOI:10.3321/j.issn:1003-5427.2007.04.005 |
| [2] | Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [3] | Gold AC, Thompson SP, Piehler MF. Nitrogen cycling processes within stormwater control measures: A review and call for research. Water Research, 2019, 149: 578-587. DOI:10.1016/j.watres.2018.10.036 |
| [4] | He JZ, Zhang LM. Key processes and microbial mechanisms of soil nitrogen transformation. Microbiology China, 2013, 40(1): 98-108. (in Chinese) 贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制. 微生物学通报, 2013, 40(1): 98-108. |
| [5] | Zhu YG, Wang XH, Yang XR, Xu HJ, Jia Y. Key microbial processes in nitrous oxide emissions of agricultural soil and mitigation strategies. Environmental Science, 2014, 35(2): 792-800. (in Chinese) 朱永官, 王晓辉, 杨小茹, 徐会娟, 贾炎. 农田土壤N2O产生的关键微生物过程及减排措施. 环境科学, 2014, 35(2): 792-800. |
| [6] | Borken W, Matzner E. Reappraisal of drying and wetting effects on C and N mineralization and fluxes in soils. Global Change Biology, 2010, 15(4): 808-824. |
| [7] | Dosskey MG, Vidon P, Gurwick NP, Allan CJ, Duval TP, Lowrance R. The role of riparian vegetation in protecting and improving chemical water quality in streams. JAWRA Journal of the American Water Resources Association, 2010, 46(2): 261-277. DOI:10.1111/j.1752-1688.2010.00419.x |
| [8] | West AW, Sparling GP, Feltham CW, Reynolds J. Microbial activity and survival in soils dried at different rates. Soil Research, 1992, 30(2): 209-222. DOI:10.1071/SR9920209 |
| [9] | Denef K, Six J, Bossuyt H, Frey SD, Elliott ET, Merckx R, Paustian K. Influence of dry-wet cycles on the interrelationship between aggregate, particulate organic matter, and microbial community dynamics. Soil Biology and Biochemistry, 2001, 33(12/13): 1599-1611. |
| [10] | Denef K, Six J, Paustian K, Merckx R. Importance of macroaggregate dynamics in controlling soil carbon stabilization: short-term effects of physical disturbance induced by dry-wet cycles. Soil Biology and Biochemistry, 2001, 33(15): 2145-2153. DOI:10.1016/S0038-0717(01)00153-5 |
| [11] | 郑莹莹.干湿交替对土壤氮素转化及生物学特性的影响.东华大学学位论文, 2013. http://d.wanfangdata.com.cn/Thesis/Y2279457 |
| [12] | Pinay G, Clément JC, Naiman RJ. Basic principles and ecological consequences of changing water regimes on nitrogen cycling in fluvial systems. Environmental Management, 2002, 30(4): 481-491. DOI:10.1007/s00267-002-2736-1 |
| [13] | Chen Y, Cheng QH, Gan CJ, Ren PP. Research and prospect on processes of nitrogen removal in bioretention systems. Environmental Engineering, 2018, 36(7): 11-16. (in Chinese) 陈垚, 程启洪, 甘春娟, 任萍萍. 雨水生物滞留系统氮素过程研究现状与展望. 环境工程, 2018, 36(7): 11-16. |
| [14] | Morugán-Coronado A, García-Orenes F, McMillan M, Pereg L. The effect of moisture on soil microbial properties and nitrogen cyclers in Mediterranean sweet orange orchards under organic and inorganic fertilization. Science of the Total Environment, 2019, 655: 158-167. DOI:10.1016/j.scitotenv.2018.11.174 |
| [15] | Chen J, Nie YX, Liu W, Wang ZF, Shen WJ. Ammonia-oxidizing archaea are more resistant than denitrifiers to seasonal precipitation changes in an acidic subtropical forest soil. Frontiers in Microbiology, 2017, 8: 1384. DOI:10.3389/fmicb.2017.01384 |
| [16] | Liu RX, He JZ, Zhang LM. Response of nitrification/denitrification and their associated microbes to soil moisture change in paddy soil. Environmental Science, 2014, 35(11): 4275-4283. (in Chinese) 刘若萱, 贺纪正, 张丽梅. 稻田土壤不同水分条件下硝化/反硝化作用及其功能微生物的变化特征. 环境科学, 2014, 35(11): 4275-4283. |
| [17] | Chen XL, Peltier E, Sturm BSM, Young CB. Nitrogen removal and nitrifying and denitrifying bacteria quantification in a stormwater bioretention system. Water Research, 2013, 47(4): 1691-1700. DOI:10.1016/j.watres.2012.12.033 |
| [18] | Rotthauwe JH, Witzel KP, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/AEM.63.12.4704-4712.1997 |
| [19] | Vilar-Sanz A, Puig S, García-Lledó A, Trias R, Balaguer MD, Colprim J, Ba?eras L. Denitrifying bacterial communities affect current production and nitrous oxide accumulation in a microbial fuel cell. PLoS One, 2013, 8(5): e63460. DOI:10.1371/journal.pone.0063460 |
| [20] | Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Gl?ckner FO. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools. Nucleic Acids Research, 2013, 41(D1): D590-D596. |
| [21] | Oren A, Jiao ZW, Janbolat, Mu GL, Yang XR. Determination of bacteria diversity of degraded grassland in Xinyuan county by high-throughput sequencing technology. Microbiology China, 2017, 44(3): 545-553. (in Chinese) 吾尔恩·阿合别尔迪, 焦子伟, 江波拉提, 木古丽, 杨晓绒. 高通量测序技术分析新疆新源县过度放牧土壤细菌多样性. 微生物学通报, 2017, 44(3): 545-553. |
| [22] | Marcos MS, Bertiller MB, Cisneros HS, Olivera NL. Nitrification and ammonia-oxidizing bacteria shift in response to soil moisture and plant litter quality in arid soils from the Patagonian Monte. Pedobiologia, 2016, 59(1/2): 1-10. |
| [23] | Di HJ, Cameron KC, Podolyan A, Robinson A. Effect of soil moisture status and a nitrification inhibitor, dicyandiamide, on ammonia oxidizer and denitrifier growth and nitrous oxide emissions in a grassland soil. Soil Biology and Biochemistry, 2014, 73: 59-68. DOI:10.1016/j.soilbio.2014.02.011 |
| [24] | Hastings RC, Butler C, Singleton I, Saunders JR, McCarthy AJ. Analysis of ammonia-oxidizing bacteria populations in acid forest soil during conditions of moisture limitation. Letters in Applied Microbiology, 2010, 30(1): 14-18. |
| [25] | Belser LW. Population ecology of nitrifying bacteria. Annual Review of Microbiology, 1979, 33(1): 309-333. DOI:10.1146/annurev.mi.33.100179.001521 |
| [26] | Liu R, Hayden HL, Suter H, Hu HW, Lam SK, He JZ, Mele PM, Chen D. The effect of temperature and moisture on the source of N2O and contributions from ammonia oxidizers in an agricultural soil. Biology and Fertility of Soils, 2017, 53(1): 141-152. DOI:10.1007/s00374-016-1167-8 |
| [27] | Luo M, Huang JF, Liu YQ, Tong C. Progress in effects of root bioturbation on dissimilatory iron reduction in the rhizosphere of wetland plants. Acta Ecologica Sinica, 2017, 37(1): 156-166. (in Chinese) 罗敏, 黄佳芳, 刘育全, 仝川. 根系活动对湿地植物根际铁异化还原的影响及机制研究进展. 生态学报, 2017, 37(1): 156-166. |
| [28] | Hinsinger P, Bengough AG, Vetterlein D, Young IM. Rhizosphere: biophysics, biogeochemistry and ecological relevance. Plant and Soil, 2009, 321(1/2): 117-152. |
| [29] | Jha N, Saggar S, Giltrap D, Tillman R, Deslippe J. Soil properties impacting denitrifier community size, structure, and activity in New Zealand dairy-grazed pasture. Biogeosciences, 2017, 14(8): 4243-4253. |
| [30] | Hamonts K, Clough TJ, Stewart A, Clinton PW, Richardson AE, Wakelin SA, O'Callaghan M, Condron LM. Effect of nitrogen and waterlogging on denitrifier gene abundance, community structure and activity in the rhizosphere of wheat. FEMS Microbiology Ecology, 2013, 83(3): 568-584. DOI:10.1111/1574-6941.12015 |
