周盈1, 魏文静2, 江宝怡1, 李金山3, 毕利军1,4


1. 佛山科学技术学院生物医学工程系, 广东 佛山 528000;
2. 广东省结核病控制中心, 广东 广州 510630;
3. 华中农业大学农业微生物学国家重点实验室, 湖北 武汉 430070;
4. 中国科学院生物物理研究所核酸生物学重点实验室, 北京 100101
收稿日期:2019-05-05;修回日期:2019-07-07;网络出版日期:2019-11-19
基金项目:佛山科学技术学院科研启动项目
*通信作者:毕利军, Tel/Fax:+86-10-64888464;E-mail:blj@sun5.ibp.ac.cn.
摘要:[目的] START家族蛋白的突变或者错误表达使哺乳动物产生肾上腺皮质增生、乳腺癌和结肠癌等疾病;START家族蛋白是植物发育过程中重要的调节因子;尚未阐明START家族蛋白作为细菌必需基因的作用机制。结核分枝杆菌必需基因Rv0164属于START家族,功能未知,研究Rv0164作用机制将为START家族分子机制增添新理论。[方法] 生物信息学方法分析Rv0164序列特征;模式菌耻垢分枝杆菌中表达Rv0164并分析蛋白的细胞定位;Co-immunoprecipitation(Co-IP)方法垂钓Rv0164的相互作用蛋白,质谱鉴定互作蛋白,酵母双杂交和Pull down验证蛋白相互作用。[结果] Rv0164的N端17个氨基酸在分枝杆菌中不保守;Rv0164无信号肽;Rv0164定位在细胞质中,受蛋白降解机制调控,该机制在细菌生长平台期比对数期活性弱;N端缺失使Rv0164在平台期和对数期均不稳定;Rv0164结合多个胞内蛋白。[结论] Rv0164的N端肽段增加了蛋白的稳定性;Rv0164是一个胞内蛋白;Rv0164能够结合细菌生存必需蛋白。
关键词:START家族Rv0164蛋白定位蛋白-蛋白相互作用
Expression and protein interaction analysis of the START family protein Rv0164 in Mycobacterium smegmatis
Ying Zhou1, Wenjing Wei2, Baoyi Jiang1, Jinshan Li3, Lijun Bi1,4


1. Department of Biomedical Engineering, Foshan University, Foshan 528000, Guangdong Province, China;
2. Center for Tuberculosis Control of Guangdong Province, Guangzhou 510630, Guangdong Province, China;
3. State Key Laboratory of Agricultural Microbiology, Huazhong Agricultural University, Wuhan 430070, Hubei Province, China;
4. Key Laboratory of RNA Biology, Institute of Biophysics, Chinese Academy of Sciences, Beijing 100101, China
Received: 5 May 2019; Revised: 7 July 2019; Published online: 19 November 2019
*Corresponding author: Lijun Bi, Tel/Fax: +86-10-64888464; E-mail: blj@sun5.ibp.ac.cn.
Foundation item: Supported by the Start-Up Research Project of Foshan University
Abstract: [Objective] Mutations or abnormal expressions of START family proteins in mammals are the basis for diseases such as adrenal hyperplasia, breast cancer and colon cancer. START family proteins are key regulators of plant development. The mechanism of the essential START proteins in prokaryote is unclear. Rv0164 belongs to the START family and is an essential gene with unknown function in Mycobacterium tuberculosis. Therefore, exploring the function of Rv0164 will add a new theory to the molecular mechanisms of START family. [Methods] Bioinformatics method was used to analyze the sequence of Rv0164. Rv0164 was expressed in the model strain Mycobacterium smegmatis and analyzed the protein localization. The interaction proteins of Rv0164 were identified by Co-IP and mass spectrometry, then verified by yeast two hybrid and pull down. [Results] The N-terminal 17 residues of Rv0164 were not conserved in Mycobacterium. Rv0164 had no signal peptide. Rv0164 was localized in cytoplasm and regulated by some proteostasis mechanisms which were weaker in stationary phase than in logarithmic phase. Deletion of the N-terminus of Rv0164 produced protein instability in both stationary phase and logarithmic phase. Rv0164 interacted with several cytosolic proteins. [Conclusion] The N-terminus of Rv0164 enhances protein stability. Rv0164 localizes in cytoplasm and can bind essential proteins.
Keywords: START familyRv0164protein localizationprotein-protein interaction
1994年Clark等首次从小鼠中鉴定并命名了StAR蛋白(steroidogenic acute regulatory protein),该线粒体蛋白负责转运脂类,参与胆固醇代谢[1]。随后的研究表明具有类似结构的蛋白广泛分布在细菌、真菌、动物和植物中,不同的序列特征决定这类蛋白结合的底物类型[2]。Iyer等将具有类似结构的蛋白归为START (StAR-related lipid transfer domain)家族[3]。越来越多的研究表明START家族蛋白不仅能够作为脂质转运蛋白,还能够结合其他结构域发挥脂类感应功能。此外,在链霉菌中还发现了一类具有酶学活性的START家族蛋白——聚酮环化酶。哺乳动物START家族蛋白的突变或者错误表达产生肾上腺皮质增生、乳腺癌和结肠癌等疾病;START家族蛋白是植物发育过程的关键调节因子;尚未阐明START蛋白作为细菌必需基因的作用机制[1, 4-7]。研究细菌START蛋白将为START家族分子机制增添新理论,并为动物和植物生理和病理研究提供新视角。
不同物种之间START家族蛋白数量相差很大,真核生物一般有多个START蛋白,例如拟南芥有59个[3]。大部分原核生物仅有一个START蛋白,例如枯草芽孢杆菌和大肠杆菌[6-7]。比较特殊的是天蓝色链霉菌和结核分枝杆菌,分别有13个和18个START蛋白,链霉菌含有大量START蛋白可能与该菌产多种次生代谢物相关[3]。结核分枝杆菌的18个START蛋白中,Rv0088、Rv0164和Rv2186c是结核分枝杆菌生存必需基因,其他为非必需基因,NCBI标注Rv0088为甲酸氢化酶复合物亚基,其他标注为hypothetical protein。结核分枝杆菌不产次生代谢物,尚不清楚结核分枝杆菌基因组包含大量START基因的原因。
Rv0164首次发现于结核分枝杆菌的培养液上清,具有B细胞和T细胞免疫原性[8-9]。我们用结核分枝杆菌全蛋白芯片和病人血清开展分子标志物研究,发现Rv0164具有诊断标志物特性,并开发了一款结核病诊断试剂盒(专利号CN201310608295)。尝试结构生物学方法探讨Rv0164功能,但是Rv0164在大肠杆菌表达体系中蛋白得率低,并且无法获得较好的晶体衍射数据。MSMEG_0129是Rv0164在耻垢分枝杆菌中的同源蛋白,MSMEG_0129的三维结构与START家族聚酮环化酶相似,但是缺少环化反应的关键氨基酸[10],说明Rv0164和MSMEG_0129的功能与已知的细菌START蛋白作用机制不同。本研究分析了Rv0164的序列组成、细胞定位和胞内互作蛋白,结合组学数据库信息,提出Rv0164的可能作用机制。
1 材料和方法 1.1 材料 添加0.5%甘油和0.05%吐温80的LB培养基37 ℃振荡培养耻垢分枝杆菌。卡那霉素使用浓度为30 μg/mL (购自Amersco)、四环素(购自Amersco)使用浓度为0–80 ng/mL。GroEL抗体购自Santa Cruz公司,HRP标记的小鼠抗体购自Promega公司。Western及IP细胞裂解液和银染试剂盒购自碧云天公司。His抗体、FLAG抗体和FLAG抗体偶联磁珠购自Sigma公司。
重组质粒pMIND-Rv0164和pMIND-Rv0164 (–17aa)的构建策略为在酶切位点BamHⅠ和EcoR Ⅴ之间连入His标签的表达序列、EcoR Ⅴ和PacⅠ之间连入目标基因,His标签融合表达在目标基因N端。重组质粒pMV261-Rv0164-FLAG的构建策略为EcoR Ⅰ和Hind Ⅲ之间连入不含终止密码子的Rv0164基因,Hind Ⅲ和SalⅠ之间连入FLAG标签的表达序列,FLAG标签融合表达在Rv0164基因C端。重组质粒经序列测定验证正确后,电转化耻垢分枝杆菌野生菌Mycobacterium smegmatis MC2 155。
1.2 蛋白定位 耻垢分枝杆菌培养液经4 ℃离心分为培养上清和细胞沉淀两部分。沉淀部分超声破碎细胞,4 ℃离心去除细胞碎片,获得胞内总蛋白。培养上清通过0.22 μm过滤器,截留分子量3 kDa浓缩管进行浓缩。直接比色法测定上清和胞内总蛋白的蛋白浓度,30 μg蛋白进行10% SDS-PAGE分离,GroEL抗体、His抗体、HRP标记的小鼠抗体进行Western blotting检测。
1.3 免疫共沉淀 4 ℃离心收集菌体,Western及IP细胞裂解液重悬菌体。超声破碎细胞,4 ℃离心去除细胞碎片,获得胞内总蛋白。FLAG抗体偶联磁珠与胞内全蛋白4 ℃孵育2 h,随后裂解液和TBS (50 mmol/L Tris HCl,150 mmol/L NaCl,pH 7.4)分别清洗磁珠5次。两种溶液分别孵育柱材料获得洗脱蛋白:(1) 0.1 mol/L glycine HCl pH 3.5溶液;(2) SDS-PAGE上样缓冲液。
1.4 蛋白胶银染 10% SDS-PAGE分离免疫共沉淀获得的洗脱蛋白,去离子水清洗凝胶,按照银染试剂盒说明书进行蛋白胶银染。
1.5 质谱鉴定蛋白质 蛋白胶银染后,切下凝胶,依次用50% (V/V) K3[Fe(CN)6]Na2S2O3清洗、100 mmol/L NH4HCO3浸泡以及胰蛋白酶trypsin消化。获得的肽段混合物用C18反相柱在配置EASY-nL 1200系统的Q Exactive组合型四极杆Orbitrap质谱仪上进行蛋白质鉴定。MS/MS谱图用MASCOT引擎搜索NCBI的Mycobacterium smegmatis MC2 155和Mycobacterium tuberculosis H37Rv数据库。质谱鉴定参数设置为Peptide mass tolerance=20 ppm、MS/MS tolerance=0.1 Da、Enzyme=Trypsin、Missed cleavage=2、Fixed modification Carbamidomethyl (C)、Variable modification Oxidation (M)。
1.6 酵母双杂交 Matchmaker? Gold yeast two Hybrid试剂盒(购自TaKaRa)用于检测蛋白质相互作用。以结核分枝杆菌标准菌株H37Rv基因组为模板,扩增Rv0164和Rv3002c基因,无缝克隆方法分别插入pGBKT7和pGADT7载体的EcoR Ⅰ和BamH Ⅰ位点之间,重组质粒送测序公司验证插入序列。按照试剂盒流程转化酵母菌,重组酵母菌涂布质粒筛选培养基SD/-Trp/-Leu/获得含有双质粒的酵母菌。挑取SD/-Trp/-Leu/培养基上的酵母单菌落,无菌生理盐水制备菌悬液,2 μL菌液分别接种质粒筛选培养基和蛋白互作筛选培养基SD/-Trp/-Leu/-Ade/- His/X-alpha-Gal,30 ℃培养至菌苔出现。
1.7 Pull down 以结核分枝杆菌标准菌株H37Rv基因组为模板扩增Rv3002c基因,以载体pGEX6p-1为模板扩增GST基因,重叠PCR获得GST-Rv3002c融合基因,无缝克隆方法插入载体pET28a的NcoⅠ和Hind Ⅲ位点之间,GST-Rv3002c的C端融合表达His标签,重组质粒送测序公司验证插入基因。重组质粒转入大肠杆菌BL21(DE3),0.4 mmol/L IPTG 16 ℃诱导重组菌12 h,收集胞内可溶蛋白,Ni2+-NTA亲和层析柱富集GST-Rv3002c。不同浓度的咪唑溶液依次洗脱蛋白,SDS-PAGE检测洗脱蛋白中GST-Rv3002c的浓度和纯度。经缓冲液A (20 mmol/L Tris pH 8.0,NaCl 100 mmol/L,5%甘油)透析和截留分子量10 kDa浓缩管,获得大量高纯度GST-Rv3002c。
GST-Rv3002c与Glutathione Sepharose 4B beads 4 ℃孵育1 h,缓冲液A清洗柱材料5次,获得GST-Rv3002c柱材料。重组质粒pMV261- Rv0164-FLAG电转化野生菌M. smegmatis MC2 155获得Rv0164过表达菌株。37 ℃振荡培养Rv0164过表达菌,收集胞内全蛋白与GST-Rv3002c柱材料4 ℃孵育1 h,缓冲液A清洗柱材料5次,SDS-PAGE上样缓冲液洗脱柱材料上结合的所有蛋白,His抗体和FLAG抗体Western blotting检测洗脱蛋白。Pull down设置两组对照,第1组为GST-Rv3002c柱材料不与过表达菌胞内全蛋白孵育,其他实验步骤与Pull down样品相同,第2组设置样品为过表达菌的胞内全蛋白。
2 结果和分析 2.1 同源蛋白Rv0164和MSMEG_0129的序列特征 结核分枝杆菌是经呼吸道传染的病原菌,在生物安全实验室才能开展研究工作,因此常用非致病菌耻垢分枝杆菌作为模式菌研究分枝杆菌的蛋白功能以及筛选分子药靶的抑制剂,大部分同源蛋白在结核分枝杆菌和耻垢分枝杆菌中执行相同的生理功能[11-12]。同源蛋白Rv0164和MSMEG_0129的序列相似性高,各自被证明是细菌生存的必需基因[10, 13],表明两者的生理功能相似或者完全相同。
NCBI标注Rv0164有161个氨基酸,与上游基因Rv0163的转录方向相同,与邻近基因Rv0162和Rv0165的转录方向相反,Rv0163和Rv0164基因相距54个碱基,以上特性表明Rv0163和Rv0164可能属于一个操纵子。Roback等分析了474份结核分枝杆菌Microarray实验数据[14],发现Rv0163和Rv0164转录水平相关性低,表明Rv0163和Rv0164不属于一个操纵子。
MSMEG_0129与Rv0164的基因组环境相似。MSMEG_0129与上游基因MSMEG_0128的转录方向相同,与邻近基因MSMEG_0127和MSMEG_0130的转录方向相反,MSMEG_0129与上游基因MSMEG_0128间隔26个碱基。NCBI标注MSMEG_0129为144个氨基酸,比Rv0164少17个氨基酸。如图 1所示,序列比对表明MSMEG_0129第2个氨基酸对应于Rv0164第19个氨基酸,随后的6个氨基酸在分枝杆菌属4个菌种中保守,表明Rv0164的N端17个氨基酸在分枝杆菌属中不保守。
 |
| 图 1 不同分枝杆菌Rv0164同源蛋白的序列比对 Figure 1 Sequence alignment of Rv0164 homologous proteins. The identity between M. tuberculosis Rv0164 and M. smegmatis MSMEG_0129 is 87/144 (60%), M. tuberculosis Rv0164 and M. marinum MMAR_0407 129/153 (84%), M. tuberculosis Rv0164 and M. leprae ML2629c 116/156 (74%). |
| 图选项 |
为什么MSMEG_0129比Rv0164序列短?三种可能性,一种是MSMEG_0129序列标注有误,一种是Rv0164的N端是信号肽,第三种是Rv0164的N端有其他功能。将NCBI标注的MSMEG_0129阅读框前移3的倍数,MSMEG_0128和MSMEG_0129阅读框之间的序列能够翻译为RYARRMPL,该肽段与Rv0164的N端无相似性,因此排除了MSMEG_0129阅读框标注有误。
进一步通过SignalP分析Rv0164是否包含信号肽[15]。Mycolyl transferase 85A (基因名称fbpA,基因编号Rv3804c)是分泌蛋白,伴侣蛋白GroEL (基因名称groEL1,基因编号Rv3417c)是胞内蛋白,分别作为SignalP的阳性对照和阴性对照。一般信号肽区域的S值高,信号肽剪切位点的C值高;当序列中预测有多个信号肽剪切位点时,剪切位点只有一个,为S值降低和C值高的位点;D值是平均S值和平均C值的平均值,判断蛋白具有信号肽的Cutoff值为0.4[16]。Rv0164、FbpA、GroEL和MSMEG_0129的D值分别为0.391、0.437、0.129和0.137。MSMEG_0129的D值小于0.4,说明为胞内蛋白。Rv0164的D值与Cutoff值相差不大。如果Rv0164是分泌蛋白,信号肽剪切位点是第30位氨基酸,但是第11–30位氨基酸之间S值低,不符合信号肽特性,因此排除了Rv0164具有信号肽。以上结果表明Rv0164的非保守N端多肽可能有其他的功能(图 2)。
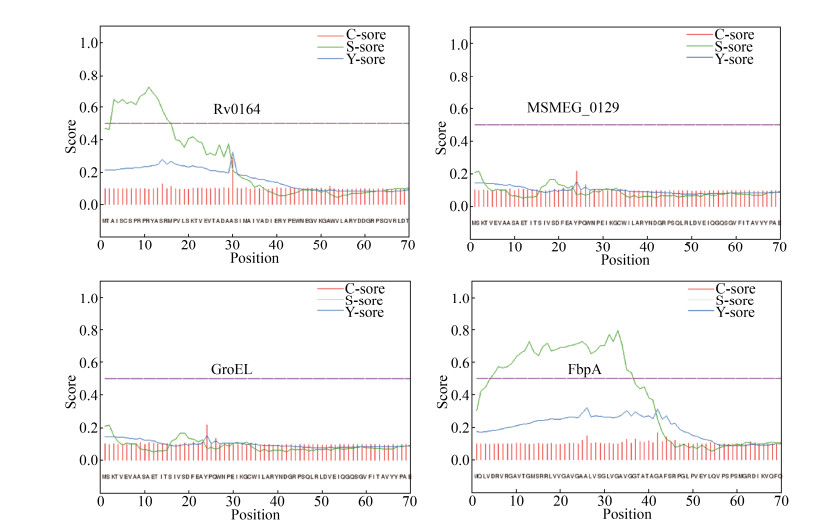 |
| 图 2 SignalP分析蛋白的信号肽 Figure 2 Signal peptide prediction by SignalP. The cytosolic protein GroEL and secretory protein FbpA were set as negative and positive control, respectively. |
| 图选项 |
2.2 Rv0164是胞内蛋白 分枝杆菌表达体系通常使用四环素或者乙酰胺作为诱导剂,或者通过高温培养诱导hsp60启动子超量表达目标基因[17-18]。本实验利用四环素诱导表达质粒pMIND在耻垢分枝杆菌中表达Rv0164,Rv0164的N端融合表达His标签用于Western blotting检测。
检测细菌生长对数期和平台期时Rv0164在胞内和胞外的分布,GroEL作为上样量和胞内定位的对照蛋白。GroEL是分枝杆菌组成性表达的胞内蛋白,在培养上清中也能检测到GroEL的可能原因是细菌裂解[19]。如图 3-A所示,对数期胞内蛋白中检测到Rv0164而胞外无Rv0164杂交条带,说明Rv0164在耻垢分枝杆菌中表达时是一个胞内蛋白,与Rv0164不含信号肽是一致的。平台期胞内蛋白和培养上清均未检测到Rv0164蛋白,可能原因是对数期添加的诱导剂到平台期时失效。
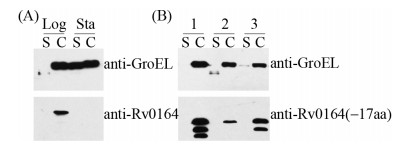 |
| 图 3 Rv0164和其截断体在耻垢分枝杆菌中的表达和定位 Figure 3 Expression and localization of Rv0164 and its truncated mutant in Mycobacterium smegmatis. A: Expression of Rv0164 in the strain M. smegmatis MC2 155/pMIND-Rv0164 at OD600=0.5 (Log) and OD600=2 (Sta) was detected by Western blotting. B: Expression of the N-terminal 17 residue truncated mutant in the strain M. smegmatis MC2 155/pMIND-Rv0164(–17aa); logarithmic phase culture of 3 colonies was detected by Western blotting. Secreted proteins in the culture supernatant and cytosolic proteins in the cell cytoplasm are labeled as S and C, respectively. GroEL was used as an internal control. |
| 图选项 |
检测缺失N端非保守肽段的Rv0164截短体的细胞定位,仅胞内蛋白样品中检测到Rv0164 (–17aa),而且不同的单菌落出现不同的杂交带型(图 3-B)。其中单菌落2的带型单一,并且该条带在单菌落1和3中重复出现,表明该条带为Rv0164 (–17aa)。单菌落1和3分别比单菌落2多了2条和1条略小的杂交条带,而His标签融合表达在基因N端,说明Rv0164(–17aa)的C端发生了部分降解。以上结果表明Rv0164(–17aa)的蛋白稳定性低于全长Rv0164,说明Rv0164的N端非保守肽段与蛋白稳定性相关。
2.3 Rv0164蛋白在平台期比对数期稳定 添加不同浓度的诱导剂,检测对数期Rv0164的动态变化特性。无诱导剂时,诱导菌M. smegmatis MC2 155/pMIND-Rv0164的胞内蛋白在检测时间内无Rv0164 (图 4),说明pMIND质粒无泄漏表达。20 ng/mL诱导剂时,除了第2小时和第3小时,其他时间点未检测到胞内Rv0164;40 ng/mL诱导剂时,除了第6小时,其他时间点均可检测到胞内Rv0164;80 ng/mL诱导剂时,6个时间点均可检测到胞内Rv0164;每个诱导剂浓度的检测时间内Rv0164呈现先增加后降低的变化趋势(图 4-A)。而诱导剂浓度越高,pMIND质粒表达目标蛋白的数量越多[17]。以上数据说明除了诱导剂的蛋白增量效应,同时存在胞内蛋白减量效应,低浓度诱导剂产生蛋白的速度低于胞内蛋白减量效应。
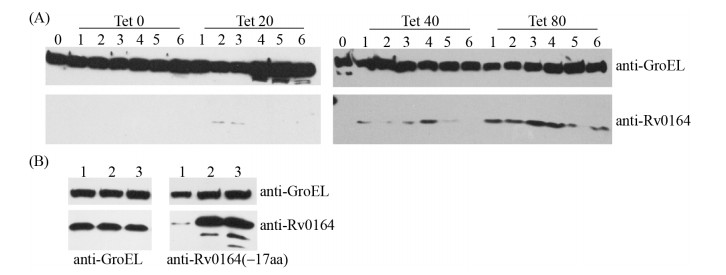 |
| 图 4 胞内Rv0164蛋白的动态变化 Figure 4 Dynamic model of intracellular Rv0164. A: Cytoplasmic Rv0164 in the strain M. smegmatis MC2 155/ pMIND-Rv0164 was detected by Western blotting using a monoclonal anti-polyHistidine antibody; logarithmic bacterial cells were applied with different concentration of tetracycline (20 ng/mL, 40 ng/mL and 80 ng/mL); samples were harvested at 1 hour intervals. B: Stationary bacterial cells (OD600=2) of the strains M. smegmatis MC2 155/pMIND-Rv0164 and M. smegmatis MC2 155/pMIND-Rv0164(–17aa) were applied with 80 ng/mL tetracycline and samples were harvested at 1 hour intervals. GroEL was used as an internal control. |
| 图选项 |
平台期诱导菌补加诱导剂80 ng/mL,检测平台期胞内Rv0164的动态变化特性。如图 4-B所示,3 h内Rv0164杂交信号相同,说明平台期添加诱导剂产生的Rv0164数量比较稳定,此时诱导剂的蛋白增量效应与胞内蛋白减量效应趋于平衡。
相同的策略检测平台期胞内Rv0164(–17aa)的动态变化特性。Rv0164(–17aa)诱导菌在补加诱导剂的第2小时、第3小时比第1小时的杂交信号强,并且随着培养时间的延长,杂交带型越多,说明诱导剂产生蛋白的速度高于胞内蛋白减量效应,较小的杂交条带是Rv0164(–17aa)的C端逐步降解产生。以上结果表明全长Rv0164在平台期比对数期稳定,Rv0164截短体在平台期也不稳定。
2.4 垂钓Rv0164互作蛋白 前面实验结果表明平台期时全长Rv0164更稳定,因此选用平台期Rv0164表达菌进行垂钓互作蛋白实验。平台期M. smegmatis MC2 155/pMV261- Rv0164-FLAG菌液,一部分继续37 ℃培养3 h,另一部分转移到42 ℃培养3 h,分别收集上清蛋白和胞内蛋白,SDS-PAGE和考马斯亮蓝染色检测全部蛋白。如图 5-A所示42 ℃比37 ℃培养上清中蛋白种类更多,并且42 ℃胞内和上清蛋白的条带相似性比37 ℃高,可能原因是高温使细菌受到损伤,部分细菌裂解,胞内蛋白溢出。42 ℃胞内蛋白与37 ℃胞内蛋白相比,蛋白种类无显著差异,蛋白丰度发生变化,例如图 5-A箭头1和2所示的蛋白条带。以上结果表明42 ℃培养使细菌胞内和胞外蛋白发生了变化。
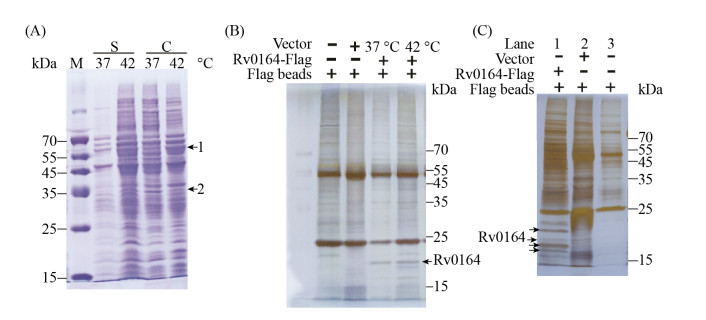 |
| 图 5 Co-IP垂钓Rv0164互作蛋白 Figure 5 Co-immunoprecipitation (co-IP) of Rv0164 interacting proteins. A: Stationary bacterial cells of the strain M. smegmatis MC2 155/pMV261-Rv0164-FLAG was cultured at 37 ℃ or 42 ℃ for 3 hours; SDS-PAGE and Coomassie Blue Staining of culture supernatant (S) and cytoplasmic (C) proteins. B–C: Immunoprecipitation and silver staining of Flag-tagged Rv0164 from cell lysates; elution buffer was loading buffer (B) or glycine HCl (C). Rv0164-Flag and interacting protein candidates are labeled with an arrow in B and C. |
| 图选项 |
用FLAG抗体偶联柱材料分别与37 ℃和42 ℃胞内蛋白孵育,富集Rv0164以及能够与Rv0164相互作用的蛋白,蛋白上样缓冲液破坏抗体与柱材料间的偶联作用,洗脱柱材料上结合的所有蛋白。设置两组阴性对照,一组是柱材料未与蛋白孵育,一组是携带pMV261的重组菌的胞内蛋白与柱材料孵育。如图 5-B所示,分子量为55 kDa和25 kDa的高丰度蛋白分别为FLAG抗体的重链和轻链,与阴性对照相比,37 ℃胞内样品富集蛋白在15 kDa和25 kDa之间多了一个蛋白条带,切割该区域进行质谱分析,质谱分析的肽段覆盖Rv0164序列55.3%。与阴性对照相比,42 ℃胞内样品在15 kDa和25 kDa之间多了几个蛋白条带,其中丰度最高的蛋白条带在37 ℃样品中也存在,切割该区域凝胶质谱鉴定蛋白组成,质谱分析的肽段覆盖Rv0164序列43.5%。以上结果表明42 ℃培养3 h的方案能够获得较多的Rv0164互作蛋白。
2.5 质谱鉴定互作蛋白 蛋白上样缓冲液洗脱柱材料上的富集蛋白时,洗脱液中含有较多FLAG抗体(图 5-B)。glycine HCl溶液作为洗脱溶液时,洗脱蛋白中FLAG抗体明显减少,Rv0164互作蛋白条带更加清晰(图 5-C)。切割互作蛋白中的3条主带(图 5-C箭头标注),质谱鉴定蛋白组成。
质谱结果搜索数据库获得互作蛋白列表,大部分蛋白的信号值和肽段覆盖度较低,设定筛选参数排除假阳性蛋白。互作蛋白中hypothetical protein LI98的Intensity值最高(约为8×109),取最高信号值的0.1%设为阈值排除假阳性蛋白,获得280个较高信号值的蛋白。其次设定肽段覆盖度阈值为40%,280个蛋白中有24个满足筛选条件。为了进一步提高互作蛋白的可信度,进行了第2次独立的垂钓实验。第2次获得的互作蛋白列表中满足Intensity值大于8×106的蛋白有211个,同时满足肽段覆盖度大于40%的蛋白有24个。2次独立实验中均满足筛选条件的互作蛋白有7个,其中3个是必需基因(表 1)。
表 1. Rv0164互作蛋白 Table 1. Rv0164 interacting proteins
| Sequence coverage/% (Twice) | kDa1 | Sequence length2 | MSMEG3 | Product | Rv4 | Essential5 | Reference | |
| 60.0 | 42.9 | 18.3 | 170 | MSMEG_2373 | Acetolactate synthase small subunit | Rv3002c | Yes | [20] |
| 48.8 | 42.4 | 17.6 | 170 | MSMEG_4940 | ATP synthase B chain AtpF | Rv1306 | Yes | NCBI |
| 54.0 | 54.0 | 16.7 | 150 | MSMEG_1557 | 30S ribosomal protein S9 RpsI | Rv3442c | Yes | NCBI |
| 56.6 | 42.2 | 18.3 | 166 | MSMEG_6913 | Transcriptional regulatory protein | Rv0042c | No | NCBI |
| 49.7 | 41.0 | 18.5 | 161 | MSMEG_0636 | Hypothetical protein | Rv0310c | No | NCBI |
| 48.0 | 40.5 | 21.1 | 200 | MSMEG_6082 | Beta-carbonic anhydrase CanB | Rv3588c | No | NCBI |
| 44.1 | 87.7 | 21.6 | 195 | MSMEG_4891 | Alkyl hydroperoxide reductase C protein AhpC | Rv2428 | No | NCBI |
| 1: Molecular weight. 2: Number of amino acid residues. 3: Gene number in M. smegmatis. 4: Gene number in M. tuberculosis. 5: Experiment data from [13]. | ||||||||
表选项
2.6 Rv0164结合Rv3002c Rv0164的互作蛋白中,有3个是结核分枝杆菌生存必需的蛋白(Rv3002c、Rv1306和Rv3442c),仅Rv3002c有研究报道[20]。酵母双杂交和Pull down验证Rv0164和Rv3002c的蛋白-蛋白相互作用。如图 6-A所示,酵母菌能够在不含Trp和Leu的基本培养基上生长,说明pGBKT7或者其携带外源基因的重组质粒、pGADT7或者其携带外源基因的重组质粒同时导入了酵母菌。当pGBKT7和pGADT7携带的外源基因发生蛋白相互作用时,激活报告基因的转录,产生ADE2、HIS3和MEL1,使酵母菌能够在不含Ade和His的基本培养基上生长,酵母菌分泌的MEL1在含有底物X-alpha-Gal的培养基中使菌苔呈蓝色。阳性对照中P53和SV40 large T-antigen是已知的一组互作蛋白,重组酵母菌在蛋白互作筛选培养基上呈深蓝色。阴性对照中Lamin和SV40 large T-antigen是已知的非互作蛋白,重组酵母菌在蛋白互作筛选培养基上无明显生长。pGBKT7和pGADT7质粒仅携带Rv0164或者Rv3002c时,重组酵母菌在蛋白互作筛选培养基上无明显生长,说明Rv0164或者Rv3002c自身不能激活报告基因的转录。pGBKT7-Rv0164和?pGADT7-Rv3002c使重组酵母菌在蛋白互作筛选培养基上生长,菌苔呈淡蓝色,说明Rv0164和Rv3002c能够发生蛋白相互作用。
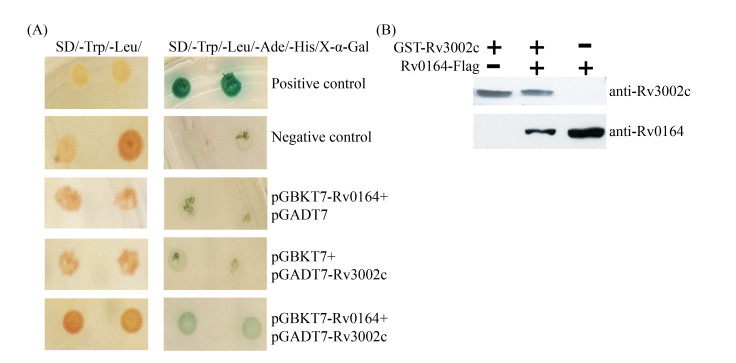 |
| 图 6 Rv0164结合Rv3002c Figure 6 Interaction between Rv0164 and Rv3002c. A: Yeast Two-Hybrid; Positive control: pGBKT7-P53?+ ?pGADT7-T; Negative control: pGBKT7-Lam?+ pGADT7-T. B: GST Pull down. |
| 图选项 |
Pull down检测中GST标签与柱材料的高亲和力使融合蛋白GST-Rv3002c被固定在柱材料上,Rv3002的互作蛋白通过蛋白-蛋白相互作用也将结合在柱材料上。如图 6-B中间泳道,Pull down样品的洗脱液中有Rv0164和Rv3002,而2个对照样品中仅检测到Rv3002 (左侧泳道)或者Rv0164 (右侧泳道),说明Rv0164和Rv3002c有相互作用。
3 讨论 结构生物学分析表明Rv0164同源蛋白是START家族的无酶活蛋白[10]。NCBI组学数据表明饥饿培养24 h和96 h[21]、感染小鼠巨噬细胞24 h以及双氧水处理24 h时结核分枝杆菌Rv0164表达量降低[22],说明Rv0164参与细菌胁迫应答。START家族的主要作用机制是:(1)作为脂质转运蛋白,在真核细胞内的不同膜结构之间转运脂类物质[23];(2)作为脂类感应蛋白,一般为多结构域蛋白,由START结构域结合脂类引起蛋白构象变化而激活其他结构域或者改变蛋白的细胞定位[24];(3)聚酮环化酶活性[5, 25]。仅脂类感应功能同时满足无酶活和参与胁迫应答的特性,表明Rv0164可能参与脂类感应。
脂类感应需要信号的接收和传递,一般由2个结构域执行。例如植物HD-Zip转录因子的START结构域结合配体后(即信号接收)调控其转录因子活性(即信号传递)[4]。Rv0164全长161个氨基酸,START功能域覆盖128个氨基酸(第25–152位),是单一结构域蛋白,能够结合配体完成脂类感应的信号接收,需要下游效应蛋白改变细菌的生理状态,例如改变蛋白的数量和种类,从而完成信号传递。Rv0164是必需基因,下游效应蛋白可能也是必需基因。Co-IP方法垂钓Rv0164的相互作用蛋白(图 5),7个互作蛋白中有3个是必需基因,分别属于蛋白质合成(MSMEG_2373和MSMEG_1557)和能量合成通路(MSMEG_4940),说明上述3个必需基因可能作为Rv0164的下游效应蛋白。
尽管在细菌培养上清发现了Rv0164,尚无研究报道该蛋白是细菌主动排出或者由于细菌裂解而释放到胞外。分枝杆菌转运系统的底物特征是:(1) Sec分泌系统,分泌蛋白的N端有信号肽;(2) Twin-arginine transporter,分泌蛋白的信号肽有2个精氨酸;(3)Ⅶ型分泌系统,分泌蛋白的C末端有T7S motif (YXXXD/E) [26-27]。SignalP分析发现Rv0164无信号肽(图 2),Rv0164也不具有上述分泌系统的特征序列。Western blotting进一步证实Rv0164定位在胞内(图 3)。以上结果表明Rv0164是一个胞内蛋白。
综合数据库信息和本研究结果,推测Rv0164的脂类感应作用机制是:细菌面临环境压力时Rv0164接收信号,通过蛋白互作影响下游效应蛋白,下游效应蛋白调控胞内大分子数量,使细菌适应环境变化。真核生物START家族蛋白的脂类感应功能通常由一个多结构域蛋白完成信号接收和传递,Rv0164与下游效应蛋白组成的“双元件”体系使得不同的下游效应蛋白可能产生不同的调控效果,值得进一步研究。
References
| [1] | Clark BJ, Wells J, King SR, Stocco DM. The purification, cloning, and expression of a novel luteinizing hormone-induced mitochondrial protein in MA-10 mouse Leydig tumor cells. Characterization of the steroidogenic acute regulatory protein (StAR). The Journal of Biological Chemistry, 1994, 269(45): 28314-28322. |
| [2] | Wong LH, ?opi? A, Levine TP. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences, 2017, 42(7): 516-530. DOI:10.1016/j.tibs.2017.05.001 |
| [3] | Iyer LM, Koonin EV, Aravind L. Adaptations of the helix-grip fold for ligand binding and catalysis in the START domain superfamily. Proteins, 2001, 43(2): 134-144. DOI:10.1002/1097-0134(20010501)43:2<134::AID-PROT1025>3.0.CO;2-I |
| [4] | Schrick K, Bruno M, Khosla A, Cox PN, Marlatt SA, Roque RA, Nguyen HC, He CW, Snyder MP, Singh D, Yadav G. Shared functions of plant and mammalian StAR-related lipid transfer (START) domains in modulating transcription factor activity. BMC Biology, 2014, 12: 70. DOI:10.1186/s12915-014-0070-8 |
| [5] | Zhang Z, Pan HX, Tang GL. New insights into bacterial type Ⅱ polyketide biosynthesis. F1000Research, 2017, 6: 172. DOI:10.12688/f1000research.10466.1 |
| [6] | Stark JL, Mercier KA, Mueller GA, Acton TB, Xiao R, Montelione GT, Powers R. Solution structure and function of YndB, an AHSA1 protein from Bacillus subtilis. Proteins, 2010, 78(16): 3328-3340. DOI:10.1002/prot.22840 |
| [7] | Zhang YL, Inouye M. RatA (YfjG), an Escherichia coli toxin, inhibits 70S ribosome association to block translation initiation. Molecular Microbiology, 2011, 79(6): 1418-1429. DOI:10.1111/j.1365-2958.2010.07506.x |
| [8] | Lim JH, Kim HJ, Lee KS, Jo EK, Song CH, Jung SB, Kim SY, Lee JS, Paik TH, Park JK. Identification of the new T-cell-stimulating antigens from Mycobacterium tuberculosis culture filtrate. FEMS Microbiology Letters, 2004, 232(1): 51-59. DOI:10.1016/S0378-1097(04)00018-7 |
| [9] | Eweda G, Suzuki D, Nagata T, Tsujimura K, Koide Y. Identification of murine T-cell epitopes on low-molecular-mass secretory proteins (CFP11, CFP17, and TB18.5) of Mycobacterium tuberculosis. Vaccine, 2010, 28(29): 4616-4625. DOI:10.1016/j.vaccine.2010.04.079 |
| [10] | Zheng SP, Zhou Y, Fleming J, Zhou YF, Zhang MT, Li SL, Li HL, Sun BQ, Liu W, Bi LJ. Structural and genetic analysis of START superfamily protein MSMEG_0129 from Mycobacterium smegmatis. FEBS Letters, 2018, 592(8): 1445-1457. DOI:10.1002/1873-3468.13024 |
| [11] | Yuan TA, Sampson NS. Hit generation in TB drug discovery: from genome to granuloma. Chemical Reviews, 2018, 118(4): 1887-1916. DOI:10.1021/acs.chemrev.7b00602 |
| [12] | Zhang B, Li J, Yang XL, Wu LJ, Zhang J, Yang Y, Zhao Y, Zhang L, Yang XN, Yang XB, Cheng X, Liu ZJ, Jiang B, Jiang HL, Guddat LW, Yang HT, Rao ZH. Crystal structures of membrane transporter MmpL3, an Anti-TB drug target. Cell, 2019, 176(3): 636-648. |
| [13] | Griffin JE, Gawronski JD, Dejesus MA, Ioerger TR, Akerley BJ, Sassetti CM. High-resolution phenotypic profiling defines genes essential for mycobacterial growth and cholesterol catabolism. PLoS Pathogens, 2011, 7(9): e1002251. DOI:10.1371/journal.ppat.1002251 |
| [14] | Roback P, Beard J, Baumann D, Gille C, Henry K, Krohn S, Wiste H, Voskuil MI, Rainville C, Rutherford R. A predicted operon map for Mycobacterium tuberculosis. Nucleic Acids Research, 2007, 35(15): 5085-5095. DOI:10.1093/nar/gkm518 |
| [15] | Petersen TN, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nature Methods, 2011, 8(10): 785-786. DOI:10.1038/nmeth.1701 |
| [16] | Gomez M, Johnson S, Gennaro ML. Identification of secreted proteins of Mycobacterium tuberculosis by a bioinformatic approach. Infection and Immunity, 2000, 68(4): 2323-2327. DOI:10.1128/IAI.68.4.2323-2327.2000 |
| [17] | Blokpoel MCJ, Murphy HN, O'Toole R, Wiles S, Runn ESC, Stewart GR, Young DB, Robertson BD. Tetracycline-inducible gene regulation in mycobacteria. Nucleic Acids Research, 2005, 33(2): e22. |
| [18] | Stover CK, de la Cruz VF, Fuerst TR, Burlein JE, Benson LA, Bennett LT, Bansal GP, Young JF, Lee MH, Hatfull GF, Snapper SB, Barletta RG, Jacobs Jr WR, Bloom BR. New use of BCG for recombinant vaccines. Nature, 1991, 351(6326): 456-460. DOI:10.1038/351456a0 |
| [19] | Stokes RW. Mycobacterium tuberculosis Hsp60 as a key virulence factor in tuberculosis//Henderson B, Pockley AG. Cellular Trafficking of Cell Stress Proteins in Health and Disease. Dordrecht: Springer Netherlands, 2012: 243-258. |
| [20] | Singh V, Chandra D, Srivastava BS, Srivastava R. Biochemical and transcription analysis of acetohydroxyacid synthase isoforms in Mycobacterium tuberculosis identifies these enzymes as potential targets for drug development. Microbiology, 2011, 157(1): 29-37. |
| [21] | Betts JC, Lukey PT, Robb LC, McAdam RA, Duncan K. Evaluation of a nutrient starvation model of Mycobacterium tuberculosis persistence by gene and protein expression profiling. Molecular Microbiology, 2002, 43(3): 717-731. DOI:10.1046/j.1365-2958.2002.02779.x |
| [22] | Schnappinger D, Ehrt S, Voskuil MI, Liu Y, Mangan JA, Monahan IM, Dolganov G, Efron B, Butcher PD, Nathan C, Schoolnik GK. Transcriptional adaptation of Mycobacterium tuberculosis within macrophages: insights into the phagosomal environment. The Journal of Experimental Medicine, 2003, 198(5): 693-704. DOI:10.1084/jem.20030846 |
| [23] | Horenkamp FA, Valverde DP, Nunnari J, Reinisch KM. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal, 2018, 37(6): e98002. |
| [24] | Prashek J, Bouyain S, Fu MG, Li Y, Berkes D, Yao XL. Interaction between the PH and START domains of ceramide transfer protein competes with phosphatidylinositol 4-phosphate binding by the PH domain. The Journal of Biological Chemistry, 2017, 292(34): 14217-14228. DOI:10.1074/jbc.M117.780007 |
| [25] | Yao YP, Wang WS, Yang KQ. Efficient production of polyketide products in Streptomyces hosts-a review. Acta Microbiologica Sinica, 2016, 56(3): 418-428. (in Chinese) 姚永鹏, 王为善, 杨克迁. 链霉菌中高效生产聚酮化合物的研究方法及进展. 微生物学报, 2016, 56(3): 418-428. |
| [26] | Champion PAD, Cox JS. Protein secretion systems in Mycobacteria. Cellular Microbiology, 2007, 9(6): 1376-1384. DOI:10.1111/j.1462-5822.2007.00943.x |
| [27] | Daleke MH, Ummels R, Bawono P, Heringa J, Vandenbroucke-Grauls CM, Luirink J, Bitter W. General secretion signal for the mycobacterial type Ⅶ secretion pathway. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(28): 11342-11347. DOI:10.1073/pnas.1119453109 |
