何新宇, 郑京, 刘琪, 冯永君


北京理工大学生命学院, 北京 100081
收稿日期:2019-04-01;修回日期:2019-07-12;网络出版日期:2019-09-04
基金项目:国家自然科学基金(31870090);国家重点研发计划(2018YFA0901800)
*通信作者:冯永君, E-mail:fengyj@bit.edu.cn.
摘要:菠萝泛菌(Pantoea ananatis)YJ76是从水稻“越富”品种中分离的优势内生菌,与宿主水稻互作时具有多种促生作用,其分泌的吲哚作为细菌种内及种间的信号分子参与调控多种生理生化行为。[目的] 筛选获得与吲哚调控相关的突变株,鉴定突变位点并研究突变基因对菌株的生存适应性以及对宿主水稻定殖和促生的影响,为研究吲哚调控通路奠定基础。[方法] 用双亲本接合法构建YJ76的mTn5转座子插入突变文库,以染色体步移TAIL-PCR技术鉴定突变基因,最后探究基因突变对菌体产生的影响。[结果] 筛选到1株吲哚产量大幅上升的YJ76突变株M04,鉴定突变位点为一个长度195bp未报道过的新基因,将其命名为ipc(indole production control),基因突变后增强了YJ76对重金属、四环素和酸的抗性,也增强了菌体对宿主水稻定殖和促生的能力。[结论] 吲哚产量上调的ipc突变株能够提高菌体生存适应性并增强其对宿主水稻定殖和促生的能力。
关键词:菠萝泛菌YJ76吲哚抗逆植物微生物相互作用
Identification and characterization of an indole production-upregulated mutant of rice endophytic Pantoea ananatis YJ76
Xinyu He, Jing Zheng, Qi Liu, Yongjun Feng


School of Life Sciences, Beijing Institute of Technology, Beijing 100081, China
Received: 1 April 2019; Revised: 12 July 2019; Published online: 4 September 2019
*Corresponding author: Feng Yongjun, E-mail:fengyj@bit.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31870090) and by the National Key Research and Development Program of China (2018YFA0901800)
Abstract: [Objective] To obtain and identify mutants related to indole regulation will provide functional genes responding for bacterial survival adaptability and in-planta colonization/association, which will be significant to the study of indole regulation pathway. [Methods] A mutant library of YJ76 was constructed by mTn5 transposon mutagenesis method, from which the mutant genes could be identified by using the high-efficiency TAIL PCR (hiTAIL-PCR) technique. [Results] A mutant strain named M04 with significantly increased indole production was selected from the mutant library of YJ76, and the mutation site was identified as a new gene (195 bp), which we named as ipc (indole production control) gene. This mutation enhanced the bacterial resistance to heavy metals, tetracycline and acids, and also enhanced the colonizing/plant growth-promoting ability of the endophyte on rice. [Conclusion] By carrying ipc mutation, the mutant strain increased indole production, resulting in the improved survival adaptability and higher association ability on the host rice.
Keywords: Pantoea ananatisindolesurvival adaptabilityplant endophyte association
植物内生菌(endophyte)是指能定殖在健康植物组织内,并与植物建立了和谐联合关系的一类微生物。内生菌在植物体内的定殖是一个主动过程,定殖后的内生菌不仅不会对植物造成实质性危害症状[1],还能够对宿主产生促生作用,其机理在于两个方面:一是分泌植物激素、固氮以促进植物生长[2];二是通过抑制其他病原菌的侵害来促进植物生长[3]。
菠萝泛菌(Pantoea ananatis)YJ76是从水稻“越富”品种中分离的优势内生菌,是吲哚分泌型菌株,可以表达色氨酸酶将L-色氨酸分解成吲哚。吲哚作为细菌种内及种间的信号分子参与调控多种生理生化行为,例如,可激活大肠杆菌(Escherichia coli)细胞的药物泵出和抗氧化保护等[4]。吲哚还可诱发细菌诸多生理生化性状的变化,包括生物被膜、趋化、抗逆、毒素、运动及细胞分裂等,其调控功能十分广泛[5-6],是细菌信号研究中最受关注的热点问题之一。但是,由于至今没有发现吲哚信号的受体,其调控机制尚不清楚[6]。
本实验室前期对YJ76的研究发现,该菌产生的吲哚可以促进生物被膜形成、提高对多种胁迫(重金属盐、酸、抗生素、干燥等)的抗性、提高定殖和促生能力[7]。本研究通过利用Tn5随机插入突变技术构建突变株文库,筛选吲哚产量发生变化的突变株,鉴定突变位点并研究突变基因对菌株的生存适应性以及对宿主水稻定殖和促生的影响,为进一步研究该突变基因的功能以及探索吲哚信号分子的调控通路奠定基础。
1 材料和方法 1.1 菌种和实验材料 菌种与培养方法:菠萝泛菌YJ76及其抗利福平突变株YJ76-Rpr由本实验室筛选并保存,培养条件为30 ℃、180 r/min;大肠杆菌SM10λpir,由比利时大学Jan Michiels教授提供,含有转座质粒pFAJ1819,卡那霉素抗性(Kmr),培养条件为37 ℃、220 r/min。本研究中对细菌菌株的培养,如无特殊说明均以1%的接种量于LB培养基(g/L):NaCl 10,胰蛋白胨10,酵母浸提物5,1×105 Pa灭菌30 min。配制固体培养基时,琼脂加入量为1.5%。
水稻与培养方法:水稻“越富”品种原种由北京市海淀区上庄镇东马坊农场惠赠。水稻种植培养基为EPA培养基(无氮培养基)[8]。
柯氏试剂(Kovac’s reagent):将10 g对二甲基氨基苯甲醛加入150 mL纯戊醇中;然后缓慢加入36%浓盐酸50 mL;X-Gluc(5-溴-4-氯-3-吲哚-β-D-半乳糖)母液(20 g/L):取X-Gluc (Sigma公司) 20 mg溶于二甲基甲酰胺(DMF) 1 mL中,–20 ℃避光保存,使用浓度为20 μg/mL;PBS缓冲液:NaCl 8.0 g,KCl 0.2 g,Na2HPO4 1.15 g,KH2PO4 0.2 g,以上试剂依次溶解在800 mL双蒸水中,用1 mol/L HCl调节pH值至7.4后,再加水定容至1000 mL;吲哚溶液母液(500 mmol/L):取吲哚(Sigma公司) 0.586 g,溶于10 mL的95%乙醇溶液中,避光保存;盐酸-戊醇溶液:30 mL的36%浓盐酸和90 mL的纯戊醇溶液。
1.2 构建YJ76的mTn5转座子突变文库并筛选阳性突变子 mTn5转座子突变文库的构建以Belas等的方法[9]为基础,将含供体菌SM10λpir与受体菌YJ76-Rpr混合液(1:1,V/V)的滤纸片放置于LB平板中,30 ℃培养24 h后,用含底物X-Gluc及利福平、卡那霉素的双抗LB平板进行筛选,在30 ℃避光培养72 h后,将变蓝的突变子接种于LB培养基,向培养好的菌液中按10:4滴加柯氏试剂,其中颜色变化与野生株呈明显差异的菌株接种于摇瓶中进行扩大培养,并进一步验证吲哚产量的变化。
1.3 TAIL-PCR法鉴定突变位点 采用染色体步移中的TAIL-PCR(热不对称交错PCR)法鉴定突变位点,以Liu等方法[10]为参照,根据插入突变株的转座子上的已知基因gusA设计3条具有高Tm值的嵌套特异性引物(gus1,gus2,gus3),以及根据细菌基因组中启动子的保守序列的特点设计低Tm值的简并引物(LAD),见表 1。
表 1. TAIL-PCR中所用引物 Table 1. Primers used in TAIL-PCR
| Primers | Sequences(5′→3′) |
| LAD1 | ACGATGGACTCCAGAGCGGCCGCVNVNNNGGAA |
| LAD2 LAD3 LAD4 | ACGATGGACTCCAGAGCGGCCGCBNBNNNGGTT ACGATGGACTCCAGAGCGGCCGCVVNVNNNCCAA ACGATGGACTCCAGAGCGGCCGCBBNBNNNCGGT |
| AC1 | ACGATGGACTCCAGAG |
| gus1 | CACTTCCTGATTGACCCACATTTGCCGT |
| gus2 | ACGATGGACTCCAGAGCGGCCGATACCAGACGTTGCCCGCATAATT |
| gus3 | GCTTTCCCACCAACGCTGATCAATTCC |
表选项
使用细菌基因组DNA提取试剂盒(天根生化科技有限公司)提取突变株的基因组,以该基因组为模板,利用3条特异性引物分别与简并引物(LAD)组合,根据引物的长短和特异性差异设计不对称的温度循环,通过三轮分级反应来扩增特异性产物[10],PCR体系与程序设置按照本课题组已发表方法[11]。将三轮获得的特异性条带回收,与pMD18 simple T载体(TaKaRa生物技术有限公司)进行连接,使用热激转化法将连接产物导入Trans-T1感受态细胞(全式金生物科技有限公司),提取质粒送至金唯智生物科技有限公司测序,使用NCBI网站Blast工具进行比对分析测序结果,确定序列所属的基因。
1.4 突变株吲哚产量以及生长曲线的测定 吲哚产量的测定以Kawamura-Sato等的方法[12]为基础,在不同生长点分别取1.5 mL菌液,12000×g离心2 min,取上清1.0 mL加入0.4 mL柯氏试剂,混合2 min,然后用盐酸-戊醇溶液10倍稀释,用分光光度计测定A540值。
生长曲线测定采用比浊法,从各组中分别取不同时间点的0.2 mL菌液至比色皿中,加入1.8 mL灭菌的PBS缓冲液并充分混匀,用分光光度计测定A600值。
1.5 基因突变对YJ76抗逆能力影响的检测 测得YJ76的重金属盐、强酸和四环素的最低灭生浓度MBC值分别为4 mmol/L、pH 3、60 μg/mL。基于以上的数据,在进行抗重金属、酸、四环素实验时,分别是终浓度为2.5 mmol/L的硫酸铜、pH设定为4、终浓度为30 μg/mL的四环素。
设置3组实验组,包括YJ76组、YJ76添加吲哚组、突变株M04组。由于相比于野生株,突变株的吲哚产量增加0.4–0.5 mmol/L,因此如未特殊说明,则本文中吲哚添加量均为0.5 mmol/L。将细菌培养至40 h后,调节各组A600值至一致,在培养基中加入四环素母液,使其终浓度为30 μg/mL,调节pH值至6.8,对照组不加四环素,取而代之的是同体积的PBS缓冲液。30 ℃、180 r/min培养4 h后,用平板计数法测得存活率。用相同的方法,在抗金属能力试验时在培养基中加入CuSO4母液,使其终浓度为2.5 mmol/L,在抗酸能力试验中在培养基中加入盐酸,使其pH值为4。
1.6 ipc基因突变菌株对水稻促生及其定殖能力的测定 水稻的种子消毒处理及接种培养参考Feng等的方法[8],将水稻种子脱壳并彻底消毒后,移入用PBS稀释至菌体浓度为1.5×107/mL菌液中侵染1 h,倾去菌液,用大量的无菌水洗涤7次(空白组在等量的PBS中浸泡1 h)。最后将种子转移到装有25 mL的EPA培养基的试管(3 cm×20 cm)中培养,每管1粒种子,试管的培养基界面以下用锡箔纸包裹,置于温度25 ℃、湿度50%的恒温光照培养箱中培养,每天光照与黑暗各12 h。水稻种子分成5组,即无菌空白组、吲哚添加组、YJ76侵染组、M04侵染组、YJ76侵染并添加吲哚组。
促生和定殖:培养至第12天,用无菌镊子拔出水稻苗,用无菌水冲洗,无菌吸水纸吸水,对水稻的整株、茎叶和根的鲜重分别进行测量和统计,以此来判断菌体侵染对水稻的促生作用。将水稻苗在无菌研钵内充分研磨,加入1 mL PBS悬浮,稀释104倍后涂布100 μL菌悬液于LB固体培养基,菌落长出后,计数统计以此来判断菌株的定殖能力。
2 结果 2.1 获得吲哚产量上调的YJ76阳性突变株 在构建YJ76的mTn5转座子突变文库中筛选到一株吲哚产量上调的YJ76阳性突变株,命名为突变株M04。使用柯氏反应分别检测YJ76和M04中的吲哚含量,吲哚含量越高,玫红色越深,如图 1所示(以黑白色显示),可以明显看出突变株的颜色更深,产量高于野生株。
 |
| 图 1 YJ76和M04吲哚产量的检测 Figure 1 Indole production ability of YJ76 and M04 are detected by Kovac's reagent. Bacterial cultures (1 mL) were incubated overnight in LB medium and then transferred to 1.5 mL tubes, and then Kovac's reagent (200 μL) was added, demonstrating that the YJ76 produces indole (rose-bengal color), and the M04 produces a larger amount of indole, which however is shown as a darker color in the black & white publication scale. |
| 图选项 |
2.2 染色体步移法成功鉴定突变位点ipc基因 用TAIL-PCR对转座片段的插入位点进行的鉴定(图 2)显示,以简并引物LAD3进行PCR扩增得到大小在700–1000 bp的单一特异性条带。将此片段回收后连接pMD18-T载体,转入Trans-T1感受态细胞中扩增,提取质粒并测序发现目标片段中1–326 bp为转座子上gusA中的一段序列。通过在NCBI上进行BLAST,327–782 bp部分序列与YJ76基因组中的一个未报道过的195 bp的开放阅读框序列同源,可以判断突变位点为YJ76中的此基因,并将此基因命名为ipc (indole production control),其基因的序列见图 5,序列中的箭头所示的位置是转座片段插入的位点。
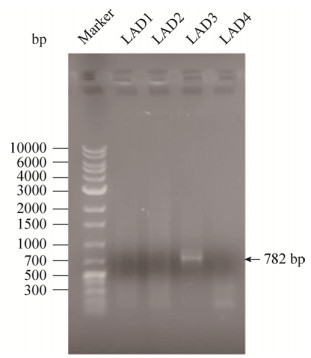 |
| 图 2 第三轮TAIL-PCR样品电泳图 Figure 2 Electropherogram of the third round TAIL-PCR samples. |
| 图选项 |
 |
| 图 5 M04中ipc基因与上、下游基因分析 Figure 5 ipc gene of mutant M04 and its upstream and downstream gene analysis. cydB: cytochrome D-panthenol oxidase II gene; yqjH: iron carrier interacting protein gene; yqjI: transcriptional regulatory gene of PadR family; the 867 bp part is AraC family transcription factor gene (unnamed gene); the middle 195 bp part is the mutant gene that we found ipc; the arrow in the sequence is where the transposon is inserted. |
| 图选项 |
根据以上获得的碱基序列设计引物,以YJ76的基因组为模板进行PCR扩增,产物进行琼脂糖凝胶电泳验证,成功得到ipc基因的电泳带,见图 3。
 |
| 图 3 鉴定得到的目标基因 Figure 3 Identification of the target gene. |
| 图选项 |
通过NCBI数据库比对,发现ipc基因在种内同源性高达98%,覆盖度高达100%;属内它种同源性最高75% (P. stewartii),属外无同源性。如图 4所示,ipc基因编码的假设蛋白在种内同源性高达98%,覆盖度高达100%,属内它种同源性最高83% (P. allii),最低43% (P. vagans),属外无匹配蛋白。
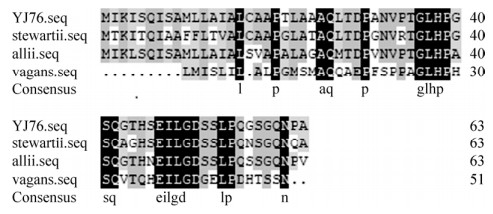 |
| 图 4 泛菌属内ipc基因所编码假设蛋白的氨基酸序列比对 Figure 4 Comparison of amino acid sequences of hypothetical proteins encoded by the ipc gene in Pantoea. |
| 图选项 |
通过分析M04突变基因上、下游序列,如图 5所示,发现该基因上、下游的4个基因中有2个基因是转录因子的基因,例如yqjI是PadR家族的转录调控基因和其上游的867 bp部分为AraC家族转录因子基因。上述生物信息学分析说明该基因在泛菌属内有独特性,表明其具有特殊的生物学功能。
2.3 突变株吲哚产量上调但没有明显影响菌体生长 如图 6-A所示,YJ76和M04的吲哚产生均在对数期开始,在稳定期保持相对稳定,并且M04的产量比YJ76高出0.4–0.5 mmol/L。
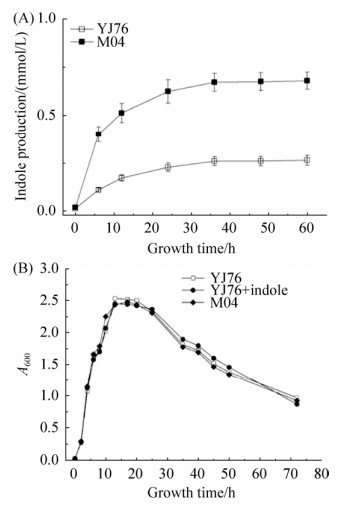 |
| 图 6 M04和YJ76的吲哚产量(A)和生长曲线(B) Figure 6 Indole production (A) and growth curve (B) of M04 and YJ76. P < 0.05. |
| 图选项 |
YJ76和M04的生长曲线如图 6-B所示,由图中可以得出基因的突变对YJ76的生长没有造成影响,并且适当浓度范围内的吲哚也不会对YJ76的生长有影响,表明吲哚产量增加并非是基因突变对菌生长造成影响而导致的。
2.4 ipc基因突变后YJ76的抗逆增强 如图 7所示,ipc基因突变后使YJ76的抗重金属、抗四环素和抗酸能力增强。在重金属盐2.5 mmol/L CuSO4的作用下,YJ76的存活率仅为9.15%,而M04的存活率为27.49%,较YJ76提高200.44%;在终浓度为30 μg/mL的四环素作用下,YJ76的存活率仅为19.30%,而M04的存活率为58.71%,较YJ76提高204.20%;在pH 4的酸性条件下,YJ76的存活率仅为24.44%,而M04的存活率为42.23%,较YJ76提高72.79%,另外,添加吲哚后YJ76的抗逆能力也显著提高,分别提高276.93%、261.20%和81.46%。
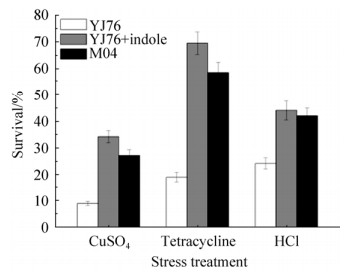 |
| 图 7 ipc基因突变后对YJ76抗逆境能力的影响 Figure 7 Effect of ipc gene mutation on YJ76 resistance to stress. P < 0.05. |
| 图选项 |
2.5 ipc基因突变后YJ76对水稻的促生和定殖能力增强 培养12 d后,各组水稻的生长情况如图 8所示,有内生菌接种的水稻比对照组长势明显要好,且M04接种的水稻比YJ76接种长势明显好,说明基因突变后能够增强YJ76对水稻促生作用。
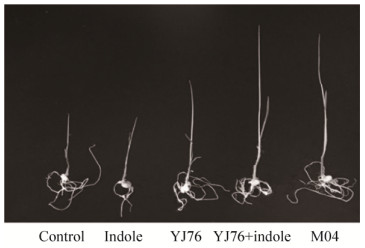 |
| 图 8 培养12 d后的水稻的长势图 Figure 8 Growth profile of rice after 12 days of cultivation. |
| 图选项 |
水稻鲜重的统计结果如图 9所示,培养12 d时水稻整株的重量为29.6 mg,接种YJ76和M04后,水稻鲜重分别为39.4 mg和48.7 mg,各增加33.11%和64.53%。无接种组的茎叶为19.5 mg,接种YJ76和M04后,茎叶分别为28.6 mg和35.3 mg,各增加46.67%和81.03%。无接种组的根为10.1 mg,接种YJ76和M04后,根分别为10.8 mg和13.4 mg,各增加6.93%和32.67%。显然,接种M04的水稻相较于接种YJ76的水稻的整株的鲜重以及茎叶、根的鲜重均增加,因此ipc基因突变后增强了细菌的促生能力。并且比较吲哚添加组和对照组可以看出吲哚本身对水稻的生长没有明显的影响,而对比YJ76吲哚双添加组和YJ76单接种组可以说明吲哚是通过YJ76发挥作用的。
 |
| 图 9 基因突变后对YJ76促生能力的影响 Figure 9 Effect of ipc gene mutation on the plant growth-promoting ability of YJ76. P < 0.05. |
| 图选项 |
培养12 d时,对水稻的茎叶、根分别进行研磨、稀释、涂板处理,待单菌落长出后,计算水稻单位质量上的细菌定殖数量。结果如表 2所示,YJ76在水稻整株中的定殖数量为1.2×104 CFU/mg,其中,根中为1.3×104 CFU/mg,茎叶中为1.1×104 CFU/mg。相比于YJ76,M04定殖能力明显上升,整株的细菌定殖数量为2.0×104 CFU/mg,比YJ76增加66.67%;茎叶为1.5×104 CFU/mg,比YJ76增加36.36%;根为2.5×104 CFU/mg,增加92.31%。YJ76添加吲哚后,定殖能力也显著增强,相比于YJ76,整株增加116.67%,茎叶增加100%,根增加130.77%。
表 2. ipc基因突变后对YJ76在水稻中定殖能力的影响 Table 2. Effect of ipc gene mutation on the colonization ability of YJ76 in rice.
| Seed treatment | Whole plant/(CFU/mg) | Roots/(CFU/mg) | Shoots/(CFU/mg) |
| YJ76 | 1.2×104a | 1.3×104a | 1.1×104a |
| YJ76+indole | 2.6×104c | 3.0×104b | 2.2×104c |
| M04 | 2.0×104b | 2.5×104b | 1.5×104b |
| 20 groups in each group, the seeds were cultured for 12 days and then counted, P<0.05, the different letters in each column indicate that there is a significant difference between them. | |||
表选项
3 讨论 本研究从菠萝泛菌YJ76出发,筛选出吲哚产量上调的M04突变株,研究发现吲哚产量的增加虽然没有影响菌体的生长,但是显著地提高了菌体的生存适应性,促进其对水稻的定殖和促生,M04突变株的获得不仅对于研究吲哚的代谢调控有所帮助,同时也为促生生物菌剂的开发显示出一定的应用价值。
我们通过TAIL-PCR法鉴定出M04的突变位点位于ipc基因,该基因还未有过相关报道,在NCBI数据库比对ipc基因序列和假设蛋白,发现在属外没有相匹配的基因和蛋白。泛菌属菌群的表型和基因型非常复杂,分类和鉴定经历了一个复杂的过程[13-15],直到1989年,Gavini根据DNA杂交结果提出一个新属泛菌属(Pantoea)[16],以收容这些DNA杂交度高但生理生化表型很不统一的菌群。该ipc基因在表型和基因型非常复杂的泛菌属内如此保守,并且属外无此基因,说明该基因在泛菌属内有独特性同时也预示着它在生存适应性等方面有着重要意义,为吲哚产量调控的机制研究提供了一条新的思路。
在自然界中,细菌常常会遭遇到各种逆境,使细菌面临严重的生存压力,如何能够提高自身生存适应性是至关重要的。本研究表明,相比于野生株,M04可以增强菌体的抗重金属、抗四环素和抗酸能力,在逆境中的存活率明显上升,并且可以推断出是因为YJ76内源性吲哚产量上升而使M04抗逆增强。另外,M04对宿主水稻的促生和定殖能力都有增加,因而同样可以证明内源性吲哚产量的上调是YJ76定殖和促生能力增强的原因。据上文的论述,吲哚信号的调控作用非常广泛,细菌细胞内吲哚的合成必须受到严格的调控,从而在各种因素引起的促进合成和抑制合成之间达到一种动态平衡,而ipc正是在吲哚合成代谢中起负调控作用的这样一个基因。我们观察到M04的定殖与促生能力要弱于添加吲哚的YJ76,这说明ipc基因除了能够控制吲哚产量外还可能有其他方面的潜在作用。ipc基因调控吲哚产量的机制以及吲哚如何调控YJ76的生理行为进而促进水稻的生长等机制,尚有待进一步研究。
References
| [1] | Kloepper JW, Beauchamp CJ. A review of issues related to measuring colonization of plant roots by bacteria. Canadian Journal of Microbiology, 1992, 38(12): 1219-1232. DOI:10.1139/m92-202 |
| [2] | Kim HJ, Lee JH, Kang BR, Rong XQ, McSpadden Gardener BB, Ji HJ, Park CS, Kim YC. Draft genome sequence of Pantoea ananatis B1-9, a nonpathogenic plant growth-promoting bacterium. Journal of Bacteriology, 2012, 194(3): 729. DOI:10.1128/JB.06484-11 |
| [3] | Torres R, Teixidó N, Usall J, Abadias M, Vi?As I. Post-harvest control of Penicillium expansum on pome fruits by the bacterium Pantoea ananatis CPA-3. The Journal of Horticultural Science and Biotechnology, 2005, 80(1): 75-81. DOI:10.1080/14620316.2005.11511895 |
| [4] | Lee HH, Molla MN, Cantor CR, Collins JJ. Bacterial charity work leads to population-wide resistance. Nature, 2010, 467(7311): 82-85. DOI:10.1038/nature09354 |
| [5] | Lee JH, Lee J. Indole as an intercellular signal in microbial communities. FEMS Microbiology Reviews, 2010, 34(4): 426-444. DOI:10.1111/j.1574-6976.2009.00204.x |
| [6] | Kim J, Park W. Indole:a signaling molecule or a mere metabolic byproduct that alters bacterial physiology at a high concentration?. Journal of Microbiology, 2015, 53(7): 421-428. DOI:10.1007/s12275-015-5273-3 |
| [7] | Zheng J, Yu JJ, Jia MQ, Zheng LP, Feng YJ. Indole enhances the survival of Pantoea ananatis YJ76 in face of starvation conditions. Journal of Basic Microbiology, 2017, 57(7): 633-639. DOI:10.1002/jobm.201700027 |
| [8] | Feng Y, Shen D, Song W. Rice endophyte Pantoea agglomerans YS19 promotes host plant growth and affects allocations of host photosynthates. Journal of Applied Microbiology, 2006, 100(5): 938-945. DOI:10.1111/j.1365-2672.2006.02843.x |
| [9] | Belas R, Erskine D, Flaherty D. Transposon mutagenesis in Proteus mirabilis. Journal of Bacteriology, 1991, 173(19): 6289-6293. DOI:10.1128/JB.173.19.6289-6293.1991 |
| [10] | Liu YG, Chen YL. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences. BioTechniques, 2007, 43(5): 649-650, 652, 654, 656. DOI:10.2144/000112601 |
| [11] | Jia MQ, Yu XM, Jiang J, Li ZH, Feng YJ. The cytidine repressor participates in the regulatory pathway of indole in Pantoea agglomerans. Research in Microbiology, 2017, 168(7): 636-643. DOI:10.1016/j.resmic.2017.04.006 |
| [12] | Kawamura-Sato K, Shibayama K, Horii T, Iimuma Y, Arakawa Y, Ohta M. Role of multiple efflux pumps in Escherichia coli in indole expulsion. FEMS Microbiology Letters, 1999, 179(2): 345-352. DOI:10.1111/j.1574-6968.1999.tb08748.x |
| [13] | Dye DW. A taxonomic study of the genus Erwinia. III. The "herbicola" group. New Zealand Journal of Agricultural Research, 1969, 12(2): 223-236. |
| [14] | Ewing WH, Fife MA. Enterobacter agglomerans (Beijerinck) comb. nov. (the Herbicola-Lathyri Bacteria). International Journal of Systematic Bacteriology, 1972, 22(1): 4-11. DOI:10.1099/00207713-22-1-4 |
| [15] | Brenner DJ, Fanning GR, Knutson JKL, Steigerwalt AG, Krichevsky MI. Attempts to classify herbicola group-Enterobacter agglomerans strains by deoxyribonucleic acid hybridization and phenotypic tests. International Journal of Systematic Bacteriology, 1984, 34(1): 45-55. DOI:10.1099/00207713-34-1-45 |
| [16] | Gavini F, Mergaert J, Beji A, Mielcarek C, Izard D, Kersters K, de Ley J. Transfer of Enterobacter agglomerans (Beijerinck 1888) Ewing and Fife 1972 to Pantoea gen. nov. as Pantoea agglomerans comb. nov. and description of Pantoea dispersa sp. nov. International Journal of Systematic Bacteriology, 1989, 39(3): 337-345. DOI:10.1099/00207713-39-3-337 |
