谢玲玲1, 宁婷婷1, 卞光凯1, 高丁2

 , 刘天罡1
, 刘天罡1

1.武汉大学药学院, 组合生物合成与新药发现教育部重点实验室, 湖北 武汉 430071;
2.中国科学院武汉病毒所, 湖北 武汉 430071
收稿日期:2017-01-09;修回日期:2017-02-21;网络出版日期:2017-03-01
基金项目:“****”青年拔尖人才项目
*通信作者:高丁, E-mail:gaoding@wh.iov.cn
刘天罡, Tel:+86-27-68755086, E-mail:liutg@whu.edu.cn
摘要: [目的]探索大肠杆菌生长分裂过程中,脂肪酸作为底物在细胞膜合成过程中的掺入模式。[方法]本研究解析了以乙酰CoA为底物,合成中间产物长链脂酰-ACP,随后合成磷脂酰乙醇胺(phosphatidylethanolamine,PE)的途径,并将合成途径中的10个关键酶与绿色荧光蛋白(enhanced green fluorescent protein,EGFP)或红色荧光蛋白(monmer Cherry,mCherry)进行融合,在大肠杆菌内表达这些融合蛋白,用激光共聚焦荧光显微镜成像的方式来获得这些融合蛋白的定位信息。[结果]宽场荧光显微镜成像结果显示,磷脂酰乙醇胺合成途径中的10个酶在不同表达水平下出现不同的定位模式。在大肠杆菌中高水平表达融合蛋白EGFP-FabA、EGFP-FabB、EGFP-FabI、EGFP-FabG、EGFP-PlsB和EGFP-PssA时,细胞两极和中部有大量蛋白聚集的现象。EGFP-FabD、EGFP-FabF、EGFP-CdsA、EGFP-PSD在不同表达水平下,均匀分散在细胞质或细胞膜上。缩时影像(Time-lapse)结果显示,合成途径中的一个关键蛋白EGFP-PlsB在细胞分裂前随着细胞膜的内陷聚集到细胞隔膜,随着细胞分裂,母细胞的隔膜成为新细胞的两极。[结论]本研究通过获取磷脂酰乙醇胺合成相关蛋白酶在大肠杆菌中的定位结果,推测脂肪酸分子是在细胞分裂隔膜和两极掺入,被催化合成PE后被运送到细胞膜其他位置。
关键词: 磷脂酰乙醇胺 (PE) 细胞膜合成 融合荧光蛋白 PlsB 脂肪酸 MreB
Fatty acid insertion mechanism during membrane synthesis in Escherichia coli
Lingling Xie1, Tingting Ning1, Guangkai Bian1, Ding Gao2

 , Tiangang Liu1
, Tiangang Liu1

1.Key Laboratory of Combinatorial Biosynthesis and Drug Discovery, Ministry of Education, School of Pharmaceutical Sciences, Wuhan University, Wuhan 430071, Hubei Province, China;
2.Wuhan Institute of Virology, Chinese Academy of Sciences, Wuhan 430071, Hubei Province, China
Received 09 January 2017; Revised 21 February 2017; Published online 01 March 2017
*Corresponding author: Ding Gao, E-mail: gaoding@wh.iov.cn
Tiangang Liu, Tel: +86-27-68755086, E-mail: liutg@whu.edu.cn
Supported by the Young Talents Program of National High level Personnel of Special Support Program (The "Ten Thousand Talent Program")
Abstract: [Objective]This study aimed to explore the mechanism of fatty acid insertion during membrane synthesis when cells grow and divide in Escherichia coli.[Methods]In the phosphatidylethanolamine (PE) synthesis pathway, acetyl-CoA was used as the substrate to synthesize long-chain acyl-ACP, followed by the synthesis of PE. The ten enzymes involved were each fused with a fluorescent protein such as enhanced green fluorescent protein (EGFP) or monmer Cherry (mCherry), and the fusion proteins were expressed in E. coli. The localizations of the fusion proteins were detected by laser scanning confocal fluorescence microscopy.[Results]Fluorescent microscope images showed that the proteins EGFP-FabA, EGFP-FabB, EGFP-FabI, EGFP-FabG, EGFP-PlsB, and EGFP-PssA accumulated in the polar and septal regions when expressed at high levels. Therefore, the ten enzymes displayed different localization patterns at different expression levels. Time-lapse imaging showed that EGFP-PlsB accumulated in the septum before cell division, then these regions of division became poles of the new cells.[Conclusion]This indicates that fatty acid inserted at the septum for PE synthesis and then PE was transported to all the other membrane regions.
Key words: phosphatidylethanolamine (PE) membrane synthesis fusing with fluorescent protein PlsB fatty acid MreB
细胞膜是细胞与外部环境分隔开的生理边界,它包裹着活细胞的细胞质,保证生命体必须的生化反应。细胞膜由内外两层膜组成,即内膜和外膜,内外膜之间由周质隔开,其中内膜是由脂质双分子层和镶嵌在其中的蛋白构成[1-2]。尽管不同类型的细胞中,细胞膜磷脂的组成有所差异,但大部分由磷脂酰乙醇胺 (phosphatidylethanolamine,PE)、磷脂酰甘油 (phosphatidylglyrecol,PG) 和卵磷脂 (cardiolipin,CL) 组成。大肠杆菌细胞膜磷脂是由80%的PE、5%-25%的PG和5%-10%的CL组成[1]。
大肠杆菌磷脂酰乙醇胺合成途径可以分为长链脂酰-ACP合成和磷脂合成2个部分。与长链脂酰-ACP合成相关的蛋白包括FabH、FabB、FabG、FabA、FabI、FabD、FabF和FabZ,乙酰CoA经过这些Fab酶的催化后得到特定长度的长链脂酰-ACP,随后进入磷脂合成阶段,由PlsB/PlsC系统催化,经过PssA、PSD和CdsA的后续催化合成磷脂酰乙醇胺[3](图 1)。
 |
| 图 1. 大肠杆菌磷脂合成途径 Figure 1. PE synthesis pathway in E. coli. Acetyl-CoA is catalyzed by three condensing enzymes, including FabH (β-ketoacyl-ACP synthase III), FabB (β-ketoacyl-ACP synthase I), and FabF (β-ketoacyl-ACP synthase II). FabG (β-ketoacyl-ACP reductase), FabA/Z (β-ketoacyl-ACP dehydratase) and FabI (trans-2-enoyl-ACP reductase) add two carbons with the completion of each turn of the cycle until a long chain acyl-ACP is generated. After the desired chain length of acyl-ACP has been synthesized, it is transferred to either sn-1 or sn-2 position of glycerol-3-phosphate (G3P) by the PlsB/PlsC system. These reactions produce phosphatidic acid (PA), which is the universal phospholipid precursor in bacteria. CdsA (phosphatidate cytidylyl-transferase) catalyzes the conversion of PA to the activated intermediate, CDP-diglyceride, which can then be utilized in the synthesis of either PE, PG or CL. PssA (CDP-diacylglycerol-L-serine phosphatidy-ltransferase) condenses serine to the phosphatidyl moiety of CDP-DAG coupled with the release of cytidine monophosphate (CMP). The final reaction in PE biosynthesis is performed by PSD (phosphatidyl-l-serine carboxylase), which decarboxylates the serine head group, resulting in the release of CO2. |
| 图选项 |
对于细胞膜合成机制的研究大都集中在阐明合成相关蛋白的生化性质[2],对于磷脂合成的位置,底物分子的掺入方式以及合成酶的亚细胞定位涉及很少。利用PE和CL的特异性荧光染料将枯草芽孢杆菌染色发现,枯草芽孢杆菌细胞膜中磷脂酰乙醇胺 (PE) 和卵磷脂 (CL) 聚集在细菌的两极和分裂隔膜位置。同时通过融合荧光蛋白表达的方式发现,合成这两种磷脂的关键蛋白也聚集在两极和中间,说明枯草芽孢杆菌中的PE和CL极有可能是在两极和细胞分裂位置合成的。而在大肠杆菌中,利用荧光染料染色的方式难以观察到磷脂清晰的定位模式[4]。蛋白的定位机制可以在一定程度上指示其在细胞中的特殊功能[5],追踪磷脂合成相关酶的亚细胞定位是探索底物和中间产物如何掺入细胞膜的一种方法。磷脂合成酶的磷脂分子合成发生在大肠杆菌什么位置,脂肪酸分子又是如何随着细胞生长掺入到大肠杆菌细胞膜中,这些关于细胞膜合成的动态分布问题,仍然不得而知。
蛋白以动态形式存在于细胞中[6],获得细胞生长过程中蛋白的动态变化非常重要。研究者们可利用超高分辨荧光显微镜成像,得到大肠杆菌内清晰的蛋白定位,利用缩时影像 (Time-lapse) 显微拍摄技术能观察到细胞生长分裂过程中蛋白的移动[7]。本研究从研究蛋白定位展开,对大肠杆菌细胞膜磷脂酰乙醇胺的生物合成机制进行探索,在大肠杆菌中表达从乙酰CoA合成长链脂酰-ACP,继而合成磷脂酰乙醇胺途径中的10个关键蛋白,获得不同表达水平下蛋白的定位模式。在大肠杆菌磷脂合成途径中,第一个阶段长链脂酰-ACP合成后,合成细胞膜磷脂的第一步反应是甘油-3-磷酸的乙酰化,PlsB是这一反应的关键蛋白。
研究发现,磷脂酰乙醇胺合成途径中关键酶与荧光蛋白的融合蛋白EGFP-FabA、EGFP-FabB、EGFP-FabI和EGFP-PlsB等在高水平表达下聚集在细胞两极,同时利用Time-lapse显微拍摄技术对EGFP-PlsB蛋白在细胞生长过程中的定位进行追踪,发现EGFP-PlsB在细胞膜上不是静止不动的,而是伴随着细菌的生长在细胞膜上以一定的规律动态移动。
1 材料和方法 1.1 质粒和菌株 1.1.1?菌株:大肠杆菌 (Escherichia coli) XL1-blue用来保存和扩增质粒,大肠杆菌 (Escherichia coli) MG1655(DE3) 用来表达蛋白。
1.1.2?质粒:本研究使用的原始载体pET28含有T7启动子和乳糖操纵子。所有表达载体均在酶切位点Nde I和Xho I之间插入融合蛋白基因,所用引物如表 1所示。绿色荧光蛋白基因gfp和樱桃红荧光蛋白基因mcherry根据已知序列合成。用overlap PCR的方法将fabB基因和gfp基因融合在一起[8],用1个24 bp的linker将目标蛋白基因和荧光蛋白基因连接在一起,荧光蛋白连在目标蛋白的N端 (图 2),linker的序列是CCTAGTTGTAGTAGTCCTAGTCCT。其他蛋白表达载体的构建均采用simple cloning的方法[9],将包含荧光蛋白基因的载体和目的基因连接起来。
 |
| 图 2. 表达融合蛋白所用重组质粒的构建示意图 (以EGFP-FabB为例) Figure 2. Construction schematic of recombinant plasmid for fusion protein expression (taking EGFP-FabB for example). |
| 图选项 |
表 1. 本研究中使用的引物 Table 1. Primers used in this study
| Plasmids | Primer names | Primer sequences (5′→3′) |
| pET28a-eGFP-FabA | pET28-GFP-FabA F FabA | GACGAGCTGTACAAGCCTAGTTGTAGTAGTCCTAGTCCTATGGTAGATAAACGCGAATC |
| pET28-GFP-FabA R FabA | CAGTGGTGGTGGTGGTGGTGCTCGAGTCAGAAGGCAGACGTATCCTGGAACAGACCGAC | |
| pET28-GFP-FabAF vec | CTGTTCCAGGATACGTCTGCCTTCTGACTCGAGCACCACCACCACCACCACTGAGATC | |
| pET28-GFP-FabA R vec | GGATTCGCGTTTATCTACCATAGGACTAGGACTACTACAACTAGGCTTGTACAGCTC | |
| pET28a-eGFP-FabI | pET28-GFP-FabI F FabI | CCTAGTTGTAGTAGTCCTAGTCCTATGGGTTTTCTTTCCGGTAAGCGCATTCTG |
| pET28-GFP-FabI R FabI | GTGGTGGTGGTGGTGGTGCTCGAGTTATTTCAGTTCGAGTTCGTTCATTGCAGC | |
| pET28-GFP-FabI R vec | GCGCTTACCGGAAAGAAAACCCATAGGACTAGGACTACTACAACTAGGCTTGTACAG | |
| pET28-GFP-FabI F vec | GAACGAACTCGAACTGAAATAACTCGAGCACCACCACCACCACCACTGAGATCCG | |
| pET28a eGFP-FabG | pET28-GFP-FabG F FabG | CCTAGTTGTAGTAGTCCTAGTCCTATGAATTTTGAAGGAAAAATCGCACTGGTAACC |
| pET28-GFP-FabG R FabG | GTGGTGGTGGTGGTGGTGCTCGAGTCAGACCATGTACATCCCGCCGTTCACATG | |
| pET28-GFP-FabG R vec | GATTTTTCCTTCAAAATTCATAGGACTAGGACTACTACAACTAGGCTTGTACAG | |
| pET28-GFP-FabG F vec | CGGCGGGATGTACATGGTCTGACTCGAGCACCACCACCACCACCACTGAGATCCG | |
| pET28a-eGFP-CdsA | GFP-CdsA R vec | CAGATATCAGGCGATACTTCAGCAAAGGACTAGGACTACTACAACTAGGCTTGTACAGC |
| GFP-CdsA R CdsA | CAGTGGTGGTGGTGGTGGTGCTCGAGTTAAAGCGTCCTGAATACCAGTAACAACAAGC | |
| GFP-CdsA F vec | GTTACTGGTATTCAGGACGCTTTAACTCGAGCACCACCACCACCACCACTGAGATCCGG | |
| GFP-CdsA F CdsA | GCCTAGTTGTAGTAGTCCTAGTCCTTTGCTGAAGTATCGCCTGATATCTGCTTTTGTG | |
| pET28a-eGFP-PSD | GFP-PsD R vec | GCGAAAGTTTAAATGAATTTAACAAAGGACTAGGACTACTACAACTAGGCTTGTACAGC |
| GFP-PSD F vec | GTTGACGACAAAAAAGACCAGGTCTAACTCGAGCACCACCACCACCACCACTGAGATCC | |
| GFP-PSD R PSD | GTGGTGGTGGTGGTGGTGCTCGAGTTAGACCTGGTCTTTTTTGTCGTCAACCAATGGGC | |
| GFP-PSD F PSD | GCCTAGTTGTAGTAGTCCTAGTCCTTTGTTAAATTCATTTAAACTTTCGCTACAGTAC | |
| pET28a-eGFP-PssA | GFP-PssA R Vec | GTTTATTACGCTTAAATTTTGACAACATAGGACTAGGACTACTACAACTAGGCTTGTAC |
| GFP-PssA F vec | CGACCGATTAATTAGCCGCATCCTGTAACTCGAGCACCACCACCACCACCACTGAGATC | |
| GFP-PssA R PssA | GTGGTGGTGGTGGTGGTGCTCGAGTTACAGGATGCGGCTAATTAATCGGTCGATGCGG | |
| GFP-PssA F PssA | GCCTAGTTGTAGTAGTCCTAGTCCTATGTTGTCAAAATTTAAGCGTAATAAACATCAAC | |
| pET28a-eGFP-PlsC | GFP-PlsC F vec | GAACGCGAAGCCGCCGGAAAAGTTTAACTCGAGCACCACCACCACCACCACTGAGATCC |
| GFP-PlsC R vec | GTAATAATAAGACGAAAGATATATAGCATAGGACTAGGACTACTACAACTAGGCTTGTA | |
| GFP-PlsC R PlsC | CAGTGGTGGTGGTGGTGGTGCTCGAGTTAAACTTTTCCGGCGGCTTCGCGTTCTGCGAC | |
| GFP-PlsC F PlsC | CCTAGTTGTAGTAGTCCTAGTCCTATGCTATATATCTTTCGTCTTATTATTACCGTG | |
| pET28a-eGFP-PlsB | GFP-PlsB R vec | GTAAATTCGTGGCCAGCCGGACATAGGACTAGGACTACTACAACTAGGCTTGTACAGC |
| GFP-PlsB F vec | GAGAGTGCGACGCAGGGCGAAGGGTAACTCGAGCACCACCACCACCACCACTGAGATCC | |
| GFP-PlsB R PlsB | GTGGTGGTGGTGGTGGTGCTCGAGTTACCCTTCGCCCTGCGTCGCACTCTCAATCGTC | |
| GFP-PlsB F PlsB | CTAGTTGTAGTAGTCCTAGTCCTATGTCCGGCTGGCCACGAATTTACTACAAATTACTG | |
| pET28a-mCherry-MreB | pET28-GFP-MreB F MreB | GCCTAGTTGTAGTAGTCCTAGTCCTATGTTGAAAAAATTTCGTGGCATGTTTTCCAATG |
| pET28-GFP-MreB F Vec | CGGCGACCTGTTCAGCGAAGAGTAACTCGAGCACCACCACCACCACCACTGAGATCCGGC | |
| pET28-GFP-MreB R MreB | CAGTGGTGGTGGTGGTGGTGCTCGAGTTACTCTTCGCTGAACAGGTCGCCGCCGTGC | |
| pET28-GFP-MreB R Vec | GAAAACATGCCACGAAATTTTTTCAACATAGGACTAGGACTACTACAACTAGGCTTGTAC | |
| pXL03 | pLQ22-F-pET28 | GAAGGCTCTCAAGGGCATCGGTCGAGATCCCGGTGCCTAATGAGTGAGCTAAC |
| pLQ22-R-gfp plsB | GAACAGCTCCTCGCCCTTGCTCACCATATGGCTGCCGCGCGGCACCAG | |
| GFP-plsB-F-pLQ22 | GCCTGGTGCCGCGCGGCAGCCATATGGTGAGCAAGGGCGAGGAGCTGTTC | |
| T7 term-R-T7 promoter | GTATTAATTTCGCGGGATCGAGATCTCAAAAAACCCCTCAAGACCCGTTTAGAGG | |
| T7 promoter-F-pET28 | CTAAACGGGTCTTGAGGGGTTTTTTGAGATCTCGATCCCGCGAAATTAATACG | |
| mcherry mreB-R-pET28 | CCTCCTCGCCCTTGCTCACCATATGGCTGCCGCGCGGCACCAG | |
| pET28-F-mreB | GGCGACCTGTTCAGCGAAGAGTAACTCGAGCACCACCACCACCACCACTGAG | |
| pET28-R-pLQ22 | CTCACTCATTAGGCACCGGGATCTCGACCGATGCCCTTGAGAGCCTTCAACC |
表选项
1.2 培养条件 本研究所用培养基为LB培养基 (tryptone 10 g /L,NaCl 10 g /L,yeast extract 5 g/L)。质粒和菌株通过在培养基中加入卡那霉素 (Kanamycin 50 mg/mL) 以实现筛选。表达质粒转化至大肠杆菌MG1655(DE3) 中,不加入抗生素,在37 ℃培养至OD600为0.6-0.8,通过加入不同浓度 (10、1、0.1、0.001 mol/L) 的IPTG,30 ℃下诱导30 min,以观察不同表达水平下蛋白的定位模式。观察大肠杆菌PlsB定位的高分辨图像时,加入0.16 μmol/L的IPTG诱导。观察PlsB动态成像时,所用的大肠杆菌用0.01 mol/L的IPTG诱导。所有蛋白表达均在30 ℃下诱导表达30 min,菌液经过离心浓缩并保存在4 ℃待用。
1.3 脂肪酸合成的体外重建 体外重建脂肪酸合成的实验方法参考文献[10],将参与脂肪酸合成的所有蛋白纯化出来,按照文献中的优化比例加入蛋白,用放射性薄层色谱法 (thin layer chromatography,TLC) 进行脂肪酸的检测。在这个反应中加入每个参与反应的Fab蛋白各1 μmol/L,加入10 μmol/L holo-ACP和TesA,1 mmol/L的NADPH、NADH和TECP,0.2 mmol/L的acetyl-CoA,0.5 mol/L的5% C14标记的malonyl-CoA (American Radiolabeled Chemicals)。为了验证融合的EGFP-FabB蛋白与FabB功能上的差异,将体外反应中的FabB蛋白用EGFP-FabB替换。
1.4 宽场荧光成像和3D-SIM成像 宽场荧光成像和3D-SIM成像都在DeltaVisionOMX V4成像系统 (GE Healthcare Company,Issaquah,USA) 进行,物镜为60X/1.42 N.A. (Olympus)。用FITC,A568过滤器设置通过单色激光来激发。用softWoRx version 6.5.2 (GE Healthcare Company) 软件进行图像的去卷积和重构。将2 μL浓缩后的样品加到载玻片上,盖上盖玻片。激发波长为488 nm或者561 nm,图像的对比度和亮度调节使用软件Adobe Photoshop CS5处理。
1.5 Time-lapse拍摄EGFP-PlsB动态成像 EGFP-PlsB的动态成像在PerkinElmer VOX转盘共聚焦显微镜进行,采用物镜100×/1.49 N.A. (Olympus),ImagEM-1KEMCCD相机 (HAMAMAT SU),以及Volocity 6.3软件 (PerkinElmer)。琼脂胶块含有1.8%的琼脂糖,用稀释了5倍的LB溶解。琼脂胶块的制备参考文献[11],制备过程如图 3。将融化的琼脂溶液滴加到载玻片上,待冷却2 min左右,用盖玻片盖住使胶块表面平整,待胶块凝固完全,移去盖玻片,将胶块切成1 cm2的正方形小块。质粒pXL03转入大肠杆菌,加入0.01 mmol/L的IPTG诱导,30 ℃诱导30 min,菌液滴加到琼脂块成像。恒温控制室提前预热到37 ℃至少30 min,菌液加到琼脂块上之后,置入恒温空间用于观察。通过减少曝光时间来减小光毒性,动态拍摄的时间间隔为2 min,拍摄总时长120 min。
 |
| 图 3. 琼脂胶块的制作 Figure 3. The process of making agarose pad. Molten agar solution (1.8%; LB diluted 1:5 in water) was added to the well of a culture slide and covered with a glass slide. After cooling, the slide was removed and two air pockets were cut out of the agar, leaving a 3-5 mm agarose bridge in the center of the well. |
| 图选项 |
2 结果和分析 2.1 体外重建脂肪酸合成的实验证明融合蛋白EGFP-FabB的活性 通过将体外脂肪酸合成反应体系中的FabB蛋白换成融合了绿色荧光蛋白的EGFP-FabB,放射性TLC检测脂肪酸含量的结果显示,EGFP-FabB的活性与FabB活性相当,如图 4所示。
 |
| 图 4. 体外重建脂肪酸合成体系验证融合蛋白EGFP-FabB的活性 Figure 4. In vitro reconstitution of fatty acid biosynthesis to verify the activity of the fused protein EGFP-FabB. Whether EGFP-FabB has a function analogous to that of FabB was tested in the in vitro reconstitution fatty acid biosynthesis system. FabB was replaced by EGFP-FabB in the reconstitution system, and the production of fatty acid was recorded at different time points. The y-axis represents the relative intensity of fatty acid on the TLC plate. And the x-axis represents the reaction time of the in vitro reconstitution of fatty acid biosynthesis. |
| 图选项 |
2.2 长链脂酰-ACP合成的相关酶在不同表达水平下的定位模式 长链脂酰-ACP合成的相关酶FabA、FabB、FabG和FabI分别融合了绿色荧光蛋白EGFP,T7启动子启动表达,浓度梯度的IPTG诱导表达这些融合蛋白。对于每一个诱导条件下的样品,我们随机选取了5个以上的成像视野进行拍摄,总共获得了超过50个细胞的定位成像图。重复试验的结果一致显示,在IPTG浓度高达10 mmol/L和0.1 mmol/L时,这些蛋白呈现明显的两极和中间位置聚集的现象,而当IPTG浓度降低到一定程度时,它们在整个细胞均匀分布。蛋白mCherry-FabH在高浓度IPTG诱导下,呈现一种中空分布模式,表达水平降低时在整个细胞均匀分布 (图 5)。
 |
| 图 5. 脂肪酸合成相关蛋白的定位 Figure 5. Localization of enzymes involved in long chain acetyl-ACP synthesis. More than five fields were captured for microscope imaging, and there were in total more than 50 cells' images were collected of every microscopy sample. After repeated trials, we concluded that the long chain acetyl-ACP synthesis enzymes, including EGFP-FabA, EGFP-FabB, EGFP-FabG, and EGFP-FabI were localized at the poles in E. coli cells when induced with 10 mmol/L or 0.1 mmol/L IPTG. When induced with 1 μmol/L IPTG, these enzymes were distributed throughout the cytoplasm. The fused protein mCherry-FabH distributed throughout the cell except for a circular region in the middle of the cell when induced with 10 mmol/L IPTG, whereas it was dispersed throughout all the cell when induced with 0.1 mmol/L IPTG or less. |
| 图选项 |
2.3 与PE合成相关的膜蛋白在不同表达水平下的定位模式 长链脂酰-ACP合成之后,进一步合成磷脂酰乙醇胺的几个膜蛋白包括PlsC、PlsB、PssA、PSD和CdsA。在不同诱导条件下表达这些膜蛋白得到的每个样品,也有超过50个单细胞被测定。重复试验一致显示,EGFP-PlsB和PssA在10 mmol/L和1 mmol/L的IPTG诱导下呈现两极和隔膜定位,然而随着IPTG加入量的减小,这种两极定位逐渐消失,EGFP-PssA均匀分布在整个细胞质,EGFP-PlsB则成簇状分散在细胞膜。EGFP-PSD、EGFP-PlsC和EGFP-CdsA在10 mmol/L到0.001 mmol/L的IPTG诱导下均在细胞膜上呈现离散分布 (图 6)。
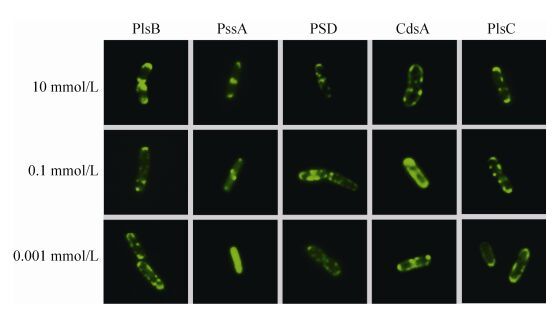 |
| 图 6. 脂酰-ACP到磷脂酰乙醇胺合成酶的定位 Figure 6. Localization patterns of enzymes involved in PE synthesis. More than 50 cells' images were obtained of each sample. It can be concluded that EGFP-PssA accumulated in the polar and septal regions when induced with 10 mmol/L or 0.1 mmol/L IPTG, but it was distributed in the cytoplasm when induced with 1 μmol/L IPTG. EGFP-PlsB accumulated at the poles in the presence of 10 mmol/L IPTG, and when the IPTG amount was decreased, EGFP-PlsB gradually scattered on the entire membrane. All the other membrane enzymes were scattered on the membrane at different expression levels. |
| 图选项 |
2.4 超高分辨荧光显微镜成像下EGFP-PlsB的定位模式以及EGFP-PlsB在细胞内移动的动态成像 荧光显微镜的应用使观测大肠杆菌内复杂的结构成为可能,蛋白在细胞内处于不断运动的状态,并会根据细胞状态定位在特定位置[12]。PlsB是利用细胞质中的底物脂酰-ACP合成磷脂酰乙醇胺反应的第一个酶,PlsB的空间定位对于解释脂肪酸掺入细胞膜模式以及PE合成发生的位置非常重要。通过超高分辨荧光显微镜观察,发现EGFP-PlsB在0.16 μmol/L的IPTG诱导下,成簇分散在整个细胞膜上 (图 7-A)。
 |
| 图 7. 超高分辨荧光显微镜下EGFP-PlsB定位成像图以及Time-lapse模式下EGFP-PlsB在不同时间点的定位成像图 Figure 7. Super-resolution microscopic images of EGFP-PlsB localization and Time-lapse images of EGFP-PlsB localization at different time points. A: Super-resolution image showed that EGFP-PlsB clustered on the entire membrane. B: Collection of six images at different time points from time-lapse images showed that the fluorescence signal spread around the cell at first, and then it became comparatively stronger at the septum of the cell when it divided. And at last the septum became the poles of the new cells along with the completion of division. |
| 图选项 |
通过优化动态拍摄的条件,经过2 h的观察,结果表明,随着细菌的生长和分裂,EGFP-PlsB沿着细胞膜移动,在分裂前集中到细胞隔膜,分裂结束后定位在新细胞的两极 (图 7-B)。这一结果说明,脂肪酸很有可能是在细胞分裂隔膜位置掺入细胞膜中的,细胞膜磷脂合成大部分发生在细胞隔膜。作为母细胞隔膜的产物,子细胞的两极也可能会掺入脂肪酸,合成新的磷脂来提供细胞膜的合成。
3 讨论 大肠杆菌细胞膜磷脂中80%的成分是磷脂酰乙醇胺,脂质双分子层构成了细胞膜的基本骨架,脂质的流动性保证细胞膜的生理功能,因而磷脂酰乙醇胺的合成是细胞膜合成的基础。本研究通过观察大肠杆菌中参与细胞膜磷脂酰乙醇胺合成相关蛋白在胞内的定位信息,揭示出脂肪酸在磷脂合成过程中掺入细胞膜的时空模式,回答细胞膜扩增发生的地点这一基本问题。
本研究提出脂肪酸作为底物参与磷脂合成过程中掺入细胞膜的2种模型 (图 8),一种模型假设脂肪酸是在细胞两极和细胞隔膜掺入,继而合成磷脂,运送到细胞膜上补充细胞膜的合成;另一种模型则假设脂肪酸随机掺入,磷脂在整个细胞膜上随机合成。
 |
| 图 8. 脂肪酸在磷脂合成过程中掺入细胞膜的2种模式 Figure 8. Proposed modes of fatty acid insertion mechanism during membrane synthesis. A: Insertion of fatty acid occurs at the septum and new synthesized molecules are transferred along the membrane to the other region of the cell. B: Fatty acid insertion occurs randomly around the entire membrane during cell growth and division. |
| 图选项 |
由于分子染料成像的有限性,本研究从磷脂合成相关的蛋白酶定位着手探索磷脂合成的位置以及脂肪酸掺入细胞膜的方式。在大肠杆菌中表达磷脂酰乙醇胺合成相关的10个关键蛋白发现,同一个蛋白在不同的表达水平下,呈现不同的定位模式,高水平表达下,蛋白出现两极和中部聚集的现象,包括EGFP-FabA、EGFP-FabB、EGFP-FabG、EGFP-FabI、EGFP-PlsB和EGFP-PssA,而EGFP-FabD、EGFP-FabF分布在整个细胞质,EGFP-PSD、EGFP-PlsC和EGFP-CdsA则成簇分布在细胞膜上,mCherry-FabH则呈现一种中空的定位模式。蛋白定位模式总结见表 2。所有磷脂酰乙醇胺合成相关蛋白,在较低水平下都呈现出均匀分散的定位模式,这种均匀分布可能是蛋白处于不断运动状态下的平衡。而过表达下的蛋白聚集可能是调节酶活性的一种方式,因为高水平表达下细胞产生了多余的酶[13]。
表 2. 大肠杆菌内磷脂酰乙醇胺相关酶高水平表达下在大肠杆菌中的定位 Table 2. Sub-cellular localization of the enzymes involved in lipid synthesis in E. coli at high expression level
| Protein | Function | Localization |
| FabA | Beta-hydroxydecanoyl-ACP-dehydratase | Polar |
| FabB | Beta-ketoacyl-ACP-synthase | Polar |
| FabG | Beta-Keto-ACP-reductase | Polar |
| FabH | Ketoacyl-ACP-synthase III | Hollow |
| FabI | Enoyl-ACP-reductase | Polar |
| PssA | Phosphatidylserine synthase | Polar and septal |
| PSD | Phosphatidylserine decarboxylase | Clustered |
| CdsA | CDP-diacylglycerol synthetase | Clustered |
| PlsB | sn-Glycerol 3-phosphate acyltransferase | Polar and septal |
| PlsC | 1-Acylglycerol 3-phosphate acyltransferase | Clustered |
| ACC | Acetyl-CoA carboxylase | Cytoplasm |
| FabD | Malonyl-CoA-[acyl-carrier-protein] transacylase | Cytoplasm |
| FabF | Beta-ketoacyl-acyl carrier protein (ACP) synthase | Cytoplasm |
表选项
在本研究中,不同的表达水平会导致大肠杆菌蛋白亚细胞定位的不同,为了探索大肠杆菌真实的蛋白亚细胞定位模式,本研究另外构建了2株突变株,将基因组上的fabB和fabH基因分别被融合基因egfp-fabB和egfp-fabH替换,利用基因组上fabB和fabH的原始启动子分别表达EGFP-FabB和mCherry-FabH。荧光显微镜成像结果显示,EGFP-FabB和mCherry-FabH都均匀分散在整个细胞内 (图 9)。由此推测,低水平表达下EGFP-FabB和mCherry-FabH更接近于细胞内真实的FabB和FabH的定位情况。本研究所用的T7启动子是较强的启动子,使用较低浓度IPTG诱导时的表达水平可能更接近于细胞内真实的基因表达水平。
 |
| 图 9. 基因组原始启动子启动表达EGFP-FabB (A) 和mCherry-FabH (B) 时蛋白在大肠杆菌内的定位 Figure 9. Localizations of EGFP-FabB (A) and mCherry-FabH (B) when they were promoted by local promoters in E. coli. Both EGFP-FabB and mCherry-FabH distributed around the whole cell when their expressions were promoted by the local promoters in the genome of E. coli. |
| 图选项 |
在研究膜蛋白的定位时,荧光蛋白融合在目标蛋白的N端还是C端是个问题,因为EGFP只有在大肠杆菌细胞质中才会发光[12]。我们将这几个膜蛋白C端和N端都融合上荧光蛋白,当N端融合EGFP时,这5个蛋白都发光,而CdsA和PssA的C端融合荧光蛋白时没有荧光,为了保持一致性,都采用N端融合荧光蛋白。
相对于静态成像,动态成像能更好地反映蛋白在细胞体内的真实变化。PlsB是以细胞质中的脂酰-ACP为底物,合成磷脂的第一个膜蛋白,是本研究中的一个关键蛋白。EGFP-PlsB在分裂前聚集到分裂位置的现象表明,脂酰-ACP极有可能是在细胞分裂隔膜掺入到细胞膜中,继而合成磷脂,细胞分裂形成的隔膜成为新细胞的两极,脂肪酸可能在新细胞两极掺入脂肪酸,继续合成磷脂,保证细胞的生长。图 8中的磷脂合成模型B更符合细胞内的真实状况。大肠杆菌和枯草芽孢杆菌细胞两极的物质合成要远远少于侧壁和分裂位置的合成,在球形金黄色葡萄球菌和链球菌物种,新的合成也主要发生在分裂隔膜[14]。
蛋白的极性定位是保证细胞正常功能的基础,通过分子标记等方法,在很多细菌中已经可以观察到蛋白在细胞中的极性定位,然而,引导和调节这种极性的一个通用的调节因子还没有找到[15]。MreB是细菌中肌动蛋白的一个同源蛋白,与Mre系列蛋白共同维持细胞的棒状形态[14]。在新月柄杆菌 (Caulobactercrescentus) 中分别敲除和高水平表达MreB,发现MreB对于染色体某些序列的极性定位,以及调控蛋白的动态定位,从而引导蛋白定位到特定细胞极至关重要[15-16]。
在枯草芽孢杆菌内,MreB细胞骨架与细胞流动性的脂质域有关,可通过干扰脂质稳态来影响膜蛋白定位[17]。在海栖热袍菌 (Thermotoga maritime) 中,MreB呈细丝状与细胞膜相连,协调细菌细胞壁的合成,RodZ直接或间接地将MreB与细胞壁合成相关的酶耦合[18]。
EGFP-PlsB在0.16 μmol/L的IPTG诱导下成簇分布在细胞膜上,与已经报道的大肠杆菌骨架蛋白MreB的螺旋分布模式相似。我们将PlsB和MreB融合不同颜色荧光构建在同一个pET28载体上表达,用T7启动表达mCherry-MreB,启动子plq22启动EGFP-PlsB表达,启动子plq22是本实验室已验证的较弱启动子,用一个较弱的启动子启动表达PlsB是为了避免强启动子启动时高水平表达下PlsB的两极聚集。在一些细胞中,MreB的聚集伴随着PlsB的聚集,但MreB和PlsB的共定位程度很低 (图 10)。
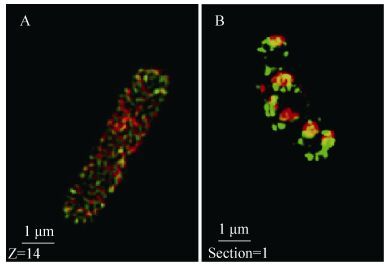 |
| 图 10. EGFP-PlsB (A) 和mCherry-MreB (B) 在大肠杆菌共表达高分辨成像图 Figure 10. Super-resolution images of E. coli in which EGFP-PlsB (A) and mCherry-MreB (B) were co-expressed. Two fluorescent colors indicated that EGFP-PlsB and mCherry-MreB were distributed around the membrane without obvious co-localization. In some cells, PlsB patches followed the orbit of the MreB focus. |
| 图选项 |
MreB作为细胞骨架处在细胞膜内膜之下,PlsB在细胞膜上,两者在空间上不在一个层面,因此共定位程度低也是正常的。而在大肠杆菌中,PlsB和MreB之间有可能存在一个因子,通过蛋白耦合或者其他未知的方式将两者间接联系起来。MreB可能通过一种间接的方式引导PlsB的移动,甚至调节整个磷脂合成相关酶的定位,从而调节大肠杆菌内磷脂的合成。
参考文献
| [1] | Zhang YM, Rock CO. Membrane lipid homeostasis in bacteria.Nature Reviews Microbiology, 2008, 6(3): 222–233DOI:10.1038/nrmicro1839. |
| [2] | Cronan JE. Bacterial membrane lipids:where do we stand.Annual Review of Microbiology, 2003, 57: 203–224DOI:10.1146/annurev.micro.57.030502.090851. |
| [3] | Parsons JB, Rock CO. Bacterial lipids:metabolism and membrane homeostasis.Progress in Lipid Research, 2013, 52(3): 249–276DOI:10.1016/j.plipres.2013.02.002. |
| [4] | Nishibori A, Kusaka J, Hara H, Umeda M, Matsumoto K. Phosphatidylethanolamine domains and localization of phospholipid synthases in Bacillus subtilis membranes.Journal of Bacteriology, 2005, 187(6): 2163–2174DOI:10.1128/JB.187.6.2163-2174.2005. |
| [5] | Hughes HV, Huitema E, Pritchard S, Keiler KC, Brun YV, Viollier PH. Protein localization and dynamics within a bacterial organelle.Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(12): 5599–5604DOI:10.1073/pnas.0909119107. |
| [6] | Johnson AS, van Horck S, Lewis PJ. Dynamic localization of membrane proteins in Bacillus subtilis.Microbiology, 2004, 150(9): 2815–2824DOI:10.1099/mic.0.27223-0. |
| [7] | Huang B, Bates M, Zhuang XW. Super-resolution fluorescence microscopy.Annual Review of Biochemistry, 2009, 78: 993–1016DOI:10.1146/annurev.biochem.77.061906.092014. |
| [8] | Bryksin AV, Matsumura I. Overlap extension PCR cloning:a simple and reliable way to create recombinant plasmids.Biotechniques, 2010, 48(6): 463–465DOI:10.2144/000113418. |
| [9] | You C, Zhang XZ, Zhang YHP. Simple cloning via direct transformation of PCR product (DNA Multimer) to Escherichia coli and Bacillus subtilis.Applied and Environmental Microbiology, 2012, 78(5): 1593–1595DOI:10.1128/AEM.07105-11. |
| [10] | Yu XY, Liu TG, Zhu FY, Khosla C. In vitro reconstitution and steady-state analysis of the fatty acid synthase from Escherichia coli.Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(46): 18643–18648DOI:10.1073/pnas.1110852108. |
| [11] | Gregory JA, Becker EC, Pogliano K. Bacillus subtilis MinC destabilizes FtsZ-rings at new cell poles and contributes to the timing of cell division.Genes & Development, 2008, 22(24): 3475–3488. |
| [12] | Rapp M, Drew D, Daley DO, Nilsson J, Carvalho T, Melén K, De Gier JW, Von Heijne G. Experimentally based topology models for E. coli inner membrane proteins.Protein Science, 2004, 13(4): 937–945DOI:10.1110/ps.03553804. |
| [13] | Beaty NB, Lane MD. The polymerization of acetyl-CoA carboxylase.The Journal of Biological Chemistry, 1983, 258(21): 13051–13055. |
| [14] | Cabeen MT, Jacobs-Wagner C. Bacterial cell shape.Nature Reviews Microbiology, 2005, 3(8): 601–610DOI:10.1038/nrmicro1205. |
| [15] | Gitai Z, Dye N, Shapiro L. An actin-like gene can determine cell polarity in bacteria.Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(23): 8643–8648DOI:10.1073/pnas.0402638101. |
| [16] | Bean GJ, Amann KJ. Polymerization properties of the Thermotoga maritima actin MreB:roles of temperature, nucleotides, and ions.Biochemistry, 2008, 47(2): 826–835DOI:10.1021/bi701538e. |
| [17] | Gowrishankar K, Ghosh S, Saha S, Rumamol C, Mayor S, Rao M. Active remodeling of cortical actin regulates spatiotemporal organization of cell surface molecules.Cell, 2012, 149(6): 1353–1367DOI:10.1016/j.cell.2012.05.008. |
| [18] | Morgenstein RM, Bratton BP, Nguyen JP, Ouzounov N, Shaevitz JW, Gitai Z. RodZ links MreB to cell wall synthesis to mediate MreB rotation and robust morphogenesis.Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(40): 12510–12515DOI:10.1073/pnas.1509610112. |
