张祥1, 方卉1, 谢东芳1, 林越呈1, 陶颖妍1, 王宏鹏1, 龚金炎1, 葛青1, 泮国荣1, 黄俊1,3

 , 尤玉如1,2
, 尤玉如1,2

1.浙江科技学院省农产品化学与生物加工技术重点实验室, 浙江 杭州 310023;
2.贝因美婴童食品股份有限公司, 浙江 杭州 310007;
3.杭州奥盛仪器有限公司, 浙江 杭州 310030
收稿日期:2016-07-29;修回日期:2016-08-30;网络出版日期:2016-09-18
基金项目:国家自然科学基金(31470793,31240054);杭州市科技计划(20140432B106);浙江科技学院研究生创新基金(2015ZUSTJSKC010)
*通信作者:黄俊,Tel/Fax:+86-571-85070370;E-mail:hjunlzr@163.com
尤玉如,Tel/Fax:+86-571-85070370;E-mail:youyuru0130@163.com
摘要: [目的]筛选H+-ATPase活性降低的植物乳杆菌突变菌,比较其与亲本菌基因表达水平的差异,进一步探索H+-ATPase的调控机制。[方法]利用硫酸新霉素诱变、筛选突变菌,并对亲本菌(ZUST)和突变菌(ZUST-1、ZUST-2)进行生长、产酸能力及H+-ATPase活性的测定。分别提取亲本菌和突变菌的基因组DNA,扩增H+-ATPase全部编码基因并测序。通过荧光定量PCR对H+-ATPase全部编码基因进行相对定量分析。[结果]突变菌的生长和产酸能力均低于亲本菌,突变菌ZUST-1和ZUST-2的H+-ATPase活性比亲本菌分别降低了10.1%和28.8%。突变菌ZUST-1和ZUST-2的atpA基因均有22个位点发生突变,而ZUST-2的atpC基因有6个位点发生突变。突变菌ZUST-1和ZUST-2的atpA在对数期基因表达水平分别比亲本菌ZUST下调了41.1%和35.7%,在稳定期分别下调了43.6%和14.2%;ZUST-1的atpC基因在对数期的表达水平比ZUST略高,在稳定期比ZUST上调了30%,而ZUST-2的atpC基因未表达。[结论]突变菌H+-ATPase活性减弱会导致其全部编码基因在稳定期表达水平上调(除ZUST-2的atpC不表达外),而且atpA和atpC基因突变导致的基因表达水平的差异是影响H+-ATPase活性的主要因素,此研究结果为进一步研究植物乳杆菌中H+-ATPase的调控机制奠定了基础。
关键词: 植物乳杆菌 H+-ATPase 突变 荧光定量PCR
Isolation, characterization and real-time RT-PCR for relative quantification of gene expression in H+-ATPase-defective mutants from Lactobacillus plantarum
Zhang Xiang1, Fang Hui1, Xie Dongfang1, Lin Yuecheng1, Tao Yingyan1, Wang Hongpeng1, Gong Jinyan1, Ge Qing1, Pan Guorong1, Huang Jun1,3

 , You Yuru1,2
, You Yuru1,2

1.Key Laboratory of Agricultural Products Chemical and Biological Processing Technology, Zhejiang University of Science and Technology, Hangzhou 310023, Zhejiang Province, China;
2.Beingmate Baby & Child Food Co., Ltd., Hangzhou 310007, Zhejiang Province, China;
3.Hangzhou Allsheng Instruments Co., Ltd, Hangzhou 310030, Zhejiang Province, China
Received 29 July 2016; Revised 30 August 2016; Published online 18 September 2016
*Corresponding author: Tel/Fax: +86-571-85070370;Jun Huang,. E-mail:hjunlzr@163.com
Tel/Fax: +86-571-85070370;Yuru You,. E-mail:youyuru0130@163.com
Supported by the National Natural Science Foundation of China (31470793, 31240054); Hangzhou Science and Technology Project of China (20140432B106); Graduate Innovation Project of Zhejiang University of Science and Technology(2015ZUSTJSKC010)
Abstract: [Objective] The aim of this study was to isolate Lactobacillus plantarum acid-sensitive mutants with lower H+-ATPase activity, and to study the mechanism of H+-ATPase regulation in Lactobacillus plantarum.[Methods] We used neomycin to isolate acid-sensitive mutants of L. plantarum, and measured H+-ATPase activity and lactic acid production of wild-type and mutants. Genomic DNA was extracted from the wild-type ZUST and two mutants (ZUST-1, ZUST-2), and used as PCR templates. H+-ATPase genes of the strain were amplified, and the PCR products were sequenced. Sequence similarity of H+-ATPase was analyzed. Real-time RT-PCR was used to evaluate the relative quantification of the H+-ATPase genes expression.[Results] The growth of the mutants was characterized in MRS broth, which revealed that their cell biomass and acid production were lower than that of the wild-type. H+-ATPase activity of the mutants ZUST-1 and ZUST-2 was 10.1% and 28.8% lower than that of the wild-type. Results showed that atpA gene of the mutants ZUST-1 and ZUST-2 existed 22 mutations by alignment of the wild-type sequence, and atpC gene of ZUST-2 existed 6 mutations. Mutants ZUST-1 and ZUST-2 atpA gene expression were 41.1% and 35.7% lower than that of the wild-type in exponential phase, 43.6% and 14.2% in stationary phase, respectively. The atpC gene expression of ZUST-1 was similar to that of the wild-type in exponential phase, and was 30% higher than that of the wild-type in stationary phase, and ZUST-2 atpC gene was not expressed.[Conclusion] The mutants with lower H+-ATPase activity were found to up-regulate the expression of H+-ATPase genes in stationary phase, except ZUST-2 atpC gene was not expressed. H+-ATPase activity has an important connection with the difference in gene expression of atpA and atpC. The results of this study will pave the way for gaining further insights into the mechanism of the H+-ATPase-defective mutants.
Key words: Lactobacillus plantarum H+-ATPase mutant real-time RT-PCR
植物乳杆菌属于乳酸杆菌属,菌体成对或成链状排列分布,不产芽孢,厌氧或兼性厌氧,革兰氏阳性菌,最适pH为6.5左右。作为一种公认的益生菌[1],植物乳杆菌与人类的生活关系密切,被广泛用于酸奶、火腿、泡菜等发酵制品[2-6]。该菌株能通过胃并定殖于肠道,维持肠道内菌群平衡,促进营养物质吸收,缓解乳糖不耐症,具有多种重要的保健功能[7-10]。本课题组前期从黄酒浸米液中筛选出1株产γ-氨基丁酸的菌株,采用生理生化实验、形态学观察及16S rRNA鉴定,结果表明该菌株为植物乳杆菌[11]。
H+-ATPase是乳酸菌产酸代谢的关键酶,既可以利用跨膜质子电化学梯度合成ATP,也可以利用ATP水解产生的能量将胞内H+排出。当外界pH下降时,H+会通过细胞膜进入胞内,对乳酸菌细胞形成酸压力,诱导细胞膜上H+-ATPase水解ATP释放自由能,推动细胞内质子泵出胞外,从而使胞内pH维持在中性附近[12-15]。刘飞等[16]利用硫酸新霉素成功筛选出H+-ATPase缺陷的德氏乳杆菌保加利亚亚种突变菌,发现突变菌的H+-ATPase活力降低,具有较低的生长速率和产酸能力。H+-ATPase由8个蛋白亚基组成,位于细胞质膜上,难以分离纯化。Koponen等[17]研究发现,将鼠李糖乳杆菌分别在最佳pH 5.8和相对酸性环境pH 4.8中进行培养,结果在pH 4.8时鼠李糖乳杆菌的H+-ATPase编码基因在对数期末期表达水平比对数期中期均上调。目前一些乳酸菌H+-ATPase的操纵子已被鉴定,但关于其具体调控机制仍鲜有报道,对植物乳杆菌的H+-ATPase调控机制也鲜有报道。本研究以植物乳杆菌ZUST作为出发菌,通过硫酸新霉素诱变筛选低H+-ATPase活性突变菌,使其产酸能力减弱,并从基因表达水平方面对H+-ATPase编码基因的调控进行研究,期望结合植物乳杆菌的功能性和弱后酸化性,使发酵的产品不仅具有良好的生理功能,而且具有弱后酸化效果,防止产品在短期内酸败,从而延长产品的保质期。
1 材料和方法 1.1 材料和主要仪器试剂 植物乳杆菌ZUST为本实验室筛选保存菌株;BacteriaGen DNA Kit,SuperQuick RT MasterMix,UltraSYBR Mixture均购自北京康为世纪生物科技有限公司;RNAstore Reagent,RNAprep Pure Bacteria Kit均购自天根生化科技有限公司;硫酸新霉素、氨苄青霉素均购自上海生工生物工程股份有限公司;ABI StepOne实时定量PCR仪;ABI PCR扩增仪;MD SpectraMax 190全波长酶标仪;UV-5500PC紫外分光光度计;Gel Doc 2000凝胶成像分析系统;Beckman超速冷冻离心机;杭州奥盛Nano-100超微量核酸分析仪;Waters高效液相色谱仪。
1.2 亲本菌株诱变及低H+-ATPase活性突变株筛选
1.2.1 菌种活化: 将-80 °C保存的植物乳杆菌接种于MRS液体培养基中,37 °C培养12 h后,在MRS液体培养基中传代2-3次。
1.2.2 生长曲线的测定: 将活化的菌株按2%的接种量接种于MRS液体培养基,37 °C培养24 h,每2 h测定OD660值,建立菌株生长曲线。
1.2.3 硫酸新霉素诱变: 将活化后的植物乳杆菌接种于MRS液体培养基中,37 °C培养8 h,使用硫酸缓冲液(PBS) (pH 7.2,10 mmol/L)稀释后,取100 μL涂布于含有硫酸新霉素的MRS平板上,硫酸新霉素的质量浓度分别为100,200,300,400和500 mg/L。
1.2.4 突变菌的筛选: 硫酸新霉素平板在37 °C培养24 h后,挑选单个菌落接种于MRS液体培养基,继续培养24 h,每隔2 h测定OD660和pH值,挑选生长产酸弱的菌株保存。 1.3 H+-ATPase活性的测定
1.3.1 建立磷浓度标准曲线: 参考Ongol等[18]报道的方法建立了磷浓度标准曲线。
1.3.2 建立蛋白含量标准曲线: 使用改良型Bradford法蛋白浓度测定试剂盒(上海生工)建立蛋白含量标准曲线。
1.3.3 H+-ATPase活性的测定: 参考Jaichumjai等[4]报道的方法稍作修改,将亲本菌及突变菌接种于MRS液体培养基中,37°C培养至对数期,收集菌体(8000×g,5min)。首先加入1mL浓度为75mmol/L的Tris-HCl缓冲液(含10mmol/L的MgSO4,pH7.0)重悬菌体,然后加入60μL甲苯和丙酮(1∶9)混合物,剧烈震荡5min,得到粗酶液。向粗酶液中加入1mL浓度为50mmol/L的Tris-maleate缓冲液(含10mmol/L的MgSO4,pH6.0),混合均匀,37°C水浴5min,加入50μL浓度为0.1mol/L的ATP,37°C水浴15min,然后加入600μL浓度为0.1mol/L的HCl终止反应,迅速放在冰上冷却,分别测定磷浓度和蛋白含量。H+-ATPase的活性用μmol/(mg·min)来表示,其中每个活力单位定义为1mgH+-ATPase的粗提取物在1min内分解ATP形成1μmol的无机磷酸盐。 1.4 乳酸含量的测定 色谱分离柱:WonCractODS-2 C18柱;检测器:UV 206 nm;流动相A:0.1 mol/L KH2PO4,流动相B:甲醇,A∶B=95∶5;流速:0.8 mL/min;进样量:10 μL。将发酵液在10000×g条件下离心10 min,取上清液经0.22 μm滤膜过滤后经适当稀释后进行色谱分析测定乳酸含量,乳酸单位用g/L来表示。
1.5 H+-ATPase编码基因的PCR
1.5.1 基因组DNA的提取及鉴定: 用基因组提取试剂盒(康为世纪)提取细菌基因组DNA,实验步骤参照说明书进行。用质量分数为1%的琼脂糖凝胶进行凝胶电泳,紫外下观察基因组条带的纯度和完整性。
1.5.2 H+-ATPase编码基因的PCR扩增: 查找GenBank中植物乳杆菌的基因组信息,发现H+-ATPase共有8个亚基,对应的编码基因分别为atpA、atpB、atpC、atpD、atpE、atpF、atpG、atpH,设计8对引物进行全酶扩增(表 1)。使用Primer Premier 5.0设计扩增引物,由通用生物系统有限公司合成。 表 1. 用于扩增H+-ATPase编码基因的PCR引物 Table 1. PCR primers for amplifying different H+-ATPase encoding genes
| Gene name | Forward primer | Reverse primer |
| atpA | ATGAGCATTAAATCTGAAGAAATCA | CTACTTCGCAGCGGTTGGC |
| atpB | GTGGGTGATCCAGTTCCTACAGTC | TTATTCCTCGTCGTTAACCTTCTGA |
| atpC | ATGGCTGACAATGCAAAATCAT | TTAATGCCGAGCAACATTCAA |
| atpD | ATGAGTACAGGTAAAGTTGTACAAGTTA | CTAATCAGTTACCATCGATTTCG |
| atpE | ATGGGAGCAATTGCTGCAGGT | TTACTTGTTCATAACCATCAAAGCA |
| atpF | ATGCTCTCGCATTTAATTATCGGT | TCAAGACTCATGCTTTCCCAACC |
| atpG | ATGGCAGAATCATTAATGGATGTC | TTATTCTTGCGCAACCAAACC |
| atpH | ATGAGTCTTGATAATCTTACAATTGC | CTAATTGTTGATGAGTGCTCGTC |
表选项
PCR反应体系:灭菌ddH2O,22 μL;基因组DNA,1 μL;正向引物,1 μL;反向引物,1 μL;Prime STAR Max Premix,25 μL。 PCR反应条件:98 °C 2 min;98 °C 10 s,55 °C 30 s,72 °C 30 s,共35个循环;72 °C 10 min。
1.5.3 PCR产物胶回收、TA克隆及测序: 采用琼脂糖凝胶纯化试剂盒(上海生工)对目的条带进行回收纯化,并电泳鉴定。用平端DNA片段添dA试剂盒(上海生工)在回收产物末端添加单个碱基A,采用T-载体PCR产物克隆试剂盒(上海生工)对其克隆,通过蓝白斑筛选挑出白色阳性克隆,接种至含有氨苄青霉素的LB试管中,培养过夜,取新鲜菌液送至通用生物系统有限公司 测序。 1.6 基因表达水平的测定
1.6.1 RNA的提取及鉴定: 将亲本菌和突变菌以2%接种量接种于MRS液体培养基中,在37 °C下培养,分别收集对数期(亲本菌ZUST及突变菌ZUST-1为8 h;突变菌ZUST-2为10 h)和稳定期(亲本菌ZUST及突变菌ZUST-1为12 h;突变菌ZUST-2为16 h)的菌体,用细菌总RNA提取试剂盒(天根)提取细胞中的总RNA,实验步骤参照说明书进行。对提取的总RNA进行1%琼脂糖凝胶电泳检测。
1.6.2 cDNA的合成: 按照SuperQuickRT MasterMix试剂盒(康为世纪)进行逆转录,步骤参照说明书。反应完毕后,-20 °C条件下保存。
1.6.3 荧光定量PCR测定基因转录情况: 采用SubGreen法测定目的基因的转录情况。根据植物乳杆菌的全部编码基因和16S rRNA基因设计荧光定量引物,其中16S rRNA基因是内参基因,引物序列如表 2。 表 2. qRT-PCR反应中所用引物 Table 2. PCR primers used in qRT-PCR
| Gene name | Forward primer | Reverse primer |
| atpA | TCGACAACGCCTTACAAGGTGAATTA | CAAAATCCCCTAAAACAACGATACCAA |
| atpB | TATGAAGCCCAAGGGTGGACAAA | TACTGCCCGACCTGAACGTGAAT |
| atpC | ATGACGAAATTGCCGTTAATGGTG | TTGGGCATTTTGAATCCGTGTTT |
| atpD | ACTTTACGGGCCAACCTG | CGGAATGCGTCTTCTGGT |
| atpE | TCTAAGATGCTTGAAGGGATGGC | CAACAACGAAGGAAATGATAGGC |
| atpF | CAACAACGAAGGAAATGATAGGC | AATGCCTGTTGGCCTTCC |
| atpG | CGTAAAGCCAAGGATAGCCGGGTAGA | GTTGACGACGAAGCATGTTCAGCAGT |
| atpH | ATTGGCGGTGTAATTGTGAA | TTGTTGATGAGTGCTCGTCTT |
| 16S rRNA | ACATCTCACGACACGAGCTG | TGAGTGCTAAGTGTTGGAGG |
表选项
用UltraSYBRMixture试剂盒(康为世纪)测定目的基因的转录水平,按说明书操作。Real-time RT PCR反应条件:95 °C 10 min;95 °C 15 s,60 °C 1 min (40个循环);熔解曲线分析:95 °C 15 s;60 °C 1 min;95 °C 15 s。 每个样品重复3次。以16S rRNA作为内参基因,根据-△△Ct法测定全部编码基因在不同时期的相对表达量。按照公式(1)、(2)和(3)计算[19]。 △Ct目的基因=Ct(目的基因)-Ct (同一样本的内参基因)?公式(1) △△Ct目的基因=实验组△Ct目的基因-参照组△Ct目的基因?公式(2) 相对倍数(实验组/对照组) = 2-△△Ct?公式(3) 其中Ct为循环阈值(cycle threshold value),代表PCR扩增过程中扩增产物的荧光信号达到设定的荧光阈值所经过的循环数。2-△△Ct代表目标基因表达量的变化倍数。 2 结果和分析 2.1 低H+-ATPase活性突变株的筛选 从每个含硫酸新霉素的MRS平板中随机挑取5个单菌落,分别接种于MRS液体培养基中,37 °C培养24 h,通过测定OD660和pH值,挑选出1株突变菌,命名为ZUST-1。再对ZUST-1进行诱变,挑选出另一株突变菌ZUST-2。
各菌株生长曲线和pH (图 1-A)测定实验表明:亲本菌ZUST和突变菌ZUST-1均在4 h进入对数期,在12 h达到稳定期,而突变菌ZUST-2在8 h进入对数期,16 h达到稳定期。突变菌ZUST-1和ZUST-2生长能力均低于亲本菌ZUST,ZUST-1在进入稳定期后与ZUST基本相近,而ZUST-2的差异最显著;pH测定结果与生长能力成对应的关系,突变菌ZUST-1在对数期与亲本菌ZUST的pH差异很小,进入稳定期后几乎相同,在24 h时pH为3.7,而突变菌ZUST-2的产酸能力最弱,在24 h时pH为4.0。发酵液乳酸含量(图 1-B)测定结果表明:亲本菌ZUST和突变菌ZUST-1在对数期和稳定期发酵液中乳酸含量差异很小,趋于一致。突变菌ZUST-2在对数期和稳定期发酵液中乳酸含量均低于亲本菌ZUST和突变菌ZUST-1,且在稳定期差异比对数期小。这与pH测定结果一致,突变菌ZUST-2的pH均高于亲本菌ZUST和突变菌ZUST-1,但在稳定期的差异比对数期小。H+-ATPase活力(图 1-C)测定结果表明:突变菌ZUST-1和ZUST-2酶活力比亲本菌ZUST分别下降了10.1%和28.8%,说明由于H+-ATPase活力的下降,导致ZUST-1和ZUST-2的生长代谢能力减弱,使突变菌OD660均低于亲本菌,pH均高于亲本菌,发酵液中乳酸含量均低于亲本菌,同时ZUST-2的酶活力比ZUST-1更低,所以ZUST-2生长产酸能力最弱。
 |
| 图 1. 生长曲线和产酸能力、乳酸含量和酶活力分析 Figure 1. The growth curve,acid production ability,the concentration of lactic acid and H+-ATPase activity assay. |
| 图选项 |
2.2 突变位点分析
2.2.1 基因组DNA的提取: 基因组DNA电泳结果(图 2)均得到单一清晰的条带,OD260/OD280为1.8,表明提取的DNA较完整,没有RNA、蛋白的污染,可直接用于PCR扩增H+-ATPase的编码基因。
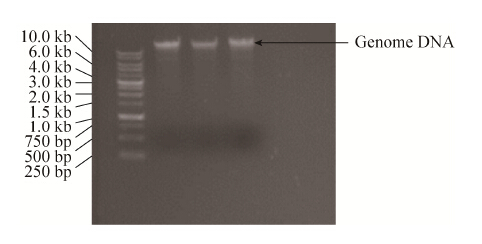 |
| 图 2. 基因组DNA琼脂糖凝胶电泳检测图 Figure 2. Agarose gel electrophoresis of genomic DNA. |
| 图选项 |
2.2.2 H+-ATPase全部编码基因的扩增: H+-ATPase全部编码基因扩增结果表明,亲本菌ZUST (图 3-A),突变菌ZUST-1 (图 3-B),突变菌ZUST-2 (图 3-C)均已全部成功扩增,但有杂带,经胶回收纯化后,亲本菌ZUST (图 4-A),突变菌ZUST-1 (图 4-B),突变菌ZUST-2 (图 4-C)都得到只含目的片段的单一条带,可以继续后续实验。
 |
| 图 3. 目的基因PCR扩增琼脂糖电泳检测图 Figure 3. Agarose gel electrophoresis of PCR amplification for the target genes. |
| 图选项 |
 |
| 图 4. 目的基因PCR产物胶回收电泳图 Figure 4. Gel extraction of PCR products for the target genes. |
| 图选项 |
2.2.3 测序结果分析: 用DNAMAN对测序结果进行比对,结果如表 3所示。突变菌ZUST-1的atpA基因上有22个突变位点,其它编码基因均无突变。突变菌ZUST-2的atpA基因上有22个位点发生突变,而且atpC基因上有6个位点发生突变,其中有2个发生移码突变。为确保测序结果准确性,每个编码基因均送测3次。 表 3. 基因序列分析结果 Table 3. The results of the gene sequence similarity
| Gene name | Sequence size | Numbers of mutations | |
| ZUST-1 | ZUST-2 | ||
| atpA | 1515 bp | 22 | 22 |
| atpB | 714 bp | 0 | 0 |
| atpC | 429 bp | 0 | 6(2) |
| atpD | 1404 bp | 0 | 0 |
| atpE | 213 bp | 0 | 0 |
| atpF | 516 bp | 0 | 0 |
| atpG | 945 bp | 0 | 0 |
| atpH | 546 bp | 0 | 0 |
表选项
2.3 亲本和突变菌H+-ATPase表达量的相对定量分析
2.3.1 总RNA非变性琼脂糖凝胶电泳结果: 总RNA电泳结果(图 5)得到两条清晰条带,分别为23S rRNA和16S rRNA,而且OD260/OD280为2.0,表明RNA提取完整,无污染,可以用于后续实验。
 |
| 图 5. 总RNA电泳检测图 Figure 5. The gel electrophoresis of total RNA. |
| 图选项 |
2.3.2 荧光定量PCR检测H+-ATPase的表达: H+-ATPase (图 6-A)由8个编码基因组成,分别编码8个蛋白亚基。其中atpB、atpE、atpF编码的a、c、b亚单位共同组成膜质子通道F0,其它5个亚单位组成编码复合体F1,负责对质子通道的装配及调控[20]。
 |
| 图 6. H+-ATPase结构以及H+-ATPase全部编码基因表达水平 Figure 6. The H+-ATPase structure and its gene expression. |
| 图选项 |
各菌株编码基因表达水平实验结果(图 6-B)表明:所有菌株的atpA和atpC基因在稳定期的表达水平均比对数期高(除ZUST-2的atpC不表达外),而亲本菌ZUST的其它6个基因在稳定期的表达水平均比对数期低,突变菌ZUST-1和ZUST-2的其它6个基因在稳定期的表达水平均比对数期高。分析认为,突变菌ZUST-1和ZUST-2由于发生基因突变,酶活力减弱,在进入稳定期后,酸胁迫环境愈加严峻,菌株为保证自身存活,产生一系列应激反应,从而导致其它6个编码基因的表达水平上调。亲本菌和突变菌atpA和atpC基因表达水平在稳定期均高于对数期(除ZUST-2的atpC不表达外),具有一致性,在稳定期为应对酸胁迫环境,基因表达水平均上调,说明atpA和atpC是调控H+-ATPase活力的主要基因。突变菌ZUST-1的atpA在对数期和稳定期的基因表达水平分别比亲本菌ZUST下调了41.1%和43.6%,突变菌ZUST-2在对数期和稳定期的基因表达水平分别比亲本菌下调了35.7%和14.2%。突变菌ZUST-1的atpC基因在对数期表达水平比亲本菌ZUST略高,在稳定期的表达水平上调了30%,而突变菌ZUST-2在对数期和稳定期均未表达。 3 讨论 植物乳杆菌是食品工业上常用的发酵剂,硫酸新霉素能够使乳酸菌H+-ATPase的某个亚基发生突变,从而使乳酸菌的生长代谢能力减弱,关于利用硫酸新霉素选育弱后酸化的乳酸菌的研究多有报道[21-23]。本研究通过利用硫酸新霉素成功诱变、筛选出2株突变菌,发现突变菌生长产酸能力均低于亲本菌。通过对H+-ATPase的酶活力进行测定,结果表明突变菌的酶活力均低于亲本菌。研究表明,H+-ATPase是控制乳酸菌生长代谢的关键酶,在正常生长条件下能够维持乳酸菌的胞内pH在中性附近,从而提高乳酸菌的耐酸性[24]。由于H+-ATPase的酶活力减弱,从而使突变菌的生长产酸能力下降,这一结果与报道内容一致。
目前关于弱后酸化乳酸菌的选育研究虽然有很多,但对弱后酸化机理的研究却鲜有报道,即使有限的报道也是观点与机理不一。崔兰[25]对弱后酸化保加利亚乳杆菌的研究发现,突变菌的atpG表达水平在对数期和稳定期分别比亲本菌下调了76%和67%。刘飞等[26]通过对弱后酸化保加利亚乳杆菌突变株和亲本菌株的H+-ATPase基因相似性进行研究,发现突变菌和亲本菌的H+-ATPase编码基因相似性为100%,认为突变株和亲本菌之间的H+-ATPase活力的差异并不是由于编码基因发生了突变,可能是转录水平的降低导致H+-ATPase蛋白的表达量降低,进而影响酶活力。本研究为进一步阐述突变菌的弱后酸化机理及H+-ATPase的调控机制,分别对亲本菌和突变菌H+-ATPase的全部编码基因进行测序,结果发现突变菌ZUST-1和ZUST-2的atpA编码基因上均有22个位点发生突变,而且ZUST-2的atpC编码基因上有6个位点发生突变,包括两个移码突变位点。对亲本菌和突变菌全部编码基因表达水平的测定,结果发现突变菌所有编码基因表达水平在稳定期均高于对数期,亲本菌和突变菌atpA及atpC基因表达水平在稳定期均高于对数期(除ZUST-2的atpC不表达外),具有一致性,而其它6个基因则恰好相反,亲本菌ZUST在稳定期表达水平均低于对数期,突变菌ZUST-1及ZUST-2在稳定期表达水平均高于对数期。突变菌ZUST-1和ZUST-2的atpA基因的表达水平均比亲本菌低,而ZUST-2的atpC不表达,综合分析认为atpA和atpC这两个编码基因对H+-ATPase的功能均有影响,而atpC的影响可能起主导作用。虽然突变菌ZUST-2atpA基因的表达水平在稳定期比突变菌ZUST-1要高,但由于atpC基因不表达,导致ZUST-2的H+-ATPase活力比ZUST-1更低,具体表现为ZUST-2的生长产酸能力最弱,发酵液乳酸含量最低。突变菌ZUST-1的atpA基因表达水平与亲本菌ZUST相比下调幅度较大,但是其atpC基因的表达水平在对数期比ZUST略高,稳定期与ZUST差异更大。所以,最终表现为突变菌ZUST-1与亲本菌ZUST的生长产酸能力差异不大,在稳定期比对数期的差异更小,与图 1实验结果相符。
本研究通过实时荧光定量PCR分析了亲本菌和突变菌的基因表达水平,结果表明突变菌H+-ATPase活力下降是由于其编码基因发生突变,进而引起该编码基因的表达水平下调或不表达,最终导致酶活力下降,具体表现为菌体生长产酸能力减弱,从而达到弱后酸化的效果。通过对两株突变菌的测序结果分析,发现atpA上发生突变的位点相同,推测H+-ATPase活性降低的植物乳杆菌突变菌的突变位点可能存在一定的规律性,而突变菌ZUST-2上的atpC发生突变是导致ZUST-2与突变菌ZUST-1差异的主要原因,说明atpC基因对该酶活力的影响更大,可能是调控H+-ATPase功能的关键基因。本研究为进一步阐述H+-ATPase在植物乳杆菌中的调控机制奠定了基础,但关于H+-ATPase中的每个编码基因的作用及其编码的蛋白亚基对H+-ATPase的单独调控作用及其协同作用有待进一步深入的研究。
参考文献
| [1] | Wassenaar TM, Klein G. Safety aspects and implications of regulation of probiotic bacteria in food and food supplements.Journal of Food Protection, 2008, 71(8): 1734–1741DOI:10.4315/0362-028X-71.8.1734. |
| [2] | Yu ZH, Zhang X, Li SY, Hou JM, Wang J, Li D, Yang ZN. Study on probiotic yoghurt.China Dairy Industry, 2013, 41(7): 27–30(in Chinese).于志会, 张雪, 李盛钰, 侯聚敏, 王辑, 李达, 杨贞耐. 植物乳杆菌C88在酸奶中的应用.中国乳品工业, 2013, 41(7): 27–30. |
| [3] | Zhang Y, Meng XC. Isolation, identification and genetic diversity analysis of lactobacilli from naturally fermented northeast sauerkraut.China Dairy Industry, 2009, 37(2): 19–22(in Chinese).张杨, 孟祥晨. 自然发酵酸菜发酵液中乳杆菌的分离鉴定与多态性分析.中国乳品工业, 2009, 37(2): 19–22. |
| [4] | Jaichumjai P, Valyasevi R, Assavanig A, Kurdi P. Isolation and characterization of acid-sensitive Lactobacillus plantarum with application as starter culture for Nham production.Food Microbiology, 2010, 27(6): 741–748DOI:10.1016/j.fm.2010.03.014. |
| [5] | Hammes WP, Bantleon A, Min S. Lactic acid bacteria in meat fermentation.FEMS Microbiology Letters, 1990, 87(1/2): 165–173. |
| [6] | Ho N, Prasad V. Probiotics, prebiotics, synbiotics and naturally fermented foods:why more may be more.Annals of Gastroenterology, 2013, 26(3): 277–278. |
| [7] | Tannock GW, Dashkevicz MP, Feighner SD. Lactobacilli and bile salt hydrolase in the murine intestinal tract.Applied and Environmental Microbiology, 1989, 55(7): 1848–1851. |
| [8] | Caramia G. Gastroenteric pathology and probiotics:from myth to scientific evidence.Minerva Gastroenterologicae Dietologica, 2009, 55(3): 237–272. |
| [9] | Pieper R, Janczyk P, Urubschurov V, Hou ZP, Korn U, Pieper B, Souffrant WB. Effect of Lactobacillus plantarum, on intestinal microbial community composition and response to enterotoxigenic Escherichia coli challenge in weaning piglets.Livestock Science, 2010, 133(1/3): 98–100. |
| [10] | Ra?i? JL, Vuji?i? IF, ?krinjar M, Vuli? M. Assimilation of cholesterol by some cultures of lactic acid bacteria and Bifidobacteria.Biotechnology Letters, 1992, 14(1): 39–44DOI:10.1007/BF01030911. |
| [11] | Gong JY, Xie T, Lou J, Hu S, Mei LH, Xie DF, Huang J. Screening and identification of lactic acid bacteria for biosynthesis of γ-aminobutyric acid from the leaching solution of Chinese rice wine.Industrial Microbiology, 2015, 45(6): 26–31(in Chinese).龚金炎, 谢湉, 楼坚, 胡升, 梅乐和, 谢东芳, 黄俊. 黄酒浸米液中产γ-氨基丁酸乳酸菌的筛选和鉴定.工业微生物, 2015, 45(6): 26–31. |
| [12] | Kobayashi H, Suzuki T, Unemoto T. Streptococcal cytoplasmic pH is regulated by changes in amount and activity of a proton-translocating ATPase.The Journal of Biological Chemistry, 1986, 261(2): 627–630. |
| [13] | Yokota A, Amachi S, Ishii S, Tomita F. Acid sensitivity of a mutant of Lactococcus lactis subsp.Biotechnology, and Biochemistry, 1995, 59(10): 2004–2007DOI:10.1271/bbb.59.2004. |
| [14] | Yamamoto N, Masujima Y, Takano T. Reduction of membrane-bound ATPase activity in a Lactobacillus helveticus strain with slower growth at low pH.FEMS Microbiology Letters, 1996, 138(2/3): 179–184. |
| [15] | Senouci-Rezkallah K, Jobin MP, Schmitt P. Adaptive responses of Bacillus cereus ATCC14579 cells upon exposure to acid conditions involve ATPase activity to maintain their internal pH.Microbiology Open, 2015, 4(2): 313–322DOI:10.1002/mbo3.2015.4.issue-2. |
| [16] | Liu F, Du P, Wang YT, Liu F, Huo GC. Screening of H+-ATPase deficient mutant of Lactobacillus delbrueckii subsp.Acta Microbiologica Sinica, 2009, 49(1): 38–43(in Chinese).刘飞, 杜鹏, 王玉堂, 刘芳, 霍贵成. 保加利亚乳杆菌H+-ATPase缺陷型菌株的筛选.微生物学报, 2009, 49(1): 38–43. |
| [17] | Koponen J, Laakso K, Koskenniemi K, Kankainen M, Savijoki K, Nyman T, de Vos WM, Tynkkynen S, Kalkkinen N, Varmanen P. Effect of acid stress on protein expression and phosphorylation in Lactobacillus rhamnosus GG.Journal of Proteomics, 2012, 75(4): 1357–1374DOI:10.1016/j.jprot.2011.11.009. |
| [18] | Ongol MP, Sawatari Y, Ebina Y, Sone T, Tanaka M, Tomita F, Yokota A, Asano K. Yoghurt fermented by Lactobacillus delbrueckii subsp.International Journal of Food Microbiology, 2007, 116(3): 358–366DOI:10.1016/j.ijfoodmicro.2007.02.019. |
| [19] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the method.Methods, 2001, 25(4): 402–408DOI:10.1006/meth.2001.1262. |
| [20] | 孙懿琳. 弱后酸化保加利亚乳杆菌菌株的筛选及其后酸化机理. 东北农业大学硕士学位论文, 2013. http://cn.bing.com/academic/profile?id=c014735a254d517ad00edcf319963035&encoded=0&v=paper_preview&mkt=zh-cn |
| [21] | Wang XN, Chen M, Sun CL, Wurentuya, Dan T, Bao QH, Zhang HP, Menghebilige. Mutation breeding of H+-ATPase deficient mutant of Lactobacillus delbrueckii subsp.China Dairy Industry, 2013, 41(8): 8–11(in Chinese).王雪妮, 陈明, 孙春玲, 乌仁图雅, 丹彤, 包秋华, 张和平, 孟和毕力格. 保加利亚乳杆菌H+-ATPase弱化菌株的诱变选育.中国乳品工业, 2013, 41(8): 8–11. |
| [22] | Wang XH, Ren HY, Liu DY, Wang B, Zhu WY, Wang W. H+-ATPase-defective variants of Lactobacillus delbrueckii subsp.Journal of Food Science, 2013, 41(8): 8–11,28. |
| [23] | Jia BH, Zhong XT, Yuan CH, Li K, Lin K, Zhang Q, Che ZM, Chen G, Xiang WL. Screening of Lactobacillus plantarum LPM21 with F1F0-ATPase β-subunit mutation used as probiotics adjunct in Sichuan pickle.Food Science and Technology Research, 2013, 19(6): 1045–1050DOI:10.3136/fstr.19.1045. |
| [24] | Nyanga-Koumou AP, Ouoba LII, Kobawila SC, Louembe D. Response mechanisms of lactic acid bacteria to alkaline environments:a review.Critical Reviews in Microbiology, 2012, 38(3): 185–190DOI:10.3109/1040841X.2011.640978. |
| [25] | 崔兰. 弱后酸化保加利亚乳杆菌的应激性及其遗传稳定性研究. 哈尔滨: 东北农业大学硕士学位论文, 2012. http://cn.bing.com/academic/profile?id=5d61603d7e9796685520d720b761b55e&encoded=0&v=paper_preview&mkt=zh-cn |
| [26] | Liu F, Jiao YH, Guo WK, Yu W, Gu CT, Huo GC. Comparison of similarity of H+-ATPase coding gene between wild strain and mutant strain with low post-acidification of Lactobacillus delbrueckii subsp.Science and Technology of Food Industry, 2013, 34(15): 127–130(in Chinese).刘飞, 焦月华, 郭文奎, 于微, 谷春涛, 霍贵成. 弱后酸化保加利亚乳杆菌突变株与亲本菌株H+-ATPase基因的相似性比较.食品工业科技, 2013, 34(15): 127–130. |
