高芝, 张梁

 , 李由然, 顾正华, 丁重阳, 石贵阳
, 李由然, 顾正华, 丁重阳, 石贵阳 江南大学粮食发酵工艺与技术国家工程实验室, 工业生物技术教育部重点实验室, 江苏 无锡 214122
收稿日期:2016-12-24;修回日期:2017-02-16;网络出版日期:2017-03-14
基金项目:国家星火计划重点项目(2015GA690004);江苏省****基金(BK20140002)
*通信作者:张梁, E-mail:zhangl@jiangnan.edu.cn
摘要:[目的]构建一个适用于Candida amazonensis抗性标记可重复使用的FLP/FRT基因敲除系统,并通过敲除C. amazonensis的丙酮酸脱羧酶基因(Pyruvate decarboxylase,PDC)对该系统进行初步验证。[方法]以gfpm(绿色荧光蛋白基因)为报告基因,通过添加相应诱导剂评估Spathaspora passalidarum来源启动子(SpXYLp、SpMAL6p、SpMAL1p、SpGAL1p)和Saccharomyces cerevisiae来源ScGAL1p启动子在C. amazonensis中的诱导调控性能。选择严格诱导型启动子调控FLP重组酶的表达,并在FLP表达盒和潮霉素(Hygromycin B)抗性标记基因(hphm)两端添加同向重复的FRT位点,以PDC基因作为靶基因构建敲除盒PRFgHRP,转化宿主菌C. amazonensis CBS 12363,筛选得到阳性转化子后,通过添加诱导剂,表达FLP重组酶,实现FRT位点间片段切除。[结果]诱导调控实验表明启动子SpGAL1p(受半乳糖诱导)和SpMAL1p(受麦芽糖诱导)是适用于C. amazonensis的严格诱导型启动子。以SpGAL1p调控FLP基因表达,构建的敲除盒PRFgHRP成功转化宿主菌,获得阳性转化子C. amazonensis PDC01,通过添加半乳糖诱导,成功切除基因组中FLP表达盒和抗性标记盒,获得突变株C. amazonensis PDC02。[结论]首次建立了一个适用于C. amazonensis抗性标记可重复使用的FLP/FRT基因敲除系统,并利用该系统成功敲除了C. amazonensis内的PDC基因,为进一步利用代谢工程改造C. amazonensis酵母奠定了良好基础。
关键词: Candida amazonensis FLP/FRT 诱导型启动子 基因敲除
Development and verification of an FLP/FRT system for gene deletion in Candida amazonensis
Zhi Gao, Liang Zhang

 , Youran Li, Zhenghua Gu, Zhongyang Ding, Guiyang Shi
, Youran Li, Zhenghua Gu, Zhongyang Ding, Guiyang Shi National Engineering Laboratory for Cereal Fermentation Technology, Key Laboratory of Industrial Biotechnology of Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu Province, China
Received 24 December 2016; Revised 16 February 2017; Published online 14 March 2017
*Corresponding author: Liang Zhang, E-mail:zhangl@jiangnan.edu.cn
Supported by the China Spark Program (2015GA690004) and by the Jiangsu Science Fund for Distinguished Young Scholars (BK20140002)
Abstract: [Objective]To develop an FLP/FRT system for gene disruption in Candida amazonensis that can repeatedly use a single selectable marker, and to verify the effectiveness of this system by deleting the PDC gene encoding pyruvate decarboxylase.[Methods]Four promoters (SpXYLp, SpMAL6p, SpMAL1p and SpGAL1p) from Spathaspora passalidarum and ScGAL1p promoter from Saccharomyces cerevisiae were amplified and fused to the reporter gene of green fluorescent protein (gfpm) to study the regulation under corresponding inducible conditions. A strictly inducible promoter was selected to control the expression of the C. amazonensis-adapted FLP gene (caFLP), encoding the site-specific recombinase FLP. The promoter-caFLP fusion fragment was used to ligated with the hphm marker gene that conferred resistance to Hygromycin B, and the ligation product was flanked by direct repeats of the FLP recognition target (FRT). Then with the addition of the homologous arms, we constructed the PDC deletion cassette (PRFgHRP). The cassette was transformed into C. amazonensis CBS 12363 and transformants with hphm were derived. When the transformants were incubated into inducible medium, FLP-mediated recombination resulted in the deletion of DNA located between the repeats.[Results]SpMAL1p (induced by maltose) and SpGAL1p (induced by galactose) were identified to be strictly inducible promoters. SpGAL1p was used to regulate the expression of the FLP, and the PDC deletion cassette (PRFgHRP) was constructed and transformed into C. amazonensis successfully. After selection of Hyg-resistant transformant (designated as C. amazonensis PDC01) in which the deletion cassette was inserted into the PDC target gene, FLP expression was induced by growth of the transformant in galactose-containing medium, and Hyg-sensitive transformant in which hphm and caFLP flippers were excised from the genome was obtained, designated as C. amazonensis PDC02.[Conclusion]It is the first time to construct an FLP/FRT system for gene disruption in C. amazonensis, and we obtained a PDC mutant without resistant marker gene successfully through this system. These research results lay a good foundation for further metabolic engineering of C. amazonensis.
Key words: Candida amazonensis FLP/FRT inducible promoters gene disruption
Candida amazonensis是近年来从亚马逊森林中新分离出的一株能够高效利用木糖的酵母,该酵母能够代谢多种糖类,同时具有发酵纤维二糖、海藻糖等的能力[1-2]。在研究中发现C. amazonensis具有快速代谢木糖的能力,并在木糖醇生产方面表现出一定优势。实验室前期研究注意到该酵母分别在高木糖浓度(350 g/L)、高温42、pH 2.5等胁迫条件下仍能有效发酵木糖并积累较高的生物量,表现出良好的耐高糖、耐高温以及耐酸性能,有望成为新型木糖发酵模式菌株[3]。
然而,良好的模式菌株,除自身具有的优良特性外,还需要基因表达与敲除等基本的分子生物学手段的有效支持,用以充分挖掘相关菌株优良信息。目前,C. amazonensis基因水平上的研究尚处于起步阶段,至今还未有全基因组测序的工作展开。此外,由于该酵母属于利用木糖类酵母,使用特殊的基因编码系统,即密码子CTG编码的是丝氨酸而非常见的亮氨酸[4],这使得常规表达系统所对应的一些载体元件包括筛选标记、报告基因等难以有效用于该CTG类酵母中。实验室前期利用定点突变后(9个CTG突变为亮氨酸密码子TTG)的潮霉素抗性基因hphm和绿色荧光蛋白基因gfpm (1个CTG突变为TTG)以及来自Spathaspora passalidarum酵母的启动子、终止子等元件成功构建了一个适用于C. amazonensis的整合型表达载体PRACTH-gfpm[5],然而,有限的筛选标记很难满足人们对于代谢工程育种的要求。因此,对于新型木糖利用酵母C. amazonensis,目前急需一个筛选标记可重复使用的基因敲除系统来对其在菌种改造方面进行更深入的研究。近年来,在CTG类酵母中发展起来的FLP/FRT位点特异性重组系统[6-10],因重组效率和靶向性高、可快速准确地实现抗性消除而受到众多研究者的青睐。
FLP/FRT系统由FLP重组酶和FRT识别位点两部分组成。FLP是一个48 kDa的多肽单体蛋白,其可以介导34 bp的FRT重复序列位点特异性重组,切除同向重复的2个FRT位点间的DNA片段和1个FRT位点,保留另一个FRT位点。早在1999年Staib等[7]就选择在Candida albicans中将密码子修饰过后(3个CTG突变为TTG)的FLP重组酶基因置于SAP2 (与分泌天门冬氨酰蛋白酶有关)的启动子下进行调控,并在FLP表达盒和霉酚酸抗性标记基因(IMH3R)两端添加同向重复的FRT位点,由此构建的敲除系统在SAP2启动子对FLP酶的调控下,实现2个FRT位点之间片段的切除。同年,Morschh?user等[8]在抗性标记被URA3营养缺陷型标记替代的基础上,使用同样的技术在C. albicans中进行了连续分别2轮敲除CDR4基因和MDR1基因,获得突变纯合株,成功实现了FLP/FRT系统在C. albicans中抗性重复使用和多基因连续敲除方面的应用。此外,Sánchez-Martínez等[9]和Reu?等[10]选择用一个C. albicans自身来源的麦芽糖诱导型启动子MAL2 (替换前面的SAP2)构建的FLP/FRT系统也成功实现了C. albicans中同向FRT位点之间序列的删除,其中FLP重组酶的表达受麦芽糖的诱导,因此通过培养基中麦芽糖的有无就可以实现对FLP表达的调控,从而可实现对FRT位点间片段切除在时空顺序上的控制。在这基础上,当Ding & Butler[11]将该敲除系统应用于Candida parapsilosis中时,发现只有将CaMAL2启动子替换成宿主自身来源的CpMAL2时,才能有效行使其功能。而让人意外的是,Gácser等[12]和Nguyen等[13]在后来相继报道称不需改变CaMAL2启动子序列,便可在C. parapsilosis中成功实现由FLP重组酶介导的敲除。
基于以上FLP/FRT系统在CTG类酵母中的研究应用,对于能够在C. amazonensis中适用的FLP/FRT敲除系统的获得,其关键在于拥有一个能严格调控FLP酶表达的启动子,即只有在诱导条件下启动活性明显而非诱导条件下几乎无活性的严格诱导型启动子。目前,已有不少调控型启动子被克隆及功能鉴定,其中研究频率比较高的诱导型启动子包括:来源于酿酒酵母的由半乳糖诱导而受葡萄糖抑制的GAL1和GAL10启动子[14];经铜离子激活的CUP1启动子[15];受葡萄糖抑制、麦芽糖诱导的MAL62 (酿酒酵母来源)[16]和MAL2 (白色假丝酵母来源)[9-10];以及受木糖诱导的XYL启动子[17]。此外还有一些像MET3和MET25等受蛋氨酸抑制的调节型启动子[18];受无机磷酸盐负调控的PHO5启动子[19]等等。这些启动子的诱导或抑制效果都比较明显,但大多数启动子在非诱导条件下甚至是抑制的条件下没有办法做到完全关闭其启动活性,而是仍然有一定量的基础表达,难以达到严格诱导的标准。此外,不同来源的启动子或者不同的表达宿主因为其遗传背景不同,可能导致启动子的行使功能有差异。
本文着重对适用于C. amazonensis的诱导型启动子进行了筛选,并利用获得的严格诱导型启动子SpGAL1p (受半乳糖诱导)和定点突变后的FLP重组酶基因构建了抗性标记可重复利用的FLP/FRT基因敲除系统,并通过C. amazonensis内PDC基因的敲除得到验证。
1 材料和方法 1.1 菌株与质粒 本研究中所使用的菌株、质粒如表 1所示,引物序列如表 2所示。
表 1. 文中所用主要菌株及质粒 Table 1. Main strains and plasmids used in this study
| Strain or plasmid names | Short names | Sources |
| Escherchia coli JM109 | E. coli JM109 | Laboratory |
| Saccharomyces cerevisiae W303-1A | S. cerevisiae | Laboratory |
| Spathaspora passalidarum NRRL Y-27907 | S. passalidarum | Louisiana State University |
| Candida amazonensis CBS 12363 | C. amazonensis | CBS (Centraalbureau voor Schimmelcultures) |
| pMD19-T simple vector | pMD19-T simple | TaKaRa Biotechnology (Dalian) Co., Ltd. |
| PRACTH-gfpm | SpADHp-gfpm | Laboratory |
| PRXCTH-gfpm | SpXYLp-gfpm | This study |
| PRM6CTH-gfpm | SpMAL6p-gfpm | This study |
| PRM1CTH-gfpm | SpMAL1p-gfpm | This study |
| PRG1CTH-gfpm | SpGAL1p-gfpm | This study |
| PRGc1CTH-gfpm | ScGAL1p-gfpm | This study |
| PRG1CTH-caFLP | SpGAL1p-caFLP | This study |
| Ts-PDCL-FRT-SpGAL1p-caFLP-hphm-FRT-PDCR | Ts-PRFgHRP | This study |
表选项
表 2. 文中所用到的引物 Table 2. Primers used in this study
| Primers | Sequences (5′→3′) | Restriction sites |
| SpXYLp-F | GCCGGAAGATCTGTGACATAGTTAACTATGGC | Bgl Ⅱ |
| SpXYLp-R | ACGCGTCGACTTTATTGTATTGTG | Sal Ⅰ |
| SpMAL6p-F | GCCGGAAGATCTGAATATCAATACGTTTTAGATCACCG | Bgl Ⅱ |
| SpMAL6p-R | GCCGCACGCGTCGACATTTAAAAAGTATTTATGATTTG | Sal Ⅰ |
| SpMAL1p-F | ATCGCGGATCCTGTGGGTATTTTTACAGCAGGATG | BamH I |
| SpMAL1p-R | CCGCACGCGTCGACATTCAACTGTTTGTTAATATATG | Sal Ⅰ |
| SpGAL1p-F | TGGAAGATCTAGGGGTTGGAAGAAGAAAAAATCGG | Bgl Ⅱ |
| SpGAL1p-R | ACGCGTCGACAGTAAGAATTTGATAAACTTTGCG | Sal Ⅰ |
| ScGAL1p-F | CGGAAGATCTGGAAACGTTGTATTGTTGCAT | Bgl Ⅱ |
| ScGAL1p-R | ACGCGTCGACTATAGTTTTTTCTCCTTGACGTT | Sal Ⅰ |
| FLP-F | GACGCGTCGACATGCCACAATTTGGTATATTATGT | Sal Ⅰ |
| FLP-R | CATAAGAATGCGGCCGCTTATATGCGTCTATTTATGTAGG | Not Ⅰ |
| FLP-1 | GTTTCGATATTGTCAATAAATCACTC | |
| FLP-2 | TCAACGAATTGCTTATGATAG | |
| FLP-3 | GAAGCCTCATTAAAGAAATTG | |
| FLP-4 | CAAAATTGTTGCTTTTTGCG | |
| FLP-5 | GAGTAATAATCCAGTGTT | |
| FLP-6 | CCAAATACTTATTTTGGAC | |
| PDC-F | ATGTCGGAAATTTCTTTAGGTAGAT | |
| PDC-R | CATCCATTCTTGGCAACATAACTTC | |
| PDC-1 | GCAAAAGGCCTGTCGGAAATTTCTTTAGGTAGAT | Stu Ⅰ |
| PDC-2 | GCACCAACAGAGAAACCAATCGGGGTACCCCGGTATGATGTAATAATAATTG | Kpn Ⅰ |
| PDC-3 | CAATTATTATTACATCATACCGGGGTACCCCGATTGGTTTCTCTGTTGGTGC | Kpn Ⅰ |
| PDC-4 | AAAAGGCCTCATCCATTCTTGGCAACATAACTTC | Stu Ⅰ |
| FRT-1 | CGGGGTACCGAAGTTCCTATTCTCTAGAAAGTATAGGAACTTCAGGGGTTGGAAGAAGAAAAAATCGG | Kpn Ⅰ |
| FRT-2 | ATAGGGGTACCGAAGTTCCTATACTTTCTAGAGAATAGGAACTTCAACTCCTTCCTTTTCGGTTAG | Kpn Ⅰ |
| The underlined are restriction enzyme cutting sites. | ||
表选项
1.2 培养基 LB培养基(g/L):蛋白胨10,酵母提取物5,氯化钠10。用于固体培养基时添加1.6%的琼脂粉;筛选转化子时添加终质量浓度为100 μg/mL的氨苄青霉素。
YEPD培养基(g/L):蛋白胨20,酵母提取物10,葡萄糖20。用于固体培养基时添加1.6%的琼脂粉;筛选转化子时添加终质量浓度为900 μg/mL的潮霉素B。
木糖诱导培养基YEPX (g/L):蛋白胨20,酵母提取物10,木糖20。用于固体培养基时添加1.6%的琼脂粉。
麦芽糖诱导培养基YEPM (g/L):蛋白胨20,酵母提取物10,麦芽糖20。用于固体培养基时添加1.6%的琼脂粉。
半乳糖诱导培养基YEPG (g/L):蛋白胨20,酵母提取物10,半乳糖20。用于固体培养基时添加1.6%的琼脂粉。
1.3 诱导型启动子的筛选
1.3.1 重组载体的构建: 采用启动子在线测评软件,对GenBank (http://www.ncbi.nlm.nih.gov/)提供的S. passalidarum NRRL Y-27907基因组序列(GenBank:NZ_AEIK00000000)中的木糖还原酶、α-麦芽糖苷酶1、α-麦芽糖苷酶6、以及β-半乳糖苷酶基因的ORF上游核苷酸序列进行在线预测,并设计引物分别扩增其上游约1200 bp左右的序列以保证启动子的启动活性。以S. passalidarum基因组DNA为模板,分别用引物SpXYLp-F/SpXYLp-R、SpMAL6p-F/SpMAL6p-R、SpMAL1p-F/SpMAL1p-R和SpGAL1p-F/SpGAL1p-R PCR扩增木糖还原酶基因启动子SpXYLp、α-麦芽糖苷酶基因启动子SpMAL6p、SpMAL1p、β-半乳糖苷酶基因启动子SpGAL1p;以S. cerevisiae基因组DNA为模板,用引物ScGAL1p-F/ScGAL1p-R PCR扩增其β-半乳糖苷酶基因启动子ScGAL1p。将以上5个启动子序列分别克隆至pMD19-T simple载体。以实验室前期构建的整合型质粒PRACTH-gfpm (简称为SpADHp-gfpm)为载体[5],经Bgl Ⅱ和Sal Ⅰ双酶切后回收大片段并分别与以上克隆至pMD19-T simple载体经相同酶切的5个启动子序列连接,获得5个在不同启动子下调控绿色荧光蛋白基因表达的重组质粒Promoter-gfpm,分别命名为SpXYLp-gfpm、SpMAL6p-gfpm、SpMAL1p-gfpm、SpGAL1p-gfpm、ScGAL1p-gfpm。
1.3.2 醋酸锂转化构建重组菌及转化子验证: 重组质粒Promoter-gfpm经Stu Ⅰ线性化后醋酸锂转化C. amazonensis,醋酸锂转化法参考文献[20]。涂布潮霉素(Hygromycin B) 900平板筛选得到转化子。在通过更高浓度的Hyg抗性平板进行转化子筛选后,提取各转化子基因组进行PCR验证,最终获取阳性转化子。
1.3.3 荧光显微镜检测: 将重组菌C. amazonensis/ Promoter-gfpm接种于YEPD培养基,30 ℃、200 r/min过夜培养18 h获得种子液后,收集菌体,用生理盐水洗涤2次,重悬后分别转接至新的YEPD和其启动子对应的诱导培养基中,其中重组菌C. amazonensis/SpXYLp-gfpm转接至木糖诱导培养基YEPX;C. amazonensis/SpMAL6p-gfpm和C. amazonensis/SpMAL1p-gfpm转接至麦芽糖诱导培养基YEPM;C. amazonensis/SpGAL1p-gfpm和C. amazonensis/ScGAL1p-gfpm转接至半乳糖诱导培养基YEPG。每次转接后的初始OD600保持在0.5左右。于30、200 r/min培养18–24 h后,收集菌体,用生理盐水洗涤2次,重悬后吸取少量菌液置于载玻片上,在荧光显微镜下观察菌体细胞并拍照,激发波长λ=485 nm。
1.3.4 荧光分光光度计定量: 分别于YEPD和诱导培养基(YEPX/YEPM/YEPG)中收集重组菌C. amazonensis/Promoter-gfpm,用生理盐水洗涤2次,重悬后稀释适当倍数至菌液OD600保持在0.5左右。利用日立荧光分光光度计F-7000在480 nm激发光下激发,测定各重组菌液在510 nm处的发射光强度。
1.4 FLP重组酶基因定点突变 以S. cerevisiae为模板,用引物FLP-F和FLP-R PCR扩增得到FLP基因序列,连接pMD19-T simple载体,转化感受态E. coli JM109,获得重组子Ts-FLP。以Ts-FLP为模板,分别使用引物FLP-1和FLP-2、FLP-3和FLP-4、FLP-5和FLP-6经过3轮PCR定点突变,定点突变参照Quickchange Lightning Multi Site-Directed Mutagenesis Kit说明书进行。(1) PCR反应体系(25 μL):5 μL 5×PrimeSTAR Buffer,2 μL dNTPs Mixture (2.5 mmol/L each),0.3 μL各引物(25 μmol/L),0.5 μL模板(10 pg–1 ng),0.3 μL PrimeSTAR Polymerase (2.5 U/μL),加双蒸水至25 μL;(2) PCR扩增条件:95;95,55,65,65,30个循环;(3)将PCR产物冰浴2 min,加入内切酶Dpn Ⅰ于37消化30 min;(4)将消化后的PCR产物于65反应10 min以失活内切酶Dpn Ⅰ,之后加入E. coli JM109感受态中进行转化;(5)随机挑取若干转化子提取质粒测序,比对测序结果,挑选阳性突变子。
1.5 caPDC基因的克隆 C. amazonensis为新近发现的木糖利用酵母,目前还没有经过全基因组测序。通过比对S. passalidarum和其他多个酵母的PDC基因序列,发现多个PDC基因的ATG密码子后的一小段序列以及距离终止密码子几十bp处的一段序列普遍具有较高的保守性,从而设计上下游引物PDC-F和PDC-R,以C. amazonensis基因组为模板,PCR反应获得一段1.7 kb左右的PDC基因产物(caPDC),插入pMD19-T simple载体中,获得重组质粒Ts-caPDC后,转化E. coli JM109,挑取阳性转化子,送生工生物工程(上海)股份有限公司进行测序,由测序结果获得caPDC的基因序列。
1.6 PDC基因敲除突变盒的构建 以Ts-caPDC质粒为模板,用引物PDC-1/PDC-2和PDC-3/PDC-4分别PCR扩增caPDC基因5′端360 bp的上游序列caPDC-L和3′端390 bp的下游序列caPDC-R,用重叠延伸PCR法将上下游序列拼接获得PDC基因同源重组片段PDCL-R。PDCL-R连接pMD19-T simple载体,转化感受态E. coli JM109,用含氨苄青霉素的LB固体培养基筛选阳性克隆,命名为Ts-PDCLR。提取质粒Ts-PDCLR,用Kpn Ⅰ酶切,去磷酸化后备用。将质粒SpGAL1p-gfpm和Ts-caFLP分别用Sal Ⅰ和Not Ⅰ进行双酶切,酶切产物经T4 DNA连接酶过夜连接,转化感受态E. coli JM109,获得的阳性克隆重组子命名为SpGAL1p-caFLP。以该重组质粒为模板,用引物FRT-1/FRT-2扩增带有FRT位点的SpGAL1p-caFLP-hphm片段,用Kpn Ⅰ酶切并纯化后,与前面备用的Ts-PDCLR经T4 DNA连接酶连接,转化感受态E. coli JM109,用含氨苄青霉素的LB固体培养基筛选、鉴定并保藏,命名为Ts-PDCL-FRT-SpGAL1p-caFLP-hphm-FRT-PDCR (简称为Ts-PRFgHRP),该同源重组载体通过Stu Ⅰ酶切后割胶回收同源重组片段PDCL-FRT-SpGAL1p-caFLP-hphm-FRT-PDCR (PRFgHRP)备用。
1.7 PDC基因敲除与潮霉素抗性标记基因(hphm)的重复利用 将敲除盒PRFgHRP醋酸锂转化C. amazonensis,涂布Hyg 900抗性平板,筛选得到阳性转化子后于YEPG固体培养基中分离纯化,30 ℃培养2–3 d后,将长出的单菌落分别划线于普通的YEPD平板和Hyg 900抗性平板中培养,挑取YEPD平板上生长而抗性板上不生长的单菌落,提取其染色体,用引物PDC-1/PDC-4和FLP-F/FLP-R进行PCR验证,验证正确后经测序获得进一步确认。
1.8 PDC比酶活测定 C. amazonensis细胞破碎及PDC酶活的测定参考文献[21],蛋白浓度用考马斯亮蓝染色法测定。
2 结果和分析 2.1 诱导型启动子的筛选
2.1.1 启动子研究载体构建: 分别克隆S. passalidarum来源的木糖还原酶启动子SpXYLp、α-麦芽糖苷酶启动子SpMAL6p和SpMAL1p、β-半乳糖苷酶启动子SpGAL1p,以及S. cerevisiae来源的β-半乳糖苷酶启动子ScGAL1p,分别替换掉原有质粒PRACTH-gfpm中的乙醇脱氢酶基因启动子SpADH1p,构建得到5个不同启动子诱导的表达质粒Promoter-gfpm,分别简称为SpXYLp-gfpm、SpMAL6p-gfpm、SpMAL1p-gfpm、SpGAL1p-gfpm、ScGAL1p-gfpm (图 1)。其中gfpm为报告基因,hphm为Hygromycin B抗性标记基因。
 |
| 图 1 不同诱导型质粒Promoter-gfpm示意图 Figure 1 The plasmids of Promoter-gfpm with different inducible promoters. |
| 图选项 |
2.1.2 备选启动子的诱导调控表达性能: 分别于YEPD和诱导培养基(YEPX/YEPM/YEPG)中收集重组菌C. amazonensis/Promoter-gfpm,用生理盐水洗涤2次后,取样用荧光显微镜观察,荧光检测结果显示(图 2),重组菌C. amazonensis/SpXYLp-gfpm和C. amazonensis/SpMAL6p-gfpm在YEPD和诱导培养基(分别是YEPX和YEPM)中均实现了绿色荧光蛋白的有效表达,且诱导培养基均较YEPD的荧光强度大,说明S. passalidarum来源的木糖还原酶基因启动子SpXYL1p和α-麦芽糖苷酶启动子SpMAL6p在C. amazonensis中均是有效的,但SpXYL1p和SpMAL6p均非严格诱导型启动子,在以葡萄糖为底物的培养环境中仍有一定量的基础表达;重组菌C. amazonensis/ScGAL1p-gfpm无论在YEPD培养基还是YEPG诱导培养基中均未观察到绿色荧光蛋白的产生,可见S. cerevisiae来源的β-半乳糖苷酶启动子ScGAL1p在C. amazonensis中并不能有效行使其功能;重组菌C. amazonensis/SpMAL1p-gfpm和C. amazonensis/ SpGAL1p-gfpm在YEPD中未观察到绿色荧光蛋白,但分别在YEPM和YEPG中明显实现了绿色荧光蛋白GFP的表达,启动子受底物诱导显著,是严格的诱导型启动子,其中重组菌C. amazonensis/SpMAL1p-gfpm在YEPM下荧光强度较弱,不及重组菌C. amazonensis/SpGAL1p-gfpm在YEPG下的荧光强度。因此,相较于SpMAL1p,严格诱导型启动子SpGAL1p更适合用于调控C. amazonensis中异源蛋白的表达。
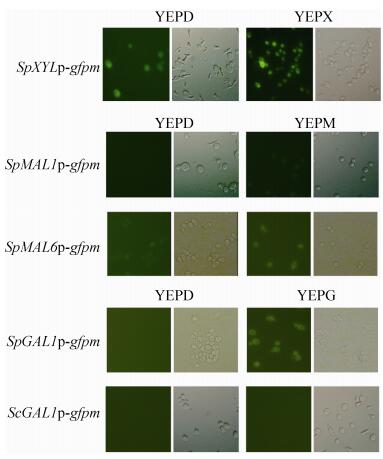 |
| 图 2 荧光显微镜检测非诱导和诱导培养基中重组菌内绿色荧光蛋白 Figure 2 Observation of GFP in recombinant strains by fluorescence microscopy under the conditions with or without induction. YEPD: non-inducing medium; YEPX: xylose-containing medium for C. amazonensis/ SpXYLp-gfpm; YEPM: maltose-containing medium for C. amazonensis/SpMAL1p-gfpm and C. amazonensis/ SpMAL6p-gfpm; YEPG: galactose-containing medium for C. amazonensis/SpGAL1p-gfpm and C. amazonensis/ ScGAL1p-gfpm. |
| 图选项 |
在荧光定量实验中,分析比较各重组菌在不同培养条件下的GFP荧光强度。如图 3所示,尽管诱导条件下重组菌C. amazonensis/SpXYL1p-gfpm和C. amazonensis/SpMAL6p-gfpm的GFP荧光强度均远远高于其在YEPD的荧光强度,但这2株重组菌在YEPD中仍然有较高的GFP荧光强度,说明二者在未诱导条件下仍然具有一定量的基础表达,并非严格调控型启动子。重组菌C. amazonensis/ ScGAL1p-gfpm无论在YEPD还是诱导培养基中均没有检测到高于野生型的荧光强度,进一步确认了ScGAL1p启动子在C. amazonensis中的不适用性;重组菌C. amazonensis/SpMAL1p-gfpm和C. amazonensis/SpGAL1p-gfpm在YEPD中几乎检测不到GFP荧光强度,与野生菌C. amazonensis一致,而在诱导条件下二者的GFP荧光强度明显,尤其是C. amazonensis/SpGAL1p-gfpm,说明SpMAL1p和SpGAL1p启动子受底物诱导显著,且在葡萄糖条件下几乎无启动活性,是严格的诱导型启动子,这与前面荧光显微镜的观察结果完全一致。后续基因敲除系统的构建以选用半乳糖诱导型启动子SpGAL1p为例。
 |
| 图 3 重组菌内GFP荧光强度定量测定 Figure 3 Quantification of GFP fluorescence intensity in recombinant strains. |
| 图选项 |
2.2 FLP重组酶基因定点突变 FLP重组酶基因全长1272 bp,分别在第202、265、631 bp处含有CTG (Ser)密码子,将其突变为TTG (Leu)。根据测序结果挑选正确的转化子,突变后的重组酶命名为caFLP,其CDS序列如图 4所示。
 |
| 图 4 定点突变后的FLP基因CDS序列 Figure 4 The CDS sequence of FLP gene after site-specific mutagenesis. TTG: the mutated codon. |
| 图选项 |
2.3 caPDC基因的克隆 以C. amazonensis基因组为模板,设计引物扩增获得一段1.7 kb的PCR产物(图 5)。对其测序,将该序列置于NCBI数据库中进行比对,结果发现caPDC与S. passalidarum和Candida dubliniensis等酵母的丙酮酸脱羧酶基因序列的一致性达到80%,进一步证明该克隆产物即为C. amazonensis的丙酮酸脱羧酶基因。
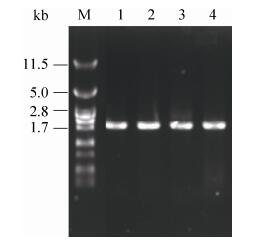 |
| 图 5 caPDC基因的PCR产物 Figure 5 PCR products of the PDC gene in C. amazonensis. M: λ DNA/Pst Ⅰ marker; lane 1–4: The PCR products of the PDC gene in C. amazonensis. |
| 图选项 |
2.4 PDC基因敲除突变盒的构建 构建同源重组质粒Ts-PRFgHRP (图 6),经Kpn Ⅰ单酶切后,凝胶电泳显示存在3.4 kb和4.6 kb 2条DNA带,分别与PDCL-Ts-PDCR片段和FRT-SpGAL1p-FLP-hphm-FRT片段大小一致(图 7)对重组质粒Ts-PRFgHRP进一步进行Kpn Ⅰ和BamH I双酶切后,得到3.4 kb、2.8 kb和1.8 kb的3条DNA带,与预期的PDCL-Ts-PDCR、FRT-SpGAL1p-caFLP和hphm-FRT片段大小一致(图 7)。结果表明基因敲除盒PRFgHRP构建成功。
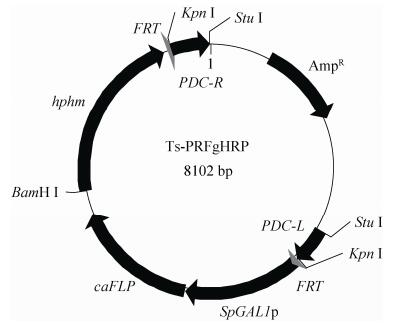 |
| 图 6 重组质粒Ts-PRFgHRP示意图 Figure 6 The recombinant plasmid of Ts-PRFgHRP. |
| 图选项 |
 |
| 图 7 重组质粒Ts-PRFgHRP的酶切验证 Figure 7 The enzymatic digestion of Ts-PRFgHRP. M: DL 15000 DNA marker; lane 1: recombinant plasmid Ts-PRFgHRP digested by Kpn Ⅰ; lane 2: recombinant plasmid Ts-PRFgHRP digested by Kpn Ⅰ and BamH I; lane 3: recombinant plasmid Ts-PRFgHRP digested by Stu Ⅰ. |
| 图选项 |
2.5 PDC基因的敲除与hphm抗性标记基因的回收 将基因敲除盒PRFgHRP (DNA结构示意图如图 8-A所示)转化入野生型酵母C. amazonensis中,涂布Hyg 900抗性平板。将长出的单菌落转接培养,提取其染色体DNA作为模板进行PCR验证,结果如图 9所示。用引物PDC-1/PDC-4扩增,获得5.4 kb的DNA条带,测序发现这个条带与PRFgHRP片段序列完全一致。而原始菌株C. amazonensis的PCR结果为1.7 kb的PDC基因条带,此外进一步用引物FLP-F/FLP-R进行PCR验证,获得1.3 kb左右的FLP基因条带。因此,可以确定基因敲除盒PRFgHRP已正确整合到染色体的PDC基因位点,其整合位点基因结构如图 8-B所示,该转化子命名为C. amazonensis PDC01。为将PDC突变株内的hphm抗性标记基因删除,将培养后的C. amazonensis PDC01划线于含半乳糖的YEPG平板中进行分离纯化,培养数代后,将长出的单菌落分别接种于普通的YEPD平板和含Hyg 900的抗性平板中培养,抗性回收株因hphm标记基因被切除而无法在抗性平板上生长(图 10),因此,挑取YEPD平板上生长而抗性板上不生长的单菌落,提取其染色体,用引物FLP-F/FLP-R进行PCR扩增,结果发现没有条带出现(图 9);用引物PDC-1/PDC-4进行PCR扩增获得一段0.8 kb左右的条带(图 9),经DNA测序分析表明其为PDCL-FRT-PDCR片段序列,说明插入PDC基因位点的caFLP重组酶和抗性标记基因hphm已成功得到切除。对该抗性回收突变株命名为C. amazonensis PDC02。
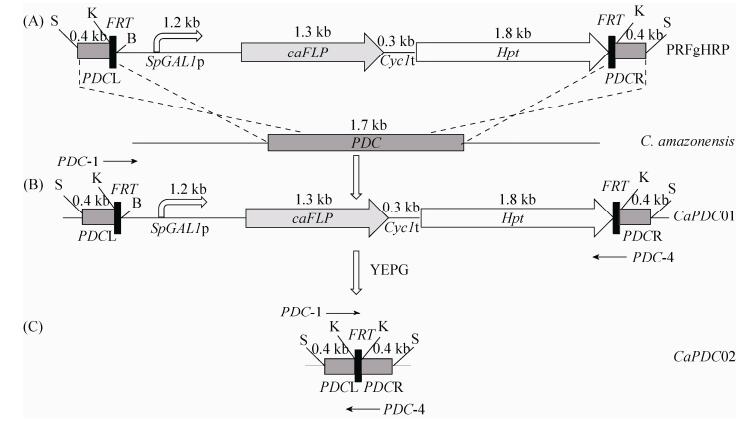 |
| 图 8 PDC基因敲除流程图 Figure 8 Disruption of the PDC gene in C. amazonensis. CaPDC01: C. amazonensis PDC01; CaPDC02: C. amazonensis PDC02; S: restriction site of Stu Ⅰ; K: restriction site of Kpn Ⅰ; B: restriction site of BamH I. |
| 图选项 |
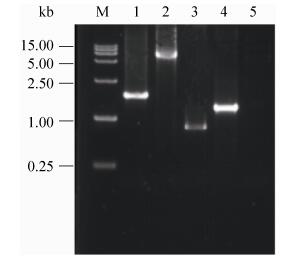 |
| 图 9 PDC突变株C. amazonensis PDC01和C. amazonens zis PDC02的PCR验证 Figure 9 Identification of the PDC mutants by PCR. M: DL 15000 DNA marker; lane 1: PCR product using primers PDC-1/PDC-4 of wild type; lane 2: PCR product using primers PDC-1/PDC-4 of C. amazonensis PDC01; lane 3: PCR product using primers PDC-1/PDC-4 of C. amazonensis PDC02; lane 4: PCR product using primers FLP-F/FLP-R of C. amazonensis PDC01; lane 5: PCR product using primers FLP-F/FLP-R of C. amazonensis PDC02. |
| 图选项 |
 |
| 图 10 原始菌株C. amazonensis CBS 12363及PDC敲除株在Hyg 900平板上的生长状况 Figure 10 Growth of the parental strain (wild-type) and PDC mutants onYEPD plates with 900 μg/mL Hygromycin B. W: wild type; 01: C. amazonensis PDC01; 02: C. amazonensis PDC02. |
| 图选项 |
2.6 PDC基因缺失的功能鉴定 为了考察PDC基因的缺失对突变株中酶活力的影响,对原始野生株C. amazonensis和PDC突变株C. amazonensis PDC02分别进行了PDC酶活的测定,结果如图 11所示。突变株较出发菌株C. amazonensis的PDC比酶活下降了41.73%,但仍然保留有一定的PDC酶活。因此,我们推测突变株中可能存在编码PDC的同工酶基因,能够在一定程度上功能互补缺失基因。
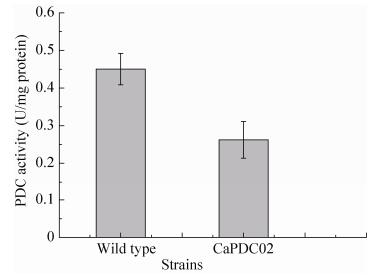 |
| 图 11 野生菌株及PDC敲除株胞内PDC比酶活分析 Figure 11 The specific enzyme activity of PDC in wild strain and PDC mutant. CaPDC02: C. amazonensis PDC02. |
| 图选项 |
3 讨论 C. amazonensis作为一株新近发现的可高效利用木糖的酵母菌株,在代谢木糖和生产木糖醇方面表现出一定优势,有望成为新型木糖发酵模式菌株。但该酵母由于缺乏可对其进行遗传操作的分子手段而限制了代谢工程育种技术对其的应用,因此,建立一个筛选标记可重复利用的基因敲除系统对代谢工程改造C. amazonensis具有重要意义。本文通过对适用于C. amazonensis诱导型启动子的筛选,获得2个严格受底物诱导的调控型启动子SpGAL1p (受半乳糖诱导)和SpMAL1p (受麦芽糖诱导)。选择启动子SpGAL1p对FLP重组酶进行调控表达,并在FLP表达盒和抗性标记基因(hphm)两端添加同向重复的FRT位点,由此构建的敲除盒待确认整合到C. amazonensis的染色体上之后,对阳性转化子添加半乳糖诱导培养,实现FLP重组酶的表达,并作用于FRT位点,使2个同向FRT位点之间的片段得到切除,最后只留下1个FRT位点。利用上述构建的FLP/FRT基因敲除系统,选择对C. amazonensis内的丙酮酸脱羧酶基因(PDC)进行敲除,丙酮酸脱羧酶可将丙酮酸催化生成乙醛,并在乙醛脱氢酶的作用下进一步代谢为乙醇,是酵母内碳代谢途径上的关键酶。因此,作为对C. amazonensis FLP/FRT基因敲除系统可行性的初步验证,本研究对C. amazonensis的PDC基因进行了敲除,并回收了抗性标记,成功实现了FLP/FRT系统在C. amazonensis中的首次应用。
本研究构建的FLP/FRT基因敲除系统在以下方面具有一定的优势:(1)只需要1次同源重组和后续的培养基诱导便可获得重组酶基因和抗性基因一同被切除的靶基因敲除株,而不需要另外转入1个携带重组酶的游离质粒,如Cre-Loxp系统中携带Cre酶的PSH47质粒[22]。(2)只需要1个可用的抗性标记便可完成多个基因的连续敲除。(3)基因敲除株与其出发菌株相比,除了目的基因的部分或完全缺失及多了1个34 bp的FRT位点外,无其他基因上的差异。(4)该敲除系统可以直接以野生型菌株为改造对象,不需要利用URA3或HIS1、ARG4等营养缺陷型菌株[23],一方面省去了营养缺陷型菌株的筛选过程,也避免了回复突变等问题;另一方面由于URA3等基因的缺失会改变菌株的表型,这可能会导致其对突变株有其他未知的影响。
另一方面,该敲除系统也存在一定的不足之处,如由于构建的敲除盒中除了同源臂序列和抗性标记基因外,还添加有FLP重组酶基因的表达盒,这就使得敲除盒片段过大,不仅增加了PCR扩增敲除盒的难度,同时还可能造成其同源重组效率的降低。此外,该敲除系统还存在一个Cre-Loxp和FLP/FRT系统普遍存在的问题,即每一次利用该系统敲除基因回收掉抗性后,都会在基因组上留下1个FRT识别位点的疤痕,因此如果对基因组进行多轮操作,留下的多个识别位点之间可能会发生重组,降低基因组的稳定性。为了避免这种情况,后续可参考Yan等使用突变型的FRT位点,从而提高遗传稳定性[24]。
总的来说,利用本研究建立的FLP/FRT基因敲除系统能够高效地连续敲除C. amazonensis内目的基因,为菌株的代谢工程改造提供了强有力的技术手段,同时也为其他酵母的遗传转化系统研究提供了借鉴意义。
References
| [1] | Cadete RM, Melo MA, Lopes MR, Pereira GMD, Zilli JE, Vital MJS, Gomes FCO, Lachance MA, Rosa CA. Candida amazonensis sp. nov., an ascomycetous yeast isolated from rotting wood in the Amazonian forest. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(6): 1438-1440. |
| [2] | Cadete RM, Melo MA, Dussán KJ, Rodrigues RC, Silva SS, Zilli JE, Vital MJ, Gomes FC, Lachance MA, Rosa CA. Diversity and physiological characterization of D-xylose-fermenting yeasts isolated from the Brazilian Amazonian forest. PLoS ONE, 2012, 7(8): e43135. DOI:10.1371/journal.pone.0043135 |
| [3] | Fan HC, Zhang L, Li Y, Li YR, Gu ZH, Ding ZY, Shi GY. Physiological and metabolic characteristics of five xylose utilizing yeasts. Acta Microbiologica Sinica, 2015, 55(8): 1026-1035. (in Chinese) 范贺超, 张梁, 李赢, 李由然, 顾正华, 丁重阳, 石贵阳. 五株木糖利用酵母的生理代谢特性. 微生物学报, 2015, 55(8): 1026-1035. |
| [4] | Wohlbach DJ, Kuo A, Sato TK, Potts KM, Salamov AA, LaButti KM, Sun H, Clum A, Pangilinan JL, Lindquist EA, Lucas S, Lapidus A, Jin MJ, Gunawan C, Balan V, Dale BE, Jeffries TW, Zinkel R, Barry KW, Grigoriev IV, Gasch AP. Comparative genomics of xylose-fermenting fungi for enhanced biofuel production. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(32): 13212-13217. DOI:10.1073/pnas.1103039108 |
| [5] | Fan HC. Evaluation of physiological and metabolic characteristics of new xylose-utilizing yeasts and the construction of genetic expression system. Master Dissertation of Jiangnan University, 2015. (in Chinese) 范贺超. 新型木糖利用酵母的评价及其遗传表达系统构建. 江南大学硕士学位论文, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10295-1015430143.htm |
| [6] | Lloyd AM, Davis RW. Functional expression of the yeast FLP/FRT site-specific recombination system in Nicotiana tabacum. Molecular and General Genetics MGG, 1994, 242(6): 653-657. |
| [7] | Staib P, Kretschmar M, Nichterlein T, K?hler G, Michel S, Hof H, Hacker J, Morschh?user J. Host-induced, stage-specific virulence gene activation in Candida albicans during infection. Molecular Microbiology, 1999, 32(3): 533-546. DOI:10.1046/j.1365-2958.1999.01367.x |
| [8] | Morschh?user J, Michel S, Staib P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiology, 1999, 32(3): 547-556. DOI:10.1046/j.1365-2958.1999.01393.x |
| [9] | Sánchez-Martínez C, Pérez-Martín J. Site-specific targeting of exogenous DNA into the genome of Candida albicans using the FLP recombinase. Molecular Genetics and Genomics, 2002, 268(3): 418-424. DOI:10.1007/s00438-002-0763-5 |
| [10] | Reu? O, Vik ?, Kolter R, Morschh?user J. The SAT1 flipper, an optimized tool for gene disruption in Candida albicans. Gene, 2004, 341: 119-127. DOI:10.1016/j.gene.2004.06.021 |
| [11] | Ding C, Butler G. Development of a gene knockout system in Candida parapsilosis reveals a conserved role for BCR1 in biofilm formation. Eukaryotic Cell, 2007, 6(8): 1310-1319. DOI:10.1128/EC.00136-07 |
| [12] | Gácser A, Trofa D, Sch?fer W, Nosanchuk JD. Targeted gene deletion in Candida parapsilosis demonstrates the role of secreted lipase in virulence. The Journal of Clinical Investigation, 2007, 117(10): 3049-3058. DOI:10.1172/JCI32294 |
| [13] | Nguyen LN, Trofa D, Nosanchuk JD. Fatty acid synthase impacts the pathobiology of Candida parapsilosis in vitro and during mammalian infection. PLoS One, 2009, 4(12): e8421. DOI:10.1371/journal.pone.0008421 |
| [14] | Guarente L, Yocum RR, Gifford P. A GAL10-CYC1 hybrid yeast promoter identifies the GAL4 regulatory region as an upstream site. Proceedings of the National Academy of Sciences of the United States of America, 1982, 79(23): 7410-7414. DOI:10.1073/pnas.79.23.7410 |
| [15] | Karpova TS, Kim MJ, Spriet C, Nalley K, Stasevich TJ, Kherrouche Z, Heliot L, McNally JG. Concurrent fast and slow cycling of a transcriptional activator at an endogenous promoter. Science, 2008, 319(5862): 466-469. DOI:10.1126/science.1150559 |
| [16] | Finley RL Jr, Zhang HM, Zhong JH, Stanyon CA. Regulated expression of proteins in yeast using the MAL61-62 promoter and a mating scheme to increase dynamic range. Gene, 2002, 285(1/2): 49-57. |
| [17] | Kopke K, Hoff B, Kück U. Application of the Saccharomyces cerevisiae FLP/FRT recombination system in filamentous fungi for marker recycling and construction of knockout strains devoid of heterologous genes. Applied and Environmental Microbiology, 2010, 76(14): 4664-4674. DOI:10.1128/AEM.00670-10 |
| [18] | Mumberg D, Müller R, Funk M. Regulatable promoters of Saccharomyces cerevisiae:comparison of transcriptional activity and their use for heterologous expression. Nucleic Acids Research, 1994, 22(25): 5767-5768. DOI:10.1093/nar/22.25.5767 |
| [19] | Rogers DT, Lemire JM, Bostian KA. Acid phosphatase polypeptides in Saccharomyces cerevisiae are encoded by a differentially regulated multigene family. Proceedings of the National Academy of Sciences of the United States of America, 1982, 79(7): 2157-2161. DOI:10.1073/pnas.79.7.2157 |
| [20] | Ko BS, Kim J, Kim JH. Production of xylitol from D-xylose by a xylitol dehydrogenase gene-disrupted mutant of Candida tropicalis. Applied and Environmental Microbiology, 2006, 72(6): 4207-4213. DOI:10.1128/AEM.02699-05 |
| [21] | Gao NF, Deng XH, Wang DP, Li L. Determination of pyruvate decarboxylase activity in Saccharomyces cerevisia. China Brewing, 2011, 30(3): 128-130. (in Chinese) 高年发, 邓旭衡, 王德培, 李磊. 酿酒酵母丙酮酸脱羧酶活性测定的研究. 中国酿造, 2011, 30(3): 128-130. |
| [22] | Laplaza JM, Torres BR, Jin YS, Jeffries TW. Sh ble and Cre adapted for functional genomics and metabolic engineering of Pichia stipitis. Enzyme and Microbial Technology, 2006, 38(6): 741-747. DOI:10.1016/j.enzmictec.2005.07.024 |
| [23] | Noble SM, Johnson AD. Strains and strategies for large-scale gene deletion studies of the diploid human fungal pathogen Candida albicans. Eukaryotic Cell, 2005, 4(2): 298-309. DOI:10.1128/EC.4.2.298-309.2005 |
| [24] | Yan X, Yu HJ, Hong Q, Li SP. Cre/lox system and PCR-based genome engineering in Bacillus subtilis. Applied and Environmental Microbiology, 2008, 74(17): 5556-5562. DOI:10.1128/AEM.01156-08 |
