侯路宽1, 李花月1,2, 李文利1,2


1.教育部海洋药物重点实验室, 中国海洋大学医药学院, 山东 青岛 266003;
2.青岛海洋科学与技术国家实验室, 海洋生物学与生物技术功能实验室, 山东 青岛 266237
收稿日期:2017-05-19;修回日期:2017-06-12;网络出版日期:2017-07-21
基金项目:国家自然科学基金(31570032);国家自然科学基金-山东省政府联合基金(U1606403)
作者简介:李文利,中国海洋大学教授,博导,教育部“新世纪优秀人才”,海洋湖沼学会药学分会理事,中国研究型医院学会空间微生物与感染委员会委员,中国微生物学会和美国微生物学会会员。2003年毕业于中国科学院微生物所,获博士学位。2004年2月加入美国威斯康星大学麦迪逊分校药学院从事博士后研究,主要开展了聚酮类抗生素生物合成研究。2009年10月受聘于中国海洋大学海洋药物教育部重点实验室。目前主要从事海洋微生物隐性基因簇激活和海洋天然产物生物合成研究。近年来在国际主流学术刊物如EnvironmentalMicrobiology、Journal of Biological Chemistry、OrganicLetters、Journal of the American Chemical Society、ChemBioChem、Microbial Cell Factories等发表SCI论文30多篇,参编专著3部。先后主持/承担了国家自然科学基金、科技部“863”、国家基金委-山东省政府联合基金等国家级课题十余项
*通信作者:李文利, Tel/Fax:+86-532-82081813, E-mail:liwenli@ouc.edu.cn
摘要:传统的"活性-化合物"天然药物发现方法导致大量已知化合物被重复分离,大大加剧了新药发现的难度。规模化基因组测序揭示了微生物基因组中存在大量的隐性(cryptic)次级代谢产物生物合成基因簇,如何激活这些隐性基因簇成为当今世界天然产物研究领域的难点与热点。本文从途径特异性和多效性两个角度综述了隐性生物合成基因簇激活策略;同时,对基因组信息指导下结构导向(structure-guided)的化合物定向分离技术进行了归纳。隐性基因簇的激活为定向发掘具有优良活性的新型天然产物提供了新的契机。
关键词: 激活 隐性生物合成基因簇 天然产物 定向发现
Activation of cryptic microbial biosynthetic gene clusters and structure-guided natural product discovery
Lukuan Hou1, Huayue Li1,2, Wenli Li1,2


1.Key Laboratory of Marine Drugs, Ministry of Education, School of Medicine and Pharmacy, Ocean University of China, Qingdao 266003, Shandong Province, China;
2.Laboratory for Marine Drugs and Bioproducts, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, Shandong Province, China
Received 19 May 2017; Revised 12 June 2017; Published online 21 July 2017
*Corresponding author: Wenli Li, Tel/Fax:+86-532-82081813, E-mail:liwenli@ouc.edu.cn
Supported by the National Natural Science Foundation of China (31570032) and by the NSFC-Shandong Joint Fund for Marine Science Research Centers (U1606403)
Abstract: Traditional bioassay-guided natural product discovery has led to re-isolation of a large quantity of known compounds, and thus dramatically stunted the process of drug development. Large-scale genome sequencing revealed the existence of numerous cryptic biosynthetic gene clusters in microbes, which have attracted increasing attention in the research field of natural product discovery. In this review, we summarized the strategies for activation of microbial cryptic gene clusters at pathway-specific and pleiotropic levels, and discussed trends in structural identification technologies used for genome-guided compound mining. Activation of cryptic gene clusters will open new era for effective discovery of novel bioactive natural products.
Key words: activation cryptic biosynthetic gene cluster natural products structure-guided discovery
微生物来源的天然产物一直是新药开发的重要来源,包括临床上许多抗细菌、抗真菌、抗寄生虫、抗肿瘤以及免疫抑制剂药物。然而,从20世纪90年代末期开始,发现结构新颖的活性化合物的进程逐渐缓慢,传统的“活性-化合物”天然药物筛选方法导致大量已知化合物被重复分离[1]。因此,如何有效地靶向分离新型天然产物已成为微生物天然产物领域的研究热点。
随着高通量测序技术的飞速发展,越来越多的微生物全基因组被测序。根据在线基因数据库GOLD (Genomes OnLine Database),截止2017年4月,已有246189种细菌、15521种真核生物和2216种古细菌的基因组测序工作完成或正在进行中(http://www.genomesonline.org/)。生物信息学分析揭示,许多微生物基因组中含有多个次级代谢产物生物合成基因簇(如许多放线菌的基因组中有8%–10%的序列与次级代谢相关),其数目远远大于从这些微生物中分离得到的化合物种类,目前发现的次级代谢产物仅仅是“冰山的一角”。例如,模式放线菌天蓝色链霉菌Streptomyces coelicolor基因组中含有29个次级代谢产物生物合成基因簇[2],但早期只有4种被分离鉴定,包括undecylprodigiosin、calcium-dependent antibiotic、actinorhodin和methylenomycin。由此可见微生物合成天然产物的能力远远超出人们的预期。然而,许多基因簇在实验室条件下表达量极低或不表达,处于隐性(cryptic)状态,激活这些基因簇的表达将为发掘新颖结构天然产物提供新的机遇。
科学家们通过培养条件优化(如营养胁迫、稀有金属、仿生培养等)和/或核糖体工程来影响次级代谢生物合成基因的表达,进而从中分离活性次级代谢产物,取得了明显的成效。然而,这些策略随机性强,被激活的基因簇很可能是已知的[3-4]。随着天然产物生物合成机制研究的不断深入,特别是对模块型的聚酮合酶(polyketide synthetase,PKS)、非核糖体肽合成酶(nonribosomal peptides,NRPS)及杂合NRPS-PKS有序组装机制的认知,结合飞速发展的生物信息学技术,往往能够预测它们可能的产物或结构特征,从而实现生物合成基因簇指导下新型天然产物的定向发现。近年来,科学家们通过启动子工程、调控基因遗传改造、异源表达/合成生物学等基因组挖掘策略有效激活了一系列隐性基因簇。本文从途径特异性和多效性两个角度对隐性基因簇的激活研究进展进行了综述,同时归纳了基因组信息指导下结构导向(structure-guided)的化合物定向分离技术,并展望了隐性基因簇激活在微生物天然产物发现中的应用前景。
1 隐性次级代谢产物生物合成基因簇的激活 隐性基因簇之所以“沉默”,一般是由于受到直接性或间接性阻遏造成的。去除或绕过阻遏因素是激活隐性基因簇的关键。
1.1 途径特异性激活 途径特异性激活指靶向激活特定基因簇[5],主要包括启动子工程、途径特异性调控基因遗传改造、异源表达和合成生物学、报告基因指导下的突变株筛选和体外重构等策略(图 1和表 1)。
 |
| 图 1 采用途径特异性激活策略分离到的天然产物 Figure 1 Compounds isolated from microbes by pathway-specific activation of cryptic gene clusters. |
| 图选项 |
表 1. 通过激活隐性生物合成基因簇分离到的天然产物 Table 1. Natural products isolated by activation of cryptic gene clusters
| Approach | Compounds | References | |
| Pathway-specific methods | Promoter engineering | burkholderic acid (1), 2-hydroxy-5-isobutyl-3-propanamidylpyrazine (2) | [6-7] |
| Manipulating pathway-specific regulators | stambomycins A-D (3–6), coelimycin P1 (7) | [8-10] | |
| Heterologous expression | AHFCA (8–12), taromycin A (13), penocin A (14), spectinabilin (15) | [11-14] | |
| Reporter-guided mutant selection | gaudimycins D-E (16–17) | [15] | |
| In vitro reconstitution | 2-methylisoborneol (18), epi-isozizaene (19), albaflavenone (20), avermitilol (21) | [16-18] | |
| Pleiotropic methods | Manipulating global regulators | violapyrone B (22), (23–28) | [19-23] |
| Epigenetic perturbation | nygerone A (29), oxylipins (30–32), mondictyphenone (33), emodins (34–38), F9775A (39), F9775B (40) | [24-26] | |
| Inactivation of competing pathways | christolane A (41), christolane B (42), christolane C (43), piericidin F (44) | [27-28] | |
| Co-culture | alchivemycin A (45) | [29-30] |
表选项
1.1.1 启动子工程:在自然环境中,微生物的次级代谢产物生物合成基因往往在一定条件下表达,大部分基因簇在实验室条件下处于“沉默”状态,将天然启动子更换为组成型或者诱导型的启动子,进行人为可控表达,往往可以激活隐性基因簇的表达,从而产生相应的次级代谢产物。Franke等将一个组成型启动子插入到Burkholderia pseudomallei基因组中编码一个杂合的PKS-NRPS生物合成途径的操纵子上游,从而激活了其表达并分离得到了化合物burkholderic acid (1)[6]。Aspergillus nidulans中包含一个负责编码fellutamide B的生物合成基因簇inp,Yeh等将该基因簇在天然启动子的控制下进行异源表达没有成功;随后,将基因簇中inpA-D基因的启动子均更换为诱导型启动子alcAp后,在环戊酮的诱导下激活了目标基因簇的表达进而产生了新化合物2-hydroxy-5-isobutyl-3-propanamidylpyrazine (2)[7]。
1.1.2 途径特异性调控基因遗传改造:途径特异性调控基因一般位于基因簇内,包括正调控基因和负调控基因,二者分别对生物合成起激活和阻遏作用。Laureti等通过高表达途径特异性LuxR家族调节蛋白,激活了处于沉默状态的Ⅰ型PKS基因簇,从Streptomyces ambofaciens ATCC23877发酵产物中发现了具有抗肿瘤活性的51元大环内酯类抗生素stambomycins A–D (3–6)[8];通过敲除途径特异性负调控基因scbR2,S. coelicolor中隐性基因簇cpk被激活[9],进一步结合核糖体工程策略使之产量提高,分离和鉴定了黄色色素coelimycin P1 (7)[10]。
1.1.3 异源表达和合成生物学手段:很多情况下,基因簇的沉默可能是由于未知的复杂性阻遏因素导致的,在遗传背景清楚的宿主中进行异源表达是解除天然菌株复杂调控网络的有效手段。Corre等将位于S. coelicolor A3 (2)中线性大质粒SCP1上的mmfLHP操纵子在不含SCP1的S. coelicolor M512中进行了异源表达,发现了5个新的抗生素产生信号分子2-烷基-4-羟甲基呋喃-3-羧酸(AHFCA) (8–12)[11];Yamanaka等将来自Saccharomonospora sp. CNQ-490中一个NRPS基因簇导入S. coelicolor M512中,从而激活了其表达,分离得到新颖的肽类抗生素taromycin A (13)[12];Pediococcus pentosaceus ATCC 25745基因组中包含多个负责合成细菌素的基因簇,但是缺少调控基因和转运基因,阻碍了基因簇的正常表达,而Lactobacillus sakei中包含这些元件,Diep等将负责合成细菌素的基因penA-peiA导入L. sakei中从而产生了新的细菌素penocin A (14)[13]。Zhao课题组根据合成生物学理念设计了即插即用(plug-and-play)的模型,他们以Streptomyces orinoci中编码spectinabilin的隐性基因簇nor为研究对象,将诱导型启动子nitAp组装至途径中的第1个关键基因norG之前,将看家基因的强启动子组装至其他nor基因之前,构建成一个新的基因簇,然后导入S. lividans中进行异源表达,从而绕过原始的复杂调控机制,可控地确保每个基因表达,成功产生了spectinabilin (15)[14]。
1.1.4 报告基因指导下的突变株筛选策略:在原始产生菌中,随机性突变是解除未知阻遏因素的有效手段,与此相应则需要一个高通量的筛选策略。Guo等建立了一种高效的报告基因指导下的突变株筛选策略(reporter-guided mutant selection,RGMS),成功激活了Streptomyces venezuelae ISP5230中jadomycin基因簇的表达和Streptomyces sp. PGA64中隐性基因簇pga的表达,分离得到了2个新的蒽醌类化合物gaudimycins D-E (16–17)[15]。该策略适用于在天然菌株中通过全基因组水平的随机突变解除复杂性阻遏调控,靶向筛选目标基因簇表达的突变株。
1.1.5 体外重构:对于前体容易获取的途径,可以将天然产物合成有关的酶进行表达,并与预测的底物、辅酶及辅因子进行体外反应,通过体外重构方法直接得到产物。这种方法受制于可以获得的前体底物,目前成功的例子主要是萜类化合物的体外重构。如S. coelicolor中的萜烯合酶SCO7700和SCO5222的编码产物2-methylisoborneol (18)[16]以及epi-isozizaene (19)[16]都是采用体外重构获得的。由于epi-isozizaene基因簇还包含了细胞色素P450 (CYP170A1) 基因sco5223,推测epi-isozizaene可能被进一步氧化,Zhao等体外表达了SCO5223,以epi-isozizaene为底物进行体外催化得到了albaflavenone (20)[17]。另外,Streptomyces avermitilis中的萜烯合酶SAV_76编码产物avermitilol (21)也是通过与其底物FPP (farnesyl diphosphate)及相关辅因子进行体外催化而得到的[18]。
1.2 多效性激活 多效性激活指一个或多个基因簇的非靶向性激活[5],主要包括全局性调控基因遗传改造、表观遗传学、阻断竞争途径和共培养等策略(图 2和表 1)。
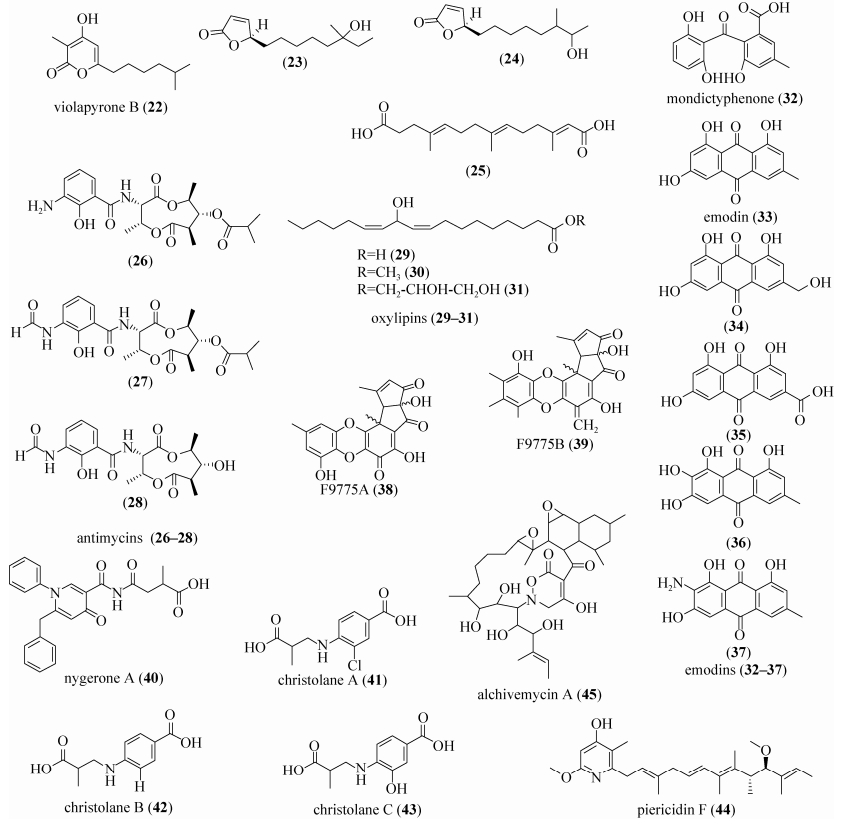 |
| 图 2 采用多效性激活策略分离到的天然产物 Figure 2 Compounds isolated from microbes by pleiotropic activation of cryptic gene clusters. |
| 图选项 |
1.2.1 全局性调控基因遗传改造:全局性调控基因一般位于生物合成基因簇之外,对多个代谢途径具有调控作用,在链霉菌中常常与形态分化相关。研究表明,在链霉菌中广泛存在的wblA(whiB-like)基因阻断不仅能够导致菌株丧失产孢能力,同时可以显著影响次级代谢过程[19-20]。如,本课题组Huang等通过阻断深海来源链霉菌Streptomyces somaliensis SCSIO ZH66中的wblAso基因,使突变株在丧失产孢能力的同时积累了多种次级代谢产物,从而发现了Ⅲ型聚酮类化合物violapyrone B (22)并初步阐明了其生物合成途径[21];进一步,从突变株中分离纯化得到了2个丁烯酸内酯类化合物(23–24)、1个新的二元酸化合物(25)[22]和3个具有抗炎活性的antimycin类型的化合物(26–28)[23]。
1.2.2 表观遗传学:染色体中组蛋白的修饰及DNA的修饰(包括甲基化、生物素化、磷酸化、糖基化和乙酰化等)会对生物合成基因簇的表达产生影响,主要包括化学表观遗传学策略,即添加能够改变组蛋白或DNA修饰的相关化学因子(如组蛋白去乙酰抑制因子trichostatin A、trapoxin B和suberoylanilide hydroxamic acid等,DNA甲基转移酶抑制因子5-azacytidine和5-aza-2′-deoxycytidine等),和分子表观遗传学策略,即直接对组蛋白或DNA修饰的相关基因(如编码甲基化H3组蛋白的基因cclA和编码翻译后修饰组蛋白的基因sumO等)进行分子改造。如Robert等采用化学表观遗传学策略,在培养Cladosporium cladosporioides时添加5-azacytidine,进而得到了化合物oxylipins (29-31)[24]。Henrikson等在Aspergillus niger的发酵过程中添加suberoylanilide hydroxamic acid,成功分离得到了新的天然产物nygerone A (32)[25]。Bok等运用分子表观遗传学策略阻断了A. nidulans中编码甲基化H3组蛋白的基因cclA,从而激活了2个隐性基因簇:其中1个新的基因簇编码mondictyphenone (33)、emodin (34)和emodin的类似物(35-38);另外1个基因簇编码了2个抗骨质疏松的聚酮类化合物F9775A (39)和F9775B (40)[26]。
1.2.3 阻断竞争途径:前体竞争是导致某些次级代谢产物合成基因簇处于隐性状态的原因之一。Gómez等研究Streptomyces lydicus NRRL 2433时发现其中一个PKS-NRPS基因簇可编码特胺酸类抗生素-链霉溶菌素streptolydigin,当他们对streptolydigin生物合成基因进行阻断后发现突变株在丧失合成streptolydigin能力的同时,积累了3个新化合物christolane A (41)、christolane B (42)和christolane C (43)[27]。最近,通过阻断大环多烯类化合物reedsmycin的生物合成正调控基因,我们使突变株积累了新的聚酮类化合物piericidin F (44)[28]。以上结果为运用阻断竞争途径手段实现隐性基因簇的激活提供了有力证据。
1.2.4 共培养(co-culture):在特定生存环境下,不同菌株的共同培养不仅会引起生存竞争,刺激菌株产生自我防御的次级代谢产物,所产生的物质也可能作为诱导因子或前体物质促进其他菌株次级代谢产物的生成。Schroeckh等发现A. nidulans基因组中包含28个PKS基因簇以及24个NRPS基因簇,但却只有少量次级代谢产物表达。当Schroeckh将A. nidulans与其原属地土壤菌Streptomyces hygroscopicus共培养时,转录组分析表明A. nidulans中395个基因呈现明显差异表达,其中248个基因表达上调,147个表达下调[29]。Onaka等发现只有当菌株Streptomyces endus S-522与可以产生霉菌酸的细菌(如Tsukamurella pulmonis)共同培养时才能产生化合物alchivemycin A (45)[30]。
2 基因组信息指导下结构导向(structure-guided)的化合物定向分离技术 基因组信息蕴含了化合物的结构特征,以此为指导靶向进行化合物分离是建立基因簇–化合物之间联系的关键环节。近年来现代波谱学技术的进步大大促进了定向分离技术的发展(图 3,4),在天然产物快速发现过程中发挥了日愈重要的作用(图 5)。
 |
| 图 3 串联质谱分子网络应用于天然产物的定向分离[38] Figure 3 Discovery of natural products guided by MS/MS molecular networking[38]. |
| 图选项 |
 |
| 图 4 基于2D-NMR波谱差别分析的天然产物发掘 Figure 4 Discovery of natural products guided by DNAS. |
| 图选项 |
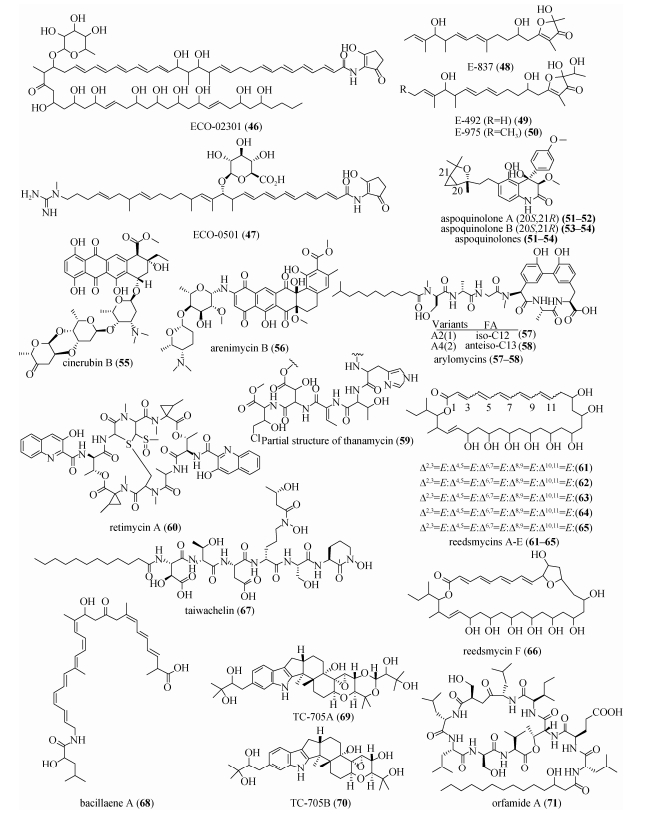 |
| 图 5 基因组信息指导下结构导向的定向分离技术分离的化合物 Figure 5 Compounds isolated by genome-guided structure mining techniques. |
| 图选项 |
2.1 基于UV特征吸收进行定向分离 天然产物中往往含有多种具有特征UV吸收的官能团,通过UV特征吸收对天然产物进行定向分离是一种比较直观而传统有效的方法。如Ecopia BioSciences采用UV-based方法从放线菌中分离得到一系列多烯类抗生素,包括抗真菌药物ECO-02301 (46)[31],抗MRSA(methicillin resistant Staphylococcus aureus)药物ECO-0501 (47)[32]和电子传递抑制剂E-837 (48)、E-492 (49)、E-975 (50)[33]等化合物。Scherlach等在分析A. nidulans的基因组序列时发现其中包括多个氨基苯甲酸合酶基因,结合芳香族生物碱的特征性UV吸收,发现了4个新颖的异戊二烯化芳香喹唑酮aspoquinolones A-D (51-54)[34]。
2.2 基于MS技术进行定向分离 高分辨质谱(high resolution mass spectrometry, HRMS)可以确定化合物的精确分子量及可能的结构式,串联质谱(tandem mass spectrometry,MS/MS)能够根据特征性碎片峰推测结构信息,结合与数据库比对结果实现已知化合物的快速排重。以Dorrestein课题组为代表的美国Scripps海洋研究所科学家在基于MS技术进行定向分离方面做出了突出贡献。他们利用MS/MS技术对发酵产物粗提物中的特征碎片信号进行快速分析,将微生物的基因型和具有特征结构(如肽类和糖基化)代谢产物的化学型联系起来,提出了肽基因组学[35](Peptidogenomics)和糖基因组学(Glycogenomics)概念,发现了抗肿瘤化合物cinerubin B (55)和抗多重耐药菌S. aureus活性的抗生素arenimycin B (56)[36]。将MALDI成像质谱(imaging mass spectrometry,IMS)技术和肽基因组学相结合,他们又将表型与化学型-基因型相联系,从Streptomyces roseosporus中发现了抗感染剂arylomycins (57-58)[37]。2012年,Dorrestein课题组进一步提出了分子网络(molecular networking)的概念,这种方法是对MS/MS得到的分子离子碎片峰的产生规律做出统计,运用软件cytoscape (www.cytoscape.org)将MS/MS碎片峰数据做成可视化的分子网络,通过分析分子网络数据可以快速探寻化合物类型及其同系物。他们采用nanoDESI (Nanospray desorption electrospray ionization) MS对微生物菌落中的代谢产物直接进行分析,通过比对Pseudomonas sp. SH-C52野生株、NRPS基因thaB突变株、卤化酶基因thaC2突变株的MS分子网络,并结合肽基因组学,检测到了抗真菌氯代非核糖体肽类化合物thanamycin (59) (图 3)[38]。随后,通过对35个Salinispora属的菌株进行系统研究,建立了一种分子网络与基于代谢产物-基因簇对应关系图(pattern-based)的基因组挖掘相结合策略,实现了同时比对多个复杂的微生物提取物,快速排除培养基及已知化合物,发现了多个不同类型的新化合物及其相应的合成基因簇,并对S. arenicola CNT-005产生的新的醌霉素类缩酚酸肽retimycin A (60)及其合成基因簇NRPS40进行了研究[39]。
2.3 基于NMR技术进行定向分离 特定化学位移能够体现化合物的结构特征,从而在发掘新的天然产物的过程中起到指导性作用。Che和Li等通过全基因组扫描从海洋来源的Streptomyces sp. CHQ-64中发现了一个编码PKS的DNA片段,对其功能结构域分析并结合其发酵粗浸膏1H NMR的特征化学位移,预测该菌可能产生多烯-多元醇类化合物,进而分离得到了6个新的抗真菌大环多烯类化合物reedsmycins A-F (61-66)[40]。通过对Cupriavidus taiwanensis的基因组进行生物信息学分析,Kreutzer鉴定出一个可能编码非核糖体肽类嗜铁素的生物合成基因簇。嗜铁素包含的异羟肟酸基团可以产生容易区分的15N NMR共振态,因此通过对C. taiwanensis的发酵粗提物进行1H-15N HMBC检测,从相应的组分中分离纯化得到了新的脂肽类化合物taiwachelin (67)[41]。高照射频率核磁共振仪(如900 MHz NMR)的出现及核磁数据分析软件(MestReNova、NMRPipe等)的开发应用,显著提高了NMR测定及分析灵敏度。2007年,哈佛医学院的Walsh和Clardy等利用2D-NMR波谱差别分析法(differential analysis of 2D-NMR spectra,DANS)在Bacillus subtilis发酵产物的粗提物中得到了具有抗菌活性的新型多烯类化合物bacillaene A (68),并初步推测了其生物合成途径[42]。随后Schroeder等利用同样的方法在Tolypocladium cylindrosporum的发酵产物中得到了新型吲哚类生物碱TC-705A (69)和TC-705B (70)[43](图 4)。
2.4 通过同位素标记进行定向分离 同位素标记是进行生物合成研究的传统策略,与基因组信息相结合能够更为有效地起到指导性作用。首先,通过生物信息学分析预测次级代谢产物生物合成可能的前体,然后用同位素标记的底物进行喂养,再采用MS和2D-NMR方法对其发酵液进行分析,最终分离得到含有标记的目标产物。Gross等通过对Pseudomonas fluorescens Pf-5基因组序列进行分析,预测了其中一个NRPS基因簇的编码产物中可能包括Leu、Ile、Glu和Asp四种氨基酸[44];然后把15N标记好的Leu加入到培养基中对P. fluorescens Pf-5进行喂养实验,采用1H-15N HMBC对其发酵液进行检测,最终追踪分离得到新颖的大环脂肽类化合物orfamide A (71)。
3 展望 基于生物信息学、遗传学、微生物学、分子生物学、天然药物化学多学科发展而诞生的基因组挖掘技术为发现新颖天然产物的提供了新的视角和手段,推动着天然产物发现进入了一个新的时代。自然生境与实验室条件的巨大差异可能是导致隐性基因簇沉默的重要原因,而自然生境的复杂性与多样性使微生物中隐性基因簇的激活机制存在独特性与差异性。基于微生物次级代谢产物生物合成调控网络的级联性和复杂性,更多的激活机制亟待人们的探索和发现,而综合采用多种策略进行隐性基因簇激活也成为必然。随着基因组和宏基因组大规模测序的广泛开展,遗传操作技术和基因编辑技术的不断完善,以及科学家对不同结构类型天然产物合成机制的理解与认知,越来越多的新颖结构化合物的隐性基因簇将不断被发掘。通过激活这些基因簇而获得活性化合物将成为新药研发的重要手段,为发现新药治疗临床多种难治性疾病带来更多希望。
References
| [1] | Koehn FE, Carter GT. The evolving role of natural products in drug discovery. Nature Reviews Drug Discovery, 2005, 4(3): 206-220. DOI:10.1038/nrd1657 |
| [2] | Bentley SD, Chater KF, Cerde?o-Tárraga AM, Challis GL, Thomson NR, James KD, Harris DE, Quail MA, Kieser H, Harper D, Bateman A, Brown S, Chandra G, Chen CW, Collins M, Cronin A, Fraser A, Goble A, Hidalgo J, Hornsby T, Howarth S, Huang CH, Kieser T, Larke L, Murphy L, Oliver K, O'Neil S, Rabbinowitsch E, Rajandream MA, Rutherford K, Rutter S, Seeger K, Saunders D, Sharp S, Squares R, Squares S, Taylor K, Warren T, Wietzorrek A, Woodward J, Barrell BG, Parkhill J, Hopwood DA. Complete genome sequence of the model actinomycete Streptomyces Streptomyces coelicolor A3(2). Nature, 2002, 417(6885): 141-147. DOI:10.1038/417141a |
| [3] | Liu G, Chater KF, Chandra G, Niu GQ, Tan HR. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. DOI:10.1128/MMBR.00054-12 |
| [4] | Zhang YH, Huang HM, Xu SS, Wang B, Ju JH, Tan HR, Li WL. Activation and enhancement of Fredericamycin A production in deepsea-derived Streptomyces somaliensis SCSIO ZH66 by using ribosome engineering and response surface methodology. Microbial Cell Factories, 2015, 14: 64. DOI:10.1186/s12934-015-0244-2 |
| [5] | Rutledge PJ, Challis GL. Discovery of microbial natural products by activation of silent biosynthetic gene clusters. Microbiology, 2015, 13(8): 509. |
| [6] | Franke J, Ishida K, Hertweck C. Genomics-driven discovery of burkholderic acid, a noncanonical, cryptic polyketide from human pathogenic Burkholderia species. Angewandte Chemie International Edition, 2012, 51(46): 11611-11615. DOI:10.1002/anie.201205566 |
| [7] | Yeh HH, Ahuja M, Chiang YM, Oakley CE, Moore S, Yoon O, Hajovsky H, Bok JW, Keller NP, Wang CCC, Oakley BR. Resistance gene-guided genome mining:serial promoter exchanges in Aspergillus nidulans reveal the biosynthetic pathway for fellutamide B, a proteasome inhibitor. ACS Chemical Biology, 2016, 11(8): 2275-2284. DOI:10.1021/acschembio.6b00213 |
| [8] | Laureti L, Song LJ, Huang S, Corre C, Leblond P, Challis GL, Aigle B. Identification of a bioactive 51-membered macrolide complex by activation of a silent polyketide synthase in Streptomyces ambofaciens. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(15): 6258-6263. DOI:10.1073/pnas.1019077108 |
| [9] | Gottelt M, Kol S, Gomez-Escribano JP, Bibb M, Takano E. Deletion of a regulatory gene within the cpk gene cluster reveals novel antibacterial activity in Streptomyces Streptomyces coelicolor A3(2). Microbiology, 2010, 156(8): 2343-2353. DOI:10.1099/mic.0.038281-0 |
| [10] | Gomez-Escribano JP, Song LJ, Fox DJ, Bibb MJ, Yeo V, Challis GL. Structure and biosynthesis of the unusual polyketide alkaloid coelimycin P1, a metabolic product of the cpk gene cluster of Streptomyces Streptomyces coelicolor M145. Chemical Science, 2012, 3(9): 2716-2720. DOI:10.1039/c2sc20410j |
| [11] | Correa C, Song LJ, O'Rourke S, Chater KF, Challis GL. 2-Alkyl-4-hydroxymethylfuran-3-carboxylic acids, antibiotic production inducers discovered by Streptomyces Streptomyces coelicolor genome mining. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(45): 17510-17515. DOI:10.1073/pnas.0805530105 |
| [12] | Yamanaka K, Reynolds KA, Kersten RD, Ryan KS, Gonzalez DJ, Nizet V, Dorrestein PC, Moore BS. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(5): 1957-1962. DOI:10.1073/pnas.1319584111 |
| [13] | Hayashi S, Ozaki T, Asamizu S, Ikeda H, ōmura S, Oku N, Igarashi Y, Tomoda H, Onaka H. Genome mining reveals a minimum gene set for the biosynthesis of 32-membered macrocyclic thiopeptides lactazoles. Chemistry & Biology, 2014, 21(5): 679-688. |
| [14] | Shao ZY, Rao GD, Li C, Abil Z, Luo YZ, Zhao HM. Refactoring the silent spectinabilin gene cluster using a plug-and-play scaffold. ACS Synthetic Biology, 2013, 2(11): 662-669. DOI:10.1021/sb400058n |
| [15] | Guo F, Xiang SH, Li LY, Wang B, Rajas?rkk? J, Gr?ndahl-Yli-Hannuksela K, Ai GM, Mets?-Ketel? M, Yang KQ. Targeted activation of silent natural product biosynthesis pathways by reporter-guided mutant selection. Metabolic Engineering, 2015, 28: 134-142. DOI:10.1016/j.ymben.2014.12.006 |
| [16] | Komatsu M, Tsuda M, Omura S, Oikawa H, Ikeda H. Identification and functional analysis of genes controlling biosynthesis of 2-methylisoborneol. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(21): 7422-7427. DOI:10.1073/pnas.0802312105 |
| [17] | Zhao B, Lin X, Lei L, Lamb DC, Kelly SL, Waterman MR, Cane DE. Biosynthesis of the sesquiterpene antibiotic albaflavenone in Streptomyces Streptomyces coelicolor A3(2). The Journal of Biological Chemistry, 2008, 283(13): 8183-8189. DOI:10.1074/jbc.M710421200 |
| [18] | Chou WK, Fanizza I, Uchiyama T, Komatsu M, Ikeda H, Cane DE. Genome mining in Streptomyces avermitilis:cloning and characterization of SAV_76, the synthase for a new sesquiterpene, Avermitilol. Journal of the American Chemical Society, 2010, 132(26): 8850-8851. DOI:10.1021/ja103087w |
| [19] | Yu P, Liu SP, Bu QT, Zhou ZX, Zhu ZH, Huang FL, Li YQ. WblAch, a pivotal activator of natamycin biosynthesis and morphological differentiation in Streptomyces chattanoogensis L10, is positively regulated by AdpAch. Applied and Environmental Microbiology, 2014, 80(22): 6879-6887. DOI:10.1128/AEM.01849-14 |
| [20] | Lu C, Liao GJ, Zhang JH, Tan HR. Identification of novel tylosin analogues generated by a wblA disruption mutant of Streptomyces ansochromogenes. Microbial Cell Factories, 2015, 14: 173. DOI:10.1186/s12934-015-0359-5 |
| [21] | Huang HM, Hou LK, Li HY, Qiu YH, Ju JH, Li WL. Activation of a plasmid-situated type Ⅲ PKS gene cluster by deletion of a wbl gene in deepsea-derived Streptomyces somaliensis SCSIO ZH66. Microbial Cell Factories, 2016, 15: 116. DOI:10.1186/s12934-016-0515-6 |
| [22] | Huang HM, Li HY, Qiu YH, Hou LK, Ju JH, Li WL. A new dioic acid from a wbl gene mutant of deepsea-derived Streptomyces somaliensis SCSIO ZH66. Marine Drugs, 2016, 14(10): 184. DOI:10.3390/md14100184 |
| [23] | Li HY, Huang HM, Hou LK, Ju JH, Li WL. Discovery of antimycin-type depsipeptides from a wbl gene mutant strain of deepsea-derived Streptomyces somaliensis SCSIO ZH66 and their effects on pro-inflammatory cytokine production. Frontiers in Microbiology, 2017, 8: 678. |
| [24] | Williams RB, Henrikson JC, Hoover AR, Lee AE, Cichewicz RH. Epigenetic remodeling of the fungal secondary metabolome. Organic & Biomolecular Chemistry, 2008, 6(11): 1895-1897. |
| [25] | Henrikson JC, Hoover AR, Joynera PM, Cichewicz RH. A chemical epigenetics approach for engineering the in situ biosynthesis of a cryptic natural product from Aspergillus niger. Organic & Biomolecular Chemistry, 2009, 7(3): 435-438. |
| [26] | Bok JW, Chiang YM, Szewczyk E, Reyes-Dominguez Y, Davidson AD, Sanchez JF, Lo HC, Watanabe K, Strauss J, Oakley BR, Wang CCC, Keller NP. Chromatin-level regulation of biosynthetic gene clusters. Nature Chemical Biology, 2009, 5(7): 462-464. DOI:10.1038/nchembio.177 |
| [27] | Gómez C, Olano C, Palomino-Sch?tzlein M, Pineda-Lucena A, Carbajo RJ, Bra?a AF, Méndez C, Salas JA. Novel compounds produced by Streptomyces lydicus NRRL 2433 engineered mutants altered in the biosynthesis of streptolydigin. The Journal of Antibiotics, 2012, 65(7): 341-348. DOI:10.1038/ja.2012.37 |
| [28] | Han XN, Liu ZZ, Zhang ZZ, Zhang XM, Zhu TJ, Gu QQ, Li WL, Che Q, Li DH. Geranylpyrrol A and piericidin F from Streptomyces sp. CHQ-64 ΔrdmF. The Journal of Natural Products, 2017, 80(5): 1684-1687. DOI:10.1021/acs.jnatprod.7b00016 |
| [29] | Schroeckh V, Scherlach K, Nützmann HW, Shelest E, Schmidt-Heck W, Schuemann J, Martin F, Hertweck C, Brakhage AA. Intimate bacterial-fungal interaction triggers biosynthesis of archetypal polyketides in Aspergillus nidulans. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(34): 14558-14563. DOI:10.1073/pnas.0901870106 |
| [30] | Onaka H, Mori Y, Igarashi Y, Furumai T. Mycolic acid-containing bacteria induce natural-product biosynthesis in Streptomyces species. Applied and Environmental Microbiology, 2011, 77(2): 400-406. DOI:10.1128/AEM.01337-10 |
| [31] | McAlpine JB, Bachmann BO, Piraee M, Tremblay S, Alarco AM, Zazopoulos E, Farnet CM. Microbial genomics as a guide to drug discovery and structural elucidation:ECO-02301, a novel antifungal agent, as an example. The Journal of Natural Products, 2005, 68(4): 493-496. DOI:10.1021/np0401664 |
| [32] | Banskota AH, McAlpine JB, S?rensen D, Ibrahim A, Aouidate M, Piraee M, Alarco AM, Farnet CM, Zazopoulos E. Genomic analyses lead to novel secondary metabolites. Part 3. ECO-0501, a novel antibacterial of a new class. The Journal of Antibiotics, 2006, 59(9): 533-542. DOI:10.1038/ja.2006.74 |
| [33] | Banskota AH, McAlpine JB, S?rensen D, Aouidate M, Piraee M, Alarco AM, Omura S, Shiomi K, Farnet CM, Zazopoulos E. Isolation and identification of three new 5-Alkenyl-3, 3(2H)-furanones from two Streptomyces species using a genomic screening approach. The Journal of Antibiotics, 2006, 59(3): 168-176. DOI:10.1038/ja.2006.24 |
| [34] | Scherlach K, Hertweck C. Discovery of aspoquinolones A-D, prenylated quinoline-2-one alkaloids from Aspergillus nidulans, motivated by genome mining. Organic & Biomolecular Chemistry, 2006, 4(18): 3517-3520. |
| [35] | Kersten RD, Yang YL, Xu YQ, Cimermancic P, Nam SJ, Fenical W, Fischbach MA, Moore BS, Dorrestein PC. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology, 2011, 7(11): 794-802. DOI:10.1038/nchembio.684 |
| [36] | Kersten RD, Ziemert N, Gonzalez DJ, Duggan BM, Nizet V, Dorrestein PC, Moore BS. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(47): E4407-E4416. DOI:10.1073/pnas.1315492110 |
| [37] | Liu WT, Kersten RD, Yang YL, Moore BS, Dorrestein PC. Imaging mass spectrometry and genome mining via short sequence tagging identified the anti-infective agent arylomycin in Streptomyces roseosporus. Journal of the American Chemical Society, 2011, 133(45): 18010-18013. DOI:10.1021/ja2040877 |
| [38] | Watrous J, Roach P, Alexandrov T, Heath BS, Yang JY, Kersten RD, van der Voort M, Pogliano K, Gross H, Raaijmakers JM, Moore BS, Laskin J, Bandeira N, Dorrestein PC. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(26): E1743-E1752. DOI:10.1073/pnas.1203689109 |
| [39] | Duncan KR, Crüsemann M, Lechner A, Sarkar A, Li J, Ziemert N, Wang MX, Bandeira N, Moore BS, Dorrestein PC, Jensen PR. Molecular networking and pattern-based genome mining improves discovery of biosynthetic gene clusters and their products from Salinispora species. Chemistry & Biology, 2015, 22(4): 460-471. |
| [40] | Che Q, Li T, Liu XF, Yao TT, Li J, Gu QQ, Li DH, Li WL, Zhu TJ. Genome scanning inspired isolation of reedsmycins A-F, polyene-polyol macrolides from Streptomyces sp. CHQ-64. RSC Advances, 2015, 5(29): 22777-22782. DOI:10.1039/C4RA15415K |
| [41] | Kreutzer MF, Nett M. Genomics-driven discovery of taiwachelin, a lipopeptide siderophore from Cupriavidus taiwanensis. Organic & Biomolecular Chemistry, 2012, 10(47): 9338-9343. |
| [42] | Butcher RA, Schroeder FC, Fischbach MA, Straight PD, Kolter R, Walsh CT, Clardy J. The identification of bacillaene, the product of the PksX megacomplex in Bacillus subtilis. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(5): 1506-1509. DOI:10.1073/pnas.0610503104 |
| [43] | Schroeder FC, Gibson DM, Churchill ACL, Sojikul P, Wursthorn EJ, Krasnoff SB, Clardy J. Differential analysis of 2D NMR spectra:new natural products from a pilot-scale fungal extract library. Angewandte Chemie International Edition, 2007, 46(6): 901-904. DOI:10.1002/(ISSN)1521-3773 |
| [44] | Gross H, Stockwell VO, Henkels MD, Nowak-Thompson B, Loper JE, Gerwick WH. The genomisotopic approach:a systematic method to isolate products of orphan biosynthetic gene clusters. Chemistry & Biology, 2007, 14(1): 53-63. |
