孙大庆1

 , 李洪飞1, 杨健2, 宋大巍2
, 李洪飞1, 杨健2, 宋大巍2 1.黑龙江八一农垦大学国家杂粮工程技术研究中心, 黑龙江 大庆 163319;
2.黑龙江八一农垦大学食品学院, 黑龙江 大庆 163319
收稿日期:2017-03-22;修回日期:2017-04-25;网络出版日期:2017-04-27
基金项目:黑龙江省青年科学基金(QC2014C020);牡丹江市科学技术计划项目(Z2016n0015);黑龙江八一农垦大学“校内培育课题资助计划”项目(XZR2016-15)
*通信作者:孙大庆, Tel:+86-459-2673850;Fax:+86-459-2673847;E-mail:sundaqing1979@163.com
摘要:[目的]为了探索植物乳杆菌天然质粒系统进化关系和起源。[方法]本文利用复制起始蛋白(replication initiation protein,Rep)系统进化树、基因组共线性、基因组GC含量和宿主范围分析方法,对植物乳杆菌75个天然质粒的系统进化关系和起源进行了详细和多角度的分析。[结果]首先,Rep系统进化树和基因组共线性分析结果均表明,植物乳杆菌所有天然质粒可以划分为6个进化关系亲密的家族、2个进化形态特殊的杂合质粒和1个独立进化质粒pLP2140。杂合质粒pMRI5.2、pLP12-1分别由家族1-2和5-6质粒融合形成,因此植物乳杆菌质粒可能起源于7个祖先。其次,基因组共线性分析可以将6个家族质粒进一步划分为17个进化关系更近的亚家族类群,并清晰、有效地揭示类群内质粒之间的系统进化关系。最后,基因组GC含量和宿主范围分析为植物乳杆菌质粒的系统进化关系和起源提供了进一步的证据。[结论]因此上述研究可以准确、有效地揭示植物乳杆菌天然质粒的系统进化关系和起源,这对植物乳杆菌天然质粒系统进化和起源的了解和研究具有重要的参考价值。通过Rep系统进化树和基因组共线性两种分析方法优缺点的比较和组合,我们提出了一种更加有效的研究思路和分析方法,同时这种方法很可能适用于所有细菌天然质粒,因此对于天然质粒进化和起源研究具有普遍的方法学意义。
关键词: 植物乳杆菌 天然质粒 系统进化树 基因组共线性 最小复制子
Phylogenetic evolution and origin of natural plasmids of Lactobacillus plantarum
Daqing Sun1

 , Hongfei Li1, Jian Yang2, Dawei Song2
, Hongfei Li1, Jian Yang2, Dawei Song2 1.National Coarse Cereals Engineering Research Center, Heilongjiang Bayi Agricultural University, Daqing 163319, Heilongjiang Province, China;
2.College of Food Science, Heilongjiang Bayi Agricultural University, Daqing 163319, Heilongjiang Province, China
Received 22 March 2017; Revised 25 April 2017; Published online 27 April 2017
*Corresponding author: Daqing Sun, Tel:+86-459-2673850;Fax:+86-459-2673847;E-mail:sundaqing1979@163.com
Supported by the Youth Science Foundation of Heilongjiang Province (QC2014C020), by the Science and Technology Project of Mudanjiang City (Z2016n0015) and by the School Cultivation Subject of Heilongjiang Bayi Agricultural University (XZR2016-15)
Abstract: [Objective]In order to explore the evolutionary relationship and origin of natural plasmid of Lactobacillus plantarum.[Methods]We analyzed phylogenetic relationships and origins of 75 natural plasmids of L. plantarum by replication initiation protein (Rep) phylogenetic tree, genomic collinearity, genomic GC content and host range.[Results]Rep phylogenetic tree and genomic collinearity analysis simultaneously showed that all natural plasmids of L. plantarum could be divided into 6 families with close evolutionary relationships, 2 complex plasmids with special evolution form and 1 independent evolution plasmid pLP2140. The complex plasmids pMRI5.2 and pLP12-1 were fused by 2 different plasmids of the family 1-2 and family 5-6. Therefore, the natural plasmids of L. plantarum could be originated from 7 ancestors. The genomic collinearity analysis showed that the 6 family plasmids could be further divided into 17 subfamily groups with closer evolutionary relationships, and the phylogenetic relationships among plasmids could be clearly and effectively revealed at the subfamily level. Finally, the analysis of genomic GC content and host range provided further evidence for the phylogenetic relationship and origin of the natural plasmid of L. plantarum.[Conclusion]Therefore, the above research can accurately and effectively reveal the phylogenetic relationship and origin of the natural plasmids of Lactobacillus plantarum, which is of great reference value for the acquaintance and study of the evolution and origin of the natural plasmids of Lactobacillus plantarum. We proposed a more effective research strategy and analytical method of the evolution and origin of natural plasmids by comparing and combining the characteristics of the analytical methods of Rep phylogenetic tree and genome collinearity, and this method may be applicable to all bacterial natural plasmids, so it may have universal methodological significance for the study of the evolution and origin of natural plasmids.
Key words: Lactobacillus plantarum natural plasmid phylogenetic tree genomic collinearity minimal replicon
随着研究的不断深入,乳杆菌与人类饮食、医疗、卫生和健康的关系越来越密切,它们一直是乳酸菌研究领域的热点和前沿。目前乳杆菌属已鉴定的菌种数量多达222个(http://www.bacterio.net/lactobacillus.html),近年研究显示,乳杆菌属菌种之间进化的复杂程度超过一般“科”的分化程度[1],因此乳杆菌是乳酸菌中遗传、进化多样性十分丰富的群体。然而由于基因丢失、环境选择压力等原因,多种乳杆菌发生了高度专一性进化,它们只能在有限的环境中生存,例如德氏乳杆菌已高度适应乳环境,嗜酸乳杆菌、约氏乳杆菌则更喜欢栖息于胃肠道中,但植物乳杆菌似乎是个例外,它在自然界广泛分布,对多种不同的生存环境具有高度的适应性,这可能主要得益于它含有乳杆菌中超大且结构多样的基因组,因此被称为“自然代谢工程师”[2]。此外据我们统计分析发现,植物乳杆菌是乳杆菌属甚至乳酸菌中天然质粒最丰富和最多样的菌种[3],这些天然质粒在不增加染色体大小情况下,可为植物乳杆菌提供多种多样的生理学和生态学表型特征,从而为植物乳杆菌适应生存环境变化提供灵活且必要的遗传物质基础。因此植物乳杆菌中丰富、多样的天然质粒群体为乳杆菌属和乳酸菌质粒研究提供了理想的研究对象,它们在植物乳杆菌生长、发育、遗传和进化过程中很可能扮演着十分重要的角色。
以往植物乳杆菌质粒研究多集中于功能基因元件分离、鉴定[4-5]和克隆、表达载体构建[6-7],而质粒系统进化和起源的研究关注很少,只有个别质粒基于序列同源性推测了起源[8],因此至今我们对于植物乳杆菌天然质粒群体的进化和起源的了解都非常有限,这极大地限制了质粒和植物乳杆菌(宿主)在生理、生态学方面互作关系的研究。本研究的目的是准确、有效地揭示植物乳杆菌天然质粒的系统进化关系和起源,增加植物乳杆菌天然质粒进化机制和演化过程的了解,为深入研究天然质粒在植物乳杆菌生长、发育、遗传和进化过程中发挥的作用奠定基础。
1 材料和方法 1.1 质粒基因组序列来源 植物乳杆菌99个质粒基因组序列来自NCBI的RefSeq数据库(截止到2017.1.24),其中质粒pLY7801全基因组序列(登录号NZ_CP016631.1)为本实验室首次测定。质粒pC194、pMV158、pUCL287登录号为NC_002013.1、NC_010096.1、X75607.1,质粒pAD1、pIP501基因组序列来自文献[9]和[10]。
1.2 质粒基因组序列的收集、整理和初步分析 通过检索获得NCBI RefSeq数据库中所有收录的植物乳杆菌质粒基因组信息,选择基因组完整且没有拼接错误的质粒,下载其基因组序列及其注释信息。筛查每个质粒Rep编码情况,对注释或疑似的Rep进行BLAST比对分析,鉴定其保守结构域,确定其所属蛋白家族。
1.3 质粒Rep系统进化树构建 利用DNAMAN 8.0.8.789软件进行多序列一致性分析。利用MEGA 7.0软件[11]进行系统进化树构建。均采用Neighbor-Joining模型构建系统进化树,并采用Bootstrap 1000计算分支置信度。
1.4 质粒基因组共线性分析 利用Mauve 2.4.0软件[12]进行质粒基因组共线性分析,所有参数为默认设置。可视化结果按照质粒大小、家族和类群进行排序。
1.5 质粒家族宿主范围分析 利用每个质粒家族共有的最小复制子核心序列进行BLAST分析,对所有检索到的同源性序列的宿主进行统计和整理。
2 结果和分析 2.1 植物乳杆菌质粒基因组基本特征 经检索、比对和统计分析,RefSeq数据库共收录99个质粒完整基因组序列,其中24个质粒序列存在完全重复的区域(大于10 kb),很可能是错误拼接造成的,因此不做后续分析。其余75个质粒基因组基本特征及编码Rep情况汇总于表 1。由表 1可知,植物乳杆菌75个质粒基因组大小为1.80-74.17 kb,GC含量33.37%-42.56%,其中51个质粒编码已知的Rep,这些Rep分属于5个Rep家族,质粒pMRI5.2、pZL3和pLP12-1编码2个Rep,24个质粒没有编码已知Rep。
表 1. 植物乳杆菌质粒基因组一般特征 Table 1. General characteristics of plasmid genome in Lactobacillus plantarum
| Plasmid name | Genome size/kb | Genome GC content/% | Rep family (size/aa) |
| pLPI8 | 1.80 | 37.49 | Rep_2/237 |
| pWCFS101 | 1.91 | 39.49 | Rep_1/319 |
| pCAUH201 | 1.94 | 37.81 | Rep_1/317 |
| pLP2000 | 2.06 | 38.23 | Rep_1/317 |
| pLR1 | 2.06 | 37.75 | Rep_1/318 |
| p200 | 2.06 | 38.22 | Rep_1/317 |
| pLD1 | 2.11 | 37.78 | Rep_1/318 |
| pLP2111 | 2.11 | 38.32 | Rep_1/317 |
| pCMPG5300.01 | 2.12 | 38.03 | Rep_1/317 |
| pC30i1 | 2.14 | 36.96 | Rep_1/317 |
| pLP2140 | 2.14 | 38.27 | N |
| pLTK2 | 2.29 | 38.56 | Rep_1/317 |
| pWCFS102 | 2.36 | 34.33 | Rep_2/219 |
| pA1 | 2.82 | 34.96 | Rep_2/196 |
| pPB1 | 2.89 | 37.74 | Rep_2/208 |
| pXY3 | 2.96 | 38.24 | Rep_2/219 |
| pR18 | 3.21 | 35.81 | Rep_1/317 |
| pZL2 | 3.26 | 38.24 | Rep_1/318 |
| pM4 | 3.32 | 38.73 | Rep_1/318 |
| pG6301 | 3.51 | 37.26 | Rep_1/318 |
| pLFE1 | 4.03 | 34.43 | Rep_2/212 |
| pMRI5.2 | 5.21 | 35.79 | Rep_1/314; Rep_2/215 |
| pLJ42 | 5.52 | 42.41 | Rep_3/308 |
| LZ206p1 | 6.11 | 37.47 | N |
| pCAUH202 | 6.40 | 33.68 | Rep_3/311 |
| Lp16L | 6.46 | 34.67 | N |
| p256 | 7.22 | 36.73 | N |
| Lp16A | 7.24 | 33.37 | Rep_3/311 |
| Lp16B | 8.63 | 35.93 | Rep_3/311 |
| LBPp6 | 8.68 | 35.98 | Rep_3/311 |
| pG6302 | 9.11 | 36.39 | Rep_3/200 |
| p100 | 9.25 | 37.37 | N |
| pLP9000 | 9.25 | 37.37 | N |
| pLY7801 | 9.34 | 34.26 | Rep_3/309 |
| pG6303 | 10.04 | 36.83 | N |
| pMD5057 | 10.87 | 36.22 | Rep_3/311 |
| pCMPG5300.02 | 10.94 | 38.59 | Rep_3/309 |
| LZ206p3 | 11.65 | 34.29 | N |
| pZL3 | 11.89 | 37.66 | Rep_3/304; Rep_3/311 |
| ZJ95p2 | 12.48 | 36.37 | N |
| pZL4 | 12.51 | 39.38 | Rep_3/311 |
| Lp16I | 13.34 | 37.91 | Rep_3/307 |
| pLP-ZJ101 | 15.16 | 40.17 | Rep_3/311 |
| LBPp7 | 15.17 | 39.68 | N |
| LBPp5 | 16.10 | 42.14 | RepA_Na/155 |
| pLP12-6 | 21.51 | 41.21 | RepA_N/357 |
| Lp16C | 27.28 | 39.86 | RepA_N/366 |
| pCMPG5300.03 | 27.83 | 40.55 | RepA_N/373 |
| pLP12-5 | 27.90 | 39.30 | RepA_Na/97 |
| LZ206p2 | 33.00 | 39.93 | RepA_N/367 |
| pLTK13 | 34.52 | 39.04 | RepA_Na/102 |
| LZ227p3 | 35.46 | 39.61 | RepA_Na/102 |
| pWCFS103 | 36.06 | 40.83 | RepA_Na/102 |
| LBPp4 | 37.04 | 41.24 | N |
| Lp16D | 37.09 | 40.79 | N |
| LZ227p5 | 38.21 | 39.80 | N |
| pLP-ZJ102 | 39.11 | 38.69 | N |
| LBPp3 | 39.46 | 42.29 | N |
| pLP12-3 | 40.05 | 41.34 | RepA_Na/102 |
| Lp16E | 40.14 | 41.36 | RepA_N/367 |
| pLP-ZJ103 | 41.50 | 39.50 | N |
| LBPp1 | 45.41 | 39.67 | RepA_N/327 |
| ZJ95p1 | 48.55 | 39.05 | N |
| LBPp2 | 49.01 | 42.01 | N |
| Lp16F | 50.19 | 39.78 | N |
| Lp16G | 51.85 | 42.56 | RepR/512 |
| LZ227p2 | 52.14 | 41.83 | N |
| pST-Ⅲ | 53.56 | 38.69 | N |
| pLP12-2 | 55.31 | 39.47 | N |
| pMF1298-2 | 55.69 | 40.05 | N |
| pMF1298-1 | 63.11 | 41.14 | RepR/510 |
| pLP12-1 | 64.10 | 41.38 | RepA_Na/102; RepR /508 |
| Lp16H | 74.07 | 41.46 | RepR/510 |
| LZ227p1 | 74.17 | 41.33 | RepR/512 |
| N: There is no known Rep. Rep_1: pfam01446. Rep_2: pfam01719. Rep_3: pfam01051. RepA_N: pfam06970. RepR (PriCT-1): pfam08708. a: The Rep belonging to the RepA_N family was deduced according to the amino acid sequence identity. | |||
表选项
2.2 植物乳杆菌编码Rep质粒的系统进化关系 为了准确、有效地分析植物乳杆菌编码Rep质粒的系统进化关系,根据5个已知的Rep家族(表 1),引入5个典型质粒家族的代表性质粒作为参照质粒,进行后续质粒进化关系分析。参照质粒pC194、pMV158、pUCL287、pAD1、pIP501分别属于pC194、pMV158、pUCL287、RepA_N和pIP501质粒家族,前2个家族属于滚环复制(rolling circle replication,RCR)质粒,后3个家族属于theta复制质粒。基于Rep氨基酸序列同源性构建51个植物乳杆菌质粒和5个参照质粒的系统进化树,结果见图 1。根据进化树的拓扑结构和分支点置信度可知,植物乳杆菌50个质粒与5个参照质粒明显聚类,质粒pG6302单独成支。由于编码2个Rep蛋白,质粒pMRI5.2同时与家族1和家族2质粒聚类,质粒pLP12-1同时与家族5和家族6质粒聚类,质粒pZL3的2个Rep均与家族4质粒聚类。由此可知,质粒pMRI5.2可能是pC194家族和pMV158家族来源的2个RCR质粒重组进化形成的,这一推测与Gyu-Sung等[11]研究结果一致;质粒pLP12-1可能由RepA_N家族和pIP501家族来源的2个theta复制质粒重组进化形成;质粒pZL3或许是pUCL287家族来源的2个质粒重组进化的结果。由于质粒pMRI5.2和pLP12-1含有2个不同家族Rep,因此它们属于特殊进化阶段的杂合质粒。质粒pG6302 Rep含有一个不完整的家族4质粒Rep普遍存在的保守结构域(pfam01051),但质粒pG6302与家族4质粒没有聚类,并且与进化树中最近的分支(家族5)具有较低的置信度(46%)和遥远的遗传距离,因此推测质粒pG6302与其他家族质粒没有显示明显的进化关系,可能具有独立的进化祖先和起源。综上所述,基于质粒Rep系统进化树分析,植物乳杆菌编码Rep的51个质粒可以划分为6个进化关系密切的家族,因此它们可能起源于6个祖先质粒。虽然每个家族质粒与参照质粒显示了一定的分歧进化程度,但与不同家族之间的遗传距离比较,参照质粒与聚类家族质粒遗传距离更近,并且分支处具有高分值的置信度,因此每个家族质粒与聚类的参照质粒很可能具有共同的进化祖先和起源。
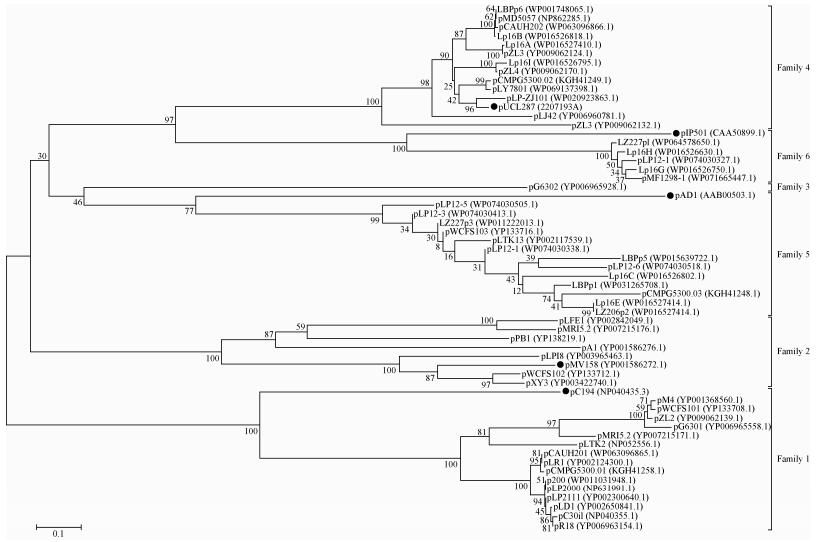 |
| 图 1 植物乳杆菌质粒Rep系统进化树 Figure 1 Phylogenetic tree of plasmid Rep in Lactobacillus plantarum. The plasmids that mark solid black dots are family-known reference plasmids. The characters in parentheses are accession numbers of the plasmid-encoded Rep amino acid sequence. |
| 图选项 |
为了进一步分析每个家族质粒的进化关系,基于Rep氨基酸序列一致性,剔除特殊的杂合质粒和质粒pG6302,分别重新构建每个家族质粒Rep系统进化树,结果见图 2。
 |
| 图 2 植物乳杆菌质粒家族Rep系统进化树 Figure 2 Phylogenetic tree of the Reps of plasmid family in Lactobacillus plantarum. A, B, C, D and E are phylogenetic trees of Lactobacillus plantarum plasmid family 1, 2, 4, 5 and 6, respectively. The characters in parentheses are accession numbers of the plasmid-encoded Rep amino acid sequence. |
| 图选项 |
2.2.1 编码Rep家族1质粒系统进化关系: 由图 2-A可知,编码Rep家族1质粒可以进一步划分为3个进化关系更近的亚家族类群。家族1质粒Rep与质粒pC194 Rep (232 aa)仅具有20.94%-24.30%一致性,因此进化树中质粒pC194与家族1质粒显示了较远的进化距离和进化关系,但序列比对分析发现,家族1质粒Rep均含有3个保守Motifs (表 2),并且这些Motifs被证实是pC194家族质粒复制起始过程中关键的功能位点[12]。因此基于3个Motifs保守性,家族1质粒可能属于RCR质粒pC194家族,与pC194家族质粒具有共同的进化祖先和起源,但它们的分歧进化时间比较久远。
表 2. 家族1质粒Rep保守基序 Table 2. Conserved motifs of the plasmid Reps in the family 1
| Term | Motif Ⅰ | Motif Ⅱ | Motif Ⅲ |
| Group 1 | FLTLTVKN | QHLHVLL/VF | TAKYEVKSAD |
| Group 2 | FLTLTVK/EN | HHMHVLLF | TAKYQVKSKD |
| Group 3 | FLTLTEEN | QHLHVLLF | TAKYQVKSAD |
| Conserved site | FLTLT**N | *H*HVL*F | TAKY*VKS*D |
| pC194 | FLTLTTPN | PHFHVLIA | MAKYSGKDSD |
| *: Not conserved site. | |||
表选项
2.2.2 编码Rep家族2质粒系统进化关系: 由图 2-B可知,编码Rep家族2质粒可以进一步划分为2个进化关系更近的类群,参照质粒pMV158与类群1质粒聚类。家族2质粒Rep与质粒pMV158 Rep (210 aa)具有20.55%-62.56%一致性。进一步序列分析发现,家族2多数质粒均含有pMV158家族Rep编码的5个保守的Motifs[13],只有质粒pA1比较特殊(表 3)。因此基于Rep大小、序列一致性,尤其Motifs序列保守性,家族2质粒很可能属于RCR质粒pMV158家族,并且与pMV158家族质粒具有共同的祖先和起源。
表 3. 家族2质粒Rep保守基序 Table 3. Conserved motifs of the plasmid Reps in the family 2
| Group | Term | Motif Ⅰ | Motif Ⅱ | Motif Ⅲ | Motif Ⅳ | Motif Ⅴ |
| Group 1 | pLPI8 | F S F I I YPESI | AISPLHDCD | KKPHYHVL--Y | VDSVENTFKYLTHESKD | YAK |
| pWCFS102 | F T F LLYPESI | AISPLHDKD | KKAHYHVI--Y | VQSMENMYLYLTHESKD | YSK | |
| pXY3 | F T F LLYPESI | AISPLHDKD | KKAHYHVI--Y | VQSMDN I YLYLTHESKN | YSK | |
| Group 2 | pLFE1 | WWIVVYPESL | AISPLHDKD | KKPHYHIVFNY | I S GLTGAVRYLTH-MDN | YDN |
| pPB1 | WSWIVYPESA | IESPLHDKD | KKHHWHII-IS | VASLQGAVQYLWHR-NN | YDK | |
| pA1 | WTFIVYPESA | IESPLHDKD | KKSHYHILLTF | - | - | |
| Conserved site | *****YPES* | **SPLHD*D | KK*H*H***** | *********Y**H**** | Y** | |
| pMV158 | F T F LLYPESI | AISPLHDKD | KKAHYHVL--Y | VLNVENMYLYLTHESKD | YDK | |
| *: Not conserved site; -: There is not conserved motif. | ||||||
表选项
2.2.3 编码Rep家族4质粒系统进化关系: 由图 2-C可知,编码Rep家族4质粒可以进一步划分为2个进化关系更近的类群,参照质粒pUCL287与类群1质粒聚类。家族4质粒Rep与质粒pUCL287 Rep (311 aa)具有高度一致性(68.17%-91.32%),因此,基于Rep大小和序列的高度保守性,家族4质粒很可能属于theta复制pUCL287家族[14],并且具有共同的祖先和起源。进化树中质粒pLJ42与家族4其他质粒遗传距离和进化关系最远,这表明与家族4类群1质粒相比,它可能经历了相对独立的进化事件或过程。
2.2.4 编码Rep家族5质粒系统进化关系: 由图 2-D可知,编码Rep家族5质粒之间具有紧密的进化关系,参照质粒pAD1 Rep (336 aa)与家族5质粒Rep具有很低的一致性(4.44%-29.07%),因此在进化树中与家族5质粒遗传距离和进化关系最远。进一步序列比对分析发现,家族5质粒较大的Rep (大于200 aa)与质粒pAD1 Rep N端显示高度一致性;家族5质粒较小的Rep (小于200 aa)与pAD1 Rep几乎没有一致性,但家族5所有质粒Rep C端具有高度一致性;较大Rep N和C末端之间序列显示了很低的一致性。家族5质粒Rep 3个区域的保守性特征与以往关于RepA_N家族质粒Rep的研究结果是一致的,这些区域特征与其负责的复制起始功能密切相关[15-16]。因此基于Rep序列不同区域的保守性特征,家族5质粒可能都属于theta复制质粒RepA_N家族,并与质粒RepA_N家族质粒具有共同的进化祖先和起源,但分歧进化时间可能比较久远。
2.2.5 编码Rep家族6质粒系统进化关系: 经BLAST比对分析发现,质粒Lp16G Rep与众多植物乳杆菌(大于90%)、其他乳杆菌和片球菌(40%-45%)和链球菌(30%-40%)质粒Rep具有一致性,这些同源Rep均含有一个保守的涉及质粒复制起始的PriCT-1结构域(pfam08708),未出现其他菌属同源Rep。家族6质粒同源性Rep系统进化树见图 2-E。由图 2-E和BLAST分析可知,家族6质粒之间的系统进化关系非常紧密,它们与植物乳杆菌、其他乳杆菌和片球菌、链球菌质粒的进化关系依次渐远,同时这些同源性质粒的进化关系与其宿主菌的系统发育关系一致,这表明家族6质粒与宿主菌具有明显的共进化特征。因此基于Rep中保守结构域的存在,家族6质粒很可能属于theta复制质粒pIP501家族,并与pIP501家族质粒具有共同的进化祖先和起源。
2.3 植物乳杆菌质粒的系统进化关系 为了全面揭示植物乳杆菌天然质粒的系统进化关系,利用Mauve软件对所有75个质粒进行了基因组共线性分析,可视化结果见图 3。质粒最小复制子是质粒基因组中最保守和必不可少的区域,最小复制子序列的微小突变即可造成质粒宿主范围的改变[17],因此本文在划分进化家族时,将是否具有同源的最小复制子作为优先原则。由图 3可知,植物乳杆菌质粒基因组中存在着大量的同源性区域(相同颜色的区块)和非同源性区域(两条垂直红线之间的空白区域)。同颜色区块表示两个区域之间至少发生过一次进化事件,同颜色区块之间序列一致性越低,表示发生的进化事件越复杂,进化关系越远,反之亦然。空白区块表示该区域与所有分析的其他质粒没有发生过进化事件,具有相对独立的进化过程和起源。因此根据质粒基因组共线性分析结果,可以清晰地反映出这些质粒之间发生的进化事件和故事情节,从而有效地揭示它们之间的系统进化关系。基于质粒基因组共线性分析结果和质粒最小复制子优先原则,植物乳杆菌所有75个质粒可以划分为6个进化关系密切的家族、2个特殊进化阶段的杂合质粒和1个独立进化质粒p2140 (图 3)。由于质粒p2140与所有分析质粒没有发现同源性区域,因此它很可能具有独立的起源和进化过程。杂合质粒pMRI5.2含有家族1和家族2质粒的复制子区域,并与家族1类群3质粒和家族2类群2质粒pPB1的基因组显示了高度同源性,因此质粒pMRI5.2很可能由家族1类群3质粒与家族2类群2质粒pPB1重组形成。杂合质粒pLP12-1不仅含有家族5质粒高同源的Rep和家族5质粒(除了LBPp5)共有的接合相关基因(下文2.3.5有具体论述),而且含有家族6质粒高同源的Rep,因此质粒pLP12-1很可能是家族5和家族6来源的两个质粒重组融合形成。
 |
| 图 3 植物乳杆菌质粒基因组共线性可视化图 Figure 3 Visualization diagram of plasmid genomic collinearity in Lactobacillus plantarum. |
| 图选项 |
2.3.1 植物乳杆菌家族1质粒系统进化关系: 经质粒基因组共线性分析,家族1含有14个质粒,均为编码Rep的RCR小型质粒,可以划分为3个进化关系更近的亚家族类群(图 3)。类群1质粒均为隐蔽性质粒,其基因组仅含有复制必需的复制子区域,具有高度的同源性和共线性。与类群1质粒比较,类群2质粒pR18额外编码了一个林肯酰胺抗性(lincosamide nucleotidyltransferase,linA)基因,该基因被报道与葡萄球菌多个质粒携带的linA基因高度一致[18]。与类群1质粒比较,类群3质粒额外编码了转移(mobilization,mob)基因和转移起点(origin of transfer,oriT),因此均为可移动质粒。进一步序列比对分析发现,家族1质粒均含有pC194家族质粒同源的复制子序列,该序列由rep、双链起点(double-stranded origin,dso)和单链起点(single-stranded origin,sso)序列组成。基于上述分析,家族1质粒之间具有十分亲密的进化关系,大部分质粒为隐蔽性质粒,只有少数(类群2和类群3)质粒发生了外源基因插入事件,因此家族1质粒很可能具有共同的进化祖先和起源。
2.3.2 植物乳杆菌家族2质粒系统进化关系: 经质粒基因组共线性分析,家族2含有6个质粒,均为编码Rep的RCR小型质粒,可以划分为3个进化关系更近的亚家族类群(图 3)。类群1质粒pLPI8为隐蔽性质粒,除了复制子没有编码其他基因。类群2质粒在复制子区域外编码了mob和/或未知基因,因此推测它们属于可移动质粒。类群3质粒pLFE1在类群2质粒框架基础上额外编码了一个红霉素抗性基因,有报道称该基因很可能来自屎肠球菌[19]。家族2质粒均含有pMV158家族质粒同源的复制子序列,该序列由拷贝数调控(copy number control,copG)、rep、dso和sso序列组成。基于上述分析,家族2质粒之间同样具有相当亲密的进化关系,因此家族2质粒也很可能具有共同的进化祖先和起源,但与家族1质粒比较,它们含有较多的非同源性区域,这提示它们经历了更复杂的进化事件和过程。
2.3.3 植物乳杆菌家族3质粒系统进化关系: 经质粒基因组共线性分析,家族3含有10个质粒,包括1个编码Rep质粒pG6302和9个非编码Rep质粒。根据基因组共线性特征和复制子序列,家族3质粒可以划分为2个进化关系更近的亚家族类群(图 3)。以往关于家族3质粒复制子的研究十分有限,目前只对质粒p256进行了初步研究,其最小复制子被定位在一个688 bp大小的DNA片段上,该区域没有编码已知的Rep,该质粒没有单链复制中间体,因此被鉴定为theta复制质粒[4]。经基因组共线性和序列比对分析,类群1质粒LZ206p1是家族3质粒中唯一不含有质粒p256复制子同源性序列的质粒,由于它大部分基因组与类群2质粒pCAUH203高度一致,且没有编码已知的Rep,因此它被归属于家族3。类群2质粒中,质粒p100和pLP9000基因组几乎完全一致,因此它们很可能是植物乳杆菌不同菌株中的同一个质粒;质粒p256与质粒LBPp7部分基因组高度同源,因此质粒LBPp7很可能由质粒p256和另一个未知质粒重组进化形成;上面提到,质粒pCAUH203含有类群1质粒LZ206p1大部分基因组,因此质粒pCAUH203很可能由类群1质粒LZ206p1和一个含有p256同源复制子的类群2质粒重组融合形成;质粒pG6302是家族3质粒中唯一编码Rep质粒,根据Xi等[20]报道,质粒pG6302可能含有一个由特殊重复子和不完整Rep (Rep_3家族)组成的新复制子,而根据基因组共线性和序列比对发现,质粒pG6302只与家族3质粒具有多个同源性区域,并且与大部分家族3质粒共有一个质粒p256同源性的非编码复制子区域,同时,质粒pG6302与其他家族质粒仅存在很少的同源性区域,更重要的是,Xi等[20]推测的复制子区域与其他74个质粒没有显示出同源性,因此我们推测质粒pG6302归属于家族3,与家族3质粒具有亲密的进化关系。基于上述分析,家族3质粒可能起源于一个含有非编码Rep的theta型复制子的祖先质粒。
2.3.4 植物乳杆菌家族4质粒系统进化关系: 经质粒基因组共线性分析,家族4含有13个质粒,包括1个非编码Rep质粒LZ206p3和12个编码Rep质粒。家族4质粒均含有一个典型的theta型复制pUCL287家族复制子序列,该复制子由ori (3-4个11 bp和3.0-4.5个22 bp正向重复序列)和rep基因组成[21]。根据基因组共线性差异,家族4质粒可以划分为2个进化关系更近的亚家族类群(图 3)。类群1质粒pLJ42是家族4质粒中最小的,除了家族共有的复制子,与家族4其他质粒没有同源性区域,这提示质粒pLJ42很可能经历了一个相对独立的进化过程,因此与家族4其他质粒进化关系较远。类群2质粒LBPp6和Lp16B基因组同源性98.99%,仅在非编码区相差50 bp,这表明它们来源于同一个质粒,并在近期发生过序列丢失或插入事件。质粒LZ206p3是家族4中唯一非编码Rep质粒,但序列分析发现,它同样含有完整的pUCL287家族复制子序列,只是其rep基因发生了移码突变(数据库中注释),导致转录提前终止,因此不能产生完整的Rep。基于上述分析,家族4质粒均含有高度同源的pUCL287家族复制子,这表明该家族质粒很可能具有共同的进化祖先和起源。此外,家族4质粒基因组中存在相当多的非同源性区域(空白区域),类群1质粒pLJ42尤为明显,这表明家族4质粒曾与植物乳杆菌外源基因(组)发生过频繁的水平基因转移事件,经历过复杂的进化事件和过程。
2.3.5 植物乳杆菌家族5质粒系统进化关系: 经质粒基因组共线性分析,家族5含有25个质粒,包括13个非编码Rep质粒和12个编码Rep质粒(属于theta复制RepA_N家族)。除了质粒LBPp5,家族5质粒均含有一个约3000 bp高度同源性共有区域,该区域编码了切口酶(nickase,traA)、2个未知蛋白和oriT,其中traA和oriT是质粒接合转化必需的基因元件。根据基因组共线性差异和复制子序列,家族5质粒可以划分为4个进化关系更近的亚家族类群。类群1唯一质粒LBPp5与家族5其他质粒几乎没有同源性区域,但与家族4质粒pLP-ZJ101具有大范围同源性区域,并缺失质粒pLP-ZJ101复制子(pUCL287家族),含有家族5质粒高度同源的Rep (RepA_N家族),因此尽管质粒LBPp5与家族4质粒pLP-ZJ101发生过大片段的基因重组事件,具有较近的进化关系,但基于复制子优先原则,将它归属于家族5。类群2含有5个质粒(4个编码Rep),除了家族共有序列,它们均含有一个由21个接合转化相关基因组成的18 kb的高同源性区域[22],这表明它们之间具有更加亲密的进化关系。类群3含有5个质粒,均为非编码Rep质粒,除了家族共有序列,它们均含有一个约18 kb的高同源性区域,该区域编码了渗透压调节、DNA合成和无机离子转运等功能蛋白[23],这提示它们之间同样具有更加亲密的进化关系。类群4含有14个质粒(7个编码Rep),除了家族共有序列,类群4质粒之间含有众多同源性和非同源性区域,没有发现大范围共有同源性区域,这表明类群4质粒经历了复杂的进化事件和过程,它们之间的进化关系不如类群2和3质粒那么亲密。基于上述分析,家族5质粒之间进化关系不是非常紧密,经历了十分复杂的进化过程,但由于普遍含有接合转移关键基因(traA和oriT),因此它们很可能起源于一个接合质粒祖先。虽然质粒LBPp5与家族5质粒缺乏同源性,但它与家族5的11个质粒编码了高度同源的RepA_N家族Rep,由此推测,质粒LBPp5可能同样起源于一个接合质粒祖先,但在与家族4质粒pLP-ZJ101发生的重组事件中丢失了接合转移基因。
2.3.6 植物乳杆菌家族6质粒系统进化关系: 经质粒基因组共线性分析,家族6含有4个质粒,均为编码Rep的大型质粒,它们均含有典型的theta复制pIP501家族复制子,该区域由rep基因和下游的ori组成。根据基因组共线性差异,家族6质粒可以清晰地划分为2个进化关系更近的亚家族类群(图 3)。类群1唯一质粒pMF1298-1基因组结构与类群2质粒明显不同,除了家族共有的复制子序列,它与类群2质粒几乎没有同源性,但与家族5类群4质粒含有众多同源性区域,并含有家族5质粒3000 bp的共有序列,因此它是一个接合质粒,尽管它与家族5质粒发生过重组进化事件,但基于复制子优先原则,它归属于家族6。类群2质粒均为非接合质粒,除了家族共有的复制子序列,它们均含有一个34 kb的高度同源性区域(大多数基因注释为未知蛋白),因此它们具有非常亲密的进化关系。基于上述分析,家族6类群1和类群2质粒进化关系较远,但鉴于它们共有pIP501家族复制子,家族6质粒很可能具有共同的进化祖先和起源。
2.4 植物乳杆菌天然质粒的起源 为了进一步探究植物乳杆菌质粒的起源,对每个家族质粒的基因组GC含量和最小复制子核心组件进行统计和BLAST分析,结果见图 4和表 4。
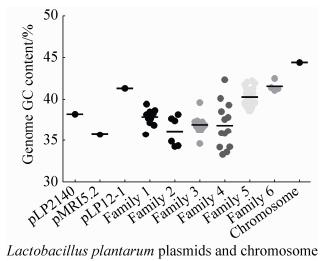 |
| 图 4 植物乳杆菌质粒和染色体基因组GC含量 Figure 4 The genome GC content of Lactobacillus plantarum plasmids and chromosome. Chromosome: The genome GC content of Lactobacillus plantarum WCFS1 chromosome (accession No. NC_004567.2) is 44.45%. |
| 图选项 |
表 4. 植物乳杆菌同源性质粒宿主范围 Table 4. Host range of homologous plasmid in Lactobacillus plantarum
| Classified family | Replication mechanism | Hypothetical family | Target sequence | Host range of homologous plasmid | |
| Gram-positive bacteria | Gram-negative bacteria | ||||
| Family 1 | RCR | pC194 | Rep | Lactobacillus | Acetobacteraceae |
| Pediococcus | Salmonella | ||||
| Weissella | |||||
| Granulicatella | |||||
| Streptococcus | |||||
| Staphylococcus | |||||
| Enterococcus | |||||
| Sporosarcina | |||||
| Listeria | |||||
| Family 2 | RCR | pMV158 | Rep | Lactobacillus | Kosakonia |
| Lactococcus | Escherichia | ||||
| Bacillus | Campylobacter | ||||
| Staphylococcus | Cetobacterium | ||||
| Weissella | Chlamydia | ||||
| Enterococcus | Mycoplasma | ||||
| Lactobacillus | |||||
| Lactococcus | |||||
| Bacillus | |||||
| Staphylococcus | |||||
| Weissella | |||||
| Enterococcus | |||||
| Paenibacillus | |||||
| Streptococcus | |||||
| Leuconostoc | |||||
| Listeria | |||||
| Streptomyces | |||||
| Jeotgalibaca | |||||
| Pediococcus | |||||
| Mycobacterium | |||||
| Aerococcus | |||||
| Carnobacterium | |||||
| Fructobacillus | |||||
| Tetragenococcus | |||||
| Pseudobutyrivibrio | |||||
| Clostrdioide | |||||
| Family 3 | Theta | p256 | Minimal replicon of plasmid p256 (688bp) | Lactobacillus | |
| Pediococcus | |||||
| Family 4 | Theta | pUCL287 | Rep | Lactobacillus | |
| Enterococcus | |||||
| Tetragenococcus | |||||
| Lactococcus | |||||
| Family 5 | Theta | RepA_N | Rep N-terminal (100aa) | Lactobacillus | |
| Pediococcus | |||||
| Oenococcus | |||||
| Tetragenococcus | |||||
| Lactococcus | |||||
| Enterococcus | |||||
| Family 6 | Theta | pIP501 | Rep | Lactobacillus | |
| Pediococcus | |||||
| Streptococcus | |||||
| The family 3 target sequence is determined by reference [3]. The family 5 target sequence is determined by reference [15]. -: No results were retrieved. | |||||
表选项
由图 4可知,家族1-6质粒基因组GC含量具有不同的变化范围和稳定性,家族6的变化最小,家族4的变化范围最大。基因组GC含量越稳定,表明该家族质粒发生水平基因转移事件越少,具有越窄的宿主范围,从而该家族质粒越趋于与宿主发生共同进化。根据Rep系统进化树(图 2E)和基因组GC含量稳定性,家族6质粒很符合这种情况。与此相反,如果基因组GC含量变化范围越大,提示该家族质粒发生水平基因转移事件越频繁,具有越宽泛的宿主范围,因此它们在广泛宿主中发生着更加频繁的水平基因转移事件,从而具有更加复杂的进化过程。根据基因组共线性分析(图 3)和基因组GC含量变化范围,家族4质粒符合这种情况。家族4质粒pLJ42即是很好的例证。Rep系统进化树(图 2-C)和基因组共线性(图 3)分析都表明质粒pLJ42与家族4其他质粒进化关系较远,具有相对独立的进化过程,此外基因组GC含量分析显示,质粒pLJ42基因组CG含量(42.41%)明显高于家族4质粒平均GC含量(36.91%),这一结果进一步证明了上述两种分析方法的结论。
家族1-6质粒基因组平均GC含量分别为37.94%、36.19%、36.98%、36.91%、40.32%和41.62%,均低于植物乳杆菌染色体(44.45%),尤其是家族1-4质粒,这提示家族1-6质粒祖先并非起源于植物乳杆菌,它们很可能起源于染色体GC含量更低的其他宿主菌。杂合质粒pMRI5.2和pLP12-1基因组GC含量分别处于家族1-2和家族5-6之间,符合Rep系统进化树(图 1)和基因组共线性(图 3)分析中关于它们由两个不同家族质粒融合进化的推测,因此它们分别与家族1-2和5-6具有共同的进化祖先和起源。根据质粒大小和基因组GC含量(38.27 %),质粒pLP2140与家族1质粒相似,然而根据基因组共线性分析,它与植物乳杆菌所有分析的其他质粒没有同源性区域,因此质粒pLP2140不属于上述6个家族,可能具有完全独立的进化过程和起源。
由表 4可知,家族1-2(RCR)同源性质粒不仅在厚壁菌门众多革兰氏阳性菌属中出现,而且可以在变形菌门多个革兰氏阴性菌属中生存,这提示家族1-2同源性质粒很可能经历了十分漫长的进化历史,尤其是家族2同源性质粒,从而获得了广泛的宿主范围,因此家族2可能是植物乳杆菌中最古老的家族,最早进入植物乳杆菌。与RCR质粒相比,theta复制家族3-6同源性质粒宿主范围较窄,尤其是家族6同源质粒仅出现在3个亲缘关系较近的宿主菌,并且它们与宿主清晰地显示了共进化现象,因此家族6可能是植物乳杆菌中出现最晚的家族。虽然家族3同源性质粒具有很窄的宿主范围,但由于该家族质粒最小复制子的研究和了解非常有限,只有质粒p256最小复制子可用于BLAST分析,因此家族3同源性质粒的宿主范围可能不限于表 4所列。家族4-5质粒比家族3、6同源性质粒宿主范围更宽,且质粒数量较多,因此家族4-5是植物乳杆菌theta复制质粒的优势家族和古老家族。基于上述分析,家族1-6质粒在不同宿主范围具有同源性质粒,它们在这些宿主范围内具有维持、复制和水平转移的潜力,因此它们与同源性质粒很可能具有共同的进化祖先和起源,而根据基因组GC含量分析,家族1-6质粒很可能不起源于植物乳杆菌,因此家族1-6质粒的祖先和起源很可能来源于这些同源性质粒及其宿主。此外,通过BLAST比对分析,质粒pLP2140全基因组序列仅与1个鼠李糖乳杆菌染色体和1个有害片球菌质粒的101 bp的非编码区域具有同源性,这进一步证明,质粒pLP2140与植物乳杆菌其他质粒的进化和起源具有完全的独立性。
3 讨论 如果两个质粒具有同源性区域,那么这两个质粒具有一定的系统进化关系;如果两个质粒具有同源性(最小)复制子,那么这两个质粒具有共同的进化祖先和起源。本文基于这两个基本假设,通过Rep系统进化树、基因组共线性、基因组GC含量和宿主范围分析方法,准确、有效地揭示了植物乳杆菌天然质粒的系统进化关系和起源,提供了分子(核苷酸和氨基酸序列)水平的佐证和依据,这对植物乳杆菌天然质粒系统进化和起源研究具有重要的参考价值,为今后植物乳杆菌天然质粒进化机制,及质粒与植物乳杆菌(宿主)互作关系研究奠定基础。
通过Rep系统进化树和基因组共线性两种方法分析,植物乳杆菌天然质粒的系统进化关系在家族水平上分析结果完全一致,这有力地证明了两种分析方法的准确性和有效性。比较两种分析方法可知,质粒Rep系统进化树方法可以简单、快速地对编码Rep质粒进行系统进化分析,可以准确划分质粒家族,并清晰地反映质粒家族之间的系统进化关系,但对于非编码Rep质粒无效。质粒基因组共线性方法可以全面、精确地对所有质粒进行系统进化分析,由于它针对质粒的全基因组进行比较分析,因此它可以在家族和亚家族水平对所有分析质粒进行准确、细致的微进化关系分析。然而,当质粒数量较多、基因组共线性较差时,该方法会产生海量的同源性区域信息和数据(本研究未列出),这对后续同源性区域的比对和鉴定造成很大困难,因此该方法进行大量质粒分析时,存在分析过程繁琐、结果数据解析费时费力的缺陷。因此基于上述分析,两种分析方法各有优缺点,Rep系统进化树分析方法更适合在家族水平对大量质粒进行快速的系统进化分析,而质粒基因组共线性分析方法更适合在亚家族水平对较少质粒进行精准的微进化分析。因此将两种方法前后结合使用,可以建立一种准确、快速、全面的质粒系统进化分析方法。此外,这种组合方法不限于编码Rep质粒,对于含有未知复制子的质粒(家族3和家族5部分质粒)同样适用,因此这种组合方法很可能适用于所有细菌天然质粒的系统进化和起源研究,可能具有普遍的方法学意义。
综合本文研究结果,我们可以推测出植物乳杆菌天然质粒大致的演化过程和一些重要进化事件。植物乳杆菌75个天然质粒起源于7个祖先,由于基因组GC含量均低于植物乳杆菌染色体,因此所有质粒的祖先并非起源于植物乳杆菌,它们可能都是通过偶然的水平基因转移事件从染色体GC含量更低的宿主菌进入植物乳杆菌。RCR小型质粒(家族1-2)属于植物乳杆菌质粒家族中的古老家族,经历了漫长的进化时间,从而适应了广泛的宿主范围,因此它们较早进入植物乳杆菌,但由于复制机制限制,基因组很小,只能携带很少的功能基因,因此含有宽宿主范围的小型质粒,对宿主细胞个体短期而言影响非常有限,但对宿主群体长期而言,意味着它们可以频繁地转移远源宿主的功能基因及其表型,促进宿主适应环境变化的能力,从而增强宿主群体的进化优势,同时质粒本身得以生存、延续和传播。theta复制质粒(家族3-6)属于植物乳杆菌天然质粒的年轻家族,家族基因组越大形成越晚,进化历史越短,从而适应的宿主范围越有限,因此这些质粒进入植物乳杆菌的时间较晚。家族6质粒即是典型的例证。家族6质粒Rep分化程度(图 1)、基因组GC含量(图 4)和宿主范围(表 4)分析均表明,家族6质粒是最后进入植物乳杆菌的家族。家族6质粒基因组巨大,可以携带多种多样的功能基因,由于形成和进化时间很短,它们通常不能跨越“物种”发生水平基因转移,因此家族6质粒与宿主表现出明显的共进化现象。然而,一旦成功发生“物种”间转移事件,新宿主可以一次性获得大量的外源基因及其多种表型,在生境发生大的变化时,更能体现进化优势,同时大质粒本身得以生存、延续和传播。基于前文分析,家族4-5比家族3、6质粒发生了更加频繁的水平基因转移,但基因组功能分析显示(结果未列出),家族4-5质粒的进化动力机制不同,家族4质粒主要依赖于转座,家族5质粒转座和接合机制共存,这表明家族5比家族4质粒可以更加频繁地进行宿主转移,宿主范围分析也支持了这个观点(表 4),因此含有家族5质粒的宿主可以获得更大的进化优势。综上所述,每个家族质粒在不同外界环境选择压力下对植物乳杆菌(宿主)个体生理学和群体生态学意味着不同的进化意义,但不论从质粒还是宿主角度分析,获得生存与进化优势或许是它们统一且最终的目标[24],因此如此丰富且多样的天然质粒很可能在植物乳杆菌生长、发育、遗传和进化过程中扮演着非常重要的角色。
References
| [1] | Sun ZH, Harris HMB, McCann A, Guo CY, Argimón S, Zhang WY, Yang XW, Jeffery LB, Cooney JC, Kagawa TF, Liu WJ, Song YQ, Salvetti E, Wrobel A, Rasinkangas P, Parkhill J, Rea MC, O'Sullivan O, Ritari J, Douillard FP, Paul Ross R, Yang RF, Briner AE, Felis GE, de Vos WM, Barrangou R, Klaenhammer TR, Caufield PW, Cui YJ, Zhang HP, O'Toole PW. Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera. Nature Communications, 2015, 6: 8322. DOI:10.1038/ncomms9322 |
| [2] | Siezen RJ, van Hylckama Vlieg JET. Genomic diversity and versatility of Lactobacillus plantarum, a natural metabolic engineer. Microbial Cell Factories, 2011, 10(Suppl 1): S3. DOI:10.1186/1475-2859-10-S1-S3 |
| [3] | Sun DQ, Li HF, Song DW, Yang J, Zhang DJ, Xu XX. Progress in research on natural plasmids in Lactobacillus. Food Science, 2015, 36(11): 251-255. (in Chinese) 孙大庆, 李洪飞, 宋大巍, 杨健, 张东杰, 许晓曦. 乳杆菌属天然质粒研究进展. 食品科学, 2015, 36(11): 251-255. DOI:10.7506/spkx1002-6630-201511047 |
| [4] | S?rvig E, Skaugen M, Naterstad K, Eijsink VGH, Axelsson L. Plasmid p256 from Lactobacillus plantarum represents a new type of replicon in lactic acid bacteria, and contains a toxin-antitoxin-like plasmid maintenance system. Microbiology, 2005, 151(2): 421-431. DOI:10.1099/mic.0.27389-0 |
| [5] | Yin S, Hao YL, Zhai ZY, Zhang W, Zhou H, Wang GH, Shi XL, Luo YB. Functional analysis of the plasmid pM4 replicon from Lactobacillus plantarum M4: determination of the minimal replicon and functionality identification of the putative sso. Plasmid, 2009, 62(3): 166-171. DOI:10.1016/j.plasmid.2009.07.004 |
| [6] | Sun ZL, Kong J, Kong WT. Characterization of a cryptic plasmid pD403 from Lactobacillus plantarum and construction of shuttle vectors based on its replicon. Molecular Biotechnology, 2010, 45(1): 24-33. DOI:10.1007/s12033-010-9242-0 |
| [7] | Rattanachaikunsopon P, Phumkhachorn P. Construction of a food-grade cloning vector for Lactobacillus plantarum and its utilization in a food model. The Journal of General and Applied Microbiology, 2012, 58(4): 317-324. DOI:10.2323/jgam.58.317 |
| [8] | de las Rivas B, Marcobal A, Mu?oz R. Complete nucleotide sequence and structural organization of pPB1, a small Lactobacillus plantarum cryptic plasmid that originated by modular exchange. Plasmid, 2004, 52(3): 203-211. DOI:10.1016/j.plasmid.2004.09.001 |
| [9] | Francia MV, Haas W, Wirth R, Samberger E, Muscholl-Silberhorn A, Gilmore MS, Ike Y, Weaver KE, An FY, Clewell DB. Completion of the nucleotide sequence of the Enterococcus faecalis conjugative virulence plasmid pAD1 and identification of a second transfer origin. Plasmid, 2001, 46(2): 117-127. DOI:10.1006/plas.2001.1533 |
| [10] | Thompson JK, Collins MA. Completed sequence of plasmid pIP501 and origin of spontaneous deletion derivatives. Plasmid, 2003, 50(1): 28-35. DOI:10.1016/S0147-619X(03)00042-8 |
| [11] | Cho GS, Huch M, Mathara JM, van Belkum MJ, Franz CMAP. Characterization of pMRI 5.2, a rolling-circle-type plasmid from Lactobacillus plantarum BFE 5092 which harbours two different replication initiation genes. Plasmid, 2013, 69(2): 160-171. DOI:10.1016/j.plasmid.2012.11.005 |
| [12] | Khan SA. Plasmid rolling-circle replication: highlights of two decades of research. Plasmid, 2005, 53(2): 126-136. DOI:10.1016/j.plasmid.2004.12.008 |
| [13] | Moscoso M, del Solar G, Espinosa M. Specific nicking-closing activity of the initiator of replication protein RepB of plasmid pMV158 on supercoiled or single-stranded DNA. Journal of Biological Chemistry, 1995, 270(8): 3772-3779. DOI:10.1074/jbc.270.8.3772 |
| [14] | Benachour A, Frère J, Novel G. pUCL287 plasmid from Tetragenococcus halophila (Pediococcus halophilus) ATCC 33315 represents a new theta-type replicon family of lactic acid bacteria. FEMS Microbiology Letters, 1995, 128(2): 167-175. DOI:10.1111/fml.1995.128.issue-2 |
| [15] | Weaver KE, Kwong SM, Firth N, Francia MV. The RepA_N replicons of Gram-positive bacteria: a family of broadly distributed but narrow host range plasmids. Plasmid, 2009, 61(2): 94-109. DOI:10.1016/j.plasmid.2008.11.004 |
| [16] | Schumacher MA, Tonthat NK, Kwong SM, Chinnam NB, Liu MA, Skurray RA, Firth N. Mechanism of staphylococcal multiresistance plasmid replication origin assembly by the RepA protein. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25): 9121-9126. DOI:10.1073/pnas.1406065111 |
| [17] | Sota M, Yano H, Hughes JM, Daughdrill GW, Abdo Z, Forney LJ, Top EM. Shifts in the host range of a promiscuous plasmid through parallel evolution of its replication initiation protein. The ISME Journal, 2010, 4(12): 1568-1580. DOI:10.1038/ismej.2010.72 |
| [18] | Jalilsood T, Baradaran A, Ling FH, Mustafa S, Yusof K, Rahim RA. Characterization of pR18, a novel rolling-circle replication plasmid from Lactobacillus plantarum. Plasmid, 2014, 73: 1-9. DOI:10.1016/j.plasmid.2014.04.004 |
| [19] | Feld L, Bielak E, Hammer K, Wilcks A. Characterization of a small erythromycin resistance plasmid pLFE1 from the food-isolate Lactobacillus plantarum M345. Plasmid, 2009, 61(3): 159-170. DOI:10.1016/j.plasmid.2009.01.002 |
| [20] | Xi XD, Fan J, Hou Y, Gu JH, Shen WJ, Li ZK, Cui ZL. Characterization of three cryptic plasmids from Lactobacillus plantarum G63 that was isolated from Chinese pickle. Plasmid, 2013, 70(3): 321-328. DOI:10.1016/j.plasmid.2013.07.004 |
| [21] | Benachour A, Frère J, Flahaut S, Novel G, Auffray Y. Molecular analysis of the replication region of the theta-replicating plasmid pUCL287 from Tetragenococcus (Pediococcus) halophilus ATCC33315. Molecular and General Genetics MGG, 1997, 255(5): 504-513. DOI:10.1007/s004380050523 |
| [22] | van Kranenburg R, Golic N, Bongers R, Leer RJ, de Vos WM, Siezen RJ, Kleerebezem M. Functional analysis of three plasmids from Lactobacillus plantarum. Applied and Environmental Microbiology, 2005, 71(3): 1223-1230. DOI:10.1128/AEM.71.3.1223-1230.2005 |
| [23] | Chen C, Ai LZ, Zhou FF, Ren J, Sun KJ, Zhang H, Chen W, Guo BH. Complete nucleotide sequence of plasmid pST-Ⅲ from Lactobacillus plantarum ST-Ⅲ. Plasmid, 2012, 67(3): 236-244. DOI:10.1016/j.plasmid.2011.12.005 |
| [24] | Kado CI. Origin and evolution of plasmids. Antonie van Leeuwenhoek, 1998, 73(1): 117-126. DOI:10.1023/A:1000652513822 |
