辛玉峰1, 赵天颖2, 曲晓华1


1.曲阜师范大学生命科学学院, 山东 曲阜 273165;
2.兰州大学萃英学院, 甘肃 兰州 730030
收稿日期:2016-09-01;修回日期:2016-11-03;网络出版日期:2016-11-29
基金项目:山东省自然科学基金青年基金(ZR2012CQ011);曲阜师范大学实验教改项目(sj201411)
*通信作者:曲晓华, Tel: +86-537-4458576; E-mail: sduxyf@126.com
摘要: [目的]为了体现亚硝酸盐还原酶在环境中氮生物循环的重要性,研究了它们的分布情况。[方法]利用现有亚硝酸盐还原酶序列在已经测序的基因组数据库中进行查找,研究该酶的分布情况,通过多序列比对比较了它们的序列相似性,通过构建系统发育树比较其进化关系,并利用宏基因组学的方法研究了它们在海洋宏基因组中的分布。[结果]分析结果显示,两类亚硝酸盐还原酶在已测序的细菌和古生菌基因组中分别有397和812个,分别占总量的8%和15.7%,几乎所有的古生菌都含有Ⅱ类酶;它们自身的序列相似性很高,在Ⅰ类酶和Ⅱ类酶中底物结合位点以及Ⅱ类酶的铜离子结合位点保守性都很高,显示该酶序列保守性与其环境功能相适应的特点;进化分析显示它们可能具有共同的进化来源;在海洋宏基因组中,平均每100000读数中分别有6个Ⅰ类和35个Ⅱ类,且2类酶都在热带南太平洋区域有最大分布。[结论]NIR在氮的生物循环及环境修复中可能起到重要作用。
关键词: 细菌 古生菌 亚硝酸盐还原酶 血红素 铜 序列比对 海洋 宏基因组学
Distribution, structure and sequence alignment, and metagenomics analysis of two nitrite reductases with NO forming
Yufeng Xin1, Tianying Zhao2, Xiaohua Qu1


1.College of Life Sciences, Qufu Normal University, Qufu 273165, Shandong Province, China;
2.Cuiying College of Lanzhou University, Lanzhou 730030, Gansu Province, China
Received 01 September 2016; Revised 03 November 2016; Published online 29 November 2016
*Corresponding author: Xiaohua Qu, Tel: +86-537-4458576; E-mail: sduxyf@126.com
Supported by the Natural Science Foundation of Shandong Province (ZR2012CQ011) and by the Experimental Innovation Project of Qufu Normal University (sj201411)
Abstract: [Objective]To reflect the importance of nitrite reductase (NIR) in the environment, we studied its distribution.[Methods]The sequences of NIR were searched in the sequenced genome database at NCBI based on previous reported NIR sequences. The sequence similarity was done by multiple sequence alignment, and phylogenetic relationship was evaluated via constructing the phylogenetic tree. Furthermore, their distribution in the marine metagenome was studied by metagenomics.[Results]The homologues of these two enzymes were 397 and 812 strains in sequenced genome, and the proportion was 8 and 15.7 percent, respectively. Almost all of archaea containing type Ⅱ NIR. They have high identity by multiple sequence alignment analysis. The cofactor, the substrate and the cooper binding sites in type Ⅱ were high conserved, suggesting that these enzymes had the specific function in denitrification. Phylogenetic analysis showed the two enzymes may have the common ancestor. In marine metagenome analysis, type Ⅰ and Ⅱ have 6 and 35 reads per 100000 reads, respectively, the two types of NIRs have the biggest proportion at the tropical south pacific area.[Conclusion]Collectively, we suggested NIR, especially type Ⅱ, play a key role in bioremediation of nitrogen contamination.
Key words: bacteria archaea nitrite reductase heme copper sequence alignment marine metagenomics
氮的地球化学循环包括氮的固定和释放。氮固定是指大气中的氮被固定成氨的形式,从而被生物所利用;而氮释放指氨或亚硝酸盐或硝酸盐被反硝化为气态的氮而重新回到大气中。一般来说,这两者是平衡的关系。但是,由于人类的活动,例如氮肥的使用以及固氮技术的提高,使得氮固定的比例远大于氮释放,据报道近年来输入到环境中的氮量,已经是回复到大气中氮量的2倍,而且还在不断增加,这样就造成了环境中的氮营养过剩[1]。过度的营养过剩会造成各种富营养环境,不利于生物地球化学循环,因此,以微生物反硝化作用为主的氮释放成为人类解决这种氮不平衡造成的环境问题的一个重要手段。
亚硝酸盐还原酶 (Nitrite reductase,NIR),是反硝化过程中一个关键的酶,也是该过程中第1个将亚硝酸盐转化产生气态氮 (NO) 的酶 (EC 1.7.1.2)。自然界中,该酶主要有3种类型,类型Ⅰ是由nirS编码的,类型Ⅱ是由nirK编码的,两者行使的功能类似,但是所需要的辅因子不同,前者是由细胞色素cd1组成,而后者则包含铜离子作为辅因子;类型Ⅲ的NIR,是一种包含1个[4Fe-4S]簇和1个西罗血红素 (siroheme) 作为辅因子的NIR,与前两类相区别的是Ⅲ类催化亚硝酸盐还原的终产物是氨 (Ammonia),而NO只是其中的中间产物[2],因此,Ⅲ类NIR不生成NO作为终产物,不属于本文的研究范围。目前分离到的酶中,据报道约有3/4为类型Ⅰ[3]。
第1类由nirS编码的亚硝酸盐还原酶 (Ⅰ类),其活性形式一般以同源二聚体 (homodimer) 的形式存在,每一个亚基包含1个c型和d1型的血红素,每个亚基约为60 kDa左右。光谱学和功能学证据表明,c型的血红素负责底物的进入,而起到催化功能的则主要是d1型的血红素[4]。关于该酶的电子供体一直存在争议,有研究指出在Pseudomonas aeruginosa中,只有细胞色素c551起到作用[5],但另外一些研究指出天青蛋白 (Azurin) 也可与该酶发生相互作用[6]。
第2类是由nirK编码的以铜离子为辅因子的酶,于2001年在古生菌Haloarcula marismortui中被纯化[7](Ⅱ类)。该类酶包括2个亚单位,估计分子量分别为46 kDa和42 kDa。有趣的是,这2个亚基都是由相同的nirK基因编码,不同的是小亚基失去了氮端的16个氨基酸。同Ⅰ类酶类似,Ⅱ类酶的生理电子供体至今仍不清楚。
两类亚硝酸盐还原酶都催化相同的反应 (公式1),该反应的逆反应也可以发生,但是比较困难。
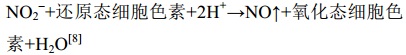 | 公式(1) |
1 材料和方法 1.1 目标氨基酸序列在NCBI数据库中的查找 在细菌和古生菌中,分别利用两类酶的初始种子氨基酸序列[Pseudomonas aeruginosa PAO1 (NP_249210.1) 和Haloarcula marismortui (WP_011224471.1)],在NCBI非冗余数据库中,以各门 (phyla) 作为搜索的限制条件,运行BLASTP程序,初步查找符合e–value≤1e–10,覆盖度 (Coverage)≥50%,并且一致性 (Identity)≥30%等3个条件的序列。将初步筛选出来的序列通过人工筛选出符合条件的匹配序列,将多余的序列去掉,找到匹配的菌株。在多序列比对,系统发育树构建以及海洋宏基因组学分析中均使用这一部分序列作为种子序列 (Seed sequences)。
1.2 多序列比对 将方法1.1中选取的氨基酸序列以FASTA格式存储于TXT文件中,利用Clustal X 2.1软件进行全局序列比对 (global alignment)[9]。利用网页软件Boxshade进行比对序列的后期着色和整理 (http://embnet.vital-it.ch/software/BOX_form.html)。
1.3 系统发育树构建 将方法1.2中进行序列比对后的文件,利用MEGA 7.0软件进行系统发育树的构建。MEGA运行的规则:配对删除 (pairwise deletion),p距离分布 (p-distance distribution),1000次重复的自展分析 (bootstrap analysis)。
1.4 宏基因组学分析 利用方法1.1中查找到的两类酶的所有同源氨基酸序列为种子,在海洋基因组数据库Global Ocean Sampling (GOS) expeditions database[10]中利用BLASTP程序查找匹配数据,查找标准为符合e–value≤1e–5的序列,将所有序列的ID与样品的ID进行对应,查找到目的序列所在的位置。计算匹配序列的含量是利用匹配序列的数量除以总序列数量。具体细节:利用Pseudomonas aeruginosa PAO1 (NP_249210.1) 和Haloarcula marismortui (WP_011224471.1) 作为种子序列,在UniProt (http://www.uniprot.org/) 中寻找与其初步匹配的序列,调取其蛋白质序号。通过建立批处理在已经下载的GOS基因组数据库中进行BlastP寻找目的序列。为了删除重复的序列,每个蛋白的PEP号与它的JCVI模板号以及mate-read号码被提取,通过EXCEL的筛选功能去除重复的序列。包含目的蛋白的菌株出现的频率通过已经报道的方法计算[11]。选择出的氨基酸序列通过建立初步的系统发育树做进一步人工筛选。
2 结果和分析 2.1 两类亚硝酸盐还原酶在NCBI数据库中的分布 利用Pseudomonas aeruginosa PAO1的NIR (NP_249210.1),在NCBI中进行结构域分析,分析结果显示,Ⅰ类NIR由2个结构域组成,一个是CccA,编码细胞色素c;另一个是细胞色素d1血红素结构域。利用Haloarcula marismortui (WP_011224471.1) 为种子序列分析Ⅱ类酶,结构域分析结果显示,它由2个相同的Cupredoxin结构域组成,以铜离子 (Copper) 为辅因子。
在NCBI截止到2016年4月6日的数据库中,已经测序的细菌和古生菌的基因组分别有4929个和242个。利用BLASTP程序进行了广泛的查找,对于两类NIR的分布进行了初步的研究。发现Ⅰ类酶主要存在于细菌中 (397个,约占细菌的8%),Ⅱ类酶在细菌 (595个,约占细菌的12%) 和古生菌 (217个,约占古生菌的90%) 中均有分布。其中Ⅱ类酶在细菌和古生菌的总和中约占15.7%(表 1和表 2)。
表 1. 两类酶在古生菌中的分布 Table 1. Hit organisms of two kinds of NIRs in archaea
| Phylum | Ⅰ | Ⅱ | Sequenced genomes |
| Korarchaeota | 0 | 0 | 1 |
| Nanoarchaeota | 0 | 0 | 2 |
| Thaumarchaeota | 0 | 43 | 43 |
| Crenarchaeota | 0 | 69 | 69 |
| Euryarchaeota | 0 | 105 | 124 |
| Others | 0 | 0 | 3 |
| Total | 0 | 217 | 242 |
表选项
表 2. 两类酶在真细菌中的分布 Table 2. Hit organisms of two kinds of NIRs in bacteria
| Phylum | Ⅰ | Ⅱ | Sequenced genomes |
| Acidobacteria | 0 | 1 | 8 |
| Actinobacteria | 0 | 1 | 531 |
| Aquificae | 10 | 0 | 16 |
| Bacteroidetes | 2 | 120 | 185 |
| Chlamydiae | 0 | 0 | 108 |
| Chlorobi | 0 | 1 | 11 |
| Chloroflexi | 10 | 15 | 25 |
| Chrysiogenetes | 0 | 0 | 1 |
| Cyanobacteria | 0 | 2 | 91 |
| Deferribacteres | 0 | 0 | 4 |
| Deinococcus-Thermus | 12 | 3 | 24 |
| Dictyoglomi | 0 | 0 | 2 |
| Fibrobacteres | 0 | 0 | 2 |
| Firmicutes | 1 | 95 | 1086 |
| Fusobacteria | 0 | 0 | 22 |
| Gemmatimonadetes | 0 | 2 | 2 |
| Nitrospirae | 3 | 20 | 23 |
| Planctomycetes | 2 | 2 | 6 |
| Poribacteria | 0 | 0 | 0 |
| Proteobacteria | 346 | 292 | 2491 |
| Spirochaetes | 1 | 14 | 60 |
| Tenericutes | 0 | 0 | 140 |
| Thermodesulfobacteria | 0 | 0 | 4 |
| Thermotogae | 0 | 0 | 29 |
| Verrucomicrobia | 0 | 10 | 10 |
| Others | 0 | 0 | 48 |
| Total | 387 | 578 | 4929 |
表选项
具体来看,Ⅰ类酶在细菌中主要分布在变形菌门 (proteobacteria) 中,这结果与蔡小龙等揭示的参与氮循环的微生物群落结构相一致[12],由于变形菌门数量基本上占整个细菌总数的50%,我们具体研究了Ⅰ类酶在变形菌门中各纲的分布,结果如表 3,在目前已知的变形菌门的5个纲 (α,β,γ,δ,ε) 中,Ⅰ类酶主要分布在γ-和β-变形菌门中,其中约占已测序γ-变形菌门基因组的16.7%,而占已测序β-变形菌门的27.7%,分别以Pseudomonas和Cupriavidus属为代表。在古生菌中未发现Ⅰ类的NIR酶。另外,在产水菌门 (Aquificae)、绿弯菌门 (Chloroflexi) 和异常球菌-栖热菌门 (Deinococcus-Thermus) 中,Ⅰ型NIR均有分布,约占各门中已测序基因组数量的1/2(表 2)。
表 3. Ⅰ类NIR在变形菌门各纲中的分布 Table 3. Distribution of type Ⅰ NIRs in every subgroups of proteobacteria
| Phylum | Alpha- | Beta- | Gamma- | Delta- | Epsilon- | total |
| Sequenced genomes | 400 | 390 | 1390 | 72 | 239 | 2491 |
| Type Ⅰ NIR | 0 | 108 | 232 | 1 | 5 | 346 |
| (NIR/Sequenced)/% | 0 | 27.7 | 16.7 | 1.4 | 2.1 | 13.9 |
表选项
Ⅱ类酶在细菌中同样广泛存在,其数量要高于Ⅰ类酶。同Ⅰ类酶一样,在变形菌门中的分布最多 (292个种),在变形菌门各纲中的分布与Ⅰ型类似 (结果未展示)。除在细菌中存在以外,Ⅱ类酶在古生菌中也有相当多的分布 (217个种),有约一半的是在广古菌门 (Euryarchaeota) 中 (105个种),由于已经测序的古生菌有242种,因此可以认为,几乎所有的古生菌都含有Ⅱ类酶。另外,在细菌中拟杆菌门 (Bacteroidetes)、绿弯菌门、厚壁菌门 (Firmicutes)、硝化螺旋菌门 (Nitrospirae)、螺旋体门 (Spirochaetes) 及疣微菌门 (Verrucomicrobia) 中有比较多的Ⅱ型NIR,有些几乎占100% (硝化螺旋菌门和疣微菌门中)(表 2)。
有趣的是,除古生菌中不含有Ⅰ类酶以外,在细菌中,含有Ⅰ类和Ⅱ类酶的细菌种类大致相同,例如在衣原体门 (Chlamydiae),脱铁杆菌门 (Deferribacteres),梭杆菌门 (Fusobacteria),网团菌门 (Dictyoglomi) 以及海绵杆菌门 (Poribacteria) 中均不含有这两类酶,而在绿弯菌门,热微菌门 (Thermomicrobia),硝化螺旋菌门,浮霉菌门 (Planctomycetes) 以及变形菌门中均含有这两类酶。
不同的细菌具有不同的生存环境,以上结果综合起来可能说明,这两类酶的存在是与生存环境更相关,而与具体物种的相关性较小。
2.2 多序列比对及结构保守性分析 通过蛋白质的多序列比对,发现两类酶与自身比较时的序列相似程度均很高 (图 1和图 2)。Ⅰ类中的酶多数具有69%以上的氨基酸序列相似性,而Ⅱ类具有60%以上的相似性。高的序列相似性预示着该酶具有相对高的功能保守性。
 |
| 图 1. Ⅰ类亚硝酸盐还原酶的氨基酸序列比对 Figure 1. Multiple sequence alignment of type Ⅰ NIR (Accession number: Pseudomonas, WP_003111532.1; Cupriavidus, AAZ64358.1; Deltaproteobacteria, OGQ63959.1; Arcobacter, WP_066402611.1). |
| 图选项 |
 |
| 图 2. Ⅱ类亚硝酸盐还原酶的氨基酸序列比对 Figure 2. Multiple sequence alignment of type Ⅱ NIR (Accession number: Haloarcula, WP_011224471.1; Salisaeta, WP_051141251.1; Acidiferrobacter, WP_065969876.1; Thermaerobacter, WP_006903597.1; Nitrososphaera, AIC14236.1). |
| 图选项 |
在Ⅰ类酶中,已经有许多酶被解析了结构,如Pseudomonas aeruginosa (PDB:1NNO)[13],该酶的2个结构域通过一段连接序列 (linker) 相连,较小的c结构域为底物的进入提供帮助,而真正起到催化作用的则是d1结构域,d1结构域以α螺旋和β折叠相互交叉的方式构成桶状结构,而该桶状结构是催化位点所在区。该酶相似程度较高且较为保守的一段区域 (350–490位氨基酸,1NNO) 与桶状结构的形成有较大的重叠性,这说明这一段序列对于该酶的功能保守性至关重要。另外,72和75位的半胱氨酸 (cysteine) 是该酶与血红素 (heme) 结合的关键位点,76位组氨酸 (histidine),113位甲硫氨酸 (methionine) 以及207位组氨酸是其结合Fe2+的关键位点,从序列比对结果看出这些位点在所有的Ⅰ类酶中是绝对保守的。352位和393位组氨酸是负责底物亚硝酸盐结合的位点[14],在所有的Ⅰ类酶中也是绝对保守的 (图 1)。以上结果说明Ⅰ类酶的底物以及辅因子结合位点均是其功能必需的。
Achromobacter cycloclastes中的Ⅱ类NIR酶 (PDB:1NIC,2BWD) 已经被解析了结构[15-16]。多个研究表明,铜的结合位点主要有2个,铜1负责将外部的电子转移给铜2,而铜2是主要的催化位点[17]。在多序列比对时 (图 2),除铜1中174位组氨酸 (Haloarcula marismortui) 有1个可能的半胱氨酸 (cysteine) 突变,其他的位点 (133位,182位组氨酸,187位甲硫氨酸在铜1中;138位,173位及332位组氨酸在铜2中) 均相对保守,说明这些位点对于Ⅱ类酶的生理功能非常重要。
综合上述结果,两类NIR的底物与辅因子结合位点都是非常保守的,这说明该酶的生理功能专一性较强,且在环境氮循环中的作用不能被替代。
2.3 系统进化关系 通过9个典型的氨基酸序列,初步构建了Ⅰ和Ⅱ类酶的系统发育树 (图 3),可以初步说明两类酶的进化关系。从整体上看,Ⅰ类和Ⅱ类之间亲缘关系较远。Ⅰ类中Pseudomonas与Cupravidus亲缘关系更近;Ⅱ类中Haloarcula与Salisaeta亲缘关系更近。就离散关系来说,Ⅰ类酶的进化距离更小,亲缘关系普遍更近;而Ⅱ类则相差较远,这可能是与Ⅱ类酶在细菌和古生菌中均有分布有关系。一般来说,异养细菌要比自养细菌进化更晚,而细菌则与古生菌具有相对平行的进化关系,因此,NIR酶在自养菌、异养菌以及古生菌中都普遍存在的情况,说明该酶可能具有从自养菌或古生菌进化至异养细菌的趋势。
 |
| 图 3. 亚硝酸盐还原酶的系统进化树 Figure 3. Phylogenetic tree of type Ⅰ and Ⅱ NIR. The accession numbers are same as shown in figure 1 and 2. |
| 图选项 |
2.4 NIR在海洋宏基因组中的分布 利用现有的海洋宏基因组GOS,分析这两类酶在宏基因组中的分布情况,以此来评价自然界中这两类酶的存在情况。尽管在现有分离到的酶中,Ⅰ类酶约占3/4,但从宏基因组分析的结果来看却并非如此,Ⅱ类酶在海洋中的数量要远大于Ⅰ类酶 (表 4)。从表 4中可以看出,每10万个读数 (reads) 中,平均Ⅱ类NIR (35个) 在海洋环境中存在的数量要远远大于Ⅰ类 (6个)。这说明:首先,分离的酶可能只限于实验条件下易于分离的酶,不代表真实情况;其次,Ⅱ类酶在宏基因组中如此广泛的分布,表明该酶在海洋氮循环中起到更重要的作用。
表 4. 亚硝酸盐还原酶的海洋宏基因组分析 Table 4. Metagenomics analysis of NIR
| Sample ID | Sample regions | description | Ⅰ NIR* | Ⅱ NIR* | |
| JCVI_SMPL_1103283000001 | Sargasso Sea | Open Ocean | 15 | 44 | |
| JCVI_SMPL_1103283000002 | Sargasso Sea | Open Ocean | 7 | 31 | |
| JCVI_SMPL_1103283000003 | Sargasso Sea | Open Ocean | 10 | 57 | |
| JCVI_SMPL_1103283000004 | Sargasso Sea | Open Ocean | 8 | 47 | |
| JCVI_SMPL_1103283000005 | Sargasso Sea | Open Ocean | 1 | 11 | |
| JCVI_SMPL_1103283000006 | Sargasso Sea | Open Ocean | 9 | 30 | |
| JCVI_SMPL_1103283000007 | Sargasso Sea | Open Ocean | 11 | 48 | |
| JCVI_SMPL_1103283000008 | North American East Coast | Coastal | 7 | 39 | |
| JCVI_SMPL_1103283000009 | North American East Coast | Coastal | 10 | 44 | |
| JCVI_SMPL_1103283000010 | North American East Coast | Coastal | 9 | 59 | |
| JCVI_SMPL_1103283000011 | North American East Coast | Embayment | 5 | 43 | |
| JCVI_SMPL_1103283000012 | North American East Coast | Estuary | 8 | 52 | |
| JCVI_SMPL_1103283000013 | North American East Coast | Coastal | 12 | 55 | |
| JCVI_SMPL_1103283000014 | North American East Coast | Coastal | 19 | 71 | |
| JCVI_SMPL_1103283000015 | North American East Coast | Coastal | 10 | 69 | |
| JCVI_SMPL_1103283000016 | North American East Coast | Coastal | 6 | 47 | |
| JCVI_SMPL_1103283000017 | North American East Coast | Estuary | 10 | 49 | |
| JCVI_SMPL_1103283000018 | North American East Coast | Estuary | 16 | 40 | |
| JCVI_SMPL_1103283000019 | North American East Coast | Coastal | 9 | 39 | |
| JCVI_SMPL_1103283000020 | North American East Coast | Coastal | 12 | 53 | |
| JCVI_SMPL_1103283000021 | Caribbean Sea | Coastal | 4 | 32 | |
| JCVI_SMPL_1103283000022 | Caribbean Sea | Coastal Sea | 4 | 35 | |
| JCVI_SMPL_1103283000023 | Caribbean Sea | Open Ocean | 7 | 42 | |
| JCVI_SMPL_1103283000024 | Caribbean Sea | Open Ocean | 8 | 44 | |
| JCVI_SMPL_1103283000039 | Galapagos Islands | Coastal | 7 | 25 | |
| JCVI_SMPL_1103283000040 | Galapagos Islands | Coastal | 10 | 45 | |
| JCVI_SMPL_1103283000041 | Galapagos Islands | Coastal | 6 | 44 | |
| JCVI_SMPL_1103283000042 | Eastern Tropical Pacific | Open Ocean | 9 | 43 | |
| JCVI_SMPL_1103283000044 | Tropical South Pacific | Open Ocean | 132 | 132 | |
| JCVI_SMPL_1103283000048 | Tropical South Pacific | Open Ocean | 0 | 141 | |
| JCVI_SMPL_1103283000049 | Tropical South Pacific | Open Ocean | 0 | 147 | |
| JCVI_SMPL_1103283000050 | Tropical South Pacific | Open Ocean | 0 | 137 | |
| JCVI_SMPL_1103283000052 | Tropical South Pacific | Open Ocean | 11 | 45 | |
| JCVI_SMPL_1103283000053 | Polynesia Archipelagos | Coral Reef | 6 | 54 | |
| JCVI_SMPL_1103283000054 | Polynesia Archipelagos | Coastal | 2 | 34 | |
| JCVI_SMPL_1103283000055 | Polynesia Archipelagos | Coral Atoll | 0 | 140 | |
| JCVI_SMPL_1103283000056 | Polynesia Archipelagos | Coral Reef Atoll | 3 | 31 | |
| JCVI_SMPL_GS048b | Polynesia Archipelagos | Coral Reef | 6 | 48 | |
| JCVI_SMPL_GS108a | Indian Ocean | Lagoon Reef | 0 | 27 | |
| JCVI_SMPL_GS108b | Indian Ocean | Lagoon Reef | 4 | 16 | |
| JCVI_SMPL_GS109 | Indian Ocean | Open Ocean | 8 | 43 | |
| JCVI_SMPL_GS110a | Indian Ocean | Open Ocean | 8 | 26 | |
| JCVI_SMPL_GS110b | Indian Ocean | Open Ocean | 2 | 4 | |
| JCVI_SMPL_GS111 | Indian Ocean | Open Ocean | 3 | 20 | |
| JCVI_SMPL_GS112a | Indian Ocean | Open Ocean | 5 | 32 | |
| JCVI_SMPL_GS112b | Indian Ocean | Open Ocean | 10 | 21 | |
| JCVI_SMPL_GS113 | Indian Ocean | Open Ocean | 15 | 45 | |
| JCVI_SMPL_GS114 | Indian Ocean | Open Ocean | 3 | 28 | |
| *reads per 100000. | |||||
表选项
就分布来说,在大部分的地区,无论是不同的海域,还是相同的海域不同的取样地点 (公开海域open或者海岸coastal),Ⅰ类和Ⅱ类的分布都比较均匀。但是,在热带南太平洋 (Tropical south pacific) 区域 (表 4),这两个酶均有最大分布,Ⅰ类比其他地区约高出10倍,而Ⅱ类约高出3倍,说明该区域的亚硝酸盐酶含量更大,这可能预示着该区域该酶的活动更重要,也可能说明该区域的亚硝酸盐含量较大[18]。
3 讨论 氮的过剩导致的水体富营养化呈现越来越严重的趋势。氮过剩其根本原因在于氮循环过程的不平衡,即进入水体环境中的氮远远高于回复到空气中的氮量。反硝化作用将硝酸盐转化为亚硝酸盐,亚硝酸盐再经进一步反硝化生成NO最终到N2,可以看出,反硝化作用在氮循环中,尤其是在当前氮循环不平衡的过程中应该扮演很重要的消除氮过剩的角色。产NO的亚硝酸盐还原酶是在反硝化作用中最重要的一种酶,研究它的分布及特点有助于更好地理解它的作用机制,发挥它的反硝化作用,解决由于氮营养过剩导致的环境污染。
本文对于两类NIR分布情况的分析,与数据库中已测序的基因组数量之间存在着一定的关系,即在每个微生物种类中,含有该酶的微生物数量所占的比例是相对不变的,但是其绝对数量会随着已测序基因组数量的变化而变化,如在变形菌门中,Ⅰ类和Ⅱ类酶各有346和292株,已测序的变形菌门的基因组数量为2491株 (表 2),因此含有Ⅰ类和Ⅱ类酶的微生物数量在变形菌门中占有的比例分别为13.9%和11.7%,随着变形菌门已测序基因组的逐渐增多,含有NIR的微生物的绝对数量会相应增加,但占有比例则不会变动太大。此外应该注意到的是,数据的准确性与样本的大小有关系,尤其是在一些已测序基因组数量并不多的种类中 (如产水菌门,绿菌门等)(表 2),现有的分析结果可能不能全部反映其真实分布情况。
广义的亚硝酸盐还原酶并不局限于本文中所分析的两类,根据辅因子及受体的不同,它至少分为3类:由细胞色素cd1构成的 (Ⅰ类),由铜离子为辅因子的 (Ⅱ类),和以铁硫蛋白和血红素为辅因子的 (Ⅲ类)[2]。其中Ⅲ类主要分布在藻类,植物和蓝细菌中,并且NO只是它催化过程中的一个中间代谢产物,氨是其终产物,这样Ⅲ类与Ⅰ类和Ⅱ类可能在环境中的作用有区别。从本文结果可以看出,Ⅰ类和Ⅱ类在蓝细菌中很少存在 (Ⅰ类0个,Ⅱ类2个),这说明Ⅲ类NIR (在蓝细菌中广泛分布) 可能是其他2种NIR的功能互补型酶,可能只是由于其产物不同导致其氮循环的路线不同。
Ⅰ类和Ⅱ类NIR酶的分布存在着明显异同点:首先,从种类上看,两类亚硝酸盐在细菌中均有存在,而且,它们具有一致性分布,如前分析的,往往在同一个门中既有Ⅰ类,也有Ⅱ类,一致性的分布说明有些细菌种类的产NO的反硝化功能是环境所必需的,细菌的种类往往是与其生活环境相关,因此,从间接意义上来说,NIR的功能与环境相关性较大;其次,在古生菌中只有Ⅱ类,古生菌一般生长在一些极端的环境下,Ⅱ类利用铜离子作为辅因子,可能与其极端环境相关;最后,Ⅱ类无论是从数量上,还是分布的广度上都要比Ⅰ类更多,这可能说明在环境中,亚硝酸盐还原以Ⅱ类为主。
近年来,国内外开展了许多氮污染环境修复的研究,涉及水、湿地、陆地和污水处理等各个区域。曾薇等、马勇等开展关于生活污水的A2O或AO工艺处理,对于氨氮的去除率达到75.4%,并且从pH值、溶解氧和水力停留时间等方面分析了生活污水脱氮处理的影响因素[19-20]。近期的综述总结了利用人工湿地进行脱氮的技术,人工湿地同时能起到去除BOD (生物化学需氧量)、COD (化学需氧量) 等目的,只是脱氮效率不稳定,还需要进一步的研究[21]。吴义锋等构建模拟河渠岸坡特定生态系统,研究其脱氮效果,发现生物脱氮率可以达到85%左右[22]。从目前已有研究来看,起到主要的氮环境污染修复功能的是植物吸收和反硝化微生物的作用,付融冰等研究发现,在水环境中植物吸收和存储仅占湿地总氮去除量的10%左右[23],因此反硝化微生物在氮环境污染修复中占的比例应该更大。本文通过几个证据说明产NO的亚硝酸盐还原酶在环境修复中具有重要的作用:首先,在已经测序的细菌和古生菌基因组中进行广泛的比对,发现具有NIR的同源蛋白的微生物占有很大的比例,Ⅰ类和Ⅱ类分别占8%和15.7%,这说明环境中具有同等功能的微生物很多;其次,序列比对发现该酶的保守性很强,包括其底物结合位点以及辅因子结合位点等,无论是细菌还是古生菌中 (Ⅱ类) 保守性都很强,这说明该酶的功能是比较专一而且重要的;最后,通过在海洋宏基因组中查找,发现该酶在环境中的比例也相对较大。综上所述,亚硝酸盐还原酶通过保守的位点以及广泛的分布说明了其在环境修复中的重要性。
鉴于水体氮营养过剩的紧迫性及其中NIR的重要性,作者认为在将来的工作中还需要重点加强以下3点研究:第一针对不同的NIR,具体研究其作用机制;第二要研究NIR在环境中的生理功能;第三,研究短程硝化以及NIR在其中发挥的具体作用,短程硝化是20世纪在国内外兴起的一种新型污水脱氮技术,相比起传统脱氮工艺以 (NH4+→NO2–→NO3–→NO2–→NO→N2) 的脱氮顺序,短程硝化通过各种理化因素等控制,使其主要脱氮路线改为 (NH4+→NO2–→NO→N2),使其反硝化速率提高了63%左右[20, 24],而从两种工艺上比较可以看出,其中亚硝酸盐氮的积累与消耗是其关键因素[25],曲洋等报道在短程硝化过程中添加功能性菌种会增强其脱氮效果[25],牟丽娉等在活性污泥中添加了活性菌剂加强了脱氮效果[26],最近,周石磊等报道在微污染水中筛选到好氧反硝化菌群,进行水库的投菌实验并取得了初步良好的脱氮效果等[27],在这些研究基础上,试验在现有工艺中添加人工构建或自然分离的高效NIR表达的菌剂,并控制合适的生长培养条件,使NIR快速消耗亚硝酸盐氮而转变为NO,有助于使脱氮过程快速的向短程硝化过程转移,这对于整个脱氮工艺将具有重要意义。
致谢: 感谢山东大学夏永振在宏基因组学分析上的指导和帮助!
参考文献
| [1] | Vitousek PM, Aber JD, Howarth RW, Likens GE, Matson PA, Schindler DW, Schlesinger WH, Tilman DG. Human alteration of the global nitrogen cycle: sources and consequences.Ecological Applications, 1997, 7(3): 737–750. |
| [2] | Kuznetsova S, Knaff DB, Hirasawa M, Lagoutte B, Sétif P. Mechanism of spinach chloroplast ferredoxin-dependent nitrite reductase: spectroscopic evidence for intermediate states.Biochemistry, 2004, 43(2): 510–517DOI:10.1021/bi035662q. |
| [3] | Gamble TN, Betlach MR, Tiedje JM. Numerically dominant denitrifying bacteria from world soils.Applied and Environmental Microbiology, 1977, 33(4): 926–939. |
| [4] | Cutruzzolà F. Bacterial nitric oxide synthesis.Biochimica et Biophysica Acta (BBA)-Bioenergetics, 1999, 1411(2/3): 231–249. |
| [5] | Vijgenboom E, Busch JE, Canters GW. In vivo studies disprove an obligatory role of azurin in denitrification in Pseudomonas aeruginosa and show that azu expression is under control of rpoS and ANR.Microbiology, 1997, 143(9): 2853–2863DOI:10.1099/00221287-143-9-2853. |
| [6] | Paraskevopoulos K, Hough MA, Sawers RG, Eady RR, Hasnain SS. The structure of the Met144Leu mutant of copper nitrite reductase from Alcaligenes xylosoxidansprovides the first glimpse of a protein-protein complex with azurin Ⅱ.Journal of Biological Inorganic Chemistry, 2007, 12(6): 789–796DOI:10.1007/s00775-007-0233-y. |
| [7] | Ichiki H, Tanaka Y, Mochizuki K, Yoshimatsu K, Sakurai T, Fujiwara T. Purification, characterization, and genetic analysis of Cu-containing dissimilatory nitrite reductase from a denitrifying halophilic archaeon, Haloarcula marismortui.Journal of Bacteriology, 2001, 183(14): 4149–4156DOI:10.1128/JB.183.14.4149-4156.2001. |
| [8] | Zumft WG. Cell biology and molecular basis of denitrification.Microbiology and Molecular Biology Reviews, 1997, 61(4): 533–616. |
| [9] | Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG. Clustal W and clustal X version 2.0.Bioinformatics, 2007, 23(21): 2947–2948DOI:10.1093/bioinformatics/btm404. |
| [10] | Rusch DB, Halpern AL, Sutton G, Heidelberg KB, Williamson S, Yooseph S, Wu DY, Eisen JA, Hoffman JM, Remington K, Beeson K, Tran B, Smith H, Baden-Tillson H, Stewart C, Thorpe J, Freeman J, Andrews-Pfannkoch C, Venter JE, Li K, Kravitz S, Heidelberg JF, Utterback T, Rogers YH, Falcón LI, Souza V, Bonilla-Rosso G, Eguiarte LE, Karl DM, Sathyendranath S, Platt T, Bermingham E, Gallardo V, Tamayo-Castillo G, Ferrari MR, Strausberg RL, Nealson K, Friedman R, Frazier M, Venter JC. The sorcerer II global ocean sampling expedition: northwest Atlantic through eastern tropical Pacific.PLoS Biology, 2007, 5(3): e77DOI:10.1371/journal.pbio.0050077. |
| [11] | Howard EC, Sun SL, Biers EJ, Moran MA. Abundant and diverse bacteria involved in DMSP degradation in marine surface waters.Environmental Microbiology, 2008, 10(9): 2397–2410DOI:10.1111/emi.2008.10.issue-9. |
| [12] | Cai XL, Luo JF, Lin WT, Tian GL. Microbial community in nitrogen cycle of aquaculture water of the Pearl River Delta.Acta Microbiologica Sinica, 2012, 52(5): 645–653(in Chinese).蔡小龙, 罗剑飞, 林炜铁, 田国梁. 珠三角养殖水体中参与氮循环的微生物群落结构.微生物学报, 2012, 52(5): 645–653. |
| [13] | Nurizzo D, Cutruzzolà F, Arese M, Bourgeois D, Brunori M, Cambillau C, Tegoni M. Conformational changes occurring upon reduction and NO binding in nitrite reductase from Pseudomonas aeruginosa.Biochemistry, 1998, 37(40): 13987–13996DOI:10.1021/bi981348y. |
| [14] | Nurizzo D, Silvestrini MC, Mathieu M, Cutruzzolà F, Bourgeois D, Fül?p V, Hajdu J, Brunori M, Tegoni M, Cambillau C. N-terminal arm exchange is observed in the 2.15 ? crystal structure of oxidized nitrite reductase from Pseudomonas aeruginosa.Structure, 1997, 5(9): 1157–1171DOI:10.1016/S0969-2126(97)00267-0. |
| [15] | Antonyuk SV, Strange RW, Sawers G, Eady RR, Hasnain SS. Atomic resolution structures of resting-state, substrate-and product-complexed Cu-nitrite reductase provide insight into catalytic mechanism.Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(34): 12041–12046DOI:10.1073/pnas.0504207102. |
| [16] | Adman ET, Godden JW, Turley S. The structure of copper-nitrite reductase from Achromobacter cycloclastes at five pH values, with NO2- bound and with type Ⅱ copper depleted.The Journal of Biological Chemistry, 1995, 270(46): 27458–27474DOI:10.1074/jbc.270.46.27458. |
| [17] | Li Y, Hodak M, Bernholc J. Enzymatic mechanism of copper-containing nitrite reductase.Biochemistry, 2015, 54(5): 1233–1242DOI:10.1021/bi5007767. |
| [18] | Martiny AC, Kathuria S, Berube PM. Widespread metabolic potential for nitrite and nitrate assimilation among Prochlorococcus ecotypes.Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(26): 10787–10792DOI:10.1073/pnas.0902532106. |
| [19] | Zeng W, Li L, Yang YY, Zhang Y, Wang SY. Short-cut nitrification and denitrification in A2O process treating domestic wastewater.China Environmental Science, 2010, 30(5): 625–632(in Chinese).曾薇, 李磊, 杨莹莹, 张悦, 王淑莹. A2O工艺处理生活污水短程硝化反硝化的研究.中国环境科学, 2010, 30(5): 625–632. |
| [20] | Ma Y, Wang SY, Zeng W, Peng YZ, Zhou L. A/O pilot-scale nitrogen removal process treating domestic wastewater Ⅰ. The study of short-cut nitrification and denitrification.Acta Scientiae Circumstantiae, 2006, 26(5): 703–709(in Chinese).马勇, 王淑莹, 曾薇, 彭永臻, 周利. A/O生物脱氮工艺处理生活污水中试 (一) 短程硝化反硝化的研究.环境科学学报, 2006, 26(5): 703–709. |
| [21] | Zhang L, Cui LH. The denitrification status and its research progress of construction wetland.Chinese Agricultural Science Bulletin, 2012, 28(5): 268–272(in Chinese).张玲, 崔理华. 人工湿地脱氮现状与研究进展.中国农学通报, 2012, 28(5): 268–272. |
| [22] | Wu YF, Lü XW, Zhong ZP, Shi J, Xu W. Influencing factors of nitrogen removal from stream water using special riverine ecosystem.Journal of Central South University (Science and Technology), 2011, 42(2): 539–545(in Chinese).吴义锋, 吕锡武, 仲兆平, 史静, 徐微. 河渠岸坡特定生态系统的脱氮效率及影响因素.中南大学学报 (自然科学版), 2011, 42(2): 539–545. |
| [23] | Fu RB, Yang HZ, Gu GW, Zhang Z. Nitrogen removal from rural sewage by subsurface horizontal-flow in artificial wetlands.Technology of Water Treatment, 2006, 32(1): 18–22(in Chinese).付融冰, 杨海真, 顾国维, 张政. 潜流人工湿地对农村生活污水氮去除的研究.水处理技术, 2006, 32(1): 18–22. |
| [24] | Peng YZ, Sun HW, Yang Q. The biochemical reaction mechanism and kinetics of partial nitrification.Acta Scientiae Circumstantiae, 2008, 28(5): 817–824(in Chinese).彭永臻, 孙洪伟, 杨庆. 短程硝化的生化机理及其动力学.环境科学学报, 2008, 28(5): 817–824. |
| [25] | Qu Y, Zhang PY, Yu DS, Guo SS, Yang RX. Bioaugmentation for shortcut nitrification in SBR treating for sewage containing sea water by nitrification-aerobic denitrification bacteria.Environmental Science, 2010, 31(10): 2376–2384(in Chinese).曲洋, 张培玉, 于德爽, 郭沙沙, 杨瑞霞. 异养硝化/好氧反硝化菌生物强化含海水污水的SBR短程硝化系统初探.环境科学, 2010, 31(10): 2376–2384. |
| [26] | Mu LP, Huang J, Gou S. Denitrification by heterotrophic nitrification microbial consortium and aerobic granular sludge.Chinese Journal of Applied & Environmental Biology, 2009, 15(3): 356–360(in Chinese).牟丽娉, 黄钧, 苟莎. 异养硝化微生物菌剂及其好氧颗粒污泥的脱氮试验.应用与环境生物学报, 2009, 15(3): 356–360. |
| [27] | Zhou SL, Huang TL, Bai SY, He XX. Nitrogen removal characteristics of mixed aerobic denitrification bacteria under in-situ biological inoculation.Acta Microbiologica Sinica, 2016, 56(4): 590–602(in Chinese).周石磊, 黄廷林, 白士远, 何秀秀. 贫营养好氧反硝化菌群的水库原位投菌脱氮特性.微生物学报, 2016, 56(4): 590–602. |
