刘伟奇1, 董旭日1, 汤荣2, 陈雯静2, 洪庆2, 刘海泉1,3,4, 潘迎捷1,3,4, 赵勇1,3,4


1.上海海洋大学食品学院, 上海 201306;
2.上海市第一人民医院检验科, 上海 201620;
3.上海水产品加工及贮藏工程技术研究中心, 上海 201306;
4.农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
收稿日期:2016-11-19;修回日期:2017-01-05;网络出版日期:2017-02-13
基金项目:国家自然科学基金(31571917,31671779);上海市科技兴农重点攻关项目(2015第4-8号2016第1-1号);上海市教育委员会科研创新计划;上海市教委“曙光计划”(15SG48)
作者简介:赵勇,博士,教授,博士生导师,上海海洋大学食品学院副院长。中国食品科学技术学会青年工作委员会委员、非热加工技术分会理事,上海市食品学会青年工作委员会主任委员、食品安全专委会副主任委员,上海市微生物学会微生物耐药防控专委会副主任委员。2005年于南京农业大学获得微生物学理学博士学位,2006年复旦大学生命科学学院访问****,2008年美国佐治亚大学食品科学与技术系访问****,2009年英国食品研究院访问****。近年主持科研项目共19项,科研总经费达千万以上;发表科研论文205篇,其中SCI论文53篇;获得各类科技奖项10余项。目前主要研究工作为:食源性致病菌快速检测技术、风险评估技术及高效防控技术的研究。
*通信作者:赵勇, Tel:+86-21-61900503, E-mail:yzhao@shou.edu.cn
摘要:[目的]检测副溶血性弧菌(Vibrio parahaemolyticus,简称VP)中规律成簇间隔的短回文序列(Clustered regularly interspaced short palindromic repeats,CRISPR),并对不同来源的VP中CRISPR位点的结构多样性进行分析。[方法]根据CRISPR DB数据库中公布的VP中确定的CRISPR结构序列CRISPR-1及文献中新发现的疑似CRISPR结构序列CRISPR-2设计引物,对不同来源的79株VP进行PCR扩增。利用CRISPR Finder分析CRISPR结构,采用生物信息学方法对不同来源VP的CRISPR位点结构多样性进行比较分析。[结果]79株VP中CRISPR-1的检出率为92.41%,CRISPR-2的检出率为96.20%,同时具有这2个位点的菌株占总数的89.87%,只有1株菌被检出不含有任何位点。分别比较不同来源的菌株CRISPR-1、CRISPR-2位点的重复序列发现不存在序列差异,而临床菌株的这2个CRISPR位点在间隔序列上比环境分离菌株存在更多的变异。2个CRISPR位点根据间隔序列的不同在VP中一共组成8种CRISPR谱型(编号A-H),除F谱型外,A-E、G谱型均只在临床分离菌株中发现,而在环境分离菌中还发现不含任何位点的H型。[结论]CRISPR在VP中普遍存在。环境分离菌株与临床分离菌株中CRISPR的结构存在差异。
关键词: 不同来源 副溶血性弧菌 CRISPR 结构
Detection and analysis of CRISPR in Vibrio parahaemolyticus
Weiqi Liu1, Xuri Dong1, Rong Tang2, Wenjing Chen2, Qing Hong2, Haiquan Liu1,3,4, Yingjie Pan1,3,4, Yong Zhao1,3,4


1.College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China;
2.Shanghai General Hospital, Shanghai 201620, China;
3.Shanghai Engineering Research Centre of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
4.Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation(Shanghai), Ministry of Agriculture, Shanghai 201306, China
Received 19 November 2016; Revised 5 January 2017; Published online 13 February 2017
*Corresponding author: Zhao Yong, Tel:+86-21-61900503, E-mail:yzhao@shou.edu.cn
Supported by the National Natural Science Foundation of China (31571917, 31671779), by the Key Project of Shanghai Agriculture Prosperity through Science and Technology [2015(4-8), 2016(1-1)], by the Innovation Program of Shanghai Municipal Education Commission and by the “Dawn” Program of Shanghai Education Commission (15SG48)
Abstract: [Objective]To detect the clustered regularly interspaced short palindromic repeats (CRISPR) in Vibrio parahaemolyticus, and analyze its structural diversities in different sources of V. parahaemolyticus strains.[Methods]The primers of convincing CRISPR structure CRISPR-1 were designed by using the sequence according to CRISPR database, while the primers of questionable CRISPR structure CRISPR-2 were designed according to the literature. PCR approach was used to detect the CRISPR locus in all 79 strains, and all CRISPR sequences were analyzed using CRISPR Finder. Furthermore, the structural diversities of CRISPR in different sources of V. parahaemolyticus were analyzed using bioinformatics methods.[Results]The positive rate of CRISPR-1 was 92.41% and of CRISPR-2 96.20%. The strains possessed with these two locus accounted for 89.87% of the total strains, and only one strain did not contain any locus. There was no difference between the repeats of CRISPR-1 and CRISPR-2 from different sources of V. parahaemolyticus, whereas the spacers of those two locus in clinical strains had more variations than those in environmental strains. Therefore, two CRISPR locus formed 8 spectral patterns (numbers A-H) according to the variations of spacers. Except type F, the patterns A-E, G only were found in the clinical strains. And the type H containing none of locus was only found in environmental strains.[Conclusion]CRISPR commonly existed in V. parahaemolyticus. There were differences in the structure of CRISPR between environmental and clinical strains.
Key words: different sources Vibrio parahaemolyticus CRISPR structure
规律成簇间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)是近年来发现的广泛分布于细菌和古细菌中的一类特殊的DNA重复序列[1],主要是由同向重复序列(repeats,R)和间隔序列(spacers,S)间隔排列组成。研究发现[2],CRISPR中的同向重复序列和间隔序列在细菌进化中均存在变异现象,使得CRISPR结构具有多态性,并且在同一物种的不同菌株间存在差异,这种差异为细菌分型溯源、进化研究提供了更加可靠、准确的依据,目前已在食源性致病菌特别是沙门氏菌的分型[3]中得到了广泛的应用;另外CRISPR结构的多态性还被发现与细菌的耐药性存在一定的关系[4]。CRISPR结构的侧翼区常存在CRISPR相关蛋白基因(CRISPR-associated,cas),这些基因与CRISPR共同构成CRISPR/cas系统,为原核生物提供对噬菌体、质粒等外源基因的获得性免疫能力,从而抵御外源基因的入侵,限制基因的水平转移[5-6]。研究发现CRISPR/cas系统除了免疫防御功能外,还可能具有调控细菌生理活动等其他功能[7]。目前,有关CRISPR在食源性致病菌中的功能及应用研究已成为热点[8-12]。
近年研究发现常见的食源性致病菌副溶血性弧菌(Vibrio parahaemolyticus,简称VP)中存在CRISPR结构,并且已有研究表明VP中CRISPR结构与细菌进化之间并无严格关系,并不适合用于细菌分型[11],而VP的CRISPR却与其毒力基因tdh之间存在显著的相关性[12],这可能表明含有CRISPR结构的VP存在潜在的致病性。VP中CRISPR/cas系统如何对外源基因进行免疫防御,是否具有其他生理功能还未可知。本文通过对44株临床分离菌株及35株环境分离菌株进行CRISPR结构的检测与分析,以期对VP中CRISPR结构的功能机制研究提供基础信息。
1 材料和方法 1.1 菌株 42株临床分离菌株由上海市第一人民医院馈赠,C5由中国科学院微生物研究所馈赠,C39由美国疾病与控制中心馈赠,35株环境分离菌株由本实验室从河虾中分离,具体信息见表 1。
表 1. 实验中所用菌株 Table 1. The strains used in this study
| Isolate | Genotype | Source of isolation |
| C1 | tdh+/trh–/tlh+ | Clinical |
| C2 | tdh+/trh+/tlh+ | Clinical |
| C15 | tdh+/trh–/tlh+ | Clinical |
| C16 | tdh+/trh–/tlh+ | Clinical |
| C17 | tdh+/trh–/tlh+ | Clinical |
| C18 | tdh–/trh+/tlh+ | Clinical |
| C19 | tdh+/trh–/tlh+ | Clinical |
| C20 | tdh+/trh–/tlh+ | Clinical |
| C21 | tdh+/trh–/tlh+ | Clinical |
| C22 | tdh+/trh–/tlh+ | Clinical |
| C25 | tdh+/trh–/tlh+ | Clinical |
| C26 | tdh+/trh–/tlh+ | Clinical |
| C27 | tdh+/trh–/tlh+ | Clinical |
| C28 | tdh+/trh–/tlh+ | Clinical |
| C29 | tdh+/trh–/tlh+ | Clinical |
| C32 | tdh+/trh–/tlh+ | Clinical |
| C33 | tdh+/trh–/tlh+ | Clinical |
| C34 | tdh+/trh–/tlh+ | Clinical |
| C35 | tdh+/trh+/tlh+ | Clinical |
| C36 | tdh–/trh+/tlh+ | Clinical |
| C37 | tdh+/trh+/tlh+ | Clinical |
| C38 | tdh+/trh+/tlh+ | Clinical |
| C40 | tdh+/trh+/tlh+ | Clinical |
| C41 | tdh+/trh–/tlh+ | Clinical |
| C42 | tdh+/trh+/tlh+ | Clinical |
| C43 | tdh+/trh–/tlh+ | Clinical |
| C44 | tdh–/trh–/tlh+ | Clinical |
| C45 | tdh+/trh–/tlh+ | Clinical |
| C46 | tdh+/trh–/tlh+ | Clinical |
| C47 | tdh+/trh–/tlh+ | Clinical |
| C48 | tdh+/trh–/tlh+ | Clinical |
| C49 | tdh+/trh–/tlh+ | Clinical |
| C50 | tdh+/trh–/tlh+ | Clinical |
| C51 | tdh+/trh+/tlh+ | Clinical |
| C54 | tdh+/trh+/tlh+ | Clinical |
| C55 | tdh+/trh–/tlh+ | Clinical |
| C85 | tdh–/trh+/tlh+ | Clinical |
| C89 | tdh+/trh–/tlh+ | Clinical |
| C90 | tdh+/trh–/tlh+ | Clinical |
| C94 | tdh–/trh+/tlh+ | Clinical |
| C97 | tdh+/trh–/tlh+ | Clinical |
| C100 | tdh+/trh–/tlh+ | Clinical |
| C5 | tdh+/trh–/tlh+ | Clinical |
| C39 | tdh+/trh+/tlh+ | Clinical |
| S1 | tdh+/trh–/tlh+ | Shrimp |
| S2 | tdh+/trh–/tlh+ | Shrimp |
| S4 | tdh+/trh–/tlh+ | Shrimp |
| S6 | tdh+/trh–/tlh+ | Shrimp |
| S8 | tdh–/trh–/tlh+ | Shrimp |
| S9 | tdh+/trh–/tlh+ | Shrimp |
| S10 | tdh–/trh+/tlh+ | Shrimp |
| S11 | tdh+/trh–/tlh+ | Shrimp |
| S12 | tdh+/trh–/tlh+ | Shrimp |
| S13 | tdh–/trh–/tlh+ | Shrimp |
| S14 | tdh–/trh–/tlh+ | Shrimp |
| S15 | tdh–/trh–/tlh+ | Shrimp |
| S16 | tdh–/trh–/tlh+ | Shrimp |
| S17 | tdh–/trh–/tlh+ | Shrimp |
| S18 | tdh–/trh–/tlh+ | Shrimp |
| S19 | tdh–/trh–/tlh+ | Shrimp |
| S20 | tdh–/trh–/tlh+ | Shrimp |
| S21 | tdh–/trh–/tlh+ | Shrimp |
| S22 | tdh+/trh–/tlh+ | Shrimp |
| S23 | tdh+/trh–/tlh+ | Shrimp |
| S24 | tdh+/trh–/tlh+ | Shrimp |
| S25 | tdh–/trh–/tlh+ | Shrimp |
| S26 | tdh+/trh–/tlh+ | Shrimp |
| S27 | tdh+/trh–/tlh+ | Shrimp |
| S28 | tdh+/trh–/tlh+ | Shrimp |
| S29 | tdh+/trh–/tlh+ | Shrimp |
| S30 | tdh+/trh–/tlh+ | Shrimp |
| S31 | tdh+/trh–/tlh+ | Shrimp |
| S32 | tdh+/trh–/tlh+ | Shrimp |
| S33 | tdh–/trh–/tlh+ | Shrimp |
| S34 | tdh–/trh–/tlh+ | Shrimp |
| S35 | tdh–/trh–/tlh+ | Shrimp |
| S36 | tdh–/trh–/tlh+ | Shrimp |
| S37 | tdh–/trh–/tlh+ | Shrimp |
| S38 | tdh–/trh–/tlh+ | Shrimp |
表选项
1.2 CRISPR检测 根据CRISPR DB数据库(http://crispr.i2bc.paris-saclay.fr/crispr/)中公布的VP中确定的CRISPR结构序列CRISPR-1,以及位于kdgR、kduD基因之间的疑似CRISPR结构序列[11]CRISPR-2设计引物(引物序列见表 2),其中CRISPR-1与cas基因组成CRISPR/cas系统,CRISPR-2为孤立位点。以试剂盒提取的VP基因组DNA为模板,对VP菌的CRISPR进行扩增和序列测定。扩增体系为50 μL,其中2×Taq PCR Mix 25 μL,ddH2O 20 μL,10 mmol/L上下游引物各2 μL,DNA模板1 μL。PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,53 ℃ 60 s,72 ℃ 60 s,35个循环;72 ℃ 10 min。取5 μL PCR产物于2%的琼脂糖凝胶电泳进行初步分析,所得阳性结果送至生工生物工程(上海)股份有限公司进行测序,利用CRISPR Finder[13],确认CRISPR的存在和结构,利用序列比对软件ClustalX 2.1及DNAMAN对检出重复序列和间隔序列进行比对。利用在线软件WebLogo (http://weblogo.berkeley.edu/logo.cgi)对重复序列进行保守性分析,所有间隔序列都通过BLAST (http:// www.ncbi.nlm.nih.gov/BLAST)在线比对分析比对,以寻找与其同源的DNA序列。
表 2. CRISPR的引物序列 Table 2. Primers of CRISPR for PCR amplification
| Genes | Primers | Sequences (5′→3′) | PCR products/bp |
| CRISPR-1 | F | AAGAAGATAGCAGCAGTGGCAATAA | 556 |
| R | TTGGCTGAGCCTGGAGATGTAA | ||
| CRISPR-2 | F | AACGCCGATGGCTTTCTGTT | 486 |
| R | TGTTGGTTCGCTGATATTTACGC |
表选项
2 结果和分析 2.1 79株VP的CRISPR位点分析
2.1.1 CRISPR的检测结果:以79株VP基因组DNA为模板,PCR扩增CRISPR-1、CRISPR-2(图 1),采用CRISPR Finder分析查找CRISPR,结果见表 3,在79株VP中大部分菌株含有2个CRISPR位点,只有1.27%的菌株未检出任何CRISPR位点,单独含有CRISPR-1、CRISPR-2位点的菌株分别占总数的2.53%、6.33%。在79株VP中确定的CRISPR结构CRISPR-1的总检出率较高(表 4),其中含2个间隔序列的CRISPR位点检出率为81.01%,11.39%的菌株含有3个间隔序列,通过比较分析临床分离菌株与环境分离菌株CRISPR-1的检出率发现,临床分离菌株CRISPR-1的检出率高于环境分离菌株,并且只在临床分离菌株中发现含有3个间隔序列。疑似CRISPR结构CRISPR-2在79株VP中普遍存在,并且都只含有1个间隔序列,而在这个疑似位点的周围没有cas基因的存在[11],推测可能是一个退化的CRISPR位点。
 |
| 图 1 2个CRISPR位点PCR扩增产物的电泳图 Figure 1 Gel electrophoresis of the two CRISPR locus. M: DL2000 DNA marker; B: blank; C1–S37: different types of strains. |
| 图选项 |
表 3. 79株VP CRISPR的分布 Table 3. Distribution of CRISPR in 79 VP strains
| Genes | Strain number | Percentage/% |
| CRISPR-1+CRISPR-2 | 71 | 89.87 |
| CRISPR-1 | 2 | 2.53 |
| CRISPR-2 | 5 | 6.33 |
表选项
表 4. 79株VP CRISPR的检出率和间隔序列数目 Table 4. The positive rate and spacer number of CRISPR in 79 VP strains
| Genes | Spacer number | Clinical strain number | Environmental strain number | Percentage/% |
| CRISPR-1 | 2 | 33 | 31 | 92.41 |
| 3 | 9 | 0 | ||
| CRISPR-2 | 1 | 43 | 33 | 96.20 |
表选项
2.1.2 CRISPR的重复序列:CRISPR Finder识别到的重复序列共2种,并未发现有新的重复序列存在。CRISPR-1的重复序列与CRISPR DB数据库中公布的一致,长度均为28 bp,通过WebLogo在线软件分析所有检出CRISPR-1的重复序列的保守性(图 2-A)发现,在重复序列的末端位点存在较多的变异。疑似位点CRISPR-2的重复序列长度均为25 bp,通过序列保守性分析(图 2-B)发现,该位点的重复序列较保守,仅有1个位点发生变异。
 |
| 图 2 VP所有CRISPR位点的重复序列 Figure 2 Sequence logo for all repeats in the CRISPR loci of VP. The height of the letters showed the relative frequency of the corresponding nucleotide at that position. |
| 图选项 |
2.1.3 CRISPR的间隔序列:CRISPR Finder识别到的间隔序列共231条,其中确定位点CRISPR-1中检测到3种不同类型的间隔序列,共155条,有2种类型的间隔序列与CRISPR DB数据库中公布的一致,另一种间隔序列为新发现的间隔序列spacer1,共10条。疑似位点CRISPR-2都只含有1种间隔序列。在同一CRISPR位点中间隔序列存在差异(图 3),CRISPR-1中3种类型的间隔序列之间同源性较低,spacer1、spacer2、spacer3这3种类型的间隔序列在一些位点存在碱基的突变其中spacer2、spacer3这两种类型间隔序列的多个碱基位点发生不同的突变。CRISPR-2的间隔序列,spacer4多样性较高,主要集中在3个位点发生碱基的突变,共6种亚型。将所有检出的间隔序列在NCBI数据库中进行BLAST分析,并未发现与之高度同源的质粒或噬菌体序列,但新获得的间隔序列spacer1.2与数据库中VP自身基因组上的假定蛋白的基因序列高度同源,这表明spacer1.2靶向了自身基因序列。
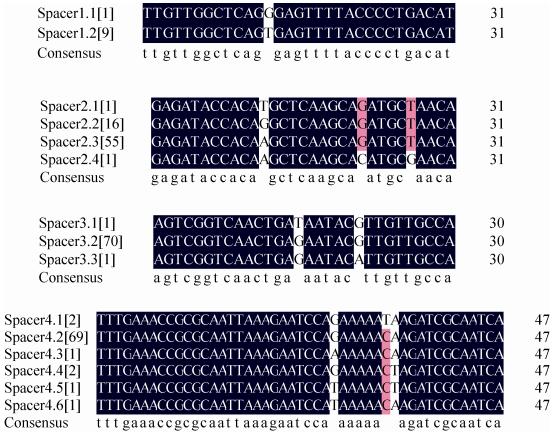 |
| 图 3 CRISPR的间隔序列比对 Figure 3 Sequence alignment of CRISPR spacers. Messages in bracket represented the number of strains with corresponding spacer. Blue and pink respectively represented 100%, 75% homology. |
| 图选项 |
2.2 不同来源的VP间CRISPR位点的比较分析
2.2.1 环境菌株与临床菌株CRISPR位点的比较分析:79株VP中,除S37外,每株至少含有1个CRISPR位点,根据CRISPR-1位点间隔序列的多样性,共组成了8种不同的CRISPR谱型,结合CRISPR-2位点间隔序列的多样性,8种谱型又包括不同的亚型,这些位点在79株VP中的分布情况见图 4。通过CRISPR位点分布图发现,大部分VP菌株主要分布在F-5亚型,比较临床分离菌株与环境分离菌株发现,环境分离菌株主要分布在F谱型,只有菌株S37为H谱型,而在剩余其他谱型中未发现有环境分离菌株,CRISPR-2位点的多样性赋予了环境分离菌株CRISPR位点的多样性,环境分离菌株的变化影响比较单一。而临床分离菌株分布较分散,除H谱型外,其他谱型中均存在临床分离菌株,在临床分离菌株中CRISPR-1、CRISPR-2位点的间隔序列均发生不同程度的碱基突变,多个位点的碱基突变赋予了临床分离菌株较环境分离菌株更多的多样性。在临床分离菌株C44、C47、C54及环境分离菌株S19、S37中均有CRISPR位点缺失的发生,而在临床菌株C36中发现该菌的CRISPR-1位点缺失较古老的间隔序列,新获得的间隔序列spacer1及间隔序列spacer2也较A、B型存在碱基的变化。这些都表明临床分离菌株与环境分离菌株CRISPR位点存在差异,也证明了在不同的生存环境下VP的CRISPR发生了微进化。
 |
| 图 4 79株VP CRISPR的结构分布 Figure 4 The CRISPR structures in the 79 VP strains A→H represented the 8 CRISPR spectral patterns. Messages in bracket represented the strains with corresponding CRISPR spectral pattern. |
| 图选项 |
2.2.2 79株VP与数据库中全基因组测序的VP的CRISPR位点的比较分析:通过CRISPR Finder查找NCBI数据库中12株全基因组测序的VP (RIMD 2210633、BB22OP、FDA_R31、CDC_K4557、UCM-V493、FORC_008、FORC_006、FORC_004、ATCC17802、FORC_014、CHN25、FORC_023) 中的CRISPR位点,由于CRISPR为非编码序列,由CRISPR Finder检测到的位于编码区的重复间隔序列不在本研究范围内。在12株菌中,FDA_R31未检到任何CRISPR序列,RIMD 2210633为上述F-5亚型,BB22OP为D-1亚型,其余9株VP均属于G型,在这9株菌中新发现了3种不同类型的疑似位点CRISPR-2。通过检测全基因组测序的VP发现,CRISPR位点只出现在VP的第2条染色体中,并未发现菌株的质粒及染色体1上具有CRISPR位点,已有研究推测VP的CRISPR结构可能是以水平基因转移的方式整合到细菌染色体上[11],而为何CRISPR位点只整合到染色体2中,这其中的获得机制还有待进一步研究。由于12株菌中检测到的确定位点CRISPR-1很少,而CRISPR-1的多样性主要出现在临床分离菌株中,通过比对NCBI数据库中全基因测序的临床分离菌株[11]并未发现新的重复序列及间隔序列,VP的CRISPR-1结构在不同年代分离的菌株中相近,这表明VP的CRISPR进化较缓慢,侧面说明了VP的CRISPR/cas系统活力较低。
3 讨论 CRISPR/cas是大多数细菌和古细菌内一种防御异源片段入侵的免疫系统,而CRISPR结构中的间隔序列在免疫过程中发挥着重要作用,间隔序列可以靶向外源基因,CRISPR/cas系统通过获得外源基因的一段标志基因为新的间隔序列,从而抵御这一外源基因的再次入侵[5-6]。而本研究中只有CRISPR-1位点周围存在cas基因,具有免疫防御功能,在检出的CRISPR-1位点中1株菌最多只含有3个间隔序列,大部分菌株只有2个间隔序列,并且这些间隔序列的序列相近,而这些间隔序列也并未发现与任何质粒或噬菌体基因同源,而通过本实验室之前对菌株耐药性分析发现,临床分离菌株都具有多重耐药基因[14],这些现象似乎与CRISPR/cas系统的功能不符,一个高活性的CRISPR/cas系统不仅具有多样性较多的间隔序列,并且应有效地抵御外源基因的入侵。近期已有研究表明,在VP中发现由移动元件表达的蛋白AcrF9是一类anti-CRISPR蛋白,并且具有很高的活性,可以有效地抑制I-F型CRISPR系统的活性,不仅是抑制VP自身CRISPR系统(I-F型[12])活性,而且能够抑制其他I-F型细菌的CRISPR系统活性,尽管这些菌株的I-F型CRISPR系统的Cas蛋白的同源性较低,但抑制活性仍然很高[15]。这或许是VP中水平基因转移频繁,CRISPR系统活性低的原因。
在比对VP间隔序列同源性时,我们发现spacer1.2靶向VP基因组蛋白,其中的功能还未可知,这种现象同样在其他菌株中也有所发现,而在一些菌中CRISPR的间隔序列靶向一些重要的蛋白家族,这些蛋白家族在菌株的生理调节活动中发挥一定的作用[16],这或许可以推测VP的CRISPR也很可能参与细菌生存方式、毒力等多项生理活动调节。在本文研究中临床分离菌株与环境分离菌株的CRISPR存在一定的差异,临床分离菌株相对于环境分离菌株在间隔序列上存在较多的变异,甚至有一些菌株的CRISPR位点缺失,已有研究[17]表明毒力或抗生素抗性等的选择压力很可能影响CRISPR/cas系统的存在,在不同生存环境的选择压力下,为何临床分离菌株CRISPR表现出较多变异性,这其中的机制还有待进一步的研究。
References
| [1] | Karginov FV, Hannon GJ. The CRISPR system:small RNA-guided defense in bacteria and archaea. Molecular Cell, 2010, 37(1): 7-19. DOI:10.1016/j.molcel.2009.12.033 |
| [2] | Wang J, Qiu SF, Song HB, Sun YS. Progress in CRISPR research on bacteria typing and evolution. Letters in Biotechnology, 2013, 24(3): 414-417. (in Chinese) 王建, 邱少富, 宋宏彬, 孙岩松. CRISPR在细菌分型和进化中的研究进展. 生物技术通讯, 2013, 24(3): 414-417. |
| [3] | Cai YQ, Li QC, Tao J, Jiang DJ, Pan ZM, Jiao XA. Advances in structure and function of CRISPR in Salmonella. Progress in Veterinary Medicine, 2015, 36(2): 97-101. (in Chinese) 蔡银强, 李求春, 陶静, 蒋道军, 潘志明, 焦新安. 沙门菌CRISPR的结构与功能研究进展. 动物医学进展, 2015, 36(2): 97-101. |
| [4] | Wang LL, Wang YF, Duan GC, Xue ZR, Guo XJ, Wang PF, Xi YL, Yang HY. Detection of CRISPR and its relationship to drug resistance in Shigella. Acta Microbiologica Sinica, 2015, 55(4): 476-483. (in Chinese) 王琳琳, 王颖芳, 段广才, 薛泽润, 郭向娇, 王鹏飞, 郗园林, 杨海燕. 志贺菌CRISPR的检测及其与耐药的关系. 微生物学报, 2015, 55(4): 476-483. |
| [5] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [6] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [7] | Medina-Aparicio L, Rebollar-Flores JE, Gallego-Hernández AL, Vázquez A, Olvera L, Gutiérrez-Ríos RM, Calva E, Hernández-Lucas I. The CRISPR/Cas immune system is an operon regulated by LeuO, H-NS, and leucine-responsive regulatory protein in Salmonella enterica serovar Typhi. Journal of Bacteriology, 2011, 193(10): 2396-2407. DOI:10.1128/JB.01480-10 |
| [8] | Liu WQ, Wang X, Liu HQ, Pan YJ, Zhao Y. Advances in structures and functions of CRISPR in common foodborne pathogenic bacterium. Food Science. (in Chinese) 刘伟奇, 王旭, 刘海泉, 潘迎捷, 赵勇. 常见食源性致病菌CRISPR系统结构与功能研究进展. 食品科学. DOI:10.7506/spkx1002-6630-201720001 |
| [9] | Fricke WF, Mammel MK, McDermott PF, Tartera C, White DG, Leclerc JE, Ravel J, Cebula TA. Comparative genomics of 28 Salmonella enterica isolates:evidence for CRISPR-mediated adaptive sublineage evolution. Journal of Bacteriology, 2011, 193(14): 3556-3568. DOI:10.1128/JB.00297-11 |
| [10] | Di HL, Yan H, Shi L. Analysis of CRISPR regions in food-borne Listeria monocytogenes. Modern Food Science and Technology, 2014, 30(8): 64-69, 237. (in Chinese) 狄慧玲, 闫鹤, 石磊. 食源性单核细胞增生李斯特菌CRISPR结构的研究. 现代食品科技, 2014, 30(8): 64-69, 237. |
| [11] | Ge CY, Lu HX, Qu DF, Zhu JL, Yuan DX, Feng LF. Comparative analysis of clustered regularly interspaced short palindromic repeats (CRISPR) in the genomes of Vibrio parahaemolyticus group. Genomics and Applied Biology, 2015, 34(8): 1712-1722. (in Chinese) 葛彩云, 陆海霞, 曲道峰, 朱军莉, 袁冬霞, 冯立芳. 副溶血性弧菌群中规律成簇间隔短回文重复序列的比较分析. 基因组学与应用生物学, 2015, 34(8): 1712-1722. |
| [12] | Sun HH, Li YH, Shi XL, Lin YM, Qiu YQ, Zhang JJ, Liu Y, Jiang M, Zhang Z, Chen QC, Sun Q, Hu QH. Association of CRISPR/Cas evolution with Vibrio parahaemolyticus virulence factors and genotypes. Foodborne Pathogens and Disease, 2015, 12(1): 68-73. DOI:10.1089/fpd.2014.1792 |
| [13] | Grissa I, Vergnaud G, Pourcel C. CRISPRFinder:a web tool to identify clustered regularly interspaced short palindromic repeats. Nucleic Acids Research, 2007, 35(Suppl 2): W52-W57. |
| [14] | Li H, Zhang ZH, Tang R, Lou Y, Zhao L, Chen WJ, Hong Q, Pan YJ, Zhao Y. Comparison of antimicrobial resistance of pathogenic Vibrio parahaemolyticus in seafood and clinical samples. Chinese Journal of Zoonoses, 2016, 32(11): 1006-1012. (in Chinese) 李欢, 张昭寰, 汤荣, 娄阳, 赵莉, 陈雯静, 洪庆, 潘迎捷, 赵勇. 食品与临床分离的致病性副溶血性弧菌耐药性比较. 中国人兽共患病学报, 2016, 32(11): 1006-1012. |
| [15] | Pawluk A, Staals RHJ, Taylor C, Watson BNJ, Saha S, Fineran PC, Maxwell KL, Davidson AR. Inactivation of CRISPR-Cas systems by anti-CRISPR proteins in diverse bacterial species. Nature Microbiology, 2016, 1(8): 16085. DOI:10.1038/nmicrobiol.2016.85 |
| [16] | Pleckaityte M, Zilnyte M, Zvirbliene A. Insights into the CRISPR/Cas system of Gardnerella vaginalis. BMC Microbiology, 2012, 12(1): 301. DOI:10.1186/1471-2180-12-301 |
| [17] | Palmer KL, Gilmore MS. Multidrug-resistant enterococci lack CRISPR-cas. mBio, 2010, 1(4): e00227-10. |
