李文均

 , 田野
, 田野 中山大学生命科学学院, 广东 广州 510275
收稿日期:2017-07-10;修回日期:2017-08-04;网络出版日期:2017-08-15
基金项目:国家自然科学基金(31470139,31600103);广东省高等学校珠江****岗位计划项目(2014)
作者简介:李文均,中山大学生命科学学院****/珠江********、博士生导师。1996年本科毕业于河南师范大学生物系;1999年硕士毕业于云南大学云南省微生物研究所;2002年博士毕业于中国科学院沈阳应用生态研究所;1999–2014年曾在云南大学云南省微生物研究所工作。现任国际原核微生物系统学委员会(International Committee on Systematics of Prokaryotes,ICSP)国际执委,伯杰氏国际系统微生物学会(BISMiS)创始会员。同时,还兼任中国青年科技工作者协会常务理事、中国微生物学会微生物教学工作委员会副主任委员、中国微生物学会普通微生物专业委员会委员、中国微生物学会分子微生物学与生物工程专业委员会委员、微生物资源专业委员会委员。Antonie van Leeuwenhoek、Frontiers in Microbiology、BioMed Research International、The Scientific World Journal、Journal of Arid Land、BISMiS Bulletin、《微生物学报》、《微生物学杂志》等杂志编委及多个国内外期刊的通讯评审专家。主要从事高温、高盐等极端环境微生物分类及基因组系统学,微生物生态学,极端酶学研究。实验室主页:http://www.liactlab.com/index.html。
*通信作者:李文均, Tel/Fax:+86-20-84111727, E-mail:liwenjun3@mail.sysu.edu.cn, liact@hotmail.com
摘要:遗传与变异体现着生命之美和生命之奥妙,前人在不断探求的过程中逐渐形成了严谨的体系和科学的方法——遗传学。在遗传学的研究中基因编辑工具的作用是不可或缺的,近年来发现的CRISPR/Cas系统作为基因编辑工具箱中的新刃,以其特异性强、靶向性好、适用性广的特点迅速成为广大科研工作者研究和开发的热点。并且,在探索生命暗物质的过程中,会有更多的新的CRISPR/Cas系统被发现并应用在遗传研究等领域。为此,本文综述目前CRISPR/Cas系统的最新研究进展,力图从其系统多样性、分子工作机制及遗传研究应用等方面,为广大科研工作者提供一个系统了解CRISPR/Cas系统及其研究现状的窗口,以期为该领域的研究与应用提供一些有益的参考。
关键词: CRISPR/Cas 基因编辑 基因调控 表观遗传学
CRISPR/Cas——a new tool for molecular genetic research
Wenjun Li

 , Ye Tian
, Ye Tian School of Life Sciences, Sun Yat-Sen University, Guangzhou 510275, Guangdong Province, China
Received 10 July 2017; Revised 4 August 2017; Published online 15 August 2017
*Corresponding author: Li Wenjun, Tel/Fax:+86-20-84111727, E-mail:liwenjun3@mail.sysu.edu.cn, liact@hotmail.com
Supported by the National Natural Science Foundation of China (31470139, 31600103) and by the Guangdong Province Higher Vocational Colleges and Schools Pearl River Scholar Funded Scheme (2014)
Abstract: Heredity and variation embody the beauty and mystery of life. In the process of continuous exploration, researchers have established genetics that is full of rigorous systematic and scientific methods. It is generally known that gene editing tools are indispensable in the study of genetics. CRISPR/Cas discovered in recent years as a new gene editing toolbox, has many characteristics, such as strong specificity and target ability, and become a hot spot of research and development. Here, we review the latest progress of CRISPR/Cas system including diversity, molecular mechanism and application.
Key words: CRISPR/Cas gene editing gene regulation genetics epigenetics
在地球生物圈中,微生物有着丰富的类群和庞大的数量。微生物在漫长的适应性进化的过程中,逐渐形成了多种抵御噬菌体和其他病毒侵染的防御系统,诸如限制-修饰系统、毒素-抗毒素系统、以及近年来发现的CRISPR/Cas系统[1-3]。CRISPR/Cas系统作为一种抵御外源核酸的获得性免疫防御手段,广泛存在于古菌和细菌中[4-8]。该系统由CRISPR阵列(CRISPR array)和Cas蛋白(CRISPR associated proteins)两部分组成[9],前者用于储存和记录外源核酸入侵的信息,后者负责免疫防御功能的具体实施。CRISPR/Cas系统的发现,不仅对于理解微生物在复杂的环境中抵御病毒的进化机制具有重大意义,更显示出该系统在分子遗传操作领域所具有的广阔应用前景。在此,本文首先系统追踪了近年来有关CRISPR/Cas系统在新类群的发现和作用机制中的研究进展,以期给大家提供详细的理论参考资料;其次,本文综述了CRISPR/Cas系统在遗传操作领域中的应用现状和应用前景,希望能给遗传学研究领域的广大科研工作者提供一些借鉴;最后,本文对现有CRISPR/Cas系统研究中存在的问题及未来的发展方向给予建议和展望,借抛砖以引玉,愿与大家携手将该领域的研究向前推进。
1 CRISPR/Cas系统的多样性 CRISPR-Cas系统依据效应蛋白的组成及其发挥功能的组织方式被分成特征明显的两大类群(Class)[10-11]:Class 1和Class 2。Class 1是以多蛋白组成的效应复合体行使免疫功能为特征(图 1),在现有报道的CRISPR-Cas系统中,90%的成员聚类此类群中,涵盖了细菌域和古菌域。Class 1包括Type Ⅰ、Type Ⅲ和Type Ⅳ,共12个亚型(Subtype) (图 1)。目前研究较为深入的是Type Ⅰ和Type Ⅲ,其中,Type Ⅰ成员的基因座中均含有特征蛋白Cas3 (部分亚型是Cas3的变体Cas3′)。Cas3蛋白含有受单链DNA(ssDNA)激活的解旋酶功能域和HD家族核酸内切酶功能域,前者用以打开DNA双链或RNA-DNA链[12-13],后者则用于DNA序列的切割。Type Ⅰ系统分为7个亚型,Ⅰ-A Subtype到Ⅰ-F Subtype及Ⅰ-U Subtype,其中Ⅰ-U Subtype是指不具代表特征的分类单元,在该类群中CRISPR array转录出来的非编码RNA (CRISPR-derived RNA,crRNA)的加工成熟机制及效应复合体的结构是未知的[14]。Ⅰ-C、Ⅰ-D、Ⅰ-E、Ⅰ-F Subtype的CRISPR基因座为单个操纵子,其中包含了Cas1、Cas2、Cas3及Cascade复合物亚基的编码基因,与之不同的Ⅰ-A和Ⅰ-B系统是一个多功能蛋白,由多个操纵子组成[15]。
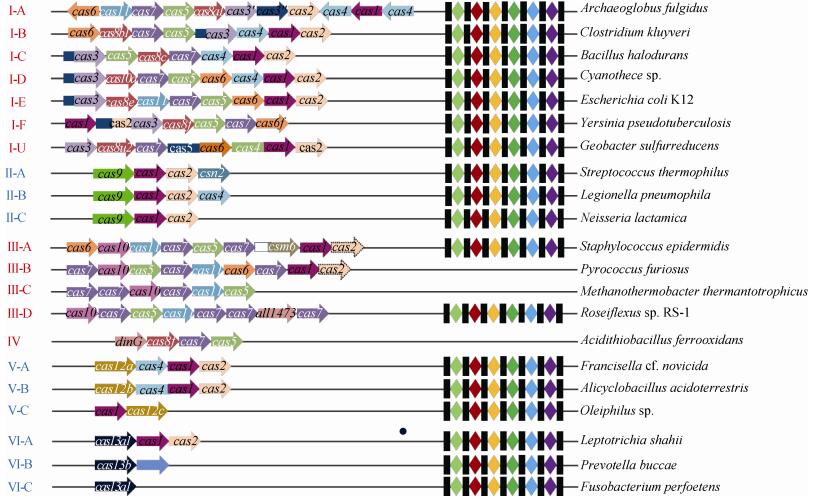 |
| 图 1 CRISPR/Cas系统分类 Figure 1 The classification of CRISPR/Cas. |
| 图选项 |
Type Ⅲ系统的特征蛋白为Cas10,Cas10蛋白包含Palm结构域、环化酶结构域、HD核酸内切酶结构域[16-17]。尽管Type Ⅰ和Type Ⅲ效应复合体的亚基组成在氨基酸序列上相似性较低,但是两者在空间结构上却惊人地相似。Ⅰ型和Ⅲ型CRISPR系统的功能发挥依赖于精密且复杂的效应蛋白复合体,其基本骨架由重复序列关联蛋白(Repeat-associated mysterious proteins,RAMPs)、RNA识别基序(RNA recognition motif,RRM)、大亚基和小亚基组成[18-19]。骨架中的RAMPs由多拷贝的Cas7蛋白和单拷贝的Cas5蛋白组成,形成一种半螺旋的新月形结构,这一结构便于容纳指导RNA (gRNA)。在效应物复合体中Cas5亚基结合在crRNA的5′端柄状区域并与大亚基相互作用(大亚基的成分,Ⅰ型中是Cas8,Ⅲ型的是Cas10),小亚基则与多拷贝的Cas7结合[20]。结构完整的效应蛋白复合体在gRNA的引导下结合靶核酸序列,进而招募Cas3蛋白行使靶位点切割的功能。Type Ⅳ系统的组成与Type Ⅲ-B接近,缺少Cas1和Cas2蛋白,并且常常缺失CRISPR阵列(CRISPR array)[10]。该系统的效应复合体由Csf1、Cas5和Cas7蛋白组成,其特征蛋白为Csf 1。
与Class 1不同的是,Class 2成员的效应模块只含有一个大分子量、多结构域的效应蛋白单体。基于效应蛋白的不同,Class 2系统分为Type Ⅱ、Type Ⅴ和Type Ⅵ,共计9个亚型,部分亚型包括一系列假定的系统,其负责免疫或免疫监视的功能还有待证明。从现有的CRISPR/Cas系统来看,Class 2倾向于分布在细菌域中,其中经典的Type Ⅱ系统包含3个亚型,效应蛋白为Cas9[21-24]。该效应蛋白被广泛应用于基因组编辑领域,是CRISPR/Cas系统中的明星成员。与Type Ⅱ效应蛋白相似,Type Ⅴ中效应蛋白是Cpf1,整个系统的其他组成元件有Cas1、Cas2和CRISPR array[25]。Cpf1是一种依赖于RNA指导的核酸内切酶,约由1300个氨基酸构成,包含RuvC样核酸酶结构域和IS605转座子家族的TnpB功能域[26]。与Cas9不同的是,Cpf1不需要附加的反式激活crRNA (Trans-activating RNA,tracrRNA)便可实现靶位点的切割,这意味着Cpf1的系统更简洁,用于基因编辑操作更方便。并且Cpf1切割靶DNA产生的是具5′突出的粘性末端,更易于引入插入突变,因此Cpf1具有成为新一代基因编辑工具的潜力。此外,张峰在2016年发现沙氏纤毛菌(Leptotrichia shahii)中存在一种新型的CRISPR/Cas系统——Type Ⅵ,在该系统中的效应蛋白Cas13a (C2c2) 具有RNA介导的RNA酶活性。在Cas13a中含有2个HEPN结构域,其活性用于切割靶RNA,这一发现为RNA编辑提供了一种新的工具。除效应蛋白质外,Class 2中的大部分CRISPR基因位点编码适应模块蛋白如Cas1、Cas2及Cas4。但仍有一些成员缺失此类蛋白,如Type Ⅵ,只由一个CRISPR array和一个效应蛋白组成。并且在Type Ⅴ中还存在一些功能未经证实的CRISPR/Cas成员,如c2c4、c2c5、c2c8、c2c9、c2c10等。这些潜在的成员往往含有分子量低的效应蛋白,小分子量效应蛋白可能在未来基因编辑工具的开发过程中更具优势。
2 CRISPR/Cas系统分子机制 作为一种在细菌和古菌中普遍存在的获得性免疫防御体系,CRISPR/Cas系统在执行功能时主要经过3个阶段:免疫获得阶段(Adaptation)、表达阶段(Expression)和免疫干扰阶段(Interference)[27] (图 2)。在免疫获得阶段,Cas蛋白通过识别入侵的核酸序列获取新的间隔序列(Spacer),并将其整合到CRISPR array中,形成免疫记忆;在表达阶段,CRISPR array转录出crRNA前体并成熟为crRNA,结合表达的效应蛋白Cas上形成crRNA-Cas复合体;在免疫干扰阶段,crRNA-Cas蛋白复合体搜寻外源核酸,通过crRNA靶向目标区域,从而实现对外源核酸的切割。
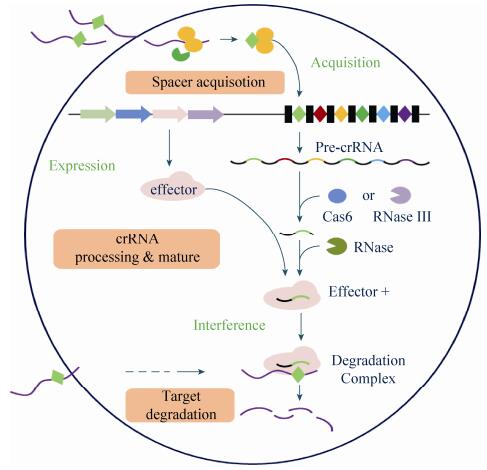 |
| 图 2 CRISPR/Cas系统分子机制 Figure 2 The molecular mechanism of CRISPR/Cas. |
| 图选项 |
2.1 免疫获得阶 段免疫获得(Adaptation)是对入侵核酸片段进行捕获和采集,使得CRISPR/Cas系统获得免疫记忆的过程。在此过程中,Cas1和Cas2蛋白扮演着重要的角色,其中具有DNA核酸酶活性的Cas1蛋白对于新spacer的插入是至关重要的[9]。以来自于大肠杆菌的Ⅰ-E Subtype系统为例,新spacer的插入是一个多步反应过程(图 3)。首先在Cas1-Cas2蛋白复合体的催化下,插入片段PAM区的3′端OH基团发生定向亲核攻击,使之结合在CRISPR array第一个重复序列的5′端。随后插入片段发生翻转,暴露的3′端羟基亲核攻击重复序列另一侧的5′端区域,使得插入序列进入到CRISPR array第一个重复区之后。最后补齐spacer插入过程中产生的缺口,完成外源核酸的免疫获得过程[28]。在Ⅱ-A Subtype系统中,需要其他Cas蛋白参与免疫获得过程,包括Csn2和Cas9蛋白。其中,Csn2用以结合双链DNA,辅助spacer的插入;Cas9负责识别外源核酸片段中的PAM区,利用其核酸内切酶的活性实现免疫获得。并且相关实验表明,Cas9在识别自己和异己核酸的过程中扮演着重要角色[23]。基于Csn2参与免疫获得这一现象,可以推测在不同的CRISPR/Cas系统中还有许多类似的蛋白参与这一过程。按照现有CRISPR/Cas系统的统计数据,7%的Cas1蛋白存在着融合其他蛋白的现象,融合的蛋白类型有Cas4、Csx1、COG4951家族的Ⅲ型限制性核酸内切酶、COG2378家族的转录调控因子、逆转录酶及Argonaute蛋白。其中最常见的组合类型是Cas1-Cas4,Cas4含有RecB样核酸外切酶结构域,暗示着其可能参与原型间隔序列(Protospacer)的加工过程。
 |
| 图 3 CRISPR/Cas系统免疫获得途径 Figure 3 The adaptation pathway of CRISPR/Cas. |
| 图选项 |
2.2 表达阶段 在CRISPR/Cas系统的表达阶段中,CRISPR arrays会转录形成一个长的转录本,称之为crRNA前体(pre-crRNA)。pre-crRNA经过加工、成熟,转变为有活性的crRNA,该过程在不同类型的CRISPR/Cas系统中是不同的。以Type Ⅱ系统为例,crRNA加工过程由细胞中常见的RNase Ⅲ在tracrRNA的辅助下对pre-crRNA进行加工,这种利用细胞内通用蛋白的方式体现了微生物基因编码产物的通用、经济原则。但也存在例外,在Type Ⅱ的部分成员中存在独立的crRNA转录系统,可以直接转录出具有生理活性的crRNA。在Type Ⅴ-A和Type Ⅵ系统中,这一过程的实现不依赖tracrRNA,而是由各自的效应蛋白独立完成crRNA的成熟[29]。Type Ⅴ-B在crRNA的加工和靶DNA切割的过程中仍依赖于tracrRNA,并且也需要RNase Ⅲ参与crRNA的成熟。但其中的详细机制现在还不清楚,还有待于进一步研究。
2.3 免疫干扰阶段
2.3.1 Type Ⅰ系统干扰机制:在Type Ⅰ系统中,干扰过程是由CRISPR抗病毒防御复合体(CRISPR-associated complex for antiviral defense,Cascade)和Cas3蛋白协同实现的。成熟的crRNA结合在Cascade蛋白复合体上使之与目标DNA序列结合,目标DNA在Cascade蛋白复合体的作用下完成解螺旋。随后,Cas3蛋白结合到目标DNA区域,利用其被激活的HD核酸酶功能域和解旋酶功能域对靶DNA序列进行切割[7]。在Type Ⅰ所有亚型中,各个成员的Cascade复合体的亚基都各不相同,Cas3蛋白也存在着差异。以来自于大肠杆菌的Cascade复合体为例,其包含分子数不同的5种蛋白,分别是Cse1、Cse2、Cas5、Cas7和Cas6,Cas7蛋白沿着crRNA的方向聚集和排列,形成新月形、半螺旋的立体结构[30]。这一结构作为Cascade复合体的骨架,便于招募其他成员,其中Cas6 (在Ⅰ-E型系统中是Cas6e)结合在crRNA的3′端发卡处,Cas5则结合在靠近5′端的柄部。小亚基通常以双拷贝的形式出现,并且结合在Cascade复合体的腹部,有利于crRNA和目标DNA的稳定[31]。大亚基(在亚型Ⅰ-E中是Cse1,在其他大多数亚型中是Cas8) 结合在crRNA的5′端(图 4-A)。干扰的初始阶段,Cascade蛋白复合体利用Cse1 (大亚基)识别外源DNA的PAM区段,随后,临近PAM的prospacer区域开始解链,形成R型的结构[32]。crRNA与靶DNA结合,形成一个稳定但结构扭曲的crRNA-靶DNA复合体。Cascade复合体在结合靶DNA后会发生构象的变化,使之有能力招募Cas3蛋白与Cse1亚基结合,对靶DNA实行切割。
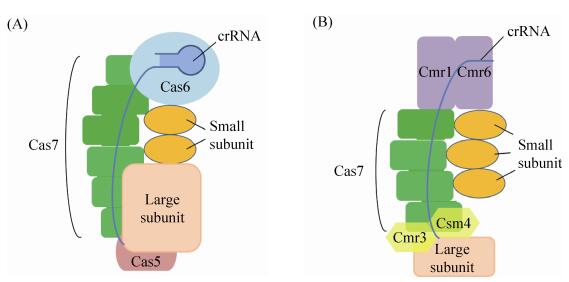 |
| 图 4 Type Ⅰ和Type Ⅲ效应复合体 Figure 4 The effect complex of Type Ⅰ (A) and Type Ⅲ (B). |
| 图选项 |
2.3.2 Type Ⅲ CRIPSR/Cas系统干扰机制:Type Ⅲ系统的效应蛋白复合体与Type Ⅰ中Cascade复合体在空间结构上非常相似。以Subtype Ⅲ-A、Subtype Ⅲ-B为例,Csm5、Cmr6和Cmr1与Type Ⅰ中的Cas7的骨架作用相似,crRNA以6碱基扭曲的方式预先结合在复合体上,靶核酸序列(既有DNA又有RNA)也以扭曲的方式结合在复合体上。Cmr3和Csm4结合在crRNA 5′端柄状结构,随后Cas10充当大亚基的角色结合在crRNA上(图 4-B),进而行使靶核酸的降解功能[33]。非常有趣的是Type Ⅲ CRISPR/Cas系统不仅可以靶向降解DNA,还可以降解RNA,例如表皮葡萄球菌(Staphylococcus epidermidis)中的Type Ⅲ系统。从此角度来看,Type Ⅲ系统为分子遗传操作提供了一种潜在的编辑转录本的可能。
2.3.3 Type Ⅱ CRIPSR/Cas系统干扰机制:与Type Ⅰ和Type Ⅲ需要形成复杂的效应蛋白复合体来实现干扰的机制不同,Type Ⅱ CRISPR/Cas的标志蛋白Cas9以单个蛋白的形式,在gRNA的引导下完成对靶基因的切割。以酿脓链球菌(Streptococcus pyogenes) Cas9蛋白为例,其含有两种核酸内切酶结构域,HNH结构域和RuvC结构域(图 5)。前者负责对目标链的切割,后者负责目标链的互补链的切割,切割位点位于PAM序列5′-NGG上游3 bp,形成DNA双链断裂的平末端,实现对靶DNA的裂解。同时,RuvC结构域除了具有内切酶活性外,还具有3′至5′的外切酶活性,能将非靶序列从切口降解5个碱基左右[34]。Type Ⅱ的干扰机制简单且高效,被广泛应用于真核及原核生物的基因编辑领域中,从而引发了新一轮基因编辑的热潮。
 |
| 图 5 Type Ⅱ、Type Ⅴ和Type Ⅵ效应蛋白结构域 Figure 5 The effect protein domain of Type Ⅱ, Type Ⅴ and Type Ⅵ. |
| 图选项 |
2.3.4 其他类型CRIPSR/Cas系统干扰机制:Type Ⅴ系统在结构上与Type Ⅱ有很大的相似性,目前Type Ⅴ系统依据不同的效应蛋白分为Subtype Ⅴ-A、Subtype Ⅴ-B和Subtype Ⅴ-C,其相应的效应蛋白分别是Cpf1、C2c2和C2c3[35]。从进化程度来看,在进化地位上来自转座子关联蛋白家族TpnB,含有C-端的RuvC结构域和富含精氨酸的桥螺旋。其中,Ⅴ-B是tracrRNA依赖型,而Ⅴ-A和Ⅴ-C则缺少tracrRNA和Cas6或Cas5相似蛋白,这两种类型的crRNA加工机制还不清楚。以弗朗西斯氏菌(Francisella novicida)中的Ⅴ-A效应蛋白为例,Cpf1通过识别靶DNA 5′端TTN为特征的PAM区,将DNA双链断裂形成5′端具有5个碱基的突出末端[36]。Cpf1是以二聚体的形式行使功能,但每个蛋白只有1个RuvC功能域在行使功能,这一谜题还需要在后续的研究中来解答。在Type Ⅵ的效应蛋白中(如Cas13),目前鉴定出来有两个HEPN结构域(图 5),它与Cas9和Cas12中的RuvC-like结构域存在着很大的差别,这是一个具有RNase活性的结构域。由此可见,Type Ⅵ系统是专性靶向RNA。另外,在细胞中表达Cas13会导致一定的细胞毒性,这表明该系统可能与细菌的程序性死亡有关。
3 CRISPR/Cas系统的应用 3.1 传统遗传学中的应用 传统遗传学的研究可整体概括为两大方向,正向遗传学和反向遗传学。其中正向遗传学是通过表型差异来探究基因差异,反向遗传学则是通过基因型的变化来研究表型的差异,两者均需要以基因突变为物质基础。CRISPR/Cas系统作为强有力的基因突变工具,为传统遗传学的研究提供一条新途径。实际应用中,将tracrRNA-crRNA复合体融合成单链指导RNA (Single guide RNA,sgRNA),提高了操作性和可控性。在sgRNA中含有保守区和可变区,其中可变区负责以Watson-Crick的方式识别靶位点序列,进而在Cas9的作用下实现切割。被切割后的DNA依靠细胞内源性的修复途径进行修复,引入突变。内源性的修复类型包括非同源重组末端修复(Non-homologous end joining,NHEJ)和同源重组修复(Homologous directed repair,HDR)[37],修复方式的选择依赖于相应修复系统的存在与完整与否,细胞周期处于的时相及是否存在同源模板等因素。其中,NHEJ修复反式倾向于向DNA断裂处引入随机突变(插入和缺失),对于功能基因而言,如果插入和缺失的碱基数目恰好是密码子碱基数目的整数倍数,往往会形成无义突变,这对于遗传学的研究是不利的。相比较而言,HDR修复方式可依赖于人为提供的同源区段(Donor)来实现精确的基因组修饰,在遗传学的研究中常基于这种策略来实现单个或多个基因的敲除或重组。值得注意的是,基于CRISPR/Cas系统本身的缺陷,在实验中仍存在许多问题,诸如基因编辑精度,存在脱靶效应等等。为解决这些问题,科研工作者开发了一系列的方法:(1) 通过对Cas9核酸内切酶活性区域RuvC D10A或HNH H840A实施突变,产生具有单链切割活性的缺口酶(Cas9 nickase,Cas9n)[38],利用成对的Cas9n在各自的sgRNA指导下对靶DNA实现切割,可以有效地减少错配的产生;(2) 利用sgRNA工程,精简sgRNA的长度,可显著降低Cas9核酸内切酶的脱靶效应;(3) 通过对Cas9中RuvC和HNH结构域同时失活,形成dCas9 (Dead Cas9),利用内切酶活性缺失的dCas9融合FokI核酸酶[39],形成类似于ZFN/TALEN的核酸酶工具,来提高Cas9系统的编辑精度。这些方法在反向遗传学的研究中,为探究基因突变型与表型的关系提供了一系列可靠的遗传操作工具,成功应用在哺乳动物细胞、斑马鱼、隐秀丽线虫、酵母、放线菌和大肠杆菌等细胞类型中,加快了相应方向的科研进程。
在正向遗传学的研究中,传统的策略是利用物理或化学诱变方法产生的随机突变体库,用以研究特定表型的基因型组成。诱变往往存在热点效应和疲劳效应,且突变点定位困难。针对这一现象,开发出了基于CRISPR/Cas系统的全基因组功能基因筛查模型,利用可以覆盖全基因组的sgRNA库构建全基因组突变文库,在筛查表型和基因型之间的关联时可以方便快捷地利用突变体含有的sgRNA进行突变基因定位,解决了传统诱变策略的弊端,为正向遗传学提供了有力的技术手段,具有广阔应用前景。
3.2 表观遗传学中的应用 CRISPR/Cas系统在表观遗传学的研究应用中,往往侧重于Cas蛋白的DNA特异结合的性质,利用内切酶活性缺失的dCas9融合多种效应基团来实现基因的转录调控功能和染色体修饰功能。例如,利用dCas9融合Kruppel-associated box (KRAB),在sgRNA的引导下可实现对靶基因的表达抑制。此外还有一些可以实现基因表达调控的元件,如dCas9融合4个拷贝的转录激活因子VP16,或融合单个拷贝的p65转录激活蛋白的结构[40]。同时,可通过设计gRNA Scaffolds来调整染色体的结构,进而实现相应基因的转录激活或转录抑制,并且通过利用dCas9融合乙酰转移酶p300或DNA甲基化酶(DNMT3A),可实现对染色体的定点乙酰化修饰和甲基化修饰[41-42](图 6)。CRISPR/Cas系统的引入使得精准表观遗传操作成为可能,这将为表观遗传研究的深入开展提供有力的技术支持。
 |
| 图 6 CRISPR/Cas技术在表观遗传学中的应用 Figure 6 The epigenetics application of CRISPR/Cas technology. |
| 图选项 |
在利用依赖sgRNA指导的Cas9蛋白实施的基因编辑研究中发现,当sgRNA长度小于16 bp时Cas9蛋白的切割活性便会受到抑制[43]。根据这一现象,通过设计截短后的sgRNA,可以实现对靶基因的转录抑制,而不会引起对靶DNA的裂解。并且,这一方法可以通过简单替换全长的sgRNA和截短的sgRNA来实现Cas9介导的基因敲除和表达调控之间轻松转换,从而避免了在传统方法中为实现Cas9介导的基因编辑和基因调控时而反复进行sgRNA的设计和组装。
以上对于转录水平的调控,是通过抑制或促进转录发生而实现的,没有涉及到RNA的加工。实际上,在CRISPR/Cas家族是存在以RNA为靶向的成员的,如来自细菌Leptotrichia shahii中的C2c2蛋白,可以实现RNA靶序列的特异切割。并且开发出缺失核糖核酸酶活性的dC2c2系统,用以实现Knock down[44]。利用RNA靶向的基因调控,也许比DNA靶向类型更安全。并且,通过发掘和鉴定自然界中新的CRISPR/Cas系统,有助于发展RNA靶向的编辑系统,相信在不久的将来会有越来越多的CRISPR/Cas新成员加入,从而将基因编辑及表达调控推向一个更高的阶段。
4 展望 目前已知的CRISPR/Cas系统共有六型,其中研究最为深入和应用最为广泛的是Type Ⅱ系统。但以Cas9为特征效应蛋白的Type Ⅱ系统本身也存在着缺陷,例如需要识别特殊的PAM区来对靶DNA实施切割。这对于不包含PAM区的DNA区段难以利用Cas9实现精确编辑,无法做到对基因组的完整覆盖,这在CRISPR/Cas系统早期的研究中是无法解决的。随着新型CRISPR/Cas系统不断被发现,尤其是Ⅴ型的CRISPR/Cpf1系统,其实现DNA切割功能所依赖的PAM区存在的位置和其中的核苷酸组成均与Cas9系统有着很大的差别,与Cas9系统相互辅助,可实现更全面覆盖基因组上可编辑位点。因此,不断发掘新型CRISPR/Cas系统资源对于解决现有系统的弊端,无疑是一条有效的途径。按照这一设想,可以从两种策略来实现对新型CRISPR/Cas系统资源的勘探和获取。其一是基于免培养的宏基因组策略(例如2017年Banfield课题组以此策略发现了CasX和CasY系统[45]),利用该策略可实现大范围、高通量的环境微生物基因组信息的调查和采集,从中发现新资源;其二是基于纯培养策略,利用不断优化的微生物分离手段,在生命暗物质中捕获新的类群,进而研究其依存的CRISPR/Cas系统。这两种策略相辅相成,其中免培养策略可以方便快捷地对环境样本的基因信息进行发掘并可指导纯培养手段的改进;纯培养策略可以在免培养无法实现的微生物细胞的生理条件下研究新型CRISPR/Cas系统的特性和作用机制。目前,本课题组正依托对全球范围内的热泉、盐湖、沙漠等极端生境样本的采集,积累了大量的宏基因组数据和种类繁多的新微生物类群,并在此基础上建立了CRISPR/Cas系统资源发掘平台。通过不断丰富平台中免培养和纯培养的信息资源,以期发现新的具有更好应用潜质的CRISPR/Cas系统,为分子遗传研究提供助力。
References
| [1] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [2] | Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(Pt 8): 2551-2561. |
| [3] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3): 653-663. DOI:10.1099/mic.0.27437-0 |
| [4] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology, 2010, 64: 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [5] | Marraffini LA, Sontheimer EJ. CRISPR interference:RNA-directed adaptive immunity in bacteria and archaea. Nature Reviews Genetics, 2010, 11(3): 181-190. DOI:10.1038/nrg2749 |
| [6] | Koonin EV, Makarova KS. CRISPR-Cas:evolution of an RNA-based adaptive immunity system in prokaryotes. RNA Biology, 2013, 10(5): 679-686. DOI:10.4161/rna.24022 |
| [7] | Makarova KS, Wolf YI, Koonin EV. The basic building blocks and evolution of CRISPR-Cas systems. Biochemical Society Transactions, 2013, 41(6): 1392-1400. DOI:10.1042/BST20130038 |
| [8] | Barrangou R, Marraffini LA. CRISPR-Cas systems:prokaryotes upgrade to adaptive immunity. Molecular Cell, 2014, 54(2): 234-244. DOI:10.1016/j.molcel.2014.03.011 |
| [9] | Yosef I, Goren MG, Qimron U. Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli. Nucleic Acids Research, 2012, 40(12): 5569-5576. DOI:10.1093/nar/gks216 |
| [10] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [11] | Sampson TR, Weiss DS. CRISPR-Cas systems:new players in gene regulation and bacterial physiology. Frontiers in Cellular and Infection Microbiology, 2014, 4: 37. |
| [12] | Sinkunas T, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. The EMBO Journal, 2011, 30(7): 1335-1342. DOI:10.1038/emboj.2011.41 |
| [13] | Gong B, Shin M, Sun JL, Jung CH, Bolt EL, van der Oost J, Kim JS. Molecular insights into DNA interference by CRISPR-associated nuclease-helicase Cas3. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(46): 16359-16364. DOI:10.1073/pnas.1410806111 |
| [14] | Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biology Direct, 2011, 6: 38. DOI:10.1186/1745-6150-6-38 |
| [15] | Vestergaard G, Garrett RA, Shah SA. CRISPR adaptive immune systems of Archaea. RNA Biology, 2014, 11(2): 156-167. DOI:10.4161/rna.27990 |
| [16] | Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV. A putative RNA-interference-based immune system in prokaryotes:computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biology Direct, 2006, 1(1): 7. DOI:10.1186/1745-6150-1-7 |
| [17] | Makarova KS, Aravind L, Grishin NV, Rogozin IB, Koonin EV. A DNA repair system specific for thermophilic Archaea and bacteria predicted by genomic context analysis. Nucleic Acids Research, 2002, 30(2): 482-496. DOI:10.1093/nar/30.2.482 |
| [18] | Haft DH, Selengut J, Mongodin EF, Nelson KE. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. PLoS Computational Biology, 2005, 1(6): e60. DOI:10.1371/journal.pcbi.0010060 |
| [19] | Samai P, Pyenson N, Jiang WY, Goldberg GW, Hatoum-Aslan A, Marraffini LA. Co-transcriptional DNA and RNA cleavage during type Ⅲ CRISPR-Cas immunity. Cell, 2015, 161(5): 1164-1174. DOI:10.1016/j.cell.2015.04.027 |
| [20] | Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biology Direct, 2011, 6: 38. DOI:10.1186/1745-6150-6-38 |
| [21] | Chylinski K, Makarova KS, Charpentier E, Koonin EV. Classification and evolution of type Ⅱ CRISPR-Cas systems. Nucleic Acids Research, 2014, 42(10): 6091-6105. DOI:10.1093/nar/gku241 |
| [22] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [23] | Heler R, Samai P, Modell JW, Weiner C, Goldberg GW, Bikard D, Marraffini LA. Cas9 specifies functional viral targets during CRISPR-Cas adaptation. Nature, 2015, 519(7542): 199-202. DOI:10.1038/nature14245 |
| [24] | Wei YZ, Terns RM, Terns MP. Cas9 function and host genome sampling in type Ⅱ-A CRISPR-Cas adaptation. Genes & Development, 2015, 29(4): 356-361. |
| [25] | Schunder E, Rydzewski K, Grunow R, Heuner K. First indication for a functional CRISPR/Cas system in Francisella tularensis. International Journal of Medical Microbiology, 2013, 303(2): 51-60. DOI:10.1016/j.ijmm.2012.11.004 |
| [26] | Makarova KS, Koonin EV. Annotation and classification of CRISPR-Cas systems//Lundgren M, Charpentier E, Fineran PC. CRISPR:Methods and Protocols. New York, NY: Humana Press, 2015: 47-75. |
| [27] | Marraffini LA. CRISPR-Cas immunity in prokaryotes. Nature, 2015, 526(7571): 55-61. DOI:10.1038/nature15386 |
| [28] | Arslan Z, Hermanns V, Wurm R, Wagner R, Pul ü. Detection and characterization of spacer integration intermediates in type Ⅰ-E CRISPR-Cas system. Nucleic Acids Research, 2014, 42(12): 7884-7893. DOI:10.1093/nar/gku510 |
| [29] | Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [30] | Hochstrasser ML, Taylor DW, Bhat P, Guegler CK, Sternberg SH, Nogales E, Doudna JA. CasA mediates Cas3-catalyzed target degradation during CRISPR RNA-guided interference. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(18): 6618-6623. DOI:10.1073/pnas.1405079111 |
| [31] | Jackson RN, Golden SM, van Erp PBG, Carter J, Westra ER, Brouns SJJ, van der Oost J, Terwilliger TC, Read RJ, Wiedenheft B. Crystal structure of the CRISPR RNA-guided surveillance complex from Escherichia coli. Science, 2014, 345(6203): 1473-1479. DOI:10.1126/science.1256328 |
| [32] | Rutkauskas M, Sinkunas T, Songailiene I, Tikhomirova MS, Siksnys V, Seidel R. Directional R-loop formation by the CRISPR-Cas surveillance complex cascade provides efficient off-target site rejection. Cell Reports, 2015, 10(9): 1534-1543. DOI:10.1016/j.celrep.2015.01.067 |
| [33] | Osawa T, Inanaga H, Sato C, Numata T. Crystal structure of the CRISPR-Cas RNA silencing Cmr complex bound to a target analog. Molecular Cell, 2015, 58(3): 418-430. DOI:10.1016/j.molcel.2015.03.018 |
| [34] | Nishimasu H, Cong L, Yan WX, Ran FA, Zetsche B, Li YQ, Kurabayashi A, Ishitani R, Zhang F, Nureki O. Crystal structure of Staphylococcus aureus Cas9. Cell, 2015, 162(5): 1113-1126. DOI:10.1016/j.cell.2015.08.007 |
| [35] | Shmakov S, Abudayyeh OO, Makarova KS, Wolf YI, Gootenberg JS, Semenova E, Minakhin L, Joung J, Konermann S, Severinov K, Zhang F, Koonin EV. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell, 2015, 60(3): 385-397. DOI:10.1016/j.molcel.2015.10.008 |
| [36] | Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [37] | Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [38] | Standage-Beier K, Zhang Q, Wang X. Targeted large-scale deletion of bacterial genomes using CRISPR-nickases. ACS Synthetic Biology, 2015, 4(11): 1217-1225. DOI:10.1021/acssynbio.5b00132 |
| [39] | Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nature Biotechnology, 2014, 32(6): 577-582. DOI:10.1038/nbt.2909 |
| [40] | Gilbert LA, Larson MH, Morsut L, Liu ZR, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [41] | Hilton IB, D'ippolito AM, Vockley CM, Thakore PI, Crawford GE, Reddy TE, Gersbach CA. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology, 2015, 33(5): 510-517. DOI:10.1038/nbt.3199 |
| [42] | Vojta A, Dobrini? P, Tadi? V, Bo?kor L, Kora? P, Julg B, Klasi? M, Zoldo? V. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research, 2016, 44(12): 5615-5628. DOI:10.1093/nar/gkw159 |
| [43] | Dahlman JE, Abudayyeh OO, Joung J, Gootenberg JS, Zhang F, Konermann S. Orthogonal gene knockout and activation with a catalytically active Cas9 nuclease. Nature Biotechnology, 2015, 33(11): 1159-1161. DOI:10.1038/nbt.3390 |
| [44] | Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DBT, Shmakov S, Makarova KS, Semenova E, Minakhin L, Severinov K, Regev A, Lander ES, Koonin EV, Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [45] | Burstein D, Harrington LB, Strutt SC, Probst AJ, Anantharaman K, Thomas BC, Doudna JA, Banfield JF. New CRISPR-Cas systems from uncultivated microbes. Nature, 2017, 542(7640): 237-241. |
