全文HTML
--> --> -->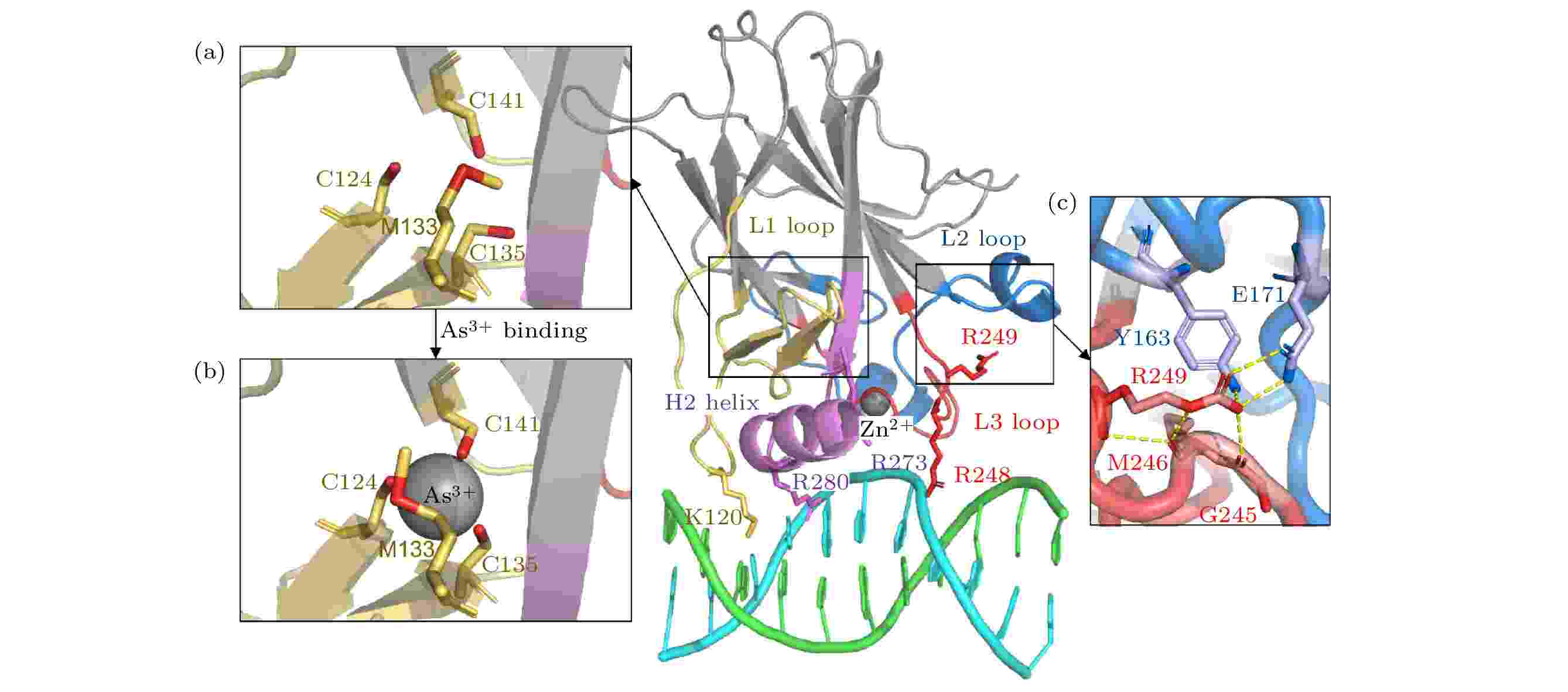 图 1 p53C蛋白晶体结构 (a)砷(III)结合位点没有砷(III)结合时的局部细节; (b)砷(III)结合位点在砷(III)结合时的局部细节; (c) R249残基与相邻区域相互作用的局部示意图. 颜色标注如下: L1环及相邻区域(残基序列112—141)为黄色, L2环及相邻区域(残基序列163—195)为蓝色, L3环(残基序列236—251)为红色, H2螺旋(残基序列271—286)为紫色, DNA为青色/绿色. 除非注明, 后图颜色标注一致
图 1 p53C蛋白晶体结构 (a)砷(III)结合位点没有砷(III)结合时的局部细节; (b)砷(III)结合位点在砷(III)结合时的局部细节; (c) R249残基与相邻区域相互作用的局部示意图. 颜色标注如下: L1环及相邻区域(残基序列112—141)为黄色, L2环及相邻区域(残基序列163—195)为蓝色, L3环(残基序列236—251)为红色, H2螺旋(残基序列271—286)为紫色, DNA为青色/绿色. 除非注明, 后图颜色标注一致Figure1. Structure of p53C: (a) The structure of As3+-binding region in the absence of As3+; (b) the structure of As3+-binding region with As3+; (c) R249 region in detail. It is colored as following: L1 loop and adjacent area (residues 112–141) in yellow, L2 loop and adjacent area (residues 163–195) in blue, L3 loop (residues 236–251) in red, H2 helix (residues 271–286) in purple and DNA in cyan/green. Unless otherwise specified, the colored regions in following figures are consistent with this.
研究发现, 蛋白中的多个突变或者小分子结合能够恢复p53的活性[5]. 最新报道显示, 三价砷(As)与包括R249S在内的多种p53突变型的结合具有恢复蛋白分子活性的变构调节作用[12](图1(a)和图1(b)). 变构作用引发的动力学性质的差异可能是砷(III)结合恢复p53蛋白活性的关键, 其中的分子动力学等物理机制尚不清楚. 分子动力学模拟是探索蛋白质动力学特征的强大计算工具, 可以为蛋白-配体相互作用引起的结构变化提供理论见解[13]. 基于主成分分析的理论研究表明, 蛋白质的构象波动中存在高度集中的太赫兹频率群体运动模式[14]. 太赫兹光谱技术的发展使得测量生物分子在太赫兹频率的低频振动成为可能, 为研究蛋白-配体相互作用提供了重要手段[15-17]. 基于弹性网络模型的正则模式分析有效地阐明了蛋白分子的低频集体振动[18,19], 以解释配体结合引起的低频振动变化[20]. 从太赫兹生物物理的角度揭示砷(III)对p53蛋白活性的恢复拯救机制, 有利于深入理解蛋白分子功能与太赫兹频率集体运动的关系.
本文对一系列蛋白分子的集体振动进行了分析, 计算了蛋白分子的低频率振动模式, 以研究砷(III)结合引起的R249S突变型p53C蛋白DNA结合域的低频振动模式恢复. 此外, 通过原子骨架波动和太赫兹频率振动模态统计分析了p53蛋白分子活性恢复的运动特征. 本研究为配体结合诱导的蛋白分子活性功能恢复提供了一种可能的物理机制阐释, 为蛋白分子的太赫兹频率运动与DNA结合活性之间的动力学关系提供了新的视角.
2.1.野生型、突变体和As结合突变体p53的结构
从蛋白质数据库(PDB)[21]中获得了未结合DNA的自由状态p53核心区域(p53C)的结构, 其中包括野生型p53 (PDB: 1TSR A), R249S突变体 (PDB: 3D06), 砷(III)结合的R249S突变体(PDB: 7DHZ), 截取其中核心区域, 即97—287残基用于模拟. 在计算前, 用Modeler软件(版本9.24)[22]补全原始结构中缺失的残基, 并用PdbViewer软件(版本4.1)[23]补全其余缺失原子. 原始结构中缺失的残基不包含本文中特殊标记出的残基.2
2.2.正则模式计算
首先评估了不同p53结构导致低频振动的差异. 通常, 正则模式分析需要通过对从PDB中获得的p53结构进行扭曲, 使用能量最小化的初始结构. 我们使用了弹性网络模型, 该模型允许使用PDB中获得的原始结构作为初始结构. 为了更精确地展示特定残基中可能存在的重原子间相互作用, 采用了Yao等[24]开发的全原子弹性网络模型(all-atom elastic network model, AAENM). 与其他粗粒度方法相比, AAENM模型包括了所有的重原子. 这一基于全原子模型的结果与基于全经验力场计算得到的正则模式有较好的一致性. 所有正则模式计算的步骤均使用基于R语言的Bio3D软件包[25]实现.2
2.3.分 析
本文中所有可视化分析使用PyMOL软件绘制, 其中, 用α碳原子的轨迹表示各残基的轨迹. 为了更好地可视化p53的运动轨迹, 各运动模态的特征向量被放大了2.5倍.原子波动统计了所有正则振动模式, 并按照下式计算[26]:
这里按照Kaynak和Doruker[27]的方法分析了正则模式频率的移动. 结构a相对于结构b的第i个正则振动模式的频率偏移百分数fs(i)按下式计算:
为比较R249S和R249S-As的频率偏移百分数, 这里定义第i个正则振动模式的频率恢复比例fr(i), 按下式计算:
3.1.砷(III)对p53C突变蛋白最低频率振动模式的恢复作用
我们使用基于ENM的计算展示了平衡位置附近的野生型p53C (Wild type, WT), R249S和R249S-As的最低频率集体振动模式. 如图2(a)所示, 野生型p53C的DNA结合域总体具有较小的振动幅度, L1环和H2螺旋区域几乎在最低振动频率保持刚性, L2环和L3环的振动特征值与整个p53C结构相比较小. 这种太赫兹频段的最低频率振动模式或许有助于p53C的DNA结合域与DNA结构分子对接的拓扑结构, 使其可以延伸到DNA的结合沟槽中. 由于R249的突变会破坏与相邻残基的氢键和配位结构, R249S的最低频率振动模式表现出显著的变化(图2(b)): L1环显示了较大的摆动运动幅度, 而H2螺旋的振动在R249S中很明显. 相比之下, L2环和L3环在振动幅度上的增加较小. 总之, R249S突变使p53C的DNA结合域最低频率振动幅度总体增加, 意味着突变导致了蛋白分子内部的太赫兹频率运动模式的变化, 蛋白分子的柔性构象变化反映了DNA结合能力的下降.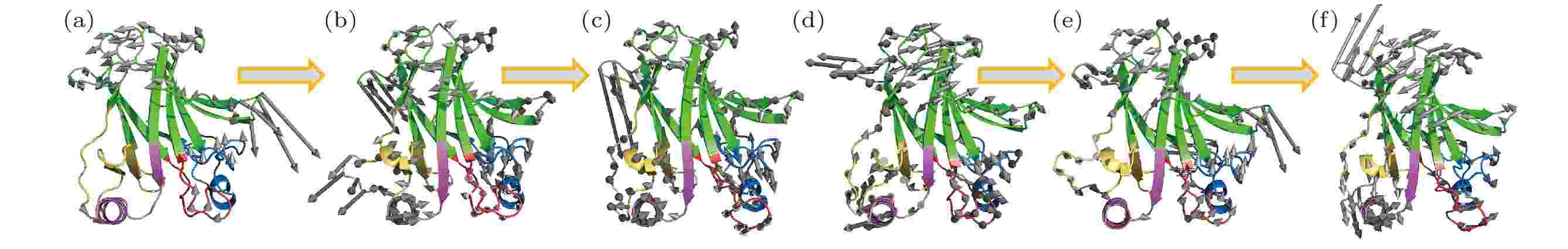 图 2 砷(III)对p53C突变体R249S的低频振动模式恢复 (a) 野生型p53C (wild type,WT) 、(b) R249S突变型p53C (R249S)、(c) 砷(III)结合的R249S突变型p53C的最低频率集体振动模式轨迹; (d)野生型p53C (wild type,WT) 、(e) R249S突变型p53C (R249S)、(f) 砷(III)结合的R249S突变型p53C的次低频率集体振动模式轨迹; 灰色箭头表示对应频率蛋白分子骨架的振动方向, 箭头长度代表相对振幅大小
图 2 砷(III)对p53C突变体R249S的低频振动模式恢复 (a) 野生型p53C (wild type,WT) 、(b) R249S突变型p53C (R249S)、(c) 砷(III)结合的R249S突变型p53C的最低频率集体振动模式轨迹; (d)野生型p53C (wild type,WT) 、(e) R249S突变型p53C (R249S)、(f) 砷(III)结合的R249S突变型p53C的次低频率集体振动模式轨迹; 灰色箭头表示对应频率蛋白分子骨架的振动方向, 箭头长度代表相对振幅大小Figure2. The trajectory of the lowest-frequency vibrations of (a) wild type (WT), (b) R249S, (c) R249S-As. The trajectory of the second lowest-frequency vibrations of (d) wild type (WT), (e) R249S, (f) R249S-As. Arrows represent the direction of motion of each residue from the initial position, with length of arrows indicating vibration amplitude, relatively.
随后, 我们检验了R249S-As的最低频率振动模式. 在最近关于三氧化二砷(III)恢复p53突变蛋白活性的研究中, 认为砷(III)是通过对相邻残基的变构作用在结构上稳定p53C来恢复蛋白活性功能的[12]. 为此, 我们研究了砷(III)结合的R249S突变型p53C (R249S-As)的最低频率运动特征, 以探索小分子结合恢复蛋白功能的物理机制. 尽管R249S-As的整体最低频率运动模式与WT相比有所变化, 但其运动轨迹显示砷(III)极大地稳定了R249S的L1环区域的向外摆动运动, 使DNA主沟结合残基序列的局部运动状态得以恢复(图2(c)). 换句话说, R249S-As整体最低频率振动模式相比WT发生了转换, 但其DNA结合域的运动显著恢复了.
此外, 在次低频振动模式中, 可观察到砷(III)结合对R249S突变体的DNA结合域运动的明显恢复作用(图2(d)—(f)). 其中, 野生型p53C结构的L1环和H2螺旋具有相对于p53C蛋白中心位置铰链开合的运动特征, 在R249S突变体中这一振动模式被抑制了, 而在砷(III)结合后, 这一铰链运动模式得到恢复; 从DNA结合域的振动特征模式方面看, 同样观察到R249S-As的运动更类似于野生型的振动模式. 可见, p53C蛋白中心最低频率的两个振动模式都显示蛋白的活性与太赫兹频率集体振动有关.
2
3.2.砷(III)对p53C突变蛋白的原子波动恢复作用
通过统计所有正则振动模式的原子波动, 可以获得蛋白分子骨架的整体波动情况. 原子波动参数用以描述p53C野生型、突变结构和活性恢复结构之间的蛋白分子内部运动的特定差异[29]. 如图3所示, WT, R249S和R249S-As的原子波动在非DNA结合区域的趋势一致, 反映了p53C蛋白分子整体柔性和形变能较为稳定, 其总体趋势取决于蛋白分子骨架构成及二级结构. 我们注意到, R249S突变蛋白在L1环和L2环区域具有较高水平的原子波动, 这可能是R249S不利于DNA结合的原因之一; R249S-As在L1环和L2环区域的原子波动恢复到了与野生型一致的水平, 而R249S-As的L3环原子波动幅度相比野生型有所增加. 综合最低频率轨迹和原子波动分析结果, 可以发现砷(III)结合明显改变L1环区域的低频运动, 意味着L1环区域的运动对DNA结合活性至关重要.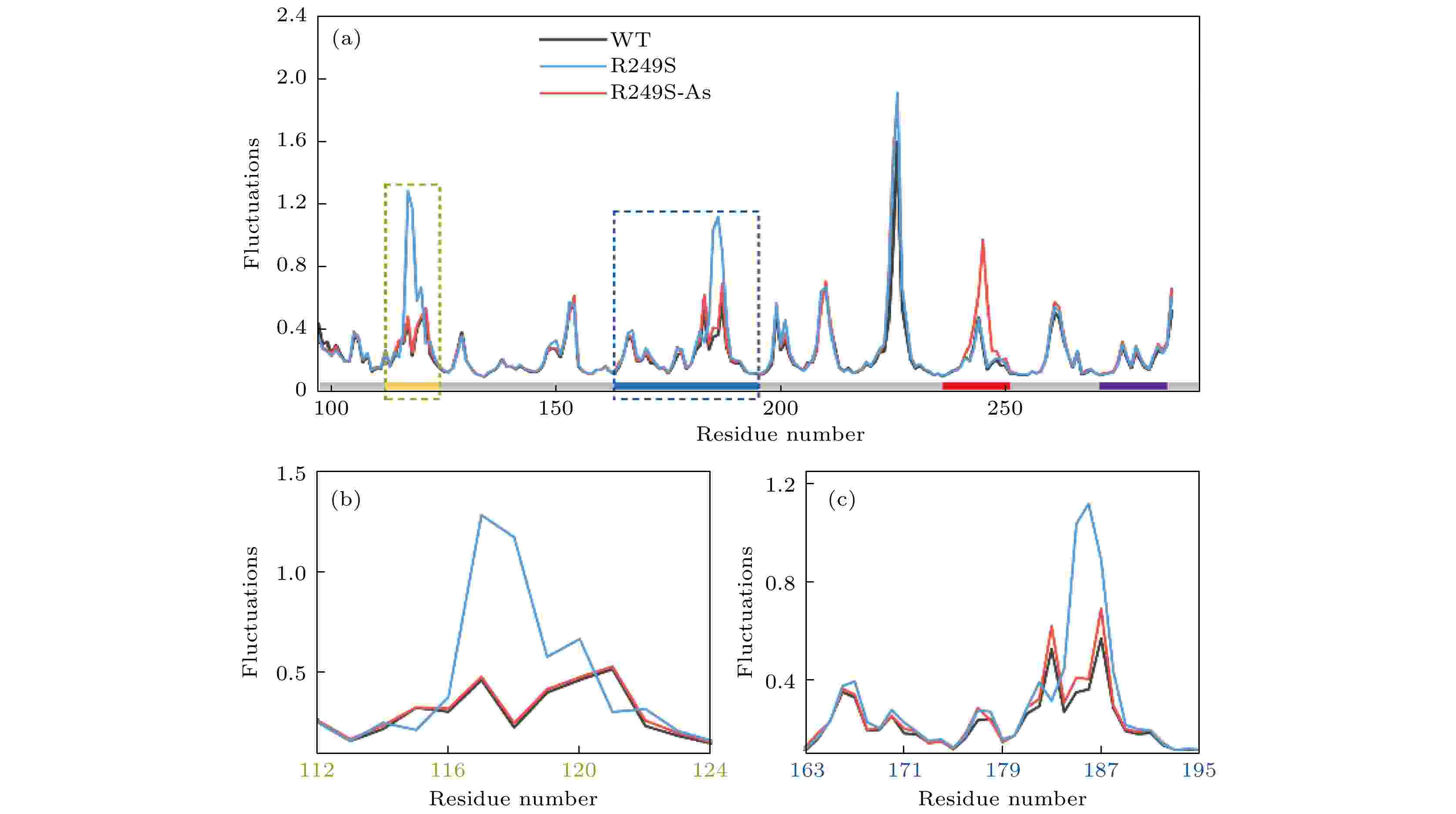 图 3 (a) p53C野生型、R249S突变型和砷(III)结合的R249S突变型p53C的原子波动分析(沿X轴的灰色条代表序列, 其中用四种不同颜色分别标记4个区域); (b) L1环区域(残基序列112—124)的局部原子波动分析; (c) L2环区域(残基序列163—195)的局部原子波动分析
图 3 (a) p53C野生型、R249S突变型和砷(III)结合的R249S突变型p53C的原子波动分析(沿X轴的灰色条代表序列, 其中用四种不同颜色分别标记4个区域); (b) L1环区域(残基序列112—124)的局部原子波动分析; (c) L2环区域(残基序列163—195)的局部原子波动分析Figure3. (a) Atomic fluctuation analysis of the DNA binding domain of the WT, R249S, and R249S-As. The gray bar above the X axis represents the sequence, where four regions are marked with four colors. (b) The details of the yellow (L1, residues 112–124) dotted areas. (c) The details of the blue (L2, residues 163–195) dotted areas.
2
3.3.砷(III)对p53C突变蛋白的振动频率恢复作用
蛋白分子的欠阻尼振动的频率决定了运动发生的时间尺度. 对蛋白分子自由状态和结合状态的模拟研究表明, 配体的结合会造成频率向高频方向偏移[30], 频率参数可能在反映蛋白-配体相互作用中有重要意义. 基于ENM获取的振动模式谱显示, p53C自最低频率振动模式的200个太赫兹振动模式处于0—1 THz的频率范围内, 并且野生型、突变体和砷(III)结合突变体振动模式可以根据频率分布区分(图4(a)). 因此, 我们统计了突变体和砷(III)结合突变体前200个太赫兹振动模式相对野生型p53C的频率偏移. 如图4(b)所示, 在这一范围内, R249S突变均造成了频率的负偏移, 而除了第7振动模式(即最低频率振动模式)外, 砷(III)结合的突变体对所有振动模式的频率展示出了明显的恢复作用. 图4(b)中插图展示了砷(III)结合对R249S突变体频率变化的恢复效果, 部分振动模式的恢复效果可达60%. 此外, 这里专门研究了特定频率太赫兹振动模式的偏移, 如图4(c)所示. 在0.3—1.3 THz范围内, R249S突变均造成了频率的负偏移, 偏移程度随频率升高而变小. 这暗示突变对太赫兹振动模式的影响可能主要体现在较低频段. 与之对应, 砷(III)结合对频率的恢复效果也主要体现在较低区域(0.3—0.8 THz), 而在更高频段恢复效果较弱. 砷(III)结合对p53C突变体振动频率的恢复, 进一步表明蛋白活性的恢复与其太赫兹频率振动属性相关.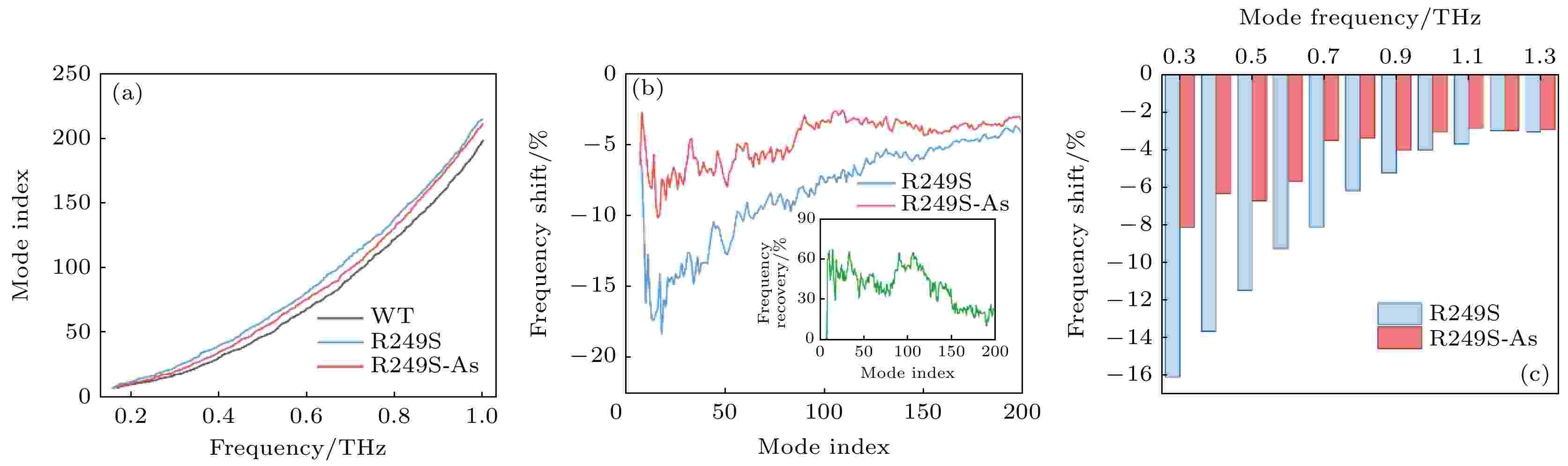 图 4 (a) 0—1.0 THz内的野生型p53C (WT), 突变体(R249S)和砷(III)结合突变体(R249S-As)的振动谱; (b) 突变体(R249S)和砷(III)结合突变体(R249S-As)相对于野生型p53 C的前200个振动模式的太赫兹振动频率偏移百分数, 插图为砷(III)结合后突变体的频率恢复比例; (c) R249S和R249S-As在野生型p53C特定频率处对应振动模式的频率偏移百分数
图 4 (a) 0—1.0 THz内的野生型p53C (WT), 突变体(R249S)和砷(III)结合突变体(R249S-As)的振动谱; (b) 突变体(R249S)和砷(III)结合突变体(R249S-As)相对于野生型p53 C的前200个振动模式的太赫兹振动频率偏移百分数, 插图为砷(III)结合后突变体的频率恢复比例; (c) R249S和R249S-As在野生型p53C特定频率处对应振动模式的频率偏移百分数Figure4. (a) Mode index spectra of wild-type p53C (WT), mutant (R249S) and As-bound mutant (R249S-As) in 0–1.0 THz; (b) the frequency shift (%) of the R249S and the R249S-As mutant from the WT in first 200 collective vibration modes, and the insert shows the proportion of frequency recovery for the As-bound R249S mutants; (c) the frequency shift (%) of corresponding mode of the R249S and the R249S-As at certain frequencies of WT.
2
3.4.p53C结构的动力学相关性分析
我们引入了动力学相关矩阵来解释突变和砷(III)结合对太赫兹振动的破坏和恢复. 如图5所示, 动力学正相关区域用黑色线框标出, 与DNA直接接触的4个残基和直接与砷(III)结合的4个残基在横轴上分别用绿色和黄色虚线标出. 动力学相关矩阵显示, L1, L2, L3和H2这4个区域是彼此动力学正相关的. 这意味着, 某一区域因突变而造成的动力学变化可以通过这种相关性网络传递到其他区域. 此外, 砷(III)结合的L1区域与本区域的K120残基, 以及H2区域的R273和R280残基都具备明显的运动相关性, 尤其是两个DNA结合残基(R273和R280)的运动与3个砷(III)结合残基(C124, M133, M135)直接正相关. 这种动力学相关性网络可能是砷(III)结合作为一种有效的p53蛋白活性恢复策略的生物物理基础. 图 5 野生型p53C的动力学相关矩阵. 每个点根据它在X轴和Y轴上的两个残基的Cab值(残基a与b之间的相关性)来着色. Cab值根据所得太赫兹振动模态计算, 其中Cab为1, –1和0, 分别表示完全相关、完全负相关和不相关. 横轴上用绿色虚线标出了4个与DNA作用的残基, 纵轴上用黄色虚线标出了4个与砷(III)结合的残基. 黑色虚线框标出了4个正相关区域, 其中L1与H2相关区域被专门表示在右边, 并与R249S突变体, 砷(III)结合R249S突变体动力学相关矩阵的对应区域比较
图 5 野生型p53C的动力学相关矩阵. 每个点根据它在X轴和Y轴上的两个残基的Cab值(残基a与b之间的相关性)来着色. Cab值根据所得太赫兹振动模态计算, 其中Cab为1, –1和0, 分别表示完全相关、完全负相关和不相关. 横轴上用绿色虚线标出了4个与DNA作用的残基, 纵轴上用黄色虚线标出了4个与砷(III)结合的残基. 黑色虚线框标出了4个正相关区域, 其中L1与H2相关区域被专门表示在右边, 并与R249S突变体, 砷(III)结合R249S突变体动力学相关矩阵的对应区域比较Figure5. Residue-residue motion correlation map of the DNA binding domain of the wild type p53C, where each point is colored according to its Cab (correlation between residues a and b) of the two residues on the X axis and Y axis. The Cab are calculated by all modes, where Cab = 1, –1, 0 means completely correlation, completely anticorrelation and no correlation, respectively. The four DNA-contact residues are marked in dark green dash curves on the X axis, and the four As-bound residues are marked in yellow dash curves on the Y axis. The black dashed boxes mark the four positively correlated regions, where the correlated regions between L1 and H2 are specifically represented on the right and compared with the corresponding regions of the correlation map of the R249S and the As-bound R249S mutant.
我们进一步具体阐释了砷(III)结合对L1-H2动力学相关网络的调控作用. 不同结构区域间运动的正相关性意味着在大多数太赫兹振动模式中, 两区域的运动是一个方向的, 这种协同振动具有重要的功能意义. 对野生型p53C, 两区域间残基的运动显示出了广泛的正相关性, 尤其是两个DNA结合残基(K120和R280)之间展示出了运动相关性, 这可能与DNA结合过程中的协同运动有关. 这种相关性在R249S突变体中被显著削弱, 而在砷(III)结合后得以恢复. 综上, 动力学相关性分析显示砷(III)的结合具有跨区域的运动耦合相关性影响, 使砷(III)结合区域(L1)的残基与H2区域的运动耦合恢复至野生型p53C状态.
