 ,2, 周钢桥
,2, 周钢桥 ,1,2,41.
,1,2,41. 2.
3.
4.
Genetic association of MKL1 gene polymorphisms with the high-altitude adaptation
Qing Zhang1, Jie Ping2, Haoxiang Zhang3, Bo Kang3,  ,2, Gangqiao Zhou
,2, Gangqiao Zhou ,1,2,41.
,1,2,41. 2.
3.
4.
通讯作者:
第一联系人:
编委: 谢小冬
收稿日期:2019-02-25修回日期:2019-05-6网络出版日期:2019-07-20
| 基金资助: |
Received:2019-02-25Revised:2019-05-6Online:2019-07-20
| Fund supported: |
作者简介 About authors
张晴,硕士研究生,专业方向:流行病与卫生统计学E-mail:zhangqingah@163.com。
平杰,博士研究生,专业方向:遗传学E-mail:pingjie46@126.com。

摘要
关键词:
Abstract
Keywords:
PDF (438KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张晴, 平杰, 张昊翔, 康波, 李元丰, 周钢桥. MKL1基因多态性与高原环境适应性的遗传关联研究[J]. 遗传, 2019, 41(7): 634-643 doi:10.16288/j.yczz.19-049
Qing Zhang, Jie Ping, Haoxiang Zhang, Bo Kang, , Gangqiao Zhou.
青藏高原是世界上海拔最高的高原,高原低压性缺氧是生活在该地区的人们面临的主要健康问题[1]。在海拔超过3000 m时,氧分压只有海平面的70%;当海拔达到5000 m时,氧分压只有海平面的50%[2]。人体对氧气供应的调节能力是影响人类高原适应性的重要因素[3]。世居平原的个体进入高原后,人体为应对低氧环境,会增加心输出量(单位时间内心脏左心室或右心室泵出的血液量)、加速血红蛋白及红细胞的生成,并加强肺血管的收缩作用,导致血液黏稠、肺动脉压升高,从而极易诱发急、慢性高原病[4,5]。相较于低海拔地区人群,藏族人群在高原经过了长期的自然选择,具有静息通气量增加、低氧诱导的肺血管收缩反应减弱、血氧饱和度升高及血红蛋白水平降低等一系列性状,表现出较好地适应高原低氧环境的能力[4,5]。
藏族人群的高原适应性具有明显的遗传学基础。有研究显示,高原病的发生具有显著的种族差异性,汉族等低海拔地区人群的高原病发生率显著高于世居高原的藏族人群[6,7,8,9]。此外,与高原适应性相关的多种性状,如静息通气量等,均被发现与遗传因素密切相关[3,10~12]。至今,基于基因组学技术,研究人员已发现多个基因(如EPAS1、EGLN1和PPARA等)与藏族人群的高原低氧适应性密切相关[13,14,15]。然而,这些基因尚不能完全解释高原适应性的遗传学基础,更多的高原适应性相关基因仍有待被发现。
巨核细胞白血病因子1(megakaryocytic leukemia1, MKL1),又被称为心肌蛋白相关转录因子A (myocardin related transcription factor A, MRTF-A),是由MKL1基因编码的蛋白质,属于转录调节因子家族,参与平滑肌细胞的表型调变[16,17]。近年来研究发现,MKL1作为一种应激蛋白,可调节细胞对外界刺激信号的反应性[18,19]。体外细胞学和小鼠在体实验等研究表明,在低氧环境下,MKL1表达增加,可上调ICAM-1等细胞黏附因子的转录水平,从而影响小鼠肺组织炎症细胞的聚集及肺血管炎症反应,进而引发低氧性肺动脉高压[20]。在上调ICAM-1基因转录水平的同时,MKL1还可下调内皮型一氧化氮合酶基因(eNOS)的转录水平[21]。eNOS编码内皮型一氧化氮合酶(eNOS),在调节血管紧张度方面发挥重要作用[22]。多项研究已报道,eNOS与高原适应性及高原病的发生存在显著的遗传学关联[23,24,25,26,27,28]。此外,MKL1还参与内皮素(ET-1)在人类血管内皮细胞中的转录激活[29]。ET-1作为一种强效的血管收缩剂,与肺动脉高压的发生密切相关[30,31,32,33]。因此推测,MKL1可能与高原低氧适应性相关。
为进一步探讨MKL1与高原适应性的关系,本研究基于595名藏族个体和442名汉族个体,分型检测了MKL1基因上的功能性单核苷酸多态性位点(single nucleotide polymorphism, SNP)rs59098711。研究发现,该SNP位点在世居高原地区的藏族人群与世居平原地区的汉族人群之间存在显著的分布差异,提示MKL1基因可能与藏族人群对高原低氧环境的适应能力有关。
1 材料与方法
1.1 研究对象
本研究招募了595名藏族个体,其平均年龄为40.08±14.82岁,世居海拔超过3500 m的青藏高原地区。还招募了442名汉族个体,其平均年龄为30.42±12.76岁,世居海拔低于200 m的平原地区。所有研究对象均签署了知情同意书。1.2 样本采集及DNA提取
采用EDTK-K2抗凝管采集肘静脉全血5 mL,离心后分离全血和血浆,储存至-80℃低温冰箱保 存。采用Sangon公司(上海)的基因组DNA提取试剂盒进行全血DNA的提取。具体步骤如下:在300 μL抗凝全血中加入600 μL缓冲液TBP,充分混匀,室温放置1 min至红细胞完全裂解,8000 r/min离心1 min,弃上清。加入180 μL裂解液和20 μL蛋白酶K溶液,振荡混匀,加入60 μL缓冲液PR充分颠倒混匀,-20℃冰箱放置5 min。室温条件下多次离心后弃去上清。重复乙醇漂洗。50 μL TE缓冲液溶解DNA,检测其浓度后,标准化至20~50 ng/μL,-20℃保存备用。1.3 SNP的选择
基于千人基因组计划数据库(1000 Genomes Project)中东亚人群的基因型数据,寻找位于MKL1基因上(Chr.22:40806292~41032690,人类基因组第19版本,hg19)的常见SNP位点(最小等位基因频率> 0.05)。在MKL1基因内共有283个常见SNP位点。采用Haploview 4.2软件挑选标签SNPs。利用HaploReg在线工具(V4.1, https://pubs.broadinstitute. org/mammals/haploreg/haploreg.php)和GTEx数据库(V7, https://gtexportal.org/)对标签SNPs进行注释,根据注释信息选择候选位点。1.4 SNP分型
采用Array Designer软件设计PCR扩增及延伸引物。rs59098711上游引物序列为5°-ACGTTGGATGACACAGGGAGACCCCCGTTT-3°,下游引物序列为5°-ACGTTGGATGTCAGCTTCCGGAGCAGCTA- 3°,引物由北京华大基因科技股份有限公司合成。采用Agena公司(美国)MassARRAY SNP分型检测方法,经过多重PCR技术、MassARRAYiPLEX单碱基延伸技术和基质辅助激光解吸附电离飞行时间质谱技术(matrix-assisted laser desorption/ionization-time of flight, MALDI-TOF)对rs59098711位点进行分型检测。最后使用Typer 4.0软件对检测结果进行分析,从而获得rs59098711的分型数据。1.5 统计学方法
采用R 3.4.4软件进行数据分析,Hardy-Weinberg (H-W)平衡检验、组间基因型及等位基因型频率分布的比较采用卡方(χ2)检验。组间基因表达水平的比较采用两独立样本t检验。以α = 0.05作为检验水准。使用Plink 1.9软件计算SNP位点的遗传分化系数(FST)。2 结果与分析
2.1 候选位点的选择
在MKL1基因全长内共找到283个常见SNP位点。使用Haploview 4.2软件,挑选出10个标签SNPs(图1)。利用HaploReg 4.1在线工具发现,所有的标签SNPs均位于MKL1基因的内含子区。其中,MKL1基因第3号内含子上的1个标签SNP rs59098711(Chr.22:40877004, hg19)位于血管内皮细胞(人脐静脉内皮细胞,human umbilical vein endothelial cell, HUVEC)基因组的增强子区域(图1)。查询GTEx数据库(V7, https://gtexportal.org/)发现,rs59098711与MKL1基因的表达水平显著相关(false discovery rate, FDR<0.05)。因此,本研究选择rs59098711作为候选位点,进行后续的遗传关联分析。图1
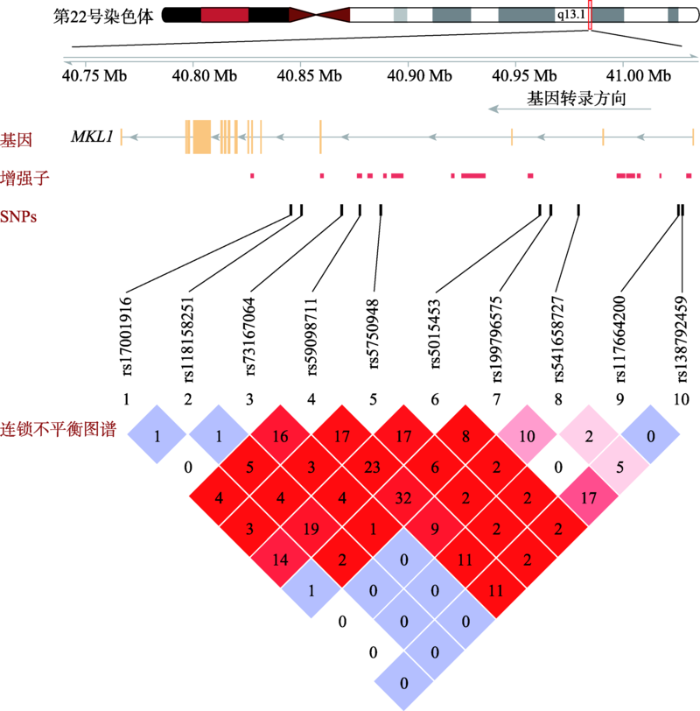 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1MKL1基因区域的标签SNPs
增强子信息来源于“人类表观基因组计划”(Roadmap Epigenomics Project)中人脐静脉内皮细胞(human umbilical vein endothelial cell, HUVEC)的染色质数据。连锁不平衡图谱中的数字等于100×r2,颜色越红代表D°值越大,越蓝代表D°值越小。
Fig. 1The tag SNPs at MKL1 locus
2.2 Hardy-Weinberg平衡检验
本研究使用MassARRAY阵列图谱技术,在藏族人群(n=595)和汉族人群(n=442)中对rs59098711位点进行了分型,所有个体均分型成功。在两组人群中该位点的基因型分布均符合Hardy-Weinberg平衡(P>0.05),说明群体基因遗传平衡,样本抽样无明显偏差(表1)。Table 1
表1
表1 rs59098711位点Hardy-Weinberg平衡检验
Table 1
| rs59098711 | 藏族人群 | 汉族人群 | ||||||
|---|---|---|---|---|---|---|---|---|
| 观察数 | 理论数 | χ2 | P | 观察数 | 理论数 | χ2 | P | |
| TT | 105 | 90.08 | 3.29 | 0.19 | 168 | 171.11 | 0.20 | 0.91 |
| TC | 253 | 282.86 | 214 | 207.80 | ||||
| CC | 237 | 222.05 | 60 | 63.09 | ||||
新窗口打开|下载CSV
2.3 在藏族与汉族人群间rs59098711位点基因型频率的比较
在藏族和汉族人群间,rs59098711位点的基因型和等位基因型频率分布存在显著差异,在藏族人群中基因型CC和等位基因C的频率显著高于汉族人群(P<0.05;表2)。根据藏族与汉族人群的基因型频率数据,计算得到rs59098711的FST值为0.10,远高于已报道的藏、汉人群间全基因组的平均FST值(0.012)[34],提示在藏族人群和汉族人群之间rs59098711位点存在遗传分化现象。Table 2
表2
表2 在藏族和汉族人群中rs59098711位点的基因型和等位基因型频率分布
Table 2
| 基因型 | 等位基因型 | ||||
|---|---|---|---|---|---|
| CC | TC | TT | C | T | |
| 藏族[例(%)] | 237(39.83) | 253(42.52) | 105(17.65) | 727(61.09) | 463(38.91) |
| 汉族[例(%)] | 60(13.57) | 214(48.42) | 168(38.01) | 334(37.78) | 550(62.22) |
| OR(95% CI) | 3.34(2.40~4.66) | 6.32(4.40~9.08) | 2.59(2.16~3.09) | ||
| χ2 | 50.71 | 99.56 | 109.36 | ||
| P | 1.07×10-12 | 1.90×10-23 | 1.35×10-25 | ||
新窗口打开|下载CSV
2.4 在藏族人群与其他种群间rs59098711位点基因型频率的比较
将藏族人群中rs59098711的基因型及等位基因型的频率分布,分别与千人基因组计划数据库(1000 Genomes Project)中来自东亚、南亚、欧洲、美洲和非洲的5个人群进行比较,结果发现,藏族人群中rs59098711的基因型及等位基因型的频率分布与5个人群均存在显著差异,在藏族人群中基因型CC和等位基因C的频率显著高于其他5个人群(表3)。为进一步验证上述结果,将藏族人群与gnomAD (Genome Aggregation Database)数据库中的5个人群也进行了比较,发现了与千人基因组计划数据库相比较的类似的结果(表4)。以上结果表明,rs59098711位点的基因型和等位基因型的频率分布在不同种族之间存在显著差异。Table 3
表3
表3 在藏族和千人基因组计划的5个人群中rs59098711位点的基因型和等位基因型的频率分布
Table 3
| 基因型 | 等位基因型 | |||||
|---|---|---|---|---|---|---|
| CC | TC | TT | C | T | ||
| 藏族[例(%)] | 237(39.83) | 253(42.52) | 105(17.65) | 727(61.09) | 463(38.91) | |
| 东亚[例(%)] | 48(9.52) | 229(45.44) | 227(45.04) | 325(32.24) | 683(67.76) | |
| 南亚[例(%)] | 36(7.36) | 177(36.20) | 276(56.44) | 249(25.46) | 729(74.54) | |
| 欧洲[例(%)] | 24(4.77) | 151(30.02) | 328(65.21) | 199(19.78) | 807(80.22) | |
| 美洲[例(%)] | 6(1.73) | 74(21.32) | 267(76.95) | 86(12.39) | 608(87.61) | |
| 非洲[例(%)] | 46(6.96) | 282(42.66) | 333(50.38) | 374(28.29) | 948(71.71) | |
| 藏族vs.东亚 | ||||||
| OR(95% CI) | 4.47(3.16~6.32) | 10.67(7.42~15.36) | 3.30(2.77~3.93) | |||
| χ2 | 71.69 | 162.76 | 180.87 | |||
| P | 2.52×10-17 | 2.83×10-37 | 3.12×10-41 | |||
| 藏族vs.南亚 | ||||||
| OR(95% CI) | 4.60(3.13~6.77) | 17.30(11.89~25.19) | 4.60(3.83~5.51) | |||
| χ2 | 60.57 | 221.46 | 273.93 | |||
| P | 7.11×10-15 | 4.34×10-50 | 1.58×10-61 | |||
| 藏族vs.欧洲 | ||||||
| OR(95% CI) | 5.89(3.81~9.12) | 30.84(20.73~45.90) | 6.37(5.29~7.67) | |||
| χ2 | 63.5 | 285.93 | 379.81 | |||
| P | 1.61×10-15 | 3.82×10-64 | 1.37×10-84 | |||
| 藏族vs.美洲 | ||||||
| OR(95% CI) | 11.55(5.66~23.56) | 100.44(58.72~171.82) | 11.10(8.82~13.97) | |||
| χ2 | 45.31 | 283.20 | 421.83 | |||
| P | 1.68×10-11 | 1.51×10-63 | 9.75×10-94 | |||
| 藏族vs.非洲 | ||||||
| OR(95% CI) | 5.74(4.08~8.08) | 16.34(11.51~23.20) | 3.98(3.38~4.69) | |||
| χ2 | 100.90 | 243.95 | 272.37 | |||
| P | 9.67×10-24 | 5.41×10-55 | 3.45×10-61 | |||
新窗口打开|下载CSV
Table 4
表4
表4 在藏族和gnomAD数据库的5个人群中rs59098711位点的基因型和等位基因型的频率分布
Table 4
| 基因型 | 等位基因型 | ||||
|---|---|---|---|---|---|
| CC | TC | TT | C | T | |
| 藏族[例(%)] | 237(39.83) | 253(42.52) | 105(17.65) | 727(61.09) | 463(38.91) |
| 东亚[例(%)] | 79(10.17) | 356(45.82) | 342(44.01) | 514(33.08) | 1040(66.92) |
| 欧洲[例(%)] | 526(5.60) | 3185(33.88) | 5689(60.52) | 4237(22.54) | 14563(77.46) |
| 非洲[例(%)] | 230(5.32) | 1,592(36.86) | 2498(57.82) | 2052(23.75) | 6588(76.25) |
| 拉丁美洲[例(%)] | 7(1.66) | 110(26.13) | 304(72.21) | 124(14.73) | 718(85.27) |
| 德系犹太人[例(%)] | 11(7.59) | 48(33.10) | 86(59.31) | 70(24.14) | 220(75.86) |
| 基因型 | 等位基因型 | ||||
| CC | TC | TT | C | T | |
| 藏族vs.东亚 | |||||
| OR(95% CI) | 4.22(3.15~5.66) | 9.77(7.10~13.44) | 3.18(2.72~3.71) | ||
| χ2 | 92.14 | 196.53 | 212.41 | ||
| P | 8.07×10-22 | 1.19×10-44 | 4.09×10-48 | ||
| 藏族vs.欧洲 | |||||
| OR(95% CI) | 5.67(4.71~6.82) | 24.41(20.31~29.34) | 5.40(4.83~6.03) | ||
| χ2 | 338.18 | 1160.80 | 889.20 | ||
| P | 1.59×10-75 | 1.99×10-254 | 2.18×10-195 | ||
| 藏族vs.非洲 | |||||
| OR(95% CI) | 6.48(5.25~8.00) | 24.51(19.82~30.33) | 5.04(4.48~5.67) | ||
| χ2 | 303.86 | 868.23 | 717.41 | ||
| P | 4.76×10-68 | 7.93×10-191 | 4.90×10-158 | ||
| 藏族vs.拉丁美洲 | |||||
| OR(95% CI) | 14.72(7.80~27.79) | 98.02(58.84~163.31) | 9.09(7.39~11.19) | ||
| χ2 | 68.83 | 310.00 | 433.60 | ||
| P | 1.07×10-16 | 2.18×10-69 | 2.67×10-96 | ||
| 藏族vs.德系犹太人 | |||||
| OR(95% CI) | 4.09(2.12~7.89) | 17.65(10.08~30.89) | 4.93(3.74~6.52) | ||
| χ2 | 17.6 | 100.93 | 126.65 | ||
| P | 2.72×10-5 | 9.52×10-24 | 2.22×10-29 | ||
新窗口打开|下载CSV
2.5 rs59098711位点变异影响MKL1的表达
SNP位点rs59098711位于MKL1基因的内含子区,不能改变MKL1蛋白的氨基酸序列。然而,非编码区的变异可通过调控基因的表达水平而发挥生物学功能[35]。本研究采用HaploReg注释,发现在肺组织和人脐静脉内皮细胞中rs59098711位点位于增强子区域,提示其具有调节肺血管基因表达水平的功能。进一步查询3DIV数据库(https://www.kobic. kr/3div/)发现,在肺组织和血管内皮细胞中,rs59098711位点所在区域与MKL1基因的启动子 区存在染色质相互作用(表5)。为进一步验证rs59098711是否能够调节MKL1基因的表达,基于GTEx数据库,查看了rs59098711基因型是否与MKL1基因的表达水平相关。结果发现,在脑组织中,rs59098711基因型与MKL1基因的表达水平存在显著关联(P=3.2×10-5)。其中,C等位基因与较低的MKL1表达水平相关。以上结果提示,rs59098711位于基因组调控区,可能在MKL1的转录过程中发挥调控作用,从而影响MKL1的表达水平,最终影响个体对高原缺氧环境的适应能力。Table 5
表5
表5 rs59098711与MKL1基因的启动子区存在染色质相互作用
Table 5
| SNP | 样本组织类型 | 相互作用片段的基因组位置(bp) | 基因* |
|---|---|---|---|
| rs59098711 | Lung | Chr.22:41030000~41035000 | MKL1 |
| HMEC | Chr.22:41030000~41035000 | MKL1 | |
| HUVEC | Chr.22:41030000~41035000 | MKL1 |
新窗口打开|下载CSV
2.6 低压性缺氧环境下MKL1表达显著上调
基于基因表达公共数据库(Gene Expression Omnibus, GEO)的数据集(GSE87602)[36]进行分析发现,与对照组(n=2)相比,在低压性缺氧环境中培养14天的小鼠(n=3),其肺泡巨噬细胞中MKL1的表达水平显著增加,表达差异倍数(fold change, FC)为1.5倍,P=0.027(图2A)。此外,进一步对进入高原环境前后人外周血单核细胞(peripheral blood mononuclear cells, PMBCs)的表达数据集(GSE103927)[37]进行分析。结果显示,与在平原环境中的样本相比(n=21),进入高原环境后(n=13),人外周血单核细胞中MKL1的表达水平显著增加(FC=1.2, P<0.0001;图2B)。以上结果提示,MKL1可能在适应高原低氧环境的过程中发挥作用。图2
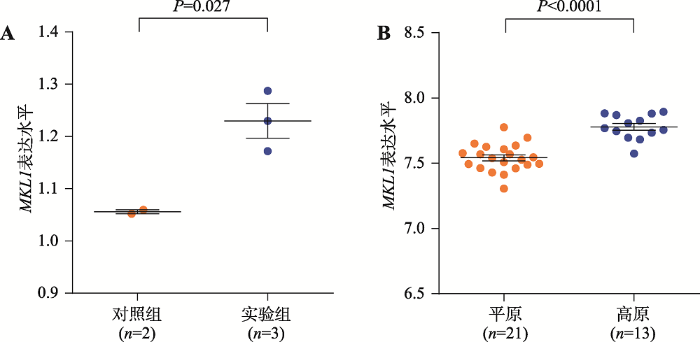 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2低压性缺氧环境下MKL1的表达水平上调
A:低压性缺氧环境下小鼠肺泡巨噬细胞中MKL1的表达水平显著增加。对照组(n=2)为常氧条件下培养的小鼠,实验组(n=3)为在低压性缺氧环境(模拟高原低氧环境)下培养的小鼠。各自培养14天后,测量并比较两组小鼠肺泡巨噬细胞中基因的表达水平;B:进入高原后人外周血单核细胞中MKL1的表达水平显著增加。分别测量研究对象在海平面高度(n=21)和进入高原后(n=13)外周血单核细胞中基因的表达水平,并进行比较。以上基因表达数据均进行了对数(log2)转换,采用两独立样本t检验分析组间MKL1基因表达差异是否具有统计学意义。
Fig. 2MLK1 expression is up-regulated by hypobaric hypoxia
3 讨论
本研究通过比较藏族与汉族人群中MKL1基因的功能性位点rs59098711的基因型和等位基因型频率分布差异发现,rs59098711的CC基因型和C等位基因型在藏族中的频率显著高于汉族人群。进一步通过与公共数据库中的数据进行比较发现,rs59098711的CC基因型和C等位基因型在藏族人群中的频率显著高于其他种族,提示rs59098711位点的C等位基因型可能在藏族人群中表现出了选择优势,有助于藏族人群适应青藏高原的生存环境。遗传分化系数FST是衡量群体间遗传分化的重要指标,可以很好的反应SNP位点在不同群体间的遗传分化程度。FST值在0~1范围内,其值越大,提示两个群体间的分化程度就越高。Wright等[38]认为,若FST值在0~0.05之间,提示群体间可能不存在遗传分化;若FST值在0.05~0.15之间,提示群体间存在中等程度的遗传分化;若FST值在0.15~0.25之间,提示群体间存在高度的遗传分化;若FST值大于0.25,则群体间有极大的遗传分化。本研究结果显示,rs59098711在藏族与汉族之间的FST值为0.10,说明该位点在藏汉人群间存在中等程度的遗传分化。以上结果提示,MKL1基因上的rs59098711位点可能与人类高原适应性相关。低压性缺氧是高原地区威胁人体健康的主要因素。与世居低海拔地区的人群相比,藏族人群已进化出多种适应性特征,使得他们能够应对高原的缺氧环境。例如,藏族人群血红蛋白水平较低,避免了高血红蛋白水平引起的多种并发症(如高黏血症)[11,13];藏族人群的缺氧性肺血管收缩程度较弱,避免了低氧性肺动脉高压和由此诱发的肺水肿[11];藏族人群的血流量,以及氧气由血液扩散进入组织细胞中的速率,均高于低海拔地区的人群[3]。有趣的是,以往遗传学研究中发现的两个最为显著的藏族高原适应性基因EPAS1和EGLN1,与上述高原适应性特征均密切相关[39]。而MKL1基因编码的蛋白属于转录因子家族成员,该蛋白可结合其靶基因(如ICAM-1、eNOS和ET-1等)的启动子区,调控靶基因的转录[20,21,29]。在缺氧条件下,肺血管中MKL1的表达增加,可促进缺氧条件下血管内皮细胞中ICAM-1、ET-1等基因的转录[20,29],并抑制eNOS的转录水平[21],从而导致血管收缩,血流量降低,肺动脉压升高。MKL1是否与藏族人群的其他适应性特征相关,目前尚未有明确报道,需要后续进一步的研究探索。根据本研究结果,藏族人群携带rs59098711的C等位基因的频率显著高于低海拔人群,而rs59098711的C等位基因型又与较低的MKL1表达水平显著相关,可以推测,大多数藏族个体MKL1的表达水平显著低于低海拔地区个体,使得藏族个体在高原低氧环境中肺血管收缩程度弱于低海拔地区个体,从而减少了低氧性肺动脉高压和肺水肿等高原病的发生率。
综上所述,本研究发现MKL1基因的多态性位点rs59098711可能与藏族人群的高原环境适应性相关。未来仍需要开展针对该基因在肺血管内皮细胞中的功能研究,以进一步探索MKL1在高原环境适应性中的作用及机制。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 3]
URL [本文引用: 2]
[本文引用: 2]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 1]
Magsci [本文引用: 1]

<p>为了研究脯氨酰羟化酶2 (EGLN1或PHD2)基因5′UTR区和内含子区的单核苷酸多态性(SNP)与高海拔低氧适应的相关性, 文章使用Sequenom MassArray基因分型系统对152例西藏藏族(海拔3 650 m)和192例湖北汉族(海拔500 m) EGLN1基因的6个单核苷酸多态性位点(rs2066140、rs2808584、rs2491405、rs2486741、rs2486734和rs21533646)进行基因分型, 并进行单倍型构建和分析。结果发现:6个SNP位点的纯合子基因型在高海拔和低海拔群体间均具有统计学差异(P<0.05)。单倍型分析显示, 单倍型G-G(rs2066140和rs2808584)和G-C(rs2486741和rs2486734)的频率在高海拔群体中高于低海拔群体(P<0.05), 单倍型C-A(rs2066140和rs2808584)和C-T(rs2486741和rs2486734)的频率在高海拔群体中低于低海拔群体(P<0.05)。研究结果提示, EGLN1基因6个SNP位点的纯合子基因型和所构建的单倍型与高海拔低氧适应相关。</p>
Magsci [本文引用: 1]

<p>为了研究脯氨酰羟化酶2 (EGLN1或PHD2)基因5′UTR区和内含子区的单核苷酸多态性(SNP)与高海拔低氧适应的相关性, 文章使用Sequenom MassArray基因分型系统对152例西藏藏族(海拔3 650 m)和192例湖北汉族(海拔500 m) EGLN1基因的6个单核苷酸多态性位点(rs2066140、rs2808584、rs2491405、rs2486741、rs2486734和rs21533646)进行基因分型, 并进行单倍型构建和分析。结果发现:6个SNP位点的纯合子基因型在高海拔和低海拔群体间均具有统计学差异(P<0.05)。单倍型分析显示, 单倍型G-G(rs2066140和rs2808584)和G-C(rs2486741和rs2486734)的频率在高海拔群体中高于低海拔群体(P<0.05), 单倍型C-A(rs2066140和rs2808584)和C-T(rs2486741和rs2486734)的频率在高海拔群体中低于低海拔群体(P<0.05)。研究结果提示, EGLN1基因6个SNP位点的纯合子基因型和所构建的单倍型与高海拔低氧适应相关。</p>
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 3]
URL [本文引用: 3]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
Magsci [本文引用: 1]

<FONT face=Verdana>低氧刺激诱导型一氧化氮合酶(Inducible nitric-oxide synthase, iNOS)催化产生NO, 增加血流, 改善组织供氧。文章采用测序和PCR-RFLP技术检测藏鸡及低地鸡iNOS基因编码区、5′侧翼区(2.0 kb片段)序列和3′侧翼序列SNP, 并测定低氧和常氧孵化时鸡胚尿囊绒毛膜组织iNOS基因表达量和酶活力。结果在iNOS基因 5′侧翼区发现一个与低氧适应相关的藏鸡高频率突变SNP位点(-870C→T), 藏鸡该突变的等位基因T频率高于低地鸡种。藏鸡iNOS基因表达量和酶活力在低氧孵化环境中多高于矮小鸡。结果表明藏鸡群体iNOS基因的突变及其低氧表达量的增加是其适应低氧环境的重要基础。</FONT>
Magsci [本文引用: 1]

<FONT face=Verdana>低氧刺激诱导型一氧化氮合酶(Inducible nitric-oxide synthase, iNOS)催化产生NO, 增加血流, 改善组织供氧。文章采用测序和PCR-RFLP技术检测藏鸡及低地鸡iNOS基因编码区、5′侧翼区(2.0 kb片段)序列和3′侧翼序列SNP, 并测定低氧和常氧孵化时鸡胚尿囊绒毛膜组织iNOS基因表达量和酶活力。结果在iNOS基因 5′侧翼区发现一个与低氧适应相关的藏鸡高频率突变SNP位点(-870C→T), 藏鸡该突变的等位基因T频率高于低地鸡种。藏鸡iNOS基因表达量和酶活力在低氧孵化环境中多高于矮小鸡。结果表明藏鸡群体iNOS基因的突变及其低氧表达量的增加是其适应低氧环境的重要基础。</FONT>
URL [本文引用: 3]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
