 ,南京总医院药理科,南京 210002
,南京总医院药理科,南京 210002Detection of UGT1A1*6 single nucleotide polymorphism in oral swap samples using the cascade invader assay-based real-time PCR
Jieyu Zhang, Yanan Chu, Bingjie Zou, Yanjie Zhang, Liying Feng ,Department of Pharmacology, Nanjing General Hospital, Nanjing 210002, China
,Department of Pharmacology, Nanjing General Hospital, Nanjing 210002, China通讯作者:
编委: 卢大儒
收稿日期:2018-03-26修回日期:2018-05-17网络出版日期:2018-08-16
| 基金资助: |
Received:2018-03-26Revised:2018-05-17Online:2018-08-16
| Fund supported: |
作者简介 About authors
张婕妤,硕士研究生,药师,研究方向:药物基因组学E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (680KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张婕妤, 初亚男, 邹秉杰, 张晏洁, 封利颖. 基于级联核酸侵入反应的real-time PCR法检测咽拭子样本中UGT1A1*6基因多态性[J]. 遗传, 2018, 40(8): 668-675 doi:10.16288/j.yczz.18-074
Jieyu Zhang, Yanan Chu, Bingjie Zou, Yanjie Zhang, Liying Feng.
伊立替康(irinotecan)是一种DNA拓扑异构酶Ⅰ抑制剂,作为抗肿瘤药在结直肠癌、肺癌、胃癌等实体瘤的化疗中发挥重要作用,但其导致腹泻和中性粒细胞减少不良反应的发生率高达20%~35%[1]。大量研究表明,在亚洲人群中其不良反应的发生与UGT1A1*6单核苷酸多态性(single nucleotide polymorphisms, SNP)相关[2, 3],因此,在用药前检测UGT1A1*6基因型是一种预防伊立替康不良反应发生的有效途径。
目前,临床用于SNP检测的方法主要有等位基因特异性PCR[4]、焦磷酸测序[5]、基于TaqMan探针的real-time PCR法[6]等。其中,等位基因特异性PCR和基于TaqMan探针的real-time PCR法可实现闭管检测,不易产生交叉污染,但为达到高特异性检测,引物或探针需要精心设计,反应条件较难优化[7,8]。焦磷酸测序法是SNP检测的金标准,但其操作繁琐、开盖检测易污染、仪器设备较昂贵[9]。级联核酸侵入反应(invader assay)因其特异性高、反应条件易优化而非常适用于SNP检测。该反应通过结构特异性内切酶flap核酸内切酶1 (flap endonucleases 1, FEN1)识别由侵入探针、检测探针和靶序列互补形成的三碱基重叠结构,并切除检测探针的5°flap片段,该片段可作为侵入探针与荧光共振转移(fluorescence resonance energy transfer probe, FRET)发卡探针参与新一轮的核酸侵入反应,生成放大的荧光信号[10]。该反应对探针与靶标形成的三碱基重叠的侵入结构具有很强的识别特异性。因此,在对SNP进行分型检测时,可将待测SNP位点置于三碱基重叠结构中,每种基因型对应不同的flap片段与不同荧光标记的FRET探针,通过双通道荧光信号检测可在单管中判断SNP的类型。虽然该方法由于灵敏度和特异性高,已被开发成为商品试剂盒广泛用于SNP检测,但现有基于级联核酸侵入反应的SNP检测大都会在PCR扩增结束后利用级联核酸侵入反应检测扩增产物中的单碱基序列差异,造成反应时间长,并且易产生非特异信号干扰结果判断[11]。
本研究以UGT1A1*6为检测对象,将级联核酸侵入反应与PCR扩增循环相偶联,在每个PCR循环中进行一段时间的级联核酸侵入反应放大扩增产物中的位点特异性信号,产生类似于荧光定量PCR的扩增曲线,通过两种荧光通道产生的扩增曲线判断UGT1A1*6的基因型,缩短了检测时间,避免了终点法产生的非特异性背景信号对结果的干扰。此外,为避免抽取患者血液进行基因检测,本研究将建立的UGT1A1*6基因分型方法与口腔咽拭子DNA提取试剂盒结合,成功建立了通过咽拭子采样的UGT1A1*6基因分型检测方法,从一定程度上简化了检测流程,提高了患者顺应性。
1 材料和方法
1.1 样本采集
采用咽拭子擦拭患者口腔内壁的10次,用于基因组DNA提取。1.2 基因组DNA提取
采用北京天根生物科技有限公司的口腔拭子基因组DNA提取试剂盒提取样本中的DNA。使用One Drop分光光度计(南京伍义科技有限公司)进行DNA的质量评估和浓度测定,合格的标本存放在-20℃ 待测。1.3 基于级联核酸侵入反应的real-time PCR反应条件
反应体系包括反应缓冲液[10 mmol/L Tris-HCl (南京Biosky公司)、0.05% (V/V) NP-40 (南京Sunshine公司)、0.05% (V/V) Tween-20 (美国Bios公司)、12 μg/mL BSA(美国Amresco公司)、50 mmol/L KCl(北京国药集团)、7.5 mmol/L MgCl2 (北京国药集团),pH 8.5],dNTPs (上海百思凯公司) 250 μmol/L,PCR正反向引物各0.5 μmol/L,侵入探针0.05 μmol/L,野生型检测探针0.125 μmol/L,突变型检测探针1 μmol/L,野生型(携带VIC荧光基团)、突变型(携带FAM荧光基团)荧光共振转移发卡探针各0.25 μmol/L,Taq DNA聚合酶1 U (美国Promega公司),FEN酶1.5 U (本实验室自表达),基因组DNA 1.5 μL,用H2O补充到20 μL。反应程序:95℃ 3 min;95℃ 20 s,67℃ 60 s,70℃ 30 s,预扩增10个循环;95℃ 20 s,63℃ 60 s,采集荧光信号,70℃ 30 s,扩增35个循环。使用Universal Invader?软件设计核酸侵入反应的引物和探针(表1),所有引物和探针由上海Invitrogen公司合成。反应在德国QIAGEN公司的Rotor-Gene Q实时荧光定量PCR仪上进行。结果判读时,将空白对照最后一个循环信号值的平均值+3SD作为阈值线,超过阈值线即为 检出。Table 1
表1
表1 基于级联核酸侵入反应的real-time PCR检测UGT1A1*6基因型的扩增引物和探针序列
Table 1
| 引物名称 | 序列(5°→3°) |
|---|---|
| 突变型荧光发卡探针FRET-1 | FAM-TCTT(BHQ1)AGCCGGTTTTCCGGCTAAGACTCCGCGTCCGT-C6-NH2 |
| 野生型荧光发卡探针FRET-2 | VIC-TCTT(BHQ1)AGCCGGTTTTCCGGCTAAGACCTCGGCGCG-C6-NH2 |
| PCR正向引物 | TGTCCTGCACCTGACGCCT |
| PCR反向引物 | ACATCCTCCCTTTGGAATGGCACA |
| 侵入探针 | GTACGTCTTCAAGGTGTAAAATGCTCA |
| 野生型检测探针 | CGCGCCGAGGCGTCTCTGATGTACAACG-PO3 |
| 突变型检测探针 | ACGGACGCGGAGTGTCTCTGATGTACAACG-PO3 |
新窗口打开|下载CSV
1.4 焦磷酸测序
PCR扩增反应采用大连TaKaRa公司的TaqTM (含Mg2+溶液),反应条件:94℃ 5 min;94℃ 30 s,55℃ 30 s,72℃ 30 s,扩增35个循环;72℃ 7 min,使用杭州朗基生产的基因扩增仪进行扩增。扩增产物用Qiagen公司的PyroMark Q24进行单链制备和焦测序。PCR扩增与焦磷酸测序引物序列见表2。Table 2
表2
表2 焦测序检测UGT1A1*6基因型的扩增引物和测序引物序列
Table 2
| 引物名称 | 序列(5°→3°) |
|---|---|
| 正向生物素 修饰扩增引物 | Biotin-CAGCAGAGGGGACATGAAATAG |
| 反向扩增引物 | CCTCCCTTTGGAATGGCAC |
| 测序引物 | GTCTTCAAGGTGTAAAATGCTC |
新窗口打开|下载CSV
2 结果与分析
2.1 基于级联核酸侵入反应的real-time PCR法检测SNP的原理
检测原理如图1所示。针对待测UGT1A1*6位点设计侵入探针与检测探针,使它们与待测靶标杂交后,侵入探针的3′端有1个碱基侵入到检测探针和待测靶标形成的双链中,形成三碱基重叠结构,flap核酸内切酶1 (FEN1)可特异地识别该结构,并将检测探针5°flap片段切下来;由于反应温度与检测探针的Tm值接近,被切割的检测探针会与靶标分离,完整的检测探针会再次与靶标退火形成切割循环,产生扩增的flap片段,该片段可在体系中与FRET探针再次杂交形成三碱基重叠结构,形成切割循环,产生放大的荧光信号。将待测位点设计在三碱基重叠结构中,当检测探针与SNP位点不匹配时,将无法形成三碱基重叠结构,不发生切割反应;而当SNP位点的碱基与检测探针匹配时,将形成侵入结构,引发切割反应,产生对应的荧光信号(突变型为FAM信号,野生型为VIC信号)。待测靶标在PCR扩增的同时会在每个循环发生级联核酸侵入反应产生荧光信号,最终形成类似于实时荧光PCR的扩增曲线,通过两种荧光信号曲线即可判断基因型。图1
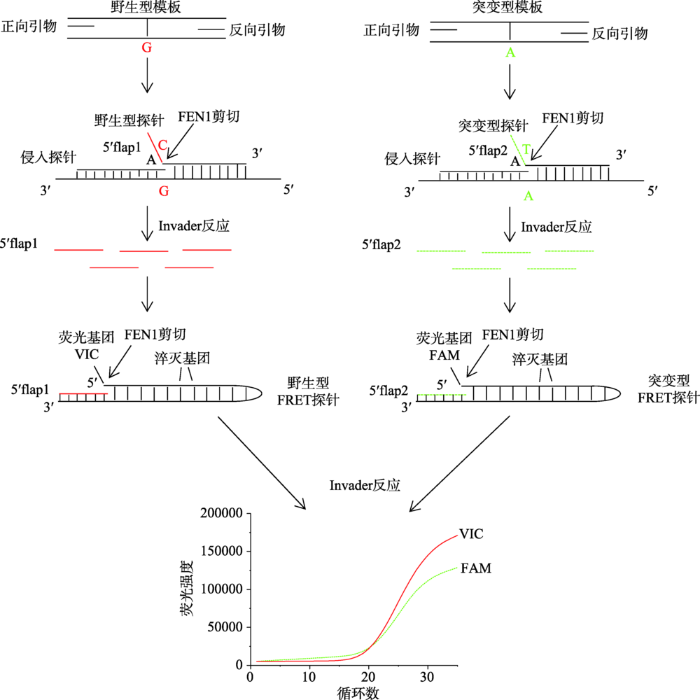 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1基于级联核酸侵入反应的实时荧光PCR检测SNP的原理
Fig. 1Schematic illustration of SNP detection using real-time PCR-based cascade invader assay
2.2 检测探针用量的优化
在级联核酸侵入反应中,影响检测特异性的最大因素为检测探针的浓度,浓度过高会造成非特异性信号,浓度过低会造成信号过低而影响曲线抬起时间,因此需要优化反应体系中野生型和突变型的检测探针。选取UGT1A1*6位点野生型(*1*1型)、杂合型(*1*6型)和突变型(*6*6型)的基因组DNA作为待测模板,浓度在10~50 ng/μL,A260/280在1.8~2.0,分型结果由焦磷酸测序确证,分别采用不同浓度的野生型和突变型检测探针进行检测。前期预实验结果发现,野生型探针会在突变型模板检测时产生较高的背景信号,而突变型探针在野生型模板检测时基本无背景信号,因此,选取低浓度野生型探针与高突变浓度型探针组合来优化探针浓度,结果如图2所示。理论上,杂合样本中野生型模板与突变型模板应该等量,两种信号应该一起出现抬起。当野生型探针浓度为0.05 μmol/L而突变型探针为1 μmol/L时,杂合型模板中突变型信号(FAM)早于野生型信号(VIC)抬起(图2A),表明野生型探针浓度偏低;增加野生型探针浓度到0.125 μmol/L后,野生型与突变型信号抬起时间基本相同,符合预期(图2B);当增加突变型探针浓度到2 μmol/L时,突变型信号抬起早于野生型信号(图2C),各浓度探针在检测纯野生或纯突变型模板时均能得到预期分型结果,因此,选择0.125 μmol/L野生型探针浓度与1 μmol/L突变型探针浓度作为最佳反应条件。图2
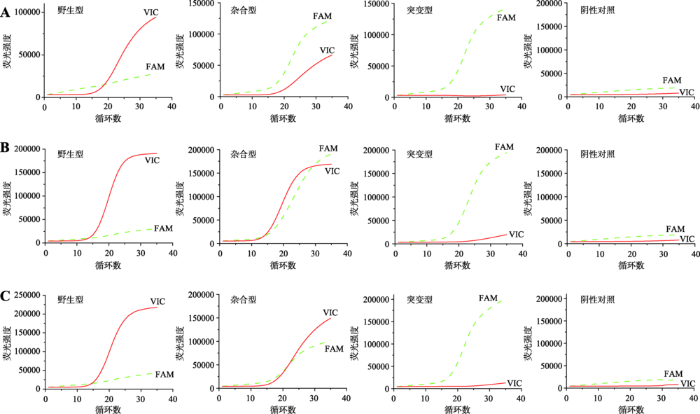 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2不同检测探针浓度对3种UGT1A1*6基因型样本的检测结果
A:野生型探针浓度为0.05 μmol/L,突变型探针浓度为1 μmol/L;B:野生型探针浓度为0.125 μmol/L,突变型探针浓度为1 μmol/L;C:野生型探针浓度为0.125 μmol/L,突变型探针浓度为2 μmol/L。
Fig. 2Detection results of three genotypes of UGT1A1*6 in different amounts of detection probes
2.3 检测口腔咽拭子DNA样本与血液DNA样本的灵敏度比较
从口腔内壁细胞中提取基因组DNA可减少对受检者的损伤,比取静脉血的应性更好。但从咽拭子中提取的DNA可能因浓度低、存在影响扩增检测的成分等原因而干扰检测结果,因此,需比较所建立方法对咽拭子样本中提取的DNA与从血液中提取的DNA的检测灵敏度是否存在差异。选取*1*6型咽拭子样本及血液提取的DNA各1例,用蒸馏水梯度稀释成10 ng/μL、2 ng/μL、0.4 ng/μL、0.08 ng/μL和0 ng/μL,每个反应加入1.5 μL DNA样本进行检测,每个浓度进行3次重复,结果如图3和图4所示。结果表明,不论对血液DNA样本还是咽拭子DNA样本,该方法均可检测低至0.6 ng的基因组DNA,咽拭子DNA可以替代血液DNA作为本研究建立方法的待测样本。图3
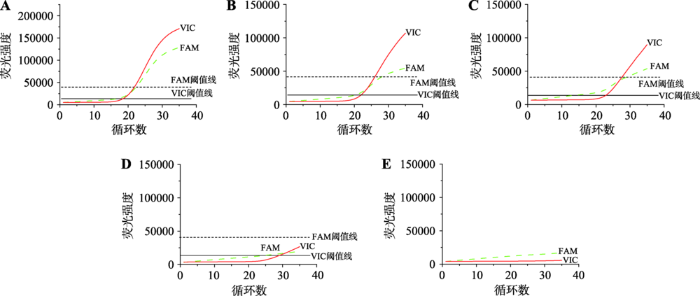 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3不同浓度*1*6型咽拭子基因组DNA的检测结果
A:15 ng基因组DNA;B:3 ng基因组DNA;C:0.6 ng基因组DNA;D:0.12 ng基因组DNA;E:空白对照。
Fig. 3Detection results of different concentrations of *1*6 type genomic DNA extracted from oral swab
图4
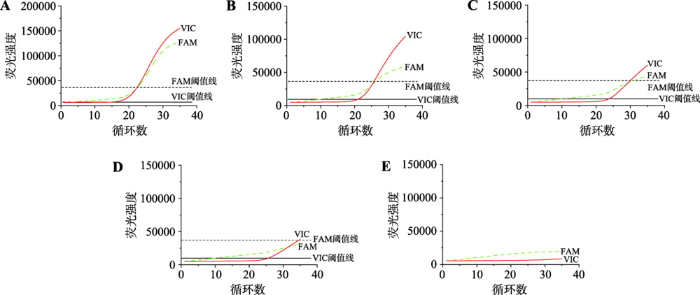 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4不同浓度*1*6型血液基因组DNA的检测结果
A:15 ng基因组DNA;B:3 ng基因组DNA;C:0.6 ng基因组DNA;D:0.12 ng基因组DNA;E:空白对照。
Fig. 4Detection results of different concentrations of *1*6 type genomic DNA extracted from blood
2.4 方法对口腔咽拭子样本检测的准确性评价
为验证所建立的方法检测口腔咽拭子样本UGT1A1*6基因型的准确性,收集28例门诊患者口腔咽拭子样本并提取了DNA,提取的DNA浓度范围在15~160 ng/μL,A260/280均在1.8~2.0。利用本研究建立的方法和焦磷酸测序法对收集的样本进行了检测,比较两种方法的分型检测结果。采用本研究建立的方法检测出UGT1A1*1*1型样本16例,*1*6型样本11例,*6*6型样本1例,该结果与焦磷酸测序法所得到的结果完全一致,因此,方法的准确性为100%,具有用于临床检测UGT1A1*6基因多态性的潜力。3 讨 论
伊立替康为半合成水溶性喜树碱,通过引起肿瘤细胞的死亡发挥抗肿瘤作用,广泛应用于多种实体瘤治疗。然而它能引起严重的不良反应包括迟发性腹泻和中性粒细胞减少,单独使用伊立替康时致死率能达到5.3%[12]。伊立替康转化成活性更强的SN-38而产生对肿瘤细胞的毒性,SN-38经肝脏中尿苷二磷酸葡萄糖醛酸基转移酶(uridine diphosphate glucuronosyl transferase, UGT)灭活,生成葡萄糖醛酸产物SN-38G,SN-38G在肠道菌群的作用下又转变成对肠道黏膜有损伤和引起迟发性腹泻的SN-38,SN-38可再经肠道中的UGT灭活,因此UGT的活性与伊立替康的不良反应息息相关[13]。药物代谢酶的基因多态性是造成个体疗效和毒性反应差异的主要因素之一,UGT的基因多态性影响SN-38和SN-38G之间的相互转化,UGT1A1*6在亚洲人群具有较高的分布频率,其基因多态性与伊立替康的不良反应相关[2,3,13]。因此,准确检测UGT1A1*6基因型可有效指导伊立替康的合理使用。基因分型检测方法很多,如焦磷酸测序法[5]、等位基因特异性PCR法[4]、基于TaqMan探针的实时荧光PCR法[6]等。然而这些方法存在开管操作易污染、特异性不够高、探针设计复杂等问题[14, 15]。虽然TaqMan探针法也是一种闭管可避免污染的操作方法,但本研究建立的基于级联核酸侵入反应的实时荧光PCR检测方法,依赖flap核酸内切酶识别侵入结构的高特异性,采用通用的FRET探针作为荧光报告探针,可以用于其他SNP位点分型检测,避免反复设计,重复合成,降低成本。该方法成功实现了对咽拭子样本中UGT1A1*6的分型检测,并且利用咽拭子样本避免了有创的静脉采血,有效提高了患者的依从性;对28例临床样本的检测结果与焦磷酸测序结果完全一致,表明方法具有很高的准确性。因此,该方法为临床进行UGT1A1*6检测来指导伊立替康的合理用药提供了一种有力工具。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]

目的:基于等位基因特异性PCR(allele-specific PCR,AS-PCR)技术,建立一种三色荧光标记复合扩增检测线粒体DNA(mtDNA) SNP的方法。方法基于AS-PCR原理,选择20个mtDNA编码区SNP位点,分为两组,分别标记FAM和HEX荧光,每个位点设计具有长度差异的两条上游(下游)等位基因特异性引物以及一条下游(上游)通用引物。结合AS-PCR技术和毛细管电泳,检测200份无关个体血样。各位点随机选取至少3个样本进行直接测序验证,并进行单倍型频率调查。结果200份血样均得到清晰分型,各位点的检测结果与直接测序结果完全一致。10μL体系下,DNA最低检测浓度为0.2 pg,当模板量为0.5~5 pg时能得到较为理想的分型图谱。在200名无关个体中,共分出15种单倍型,单倍型多样性为0.9060。结论 AS-PCR技术是一种简单、快速且有效的mtDNA SNP分型方法,适用于法庭科学检验的需求。
URL [本文引用: 2]

目的:基于等位基因特异性PCR(allele-specific PCR,AS-PCR)技术,建立一种三色荧光标记复合扩增检测线粒体DNA(mtDNA) SNP的方法。方法基于AS-PCR原理,选择20个mtDNA编码区SNP位点,分为两组,分别标记FAM和HEX荧光,每个位点设计具有长度差异的两条上游(下游)等位基因特异性引物以及一条下游(上游)通用引物。结合AS-PCR技术和毛细管电泳,检测200份无关个体血样。各位点随机选取至少3个样本进行直接测序验证,并进行单倍型频率调查。结果200份血样均得到清晰分型,各位点的检测结果与直接测序结果完全一致。10μL体系下,DNA最低检测浓度为0.2 pg,当模板量为0.5~5 pg时能得到较为理想的分型图谱。在200名无关个体中,共分出15种单倍型,单倍型多样性为0.9060。结论 AS-PCR技术是一种简单、快速且有效的mtDNA SNP分型方法,适用于法庭科学检验的需求。
[本文引用: 2]
[本文引用: 2]
URLPMID:27298347 [本文引用: 1]

Abstract Single-nucleotide polymorphisms (SNPs) in a gene sequence are markers for a variety of human diseases. Detection of SNPs with high specificity and sensitivity is essential for effective practical implementation of personalized medicine. Current DNA sequencing, including SNP detection, primarily uses enzyme-based methods or fluorophore-labeled assays that are time-consuming, need laboratory-scale settings, and are expensive. Previously reported electrical charge-based SNP detectors have insufficient specificity and accuracy, limiting their effectiveness. Here, we demonstrate the use of a DNA strand displacement-based probe on a graphene field effect transistor (FET) for high-specificity, single-nucleotide mismatch detection. The single mismatch was detected by measuring strand displacement-induced resistance (and hence current) change and Dirac point shift in a graphene FET. SNP detection in large double-helix DNA strands (e.g., 47 nt) minimize false-positive results. Our electrical sensor-based SNP detection technology, without labeling and without apparent cross-hybridization artifacts, would allow fast, sensitive, and portable SNP detection with single-nucleotide resolution. The technology will have a wide range of applications in digital and implantable biosensors and high-throughput DNA genotyping, with transformative implications for personalized medicine.
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:20129472 [本文引用: 1]

We have developed a multiplex, single nucleotide polymorphism (SNP) typing system for forensic identification, based on the Invader assay. Using this system, fluorescence data for 21 SNP loci were collected and analyzed. We used endpoint genotyping, commonly used with the Invader assay, and also developed more reliable typing criteria because endpoint typing was occasionally unable to clearly identify the genotype of some loci. Analysis of the fluorescence data identified criteria that had high reproducibility for each genotype. One such criterion is the climbing angle of the curve resulting from two-dimensional plots of the two kinds of fluorescence used for the Invader assay. The climbing angle was observed at the time during the reaction when either or both of the fluorescence intensities increased most significantly. The angles were remarkably associated with homozygous genotypes. On the other hand, all heterozygote endpoint fluorescence ratios were highly reproducible and had very little aberration. These values enabled SNP typing to be more clearly defined compared with typing using only the endpoint fluorescence ratio, which is commonly used with the Invader assay. The values suggested in this study are easily calculated from raw fluorescence data and will be useful for multiplex SNP typing based on the Invader assay.
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 2]
URLMagsci [本文引用: 1]

为了避免四酶焦测序体系中由于三磷酸腺苷双磷酸酶(apyrase)造成的测序结果偏差,文章建立了一种定量性能好的无三磷酸腺苷双磷酸酶的三酶焦测序体系。方法是将生物素修饰的DNA模板、荧光素酶和ATP硫酸化酶固定在磁性微球表面进行焦测序反应,当加入一种dNTP进行焦测序反应完后,采用磁性分离技术,除去焦测序反应产生的ATP和剩余的dNTP,然后加入另一种dNTP进行测序,按同样的方法去除影响下一轮测序反应的成分,实现循环测序。此体系能准确判读待测DNA的碱基序列,且可定量测定单核苷酸序列多态性(SNP)中两种等位基因型的相对比值。文章成功检测了16例正常人和8例唐氏综合征患者样本中21号染色体上两个杂合率较高位点(rs1042917和rs4818219)的等位基因型比值,所得结果能够明确说明待测样本中来自于父方和母方的21号染色体数目是否相等。该法具有良好的定量性能,适合于SNP等位基因型的定量分析,可以用于唐氏综合征的快速检测。
URLMagsci [本文引用: 1]

为了避免四酶焦测序体系中由于三磷酸腺苷双磷酸酶(apyrase)造成的测序结果偏差,文章建立了一种定量性能好的无三磷酸腺苷双磷酸酶的三酶焦测序体系。方法是将生物素修饰的DNA模板、荧光素酶和ATP硫酸化酶固定在磁性微球表面进行焦测序反应,当加入一种dNTP进行焦测序反应完后,采用磁性分离技术,除去焦测序反应产生的ATP和剩余的dNTP,然后加入另一种dNTP进行测序,按同样的方法去除影响下一轮测序反应的成分,实现循环测序。此体系能准确判读待测DNA的碱基序列,且可定量测定单核苷酸序列多态性(SNP)中两种等位基因型的相对比值。文章成功检测了16例正常人和8例唐氏综合征患者样本中21号染色体上两个杂合率较高位点(rs1042917和rs4818219)的等位基因型比值,所得结果能够明确说明待测样本中来自于父方和母方的21号染色体数目是否相等。该法具有良好的定量性能,适合于SNP等位基因型的定量分析,可以用于唐氏综合征的快速检测。
URLMagsci [本文引用: 1]

氯吡格雷是一种广泛用于预防静脉血栓形成的抗血小板药物。研究表明, 携带有CYP2C19基因功能缺失型等位基因CYP2C19*2、CYP2C19*3的病人, 其体内代谢氯吡格雷成为其活性形式的能力降低, 导致氯吡格雷抑制血小板聚集功能减弱。文章旨在建立一种利用高分辨率熔解曲线分析(High-resolution melting curve analysis,HRM)技术在闭合单管中同时对CYP2C19*2、CYP2C19*3两个多态性位点进行简便、准确分型的方法。本实验针对两个SNP位点分别设计特异性的HRM引物, 并在两个位点引物的5′端分别加上富含AT和GC的序列, 保证两个位点的扩增产物熔解峰无重叠。利用HRM技术, 快速、灵敏地对64例随机DNA样本的CYP2C19*2 、CYP2C19*3两个多态性位点进行了基因分型, 且HRM方法的分型结果与测序验证结果完全一致。因此, 利用HRM技术可以实现在闭合单管中简便、准确地对CYP2C19*2 、CYP2C19*3两个多态性位点同时进行基因分型。该方法有望应用于临床, 指导氯吡格雷的个体化用药。
URLMagsci [本文引用: 1]

氯吡格雷是一种广泛用于预防静脉血栓形成的抗血小板药物。研究表明, 携带有CYP2C19基因功能缺失型等位基因CYP2C19*2、CYP2C19*3的病人, 其体内代谢氯吡格雷成为其活性形式的能力降低, 导致氯吡格雷抑制血小板聚集功能减弱。文章旨在建立一种利用高分辨率熔解曲线分析(High-resolution melting curve analysis,HRM)技术在闭合单管中同时对CYP2C19*2、CYP2C19*3两个多态性位点进行简便、准确分型的方法。本实验针对两个SNP位点分别设计特异性的HRM引物, 并在两个位点引物的5′端分别加上富含AT和GC的序列, 保证两个位点的扩增产物熔解峰无重叠。利用HRM技术, 快速、灵敏地对64例随机DNA样本的CYP2C19*2 、CYP2C19*3两个多态性位点进行了基因分型, 且HRM方法的分型结果与测序验证结果完全一致。因此, 利用HRM技术可以实现在闭合单管中简便、准确地对CYP2C19*2 、CYP2C19*3两个多态性位点同时进行基因分型。该方法有望应用于临床, 指导氯吡格雷的个体化用药。
