 ,*, 高彩吉
,*, 高彩吉 ,*华南师范大学生命科学学院, 广东省植物发育生物工程重点实验室, 广州 510631
,*华南师范大学生命科学学院, 广东省植物发育生物工程重点实验室, 广州 510631Methods for TurboID-based Proximal Labeling in Plants
Jiayi Kuang, Hongqing Li, Wenjin Shen ,*, Caiji Gao
,*, Caiji Gao ,*Guangdong Provincial Key Laboratory of Biotechnology for Plant Development, School of Life Sciences, South China Normal University, Guangzhou 510631, China
,*Guangdong Provincial Key Laboratory of Biotechnology for Plant Development, School of Life Sciences, South China Normal University, Guangzhou 510631, China通讯作者: * E-mail:shenwenjin@m.scnu.edu.cn;gaocaiji@m.scnu.edu.cn
责任编辑: 白羽红
收稿日期:2021-07-1接受日期:2021-08-9
| 基金资助: |
Corresponding authors: * E-mail:shenwenjin@m.scnu.edu.cn;gaocaiji@m.scnu.edu.cn
Received:2021-07-1Accepted:2021-08-9

摘要
邻近标记作为近些年发展起来的一项检测活细胞内蛋白互作关系和亚细胞结构蛋白组的新型技术, 已成功应用于多种动植物体系的研究。该技术通过给诱饵蛋白融合一个具有特定催化连接活性的酶, 在酶的催化作用下将小分子底物(如生物素)共价连接到酶邻近的内源蛋白, 通过富集和分析被标记的蛋白可获得与诱饵互作的蛋白组。经定向进化产生的生物素连接酶TurboID具有无蛋白毒性及催化效率高的优势。利用TurboID介导的邻近标记技术分析感兴趣蛋白的邻近蛋白组, 可研究细胞内瞬时发生或微弱的蛋白互作网络, 进而解析复杂的生物学过程。该文详细描述了在拟南芥(Arabidopsis thaliana)中基于TurboID的邻近标记实验方法及注意事项, 旨在为利用这一新技术研究植物蛋白互作关系提供参考。
关键词:
Abstract
Proximity labeling (PL), a recently developed technique to detect protein-protein interactions and subcellular structural proteomes in living cells, has been successfully applied in various animal and plant systems. Proximity labeling is conducted by fusing an engineered enzyme with catalytic activity to a protein of interest (bait protein). With the catalysis of the enzyme, small molecular substrates such as biotin are covalently linked to endogenous proximal proteins, which can be further enriched and analyzed to identify the interactome of the bait protein. TurboID, a biotin ligase produced by directed evolution, has the advantages of non-toxicity and high catalytic efficiency. Using TurboID-based proximity labeling to analyze proximal proteins of bait proteins, we can study transient or weak protein interactions, which helps to understand the complex biological processes occurring inside cells. Here, we describe methods and related tips for TurboID-based proximal labeling in Arabidopsis thaliana, and hope to provide a reference for studying plant protein-protein interactions.
Keywords:
PDF (3110KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
邝嘉怡, 李洪清, 沈文锦, 高彩吉. 基于TurboID的植物蛋白邻近标记实验方法. 植物学报, 2021, 56(5): 584-593 doi:10.11983/CBB21104
Kuang Jiayi, Li Hongqing, Shen Wenjin, Gao Caiji.
蛋白互作是细胞内各项生命活动的重要基础, 研究细胞内蛋白之间的互作关系对于理解细胞内各类生物学过程的调控机制具有重要意义。近年来, 邻近标记(proximity labeling, PL)成为研究活细胞内互作蛋白组和亚细胞结构蛋白组的重要技术。该方法的原理是将一个具有特定催化连接活性的工具酶与目的蛋白融合, 通过添加小分子底物(如生物素(biotin)), 在酶的催化作用下小分子底物被活化并共价连接到酶邻近的内源蛋白, 被标记的蛋白经富集后进行质谱分析, 可鉴定目的蛋白的互作或邻近的蛋白信息(苏田等, 2020)。与传统的免疫共沉淀(Co-imunoprecipitation)等蛋白互作研究方法相比, 邻近标记可直接在自然条件下的活细胞内进行, 有利于捕获体内瞬时发生或微弱的蛋白互作关系, 能更好地理解细胞内复杂的生物学过程。在动植物中, 邻近标记技术已广泛应用于蛋白互作网络、细胞器膜接触位点和非膜性细胞器的蛋白组、蛋白拓扑结构以及蛋白与核酸的互作关系等(Lin et al., 2017; Cho et al., 2020; Yang et al., 2021)。
目前, 邻近标记技术常用的工具酶包括抗坏血酸过氧化物酶(engineered ascorbate peroxidase, APEX)与大肠杆菌生物素连接酶BirA的突变体(BirA- R118G, BioID) (Roux et al., 2012; Martell et al., 2012), 但它们在实际应用中均具有一定的局限性。其中, APEX催化效率高, 但其发挥作用需要添加底物分子过氧化氢(易对细胞产生毒性)。BioID简单无毒, 但催化效率低, 通常需要18-24小时的标记时间, 时间分辨率差, 难以研究细胞内瞬时发生的动态过程。此外, BioID的最适催化活性温度为37°C, 该温度下植物受到热胁迫, 限制了其在植物体系中的应用。2018年, 斯坦福大学的Alice Y Ting课题组通过酵母表面展示技术对BirA进行定向进化, 获得了新的生物素连接酶TurboID (Branon et al., 2018)。相比BioID, TurboID催化效率更高且对温度的要求较低, 25°C时10分钟内即可在活细胞中快速完成邻近蛋白的生物素化标记。通过将TurboID与感兴趣的目的蛋白融合表达于细胞内, 在ATP与生物素的参与下, TurboID催化生物素形成biotin-5°-AMP, biotin-5°-AMP从活性位点扩散后与邻近内源蛋白的赖氨酸结合, 从而对它们进行生物素化标记。TurboID可在活细胞中对约10 nm范围内的邻近蛋白进行生物素标记, 最后利用偶联链霉亲和素(streptavidin)的磁珠富集纯化标记的蛋白并进行质谱分析, 从而鉴定目的蛋白在物理空间上的邻近蛋白(Branon et al., 2018)。
截至目前, 基于TurboID的邻近标记技术已成功应用于植物体系。用于植物的首例研究是Zhang等(2019)利用基于TurboID的邻近标记技术在本氏烟草(Nicotiana benthamiana)中鉴定出大量可能与抗烟草花叶病毒免疫受体蛋白N发生互作的蛋白, 从中发现了新的植物免疫受体负调控蛋白UBR7。随后, Mair等(2019)将TurboID邻近标记技术应用于烟草瞬时转化体系与拟南芥(Arabidopsis thaliana)稳定转化体系, 发现TurboID可在22°C下快速催化完成植物体内蛋白的生物素标记, 且催化效率显著高于BioID。此外, Mair等(2019)还以拟南芥气孔保卫细胞特异性转录因子FAMA为目的蛋白, 利用FAMA基因特异性启动子驱动表达FAMA-TurboID融合蛋白或核定位的TurboID-NLS蛋白, 进而利用邻近标记技术鉴定了FAMA互作蛋白组和拟南芥幼苗气孔保卫细胞的核蛋白组, 进一步表明TurboID可用于植物中低水平表达蛋白的邻近蛋白组以及特定细胞器蛋白组的研究。Kim等(2019)通过将油菜素甾醇(brassinosteroid, BR)信号通路中的关键调控因子BIN2激酶与TurboID融合表达于烟草瞬时转化体系, 发现BIN2的下游底物转录因子BZR1可被生物素标记和高效检测, 且经对比发现该方法比传统免疫共沉淀技术在检测蛋白互作实验中更高效。同时, 他们利用邻近标记方法在拟南芥中鉴定了BIN2的邻近蛋白, 其中包括先前已被证明与BIN2互作的蛋白及一些未报道的邻近蛋白, 进一步丰富了BIN2介导的信号网络。Arora等(2020)应用基于TurboID的邻近标记方法在番茄(Solanum lycopersicum)毛状根培养细胞和拟南芥稳定转化悬浮细胞体系中成功鉴定了多个互作蛋白组, 证明该技术在不同类型的植物材料中均能有效捕获膜蛋白的互作蛋白。由此可见, TurboID作为一种催化效率高的生物素连接酶介导邻近标记, 已成功应用于多种植物体系的互作蛋白组和细胞器蛋白组研究。本文将介绍在拟南芥稳定转化体系中通过TurboID鉴定邻近蛋白的详细实验流程(图1), 以期为今后该方法在植物材料中的应用和推广提供参考。
图1
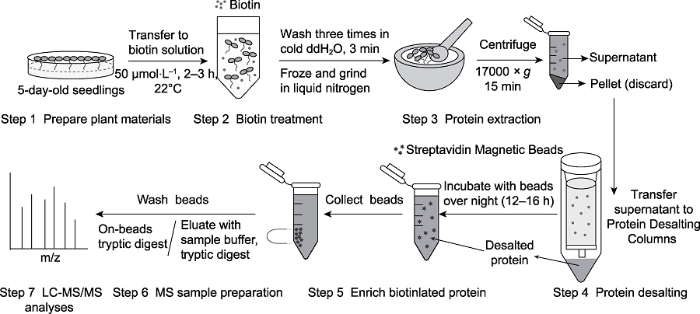 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1基于TurboID的植物蛋白邻近标记实验流程
Figure 1Experimental procedure of TurboID-based proximal labeling in plant
1 实验材料
载体: pCAMBIA1300-UBQpro::TurboID-EYFP, pCAMBIA1300-UBQpro::TurboID-EYFP-bait。菌株: 农杆菌(Agrobacterium tumefaciens)菌株GV3101。
植物材料: 表达TurboID融合蛋白的纯合转基因拟南芥(Arabidopsis thaliana L.), 野生型拟南芥(Col-0)。
2 试剂
• Sigma-Aldrich试剂: Biotin (Cat No.B4501)、EDTA (ethylenedinitrilotetraacetic acid) (Cat No.V900- 106)、KCl (Cat No.V900068)、Leupeptin (Cat No. L2884.)、NaCl (Cat No.V900058)、Na2CO3 (Cat No.V900801)、TritonTM X-100 (Cat No.V900502)和β-mercaptoethanol (Cat No.M6250)。• Sangon Biotech试剂: Bromophenol blue (Cat No.A602230)、DTT (Cat No.A620058)、Glycerol (Cat No.A501745)、Tween-20 (Cat No.A600560)、NON-Fat Powdered Milk (Cat No.A600669)、Na2 HPO4∙12H2O (Cat No.A501725)、NaH2PO4·2H2O (Cat No.A502805)、PMSF (Cat No.A610425)、Sodium deoxycholate (Cat No.A600150)、Sodium dodecyl sulfate (SDS) (Cat No.A600485)、Tris (hydroxymethyl) aminomethane (Cat No.A600194)和Urea (Cat No.A600148)。
• Caisson试剂: Murashige & Skoog (Cat No.MSP- 09-100LT)。
• Roche试剂: cOmplete™ EDTA-free Protease Inhibitor Cocktail (Cat No.04693132001)。
• Thermo试剂: Zeba™ Spin Desalting Columns (Cat No.89893)。
• MedChemExpress试剂: Streptavidin Magnetic Beads (Cat No.HY-K0208)。
• GenScript试剂: Streptavidin-HRP (Cat No.M000- 91)。
• TransGen Biotech试剂: Anti-GFP (Cat No.HT801)。
3 试剂配方
本实验中所用试剂配方见表1。Table 1
表1
表1实验所用相关试剂配方
Table 1
| Solution | Composition | Working concentration |
|---|---|---|
| Protein Extraction Buffer | Tris-HCl (pH7.5) | 50 mmol∙L-1 |
| NaCl | 150 mmol∙L-1 | |
| Sodium deoxycholate | 0.5% (w/v) | |
| SDS | 0.1% (w/v) | |
| EDTA | 1 mmol∙L-1 | |
| Triton X-100 | 1% (v/v) | |
| DTT | 1 mmol∙L-1 | |
| PMSF | 1 mmol∙L-1 | |
| Leupeptin | 10 μg∙mL-1 | |
| Protease Inhibitor Cocktail | 1× | |
| 4×SDS Sample Buffer | Tris-HCl (pH6.8) | 200 mmol∙L-1 |
| SDS | 8% (w/v) | |
| Glycerol | 40% (v/v) | |
| β-mercaptoethanol | 20% (v/v) | |
| Bromophenol blue | 0.1% (w/v) | |
| 1×PBS buffer (pH7.4) | NaH2PO4∙2H2O | 0.263 g∙L-1 |
| Na2HPO4∙12H2O | 1.856 g∙L-1 | |
| NaCl | 8 g∙L-1 | |
| KCl | 0.2 g∙L-1 | |
| 1×PBST | 1×PBS buffer containing 0.05% Tween-20 | |
| Biotin stock (stored at -20°C) | Dissolve biotin in DMSO to a final concentration of 50 mmol∙L-1 | |
新窗口打开|下载CSV
4 仪器设备
仪器设备包括植物生长培养箱(DGX-260, 宁波赛福实验仪器有限公司)、台式离心机(Fresco17/17R, Multifuge X1R, Thermo)、恒温摇床(ZWY-240, 上海智诚)、蛋白电泳系统(Bio-Rad)、化学发光成像系统(Bio-Rad)、往复式脱色摇床(TS.B-108, 其林贝尔仪器制造有限公司)、旋转混匀仪(QB-128, 其林贝尔仪器制造有限公司)和恒温金属浴(DKT200-4, 杭州米欧仪器有限公司)。5 实验设计思路
根据实验需要, 选择诱饵蛋白(bait)构建表达TurboID- bait融合蛋白的载体, 载体中可以融合荧光蛋白标签(如EYFP), 便于观察融合蛋白的亚细胞定位。本实验以目的蛋白ATG8a为例构建融合蛋白表达载体pCAMBIA1300-UBQpro::TurboID-EYFP-ATG8a, 以pCA MBIA1300-UBQpro::TurboID-EYFP作为实验对照组。将上述质粒转入农杆菌, 并利用农杆菌侵染法获得稳定表达上述融合蛋白的转基因拟南芥植株, 筛选获得单插入的纯合体株系。为获得有效的邻近蛋白信息, 需确保转基因材料内表达的融合蛋白具有正常的生物学功能。一方面, 可通过观察确定融合蛋白是否具有正确的亚细胞定位。如果融合蛋白中含荧光蛋白标签, 可在共聚焦显微镜下直接观察蛋白定位; 如果不含荧光蛋白标签, 则可利用特异识别TurboID的抗体进行免疫荧光蛋白定位分析。例如, 本实验选用的目的蛋白为拟南芥自噬调控蛋白ATG8a, 该蛋白在自噬发生后会定位到自噬体(刘洋等, 2018; Yang et al., 2020; 李喜豹等, 2021)。通过缺氮(MS-N)处理表达融合蛋白的转基因拟南芥, 并使用液泡膜v-ATPase抑制剂ConcA (concanamycinA)抑制液泡内自噬小体的降解, 在共聚焦显微镜下可观察到液泡内累积的被TurboID- EYFP-ATG8a标记的自噬小体(图2), 表明该融合蛋白TurboID-EYFP-ATG8a具有正常的生物学功能。此外, 还可通过植物材料的相应表型分析融合蛋白的生物学功能。例如, 将融合蛋白回补至相应基因功能缺陷的突变体并进行表型分析, 若可回补突变体表型, 则表明该融合蛋白具有正常的生物学功能。然后通过生物素处理上述转基因植株, 进而利用偶联链霉亲和素的磁珠富集生物素化的蛋白, 通过质谱分析可鉴定诱饵蛋白的邻近蛋白组。
图2
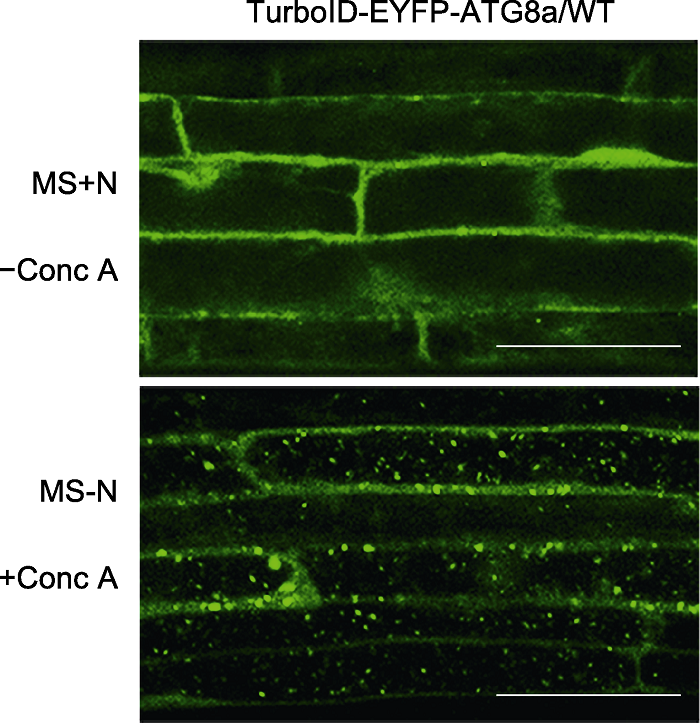 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2稳定转化拟南芥中TurboID-EYFP-ATG8a的亚细胞定位
将6日龄转TurboID-EYFP-ATG8a拟南芥幼苗转移至含1 µmol∙L-1 Conc A的MS缺氮液体培养基(MS-N)处理8小时, 在激光共聚焦荧光显微镜下观察亚细胞定位。Bars=50 μm
Figure 2Subcellular localization of TurboID-EYFP-ATG8a in stable Arabidopsis lines
6-day-old transgenic Arabidopsis seedlings overexpressing TurboID-EYFP-ATG8a were transferred to nitrogen (N)- deficient medium with 1 µmol∙L-1 Conc A for 8 h followed by confocal microscopic analysis. Bars=50 μm
6 实验程序
6.1 生物素化蛋白的检测
当融合蛋白在拟南芥中稳定表达时, TurboID可对邻近蛋白进行生物素化修饰。在材料的筛选过程中, 除抗性筛选外, 需在蛋白水平上检测转基因植物材料中融合蛋白的表达水平及TurboID的催化活性。由于链霉亲和素与生物素可高度特异性结合, 使用辣根过氧化物酶标记链霉亲和素(Streptavidin-HRP, SA-HRP)进行免疫印迹可检测生物素标记的蛋白。以野生型拟南芥(Col-0)为对照, 分析有无生物素处理时不同转基因株系生物素标记蛋白的表达水平, 从而判断不同株系TurboID的活性强弱。同时, 使用标签抗体(anti- GFP)检测融合蛋白的表达水平。此外, 使用荧光分子标记的链霉亲和素(Streptavidin-fluorophore)对生物素处理前后的植物材料进行免疫荧光标记, 检测细胞内的生物素化蛋白, 并结合融合蛋白的亚细胞定位特点进一步分析生物素标记蛋白与诱饵蛋白之间的空间分布关联性。6.1.1 生物素处理植物材料
(1) 对拟南芥种子进行表面消毒后, 4°C低温处理2- 3天。
(2) 将种子点播于1/2MS固体培养基, 放入植物生长培养箱内(22°C, 16小时光照/8小时黑暗), 培养5天。
(3) 将50 µmol∙L-1生物素溶液(用ddH2O配制)预先加入干净的12孔板内, 每孔约1 mL。
(4) 每份样品选取20 mg植物材料(约20株幼苗), 放入ddH2O中轻轻晃动以洗去多余的培养基, 用干净的纸巾或滤纸吸干幼苗表面水分。
(5) 将幼苗迅速转移至生物素溶液中, 轻轻摇晃12孔板使其被充分浸泡。
(6) 将12孔板放置于植物生长培养箱内(22°C, 全日照), 处理相应时间(一般处理30-60分钟)。
(7) 处理结束后, 迅速将幼苗转移至冰冷的ddH2O中洗涤3次, 每次1分钟。
(8) 吸干幼苗表面多余的水分, 速冻于液氮, 研磨抽提蛋白或冻存于-80°C冰箱。
(9) 无生物素处理的对照组直接取样后冻存。
6.1.2 蛋白提取和Western blotting检测生物素标记蛋白
(1) 将植物材料迅速研磨成粉末, 重悬于100 μL Protein Extraction Buffer。
(2) 4°C, 17 000 ×g离心15分钟, 取上清蛋白与4× Protein Sample Buffer (3:1, v/v)充分混合, 95°C加热5分钟, 瞬时离心后直接进行SDS-PAGE电泳或保存于-80°C冰箱。
(3) 制备5%的浓缩胶和10% (或12%)的分离胶, 每份样品取10 μL用于电泳。
(4) 用干或湿转法将凝胶上的蛋白转移至醋酸纤维膜(NC膜), 55 V, 转膜120分钟。
(5) 将硝酸纤维素(NC)膜取出, 放入约20 mL 5%脱脂牛奶(用1× PBS buffer配制), 4°C封闭过夜。
(6) 第2天, 取出NC膜放置于脱色摇床上, 室温下继续封闭30-40分钟。
(7) 倒掉5%脱脂牛奶, 加入1× PBST buffer浸没NC膜, 洗膜2次, 每次水平摇晃5分钟, 室温。
(8) 加入Streptavidin-HRP (SA-HRP, 1:2 000- 1:3 000, 用1× PBST buffer配制), 于水平摇床孵育1小时, 室温。
(9) 加入1× PBST buffer洗膜3次, 每次6-8分钟, 室温。
(10) 洗膜后加入发光液进行蛋白显影。由于链霉亲和素与生物素高度特异性结合, 蛋白显影的信号强度通常较高, 可先进行1次短时间曝光(10-30秒), 再根据实际的显影结果缩短或延长曝光时间, 以免信号过度曝光。
(11) 同时, 利用标签抗体anti-GFP (1:5 000)进行免疫印迹检测, 分析融合蛋白的表达水平, 并通过考马斯亮蓝染色(CBB)或丽春红染色(Ponceau S)分析蛋白上样情况。
正常条件下, 拟南芥含有内源生物素与少量本底生物素化蛋白。在细胞中, 生物素常作为一些羧化酶的重要辅酶参与细胞代谢, 如乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACCase)和3-甲基巴豆酰辅酶A羧化酶(3-methylcrotonoyl-CoA carboxylase, MCCase) (Alban et al., 2000)。结合本实验结果与已有文献数据, 拟南芥本底生物素化蛋白条带主要位于75 kDa以及25-37 kDa之间, 但目前尚不确定它们是哪些蛋白(Mair et al., 2019)。对比生物素处理后野生型与转基因材料中生物素标记蛋白的条带数目与大小, 后者被检测到的条带数目增多且大小明显区别于野生型, 可见TurboID对细胞内多个不同分子量的内源蛋白进行了生物素标记, 说明TurboID在这些稳定转化材料内具有催化活性。以未进行生物素处理的样品为对照, 可分析仅有内源生物素存在时TurboID的活性。经生物素处理后, 与野生型(WT)植物相比, 表达TurboID-EYFP-ATG8a与表达TurboID-EYFP的转基因拟南芥植株中生物素化蛋白条带数显著增多, 表明上述植物材料中TurboID融合蛋白有催化活性, 可以对邻近蛋白进行生物素化标记(图3A)。此外, 若材料中TurboID融合蛋白的表达水平较低, 即使进行长时间的生物素处理, 同时在显影时延长曝光时间, 可检测到的生物素标记蛋白条带仍非常少且信号弱(图3B)。因此, 对于邻近标记实验, 材料中融合蛋白的表达水平非常重要, 需筛选融合蛋白表达水平较高且TurboID活性较高的稳定转化株系用于后续实验。
图3
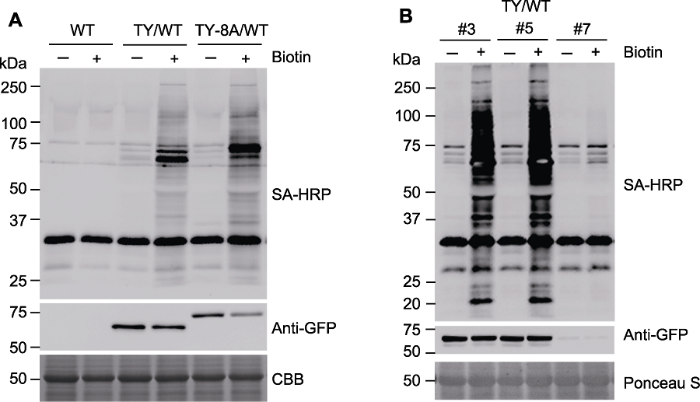 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3生物素标记蛋白的免疫印迹检测
(A) 对野生型(WT)、转TurboID-EYFP (TY/WT)和转TurboID-EYFP-ATG8a (TY-8A/WT)拟南芥幼苗分别进行生物素处理(+, 50 µmol∙L-1, 3小时)或不进行处理(-), 使用SA-HRP与anti-GFP抗体进行免疫印迹检测, 分析TurboID-EYFP的活性与表达水平; (B) 对不同株系的转TurboID-EYFP (TY/WT)拟南芥幼苗进行生物素处理(+, 50 µmol∙L-1, 3小时)或不进行处理(-), 进行免疫印迹检测。SA-HRP: 辣根过氧化物酶标记链霉亲和素; CBB: 考马斯亮蓝
Figure 3Immunoblot analysis of biotinylated protein
(A) Wild-type (WT), transgenic Arabidopsis seedlings overexpressing TurboID-EYFP (TY/WT) or TurboID-EYFP-ATG8a (TY-8A/WT) were treated with 50 µmol∙L-1 biotin (+) for 3 h or 0 (-), activity and expression of TurboID-EYFP construct were analyzed by immunoblots with SA-HRP and anti-GFP antibodies; (B) Different lines of transgenic Arabidopsis seedlings overexpressing TurboID-EYFP (TY/WT) were treated with 50 µmol∙L-1 biotin (+) for 3 h or 0 (-), followed by immunoblots. SA-HRP: Streptavidin-HRP; CBB: Coomassie Brilliant Blue
6.2 生物素处理条件的优化
在邻近标记实验中, 生物素处理时间不足或生物素浓度过低可能导致仅有极少数的邻近蛋白被生物素标记, 而处理时间过长或生物素浓度过高则可能导致许多非特异性蛋白被标记。在实验过程中, 选择合适的处理条件, 包括温度、处理时间与生物素浓度, 对成功鉴定TurboID-bait的邻近蛋白非常关键。TurboID催化的最适温度为30°C, 已有实验证明TurboID在22°C时的催化效率几乎与30°C时相同(Mair et al., 2018)。因此, 在实验中无需考虑改变植物生长的温度以改善TurboID的催化效率, 而所使用的生物素浓度和处理时间则是影响生物素标记效率的关键因素。针对不同材料, 需对生物素处理条件进行优化。选择融合蛋白表达水平适中以及TurboID活性良好的转基因植物材料, 以野生型为对照, 通过生物素浓度梯度与时间梯度实验, 选取适宜的生物素浓度与处理时间。实验中可设计生物素浓度梯度与处理时间梯度, 以野生型拟南芥(Col-0)为对照, 对表达TurboID-bait融合蛋白的转基因幼苗进行不同浓度相同时间的生物素处理, 或者相同浓度不同时间的生物素处理。提取蛋白后使用Streptavidin-HRP和anti-GFP进行免疫印迹检测, 同时进行考马斯亮蓝染色。如图4所示, 随着生物素浓度或处理时间的增加, 生物素标记的内源蛋白逐渐增多至饱和。尽管短时间的生物素处理即可检测到生物素化的内源蛋白, 但若进行质谱分析, 我们建议适当延长处理时间。根据已有文献报道和本实验结果, 我们推荐使用50 μmol∙L-1生物素处理材料2-3小时, 具体的浓度与处理时间可根据实验结果进行适当调整。
图4
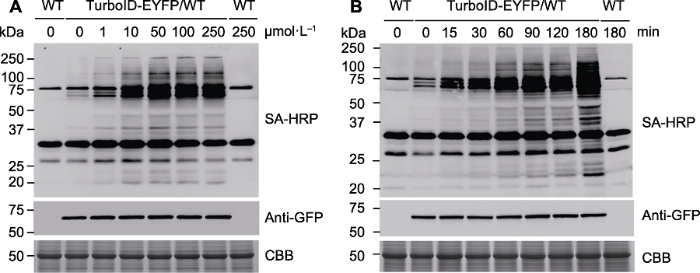 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4邻近标记中生物素浓度与处理时间的优化
(A) 使用不同浓度的生物素溶液分别对野生型和转TurboID-EYFP拟南芥处理1小时, 使用SA-HRP和anti-GFP抗体进行免疫印迹检测, 分析TurboID-EYFP的活性与表达水平; (B) 用50 µmol∙L-1生物素溶液处理野生型和转TurboID-EYFP拟南芥不同时间, 然后抽提蛋白并进行免疫印迹分析。SA-HRP: 辣根过氧化物酶标记链霉亲和素; CBB: 考马斯亮蓝
Figure 4The optimization of biotin concentration and labeling time used in proximity labeling
(A) Wild-type (WT) and transgenic Arabidopsis seedlings overexpressing TurboID-EYFP were treated with different concentrations of biotin solution for 1 h, followed by immunoblots with SA-HRP and anti-GFP antibodies to detect the proximity ligation activity and protein expression level of TurbolD-EYFP; (B) Wild-type (WT) and transgenic Arabidopsis seedings overexpressing TurboID-EYFP were treated with 50 µmol∙L-1 biotin for different times, followed by immunoblots with appropriate antibodies. SA-HRP: Streptavidin-HRP; CBB: Coomassie Brilliant Blue
6.3 邻近标记蛋白质谱检测样品的制备
由于生物素-链霉亲和素相互作用的强亲和力, 可使用偶联链霉亲和素的磁珠(Streptavidin Magnetic Beads)富集生物素标记的蛋白, 进而通过质谱鉴定生物素标记的邻近蛋白。6.3.1 生物素处理植物材料与蛋白粗提取
(1) 选取5日龄拟南芥幼苗3份, 每份0.8 g, 用于3次生物学重复。将小苗充分浸泡于50 µmol∙L-1生物素溶液中, 放置于植物生长培养箱内(22°C)处理3小时。
(2) 将幼苗迅速转移至冰冷的ddH2O中洗涤3次, 每次1分钟, 终止反应。用干净的纸巾或滤纸吸干幼苗表面的水分, 速冻于液氮, 进行蛋白抽提或冻存于-80°C冰箱。
(3) 在预冷的研钵中加入液氮, 将植物材料迅速研磨至粉末。
(4) 向每份样品加入4 mL Protein Extraction Buffer, 继续研磨均匀, 然后转移至离心管, 4°C, 17 000 ×g 离心15分钟。
(5) 将上清转移至新的1.5 mL离心管中, 4°C, 17 000 ×g再次离心15分钟。
(6) 收集上清用于后续蛋白脱盐实验, 取少许(50 μL)上清蛋白液(Extract)用于免疫印迹检测。
6.3.2 蛋白脱盐
(1) 取脱盐柱(Zeba™ Spin Desalting Columns), 剪去其底部, 放入50 mL离心管中。
(2) 拧松脱盐柱的盖子, 4°C, 1 000 ×g离心2分钟, 弃去收集的储存液。
(3) 向脱盐柱加入5 mL Protein Extraction Buffer。松盖, 4°C, 1 000 ×g离心2分钟, 弃去缓冲液。重复此步骤3次彻底洗涤脱盐柱。
(4) 向脱盐柱中心缓慢加入6.3.1节中第6步收集的蛋白样品, 待样品逐渐吸收。
(5) 松盖, 4°C, 1 000 ×g离心2分钟, 收集样品。取少许(50 μL)脱盐后的样品(After Desalting Column)用于免疫印迹检测。
6.3.3 生物素标记蛋白的富集
(1) 向5 mL离心管中加入1 mL Protein Extraction Buffer, 然后加入120 μL Streptavidin磁珠悬浊液。
(2) 轻轻晃动离心管混合磁珠与溶液, 置于磁力架进行磁性分离, 利用1 mL移液枪头套着10 μL移液枪头小心吸取上清并弃去。加入1 mL Protein Extraction Buffer, 重复洗涤1次, 磁性分离, 弃上清。
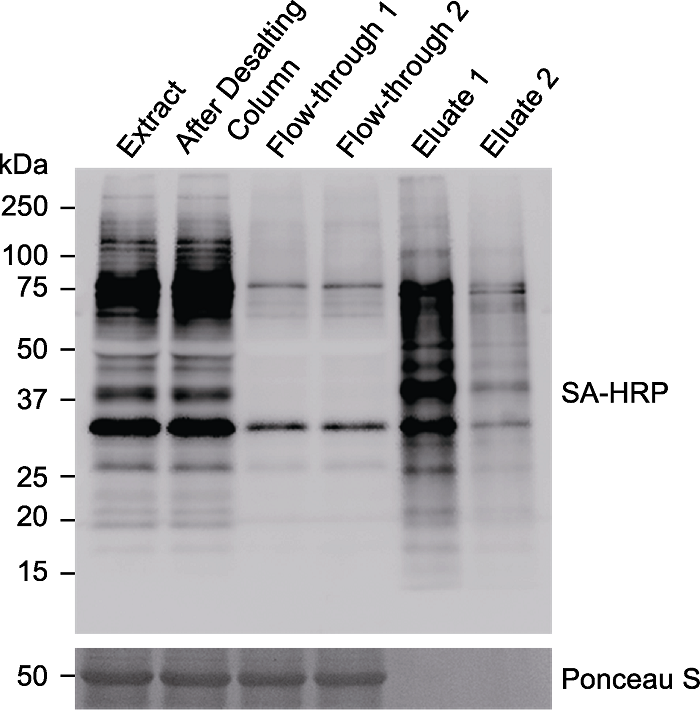
(3) 将脱盐后的蛋白加入上述洗涤后的Streptavidin磁珠中, 置于旋转混合仪上, 于4°C冷室中低速旋转孵育12-16小时。此时可将Extract和After Desalting Column分别与4× Protein Sample Buffer充分混合(3:1, v/v), 95°C加热5分钟, 保存于-80°C冰箱, 用于Western blotting分析, 检测脱盐对生物素化蛋白样品的影响。如图5所示, 脱盐后生物素化蛋白量总体上无明显损失。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5免疫印迹检测被磁珠富集的生物素化蛋白
对表达TurboID-EYFP-ATG8a的拟南芥植株进行生物素处理(50 µmol∙L-1, 3小时), 提取蛋白后使用脱盐柱进行蛋白脱盐, 将脱盐后的样品等体积分成2份, 分别与20 μL Streptavidin Magnetic Beads过夜孵育, 最后分别用20 μL或200 μL 4× SDS Sample Buffer进行洗脱。Extract: 离心后的蛋白上清; After Desalting Column: 脱盐后的蛋白样品; Flow-through 1/2: 与链霉亲和素磁珠过夜孵育后的上清; Eluate 1: 使用20 μL buffer洗脱的蛋白样品; Eluate 2: 使用200 μL buffer洗脱的蛋白样品; SA-HRP: 辣根过氧化物酶标记链霉亲和素
Figure 5Immunoblotting analyses of the biotinylated proteins enriched by Streptavidin Magnetic Beads
Arabidopsis seedlings overexpressing TurboID-EYFP-ATG8a were treated with 50 µmol∙L-1 biotin for 3 hours. The protein was extracted and desalted through Desalting Column. The desalted sample was split into two parts and incubated with 20 μL Streptavidin Magnetic Beads overnight, followed by protein elution with 20 μL or 200 μL 4× SDS Sample Buffer, respectively. Extract: Protein supernatant after centrifugation; After Desalting Column: Protein sample after desalting; Flow-through 1 or 2: Supernatant after overnight incubation with streptavidin (SA) beads; Eluate 1: Protein sample eluated with 20 μL buffer; Eluate 2: Protein sample eluated with 200 μL buffer; SA-HRP: Streptavidin-HRP
(4) 孵育完毕后, 取下离心管100-200 ×g离心30秒, 然后置于磁力架进行磁性分离。收集少量(50 μL)上清(Flow-through)与4× Protein Sample Buffer充分混合(3:1, v/v), 95°C加热5分钟, 保存于-80°C冰箱。
(5) 加入1 mL预冷的Protein Extraction Buffer至上一步的磁珠沉淀中, 温和旋转离心管, 洗涤磁珠约1分钟。磁性分离, 弃上清。加入1 mL预冷的Protein Extraction Buffer, 重复洗涤1次。
(6) 加入1 mL预冷的1 mol∙L-1 KCl溶液, 洗涤磁珠1-2分钟。磁性分离, 弃上清。
(7) 将离心管放置于磁力架。吸取1 mL冰冷的100 mmol∙L-1 Na2CO3 (现配现用)溶液沿着离心管壁缓缓加入离心管中, 然后立即吸取该溶液并弃去, Na2CO3洗涤时间需少于10秒。
(8) 沿管壁缓缓加入1 mL常温的2 mol∙L-1 Urea到10 mmol∙L-1 Tris-HCl (pH8.0, 现配现用), 加入后立即吸取该溶液并弃去, Urea洗涤时间需少于10秒。
(9) 加入1 mL预冷的Protein Extraction Buffer, 不需混匀, 磁性分离, 弃上清。然后再次加入1 mL预冷的Protein Extraction Buffer, 温和混匀, 磁性分离, 弃上清, 并重复此洗涤步骤2次。
(10) 此时可直接取少量(2%)磁珠, 洗脱蛋白用于免疫印迹检测, 其余磁珠送公司进行胰蛋白酶酶解处理(On-Beads Trypsin Digestion)和质谱分析; 也可将磁珠上的蛋白洗脱, 样品跑胶后切下目标蛋白所处位置的胶条送公司进行质谱分析。
(11) 若需切胶送样, 加入20 μL 4× SDS Sample Buffer (含2 mmol∙L-1生物素和20 mmol∙L-1 DTT), 充分涡旋振荡磁珠5分钟, 瞬时离心, 95°C加热10分钟。磁性分离, 收集上清。
(12) 再次加入20 μL 4× SDS Sample Buffer (含2 mmol∙L-1生物素和20 mmol∙L-1 DTT), 重复洗脱1次。磁性分离, 收集上清。
(13) 混合2次洗脱后收集的上清, 取少许样品(Eluate)用于免疫印迹检测, 其余样品进行SDS- PAGE电泳。制备8%浓缩胶, 加入样品, 在90 V电压下电泳15-20分钟, 然后切下蛋白胶条送公司进行蛋白酶酶解处理和质谱分析。
(14) 对保存的Extract、After Desalting Column、Flow-through和Eluate进行免疫印迹检测, 分析pull down效果。如图5所示, Streptavidin磁珠可以成功富集生物素化的蛋白(Eluate洗脱组分)。
7 注意事项
(1) 在本实验中, 如果融合蛋白的定位或功能有误, 可尝试改用生物素连接酶miniTurboID。与TurboID相比, miniTurboID分子量更小, 催化活性也较低(Branon et al., 2018)。此外, 可对载体进行优化, 如在融合部分添加连接子(linker)或调整linker的长度与氨基酸种类, 或更换载体所用的标签或连接顺序, 以帮助融合蛋白正确折叠和行使功能。在载体构建时, 添加荧光蛋白标签便于观察蛋白的亚细胞定位。若需使用其它标签, 应避免使用富含赖氨酸的标签, 如FLAG, 该标签容易被生物素修饰。(2) 稳定转化植物材料的筛选过程耗时较长。对于每种融合蛋白, 可留用3-4条转基因株系进行筛选。即使未取得纯合材料, 也可对杂合材料进行生物素处理, 提取蛋白进行免疫印迹检测, 及时分析该株系中融合蛋白的表达水平与生物素连接酶TurboID的活性。对于转基因T0代拟南芥, 可选取未抽薹植株的莲座叶进行处理, 注意需将叶片浸泡于生物素溶液真空渗透5分钟后再转移至培养箱, 便于外源生物素充分渗入叶片内。对于T1代材料, 可选取抗性幼苗直接浸泡于生物素溶液。
(3) 生物素处理结束时, 需迅速将植物材料转移至预冷的ddH2O中洗涤, 从而终止植物体内的生物素标记反应, 并去除表面的部分生物素。
(4) 粗提取的蛋白溶液中含有大量的游离生物素。与磁珠孵育过程中, 游离生物素会与生物素标记的蛋白分子竞争结合于磁珠上。为有效富集生物素化蛋白, 蛋白必须经脱盐去除游离生物素后再与磁珠进行孵育。
(5) 生物素-链霉亲和素相互作用的亲和力较强, 使用偶联链霉亲和素的磁珠可方便有效地纯化生物素标记蛋白, 但洗脱时需采用严格的变性条件。在磁珠洗涤过程中, 注意所用的Na2CO3溶液与尿素溶液需现配现用, 加入这2种溶液后要严格控制时间, 否则可能导致磁珠表面电荷发生变化而产生聚集。
(6) TurboID标记的蛋白在物理空间上靠近诱饵蛋白, 并不代表它们与诱饵蛋白间有直接相互作用, 两者之间的蛋白互作关系仍需进一步验证。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1146/arplant.2000.51.issue-1URL [本文引用: 1]
DOI:10.1105/tpc.20.00235URL [本文引用: 1]
DOI:10.1038/nbt.4201PMID:30125270 [本文引用: 3]

Protein interaction networks and protein compartmentalization underlie all signaling and regulatory processes in cells. Enzyme-catalyzed proximity labeling (PL) has emerged as a new approach to study the spatial and interaction characteristics of proteins in living cells. However, current PL methods require over 18 h of labeling time or utilize chemicals with limited cell permeability or high toxicity. We used yeast display-based directed evolution to engineer two promiscuous mutants of biotin ligase, TurboID and miniTurbo, which catalyze PL with much greater efficiency than BioID or BioID2, and enable 10-min PL in cells with non-toxic and easily deliverable biotin. Furthermore, TurboID extends biotin-based PL to flies and worms.
DOI:10.1073/pnas.1919528117URL [本文引用: 1]
[本文引用: 1]
DOI:10.3389/fpls.2017.00749URL [本文引用: 1]
DOI:10.7554/eLife.47864URL [本文引用: 4]
DOI:10.1038/nbt.2375URL [本文引用: 1]
DOI:10.1083/jcb.201112098URL [本文引用: 1]
DOI:10.1016/j.molp.2020.02.011URL [本文引用: 1]
DOI:10.1016/j.xplc.2020.100137URL [本文引用: 1]
DOI:10.1038/s41467-019-11202-zURL [本文引用: 1]
植物细胞自噬基因的功能与转录调控机制
1
2021
... 为获得有效的邻近蛋白信息, 需确保转基因材料内表达的融合蛋白具有正常的生物学功能.一方面, 可通过观察确定融合蛋白是否具有正确的亚细胞定位.如果融合蛋白中含荧光蛋白标签, 可在共聚焦显微镜下直接观察蛋白定位; 如果不含荧光蛋白标签, 则可利用特异识别TurboID的抗体进行免疫荧光蛋白定位分析.例如, 本实验选用的目的蛋白为拟南芥自噬调控蛋白ATG8a, 该蛋白在自噬发生后会定位到自噬体(
植物细胞自噬研究进展
1
2018
... 为获得有效的邻近蛋白信息, 需确保转基因材料内表达的融合蛋白具有正常的生物学功能.一方面, 可通过观察确定融合蛋白是否具有正确的亚细胞定位.如果融合蛋白中含荧光蛋白标签, 可在共聚焦显微镜下直接观察蛋白定位; 如果不含荧光蛋白标签, 则可利用特异识别TurboID的抗体进行免疫荧光蛋白定位分析.例如, 本实验选用的目的蛋白为拟南芥自噬调控蛋白ATG8a, 该蛋白在自噬发生后会定位到自噬体(
邻近标记在蛋白质组学中的发展及应用
1
2020
... 蛋白互作是细胞内各项生命活动的重要基础, 研究细胞内蛋白之间的互作关系对于理解细胞内各类生物学过程的调控机制具有重要意义.近年来, 邻近标记(proximity labeling, PL)成为研究活细胞内互作蛋白组和亚细胞结构蛋白组的重要技术.该方法的原理是将一个具有特定催化连接活性的工具酶与目的蛋白融合, 通过添加小分子底物(如生物素(biotin)), 在酶的催化作用下小分子底物被活化并共价连接到酶邻近的内源蛋白, 被标记的蛋白经富集后进行质谱分析, 可鉴定目的蛋白的互作或邻近的蛋白信息(
Biotin metabolism in plants
1
2000
... 正常条件下, 拟南芥含有内源生物素与少量本底生物素化蛋白.在细胞中, 生物素常作为一些羧化酶的重要辅酶参与细胞代谢, 如乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACCase)和3-甲基巴豆酰辅酶A羧化酶(3-methylcrotonoyl-CoA carboxylase, MCCase) (
Establishment of proximity-dependent biotinylation approaches in different plant model systems
1
2020
... 截至目前, 基于TurboID的邻近标记技术已成功应用于植物体系.用于植物的首例研究是
Efficient proximity labeling in living cells and organisms with TurboID
3
2018
... 目前, 邻近标记技术常用的工具酶包括抗坏血酸过氧化物酶(engineered ascorbate peroxidase, APEX)与大肠杆菌生物素连接酶BirA的突变体(BirA- R118G, BioID) (
... ).相比BioID, TurboID催化效率更高且对温度的要求较低, 25°C时10分钟内即可在活细胞中快速完成邻近蛋白的生物素化标记.通过将TurboID与感兴趣的目的蛋白融合表达于细胞内, 在ATP与生物素的参与下, TurboID催化生物素形成biotin-5°-AMP, biotin-5°-AMP从活性位点扩散后与邻近内源蛋白的赖氨酸结合, 从而对它们进行生物素化标记.TurboID可在活细胞中对约10 nm范围内的邻近蛋白进行生物素标记, 最后利用偶联链霉亲和素(streptavidin)的磁珠富集纯化标记的蛋白并进行质谱分析, 从而鉴定目的蛋白在物理空间上的邻近蛋白(
... (1) 在本实验中, 如果融合蛋白的定位或功能有误, 可尝试改用生物素连接酶miniTurboID.与TurboID相比, miniTurboID分子量更小, 催化活性也较低(
Split-TurboID enables contact-dependent proximity labeling in cells
1
2020
... 蛋白互作是细胞内各项生命活动的重要基础, 研究细胞内蛋白之间的互作关系对于理解细胞内各类生物学过程的调控机制具有重要意义.近年来, 邻近标记(proximity labeling, PL)成为研究活细胞内互作蛋白组和亚细胞结构蛋白组的重要技术.该方法的原理是将一个具有特定催化连接活性的工具酶与目的蛋白融合, 通过添加小分子底物(如生物素(biotin)), 在酶的催化作用下小分子底物被活化并共价连接到酶邻近的内源蛋白, 被标记的蛋白经富集后进行质谱分析, 可鉴定目的蛋白的互作或邻近的蛋白信息(
Application of TurboID mediated proximity labeling for mapping a GSK3 kinase signaling network in Arabidopsis
1
2019
... 截至目前, 基于TurboID的邻近标记技术已成功应用于植物体系.用于植物的首例研究是
Screening of proximal and interacting proteins in rice protoplasts by proximity-dependent biotinylation
1
2017
... 蛋白互作是细胞内各项生命活动的重要基础, 研究细胞内蛋白之间的互作关系对于理解细胞内各类生物学过程的调控机制具有重要意义.近年来, 邻近标记(proximity labeling, PL)成为研究活细胞内互作蛋白组和亚细胞结构蛋白组的重要技术.该方法的原理是将一个具有特定催化连接活性的工具酶与目的蛋白融合, 通过添加小分子底物(如生物素(biotin)), 在酶的催化作用下小分子底物被活化并共价连接到酶邻近的内源蛋白, 被标记的蛋白经富集后进行质谱分析, 可鉴定目的蛋白的互作或邻近的蛋白信息(
Proximity labeling of protein complexes and cell- type-specific organellar proteomes in Arabidopsis enabled by TurboID
4
2019
... 截至目前, 基于TurboID的邻近标记技术已成功应用于植物体系.用于植物的首例研究是
... )稳定转化体系, 发现TurboID可在22°C下快速催化完成植物体内蛋白的生物素标记, 且催化效率显著高于BioID.此外,
... 正常条件下, 拟南芥含有内源生物素与少量本底生物素化蛋白.在细胞中, 生物素常作为一些羧化酶的重要辅酶参与细胞代谢, 如乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACCase)和3-甲基巴豆酰辅酶A羧化酶(3-methylcrotonoyl-CoA carboxylase, MCCase) (
... 在邻近标记实验中, 生物素处理时间不足或生物素浓度过低可能导致仅有极少数的邻近蛋白被生物素标记, 而处理时间过长或生物素浓度过高则可能导致许多非特异性蛋白被标记.在实验过程中, 选择合适的处理条件, 包括温度、处理时间与生物素浓度, 对成功鉴定TurboID-bait的邻近蛋白非常关键.TurboID催化的最适温度为30°C, 已有实验证明TurboID在22°C时的催化效率几乎与30°C时相同(
Engineered ascorbate peroxidase as a genetically encoded reporter for electron microscopy
1
2012
... 目前, 邻近标记技术常用的工具酶包括抗坏血酸过氧化物酶(engineered ascorbate peroxidase, APEX)与大肠杆菌生物素连接酶BirA的突变体(BirA- R118G, BioID) (
A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells
1
2012
... 目前, 邻近标记技术常用的工具酶包括抗坏血酸过氧化物酶(engineered ascorbate peroxidase, APEX)与大肠杆菌生物素连接酶BirA的突变体(BirA- R118G, BioID) (
HY5-HDA9 module transcriptionally regulates plant autophagy in response to light-to-dark conversion and nitrogen starvation
1
2020
... 为获得有效的邻近蛋白信息, 需确保转基因材料内表达的融合蛋白具有正常的生物学功能.一方面, 可通过观察确定融合蛋白是否具有正确的亚细胞定位.如果融合蛋白中含荧光蛋白标签, 可在共聚焦显微镜下直接观察蛋白定位; 如果不含荧光蛋白标签, 则可利用特异识别TurboID的抗体进行免疫荧光蛋白定位分析.例如, 本实验选用的目的蛋白为拟南芥自噬调控蛋白ATG8a, 该蛋白在自噬发生后会定位到自噬体(
Proximity labeling: an emerging tool for probing in planta molecular interactions
1
2021
... 蛋白互作是细胞内各项生命活动的重要基础, 研究细胞内蛋白之间的互作关系对于理解细胞内各类生物学过程的调控机制具有重要意义.近年来, 邻近标记(proximity labeling, PL)成为研究活细胞内互作蛋白组和亚细胞结构蛋白组的重要技术.该方法的原理是将一个具有特定催化连接活性的工具酶与目的蛋白融合, 通过添加小分子底物(如生物素(biotin)), 在酶的催化作用下小分子底物被活化并共价连接到酶邻近的内源蛋白, 被标记的蛋白经富集后进行质谱分析, 可鉴定目的蛋白的互作或邻近的蛋白信息(
TurboID-based proximity labeling reveals that UBR7 is a regulator of N NLR immune receptor-mediated immunity
1
2019
... 截至目前, 基于TurboID的邻近标记技术已成功应用于植物体系.用于植物的首例研究是
