 ,*上海交通大学生命科学技术学院, 上海 200240
,*上海交通大学生命科学技术学院, 上海 200240Evolution and Functional Analysis of Gene Clusters in Anther Tapetum Cells of Arabidopsis thaliana
Zeyuan Zuo, Wanlin Liu, Jie Xu ,*School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China
,*School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China通讯作者:
责任编辑: 白羽红
收稿日期:2019-05-30接受日期:2019-09-19网络出版日期:2020-03-01
| 基金资助: |
Corresponding authors:
Received:2019-05-30Accepted:2019-09-19Online:2020-03-01

摘要
关键词:
Abstract
Keywords:
PDF (3514KB)摘要页面多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
左泽远, 刘琬琳, 许杰. 拟南芥花药绒毡层细胞中具有基因簇特征的基因进化和功能分析. 植物学报, 2020, 55(2): 147-162 doi:10.11983/CBB19103
Zuo Zeyuan, Liu Wanlin, Xu Jie.
基因簇(gene cluster)是指在染色体上成簇出现并协同转录的一类同源或非同源基因, 在细菌基因组中广泛存在(Worthen, 2008)。Cimermancic等(2014)利用隐马尔可夫概率模型算法从高通量微生物测序的数据中系统识别生物合成基因簇(biosynthetic gene clusters, BGC), 发现芳烯类化合物占据BGC的主导地位。基因簇通常编码一些特定化合物生物合成途径中催化某些步骤的酶, 因此在特异性化合物合成相关的合成生物学中备受关注。例如, Guenzi等(1998)解析了大肠杆菌(Escherichia coli)中丁香霉素合成酶基因簇的特性, 发现该基因簇不遵从共线性规则, 即染色体上的氨基酸结合顺序和最后多肽的氨基酸顺序不同, 更加接近真核生物多肽的表达模式。Nishida等(1999)揭示了嗜热细菌(Thermus thermophilus)通过氨基己二酸代谢路径合成赖氨酸的基因簇各基因的功能, 为探明氨基酸生物合成路径的进化历程提供了线索。
除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(Slot, 2017)。例如, 海洋放线菌(Actinoalloteichus cyanogriseus)可以产生浅蓝霉素A, 其具有良好的免疫抑制活性, 有极大的临床应用价值。Zhu等(2016)解析了海洋放线菌合成浅蓝霉素A的生物合成路径及分子机理, 并发现其相关合成基因呈现基因簇形式(Zhu et al., 2012, 2016)。著名的青霉素和头孢霉素的合成路径相关基因也以基因簇的形式存在(Díez et al., 1990; MacCabe et al., 1990)。动物中存在的主要组织相容性复合体基因簇在其固有免疫系统及适应性免疫过程中均发挥抗病原菌的作用(Klein and Figueroa, 1986)。
植物基因组中序列高度相似的同源基因成簇的现象非常普遍, 众多植物基因簇中有许多已确定的基因编码参与次生代谢途径的酶。例如, 番茄(Lycopersicon esculentum)中存在合成单萜类化合物的基因簇(Falara et al., 2011), 部分茄类植物中存在生物碱合成基因簇(Itkin et al., 2013), 在燕麦(Avena sativa)中发现了合成抗微生物的三萜类化合物的基因簇(Qi et al., 2004), 以及在玉米(Zea mays)、小麦(Triticum aestivum)和拟南芥(Arabidopsis thaliana)等大量植物中存在细胞色素P450合成基因簇(吕海舟等, 2017)。绝大部分基因簇中已鉴定的基因均可编码参与次生代谢途径的酶。而在植物发育过程中, 基因簇基因的功能(尤其是异源基因簇基因的功能)尚缺少明确报道。
植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(Mascarenhas, 1990)。同时, 花粉的产生也是植物进化过程中的一个飞跃, 是种子植物特有且高度保守的生殖发育中的核心过程(Lora et al., 2009; Wilson and Zhang, 2009)。花粉的形成依赖于花药2种细胞的分化和作用: 生殖细胞(小孢子母细胞)和形成绒毡层细胞的体细胞(Yang et al., 2003)。不同物种的形态和功能出现是新基因产生和进化的结果, 特别是基因重复(gene duplication)在基因的新功能进化和物种的丰富多样性中发挥了关键作用(朱子超等, 2009; 孙红正和葛颂, 2010)。在开花植物花药进化过程中, 花药特异表达基因的产生是否也与基因重复相关? 是否以基因簇的形式存在? 以及它们具有哪些生物学功能? 这些问题都是我们关注的重点。
因此, 本研究通过筛选拟南芥花药发育中特异表达且有基因簇特征的基因, 分析候选基因的分子特征、表达调控、基因组进化层(genomic phylostratigraphy)、基因重复以及基因功能, 以期从基因来源进化和功能预测两个角度来阐明植物花药发育中基因簇基因的产生和生物学功能, 为深入研究植物基因簇基因的遗传学功能、基因进化和物种进化关系提供科学依据。
1 材料与方法
1.1 花药发育阶段高表达基因的筛选
开花植物雄性生殖发育过程中, 绒毡层作为花药最内层细胞, 为雄配子体(花粉)的形成发育提供所需的营养物质及细胞环境。Li等(2017)使用激光显微切割捕获技术(laser microdissection and pressure catapulting, LMPC)获得了拟南芥绒毡层细胞在花药发育6-7和8-10阶段的转录组数据(RNA-Seq)。我们在该研究的转录组数据基础上, 将花药发育6-7和8-10阶段的数据合并, 选择FPKM (fragments per kilobase of transcript per million fragments mapped)大于100的基因作为花药发育阶段的高表达基因。1.2 基因簇基因的筛选
从TAIR (The Arabidopsis Information Resource)数据库中获取拟南芥全基因组基因ID, 将其按照物理位置的顺序从1开始以自然数进行编码。假设基因A和基因B的编码数为a和b, 则基因A和基因B之间间隔|a-b-1|个基因。我们据此筛选出间隔基因数≤3的基因并分类, 认为其具有潜在的基因簇形式。1.3 花药特异性基因的筛选
从BAR (The Bio-analytic Resource for Plant Biology)数据库中下载前期分析得到的拟南芥花药发育相关基因的各时期和组织表达量数据, 确定花粉发育期间相关基因的特异性表达情况, 进一步筛选出潜在的特异性表达基因。然后将其匹配到对应的基因簇中, 并对基因簇两端和中间进行一定程度的扩大筛选, 将扩大筛选后的基因并入基因簇中, 最终得到与花药发育相关的特异性表达的基因簇基因。1.4 染色质物理位置及基因结构分析
利用TAIR数据库中拟南芥基因物理位置的相关信息, 将相关数据下载后, 利用TB-tools工具进行可视化处理(Chen et al., 2018)。此外, 对属于同一基因簇的基因绘制基因结构。基因结构由非编码区、内含子和外显子组成, 还包括基因注释及转录方向。1.5 表达聚类及共表达分析
从BAR数据库中下载候选基因在各组织和各时期的表达量数据, 将相似组织和时期的表达量取平均值, 同时去除对照组(control group)数据, 进而得出主要组织和部位的表达量, 绘制表达量热图。随后取所有基因的表达量数值计算皮尔森相关系数矩阵, 筛选出相关系数大于0.9的基因, 绘制共表达网络。用HEML和CYTOSCAPE软件绘制相关图表。1.6 进化树构建
从TAIR数据库中下载所有候选基因的DNA序列, 用Cluster W程序进行序列比对。使用MEGA7软件通过极大似然法分别对所有候选基因以及基因簇内部基因构建进化树, 使用bootstrap=1 000进行验证, 其余参数均为默认值(Kumar et al., 2016)。1.7 基因组进化层分析
新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环。如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(Domazet-Lošo et al., 2007; Domazet-Lošo and Tautz, 2010)。该方法基于以下假设: 至少有1个基因的重要分支在产生之后保持其原有功能。而基因重复后产生的完全新功能化(neofunctionalization)是较小概率事件, 可以认为只会出现在原始基因(founder gene)中。相对而言, 亚功能化(sub-functionalization)和部分新功能化更为常见(Force et al., 1999; Prince and Pickett, 2002; Thornton, 2006)。我们将筛选出的序列与NCBI的BLAST非冗余数据库(non-redundant database) (E值为10-3)进行比对, 并将获取其中每类最古老的物种作为其原始基因的层级(phylostratum), 从而确定基因出现的时间。1.8 基因重复分析
将拟南芥全基因组序列通过本地BLAST进行比对, 使用MCScan X软件进行基因重复事件、串联重复以及共线性分析。相关数据来源于TAIR, 所有参数均为默认值(Wang et al., 2012)。1.9 GO富集分析
将候选基因提交到PATHER数据库中, 选择背景数据库为拟南芥, 对相关拟南芥基因进行分子功能(molecular function)、细胞结构(cellular components)和生物过程(biological process) 3方面的注释来分析基因功能。P值用于显著性判断, P<0.05为显著(Carbon et al., 2009; Mi et al., 2017)。2 结果与讨论
2.1 花粉发育过程中特异性表达基因簇基因的筛选
为获得拟南芥花药发育过程中特异高表达及具有基因簇特征的候选基因, 我们设计并确定筛选过程。首先, 在已发表文献的转录组数据基础上(Li et al., 2017), 筛选FPKM值大于100的基因为花药高表达基因, 共获得花药6-10阶段高表达基因1 045个, 其中272个基因具有基因簇特征, 分属126个基因簇。其次, 为进一步筛选花药特异性表达基因, 我们通过BAR数据库中的表达特征信息, 确定其中24个基因(属于13个基因簇)在花药绒毡层细胞中相对特异性表达。同时, 为了防止潜在基因遗漏(检测缺失或基因表达量较低导致的漏筛), 及保证基因的连贯性, 我们又分别对这13个基因簇向两侧及中间进行扩大基因筛选, 确保每个基因簇包含至少2个基因。最终筛选出84个在花药发育阶段特异性高表达的基因(分属13个基因簇)作为候选基因(图1)。图1
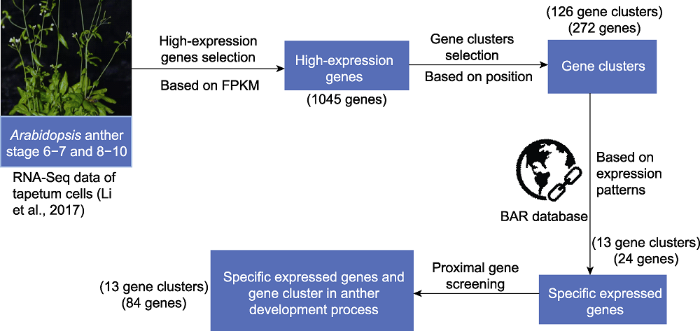 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1拟南芥花药发育中特异性表达基因簇基因的筛选流程
Figure 1Screening process of anther-specific expressed clustered genes during Arabidopsis anther development
在获得花药发育中特异性高表达和具有基因簇特征的基因后, 我们进一步利用TAIR数据库对候选基因的基本信息进行注释, 包括基因ID、基因簇序号、蛋白质分子量、等电点、细胞位置和跨膜域数量(表1)。结果表明, 候选基因的蛋白质分子量(MW)差异较大, 范围在5 641.3-232 851.8 kDa之间, 即使属于同一基因簇的基因也是如此; 等电点(PI)大多集中在7-10之间, 少部分蛋白质出现了3和4偏酸性的等电点。这些等电点偏酸性的蛋白质大多数为分泌蛋白(secreted); 候选基因的跨膜域数量(TM)基本为0或1, 只有3个基因跨膜域数量≥10。跨膜域数量和蛋白质分子量以及等电点之间并无直接关系。
Table 1
表1
表1拟南芥花药发育中特异性表达基因簇基因的基本信息
Table 1
| Cluster No. | AGI | MW (kDa) | PI | Location | TM | Description |
|---|---|---|---|---|---|---|
| 1 | AT1G12060 | 24786.8 | 9.09 | Undefined | 0 | BCL-2-associated athanogene 5 |
| 1 | AT1G12064 | 11961.3 | 9.24 | Secreted | 1 | Unknown protein |
| 1 | AT1G12070 | 25456.4 | 4.52 | Other | 0 | Immunoglobulin E-set superfamily protein |
| 1 | AT1G12080 | 15381.7 | 3.74 | Undefined | 0 | Vacuolar calcium-binding protein-related |
| 1 | AT1G12090 | 13912.3 | 8.15 | Secreted | 0 | Extensin-like protein |
| 1 | AT1G12100 | 13909.2 | 6.90 | Secreted | 0 | Bifunctional inhibitor/lipid-transfer protein/seed storage |
| 1 | AT1G12110 | 64921.2 | 8.57 | Other | 11 | Nitrate transporter 1.1 |
| 2 | AT1G61560 | 67225.5 | 10.06 | Undefined | 8 | Seven transmembrane MLO family protein |
| 2 | AT1G61563 | 8906.4 | 9.93 | Secreted | 1 | Ralf-like 8 |
| 2 | AT1G61566 | 8259.6 | 9.93 | Secreted | 1 | Ralf-like 9 |
| 2 | AT1G61570 | 9411.6 | 5.03 | Undefined | 0 | Translocase of the inner mitochondrial membrane 13 |
| 2 | AT1G61575 | 5641.3 | 4.50 | Undefined | 0 | Unknown protein |
| 3 | AT1G74540 | 56740.6 | 8.36 | Undefined | 1 | Cytochrome P450, family 98 |
| 3 | AT1G74550 | 55418.9 | 8.88 | Undefined | 0 | Cytochrome P450, family 98 |
| 4 | AT1G75880 | 41333.3 | 8.23 | Secreted | 1 | SGNH hydrolase-type esterase |
| 4 | AT1G75890 | 42126.2 | 8.44 | Secreted | 0 | GDSL-like lipase/Acylhydrolase |
| 4 | AT1G75900 | 39815.4 | 5.47 | Secreted | 0 | GDSL-like lipase/Acylhydrolase |
| 4 | AT1G75910 | 37910.6 | 10.30 | Secreted | 0 | Extracellular lipase 4 |
| 4 | AT1G75920 | 39254.5 | 10.06 | Secreted | 0 | GDSL-like lipase/Acylhydrolase |
| 4 | AT1G75930 | 38595.7 | 9.89 | Secreted | 0 | Extracellular lipase 6 |
| 4 | AT1G75940 | 61674.7 | 6.04 | Secreted | 1 | Glycosyl hydrolase superfamily protein |
| 5 | AT2G47010 | 55049.8 | 6.50 | Secreted | 0 | Unknown protein |
| 5 | AT2G47020 | 46827.8 | 7.36 | Mitochondrion | 0 | Peptide chain release factor 1 |
| 5 | AT2G47030 | 64137.1 | 9.44 | Secreted | 1 | Plant invertase/pectin methylesterase inhibitor superfamily protein |
| 5 | AT2G47040 | 64727.7 | 9.32 | Secreted | 1 | Plant invertase/pectin methylesterase inhibitor superfamily protein |
| 5 | AT2G47050 | 23775.5 | 9.35 | Secreted | 1 | Plant invertase/pectin methylesterase inhibitor superfamily protein |
| 5 | AT2G47060 | 43753.3 | 7.67 | Other | 0 | Protein kinase superfamily protein |
| 6 | AT3G07810 | 52055.5 | 8.81 | Other | 0 | RNA-binding (RRM/RBD/RNP motifs) family protein |
| 6 | AT3G07820 | 41681.1 | 7.34 | Secreted | 1 | Pectin lyase-like superfamily protein |
| 6 | AT3G07830 | 42912.9 | 8.69 | Secreted | 0 | Pectin lyase-like superfamily protein |
| 6 | AT3G07840 | 42591.5 | 8.08 | Secreted | 0 | Pectin lyase-like superfamily protein |
| 6 | AT3G07850 | 45599.9 | 8.43 | Secreted | 1 | Pectin lyase-like superfamily protein |
| 6 | AT3G07860 | 18672.5 | 10.45 | Other | 0 | Ubiquitin-like superfamily protein |
| 7 | AT3G28730 | 71645.6 | 5.56 | Undefined | 0 | High mobility group |
| 7 | AT3G28740 | 57691.8 | 9.23 | Secreted | 1 | Cytochrome P450 superfamily protein |
| 7 | AT3G28750 | 35478.4 | 10.45 | Secreted | 0 | Unknown protein |
| 7 | AT3G28760 | 49598.7 | 6.72 | Undefined | 0 | Unknown protein |
| 7 | AT3G28770 | 232851.8 | 4.73 | Secreted | 1 | Protein of unknown function (DUF1216) |
| 7 | AT3G28780 | 61329 | 3.92 | Secreted | 1 | Protein of unknown function (DUF1216) |
| 7 | AT3G28790 | 62765.8 | 10.04 | Secreted | 1 | Protein of unknown function (DUF1216) |
| 7 | AT3G28810 | 47872.2 | 9.70 | Secreted | 0 | Protein of unknown function (DUF1216) |
| 7 | AT3G28820 | 47786 | 9.38 | Secreted | 0 | Protein of unknown function (DUF1216) |
| 7 | AT3G28830 | 55500.6 | 10.42 | Secreted | 1 | Protein of unknown function (DUF1216) |
| 7 | AT3G28840 | 36875.3 | 10.06 | Secreted | 0 | Protein of unknown function (DUF1216) |
| 7 | AT3G28850 | 48178.4 | 4.72 | Undefined | 0 | Glutaredoxin family protein |
| 7 | AT3G28860 | 136786.9 | 8.37 | Other | 10 | ATP binding cassette subfamily B19 |
| 8 | AT4G29290 | 8554.9 | 6.92 | Secreted | 1 | Low-molecular-weight cysteine-rich 26 |
| 8 | AT4G29300 | 8510.8 | 4.40 | Secreted | 1 | Low-molecular-weight cysteine-rich 27 |
| 8 | AT4G29305 | 8359.7 | 7.91 | Secreted | 1 | Low-molecular-weight cysteine-rich 25 |
| 8 | AT4G29310 | 45655.6 | 9.63 | Undefined | 0 | Protein of unknown function (DUF1005) |
| 8 | AT4G29330 | 29213.2 | 10.33 | Other | 6 | DERLIN-1 |
| 8 | AT4G29340 | 14418.3 | 4.84 | Other | 0 | Profilin 4 |
| 8 | AT4G29350 | 13997.8 | 4.67 | Other | 0 | Profilin 2 |
| 9 | AT5G07410 | 39336.9 | 8.70 | Secreted | 1 | Pectin lyase-like superfamily protein |
| 9 | AT5G07420 | 39658 | 9.40 | Secreted | 0 | Pectin lyase-like superfamily protein |
| 9 | AT5G07430 | 39910.3 | 9.11 | Secreted | 0 | Pectin lyase-like superfamily protein |
| 9 | AT5G07440 | 44698.7 | 6.51 | Undefined | 0 | Glutamate dehydrogenase 2 |
| 9 | AT5G07450 | 25004.5 | 6.01 | Undefined | 0 | Cyclin p4;3 |
| 10 | AT5G07510 | 18462.4 | 12.43 | Secreted | 3 | Glycine-rich protein 14 |
| 10 | AT5G07520 | 21482.5 | 10.55 | Secreted | 3 | Glycine-rich protein 18 |
| 10 | AT5G07530 | 53192.9 | 11.13 | Undefined | 3 | Glycine rich protein 17 |
| 10 | AT5G07540 | 22300.1 | 10.85 | Secreted | 3 | Glycine-rich protein 16 |
| 10 | AT5G07550 | 10682.5 | 10.13 | Secreted | 2 | Glycine-rich protein 19 |
| 10 | AT5G07560 | 15458.3 | 11.20 | Other | 3 | Glycine-rich protein 20 |
| 10 | AT5G07570 | 148459.7 | 3.36 | Secreted | 10 | Glycine/proline-rich protein |
| 11 | AT5G17450 | 16995.5 | 10.18 | Undefined | 0 | Heavy metal transport/detoxification |
| 11 | AT5G17460 | 35918.8 | 8.34 | Undefined | 0 | Unknown protein |
| 11 | AT5G17470 | 16320.1 | 4.01 | Other | 0 | EF hand calcium-binding protein family |
| 11 | AT5G17480 | 9048 | 4.30 | Other | 0 | Pollen calcium-binding protein 1 |
| 11 | AT5G17490 | 57326.1 | 4.49 | Other | 0 | RGA-like protein 3 |
| 11 | AT5G17500 | 59551 | 8.19 | Secreted | 1 | Glycosyl hydrolase superfamily protein |
| 12 | AT5G53800 | 41786.3 | 10.38 | Other | 0 | Unknown protein |
| 12 | AT5G53810 | 41944.2 | 5.17 | Other | 0 | O-methyltransferase family protein |
| 12 | AT5G53820 | 7028.7 | 10.26 | Undefined | 0 | Late embryogenesis abundant protein (LEA) family protein |
| 12 | AT5G53830 | 26918.8 | 10.18 | Undefined | 0 | VQ motif-containing protein |
| 12 | AT5G53840 | 50906.6 | 7.31 | Other | 0 | F-box/RNI-like/FBD-like domains-containing pro- tein |
| 12 | AT5G53850 | 56520.1 | 6.14 | Undefined | 0 | Haloacid dehalogenase-like hydrolase family protein |
| 13 | AT5G67460 | 41995.4 | 8.16 | Secreted | 0 | O-glycosyl hydrolases family 17 protein |
| 13 | AT5G67470 | 98648.8 | 9.91 | Secreted | 2 | Formin homolog 6 |
| 13 | AT5G67480 | 43817.7 | 9.59 | Undefined | 0 | BTB and TAZ domain protein 4 |
| 13 | AT5G67490 | 11869.8 | 4.25 | Undefined | 0 | Unknown protein |
| 13 | AT5G67500 | 32904.9 | 7.68 | Other | 0 | Voltage dependent anion channel 2 |
| 13 | AT5G67510 | 16790.3 | 11.74 | Undefined | 0 | Translation protein SH3-like family protein |
| 13 | AT5G67520 | 34064.3 | 9.29 | Undefined | 0 | Adenosine-5'-phosphosulfate (APS) kinase 4 |
新窗口打开|下载CSV
2.2 染色质位置及基因结构
为了更直观地展现花药发育过程中特异性高表达基因和基因簇之间的位置效应, 我们通过TAIR数据库对筛选出的84个候选基因进行染色质位置效应分析。结果(图2A)表明, 分属13个基因簇的84个候选基因在染色体上具有明显相对集中的基因簇特征。候选基因在各条染色体上均有分布, 但主要分布于1、3和5号染色体, 在2和4号染色体仅分别存在1个基因簇, 基因数目也分别仅占总数的7.14%和8.33% (图2B)。
图2
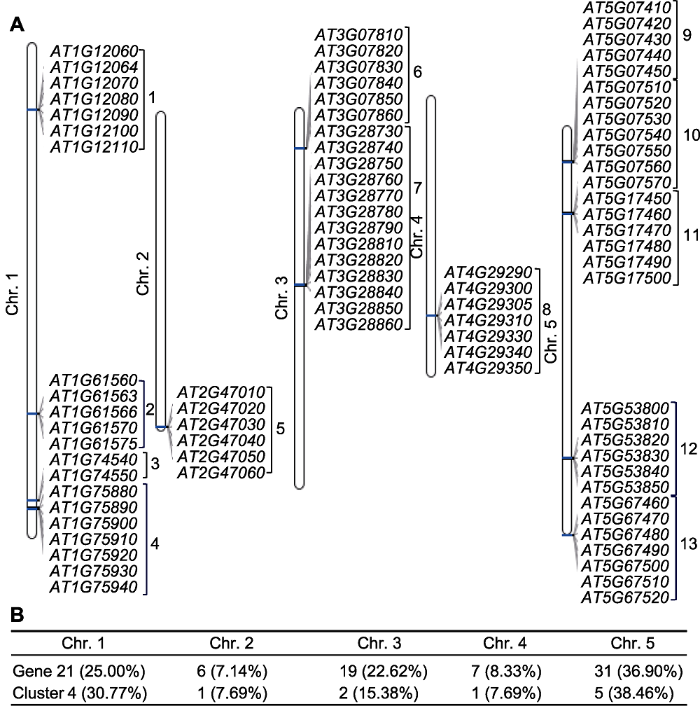 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2拟南芥花药发育特异表达的84个候选基因的染色体分布
(A) 候选基因在1-5号染色体上的分布情况(数字代表基因所属的基因簇); (B) 候选基因在染色体上具体分布的数目及比例
Figure 2Chromosome distribution of anther-specific expressed 84 candidate genes during Arabidopsis anther development
(A) Distribution of candidate genes on 1-5 chromosome (Numbers represent which cluster genes belong to); (B) Number and proportion of candidate genes distributed on chromosome
为进一步了解这些具有共表达特性的基因簇基因在转录调控和基因结构上的内在关系, 我们分析了候选基因簇基因的基因结构(图3)。结果表明, 形成同一基因簇的同一家族基因往往具有非常相似的基因结构和转录方向, 甚至基因的长度也十分相近。例如, 第3号染色体上的Ralf-like家族的AT1G61563和AT1G61566 (图3A)以及Pectin lyase-like家族的AT3G07820、AT3G07830、AT3G07840和AT3G07850 (图3B)。但来自不同家族异源基因的基因簇各基因, 其基因结构和转录方向差异较大。例如, 第1号染色体上的AT1G12060、AT1G12064、AT1G12070、AT1G12080、AT1G12090、AT1G12100和AT1G12110 (图3C)。同一基因簇内部基因转录方向的差异暗示, 尽管这些候选基因簇基因在转录水平上具有类似的表达模式, 但它们之间的转录调控机制可能不同, 同源家族基因的转录调控比较一致, 但异源基因簇基因的转录调控相对复杂。
图3
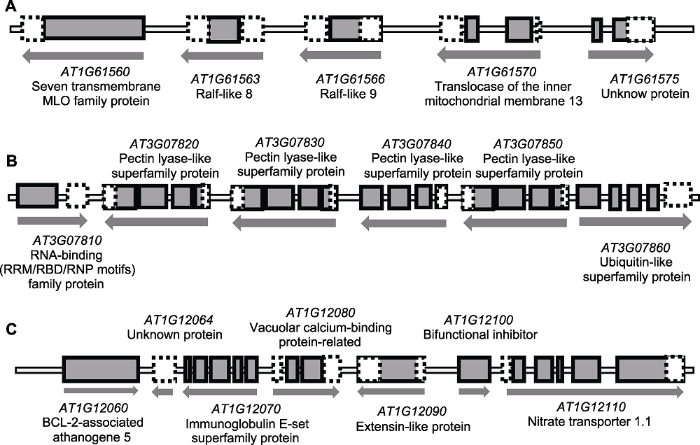 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3拟南芥花药发育特异表达基因簇基因的结构和转录方向
(A) Ralf-like家族基因簇基因结构; (B) Pectin lyase-like家族基因簇基因结构; (C) AT1G12060-AT1G12110基因簇基因结构。图中包含非编码区域和未知蛋白(虚线白框)、外显子(黑边灰框)、内含子(外显子之间的间隔)、转录方向(下方黑色箭头)和基因名注释。长度按照实际长度等比例近似缩放。
Figure 3Gene structure and transcriptional direction of anther-specific expressed clustered genes during Arabidopsis anther development
(A) Gene structure of Ralf-like family gene cluster; (B) Gene structure of pectin lyase-like family gene cluster; (C) Gene structure of AT1G12060-AT1G12110 gene cluster. Dotted line with white square: untranslated region and unknown protein; Black side with grey square: exon; Interval between exons: intron; Black arrow below: transcription direction and gene annotations. Size of gene is approximately proportional to the actual size.
2.3 基因表达聚类及共表达特性
为进一步全面分析84个花药高表达基因簇基因在整个植物发育中的表达特性, 我们在BAR数据库中获取候选基因的表达量数据, 合并为主要的12个组织部位, 然后绘制了表达量聚类热图。结果(图4A)表明, 有24个候选基因(黑框部分)相对特异地在植物生殖发育过程中的花(flower)、花粉管(pollen tubes)和花粉(pollen)阶段高表达; 13个候选基因(白框部分)在花发育中有特异性高表达。表达聚类热图(图4A)结果暗示, 37个在植物雄性生殖发育中相对特异高表达的基因可能对植物开花功能的出现或者维持是必需的。同时, 我们还分析了这些候选基因在转录水平上的基因共表达情况, 结果表明, 无论是24个特异性基因, 还是扩大筛选的候选基因, 它们之间均存在共同的共表达网络, 其中有2个最主要共表达网络(图4B)。候选基因的共表达分析表明, 这些基因至少在转录水平上存在一定的关联, 为后续探明它们之间的调控关系和功能互作提供一定的科学依据。图4
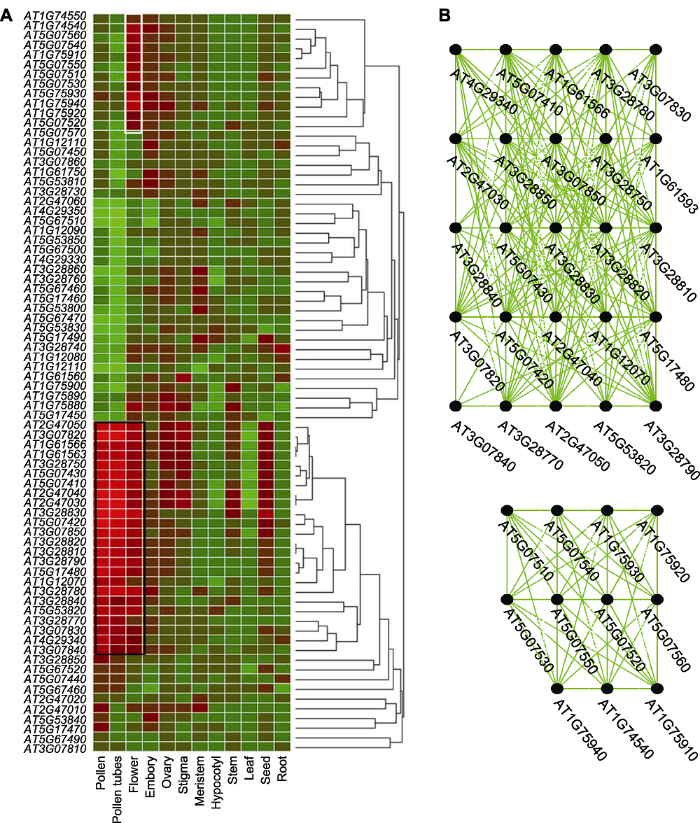 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4拟南芥花药发育特异表达候选基因的表达分析
(A) 表达聚类热图, 组织部位包括根、种子、叶、茎、子叶下轴、分生组织、柱头、子房、胚、花、花粉管和花粉; (B) 共表达网络
Figure 4Expression-pattern analysis of anther-specific expressed candidate genes during Arabidopsis anther development
(A) Clustered expression heatmap, containing tissues such as root, seed, leaf, stem, hypocotyl, meristem, stigma, ovary, embryo, flower, pollen tube, and pollen; (B) Co-expression network
2.4 进化树构建
为进一步探索候选基因之间的进化和亲缘关系, 我们根据DNA序列构建基因进化树并分析基因之间的亲缘关系。结果(图5A)显示, 属于同一基因簇的基因通常具有较近的亲缘关系, 如Cluster 3、Cluster 7、Cluster 9和Cluster 10; 但也存在部分基因簇内部基因亲缘关系较远的特例, 如Cluster 1和Cluster 13。其中, AT1G12070和AT1G12090是我们最初筛选出的24个花药发育阶段特异性高表达基因, 但它们在1号基因簇内部的亲缘关系相对较远(图5B)。此外, 结合表达聚类热图(图4A), 我们还发现AT1G12110和AT1G12080的亲缘关系在基因簇内部较远, 但是表达特征却较为相似。因此, 我们初步推测二者在转录调控中可能相对保守。图5
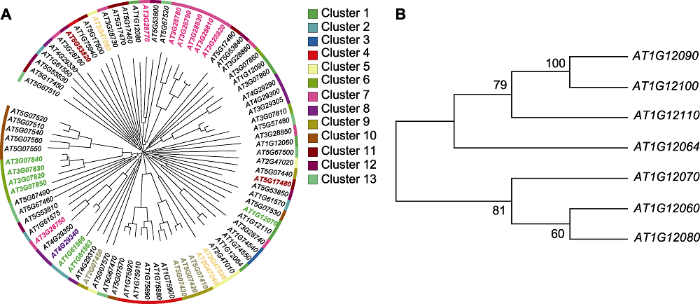 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5拟南芥花药发育特异表达候选基因进化树
(A) 候选基因进化树上彩色的基因ID代表初步筛选的24个特异性高表达基因, 外侧的颜色条代表筛选的84个基因, 同一颜色带属于同一基因簇; (B) 1号基因簇进化树, 分支上的数字来自bootstrap验证。
Figure 5Phylogenetic trees of anther-specific expressed candidate genes during Arabidopsis anther development
(A) Phylogenetic tree of candidate genes, the colored gene ID in phylogenetic tree represents 24 primarily screened specific high-expression genes, outside color strips represent 13 clusters, with 84 screened genes, and genes under the same color belonging to same cluster; (B) Phylogenetic tree of gene cluster 1, numbers on branches represent bootstrap validation.
2.5 基因组进化层分布特征
基因组进化层可以揭示宏观进化中重要的适应性事件, 并对基因功能做出一定的解释, 是从宏观层面上分析基因起源和进化的重要手段(Domazet-Lošo et al., 2007; Domazet-Lošo and Tautz, 2010)。因此, 为进一步探索筛选出的84个花药高表达基因簇候选基因的出现时间, 我们从NCBI分类学数据库(https:// www.ncbi.nlm.nih.gov/taxonomy)以及依据文献Cui等(2015)收集的相关种系数据绘制基因组进化层图。结果表明, 所有84个基因的起源都集中在PS1-PS2以及PS9-PS11 (图6)。其中PS1和PS2是原核生物层和真核生物层, 代表了生命体存在所需的最基本功能。在84个候选基因中有20个(23.81%)属于这一层, 结合之前的基因表达特征分析(图4A), 表明这些基因具有广泛表达的特点。因此, 我们初步推测这些基因在植物形成最初阶段就已经存在并具有重要的基本功能, 同时, 在植物的生殖发育阶段, 基因有可能分化出新的功能参与花药的发育过程。其余大部分基因属于代表开花植物出现和进化历程的PS9-PS11阶段。其中, 分别有35个(41.67%)、8个(9.52%)和18个(21.43%)基因属于真双子叶植物层(PS9阶段)、蔷薇类植物层(PS10阶段)和锦葵类植物层(PS11阶段)。同时, 结合这些基因的表达特征(图4A), 我们初步推断在植物生殖发育过程中, 相对特异高表达的基因可能伴随着开花植物进化过程中花器官形成和新功能出现而产生。图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6拟南芥系统发生断层学分析结果
(A) 拟南芥基因组进化层图, 包括原核生物、真核生物、绿色植物、胚胎植物、被子植物和蔷薇植物; (B) 候选基因在PS1-PS13层级的分布及比例
Figure 6Phylostratigraphic profiles of Arabidopsis genes
(A) Phylostratigraphic map of Arabidopsis, include prokaryotes, eukaryotes, green plants, embryo plants, angiosperm and rosids; (B) Distribution and proportion of candidate genes in Phylostratum 1-13
2.6 基因重复事件
基因重复事件为基因进化提供了最原始的材料, 也是生物进化的重要推动力。分析基因重复事件能够在宏观和微观层面上对基因进化进行比较全面的诠释(Zhang, 2003; 李鸿健和谭军, 2006)。为进一步了解花药高表达基因簇基因的复制来源, 我们将84个候选基因对比拟南芥全基因组分别进行了基因重复事件分析(图7), 结果表明, 无论是候选基因还是拟南芥全基因组, 邻域重复事件占比均非常少(分别只有1%和3%); 非重复事件占比也相对较少(分别只有5%和11%)。这说明样本基因与拟南芥全基因组基因类似, 主要由基因重复事件产生, 这与目前的基因重复事件创造了生物进化基因原材料的理论相吻合。串联重复、全基因组重复和分散重复3种重复事件分别占样本基因的96%及拟南芥全基因组基因的86%, 是基因重复事件的主要来源。其中分散重复事件分别占24%和27%, 在两者中占比相似。候选基因和拟南芥基因组基因主要的差异来自串联重复和全基因组重复, 候选基因中串联重复事件占较大比例, 而在拟南芥全基因组中全基因组重复占较大比例。串联重复分别占候选基因和拟南芥全基因组的48%和22%; 全基因组重复分别占候选基因和拟南芥全基因组的19%和40% (图7)。据此我们推断候选基因来自具有基因簇特征的基因, 这导致候选基因和拟南芥全基因组在基因重复事件上的占比不同。其中具有基因簇形式的候选基因其形成主要来自串联重复事件, 而非全基因组重复事件。图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7拟南芥基因重复事件
(A) 候选基因重复事件; (B) 拟南芥基因组中基因重复事件。Proximal: 邻域重复事件; Dispersed: 分散重复事件; Singleton: 非重复事件; Tandem: 串联重复事件; WGD or segmental: 全基因组或片段重复事件
Figure 7Gene duplication events in Arabidopsis
(A) Gene duplication events of candidate genes; (B) Gene duplication events in Arabidopsis genome. Proximal: Proximal duplication event; Dispersed: Dispersed duplication event; Singleton: No duplication event; Tandem: Tandem duplication event; WGD or segmental: Whole genome duplication or segmental duplication event
现有研究表明, 第3次全基因组重复发生在2.4-4千万年前, 大约在PS11-PS12之间(Mascarenhas, 1990); 而第2次全基因组重复很可能发生在单子叶植物和双子叶植物分化之后, 大约在PS8-PS9之间; 第1次全基因组重复则由于数据的稀疏性无法准确估计, 只能大致确定为被子植物出现之前, 即大约PS7- PS8之前(Mascarenhas, 1990)。为在宏观层面研究基因重复事件和种系进化之间的关系, 我们对基因组进化层和基因重复事件的关系进行了分析。结果表明, 全基因组重复事件和串联重复事件发生时间集中在PS9- PS11 (表2)。本研究结果与目前相关理论的基础基本一致, 说明我们数据分析结果合理。
Table 2
表2
表2拟南芥基因进化层与基因重复事件的关系
Table 2
| Phylostratum | WGD | Tandem | Proximal | Dispersed | Singleton |
|---|---|---|---|---|---|
| PS13 | 0 | 0 | 0 | 0 | 0 |
| PS12 | 0 | 0 | 0 | 0 | 0 |
| PS11 | 4 (21.05%) | 13 (32.50%) | 0 | 1 (5.00%) | 0 |
| PS10 | 4 (21.05%) | 2 (5.00%) | 0 | 1 (5.00%) | 1 (25.00%) |
| PS9 | 7 (36.84%) | 15 (37.50%) | 0 | 11 (55.00%) | 2 (50.00%) |
| PS8 | 0 | 1 (2.50%) | 0 | 0 | 0 |
| PS7 | 1 (5.26%) | 0 | 0 | 1 (5.00%) | 0 |
| PS6 | 0 | 0 | 0 | 0 | 0 |
| PS5 | 0 | 0 | 0 | 0 | 0 |
| PS4 | 0 | 0 | 0 | 0 | 0 |
| PS3 | 0 | 0 | 0 | 0 | 0 |
| PS2 | 3 (15.79%) | 7 (17.50%) | 0 | 6 (30.00%) | 0 |
| PS1 | 0 | 2 (5.00%) | 1 (100%) | 0 | 1 (25.00%) |
新窗口打开|下载CSV
为进一步研究候选基因内部的重复关系, 我们对其进行全基因组重复和串联重复事件分析(图8)。结果表明, 有串联重复关系的基因相互毗邻, 这是形成基因簇的天然条件之一。没有串联重复关系的基因有一部分由其它类型的基因重复事件产生, 也有一部分由其它候选基因的串联重复事件产生, 说明串联重复事件并不是严格毗邻的。同时, 为研究候选基因内部全基因组重复事件的联系, 我们对候选基因进行了共线性分析, 结果表明没有任何1对基因由全基因组重复事件产生。
图8
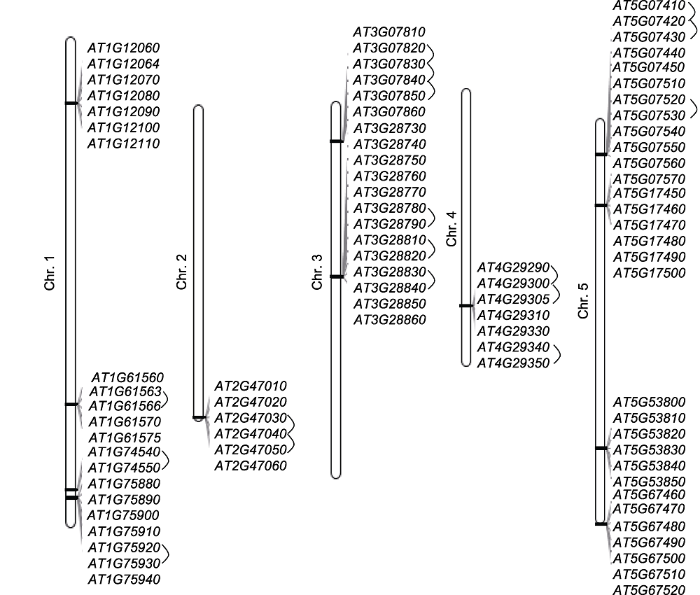 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8拟南芥花药发育特异表达候选基因基因簇分布及内部串联重复关系
Chr.1-5代表1-5号染色体。黑色连线代表存在串联重复关系。
Figure 8Distribution of gene clusters and tandem duplication relationships of anther-specific expressed candidate genes in Arabidopsis anther development
Chr.1-5 represents 1-5 chromosome. Black links represent they have tandem duplication relationships.
2.7 GO富集效应
为了探明候选基因的生物学功能, 我们进一步对候选基因进行GO富集分析。84个候选基因在生物过程(biological process)和细胞组分(cellular components)上有明显的功能富集(图9A, B)。结果表明, 图9A显著性位列前10的项目中, 植物生殖发育过程出现次数较多, 包括有性生殖过程(sexual reproduction)、多器官生殖过程(multi-organism reproductive process)、生殖过程(reproductive process)和生殖(reproduction)过程, 说明候选基因在生殖发育相关的生物过程中有强烈的富集效应。同时, 这些基因在脂类物质的储存和地位上也有较高的富集倍数, 包括脂质储存(lipid storage)和脂质定位(lipid localization), 说明脂类物质的相关代谢路径可能在生殖发育过程中具有非常重要的作用。图9B显示, 候选基因在花粉鞘和脂类物质, 包括单层包裹的脂质存储体(monolayer-surrounded lipid storage body)和脂质液滴(lipid droplet)中有高度的富集。花粉鞘的主要成分之一是脂类物质, 有促进花粉传播、帮助花粉粘附于柱头和防止紫外线伤害等重要作用(Carbon et al., 2009)。图9C显示了细胞组成项目中, 候选基因与拟南芥全基因组中拥有相关功能的基因的比值。值得注意的是, 花粉鞘的比值为100%, 说明拟南芥数据库中所有与花粉鞘形成相关的基因都在候选基因中。我们进一步推断, 筛选出的基因确实与拟南芥花药发育具有非常高的相关性。图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9拟南芥花药发育特异表达候选基因GO (gene ontology)富集分析
(A) GO生物过程富集分析; (B) GO细胞组分富集分析(图(A)和(B)纵坐标为GO项目注释, 括号中是富集倍数, 横坐标为P值负对数); (C) GO细胞组成后续分析(纵坐标为候选基因中存在GO项目的基因数目和拟南芥全基因组中存在GO项目的基因数目比值, 横坐标为GO项目的注释)
Figure 9Gene ontology (GO) enrichment analysis of anther-specific expressed candidate genes during Arabidopsis anther development
(A) GO biological process enrichment analysis; (B) GO cellular component enrichment analysis (Figures (A) and (B) y-axis represents annotations of GO terms, number in parenthesis represents GO enrichment fold and x-axis represents minus logarithm of P value); (C) Subsequent analysis of GO cellular components (y-axis represents number of genes in a GO term in candidate genes to number of genes in a GO term in Arabidopsis genome and x-axis represents annotation of GO terms)
2.8 讨论
花药发育和花粉形成是开花植物在进化中产生的最重要的生物学过程, 花粉形成(尤其是花粉外壁的建成和营养供给)主要依赖于花药绒毡层细胞的形成和功能。本研究通过对花药绒毡层特异性高表达基因的筛选(图1), 获得了拟南芥花药发育阶段的特异性高表达且具有基因簇特征的24个基因, 经扩大筛选后获得84个基因(属于13个基因簇)。这些候选基因的筛选和获得, 表明植物生殖发育过程中存在以基因簇形式的花药特异高表达基因, 它们可能在植物生殖发育过程中起至关重要的作用, 可为进一步研究这些基因的遗传学功能提供依据。我们首先通过生物信息学分析候选基因的特性, 结果表明这些候选基因在染色体分布、分子量、等电点、细胞位置和跨膜域数量上的一些特征与其它基因均无明显差异(表1)。进一步分析候选基因的基因结构和转录方向(图3), 结果表明同一基因簇内部同一家族基因的基因结构往往相似性较高, 且转录方向相同, 基因簇内部来自不同家族的异源基因的基因结构和转录方向差异较大。这暗示同源家族基因的转录调控相对比较一致, 但异源基因簇基因的转录水平的调控相对复杂。结合候选基因的表达聚类以及共表达网络分析(图4), 证明37个在植物雄性生殖发育中相对特异高表达的基因可能对开花植物的功能出现或者维持是必需的, 它们之间均存在共同的共表达网络, 说明这些基因至少在转录水平上存在一定的关联, 从而为后续研究它们之间的调控关系和功能互作提供一定的依据。
不同物种的形态和新功能出现可能是新基因产生和长期进化的结果, 基因重复在基因的新功能进化和物种的多样性中发挥关键作用(Cui et al., 2015)。因此, 我们利用基因组进化层分析, 推测候选基因出现的阶段。结果表明, 候选基因起源主要集中在PS1- PS2和PS9-PS11 (图6)。PS1-PS2阶段的基因在植物形成最初阶段就已经存在, 只是进一步在植物的生殖发育阶段, 可能分化出新的功能参与花药的发育过程。PS9-PS11是开花植物出现的阶段, 此阶段出现的基因伴随着开花植物的出现和进化, 即伴随开花植物进化过程中花器官形成和新功能出现而产生。
基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(Zhang, 2003; 李鸿健和谭军, 2006; 孙红正和葛颂, 2010)。已有研究表明, 拟南芥在过去的2.5亿年中共发生3次全基因组重复事件(Kondrashov et al., 2002), 其中第2次和第3次明确发生在开花植物出现之后, 分别在PS8-PS9和PS11-PS12阶段(Koch et al., 2000; De Bodt et al., 2005)。虽然全基因组重复后产生的基因会以较高的频率丢失, 但是依旧在发育、调控和信号转导等方面发挥关键性的进化作用(Mascarenhas, 1990)。本研究候选基因来自对基因簇特征的基因筛选, 这样导致候选基因和拟南芥全基因组在基因重复事件上的占比不同(图7)。在候选基因中, 我们推测串联重复事件是基因簇形成的主要推动力, 串联重复的候选基因占较大比例, 且能够产生毗邻的基因, 这和基因簇的形式非常吻合。在候选基因内部串联重复和共线性分析中, 有不少基因簇的形成直接来自某一基因的串联重复(图8), 但是并没有发现这些基因之间有全基因组重复事件发生的联系。
最后, 我们利用GO富集分析预测了候选基因的生物功能。结果表明, 候选基因在生殖发育相关功能以及脂类物质的生物代谢和组成有所富集, 而花粉鞘的组成与脂类物质有密切关系(图9)。这些基因的预测功能都与花药绒毡层在生殖发育过程中作为小孢子(花粉)发育的重要物质基础密切相关, 尤其对花粉外壁的建成过程提供脂类物质必不可少。因此, 我们推测这些在花药中特异性表达的基因簇基因可能在花药发育和花粉形成过程中有重要作用, 但尚需进一步的遗传学实验验证。
综上所述, 我们确认在花药发育过程中存在基因簇形式, 证实这些基因簇基因主要来源于串联重复事件, 并在进化上随着开花植物的出现产生了特定的功能。本研究初步解析了拟南芥花药发育中基因簇基因的基本特征、生物学功能和基因进化机制, 为深入研究基因簇基因的遗传学功能奠定了基础。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
In: Fox CW, Wolf JB, eds. Evolutionary Genetics: Concepts and Case Studies.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
基因倍增研究进展
2
2006
... 基因重复事件为基因进化提供了最原始的材料, 也是生物进化的重要推动力.分析基因重复事件能够在宏观和微观层面上对基因进化进行比较全面的诠释(
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
植物次生代谢基因簇研究进展
1
2017
... 植物基因组中序列高度相似的同源基因成簇的现象非常普遍, 众多植物基因簇中有许多已确定的基因编码参与次生代谢途径的酶.例如, 番茄(Lycopersicon esculentum)中存在合成单萜类化合物的基因簇(
重复基因的进化——回顾与进展
2
2010
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
查尔酮合酶基因重复-歧化的遗传学效应
1
2009
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
AmiGO: online access to ontology and annotation data
2
2009
... 将候选基因提交到PATHER数据库中, 选择背景数据库为拟南芥, 对相关拟南芥基因进行分子功能(molecular function)、细胞结构(cellular components)和生物过程(biological process) 3方面的注释来分析基因功能.P值用于显著性判断, P<0.05为显著(
... 为了探明候选基因的生物学功能, 我们进一步对候选基因进行GO富集分析.84个候选基因在生物过程(biological process)和细胞组分(cellular components)上有明显的功能富集(
TBtools, a toolkit for biologists integrating various HTS-data handling tools with a user-friendly interface
1
2018
... 利用TAIR数据库中拟南芥基因物理位置的相关信息, 将相关数据下载后, 利用TB-tools工具进行可视化处理(
Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters
1
2014
... 基因簇(gene cluster)是指在染色体上成簇出现并协同转录的一类同源或非同源基因, 在细菌基因组中广泛存在(
Young genes out of the male: an insight from evolutionary age analysis of the pollen transcriptome
2
2015
... 基因组进化层可以揭示宏观进化中重要的适应性事件, 并对基因功能做出一定的解释, 是从宏观层面上分析基因起源和进化的重要手段(
... 不同物种的形态和新功能出现可能是新基因产生和长期进化的结果, 基因重复在基因的新功能进化和物种的多样性中发挥关键作用(
Genome duplication and the origin of angiosperms
1
2005
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
The cluster of penicillin biosynthetic genes. Identification and characterization of the pcbAB gene encoding the alpha-aminoadipyl-cysteinyl- valine synthetase and linkage to the pcbC and penDE genes
1
1990
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
A phylostratigraphy approach to uncover the genomic history of major adaptations in metazoan lineages
2
2007
... 新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环.如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(
... 基因组进化层可以揭示宏观进化中重要的适应性事件, 并对基因功能做出一定的解释, 是从宏观层面上分析基因起源和进化的重要手段(
A phylogenetically based transcriptome age index mirrors ontogenetic divergence patterns
2
2010
... 新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环.如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(
... 基因组进化层可以揭示宏观进化中重要的适应性事件, 并对基因功能做出一定的解释, 是从宏观层面上分析基因起源和进化的重要手段(
The tomato terpene synthase gene family
1
2011
... 植物基因组中序列高度相似的同源基因成簇的现象非常普遍, 众多植物基因簇中有许多已确定的基因编码参与次生代谢途径的酶.例如, 番茄(Lycopersicon esculentum)中存在合成单萜类化合物的基因簇(
Preservation of duplicate genes by complementary, degenerative mutations
1
1999
... 新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环.如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(
Characterization of the syringomycin synthetase gene cluster. A link between prokaryotic and eukaryotic peptide synthetases
1
1998
... 基因簇(gene cluster)是指在染色体上成簇出现并协同转录的一类同源或非同源基因, 在细菌基因组中广泛存在(
Biosynthesis of antinutritional alkaloids in solanaceous crops is mediated by clustered genes
1
2013
... 植物基因组中序列高度相似的同源基因成簇的现象非常普遍, 众多植物基因簇中有许多已确定的基因编码参与次生代谢途径的酶.例如, 番茄(Lycopersicon esculentum)中存在合成单萜类化合物的基因簇(
Evolution of the major histocompatibility complex
1
1986
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
Comparative evolutionary analysis of chalcone synthase and alcohol dehydrogenase loci in Arabidopsis, Arabis, and related genera (Brassicaceae)
1
2000
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
Selection in the evolution of gene duplications
1
2002
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets
1
2016
... 从TAIR数据库中下载所有候选基因的DNA序列, 用Cluster W程序进行序列比对.使用MEGA7软件通过极大似然法分别对所有候选基因以及基因簇内部基因构建进化树, 使用bootstrap=1 000进行验证, 其余参数均为默认值(
Gene regulatory network for tapetum development in Arabidopsis thaliana
2
2017
... 开花植物雄性生殖发育过程中, 绒毡层作为花药最内层细胞, 为雄配子体(花粉)的形成发育提供所需的营养物质及细胞环境.
... 为获得拟南芥花药发育过程中特异高表达及具有基因簇特征的候选基因, 我们设计并确定筛选过程.首先, 在已发表文献的转录组数据基础上(
Pollen development in Annona cherimola Mill.(Annonaceae). Implications for the evolution of aggregated pollen
1
2009
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
The Aspergillus nidulans npeA locus consists of three contiguous genes required for penicillin biosynthesis
1
1990
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
Gene activity during pollen development
4
1990
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
... 现有研究表明, 第3次全基因组重复发生在2.4-4千万年前, 大约在PS11-PS12之间(
... ); 而第2次全基因组重复很可能发生在单子叶植物和双子叶植物分化之后, 大约在PS8-PS9之间; 第1次全基因组重复则由于数据的稀疏性无法准确估计, 只能大致确定为被子植物出现之前, 即大约PS7- PS8之前(
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
PANTHER version 11: expanded annotation data from gene ontology and reactome pathways, and data analysis tool enhancements
1
2017
... 将候选基因提交到PATHER数据库中, 选择背景数据库为拟南芥, 对相关拟南芥基因进行分子功能(molecular function)、细胞结构(cellular components)和生物过程(biological process) 3方面的注释来分析基因功能.P值用于显著性判断, P<0.05为显著(
A prokaryotic gene cluster involved in synthesis of lysine through the amino adipate pathway: a key to the evolution of amino acid biosynthesis
1
1999
... 基因簇(gene cluster)是指在染色体上成簇出现并协同转录的一类同源或非同源基因, 在细菌基因组中广泛存在(
Splitting pairs: the diverging fates of duplicated genes
1
2002
... 新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环.如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(
A gene cluster for secondary metabolism in oat: implications for the evolution of metabolic diversity in plants
1
2004
... 植物基因组中序列高度相似的同源基因成簇的现象非常普遍, 众多植物基因簇中有许多已确定的基因编码参与次生代谢途径的酶.例如, 番茄(Lycopersicon esculentum)中存在合成单萜类化合物的基因簇(
Fungal gene cluster diversity and evolution
1
2017
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
New genes, new functions: gene family evolution and phylogenetics
1
2006
... 新基因的形成是一个不连续的过程, 最初以非常快的速度进化, 直到成为某个代谢路径中的一环.如果这些基因之后也在某个代谢路径中发挥作用, 那么就可以通过它在现有物种中的功能以及系统发生起源的估计来综合推断它的进化起源(
MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity
1
2012
... 将拟南芥全基因组序列通过本地BLAST进行比对, 使用MCScan X软件进行基因重复事件、串联重复以及共线性分析.相关数据来源于TAIR, 所有参数均为默认值(
From Arabidopsis to rice: pathways in pollen development
1
2009
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
Streptomyces in nature and medicine: the antibiotic makers
1
2008
... 基因簇(gene cluster)是指在染色体上成簇出现并协同转录的一类同源或非同源基因, 在细菌基因组中广泛存在(
TAPETUM DETERMINANT 1 is required for cell specialization in the Arabidopsis anther
1
2003
... 植物花药发育过程是产生雄配子体(花粉粒)的重要阶段, 在植物世代交替的生活史中扮演了重要角色(
Evolution by gene duplication: an update
2
2003
... 基因重复事件为基因进化提供了最原始的材料, 也是生物进化的重要推动力.分析基因重复事件能够在宏观和微观层面上对基因进化进行比较全面的诠释(
... 基因重复事件为进化提供原始材料, 是进化过程中非常重要的过程(
Identification of caerulomycin A gene cluster implicates a tailoring amidohydrolase
1
2012
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
Flavoenzyme CrmK-mediated substrate recycling in caerulomycin biosynthesis
2
2016
... 除细菌外, 其它真核生物(如真菌、昆虫及脊椎动物)的基因组中也有基因簇(
... ,
备案号: 京ICP备16067583号-21
版权所有 © 2021 《植物学报》编辑部
地址:北京香山南辛村20号 邮编:100093
电话:010-62836135 010-62836131 E-mail:cbb@ibcas.ac.cn
本系统由北京玛格泰克科技发展有限公司设计开发
