Bioinformatics Analysis and Chromosome Location of Nuclear Integrants of Plastid DNA in Asparagus officinalis
Guangqian Cheng1,?, Keli Jia1,2,?, Na Li1, Chuanliang Deng1, Shufen Li1, Wujun Gao1,*通讯作者:
收稿日期:2018-05-11接受日期:2018-08-6网络出版日期:2019-07-01
| 基金资助: |
Corresponding authors:
Received:2018-05-11Accepted:2018-08-6Online:2019-07-01

摘要
关键词:
Abstract
Keywords:
PDF (1351KB)摘要页面多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
程广前, 贾克利, 李娜, 邓传良, 李书粉, 高武军. 石刁柏核质体DNA的生物信息学分析及染色体定位. 植物学报, 2019, 54(3): 328-334 doi:10.11983/CBB18117
Cheng Guangqian, Jia Keli, Li Na, Deng Chuanliang, Li Shufen, Gao Wujun.
植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(Michalovova et al., 2013)。在基因组演化过程中, 细胞器基因组可以向核基因组中转移, 其中由叶绿体DNA转移到核基因组中的序列称为核质体DNA (nuclear integrants of plastid DNAs, NUPTs), 而由线粒体DNA转移到核基因组中的序列称为核线粒体DNA (nuclear integrants of mitochondria DNAs, NUMTs) (Richly and Leister, 2004; Timmis et al., 2004; Kleine et al., 2009; Yoshida et al., 2014)。
目前, 已在多种植物的核基因组中发现了NUPTs (Kejnovsky et al., 2006; Guo et al., 2008; Steflova et al., 2014)。NUPTs是组成植物基因组的一种特殊类型的重复序列, 包括NUPTs和转座子在内的重复序列的插入会引起基因组不稳定, 如引起染色体结构重排、体积膨胀和异染色质化, 在基因组和染色体进化过程中具有重要作用(高东迎等, 2007; Feschotte and Pritham, 2007; Li et al., 2016)。细胞学研究表明, NUPTs多集中在染色体异染色质区域, 如着丝粒及其侧翼等。在水稻(Oryza sativa)中, 大片段的NUPTs主要集中在着丝粒及侧翼区, 随着演化的进行NUPTs长度逐渐衰减, 并且伴随着拷贝数的增加(Guo et al., 2008)。有研究表明, NUPTs在雌雄异株植物的性染色体上累积较多。Kejnovsky等(2006)在白麦瓶草(Silene latifolia) Y染色体上发现了NUPTs, 这些NUPTs也多位于Y染色体上的着丝粒及侧翼区, 且NUPTs的累积可能是白麦瓶草Y染色体体积膨胀的主要原因之一。在番木瓜(Carica papaya)中, Y染色体的雄性特异区上累积的NUPTs比X染色体高12倍, 比其它常染色体高4倍, 这种差异是由于NUPTs的复制所致(VanBuren and Ming, 2013)。上述研究表明, NUPTs的插入与植物性染色体的演化密切相关。
石刁柏(Asparagus officinalis)隶属百合科天门冬属, 是重要的雌雄异株植物。其基因组大小约为1.3 Gb, 染色体组成为2n=2x=20, 具有X和Y同型性染色体(L?ptien, 1979), 是研究性染色体起源和早期演化的理想材料之一。本实验室利用基因组消减杂交和PCR获得了一些石刁柏性别差异NUPTs (李书粉等, 2016), 但石刁柏全基因组NUPTs的分布规律和染色体分布模式尚未见报道。本研究对石刁柏基因组的NUPTs进行注释和分析, 并采用荧光原位杂交对2个序列进行染色体定位, 旨在为进一步分析NUPTs在石刁柏染色体上的分布以及在性染色体的起源和演化中的作用提供证据。
1 材料与方法
1.1 石刁柏基因组NUPT的生物信息学分析
1.1.1 数据来源本研究所用的生物信息学分析数据包括石刁柏(Asparagus officinalis L.)叶绿体基因组(下载自
1.1.2 基因组NUPTs的注释
采用本地Blast (Altschul et al., 1990)软件, 以石刁柏叶绿体基因组作为query序列, 对石刁柏核基因组进行Blast分析, e value设置为e-5, 一致性≥80%。在结果中去除50 bp以下的序列, 即得到注释为石刁柏基因组NUPTs的序列。根据Blast结果, 计算不同染色体上不同位置NUPT的数量, 滑动窗口设为5 M, 采用R语言作图。
1.1.3 基因组NUPTs在叶绿体基因组中的来源分析
NUPTs起源的叶绿体基因组区域通过将每个识别的NUPT分配(mapping)到叶绿体基因组的序列来确定。由于叶绿体基因组的2个IR (inverted repeat region)区序列互为反向互补序列, 故如果NUPT被mapping到IR区, 只计数一半。根据Blast结果, 计算叶绿体基因组不同位置的NUPT数量, 滑动窗口设为500 bp。
1.2 石刁柏叶绿体基因组IR区2个序列的染色体定位
1.2.1 基因组DNA的提取取石刁柏的幼嫩叶片, 采用传统的CTAB法(Doyle and Doyle, 1987)提取基因组DNA。由于所取的叶片材料含有大量的叶绿体, 因此所提取的基因组DNA中除了核基因组外, 也包含叶绿体基因组DNA (李巧丽等, 2018)。
1.2.2 叶绿体DNA序列扩增及回收纯化
根据石刁柏叶绿体IR区序列, 随机选取2个序列区域并设计引物, 引物序列分别为AocpIR1F: 5'-AGATAGGAAGGGCTGTTGC-3', AocpIR1R: 5'-AGCAGGGTCAGGAACAACG-3'; AocpIR2F: 5'-GATCTCAATTCCAGCTACGG-3', AocpIR2R: 5'-GTAGGTATCTGGTTCACTGC-3'。AocpIR1和AocpIR2扩增序列长度分别为4 118 bp和3 614 bp。利用这2对特异引物进行PCR扩增,反应体系如下: 2.5 μL 10×PCR buffer, 正反向引物各1 μL, 1.3 μL dNTP, 0.2 μL Taq DNA聚合酶, 1 μL石刁柏模板DNA, 用去离子水补充至25 μL。PCR反应条件如下: 95°C2分钟; 94°C30秒, 57°C50秒, 72°C4分钟, 35个循环; 72°C10分钟。
将扩增得到的PCR产物用0.8%琼脂糖凝胶进行电泳, 并用EasyPure Quick Gel Extraction Kit试剂盒进行切胶回收, 再利用EasyPure PCR Purification Kit试剂盒将切胶产物回收纯化(纯化后浓度不低于200 ng·μL-1)后保存备用。
1.2.3 探针标记
为了辅助石刁柏染色体核型分析, 采用玉米(Zea mays) 45S rDNA (质粒由中国科学院遗传与发育生物学研究所胡赞民实验室馈赠)和NUPT标记探针进行双色荧光原位杂交。45S rDNA质粒和NUPT序列回收纯化产物采用缺口平移法标记探针。NUPT片段标记红色荧光素, 45S rDNA标记绿色荧光素。探针标记体系为: 2 μg DNA, 2 μL 10×Nick translation buffer, 0.5 μL荧光素标记的dNTPs, 2 μL未标记的dNTPs, 5 μL 10 U·μL-1 DNA polymerase I, 0.5 μL 100 mU·μL-1 DNase I, 用去离子水补充至20 μL。依次加入上述试剂后充分离心混匀, 置于15°C金属浴中孵育2小时, 取出置于4°C冰箱5小时, 之后加入160 μL 5×TAE+140 ng·μL-1鲑精DNA和500 μL 90%乙醇- 10%乙酸钠, 充分混匀后置于-20°C过夜。隔日放入预冷的离心机中, 4°C 16 000 ×g离心30分钟, 弃上清; 加入1 000 μL 70%乙醇并于4°C 16 000 ×g离心2分钟, 弃上清; 加入1 000 μL无水乙醇并于4°C 16 000 ×g离心2分钟, 室温放置30分钟至1小时, 直至无水乙醇完全挥发; 加20 μL 2×SSC+1×TE充分溶解, -20°C保存备用。
1.2.4 中期染色体玻片标本制备
于25°C培养石刁柏种子, 待种子根尖长至长约1.0-1.5 cm时, 剪取根尖放置在1.01 MPa的含N2O密闭容器内处理2小时, 后用90%的冰乙酸固定10分钟, 用70%乙醇清洗4遍, 将根尖保存至70%乙醇中, 置于-20°C备用。
取出根尖, 用1×柠檬酸缓冲液清洗根尖10分钟, 取根尖分生组织置于纤维素果胶酶液中, 37°C孵育2小时后用1×TE处理2遍, 然后用无水乙醇清洗至无TE残留, 用金属针将其捣碎制成悬浮液, 低速离心40-50秒, 弃上清。加入35 μL冰乙酸, 混匀, 吸取5 μL滴片并观察, 将染色体形态好的片子保存备用。
1.2.5 荧光原位杂交
将待杂交切片置于紫外交联仪中按每平方米0.125 J的能量进行交联。取3.5 μL 2×SSC+1×TE buffer、2 μL已标记的叶绿体DNA红色探针、0.5 μL已标记的45S rDNA绿色探针, 充分混匀后离心。
将载玻片放在冰上, 在有细胞的地方滴加6 μL杂交液, 盖上塑料盖玻片, 置于沸水中5分钟, 然后放入预热的密闭湿盒中55°C杂交过夜。将载玻片从湿盒中取出, 放入2×SSC中使盖玻片滑落, 放入55°C水浴锅中15-20分钟, 之后用吸水纸把片子背面擦干, 滴上1滴含DAPI的抗淬灭剂, 盖上24 mm×50 mm的盖玻片, 使用BX63荧光显微镜进行镜检, Andor CCD拍照, 采用Adobe PhotoShop CS6软件对图片进行亮度、对比度的微调和剪切处理。
2 结果与讨论
2.1 石刁柏基因组NUPTs在染色体上的长度分布模式
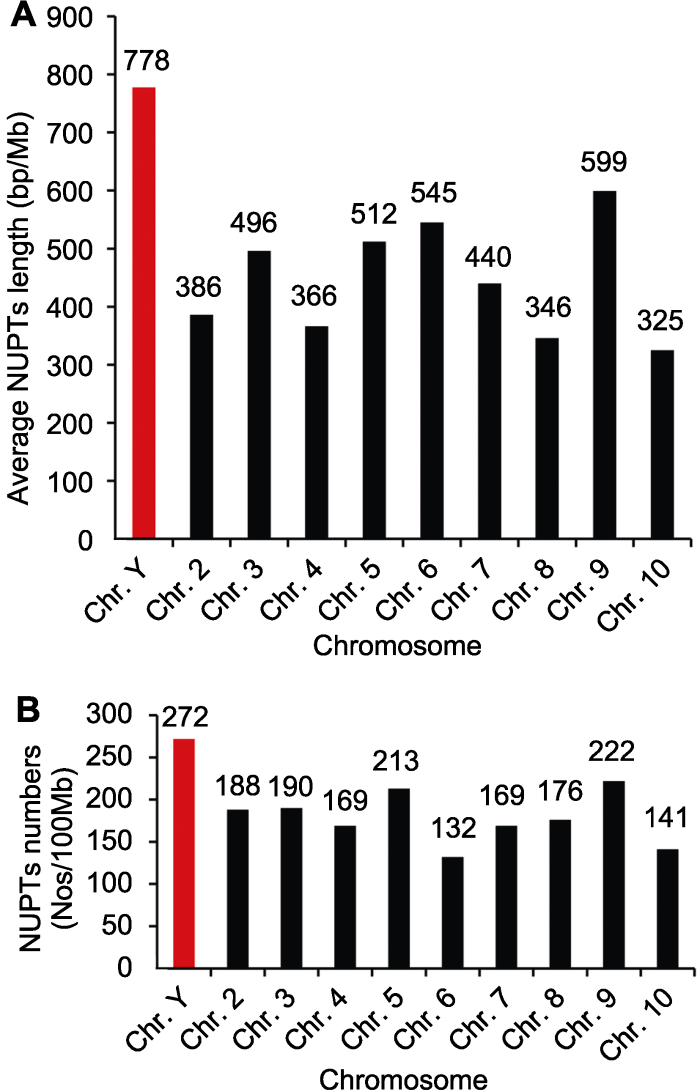
为了探明石刁柏基因组NUPTs的分布特征, 我们利用已发表的石刁柏基因组测序数据进行基因组NUPTs注释。结果表明, 目前发表的石刁柏核基因组中共有2 239个NUPTs序列的插入, 总长度为565 970 bp, 占核基因组的0.047%。我们进一步对NUPTs在染色体上的分布进行分析(图1), 发现所有染色体上均有NUPTs分布, 其中1号染色体(Y染色体)的NUPTs插入密度为272个/100 Mb, 平均插入长度为778 bp/Mb, 而其余常染色体NUPTs平均插入密度和平均插入长度分别为178个/100 Mb和446 bp/Mb, 表明Y染色体上NUPTs插入密度和长度均明显高于常染色体。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1石刁柏不同染色体核质体DNA (NUPTs)长度(A)和数目(B)
Figure 1The length (A) and number (B) of nuclear integrants of plastid DNAs (NUPTs) in different chromosomes of Asparagus officinalis
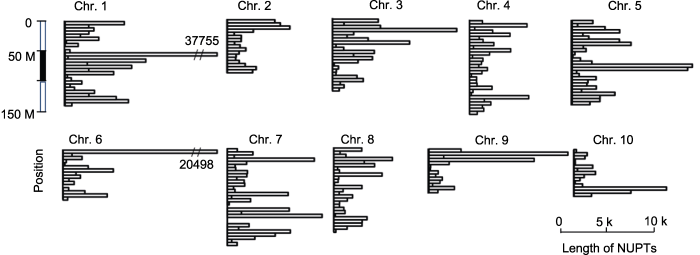
我们对NUPTs在不同染色体上的分布位置进行分析, 结果表明, NUPTs在染色体上的分布不是随机的, 而是存在一些插入热点区域。如在1号染色体的近中部和6号染色体的端部均包含1个NUPTs富集的区域, 在5 M的序列内分别有37 755 bp和20 498 bp的NUPTs序列(图2)。
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2石刁柏基因组核质体DNA (NUPTs)在染色体上的分布模式
Figure 2The chromosome distribution pattern of nuclear integrants of plastid DNAs (NUPTs) of Asparagus officinalis genome
2.2 石刁柏核基因组NUPTs来源分析
为了解叶绿体基因组序列向核基因组的转移特点, 我们对石刁柏基因组中的NUPTs数量及来源进行分析, 结果表明, 在2 239个石刁柏核基因组NUPTs中, 来源于IR区、大单拷贝区(large single copy region, LSC)和小单拷贝区(small single copy region, SSC)的NUPTs数量分别为726、1 227和286个。由于石刁柏叶绿体基因组单个IR、LSC和SSC区的长度分别为26 471 bp、83 821 bp和17 901 bp, 因此其密度分别为27.9个/kb、14.6个/kb和15.9个/kb (表1)。此外, IR、LSC和SSC区转移片段的平均长度分别为241 bp、258 bp和246 bp, 表明三者转移的平均长度没有明显差异(表1)。上述研究结果表明, 石刁柏叶绿体基因组向核基因组的转移不是随机的, 来源于IR区的NUPTs密度明显高于其它2个区域, 说明叶绿体基因组中的IR区序列更容易向核基因进行水平转移。Table 1
表1
表1石刁柏核基因组核质体DNA (NUPTs)的叶绿体基因组来源分析
Table 1
| IR | LSC | SSC | |
|---|---|---|---|
| Total sequence length (bp) | 26471 | 83821 | 17904 |
| Number of NUPTs | 726 | 1227 | 286 |
| Average density (No./kb) | 27.9 | 14.6 | 15.9 |
| Average length of NUPTs (bp) | 241 | 258 | 246 |
| The length of NUPTs (bp) | 175215 | 316626 | 70394 |
新窗口打开|下载CSV
2.3 2个IR区序列的获得
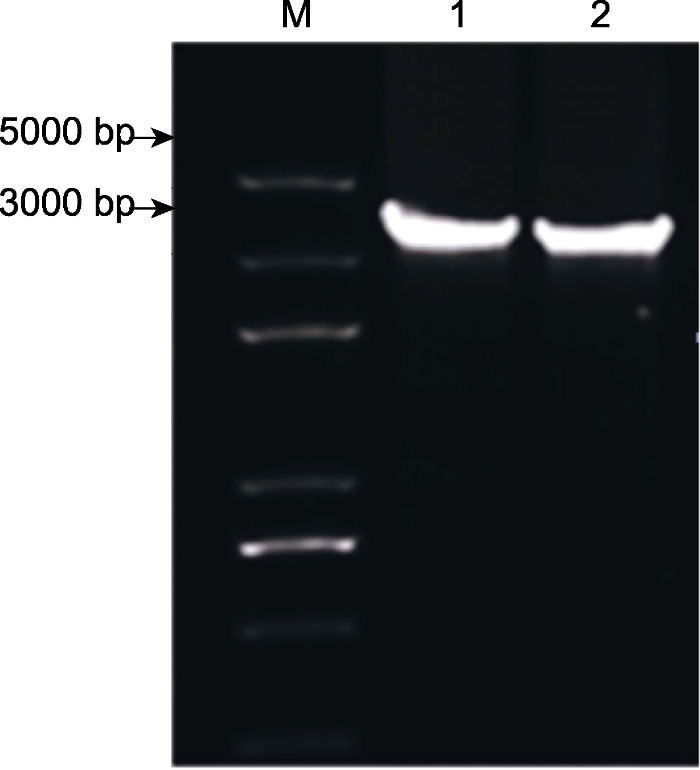
根据IR区序列设计引物, 以石刁柏基因组(包含叶绿体DNA)为模板进行PCR扩增。如图3所示, 均扩增得到约3-5 kb之间的单一条带, 大小与预期一致。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3石刁柏2个叶绿体反向重复区(IR)序列的PCR扩增
1, 2分别代表AocplR1和AocplR2的扩增结果。
Figure 3PCR amplification of inverted repeat region (IR) two sequences of region in Asparagus officinalis
1, 2 respresents the amplification results of AocplR1 and AocplR2, respectively.
2.4 2个IR区序列的染色体定位
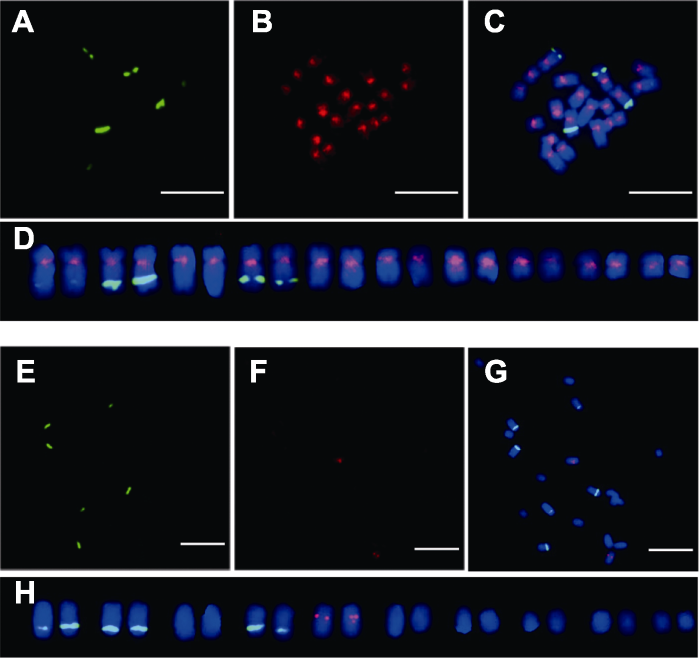
将扩增获得的2个IR序列进行回收纯化后标记探针, 与45S rDNA同时进行双色荧光原位杂交。结果表明, 45S rDNA共有6个信号, 分布在3对同源染色体上(图4A, C, E)。AocpIR1在所有染色体上都有分布, 信号主要分布在染色体着丝粒位置, 但在着丝粒侧翼位置也有微弱信号(图4B)。AocpIR2特异定位在2条染色体的着丝粒部位(图4F, G), 核型分析表明这2条染色体为石刁柏核型的第5对染色体(图4D, H), 也正是石刁柏的性染色体(L?ptien, 1979)。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4AocpIR1和AocpIR2在石刁柏有丝分裂中期染色体上的定位
(A), (E) 45S rDNA杂交信号图; (B), (F) AocpIR1和AocpIR2杂交信号图; (C), (G) 合成图; (D), (H) 核型图。Bars=10 μm
Figure 4Mitosis metaphase chromosome locations of AocpIR1 and AocpIR2 of Asparagus officinalis
(A), (E) FISH result of 45S rDNA; (B), (F) FISH result of AocpIR1 and AocpIR2; (C), (G) Merged picture; (D), (H) Karyotype analysis. Bars=10 μm
2.5 讨论
本研究表明, 叶绿体DNA序列不同区域向核基因组转移的频率存在差异, 石刁柏叶绿体基因组中IR区序列更易向核基因组中进行水平转移, 这与在白麦瓶草(Steflova et al., 2014)和水稻(Matsuo et al., 2005)中的研究结果一致。这种转移偏向性与IR区序列的独特特征关系密切。IR区为反向重复序列, 在叶绿体基因组中以相反的方向重复存在。这种结构可能有助于核基因组的插入重组。Wang等(2008)研究表明, 被子植物的IR区包含1个polyA区域, 这个区域可能是重组的热点。通过这种机制, 其中1个IR区域利用第2个IR区中同源序列的修复/重组过程延伸到相邻的SC区中(Wang et al., 2008)。因此, 推测类似的基因重组过程可促进IR区从叶绿体基因组中优先整合到核基因组中。也有观点认为大多数细胞器DNA通过非末端连接修复双链断裂机制转移到细胞核中, 但是在其它一些植物, 如酸模(Rumex acetosa)基因组中, IR区比叶绿体基因组中其它位置的序列更难转移形成NUPTs (Steflova et al., 2014)。这也表明核基因中NUPTs的插入机制更为复杂, 仍需深入研究。研究表明, 植物中NUPTs更倾向于插入和累积在着丝粒及其侧翼区等染色体异染色质区域(Michalovova et al., 2013; Steflova et al., 2014)。在黑麦(Secale cereale) B染色体中, NUPTs也在基因含量很低的异染色质区域累积(Martis et al., 2012)。本研究定位的2个IR区序列中, 其中1个定位于所有染色体的着丝粒及其侧翼区, 而另1个特异定位于性染色体的着丝粒区, 表明石刁柏的NUPTs也更倾向于插入到染色体的着丝粒及其侧翼区。这可能是由于这些异染色质区重组频率较低, 而低重组率更有利于重复序列高度一致性的维持。Matsuo等(2005)研究表明, 着丝粒及其侧翼区等异染色质区能更好地接纳大的叶绿体DNA或线粒体DNA序列, 具有更适合形成NUPT/NUMT的基因组环境。但是, 采用序列比对对NUPTs在核基因组位置的分析未显示NUPTs更倾向于插入着丝粒及其周围的特性。原因主要有2个: (1) 基因组组装过程中, 对于着丝粒及侧翼区域更不容易组装出来, 导致这些区域中的NUPT序列没有在基因组序列中体现出来; (2) 本研究只对IR区的2个序列进行FISH定位, 可能IR序列的转移位置相比LSC和SSC具有更高的特异性。后续可以全面地对石刁柏叶绿体基因组的各个区域进行FISH定位, 从而更全面地获得各个区域在染色体上的分布特征及转移特性。
目前普遍认为, 性染色体最初起源于1对常染色体, 在出现原始的性别决定基因之后, 包括性别决定基因在内的性别决定区的重组抑制是性染色体演化过程的关键步骤(Kejnovsky et al., 2009; Li et al., 2016; Abbott et al., 2017)。包括NUPTs在内的重复序列的插入造成异染色质化或倒位是重组抑制形成的重要原因, 而重组抑制区还可以招募更多的NUPTs等重复序列, 使重组抑制区进一步扩展, 最终形成异型的性染色体(Li et al., 2016)。本研究定位的2个序列中, 其中1个位于性染色体的着丝粒部位, 而该区域正是染色体不发生重组或很少发生重组的位置, 表明在雌雄异株植物石刁柏中, NUPTs的插入在其性染色体的演化过程中可能发挥作用。期待今后发现更多的性染色体特异的NUPTs, 并利用高分辨率的DNA纤维原位杂交结合免疫荧光来分析NUPTs插入区域附近染色质的DNA和组蛋白的表观修饰模式特征, 以深入揭示NUPTs在植物性染色体演化过程中所起的作用及其作用机制。
(责任编辑: 朱亚娜)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.3969/j.issn.1674-3466.2007.05.016URL [本文引用: 1]

转座子是植物基因组的重要组成部分,对于研究植物基因组进化等具有重要意义。随着水稻全基因组测序计划的开展和完成,水稻转座子研究取得了极大进展,目前已经在水稻基因组中发现了几乎所有类型的转座子,约占水稻基因组的35%。在正常情况下,大多数水稻转座子不具有转座活性,但是在特定的条件下(如组织培养或辐射等),水稻基因组中沉默的转座子可以被激活,从而可能导致插入突变并影响基因的表达。在水稻中已鉴定出6个有活性的转座子,其中Tos17已被应用到水稻功能基因组研究中。转座子序列的新的分子标记转座子展示(transposon display,TD)现已被开发,并在水稻遗传作图和遗传分化研究中得到应用。
DOI:10.11983/CBB16247URL [本文引用: 1]

鲁桑(Morus multicaulis)是亚洲地区栽培的重要经济作物。以鲁桑品种日本胡橙为实验材料,利用高通量测序技术对鲁桑叶绿体基因组进行测序,获得NCBI登录号(KU355297),并研究鲁桑的叶绿体基因组结构。结合前人对蒙桑(M.mongolica)、印度桑(M.indica)和川桑(M.notabilis)的研究结果,对鲁桑的系统进化关系进行了探讨。研究结果表明:鲁桑叶绿体基因组是一个典型的四部分结构,全长159 154 bp,共注释130个基因,包含85个蛋白质编码基因(18个基因在反向重复区重复)、37个转运RNA(tRNA)基因和8个核糖体RNA(rRNA)基因。生物信息学分析表明,在鲁桑中共搜索到82个SSR位点,单核苷酸、二核苷酸、三核苷酸、四核苷酸和五核苷酸重复基序个数分别为63、7、2、9和1个,并没有发现六核苷酸;其中单核苷酸重复在鲁桑的叶绿体基因组SSR中占76.8%。采用MEGA 6.0软件,通过最大似然法和近邻结合法对包括4个桑属物种在内的15个物种的叶绿体基因组序列进行聚类分析,2种方法得到的聚类结果均为鲁桑和蒙桑聚在一起。研究结果对叶绿体基因组工程研究及桑属种间的分子标记开发和优良品种培育具有一定的参考价值。
DOI:10.7606/j.issn.1000-4025.2016.12.2385URL [本文引用: 1]

该研究以雌雄异株植物石刁柏为材料,利用基因组消减杂交技术对石刁柏雌雄核基因组中的性别差异核质体DNA(nuclear plastid DNA ,NUPTs)进行了分离和分析。结果表明:(1)通过构建消减杂交文库共获得了52个雄性偏向序列,序列长度分布在63~297 bp之间,其中有19个差异序列属于叶绿体来源序列(命名为 Ao1~Ao19),且这些序列与石刁柏叶绿体基因组的相似性均大于84%,Ao19与石刁柏叶绿体基因组相似性为100%。(2)利用基因组半定量PCR对19个NUPTs序列的性别差异分析表明,有4条序列为稳定的雄性偏向 NUPTs序列,分别为Ao1、Ao3、Ao10和Ao18。(3)序列比对表明,转移到核基因组的NUPTs主要来源于叶绿体基因组的反向重复区(包含IRa和IRb区),说明石刁柏叶绿体基因组重复区序列更容易向核基因组进行转移形成雄性偏向的NUPTs序列。
DOI:10.1098/rspb.2016.2806URL [本文引用: 1]
DOI:10.1016/S0022-2836(05)80360-2URL [本文引用: 1]

ABSTRACT A new approach to rapid sequence comparison, basic local alignment search tool (BLAST), directly approximates alignments that optimize a measure of local similarity, the maximal segment pair (MSP) score. Recent mathematical results on the stochastic properties of MSP scores allow an analysis of the performance of this method as well as the statistical significance of alignments it generates. The basic algorithm is simple and robust; it can be implemented in a number of ways and applied in a variety of contexts including straightforward DNA and protein sequence database searches, motif searches, gene identification searches, and in the analysis of multiple regions of similarity in long DNA sequences. In addition to its flexibility and tractability to mathematical analysis, BLAST is an order of magnitude faster than existing sequence comparison tools of comparable sensitivity.
[本文引用: 1]
DOI:10.1146/annurev.genet.40.110405.090448URLPMID:18076328 [本文引用: 1]

Transposable elements are mobile genetic units that exhibit broad diversity in their structure and transposition mechanisms. Transposable elements occupy a large fraction of many eukaryotic genomes and their movement and accumulation represent a major force shaping the genes and genomes of almost all organisms. This review focuses on DNA-mediated or class 2 transposons and emphasizes how this class of elements is distinguished from other types of mobile elements in terms of their structure, amplification dynamics, and genomic effect. We provide an up-to-date outlook on the diversity and taxonomic distribution of all major types of DNA transposons in eukaryotes, including Helitrons and Mavericks. We discuss some of the evolutionary forces that influence their maintenance and diversification in various genomic environments. Finally, we highlight how the distinctive biological features of DNA transposons have contributed to shape genome architecture and led to the emergence of genetic innovations in different eukaryotic lineages.
DOI:10.1007/s10142-007-0067-2URL [本文引用: 2]
DOI:10.1038/s41467-017-01064-8URLPMID:29093472 [本文引用: 1]

Sex chromosomes evolved from autosomes many times across the eukaryote phylogeny. Several models have been proposed to explain this transition, some involving male and female sterility mutations linked in a region of suppressed recombination betweenXandY(orZ/W,U/V) chromosomes. Comparative and experimental analysis of a reference genome assembly for a double haploidYYmale garden asparagus (Asparagus officinalisL.) individual implicates separate but linked genes as responsible for sex determination. Dioecy has evolved recently withinAsparagusand sex chromosomes are cytogenetically identical with theY, harboring a megabase segment that is missing from theX. We show that deletion of this entire region results in a male-to-female conversion, whereas loss of a single suppressor of female development drives male-to-hermaphrodite conversion. A single copy anther-specific gene with a male sterileArabidopsisknockout phenotype is also in theY-specific region, supporting a two-gene model for sex chromosome evolution. Several models have been proposed to explain the emergence of sex chromosomes. Here, through comparative genomics and mutant analysis, Harkess et al. show that linked but separate genes on theYchromosome are responsible for sex determination inAsparagus, supporting a two-gene model for sex chromosome evolution.
DOI:10.1038/hdy.2009.17 [本文引用: 1]
DOI:10.1007/s10709-005-5701-0URLPMID:17028949 [本文引用: 2]

Silene latifolia is a model dioecious plant with heteromorphic sex chromosomes. The Y chromosome is the largest in this species. Theoretical models propose an accumulation of repetitive DNA sequences in non-recombining parts of the Y chromosome. In this study, we isolated a BAC7H5 clone preferentially hybridizing to the Y chromosome of S. latifolia. Sequence analysis revealed that this BAC7H5 contains part of the chloroplast genome, indicating that these chloroplast sequences have accumulated on the Y chromosome and also may contribute to its large size. We constructed Y chromosome- and X chromosome-specific libraries and screened them to find Y- and/or X-linked copies of chloroplast sequences. Sequence analysis revealed higher divergence of a non-genic region of the chloroplast sequences located on the Y chromosome while genic regions tested showed only very low (max 0.9%) divergence from their chloroplast homologues.
DOI:10.1146/annurev.arplant.043008.092119URLPMID:19014347 [本文引用: 1]

In eukaryotes, DNA is exchanged between endosymbiosis-derived compartments (mitochondria and chloroplasts) and the nucleus. Organelle-to-nucleus DNA transfer involves repair of double-stranded breaks by nonhomologous end-joining, and resulted during early organelle evolution in massive relocation of organelle genes to the nucleus. A large fraction of the products of the nuclear genes so acquired are retargeted to their ancestral compartment; many others now function in new subcellular locations. Almost all present-day nuclear transfers of mitochondrial or plastid DNA give rise to noncoding sequences, dubbed nuclear mitochondrial DNAs (NUMTs) and nuclear plastid DNAs (NUPTs). Some of these sequences were recruited as exons, thus introducing new coding sequences into preexisting nuclear genes by a novel mechanism. In organisms derived from secondary or tertiary endosymbiosis, serial gene transfers involving nucleus-to-nucleus migration of DNA have also occurred. Intercompartmental DNA transfer therefore represents a significant driving force for gene and genome evolution, relocating and refashioning genes and contributing to genetic diversity.
DOI:10.1007/s00425-016-2485-7URL [本文引用: 3]
[本文引用: 2]
DOI:10.1073/pnas.1204237109URL [本文引用: 1]
DOI:10.1105/tpc.104.027706URL [本文引用: 2]
DOI:10.1038/hdy.2013.51URLPMID:23715017 [本文引用: 2]

We analysed the size, relative age and chromosomal localization of nuclear sequences of plastid and mitochondrial origin (NUPTs-nuclear plastid DNA and NUMTs-nuclear mitochondrial DNA) in six completely sequenced plant species. We found that the largest insertions showed lower divergence from organelle DNA than shorter insertions in all species, indicating their recent origin. The largest NUPT and NUMT insertions were localized in the vicinity of the centromeres in the small genomes of Arabidopsis and rice. They were also present in other chromosomal regions in the large genomes of soybean and maize. Localization of NUPTs and NUMTs correlated positively with distribution of transposable elements (TEs) in Arabidopsis and sorghum, negatively in grapevine and soybean, and did not correlate in rice or maize. We propose a model where new plastid and mitochondrial DNA sequences are inserted close to centromeres and are later fragmented by TE insertions and reshuffled away from the centromere or removed by ectopic recombination. The mode and tempo of TE dynamism determines the turnover of NUPTs and NUMTs resulting in their species-specific chromosomal distributions.
DOI:10.1093/molbev/msh110URLPMID:15014143 [本文引用: 1]

Abstract Mitochondrial DNA sequences are frequently transferred to the nucleus giving rise to the so-called nuclear mitochondrial DNA (NUMT). Analysis of 13 eukaryotic species with sequenced mitochondrial and nuclear genomes reveals a large interspecific variation of NUMT number and size. Copy number ranges from none or few copies in Anopheles, Caenorhabditis, Plasmodium, Drosophila, and Fugu to more than 500 in human, rice, and Arabidopsis. The average size is between 62 (baker's yeast) and 647 bps (Neurospora), respectively. A correlation between the abundance of NUMTs and the size of the nuclear or the mitochondrial genomes, or of the nuclear gene density, is not evident. Other factors, such as the number and/or stability of mitochondria in the germline, or species-specific mechanisms controlling accumulation/loss of nuclear DNA, might be responsible for the interspecific diversity in NUMT accumulation.
URL [本文引用: 1]
[本文引用: 4]
[本文引用: 1]
DOI:10.1007/s00438-013-0747-7URL [本文引用: 1]
DOI:10.1186/1471-2148-8-36URLPMID:2275221 [本文引用: 2]

pAbstract/p pBackground/p pVarious expansions or contractions of inverted repeats (IRs) in chloroplast genomes led to fluxes in the IR-LSC (large single copy) junctions. Previous studies revealed that some monocot IRs contain a ittrnH-rps19 /itgene cluster, and it has been speculated that this may be an evidence of a duplication event prior to the divergence of monocot lineages. Therefore, we compared the organizations of genes flanking two IR-LSC junctions in 123 angiosperm representatives to uncover the evolutionary dynamics of IR-LSC junctions in basal angiosperms and monocots./p pResults/p pThe organizations of genes flanking IR-LSC junctions in angiosperms can be classified into three types. Generally each IR of monocots contains a ittrnH-rps19 /itgene cluster near the IR-LSC junctions, which differs from those in non-monocot angiosperms. Moreover, IRs expanded more progressively in monocots than in non-monocot angiosperms. IR-LSC junctions commonly occurred at polyA tract or A-rich regions in angiosperms. Our RT-PCR assays indicate that in monocot IRsubA /subthe ittrnH-rps19 /itgene cluster is regulated by two opposing promoters, itS10/itsubitA /it/suband itpsbA/it./p pConclusion/p pTwo hypotheses are proposed to account for the evolution of IR expansions in monocots. Based on our observations, the inclusion of a ittrnH-rps19 /itcluster in majority of monocot IRs could be reasonably explained by the hypothesis that a DSB event first occurred at IRsubB /suband led to the expansion of IRs to ittrnH/it, followed by a successive DSB event within IRsubA /suband lead to the expansion of IRs to itrps19 /itor to itrpl22 /itso far. This implies that the duplication of ittrnH-rps19 /itgene cluster was prior to the diversification of extant monocot lineages. The duplicated ittrnH /itgenes in the IRsubB /subof most monocots and non-monocot angiosperms have distinct fates, which are likely regulated by different expression levels of itS10/itsubitA /it/suband itS10/itsubitB /it/subpromoters. Further study is needed to unravel the evolutionary significance of IR expansion in more recently diverged monocots./p
DOI:10.1093/dnares/dst045URLPMID:3989485 [本文引用: 1]

The transfer of organelle DNA fragments to the nuclear genome is frequently observed in eukaryotes. These transfers are thought to play an important role in gene and genome evolution of eukaryotes. In plants, such transfers occur from plastid to nuclear [nuclear plastid DNAs (NUPTs)] and mitochondrial to nuclear (nuclear mitochondrial DNAs) genomes. The amount and genomic organization of organelle DNA fragments have been studied in model plant species, such as Arabidopsis thaliana and rice. At present, publicly available genomic data can be used to conduct such studies in non-model plants. In this study, we analysed the amount and genomic organization of NUPTs in 17 plant species for which genome sequences are available. The amount and distribution of NUPTs varied among the species. We also estimated the distribution of NUPTs according to the time of integration (relative age) by conducting sequence similarity analysis between NUPTs and the plastid genome. The age distributions suggested that the present genomic constitutions of NUPTs could be explained by the combination of the rapidly eliminated deleterious parts and few but constantly existing less deleterious parts.<br>
水稻转座子研究进展
1
2007
... 目前, 已在多种植物的核基因组中发现了NUPTs (
鲁桑叶绿体基因组序列及特征分析
1
2018
... 取石刁柏的幼嫩叶片, 采用传统的CTAB法(
石刁柏雄性偏向核质体DNA的克隆与分析
1
2016
... 石刁柏(Asparagus officinalis)隶属百合科天门冬属, 是重要的雌雄异株植物.其基因组大小约为1.3 Gb, 染色体组成为2n=2x=20, 具有X和Y同型性染色体(
Sex chromosome evolution: historical insights and future perspectives
1
2017
... 目前普遍认为, 性染色体最初起源于1对常染色体, 在出现原始的性别决定基因之后, 包括性别决定基因在内的性别决定区的重组抑制是性染色体演化过程的关键步骤(
Basic local alignment search tool
1
1990
... 采用本地Blast (
A rapid DNA isolation procedure for small quantities of fresh leaf tissue
1
1987
... 取石刁柏的幼嫩叶片, 采用传统的CTAB法(
DNA transposons and the evolution of eukaryotic genomes
1
2007
... 目前, 已在多种植物的核基因组中发现了NUPTs (
Chloroplast DNA insertions into the nuclear genome of rice: the genes, sites and ages of insertion involved
2
2008
... 目前, 已在多种植物的核基因组中发现了NUPTs (
... )中, 大片段的NUPTs主要集中在着丝粒及侧翼区, 随着演化的进行NUPTs长度逐渐衰减, 并且伴随着拷贝数的增加(
The asparagus genome sheds light on the origin and evolution of a young Y chromosome
1
2017
... 本研究所用的生物信息学分析数据包括石刁柏(Asparagus officinalis L.)叶绿体基因组(下载自
The role of repetitive DNA in structure and evolution of sex chromosomes in plants
1
2009
... 目前普遍认为, 性染色体最初起源于1对常染色体, 在出现原始的性别决定基因之后, 包括性别决定基因在内的性别决定区的重组抑制是性染色体演化过程的关键步骤(
Accumulation of chloroplast DNA sequences on the Y chromosome of Silene latifolia
2
2006
... 目前, 已在多种植物的核基因组中发现了NUPTs (
... ).有研究表明, NUPTs在雌雄异株植物的性染色体上累积较多.
DNA transfer from organelles to the nucleus: the idiosyncratic genetics of endosymbiosis
1
2009
... 植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(
Repetitive sequences and epigenetic modification: inseparable partners play important roles in the evolution of plant sex chromosomes
3
2016
... 目前, 已在多种植物的核基因组中发现了NUPTs (
... 目前普遍认为, 性染色体最初起源于1对常染色体, 在出现原始的性别决定基因之后, 包括性别决定基因在内的性别决定区的重组抑制是性染色体演化过程的关键步骤(
... ).包括NUPTs在内的重复序列的插入造成异染色质化或倒位是重组抑制形成的重要原因, 而重组抑制区还可以招募更多的NUPTs等重复序列, 使重组抑制区进一步扩展, 最终形成异型的性染色体(
Identification of the sex chromosome pair in asparagus ( Asparagus officinalis L.)
2
1979
... 石刁柏(Asparagus officinalis)隶属百合科天门冬属, 是重要的雌雄异株植物.其基因组大小约为1.3 Gb, 染色体组成为2n=2x=20, 具有X和Y同型性染色体(
... 将扩增获得的2个IR序列进行回收纯化后标记探针, 与45S rDNA同时进行双色荧光原位杂交.结果表明, 45S rDNA共有6个信号, 分布在3对同源染色体上(
Selfish supernumerary chromosome reveals its origin as a mosaic of host genome and organellar sequences
1
2012
... 研究表明, 植物中NUPTs更倾向于插入和累积在着丝粒及其侧翼区等染色体异染色质区域(
The rice nuclear genome continuously integrates, shuffles, and eli- minates the chloroplast genome to cause chloroplast- nuclear DNA flux
2
2005
... 本研究表明, 叶绿体DNA序列不同区域向核基因组转移的频率存在差异, 石刁柏叶绿体基因组中IR区序列更易向核基因组中进行水平转移, 这与在白麦瓶草(
... 研究表明, 植物中NUPTs更倾向于插入和累积在着丝粒及其侧翼区等染色体异染色质区域(
Analysis of plastid and mitochondrial DNA insertions in the nucleus (NUPTs and NUMTs) of six plant species: size, relative age and chromosomal localization
2
2013
... 植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(
... 研究表明, 植物中NUPTs更倾向于插入和累积在着丝粒及其侧翼区等染色体异染色质区域(
NUMTs in sequenced eukaryotic genomes
1
2004
... 植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(
Complete chloroplast genome sequence of asparagus ( Asparagus officinalis L.) and its phylogenetic position within asparagales
1
2017
... 本研究所用的生物信息学分析数据包括石刁柏(Asparagus officinalis L.)叶绿体基因组(下载自
Strong accumulation of chloroplast DNA in the Y chromosomes of Rumex acetosa and Silene latifolia
4
2014
... 目前, 已在多种植物的核基因组中发现了NUPTs (
... 本研究表明, 叶绿体DNA序列不同区域向核基因组转移的频率存在差异, 石刁柏叶绿体基因组中IR区序列更易向核基因组中进行水平转移, 这与在白麦瓶草(
... )基因组中, IR区比叶绿体基因组中其它位置的序列更难转移形成NUPTs (
... 研究表明, 植物中NUPTs更倾向于插入和累积在着丝粒及其侧翼区等染色体异染色质区域(
Endosymbiotic gene transfer: organelle genomes forge eukaryotic chromosomes
1
2004
... 植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(
Organelle DNA accumulation in the recently evolved papaya sex chromosomes
1
2013
... 目前, 已在多种植物的核基因组中发现了NUPTs (
Dynamics and evolution of the inverted repeat-large single copy junctions in the chloroplast genomes of monocots
2
2008
... 本研究表明, 叶绿体DNA序列不同区域向核基因组转移的频率存在差异, 石刁柏叶绿体基因组中IR区序列更易向核基因组中进行水平转移, 这与在白麦瓶草(
... 研究表明, 被子植物的IR区包含1个polyA区域, 这个区域可能是重组的热点.通过这种机制, 其中1个IR区域利用第2个IR区中同源序列的修复/重组过程延伸到相邻的SC区中(
Patterns of genomic integration of nuclear chloroplast DNA fragments in plant species
1
2014
... 植物细胞除了含有细胞核基因组之外, 还包含细胞器基因组, 主要包括线粒体基因组和叶绿体基因组(
备案号: 京ICP备16067583号-21
版权所有 © 2021 《植物学报》编辑部
地址:北京香山南辛村20号 邮编:100093
电话:010-62836135 010-62836131 E-mail:cbb@ibcas.ac.cn
本系统由北京玛格泰克科技发展有限公司设计开发
