 ,1,*
,1,*Phylogeny and Tissue-specific Expression of the GBSS Genes in Oryza officinalis
Xia Zhang1, Xiang Jing1, Guangcai Zhou2, Ying Bao ,1,*
,1,*通讯作者:
收稿日期:2018-04-26接受日期:2018-12-10网络出版日期:2019-07-01
| 基金资助: |
Corresponding authors:
Received:2018-04-26Accepted:2018-12-10Online:2019-07-01

摘要
关键词:
Abstract
Keywords:
PDF (1243KB)摘要页面多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
张霞, 景翔, 周光才, 包颖. 药用野生稻GBSS基因的系统发育及组织特异性表达. 植物学报, 2019, 54(3): 343-349 doi:10.11983/CBB18106
Zhang Xia, Jing Xiang, Zhou Guangcai, Bao Ying.
淀粉作为自然界天然生成的重要碳水化合物, 不仅为植物生长发育提供足够的营养和能量, 而且为人类和动物提供必要的食物(包颖等, 2015)。植物中的淀粉合成可以发生在不同的器官, 并基本可以划分为2个不同的阶段: 其一发生在光合器官(如叶), 由叶绿体通过光合作用进行首次的淀粉合成, 此为瞬时淀粉合成阶段; 但瞬时合成的淀粉在夜晚会解体成为麦芽糖和葡萄糖, 并进一步合成蔗糖, 其中一部分参与代谢, 而另一部分则运输到植物储藏器官(如根、茎、果实和种子), 由造粉体等质体重新合成淀粉, 此为储藏淀粉的合成阶段(包颖等, 2015)。
两个阶段的淀粉合成均至少需要5种酶参与, 它们分别是ADP焦磷酸化酶(AGPase)、淀粉合酶(SSS)、束缚态淀粉合酶(GBSS)、淀粉分支酶(SBE)和淀粉去分支酶(SBE)。其中, 束缚态淀粉合酶又被称为颗粒结合型淀粉合酶(granule-bound starch synthase), 该酶被认为与直链淀粉的合成有关(Shure et al., 1983)。由于直链淀粉与支链淀粉的相对比例决定淀粉糯性, 而糯性比例又直接影响淀粉的口感和营养价值, 因此GBSS的重要性不言而喻。在栽培水稻(Oryza sativa)中, GBSS具有2种异构型(张鹏, 2008)。其中, 相对分子量约为60 kDa的GBSSI也被称为Waxy蛋白, 其编码基因GBSSI或Waxy被称为糯性基因或蜡质基因, 该基因在水稻基因组中位于第6号染色体的短臂, 由14个外显子和13个内含子组成(Wang et al., 1990)。此外, Dian等(2005)在研究水稻淀粉合成的过程中, 发现水稻基因组中还存在分子量为70-100 kDa的GBSSII, 该酶的编码基因为GBSSII。GBSSII和GBSSI的结构相似, 二者在氨基酸序列水平的一致度达66% (顾燕娟, 2006)。尽管GBSS基因在水稻中的研究已经比较深入, 但在水稻野生亲缘种内的研究, 尤其是2个异构酶基因在不同淀粉合成阶段的表达状态等鲜有报道, 极大地限制了我们对GBSS基因在禾本科内整体进化的全面了解, 也不利于今后从遗传的角度对此类基因进行有效利用。因此, 非常有必要扩展范围, 将尽可能多的野生种类纳入到研究之中。
药用野生稻(O. officinalis)是我国自然分布的3种野生稻之一。与栽培稻的AA基因组不同, 药用野生稻具有CC基因组(杨学明, 2003), 相异的基因组类型使该物种具有与栽培稻不一样的遗传多样性, 因此是用于水稻遗传改良的良好种质资源。
本研究以药用野生稻为研究对象, 通过序列相似性比较、进化分析和荧光实时定量PCR等方法, 定性并定量地对GBSSI和GBSSII基因的编码序列、与其它植物同源基因的系统发育关系以及在代表淀粉合成不同阶段的器官(叶和种子)中的表达情况进行深入探究, 旨在回答如下问题: (1) 在DNA序列水平, 药用野生稻GBSSI和GBSSII彼此之间以及与其它植物的同源基因之间的序列相似度和分子进化关系如何? (2) 在基因表达水平, 药用野生稻GBSSI和GBSSII在相同或不同的淀粉合成阶段是否存在差异表达? 其表达模式在植物中是否保守?
1 材料与方法
1.1 材料
药用野生稻(Oryza officinalis Wall. ex Watt)种子来自国际水稻研究所(International Rice Research Insti- tute)种子库(编号104973)。为打破种子休眠, 将去壳的药用野生稻种子在50°C恒温箱内处理5天; 之后将其移入铺有湿润滤纸的培养皿中, 在30°C的培养箱中萌发。萌发后的种子直接播种在大花盆中, 并移入温室中培养至成年植株, 分别选取午后充分开展的全叶和完全成熟的种子用于RNA提取。1.2 目标基因获取
以栽培稻GBSSI (GenBank No.FJ750947.1)和GBSSII (GenBank No.AY069940.1)为参考序列, 利用本实验室早期的药用野生稻叶转录组数据(Bao et al., 2015)进行对位排列和组装, 具体方法详见包颖等(2015)。同时, 以GenBank (https://www.ncbi.nlm. nih.gov/)中栽培稻的GBSSI和GBSSII基因作为搜索序列, 生物选项中填入“Oryza officinalis (taxid: 4535)”, 进行BLAST直接查询。1.3 药用野生稻与其它植物GBSS同源基因的序列比较和系统发育分析
为深入了解药用野生稻GBSS基因和其它植物在DNA序列水平上的遗传差异并探究该基因在植物中的分子进化趋势, 我们利用NCBI (https://www.ncbi. nlm.nih.gov/)和Uniprot (https://www.uniprot.org/)数据库, 结合注释查询和以目标基因为搜索项的BLAST搜寻(阈值设为1e-5), 对具有全基因组数据的18种植物(表2)的GBSS同源基因的核苷酸或其编码的氨基酸序列进行全面查找。将获取的所有序列全部转换成蛋白序列并导入Seaview软件(Gouy et al., 2010)。采用Clustal W方法进行对位排列后, 利用PhyML v3.0.1软件(Guindon and Gascuel, 2003)在默认模式下构建最大似然性(maximum likelihood, ML)系统发育树。以藻类植物莱茵衣藻(Chlamydo- monas reinhardtii P. A. Dang.)作为外类群。1.4 GBSS基因相对表达分析
分别选取药用野生稻的叶和种子各3份生物学重复样品混合后, 采用超纯RNA提取试剂盒(CWbio, Cat No.cw0581)提取总RNA, 然后采用AMV Reverse Transcriptase XL反转录酶(TaKaRa, Cat No.2630A)将RNA反转录成cDNA。以药用野生稻GBSSI和II序列为依据, 应用Primer Premier 5.0软件在其保守区域设计定量引物(表1)。为保证扩增产物的准确性, 我们以cDNA作为模板, 对目的基因进行RT-PCR扩增, 然后将得到的PCR产物回收后测序。测序工作在3100-Avant Genetic Analyzer (Life Technologies)自动测序仪上完成。
Table 1
表1
表1GBSSI和GBSSII的引物序列
Table 1
| Gene | Primer name | Primer sequence (5'-3') |
|---|---|---|
| GBSSI | GBSSI-F | AACGTGGCTGCTCCTTGAA |
| GBSSI-R | TTGGCAATAAGCCACACACA | |
| GBSSII | GBSSII-F | AGGCATCGAGGGTGAGGAG |
| GBSSII-R | CCATCTGGCCCACATCTCTA |
新窗口打开|下载CSV
为定量了解2个GBSS基因在O. officinalis不同淀粉合成阶段的表达差异, 我们分别选取作为光合器官的叶和作为储存器官的种子进行定量分析, 以Actin为内参基因, 引物序列参照陈凤花等(2005)。定量反应在RoChe Light Cycler? 480II荧光定量PCR仪上完成。GBSSI和GBSSII在2种器官内的相对定量采用2-ΔΔCt算法进行计算。
2 结果与讨论
2.1 GBSSI和GBSSII的序列特点
通过对实验室前期的叶转录组数据进行分析, 我们只找到了GBSSII基因(GenBank No.KM264356.1), 这可能是由于GBSSI在药用野生稻叶中不表达或表达量过低造成。但是, 我们幸运地在药用野生稻中通过GenBank BLAST搜索到Waxy基因, 即GBSSI (GenBank No.GU977195.1)序列全长。该GBSSI长 3 071 bp, 包括14个外显子和13个内含子(图1), 结构和栽培稻同源基因(GenBank No.X64108.1)完全一致。对于GBSSII, 由于在叶转录组数据中, 我们只得到其mRNA序列, 为了解该基因的结构, 我们将其mRNA序列与栽培稻同源基因(GenBank No.GQ150862.1)的DNA和CDS序列同时进行对位排列。排列结果显示, 药用野生稻GBSSII基因具有与栽培稻同源基因一致的基因结构, 即包括13个外显子和12个内含子(图1)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1药用野生稻GBSSI和GBSSII基因结构
Figure 1Gene structures of the GBSSI and GBSSII in Oryza officinalis
编码区序列分析表明, 尽管药用野生稻GBSSI和GBSSII的CDS长度非常接近, 分别为1 830 和1 827 bp, 但彼此的相似程度并不高, 二者的序列一致性在核苷酸水平为63.80%, 氨基酸水平为61.97%。进一步对这2个GBSS与其它植物的同源基因进行比较, 发现其同源对之间的一致度随物种的不同而存在差异(表2)。例如, 就蛋白质序列而言, 药用野生稻与栽培稻的籼稻亚种(O. sativa subsp. indica) GBSSI (GBSSII)直系同源对之间有98% (97%)的氨基酸一致度; 而与莱茵衣藻(Chlamydomonas reinhardtii)之间的一致度则下降到48% (47%)。 此外, 除栽培稻外, 其它同样隶属禾本科的植物与药用野生稻的GBSS直系同源对之间也都具有较高的相似性(79%- 93%)。但是, 对于非禾本科植物, 其GBSS基因与药用野生稻GBSSI或GBSSII的一致度均下降到62%- 68%之间。
Table 2
表2
表2药用野生稻和其它植物的GBSS氨基酸序列一致度
Table 2
| Plants | Gene (GenBank/UniProt No.) | Sequence identity (%) | |
|---|---|---|---|
| Oryza officinalis | |||
| GBSSI | GBSSII | ||
| Oryza sativa subsp. japonica | GBSSI (XP_025882300.1) | 95.22 | 60.35 |
| GBSSII (XP_015647210.1) | 62.30 | 96.72 | |
| O. sativa subsp. indica | GBSSI (AAN77100.1) | 97.87 | 62.30 |
| GBSSII (ACY56079.1) | 62.46 | 97.04 | |
| Leersia perrieri | GBSSI (A0A0D9WLF6) * | 93.01 | 62.15 |
| GBSSII (A0A0D9WED4) * | 58.44 | 85.62 | |
| Zizania latifolia | GBSSI (ASSH01051725.1) ** | 90.80 | 62.01 |
| GBSSII (ASSH01023543.1) ** | 59.46 | 87.47 | |
| Brachypodium distachyon | GBSSI (XP_003557139.1) | 79.31 | 61.16 |
| GBSSII (XP_003569238.1) | 60.33 | 85.43 | |
| Hordeum vulgare | GBSSI (BAC41202.1) | 81.97 | 60.76 |
| GBSSII (BAJ99426.1) | 61.85 | 86.12 | |
| Setaria italica | GBSSI (AGW27658.1) | 86.72 | 61.90 |
| GBSSII (XP_004956034.1) | 62.40 | 87.34 | |
| Sorghum bicolor | GBSSI (XP_002436418.1) | 82.52 | 61.90 |
| GBSSII (XP_002461889.1) | 62.62 | 86.18 | |
| Triticum aestivum | GBSSI (XP_020146905.1) | 81.37 | 60.36 |
| GBSSII (AAG27624.1) | 61.79 | 86.09 | |
| Zea mays | GBSSI (NP_001105001) | 82.08 | 62.13 |
| GBSSII (NP_001334833) | 62.36 | 85.88 | |
| Musa acuminata | GBSS1 (KF512020.1) | 66.39 | 65.35 |
| GBSS2 (KF512021.1) | 67.96 | 63.99 | |
| GBSS3 (KF512023.1) | 63.93 | 62.91 | |
| Arabidopsis thaliana | GBSS (XP_020866584.1) | 62.48 | 63.88 |
| Gossypium raimondii | GBSS1 (XP_012474755.1) | 63.89 | 66.01 |
| GBSS2 (XP_012439861.1) | 63.11 | 66.77 | |
| GBSS3 (XP_012486622.1) | 62.75 | 64.01 | |
| Solanum lycopersicum | GBSS (NP_001311457.1) | 67.05 | 62.03 |
| Carica papaya | GBSS (XP_021900468.1) | 65.91 | 66.45 |
| S. tuberosum | GBSS (XP_006343763.1) | 62.89 | 67.27 |
| Vitis vinifera | GBSS1 (XP_010660257.1) | 65.52 | 66.23 |
| GBSS2 (XP_019081062.1) | 64.07 | 67.76 | |
| Amborella trichopoda | GBSS (XP_006837847.1) | 62.81 | 66.17 |
| Chlamydomonas reinhardtii | GBSS (XP_001697117.1) | 47.79 | 47.05 |
新窗口打开|下载CSV
2.2 系统发育分析
在上述18种植物中, 我们共筛选到34条同源基因(表2), 利用这些基因和药用野生稻的2个GBSS基因的氨基酸序列, 我们构建了最大似然性(ML)系统发育关系树(图2)。该树所展示的拓扑结构和已知植物的系统进化关系高度一致。单子叶植物和真双子叶植物首先以较高的支持率(97%和91%)并列成2个大支, 而作为被子植物基部类群的无油樟(Amborella trichopoda)毫无例外地位于这2大支的外围基部。在单子叶植物分支内部, 比较突出的特点是禾本科的所有植物都包含2个GBSS基因, 即GBSSI和GBSSII, 并且这2个基因的同源序列各自聚合在一起, 进一步构成2个平行的分支。其中, 值得注意的是, 芭蕉科植物M. acuminata的3个GBSS基因并没有分布在不同的分支, 而是单独聚在一起, 并于最后归入禾本科的GBSSI分支。此外, 在一些小的分支上, ML树也同样展示了物种之间清晰的亲缘关系。例如, 同为稻属的药用野生稻与栽培稻(O. sativa subsp. japonica和O. sativa subsp. indica)在GBSSI和GBSSII同源序列的2个分支内均呈现一致的拓扑结构, 即均是首先聚合在一起, 然后先后与其近缘的假稻属植物Leersia perrieri, 以及菰属植物Zizania latifolia聚成高支持率(98%)的递进分支, 最后与以玉米(Zea mays)和小麦(Triticum aestivum)为代表的各自小分支形成并列关系。相比单子叶植物, 真双子叶植物GBSS多为单个基因, 即使少数植物内出现多拷贝, 如雷蒙德氏棉(Gossypium raimondii)内出现3个GBSS, 但这3个GBSS均为物种特异, 没有进入其它物种分支, 而是单独聚合在一起。 因此, 双子叶植物分支中没有出现类似禾本科GBSSI和GBSSII的并列的同源序列分支。图2
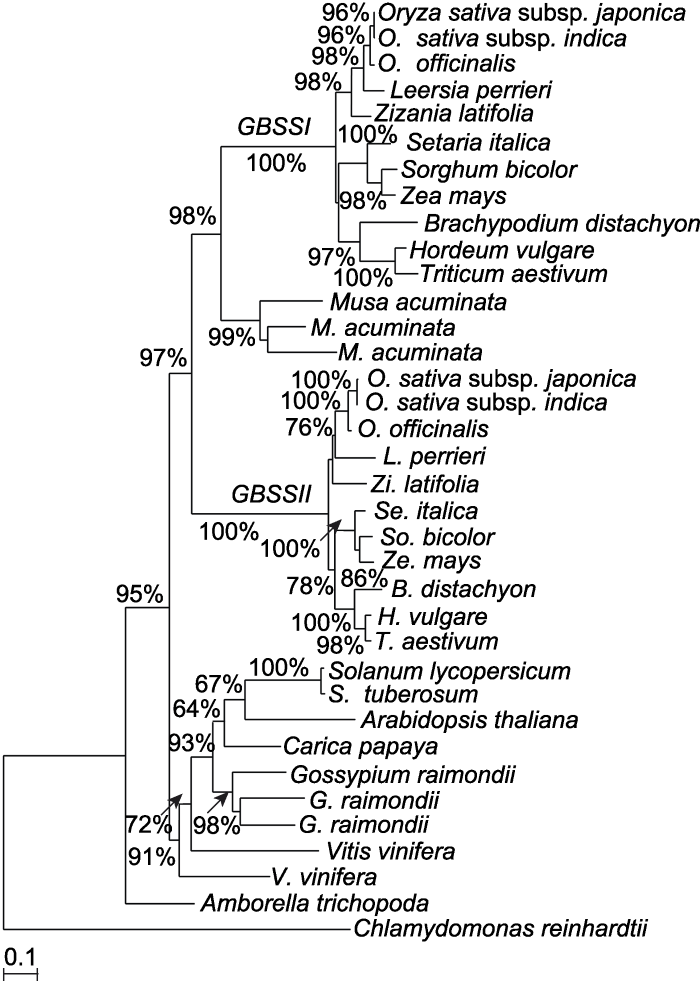 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2利用氨基酸序列构建的淀粉合酶的最大似然性系统发育树分支旁的数字代表自展支持率。
Figure 2A maximum likelihood phylogenetic tree of the GBSS based on amino acid sequences Numbers near branches indicate bootstrap value.
2.3 GBSSI和GBSSII在不同器官中的相对定量表达
通过定量PCR分析, 我们发现在光合器官叶中, 2个GBSS基因均有表达, 但GBSSII的表达量较高, 约比GBSSI高2倍, 说明在淀粉合成的第1个阶段即瞬时淀粉合成阶段, GBSSII基因比GBSSI基因发挥更为重要的作用(图3A, B)。但是, 这种情况并没有持续, 在淀粉合成的第2个阶段即储存淀粉的合成阶段(在本研究中选取的是种子), 2个GBSS基因的表达出现两极分化, 其中GBSSI表达量上升极快, 而GBSSII的表达量却急速下降, 接近不表达。此外, 对比在叶和种子内均表达的GBSSI的相对表达量, 我们发现种子中GBSSI的表达量较叶中提高了1 221倍。因此不难发现, 在药用野生稻中, GBSS的表达存在时空分化现象, 其中,GBSSII在叶中优势表达, 而GBSSI在种子中高效表达(图3C, D), 这可能与淀粉合成的第2个阶段, 即种子胚乳中营养快速累积有关。
图3
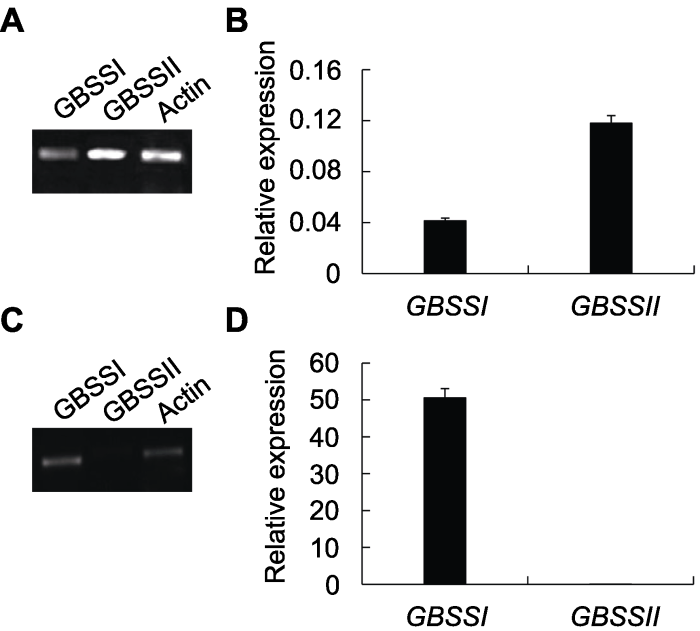 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3GBSSI和GBSSII在药用野生稻叶和种子中的相对表达
(A), (B) GBSS基因在叶中的RT-PCR和qRT-PCR扩增结果; (C), (D) GBSS基因在种子中的RT-PCR和qRT-PCR扩增结果。
Figure 3Relative expression of GBSSI and GBSSII in leaves and seeds of Oryza officinalis
(A), (B) Amplification results of RT-PCR and qRT-PCR for GBSS genes in leaves; (C), (D) Amplification results of RT-PCR and qRT-PCR for GBSS genes in seeds.
2.4 讨论
淀粉合酶GBSS是直链淀粉合成中最为关键的酶, 在药用野生稻中, 我们共鉴定出2个控制直链淀粉合成的基因(GBSSI和GBSSII)。作为旁系同源基因, 这2个基因彼此之间并不十分相似, 其CDS序列存在36.2%的核苷酸差异。但是, 对于物种间的GBSS直系同源基因, 其相似性却与选取的物种密切相关, 其亲缘关系较近的物种相似性较高(表2)。非常有意思的是, 似乎仅在禾本科内发现同时拥有2个旁系同源GBSS基因I和II的情况。最近, 基于20种陆生植物和2种藻类植物的基因组数据, 王倩等(2017)分析了GBSS基因家族的构成, 结果与我们目前的发现一致, 即所选的所有禾本科植物, 无一例外地同时具有2个GBSS拷贝。我们的序列一致度分析进一步表明, 只有禾本科内的直系同源基因之间的一致度能达到80%以上; 而非禾本科植物, 即使同时包括2个以上的拷贝, 它们的GBSS与禾本科内的2个基因的相似度也差异不大(均为60%以上)。这种情况一方面表明GBSS基因在植物中的高度保守性; 另一方面也表明非禾本科植物的GBSS基因, 无论拷贝数目多少, 基本上都属于同一类型。Cheng等(2012)根据比较系统的进化分析推断, 被子植物中的GBSS基因大约在2 510万年前发生了重复, 并由此产生了GBSSI和GBSSII两类基因, 但在后续的进化中, GBSSI仅在单子叶植物中被保留下来。因此暗示目前双子叶植物中的GBSS均为原始的GBSSII谱系直接保留或继续复制形成。从基因表达水平来看, 目前的研究表明, 药用野生稻GBSSI与GBSSII在叶和种子中具有非常不一致的表达模式。在叶中, GBSSII占据表达优势, 但在种子中, GBSSI出现强势表达。这种表达方式与栽培稻中2个同源基因的表达方式非常类似, 但在栽培稻中, GBSSI完全在种子中特异表达(Ohdan et al., 2005)。这说明相比野生稻, 栽培稻GBSSI和GBSSII基因表达的时空特异性更为明显, 功能的亚分化更加彻底。本研究中, 2个GBSS基因在淀粉合成不同阶段的差异表达显示, 至少从野生稻开始出现了稻属GBSSI和GBSSII的功能亚分化现象。这种亚分化不仅体现在同一基因在不同淀粉合成阶段的不同表达水平(如GBSSII的有和无的差异)上, 也体现在不同GBSS基因在同一器官中的表达差异(图3)。
当然, 已有研究表明, GBSSI和GBSSII分化表达的现象不仅局限于稻属。例如, Vrinten和Nakamura (2000)的研究证明, 小麦GBSSI在胚乳中高表达, 而GBSSII仅在叶、杆和果皮中表达。但是, 在非禾本科植物中, GBSS基因却承担了禾本科植物中2类GBSS的全部功能。例如, 在马铃薯(Solanum tuberosum)中, GBSS基因是单拷贝, 它在植物光合和储藏器官均有表达, 但在块茎中的表达水平明显高于叶(Van Harsselaar et al., 2017)。结合前人基于全基因组的搜索和系统发育分析(Cheng et al., 2012; 王倩等, 2017), 以及本研究结果, 即GBSSI只出现在禾本科植物中, 我们推测禾本科植物的GBSS基因之所以发生功能亚分化, 由GBSSI取代GBSS在双子叶植物内全效表达的部分功能, 成为存储器官特异基因, 可能与其为适应淀粉在胚乳中的大量积累所产生的谱系特异进化密切相关。更多有关淀粉代谢途径在禾本科植物进化中作用的详细研究将有助于进一步阐明这种功能分化的意义。
致谢 感谢国际水稻研究所(International Rice Research Institute, IRRI)提供药用野生稻种子样品。
(责任编辑: 朱亚娜)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.11983/CBB14147URL [本文引用: 2]

淀粉不仅是植物自身和后代生长繁殖的重要营养与能量储备,而且是人类膳食中碳水化合物的主要来源.植物中淀粉合成主要发生在两个阶段,一是在形成临时淀粉的光合作用阶段,另一个则是在成为贮藏淀粉的营养积累阶段.相对于最后的淀粉贮藏阶段,临时淀粉的形成阶段在植物整个碳水化合物代谢过程中扮演着更为重要的角色,然而却一直少有关注.为深入研究初始淀粉合成过程中相关酶在植物中的进化模式,选取了药用野生稻(Oryza officinalis)为研究对象,通过对其全叶转录组的重测序,定性、定量地调查了淀粉合成酶基因家族在稻属野生物种光合器官中的基因类型和表达变化.共有8个淀粉合成酶基因的完整编码序列在药用野生稻的叶中首次被识别.系统发育分析表明,这8个基因分别隶属SSⅠ、SSⅡ、SSⅢ、SSⅣ、SSⅤ和GBSSⅡ基因家族.序列比较和相对表达定量分析显示,药用野生稻与栽培稻的淀粉合成酶基因家族的进化模式具有高度的一致性,两个物种的同源基因在mRNA水平的序列相似度达到95%-98%.基于非同义置换和同义置换比率的统计检验表明,8个基因在两个物种间均经历了严格的纯化选择.另外,3个在栽培稻胚乳中特异表达的基因在药用野生稻的叶转录组中未筛查出来,而4个在栽培稻叶中优势表达的基因在药用野生稻叶中同样呈现相对较高水平的表达.
DOI:10.3969/j.issn.1001-764X.2005.05.039URL [本文引用: 1]

实时荧光定量RT-PCR(real-time fluorescent quantitativereverse transcription-polymerase chain reaction,FQ RT-PCR)是检测低丰度mRNA的敏感方法,为了去除不同标本在RNA的产量、质量以及逆转录效率上可能存在的差别而获得目标基因特异性表达的真正差异,通常选择一定的内
DOI:10.7666/d.y927534URL [本文引用: 1]

为了解支链淀粉合成相关基因等位基因间的差异对稻米淀粉理化特性的影响,试验利用苏御糯为供体亲本、桂朝2号为受体亲本的BC2F4群体,将检测到的只含有1个来自苏御糯品质基因的单株进行相互杂交,经加代得到遗传背景相近的3个F_2群体,继而通过发展分子标记进行基因型检测、分析不同纯合基因型植株胚乳淀粉的理化特性、定量分析水稻胚乳发育时期相关基因的mRNA在胚乳中的表达量,研究了5个支链淀粉合成相关基因等位基因间的差异对稻米胚乳淀粉理化特性的影响,主要结果如下: 1、依据水稻籼粳亚种间序列差异发展了SssⅢ-1和SssIV-1的分子标记。 2、Sbe1等位基因的差异对表观直链淀粉含量和胶稠度的影响达到极显著,对糊化温度没有明显影响。当苏御糯的Sbe1s导入桂朝2号替换了Sbe1g时,使桂朝2号直链淀粉含量显著降低、胶稠度变硬。不同Sbe1基因的单株RVA谱也有差异,带有Sbe1g基因的单株的最高粘度、热浆粘度和冷胶粘度总体上高于带有Sbe1s基因的单株。 3、SssⅢ-1等位基因的差异对直链淀粉含量和糊化温度的影响不显著,但对胶稠度和淀粉粘滞特性有显著影响,当苏御糯的SssⅢ-1s导入桂朝2号替换了SssⅢ-1g时,会使稻米淀粉的胶稠度变软,使最高粘度、热浆粘度、冷胶粘度3个一级特征值及回复值变大。 4、SssⅣ-1基因、Isa基因和Pul基因的等位性差异对稻米淀粉理化特性无明显影响。 5、在Sbe1/SssⅢ-1F_2群体中,Sbe1s基因单株的mRNA表达量明显大于Sbe1g单株的的表达量,由于Sbe1s基因单株种子胚乳直链淀粉含量也较Sbe1g单株显著降低,似乎暗示着Sbe1的mRNA表达强度与支链淀粉的合成有着较为密切的关系。 6、不同的支链淀粉合成相关基因之间存在互作,其中Sbe1和SssⅢ-1之间的基因互作,对糊化温度和冷胶粘度、回复值、消减值的效应达到显著或极显著。SssⅢ-1基因对Sbe1s的mRNA表达、Sbe1基因对SssⅢ-1的mRNA表达、Isa对SssⅣ-1的mRNA表达均有一定影响。
DOI:10.11983/CBB16041URL [本文引用: 2]

为全面理解植物颗粒结合淀粉合酶(GBSS)基因在植物中的进化模式并重建其进化历史,利用20种陆生植物和2种藻类植物的基因组数据,通过生物信息学手段,深入挖掘和分析植物类群基因组中GBSS基因家族的构成和基因特点,推测其可能的扩增和丢失规律.结果共识别42条同源序列.系统发育和进化分析表明,GBSS基因起源古老,可能在所有绿色植物的祖先中就已经出现,之后在进化过程中不断发生谱系的特异扩张和拷贝丢失,并最终通过功能分化的形式在植物类群中被固定.
DOI:10.7666/d.y564718URL [本文引用: 1]

稻属包含有亚洲栽培稻和非洲栽培稻两个栽培种及20余个野生稻种,迄今已发现有10个染色体组(A-H,J-K),构成了10个染色体组型(AA、BB、BBCC、CC、CCDD、EE、FF、GG、HHJJ、HHKK)。重复序列是研究物种起源和进化最有效的手段之一,在水稻基因组中大约有50%的DNA为重复序列,因而研究水稻中的重复序列在不同稻种中的分布,有助于在分子水平上研究亚洲栽培稻的起源与分化、普通野生稻的分化以及不同稻种之间的亲缘关系等。45S rDNA是水稻中与核糖体RNA合成有关的一个高度串联重复序列,已被定位于亚洲栽培稻的核仁组织区(Nucleolus Organizer Region,简称NOR);Os48是水稻AA染色体组亚端粒的一个串联重复序列。利用荧光原位杂交(FISH)技术,可准确地分析这两个重复序列在不同稻种染色体上的分布情况,结果如下。 (1)在籼稻品种中一般具有2对NOR、5-7对Os48位点,在偏籼的广亲和品种Dular和Aus373中有2对NOR、5对Os48位点,NOR位于第9和第10染色体上,Os48杂交信号强度之间有较大差异。在粳稻品种中一般具有1对NOR、2-3对Os48位点,其中广亲和品种02428具有1对NOR、3对Os48,NOR位于第9染色体上,Os48杂交信号均较强,这两个重复序列的表现与粳稻非常类似。 扬州大学硕士学位论文 (2)一年生普通野生稻具有1一2对NOR、2一5对0548位点,NOR信 号强度第9染色体强于第10染色体,0548杂交信号强度均较强。存在1 对NOR、2对0548位点的一年生普通野生稻,这与粳稻品种相近,因而粳 稻可能与一年生普通野生稻亲缘关系较近。 (3)多年生野生稻一般具有2对NOR、7一8对0548位点,NOR位于 第9和第10染色体上。从这两序列的数目与杂交信号强度分析,粕稻品种 与多年生普通野生稻相近,多年生野生稻可能是釉稻的直接祖先。多年生 普通野生稻中也存在1对NOR、9一10对0548位点的材料,而且在一对染 色体的两个末端都存在0548位点,这与其它类型的水稻均有区别。 (4)非洲栽培稻具有1对NOR,而0548位点数目因材料来源不同而 不同,有3种类型,即2对、4对和10对。巴蒂野生稻具有1对NOR、4 对0548。长药野生稻具有2对NOR、5对0548。 (5)来自哥伦比亚的1份展颖野生稻具有1对NOR,没有0548杂交 信号,由于0548是AA组染色体的一个串联重复序列,因而这份材料是否 为AA组野生稻还待进一步研究。 (6)在非AA组野生稻中,没有0548杂交位点。斑点野生稻具有3 对NOR。药用野生稻也具有3对NOR,但其中有1对较弱的杂交信号位于 染色体的近末端。小粒野生稻具有3对或5对NOR。宽叶野生稻具有2对 或5对NOR。澳洲野生稻和短药野生稻均具有1对NOR。
DOI:10.7666/d.y1261684URL [本文引用: 1]

淀粉是农业中最重要的加工原料。通过酶处理,淀粉可被加工成各种甜料,并且可以被用来作为发酵的原料,同时,可作为干淀粉用于各种不同用途。天然的或化学修饰过的淀粉广泛用于食品和非食品工业,而特定的理化特性是决定淀粉特殊用途的最主要因素。淀粉是由直链淀粉与支链淀粉两种成分组成,为了有效利用这两种成分的各自特性,培育出仅含有直链淀粉或支链淀粉的植物将是很有必要的。目前,科学家们已经利用基因工程等技术,对控制支链淀粉合成的分支酶基因进行遗传操作,在玉米和马铃薯等作物中培育出了直链淀粉高达70%以上的新品种,然而在水稻中还没有相关报导。本研究利用根癌农杆菌介导的转基因技术将Sbe1和/或Sbe3的反义或RNA干扰结构分别导入具有不同直链淀粉含量的水稻受体品种中,有效地调控了分支酶基因的表达,进而研究抑制分支酶类基因表达对稻米品质的影响,为利用基因工程技术改良稻米提供依据和材料。主要研究结果如下: 1、利用已获得的武香9915和广陵香糯来源的转反义Sbe1/Sbe3,Sbe1-RNAi和Sbe3-RNAi基因材料,对其自交后代进行分子检测,从其自交后代中分别选育到了纯合系材料。 2、对转基因纯合系成熟种子胚乳分支酶表达的Western杂交分析表明,转反义Sbe1/Sbe3纯合系成熟种子中SBE1和SBE3的表达量均明显低于未转化对照;转Sbe1-RNAi纯合系成熟种子中SBE1的表达量明显下降,SBE3的表达量也略低于未转化对照;转Sbe3 -RNAi纯合系成熟种子中SBE3的表达量明显下降,而SBE1的表达量较未转化对照有所增加。同时,我们还比较了不同转基因系中成熟种子胚乳中GBSS的表达量,结果显示,转反义Sbe1/Sbe3纯合系成熟种子中GBSS的表达量较未转化对照略有提高。 3、对选育到的纯合系材料种子中的表观直链淀粉含量进行测定,结果显示,转反义Sbe1/Sbe3和Sbe3-RNAi种子表观直链淀粉含量较未转化对照都有明显的提高,其中武香9915来源的转反义Sbe1/Sbe3基因的纯合系中,直链淀粉含量最高达到26.96%,比未转化对照提高了70.22%。广陵香糯来源的转Sbe3-RNAi基因的纯合系中,直链淀粉含量最高达到19.73%,比未转化对照提高了12.5倍。而对于转Sbe1-RNAi基因的纯合系植株,直链淀粉含量较未转化对照略有下降。 4、转基因水稻种子中内源分支酶表达受抑制和表观直链淀粉含量改变后,各类转基因纯合系种子的其它淀粉品质性状也有明显的变化。统计分析结果表明随着表观直链淀粉含量的提高,胶稠度有变硬的趋势,某些转基因纯合系的糊化温度下降,但与直链淀粉含量的相关性不高,淀粉粘滞性谱(RVA)特性也有一些变化,且淀粉粘滞性各特征值同直链淀粉含量有不同程度的相关性。 5、当转基因水稻种子中内源分支酶表达受抑制后,淀粉粒的形状、大小和结构有较明显的变化。转反义Sbe1/Sbe3和Sbe3-RNAi基因纯合系糙米的外观和粒重出现很大变化,表现为籽粒皱缩、垩白增多、粒重变小。转基因稻米中表观直链淀粉含量、粒重与籽粒厚度间呈一定的相关性。
[本文引用: 1]
.
DOI:10.1371/journal.pone.0030088URLPMID:3264551 [本文引用: 1]

Starch is one of the major components of cereals, tubers, and fruits. Genes encoding granule-bound starch synthase (GBSS), which is responsible for amylose synthesis, have been extensively studied in cereals but little is known about them in fruits. Due to their low copy gene number,GBSSgenes have been used to study plant phylogenetic and evolutionary relationships. In this study,GBSSgenes have been isolated and characterized in three fruit trees, including apple, peach, and orange. Moreover, a comprehensive evolutionary study ofGBSSgenes has also been conducted between both monocots and eudicots. Results have revealed that genomic structures ofGBSSgenes in plants are conserved, suggesting they all have evolved from a common ancestor. In addition, theGBSSgene in an ancestral angiosperm must have undergone genome duplication 65251 million years ago (MYA) to generate two families,GBSSIandGBSSII. BothGBSSIandGBSSIIare found in monocots; however,GBSSIis absent in eudicots. The ancestralGBSSIImust have undergone further divergence when monocots and eudicots split 65165 MYA. This is consistent with expression profiles ofGBSSgenes, wherein these profiles are more similar to those ofGBSSIIin eudicots than to those ofGBSSIgenes in monocots. In dicots,GBSSIImust have undergone further divergence when rosids and asterids split from each other 65126 MYA. Taken together, these findings suggest that it isGBSSIIrather thanGBSSIof monocots that have orthologous relationships withGBSSgenes of eudicots. Moreover, diversification ofGBSSgenes is mainly associated with genome-wide duplication events throughout the evolutionary course of history of monocots and eudicots.
.
DOI:10.1093/jxb/eri065URLPMID:15642712

Plants contain at least five subfamilies of starch synthases, granule bound starch synthase (GBSS) and starch synthases I, II, III, and IV (SSI, SSII, SSIII, SSIV). In this work, two members of SSIII and SSIV, respectively, were cloned and designated OsSSIII-1/-2 and OsSSIV-1/-2 in rice. Together with six other previously reported genes, the SS gene family in rice therefore is known to be duplicated and to comprise ten SS genes distributed among the five subfamilies. The starch synthase activity of each SS was confirmed by expression and enzyme activity assay in E. coli. Expression profile analysis with reverse transcription-PCR, western blotting and zymogram, indicates that OsSSIII-2 and OsSSIV-1 are mainly expressed in endosperm, while OsSSIII-1 and OsSSIV-2 are mainly expressed in the leaves. With a similar pattern of genes encoding other enzymes for starch synthesis, (such as GBSS, SSII, ADP-glucose pyrophosphorylases, and branching enzymes), it is suggested that two divergent groups of these genes should be classified in rice. Group I genes are preferentially expressed in the endosperm and function on storage starch synthesis. Group II genes are mainly expressed in leaves and some of them in the early developing endosperm, and function on transient starch synthesis in rice.
.
DOI:10.1093/molbev/msp259URLPMID:19854763 [本文引用: 1]

We present SeaView version 4, a multiplatform program designed to facilitate multiple alignment and phylogenetic tree building from molecular sequence data through the use of a graphical user interface. SeaView version 4 combines all the functions of the widely used programs SeaView (in its previous versions) and Phylo_win, and expands them by adding network access to sequence databases, alignment with arbitrary algorithm, maximum-likelihood tree building with PhyML, and display, printing, and copy-to-clipboard of rooted or unrooted, binary or multifurcating phylogenetic trees. In relation to the wide present offer of tools and algorithms for phylogenetic analyses, SeaView is especially useful for teaching and for occasional users of such software.
.
DOI:10.1080/10635150390235520URLPMID:14530136 [本文引用: 1]

Abstract The increase in the number of large data sets and the complexity of current probabilistic sequence evolution models necessitates fast and reliable phylogeny reconstruction methods. We describe a new approach, based on the maximum- likelihood principle, which clearly satisfies these requirements. The core of this method is a simple hill-climbing algorithm that adjusts tree topology and branch lengths simultaneously. This algorithm starts from an initial tree built by a fast distance-based method and modifies this tree to improve its likelihood at each iteration. Due to this simultaneous adjustment of the topology and branch lengths, only a few iterations are sufficient to reach an optimum. We used extensive and realistic computer simulations to show that the topological accuracy of this new method is at least as high as that of the existing maximum-likelihood programs and much higher than the performance of distance-based and parsimony approaches. The reduction of computing time is dramatic in comparison with other maximum-likelihood packages, while the likelihood maximization ability tends to be higher. For example, only 12 min were required on a standard personal computer to analyze a data set consisting of 500 rbcL sequences with 1,428 base pairs from plant plastids, thus reaching a speed of the same order as some popular distance-based and parsimony algorithms. This new method is implemented in the PHYML program, which is freely available on our web page: http://www.lirmm.fr/w3ifa/MAAS/.
.
DOI:10.1093/jxb/eri292URLPMID:16275672 [本文引用: 1]

A comprehensive analysis of the transcript levels of genes which encode starch-synthesis enzymes is fundamental for the assessment of the function of each enzyme and the regulatory mechanism for starch biosynthesis in source and sink organs. Using quantitative real-time RT-PCR, an examination was made of the expression profiles of 27 rice genes encoding six classes of enzymes, i.e. ADPglucose pyrophosphorylase (AGPase), starch synthase, starch branching enzyme, starch debranching enzyme, starch phosphorylase, and disproportionating enzyme in developing seeds and leaves. The modes of gene expression were tissue-and developmental stage-specific. Four patterns of expression in the seed were identified: group 1 genes, which are expressed very early in grain formation and are presumed to be involved in the construction of fundamental cell machineries, de novo synthesis of glucan primers, and initiation of starch granules; group 2 genes, which are highly expressed throughout endosperm development; group 3 genes, which have transcripts that are low at the onset but which rise steeply at the start of starch synthesis in the endosperm and are thought to play essential roles in endosperm starch synthesis; and group 4 genes, which are expressed scantly, mainly at the onset of grain development, and might be involved in synthesis of starch in the pericarp. The methodology also revealed that the defect in the cytosolic AGPase small subunit2b (AGPS2b) transcription from the AGPS2 gene in endosperm sharply enhanced the expressions of endosperm and leaf plastidial AGPS1, the endosperm cytosolic AGPase large subunit2 (AGPL2), and the leaf plastidial AGPL1.
[本文引用: 1]
[本文引用: 1]
.
DOI:10.1104/pp.122.1.255URL
[本文引用: 1]
药用野生稻叶中淀粉合成酶基因家族的序列分化和特异表达
2
2015
... 淀粉作为自然界天然生成的重要碳水化合物, 不仅为植物生长发育提供足够的营养和能量, 而且为人类和动物提供必要的食物(
... ).植物中的淀粉合成可以发生在不同的器官, 并基本可以划分为2个不同的阶段: 其一发生在光合器官(如叶), 由叶绿体通过光合作用进行首次的淀粉合成, 此为瞬时淀粉合成阶段; 但瞬时合成的淀粉在夜晚会解体成为麦芽糖和葡萄糖, 并进一步合成蔗糖, 其中一部分参与代谢, 而另一部分则运输到植物储藏器官(如根、茎、果实和种子), 由造粉体等质体重新合成淀粉, 此为储藏淀粉的合成阶段(
实时荧光定量RT-PCR内参基因的选择
1
2005
... 为定量了解2个GBSS基因在O. officinalis不同淀粉合成阶段的表达差异, 我们分别选取作为光合器官的叶和作为储存器官的种子进行定量分析, 以Actin为内参基因,
支链淀粉合成相关基因等位基因间的差异对稻米淀粉理化特性的影响
1
2006
... 两个阶段的淀粉合成均至少需要5种酶参与, 它们分别是ADP焦磷酸化酶(AGPase)、淀粉合酶(SSS)、束缚态淀粉合酶(GBSS)、淀粉分支酶(SBE)和淀粉去分支酶(SBE).其中, 束缚态淀粉合酶又被称为颗粒结合型淀粉合酶(granule-bound starch synthase), 该酶被认为与直链淀粉的合成有关(
植物颗粒结合淀粉合酶GBSS基因家族的进化
2
2017
... 淀粉合酶GBSS是直链淀粉合成中最为关键的酶, 在药用野生稻中, 我们共鉴定出2个控制直链淀粉合成的基因(GBSSI和GBSSII).作为旁系同源基因, 这2个基因彼此之间并不十分相似, 其CDS序列存在36.2%的核苷酸差异.但是, 对于物种间的GBSS直系同源基因, 其相似性却与选取的物种密切相关, 其亲缘关系较近的物种相似性较高(
... 当然, 已有研究表明, GBSSI和GBSSII分化表达的现象不仅局限于稻属.例如, Vrinten和Nakamura (2000)的研究证明, 小麦GBSSI在胚乳中高表达, 而GBSSII仅在叶、杆和果皮中表达.但是, 在非禾本科植物中, GBSS基因却承担了禾本科植物中2类GBSS的全部功能.例如, 在马铃薯(Solanum tuberosum)中, GBSS基因是单拷贝, 它在植物光合和储藏器官均有表达, 但在块茎中的表达水平明显高于叶(
几个重复序列在不同稻种中的分布及其与稻种分化关系的研究
1
2003
... 药用野生稻(O. officinalis)是我国自然分布的3种野生稻之一.与栽培稻的AA基因组不同, 药用野生稻具有CC基因组(
抑制淀粉分支酶类基因表达对稻米品质影响的研究
1
2008
... 两个阶段的淀粉合成均至少需要5种酶参与, 它们分别是ADP焦磷酸化酶(AGPase)、淀粉合酶(SSS)、束缚态淀粉合酶(GBSS)、淀粉分支酶(SBE)和淀粉去分支酶(SBE).其中, 束缚态淀粉合酶又被称为颗粒结合型淀粉合酶(granule-bound starch synthase), 该酶被认为与直链淀粉的合成有关(
De novo assembly and characterization of
1
2015
... 以栽培稻GBSSI (GenBank No.FJ750947.1)和GBSSII (GenBank No.AY069940.1)为参考序列, 利用本实验室早期的药用野生稻叶转录组数据(
Diversification of genes encoding granulebound starch synthase in monocots and dicots is marked by multiple genome-wide duplication events
1
2012
... 当然, 已有研究表明, GBSSI和GBSSII分化表达的现象不仅局限于稻属.例如, Vrinten和Nakamura (2000)的研究证明, 小麦GBSSI在胚乳中高表达, 而GBSSII仅在叶、杆和果皮中表达.但是, 在非禾本科植物中, GBSS基因却承担了禾本科植物中2类GBSS的全部功能.例如, 在马铃薯(Solanum tuberosum)中, GBSS基因是单拷贝, 它在植物光合和储藏器官均有表达, 但在块茎中的表达水平明显高于叶(
Evolution and expression analysis of starch synthase III and IV in rice
0
2005
SeaView version 4: a multiplatform graphical user interface for sequence alignment and phylogenetic tree building
1
2010
... 为深入了解药用野生稻GBSS基因和其它植物在DNA序列水平上的遗传差异并探究该基因在植物中的分子进化趋势, 我们利用NCBI (https://www.ncbi. nlm.nih.gov/)和Uniprot (https://www.uniprot.org/)数据库, 结合注释查询和以目标基因为搜索项的BLAST搜寻(阈值设为1e-5), 对具有全基因组数据的18种植物(
A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood
1
2003
... 为深入了解药用野生稻GBSS基因和其它植物在DNA序列水平上的遗传差异并探究该基因在植物中的分子进化趋势, 我们利用NCBI (https://www.ncbi. nlm.nih.gov/)和Uniprot (https://www.uniprot.org/)数据库, 结合注释查询和以目标基因为搜索项的BLAST搜寻(阈值设为1e-5), 对具有全基因组数据的18种植物(
Expression profiling of genes involved in starch synthesis in sink and source organs of rice
1
2005
... 从基因表达水平来看, 目前的研究表明, 药用野生稻GBSSI与GBSSII在叶和种子中具有非常不一致的表达模式.在叶中, GBSSII占据表达优势, 但在种子中, GBSSI出现强势表达.这种表达方式与栽培稻中2个同源基因的表达方式非常类似, 但在栽培稻中, GBSSI完全在种子中特异表达(
Molecular identification and isolation of the
1
1983
... 两个阶段的淀粉合成均至少需要5种酶参与, 它们分别是ADP焦磷酸化酶(AGPase)、淀粉合酶(SSS)、束缚态淀粉合酶(GBSS)、淀粉分支酶(SBE)和淀粉去分支酶(SBE).其中, 束缚态淀粉合酶又被称为颗粒结合型淀粉合酶(granule-bound starch synthase), 该酶被认为与直链淀粉的合成有关(
Genome-wide analysis of starch metabolism genes in potato (
1
2017
... 当然, 已有研究表明, GBSSI和GBSSII分化表达的现象不仅局限于稻属.例如, Vrinten和Nakamura (2000)的研究证明, 小麦GBSSI在胚乳中高表达, 而GBSSII仅在叶、杆和果皮中表达.但是, 在非禾本科植物中, GBSS基因却承担了禾本科植物中2类GBSS的全部功能.例如, 在马铃薯(Solanum tuberosum)中, GBSS基因是单拷贝, 它在植物光合和储藏器官均有表达, 但在块茎中的表达水平明显高于叶(
Wheat granule-bound starch synthase I and II are encoded by separate genes that are expressed in different tissues
0
2000
Nucleotide sequence of rice
1
1990
... 两个阶段的淀粉合成均至少需要5种酶参与, 它们分别是ADP焦磷酸化酶(AGPase)、淀粉合酶(SSS)、束缚态淀粉合酶(GBSS)、淀粉分支酶(SBE)和淀粉去分支酶(SBE).其中, 束缚态淀粉合酶又被称为颗粒结合型淀粉合酶(granule-bound starch synthase), 该酶被认为与直链淀粉的合成有关(
备案号: 京ICP备16067583号-21
版权所有 © 2021 《植物学报》编辑部
地址:北京香山南辛村20号 邮编:100093
电话:010-62836135 010-62836131 E-mail:cbb@ibcas.ac.cn
本系统由北京玛格泰克科技发展有限公司设计开发
