0 引言
【研究意义】MicroRNA(miRNA)是一类内源性小分子非编码的单链RNA,其长度范围在19—25 nt。植物中的miRNA高度保守,在转录水平通过碱基互补配对的方式识别并调控靶基因的表达,进而参与植物抗逆性、生长发育、新陈代谢等生物学过程[1,2]。动植物中miRNA表达受病毒感染调控,miRNA表达能够引起靶基因及其后续信号途径、代谢和免疫应答变化,引导细胞逃避或者抵抗病毒感染带来的损伤[3,4,5],另外,病毒感染可导致农作物产量和品质的下降[6,7],因此,鉴定植物中由病毒感染引起的miRNA表达差异,能够促进对植物抗病毒感染机制的深入了解。【前人研究进展】高通量测序技术的发展推动miRNA的挖掘分析,目前为止在植物中被鉴定出来miRNA已包含40多个miRNA家族。刘辛[8]报道了黄瓜花叶病毒(Cucumber mosaic virus,CMV)和番茄不孕病毒感染番茄植株后,对miRNA及其靶基因的表达有不同程度的影响;陈莎[9]采用深度测序方法挖掘并鉴定云南和贵州省玉米病毒的种类;王园龙等[10]利用高通量测序技术鉴定白花泡桐中与盐胁迫相关的差异miRNA,发现33个已知miRNA和80个新的miRNA表达具有差异性;陈洁[11]利用Solexa深度测序分析获得玉米根系中与重金属Pb胁迫相关的92个已知的miRNA和378个新的miRNA及其靶基因;马骢毓[12]运用高通量测序方法及生物信息学分析手段,鉴定出与马铃薯抗旱相关的89个已知miRNA和239个未知miRNA及其靶基因,因此,利用高通量测序是一种筛选与抗病毒相关miRNA的高效技术手段[13]。【本研究切入点】甘薯褪绿矮化病毒(Sweet potato chlorotic stunt virus,SPCSV)、甘薯羽状斑驳病毒(Sweet potato feathery mottle virus,SPFMV)、甘薯曲叶病毒(Sweet potato leaf curl virus,SPLCV)等10多种病毒是甘薯(Ipomoea batatas)中常见的感染病原[14],这些病毒严重影响了甘薯的性状、产量以及品质。但目前为止,尚未有关于甘薯病毒相关的miRNA鉴定和分析的报道。【拟解决的关键问题】通过比较被病毒感染的甘薯样品之间的差异表达miRNA,鉴定不同病毒感染引起的差异表达miRNA及其相关应答机制,明确与甘薯病毒感染相关的miRNA,为miRNA参与感染抗病毒胁迫的机制研究打下基础。1 材料与方法
试验于2016年4月至2017年10月在福建省农业科学院作物研究所薯类中心试验室进行。1.1 材料
甘薯品种龙薯9号材料均于2017年5月采自福建泉州地区,时间为植株自然发病。4个材料的病理症状包含了畸形黄化、疱疹、褪绿矮化、曲叶等症状(图1)。试验验证时,采用脱毒苗作为无病毒对照样本。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1甘薯病毒感染后不同症状
-->Fig. 1Different symptoms after sweet potato virus infection
-->
1.2 方法
1.2.1 RNA抽提、Illumina文库构建及测序 于甘薯生长期采样,每个症状的植株采样3株,收集等量叶片后混合,采用UNIQ-10柱式总RNA抽提试剂盒(上海生工)抽提叶片RNA。总RNA的质量检测由NanoDrop 2000超微量分光光度计(上海美普达仪器有限公司)和Agilent 2100生物分析仪(Agilent,美国)进行。miRNA测序文库的构建按照Illumina公司TruSeqTM Small RNA Sample Prep Kits(Illumina,美国)试剂盒提供的标准流程进行。采用Illumina Hiseq 2500平台进行small RNA测序,读长为单端1×50 bp。1.2.2 数据质控及分析 获得clean small RNA数据后,对其长度、种类及丰度分析,运用Blast软件(http://blast.ncbi.nlm.nih.gov/)与Rfam11数据库(http://Rfam.sanger.ac.uk/),过滤、剔除非small RNA序列(mRNA、rRNA、tRNA、snRNA和snoRNA等)。最后运用Bowtie 软件(http://bowtie-bio.sourceforge. net/index.shtml)分析small RNA序列在基因组上的分布情况。通过与甘薯病毒库(http://www.ncbi.nlm.nih. gov/genome/viruses)比对,鉴定测序数据中的与病毒种类。
1.2.3 已知miRNA的鉴定 将small RNA序列与甘薯参考基因组中miRNA前体及成熟体序列进行比对,进行已知miRNA的鉴定,运用RNAfold软件(http://www.tbi.univie.ac.at/RNA/)和Mireap 软件(http://mireap.sourceforge.net/)预测和分析miRNA二级结构。
1.2.4 新miRNA的鉴定 将未注释的small RNA序列数据运用Mireap软件(http://mireap.sourceforge.net/)进行序列二级结构分析和新miRNA的预测,新miRNA预测标准参考MEYERS等描述,参考预测结果结合Dicer酶切位点信息、能量值等特征预测、鉴定新miRNA[10,15]:1、miRNA前体具有标志性发夹结构;2、序列长度在18—32 nt;3、最低自由能(MFE)≤-75 kJ·mol-1,且对称度为7 nt,最长距离100 nt,碱基配对数10 nt。
1.2.5 差异表达miRNA鉴定及靶基因预测 根据每个miRNA分别在样本文库中的相对表达丰度(TPTM),运用DEGseq和DEseq R语言包中Expdiff方法选择差异表达miRNA及其显著性,差异表达miRNA的鉴定标准为│log2 (TPTM1/TPTM2 ratio)│≥1且P-value≤0.05。运用TargetFinder对差异表达miRNA靶基因预测[16]。
1.2.6 差异表达miRNA靶基因GO和KEGG pathway富集分析 利用Goatools (https://github.com/tanghaibao/ GOatools)软件进行GO分析,将miRNA靶基因按照生物学过程、构成细胞的组分,实现的分子功能等进行分类,Fisher精确检验,多重检验方法(Bonferroni, Holm, Sidak和false discovery rate,BH(FDR))对P值进行校正,校正后P值(P-FDR/ corrected P-value)≤0.05视为显著富集的GO功能。采用KOBAS(http://kobas.cbi.pku.edu. cn/home.do)进行差异表达miRNA靶基因KEGG pathway富集分析,满足校正后P值(corrected P-value)≤0.05的KEGG通路视为显著富集的KEGG通路。
1.2.7 差异表达miRNA的实时荧光定量PCR检测 miRNA的相对表达量检测采用stem-loop方法进行检测,设计miRNA的stem-loop引物、miR通用反向引物、PCR的正向引物(表1),用TaKaRa公司的荧光定量PCR试剂盒进行检测,使用ABI PRISM 7500实时荧光定量PCR系统进行扩增。扩增条件:95℃预变性5 min,然后95℃ 15 s,65℃ 45 s,40个循环,65℃保存。以actin作为内参,miRNA的相对表达量以2-∆∆Ct公式进行计算。
1.2.8 半定量PCR及一代测序检测 半定量PCR检测病毒的表达,病毒相关序列及引物名称如表1,扩增体系为20 μL(含10 μL扩增混合物),扩增产物经1.2%琼脂糖凝胶电泳检测,回收阳性条带后进行一代测序,对所得序列与NCBI数据库进行比对分析。
Table 1
表1
表1实时定量PCR引物序列
Table 1The primers for real-time PCR
| miRNA/病毒名称 miRNA/virus | 引物序列Primer sequence (5′-3′) |
|---|---|
| Actin正向引物 Actin forward primer | ACTCAGTGGCGGGACTAC |
| Actin反向引物Actin reverse primer | CTGTGAACAATTGACGGACC |
| miRNA通用反向引物 miRNA universal reverse primer | GTGCAGGGTCCGAGGTATTC |
| miR 156序列miR 156 sequence | TGACAGAAGAGAGTGAGCAC |
| miR 156-5p逆转录引物miR 156-5p reverse transcription primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACGTGCTC |
| miR 156-5p正向引物miR 156-5p forward primer | GCGCGTGACAGAAGAGAGT |
| N-miR-40序列N-miR-40 sequence | TAATCTGCATCCTGAGGTTTA |
| N-miR-40逆转录引物N-miR-40 reverse transcription primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTAAACC |
| N-miR-40正向引物N-miR-40 forward primer | GCGCTAATCTGCATCCTGA |
| miR-319m序列miR-319m sequence | TTGGACTGAAGGGAGCTCCCT |
| miR-319m逆转录引物 miR-319m reverse transcription primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACAGGGAG |
| miR-319m正向引物miR-319m forward primer | GCGTTGGACTGAAGGGAG |
| miR-160a序列miR-160a sequence | TGCCTGGCTCCCTGTATGCCA |
| miR-160a逆转录引物miR-160a reverse transcription primer | GCGTGCCTGGCTCCCT |
| miR-160a正向引物miR-160a forward primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTGGCAT |
| miR-2096序列miR-2096 sequence | TGCCGATTTCCCCCTCGGGCG |
| miR-2096逆转录引物miR-2096 reverse transcription primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACCGCCCG |
| miR-2096正向引物miR-2096 forward primer | CGTGCCGATTTCCCCCT |
| miR-387b序列miR-387b sequence | CGTGGCTCTGACCGGTGCTAAAGG |
| miR-387b逆转录引物miR-387b reverse transcription primer | GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACCCTTTA |
| miR-387b正向引物miR-387b forward primer | CGTGGCTCTGACCGGTGC |
| SPFMV正向引物SPFMV forward primer | AAGGAGGAATACGAAGGACTGC |
| SPFMV反向引物SPFMV reverse primer | ACATAAGTCTGTTGCTCTGTCGC (NCBI: 25013775) |
| SPVC正向引物SPVC forward primer | ACGCTTCCTGCGAACGATA |
| SPVC反向引物SPVC reverse primer | GCATTCTGAGTGTAGTTTGAACCATA (NCBI: 313139318) |
新窗口打开
2 结果
2.1 样本症状、测序数据质控及比对分析
如图1所示,4个样本之间的症状各有不同,分别表现为典型的畸形黄化、疱疹、褪绿矮化以及曲叶症状。4个样本所得测序数据clean reads Q20%值>98%,Q30%值>95%(表2)。对small RNA的长度分析显示3个文库中small RNA的长度在21、22和24 nt的丰度较高(图2),而Fj01样本的small RNA长度在18—21 nt间分布较多,各样本之间small RNA的长度峰值稍有不同。Table 2
表2
表2Illumina测序数据统计表
Table 2Summary of the Illumina sequencing results
| 样品 Sample | 原始数据Raw data | 净数据Clean data | Q20 (%) | Q30 (%) | ||
|---|---|---|---|---|---|---|
| 总reads数 Total reads | 总碱基数 Total bases | 总reads数 Total reads | 总碱基数 Total bases | |||
| Fj01 | 15107984 | 755399200 | 15040168 | 749810497 | 99.06 | 97.88 |
| Fj02 | 16439476 | 821973800 | 16350685 | 814862130 | 98.98 | 97.84 |
| Fj03 | 11756965 | 587848250 | 11558880 | 573793032 | 98.14 | 95.54 |
| 1H | 14471291 | 723564550 | 14404124 | 718156094 | 99.21 | 98.44 |
新窗口打开
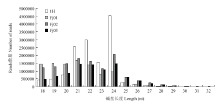 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2Small RNA的长度分布
横坐标表示small RNA的reads长度,纵坐标表示该reads长度下的频数
-->Fig. 2Distribution of small RNA length
The transverse coordinates represent the reads length of the small RNA, and the longitudinal coordinates represent the frequencies at the length of the reads
-->
4个文库数据中非small RNA序列占比在0.31%—1.32%,比对到参考基因组的比率为70.59%—92.34%,表明本次测序所得的数据质量较高(表3)。
Table 3
表3
表3Small RNA注释及参考基因组比对率
Table 3Annotation of small RNA and mapped rate to reference genome
| 样品 Sample | 小RNA Small RNA (%) | 非小RNA Non-small RNA (%) | 总RNA reads Total small RNA | Unique序列 Unique small RNA | 基因组匹配序列 Mapped small RNA | 比对率 Mapped rate (%) |
|---|---|---|---|---|---|---|
| Fj01 | 99.40 | 0.60 | 7992287 | 338723 | 7380053 | 92.34 |
| Fj02 | 98.68 | 1.32 | 9685110 | 440137 | 8329797 | 86.01 |
| Fj03 | 99.13 | 0.87 | 6601808 | 414924 | 6093029 | 92.29 |
| 1H | 99.69 | 0.31 | 7566559 | 1102719 | 5340903 | 70.59 |
新窗口打开
2.2 病毒库比对结果分析
将所得的small RNA reads与甘薯病毒库进行比对(BLASTn),4个样本与病毒库匹配度为0.0002%—0.0048%(附表1),除1H外,均感染多种甘薯病毒,包括甘薯常见病毒甘薯G病毒(Sweet potato virus G,SPVG)、甘薯病毒2号(Sweet potato virus 2,SPV2)、甘薯C病毒、甘薯褪绿矮化病毒、甘薯羽状斑驳病毒、甘薯潜隐病毒(Sweet potato latent virus,SPLV)等。另外还有落葵皱纹花叶病毒(Basella rugose mosaic virus,BaRMV)、豇豆蚜传花叶病毒(Cowpea aphid-borne mosaic virus,CAMV)、葱花叶病毒(Scallion mosaic virus,ScaMV)、人参Y病毒(Panax virus Y,PanVY)等非甘薯常见病毒。Table S1
附表1
附表1Small RNA中与甘薯病毒库比对结果
Table S1Alignment of small RNA reads to sweet potato virus database
| 样品编号 Sample number | 总RNA reads Total small RNA | 病毒序列 Reads aligned to virus | 百分比 Percent (%) | 常见甘薯病毒类型 Common viruses in sweet potato | 其他病毒类型 Other viruses | 噬菌体(细菌病毒) Bacteriophage |
|---|---|---|---|---|---|---|
| 不同地域(地域组)Different regions (geographical groups) | ||||||
| PTQZ | 10007500 | 1086 | 0.0108 | BaRMV, CAMV, KoMV, HarMV, PepMV, SCBMOV, SCBV, SPBVB, SPCSV, SPFMV, SPGVaV, SPLCV, SPLV, SPV2, SPVC, SPVG, SrMV, TYLCV | AHEACV2, AMoV, ATV, BHV-6, BnYDV, CoYV, DOVA, NDV, NSV, OrMV, PandoraV, PanVY, PenMV, PMV, PPV, PvEV-1, SiGMCRV, SPLCGxV, SPLCShV, SYSV, WMV | Acinetobacter phage Acj61, Bacillus phage phBC6A51, Citrobacter phage CR44b, Mycobacterium phage Redi/Nigel, Rhizobium phage vB_RleM_P10VF |
| 4 | 9477333 | 586 | 0.0062 | BaRMV, CAMV, HarMV, PepMV, ScaMV, PVV, SCBMOV, SCBV, SPBVA, SPBVB, SPCSV, SPFMV, SPGVaV, SPLV, SPV2, SPVC, SPVG, SrMV | ATV, BCMV, BnYDV, BHV-6, BYMV, CoYV, DOVA, LYSV, OrMV, PanVY, PandoraV, PenMV, PMaV, PSLDV, RLV, SiGMCRV, SPLCBeV, SPLCGxV, SPLCHnV, SPLCLaV, SPLCShV, SPLCSPV, TEV, WMV | Acinetobacter phage Acj61, Aeromonas phage pAh6-C, Bacillus phage phBC6A51, Citrobacter phage CR44b, Enterobacter phage IME11, Mycobacterium phage Redi/Nigel, Pseudomonas phage OBP, Rhizobium phage vB_RleM_P10VF, Prochlorococcus phage, Synechococcus phage |
| 2H | 13790960 | 1286 | 0.0093 | BaRMV, CAMV, HarMV, PepMV, PepVMV, ScaMV, SCBMOV, SCBV, SPBVA, SPBVB, SPCSV, SPFMV, SPGVaV, SPLV, SPV2, SPVC, SPVG, SrMV | AHEACV2, ATCV-1, AMoV, ATV, BPMV, BnYDV, BHV-6, CoYV, DOVA, MV, FreMV, SYSV, OrMV, PanVY, RLV, PaLCuGuV,PandoraV, PenMV, PPV, PSLDV, SiGMCRV, SLCMV-[Col], SPLCBeV, SPLCHnV, SPLCLaV, SPLCSPV, TCV, TEV, WMV | Acinetobacter phage Acj61, Bacillus phage phBC6A51, Citrobacter phage CR44b, Enterobacter phage IME11, Loktanella phage pCB2051-A, Mycobacterium phage Redi/Nigel, Pseudomonas phage OBP/UFV-P2, Synechococcus phage |
| 3H | 12635959 | 1245 | 0.0098 | BaRMV, CAMV, HarMV, PepMV, PepVMV, ScaMV, SCBMOV, SPBVA, SPBVB, SPFMV, SCBV, SPCSV, SPLV, SPV2, SPVC, SPVG, SrMV | AHEACV2, ATCV-1, AMoV, ATV, BHV-6, BnYDV, BPMV, DOVA, FreMV, MV, OrMV, PandoraV, PanVY, PenMV, PPV, RLV, SYSV, TCV, TEV | Acinetobacter phage Acj61, Bacillus phage, Citrobacter phage CR44b, Lactobacillus phage LBR48, Loktanella phage pCB2051-A, Mycobacterium phage Redi/Nigel/PMC, Pseudomonas phage LIT1/OBP/UFV-P2 |
| 不同性状(性状组)Different traits (trait groups) | ||||||
| Fj01 | 7992287 | 235 | 0.0029 | BaRMV, CAMV, HarMV, PepMV, PepVMV, ScaMV, SCBMOV, SCBV, SPBVB, SPCSV, SPGVaV, SPFMV, SPLV, SPV2, SPVC, SPVG, SrMV | AHEACV2, BHV-6, BPMV, DOVA, MV, PandoraV, PanVY, PenMV, PLV, PMV, SPLCShV | Citrobacter phage CR44b, Enterobacteria phage phiX174 sensu lato, Mycobacterium phage Redi/PMC, Synechococcus phage ACG-2014h, Xanthomonas phage phiL7 |
| Fj02 | 9685110 | 362 | 0.0038 | BaRMV, CAMV, HarMV, PepMV, PepVMV, PVV, ScaMV, SCBV, SPBVB, SPCFV, SPFMV, SPGVaV, SPLV, SPV2, SPVC, SPVG, SrMV | AHEACV2, BHV-6, BYMV, ChocGV, DOVA, LYSV, MpV12T, MV, PandoraV, PanVY, PenMV, PLV, PPV, TEV, YMMV | Acinetobacter phage Acj61, Bacillus phage phBC6A51/G, Cyanophage NATL1A-7/S-TIM5, Enterobacter phage IME11, Mycobacterium phage Redi/PMC, Synechococcus phage ACG-2014b/h/S-SM2/syn9/S-SSM4, Pseudomonas phage LIT1, Prochlorococcus phage P-SSM2/3, Xanthomonas phage phiL7 |
| 样品编号 Sample number | 总RNA reads Total small RNA | 病毒序列 Reads aligned to virus | 百分比 Percent (%) | 常见甘薯病毒类型 Common viruses in sweet potato | 其他病毒类型 Other viruses | 噬菌体(细菌病毒) Bacteriophage |
| Fj03 | 6601808 | 316 | 0.0048 | BaRMV, CAMV, HarMV, PepMV, ScaMV, SCBV, SCBMOV, SPBVB, SPCSV, SPFMV, SPGVaV, SPLV, SPV2, SPVC, SPVG | AHEACV2, BCMV, BHV-6, ClYVV, CoYV, DOVA, MV, PandoraV, PanVY, PenMV, SiGMCR, SiYMA, SPLCLaV, SPLCBeV, SPLCShV | Aeromonas phage 25/65/pAh6-C, Cyanophage NATL1A-7/S-TIM5/P-TIM40, Enterobacter phage IME11, Mycobacterium phage Redi/Nigel, Synechococcus phage ACG-2014b/h/S-SM2/S-SSM4/S-SSM7, Prochlorococcus phage P-SSM2/P-TIM68 |
| 1H | 7566559 | 16 | 0.0002 | SHAV-N/NSs, ChocGV | Enterobacteria phage phiX174 sensu lato | |
新窗口打开
1H样本除西部云杉色卷蛾颗粒体病毒(ChocGV)、沙门达病毒N/NSs(SHAV-N/NSs)和肠杆菌噬菌体病毒(Enterobacteria phage virus)外并无其他病毒感染。而其他3个样本之间的病毒感染相差不大,但种类特殊,如Fj02样本(疱疹)特异性感染了山药温和花叶病毒(Yam mild mosaic virus,YMMV)、马铃薯潜隐病毒(Potato latent virus,PLV)等,而Fj03样本中甘薯双生病毒种类较多,如甘薯上海曲叶病毒(Sweet potato leaf curl Shanghai virus,SPLCShV)和甘薯孟加拉曲叶病毒(Sweet potato leaf curl Bengal virus,SPLCBeV)。
采用半定量PCR检测了SPFMV和SPVC两个病毒在症状样本及无症状(脱毒苗,对照)样本中表达情况,发现SPFMV和SPVC两个病毒在对照样本中无表达,在4个症状样本中具有不同程度的表达(图3),一代测序结果与NCBI数据库比对结果显示,Fj01-03样本中均检测到了SPFMV和SPVC病毒序列,但1H样本序列与NCBI数据库不匹配,与二代测序结果基本一致。
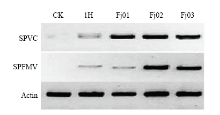 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3PCR验证甘薯病毒在样本间的表达
CK:对照样本,为无病毒感染的脱毒苗。图6同
-->Fig. 3Expression of sweet potato virus by PCR analysis
Control sample of virus-free seedling. The same as Fig. 6
-->
2.3 已知miRNA和新miRNA的鉴定
通过与miRBase20数据库(http://www.mirbase. org/)中植物miRNA前体序列比对,共鉴定到679个已知miRNAs,其中包括常见的miRNA家族成员,如miR156、miR166、miR169等家族。另外运用Mireap软件鉴定到1 004个新的miRNA。2.4 差异表达miRNA筛选及其靶基因预测
通过对不同症状样本数据的比较分析,共筛选到288个已知的差异miRNA和433个新的差异miRNA(图4、表4)。这288已知差异miRNA包括miR156、miR157、miR166、miR172等家族的全部或者部分成员,并预测到417个靶基因;433个新的差异miRNA有1 856个靶基因。这些靶基因多为转录因子家族成员,其编码蛋白多含有ZFP、bHLH、SPL、lectin和homeobox-leucine zipper protein等功能区域。通过对已知的差异miRNA进行比较,发现miRNA家族部分或者全体均为差异miRNA,如miR156家族、miR157家族全体成员等在样本之间表达均有差异,部分miRNA的成员,如miR166d/e/t,miR169i/o/u、miR395b/c/d等在不同症状样本之间表达也具有差异,而且这些差异miRNA在马铃薯、拟南芥、水稻、大豆、番茄中的保守性比较好。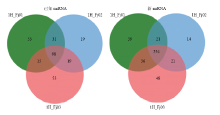 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4差异表达miRNA的Venn图
-->Fig. 4Venn graphics of differentially expressed miRNA
-->
Table 4
表4
表4样本间的差异表达miRNA
Table 4Differentially expressed miRNA between samples
| 已知miRNA Known miRNA | 全部 All | 上调 Up | 下调 Down | 新miRNA Novel miRNA | 全部 All | 上调 Up | 下调 Down | |
|---|---|---|---|---|---|---|---|---|
| 1H.vs.Fj01 | 197 | 157 | 40 | 1H.vs.Fj01 | 350 | 106 | 244 | |
| 1H.vs.Fj02 | 167 | 113 | 54 | 1H.vs.Fj02 | 310 | 62 | 248 | |
| 1H.vs.Fj03 | 185 | 134 | 51 | 1H.vs.Fj03 | 359 | 97 | 262 | |
| Fj01.vs.Fj02 | 66 | 11 | 55 | Fj01.vs.Fj02 | 62 | 12 | 50 | |
| Fj01.vs.Fj03 | 107 | 46 | 61 | Fj01.vs.Fj03 | 72 | 36 | 36 | |
| Fj02.vs.Fj03 | 82 | 52 | 30 | Fj02.vs.Fj03 | 78 | 54 | 24 |
新窗口打开
通过stem-loop PCR,检测了已知差异miRNA(miR-156和miR-319m)和新的差异miRNA(novel-miR-40)的表达,结果显示这3个miRNA的表达模式与sRNA-seq分析结果相似(图5),这表明测序数据真实度较高。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5差异表达miRNA的PCR验证与RNA-seq分析
-->Fig. 5Comparative analysis of the expression profiles of the differentially expressed miRNAs by PCR and RNA-seq
-->
2.5 病毒相关miRNA的差异表达
为了分析不同症状样本之间的差异miRNA,对4个样本进行配对比较分析,与1H样本比较,Fj01、02和03样本中的miR-156和miR-157表达量分别显著下降,miR-156家族的成员miR-156a/d/e/g/o和miR-157家族成员miR-157c在Fj01、02和03样本中的表达量均显著高于1H样本,而miR-156f/k和miR-157b/d/r的表达量在1H样本中显著高于其他样本,对Fj01、02和03这3个样本之间两两比较发现,miR-156a/d/g/k在Fj01或Fj02中的表达量最高,Fj03中最低,miR-156e在Fj02中表达量最高,Fj01中次之,Fj03中最低,同样miR-157家族成员在3个样本中分别具有不同程度的上调或下调趋势,表明病毒感染能够引起miR-156和miR-157的差异表达。miR-159、miR-166、miR-167、miR-169、miR-319、miR-395、miR-396、miR-2630等家族的成员在4个样本之间的表达量也存在显著差异。为了检测miRNA表达与病毒感染的相关性,运用实时定量PCR方法检测随机选择的3个miRNA(miR-160a、miR-2096和miR-5387b)在4个症状样本及对照(无病毒感染)样本中的表达情况,结果表明,与对照样本相比,这几个miRNA在1H和Fj01-03几个样本中表达量有不同程度的上调(图6),与上述分析结果基本一致,这表明病毒感染能够引起miRNA的差异表达,但由于田间感染病毒类型的多样性和症状的复杂性,病毒种类与差异miRNA的相关性需要进一步的分析和验证。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6PCR验证差异表达miRNA在症状样本和对照之间的表达情况
-->Fig. 6The relative expression level of 3 miRNAs in sequenced data and control samples by PCR
-->
2.6 差异表达miRNA的GO和KEGG pathway富集分析
2.6.1 已知的差异miRNA靶基因富集分析 通过对差异表达miRNA靶基因的GO富集分析,已知的差异miRNA靶基因的富集到19个显著富集的GO功能团(P-FDR≤0.05),表明这些差异miRNA的靶基因均与植物的生长发育、细胞分化、形态形成等相关(图7)。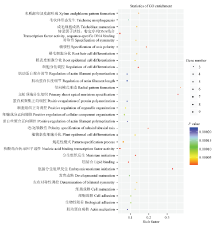 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7已知差异表达miRNA靶基因的前30个GO富集分析结果
横轴表示富集因子,即富集到某个GO term的差异基因个数占测序得到的背景基因个数的比值;纵坐标表示该GO term富集到的功能:圆圈越大代表富集到此功能的差异基因个数相对越多。从蓝色到红色的色谱代表未修正的P-value
-->Fig. 7The top 30 GO enrichment of known differentially expressed miRNA targets
X-axis indicates the ratio of genes in corresponding terms to total reference gene number. Y-axis represents the GO terms. Cycle size indicates number of genes involved in the corresponding term. Colored column (right) notes the P value
-->
KEGG pathway富集结果显示已知的差异miRNA靶基因没有显著富集的pathways(corrected P-value<0.05)。因此仅对前10个pathways分析,发现已知的差异miRNA靶基因与植物“病原加工与呈递”(ko04612)、“内质网蛋白加工”(ko04141)以及MAPK、mTOR等信号通路有关(附表2),如叶绿体热激蛋白(chloroplast heat shock protein 70-2,cpHSP70-2)。
Table S2
附表2
附表2差异表达miRNA靶基因的KEGG pathway富集分析
Table S2KEGG pathway enrichment analysis for target of differentially expressed miRNAs
| 不同地域之间已知差异表达miRNA Known miRNA within 2H/3H/4/PTQZ comparison analysis | ||||
|---|---|---|---|---|
| #Term | ID | Input number | P-Value | Corrected P-Value |
| Central carbon metabolism in cancer | ko05230 | 4 | 0.001687826 | 0.06930757 |
| Viral carcinogenesis | ko05203 | 5 | 0.002235728 | 0.06930757 |
| Selenocompound metabolism | ko00450 | 2 | 0.008480408 | 0.13838769 |
| Glycolysis / Gluconeogenesis | ko00010 | 4 | 0.01175177 | 0.13838769 |
| Notch signaling pathway | ko04330 | 2 | 0.012630203 | 0.13838769 |
| Sulfur metabolism | ko00920 | 2 | 0.013392357 | 0.13838769 |
| Purine metabolism | ko00230 | 4 | 0.018586957 | 0.147520886 |
| Biosynthesis of amino acids | ko01230 | 5 | 0.019034953 | 0.147520886 |
| Chronic myeloid leukemia | ko05220 | 2 | 0.026014451 | 0.179210664 |
| Transcriptional misregulation in cancer | ko05202 | 2 | 0.037051692 | 0.222301996 |
| 不同性状之间已知差异表达miRNA Known miRNA within 1H/Fj01/Fj02/Fj03 comparison analysis | ||||
| #Term | ID | Input number | P-Value | Corrected P-Value |
| Antigen processing and presentation | ko04612 | 4 | 0.000965935 | 0.108185 |
| Protein processing in endoplasmic reticulum | ko04141 | 8 | 0.005613585 | 0.314361 |
| Spliceosome | ko03040 | 6 | 0.019368052 | 0.402325 |
| MAPK signaling pathway | ko04010 | 3 | 0.020848151 | 0.402325 |
| Adipocytokine signaling pathway | ko04920 | 3 | 0.021849719 | 0.402325 |
| Insulin signaling pathway | ko04910 | 5 | 0.024195509 | 0.402325 |
| Estrogen signaling pathway | ko04915 | 3 | 0.027821282 | 0.402325 |
| Selenocompound metabolism | ko00450 | 2 | 0.031081592 | 0.402325 |
| Legionellosis | ko05134 | 3 | 0.039269254 | 0.402325 |
| mTOR signaling pathway | ko04150 | 3 | 0.045012535 | 0.402325 |
| 不同地域之间新的差异表达miRNA Novel miRNA within 2H/3H/4/PTQZ comparison analysis | ||||
| #Term | ID | Input number | P-Value | Corrected P-Value |
| Plant-pathogen interaction | ko04626 | 35 | 9.57E-05 | 0.023920418 |
| Linoleic acid metabolism | ko00591 | 8 | 0.000459895 | 0.057486835 |
| Basal transcription factors | ko03022 | 9 | 0.001475081 | 0.122923436 |
| Calcium signaling pathway | ko04020 | 8 | 0.002552465 | 0.129892978 |
| Protein export | ko03060 | 9 | 0.00259786 | 0.129892978 |
| Pathogenic Escherichia coli infection | ko05130 | 6 | 0.003891975 | 0.162165639 |
| AMPK signaling pathway | ko04152 | 16 | 0.007511927 | 0.249017589 |
| Transcriptional misregulation in cancer | ko05202 | 9 | 0.007968563 | 0.249017589 |
| Ribosome biogenesis in eukaryotes | ko03008 | 17 | 0.012774227 | 0.34213434 |
| Monoterpenoid biosynthesis | ko00902 | 5 | 0.013731709 | 0.34213434 |
| 不同性状之间新的差异表达miRNA Novel miRNA within 1H/Fj01/Fj02/Fj03 comparison analysis | ||||
| #Term | ID | Input number | P-Value | Corrected P-Value |
| Synaptic vesicle cycle | ko04721 | 7 | 0.001522748 | 0.213649375 |
| Endocrine and other factor-regulated calcium reabsorption | ko04961 | 5 | 0.003870463 | 0.213649375 |
| Plant-pathogen interaction | ko04626 | 14 | 0.004058889 | 0.213649375 |
| Notch signaling pathway | ko04330 | 4 | 0.00505679 | 0.213649375 |
| Basal transcription factors | ko03022 | 4 | 0.014223099 | 0.37856107 |
| Phosphatidylinositol signaling system | ko04070 | 5 | 0.014283309 | 0.37856107 |
| Chronic myeloid leukemia | ko05220 | 4 | 0.018292158 | 0.37856107 |
| Huntington's disease | ko05016 | 7 | 0.021145413 | 0.37856107 |
| Bacterial invasion of epithelial cells | ko05100 | 3 | 0.022785996 | 0.37856107 |
| Thyroid hormone signaling pathway | ko04919 | 5 | 0.024251626 | 0.37856107 |
新窗口打开
2.6.2 新的miRNA靶基因富集分析 新的差异miRNA靶基因并没有显著富集的GO和KEGG pathway(corrected P-value<0.05)。同样,对前10个相对比较显著的pathway富集结果分析,发现新的miRNA靶基因与植物“植物病原菌互作”有关,包括novel-miR-808、838和1157的靶蛋白:钙依赖性蛋白激酶或钙结合蛋白(calcium-binding protein,CML22、CML31和CML45)、novel-miR-300 靶蛋白NBS-NBS-LRR型抗病蛋白(NBS-NBS-LRR type disease resistance protein)等,这些靶蛋白通过直接或者间接的识别抗原,再通过不同的通路传递信号,参与植物的防御和抗病机制。
3 讨论
本研究中采用Illumina的高通量测序技术对具有不同病毒感染症状的4个甘薯样本的叶片(龙薯9号)进行miRNA测序,从中筛选与病毒类型相关的差异miRNA。得到的数据与参考基因组比对率较高(70.59%—92.34%),不同样本RNA的长度分布具有差异性,表明small RNA长度分布可能与病毒感染有关。不同于无菌实验室的单一病毒接种,大田自发病毒感染的样本中病毒种类比较复杂,类型多样,但不同症状样本感染的病毒类型大体相同,如3个样本(除1H)都感染了常见的甘薯病毒(如SPVG、SPV2、SPVC、SPFMV、SPBV-B等甘薯常见病毒)[17,18,19],同时本研究中还鉴定到甘薯中不常见的病毒感染(DOVA、PanVY、ScaMV、PenMV等以及几类细菌噬菌体),这表明甘薯的病原物种类繁多,大田种植需要加紧防护病毒交叉感染,以提高甘薯品质和产量。
通过对病毒类型的分析,笔者推测导致感染性状之间差异的可能为少数几个病毒,如Fj01-03这3个样本都感染了甘薯常见病毒,但性状却并不一致,而3个样本均感染了SPCSV,样本表现的明显症状也不一致,只有Fj03表现出明显的褪绿矮化。因此,推断这些病毒对植株性状的影响可能取决于病毒的感染程度(病毒浓度),张爱红等[20]研究表明,玉米粗缩病株叶片内病毒的浓度与植株的病毒症状呈现正相关,病毒浓度越高植株病情越严重,并且其他研究表明病毒浓度的增加是导致病毒症状和代谢干扰程度加深的直接诱因[21,22],季志强等[22]研究表明,随着病毒浓度的加深,马铃薯脱毒苗的叶片含水量、叶绿素含量、光合和呼吸强度以及代谢干扰程度均下降。因此,高通量测序结合病毒浓度检测,精准的判定病毒感染种类以及感染程度,对大田农作物病害的诊断和防控具有重要意义。
通过对差异miRNA的分析,笔者发现这些miRNA的差异表达与病毒感染有关,这与其他文献报道的miRNA参与调控植物抵御病原微生物的结果一致[23]。部分miRNA(家族)的差异性表达可能与症状相关,如miR-156家族和miR-157家族全部成员同时存在差异性表达,而miR-166d/e/t、miR-169i/o/u、miR-395b/c/d等miRNA家族的部分成员在不同症状样本之间具有表达差异。miR-156和miR-157家族的靶蛋白多含SPL结构域(包含SBP盒子),据报道miR-156b的过表达会导致植物高度降低、矮化、分蘖数量增加,并且多与植物的生长发育、胁迫应答有关[24,25,26]。miR-166家族和miR-169家族的靶基因分别编码含有HD-Zip结构域转录因子和K Homology结构域转录因子,而这些转录因子通过各种途径参与植物的生长发育、信号传导、形态建成以及应激反应[26,27,28,29],表明这些miRNA(家族)的靶基因功能具有多样性。MiR-169家族是植物中最大的miRNA家族,其家族成员与植物的生长发育过程、抗胁迫性明显相关[30];HU等[31]研究表明,番茄(Solanum lycopersicum)的HD-ZipI型转录因子SIH24能够正向调控抗坏血酸盐的累积,从而增加植物氧化应激耐受性;另外CHEN等[27]研究发现大豆(Glycine max)中59个HD-Zip家族基因对盐胁迫或者冷胁迫具有应激性表达。另外,通过对部分差异miRNA的保守性分析,本研究所得的差异miRNA在马铃薯、拟南芥、番茄等作物中的保守性较高,因此,笔者推测这些差异miRNA通过调控靶基因在甘薯致病性感染和应激过程中发挥重要的作用。
此外,本研究还筛选了新的差异表达miRNA,这些miRNA的靶基因大多为ZFP、WD、Myb、SPL等转录因子家族成员,均参与植物的生长发育、信号传导、离子平衡等,参与并调控植物胁迫、病毒感染、抗逆性等多种生物和非生物胁迫[32,33,34]。运用GO功能富集分析发现这些差异基因大多与细胞分裂和分化、植株的生长和形态发育相关,另外,这些新的miRNA的靶基因与“植物病原菌互作”相关,这些靶蛋白包括钙离子依赖性激酶、钙结合蛋白,或者NBS-NBS-LRR型抗病蛋白,其中钙结合蛋白参与植物的抗逆性机制[35,36,37],NBS-NBS-LRR型抗病蛋白直接或者间接的与病原体因子结合后,激活下游信号,诱导植物细胞的程序性死亡并产生快速防御机制[38]。
4 结论
甘薯病毒感染与病理症状以及miRNA差异表达存在一定相关性,对差异miRNA靶基因的分析表明病毒感染引起的差异miRNA靶基因多为转录调控因子,这些转录因子通过多种形式参与调控植物的生长发育和抗胁迫。通过对病毒种类的差异分析,笔者推测导致不同甘薯样本之间症状差异的因素,不仅与病毒种类有关,还应该与病毒的感染程度有关,因此判定病毒感染种类以及感染程度对甘薯病害的诊断和防控具有重要意义。尽管有许多文献表明差异表达的miRNA与植物生长和胁迫等相关,但针对植物的生长发育、胁迫应激反应等复杂机制,miRNA挖掘和验证仍有待进一步研究。The authors have declared that no competing interests exist.
