0 引言
【研究意义】苹果作为世界上最重要的温带水果之一,实现高产是苹果育种的主要目标之一[1,2]。矮化砧木被证实可以显著减小地上部的树体大小、提高成花率,增加单位面积内果实产量[3,4]。矮化砧木是通过诱导地上部形成良好的树体结构,从而实现苹果丰产、稳产的目的[5]。但是,苹果属植物大多属于多年生木本植物,生长周期长,遗传高度杂合,在一定程度上阻碍了苹果砧木育种研究[6]。苹果砧木育种主要依靠常规育种方法,常规育种方法周期长、效率低,这些也严重制约了苹果砧木育种进程的发展[7,8,9]。因此,如何缩短苹果砧木的育种周期和提高育种效率,成为育种工作者长期的主要目标。【前人研究进展】关于苹果砧木诱导地上部生长相关的QTL早有报道。一些形态学的性状,例如株高、茎干直径,已经被证实与苹果树体结构相关[10,11,12]。PILCHER等[13]第一次找到了控制苹果矮化性状的Dw1位点,并将该位点定位于‘M.9’第5号连锁群顶部2.5 cM范围内。FAZIO等[14]以亲本‘Ottawa 3’‘Robusta 5’为试材,证明了Dw1位点的存在,且发现位于11号连锁群与之互作的Dw2位点。随后,FAZIO等[14]以‘Geneva 935’‘Budagovsky 9’为材料,构建了包含1 841个SNP标记的高密度遗传图谱,证明Dw1与Dw2位点间的互作,同时在16号连锁群发现另一个与植株生长相关的微效QTL。FOSTER等[15]以‘M.9’和‘Robusta 5’为试验材料,分别在5、11号连锁群上找到砧木诱导矮化的Dw1位点和Dw2位点,同时在LG6、LG9、LG10和LG12上存在4个较弱的QTL位点。HARRISON等[16]以‘M432’苹果砧木作图群体为试材,找到了3个与根皮率相关的QTL,其中Dw1、Dw2与前人研究一致,Dw3位于13号连锁群,该文章指出根皮率与苹果砧木诱导矮化相关,并与接穗直径呈显著负相关[16,17]。【本研究切入点】目前,关于苹果砧木诱导接穗矮化相关基因的精细定位及其调控基因的研究在国内报道较少,相对于玉米[18]、油菜[19]、水稻[20,21]、黄瓜[22]等农作物而言,研究进展缓慢。【拟解决的关键问题】本研究以矮化苹果砧木‘G.41’和乔化苹果砧木新疆野苹果为亲本构建的F1代为试材,在前人研究的基础上,利用SSR分子标记技术构建苹果5号染色体的遗传图谱,对生长相关性状进行精细定位,深入挖掘调控苹果树体生长的候选基因。1 材料与方法
试验于2014—2015年在西北农林科技大学进行。1.1 试验材料
以对接穗品种生长具有显著差异的苹果砧木‘G.41’和新疆野苹果为亲本及其杂交F1代188株为试验材料(‘G.41’为矮化苹果砧木,新疆野苹果是在我国广泛应用的乔化苹果砧木)。2014年春播种,于4月进行盆栽(250 mm×180 mm),培养土为草炭和表层土以1﹕2体积比的混合物。于7月取单株幼叶用于DNA的提取。当年9月将接穗‘富士冠军’嫁接到杂交F1代植株上。2015年7月,对植株的接穗高度、接穗横截面积等生长性状进行调查。1.2 表型数据的调查及分析
参照HARRISON等[16]的方法对生长相关性状进行调查,调查的指标包括:接穗高度(sciont height,SH)、接穗横截面积(trunk cross-sectional area,TCA)。接穗高度的测量:利用卷尺对植株高度进行多次测量,求平均值。
接穗直径的测量:利用数显电子游标卡尺,测定距离嫁接口以上5 cm位置接穗部分的主干直径,然后通过圆面积公式S=πr2计算接穗横截面积。在同一位置,测量两次,两次测量之间呈90度,求取平均值作为最终直径。
将上述调查表型数据录入Excel 2010软件,利用IBM SPSS Statistics 20软件对其表型数据进行描述性统计和相关性分析。
1.3 SSR标记筛选
参考LODHI等[23]的CTAB法提取两亲本及F1群体各单株基因组DNA。本研究中,基因初步定位使用的69对SSR引物来源于苹果公共数据库(http://www.hidras.unimi.it/)公布的引物。SSR反应体系为20 μL:模板DNA(80 ng∙μL-1)1.5 μL,正向、反向引物(10 μm∙L-1)各1.5 μL,Taq DNA聚合酶(500 U,Thermo Fisher Scientific)0.2 μL,dNTPs(2.5 mmol∙L-1)1.6 μL,MgCl2(25 mmol∙L-1)1.6 μL,10×PCR Buffer 2 μL,加ddH20至20 μL。PCR反应程序:94℃预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸30 s,32个循环;72℃保温5 min,4℃保存。扩增产物用6.0%变性聚丙烯酰胺凝胶电泳,电泳前,PCR产物中加入5 μL 6×loading buffer,置于PCR仪上,94℃,变性5 min,迅速置于冰上,备用。采用1 400 V恒电压电泳分离2 h,银染显色后在胶片观察灯下进行带型统计分析。SSR引物由生工生物工程(上海)股份有限公司合成,500 bp Marker购自广州东盛生物科技有限公司,其他分子试剂均购自宝鑫试剂公司。
1.4 遗传图谱的构建及QTL初步定位
根据“拟测交”策略,利用Joinmap 4.0[24]软件中CP群体分离模型,构建苹果遗传图谱,作图函数为Kosambi’s。应用Map QTL 5.0[25]作图软件,在构建的遗传图谱基础上,结合后代F1群体的表型数据,对接穗高度和接穗横截面积生长性状进行QTL定位。采用区间作图方法对2个生长相关性状进行分析,当LOD≥2.5时,则认为可能存在与性状相关的QTL位点。
1.5 SSR标记开发及精细定位
根据苹果属基因组数据库(https://www.rosaceae. org/species/malus/all)基因组序列信息,确定与生长基因连锁的两侧翼标记在基因组中的位置,截取相应的基因组序列,利用Microsatellite repeats finder(http:// insilico.ehu.es/mini_tools/microsatellites/)在线软件对获得的基因组序列进行SSR位点的寻找。具体的参数设置为:二核苷酸(Dinucleotide)重复次数不少于9次;三核苷酸(Trinucleotide)重复次数不少于5次、四核苷酸(Tetranucleotide)重复次数不少于4次。利用Primer Premier 5.0软件,设计新的SSR引物,其参数主要采用默认值。新设计的引物在上海生工合成,将这些引物多态性进行筛选并对5号染色体遗传图谱上的接穗高度和接穗横截面积相关的QTL位点进行精细定位。1.6 候选基因预测
根据精细定位的结果,确定基因预测的区间,一般而言选择LOD值高、贡献率大的区域。首先找到与接穗高度、横截面积生长相关性状紧密连锁的SSR标记,从而确定QTL区域在遗传图谱上的位置。然后通过SSR标记在‘金冠’苹果参考基因组物理图谱的位置,确定候选基因在物理图谱上的位置,通过GDR网站(http://www.rosaceae.org/species/malus/malus x domestica/genome v1.0),在‘金冠’苹果基因组相应区域,根据所有基因的功能注释信息,预测与植株生长性状相关的候选基因。2 结果
2.1 生长性状的遗传分析
对亲本G.41和新疆野苹果的F1代188株个体进行表型鉴定,发现F1代发生较大变异,其表型变异均呈连续变异分布符合正态分布的特点(图1、表1)。接穗高度(SH)偏度为-0.367,峰度为-0.478;接穗横截面积(TCA)偏度为-0.217,峰度为-0.795。进一步证明了该生长性状是由微效多基因控制的数量性状。相关性分析发现,接穗高度与接穗横截面积之间存在极显著的正相关关系,R=0.606(P<0.01)。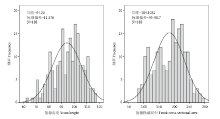 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1生长性状在作图群体中的频率分布图
-->Fig. 1The frequency distribution of growth traits in mapping population
-->
Table 1
表1
表1作图群体生长性状正态分布检测
Table 1Test of normal distribution for growth trait in mapping population
| 性状 Traits | 最大值 Maximum | 最小值 Minimum | 均值 Mean | 标准差 Std. deviation | 偏度 Kurtosis | 峰度 Skewness |
|---|---|---|---|---|---|---|
| 接穗高度 SH (cm) | 116.60 | 63.00 | 94.330 | 11.576 | -0.367 | -0.478 |
| 接穗横截面积 TCA (mm2) | 294.23 | 54.60 | 181.285 | 51.869 | -0.217 | -0.795 |
新窗口打开
2.2 遗传图谱的构建及生长相关性状QTL初步定位
利用亲本及6个F1代个体进行多态性引物的筛选,361对SSR引物中108对引物具有多态性,占总引物的29.9%。将108对多态性引物全部用于F1群体多态性分析,其中95对SSR引物能够成功应用于群体遗传图谱构建,最终构建了一张包含15个连锁群,95个SSR标记的遗传图谱(附图1)。遗传图谱总长度为897.2 cM,平均图距为9.4 cM。5号连锁群中SSR标记分布较多,有18个。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT附图1G.41×新疆野苹果遗传连锁图谱
-->Attached Fig. 1The genetic linkage map of G.41×M.sieversii
-->
利用MapQTL5.0对植株生长相关性状的QTL进行分析,共检测到4个与植株生长性状相关 的QTL(图2、表2)。其中,与接穗高度性状相关的QTL位点SH-1,定位在SSR标记CH04g09和C12371之间,LOD=7.03,解释的表型变异率为25.5%(表2);与接穗高度性状相关的QTL位点SH-2,定位在SSR标记C21057和CH03a09之间,LOD=7.57,解释的表型变异率为19.3%(表2)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2遗传连锁图谱构建与苹果生长相关性状QTL定位
-->Fig. 2QTL mapping for apple growth traits on genetic linkage map
-->
与接穗横截面积性状相关的QTL位点TCA-1,定位在SSR标记CH04g09和C12371之间,LOD=17.55,解释的表型变异率为55.1%(表2)。与接穗横截面积性状相关的QTL位点TCA-2,定位在SSR标记C21057和CH03a09之间,LOD=24.32,解释的表型变异率为51.9%(表2)。
Table 2
表2
表2苹果生长性状的QTL位点分布
Table 2QTLs of agronomic traits using Map QTL 5.0
| 性状 Traits | LOD | 左标记 Left marker | 右标记 Right marker | 表型变异率PVE (%) |
|---|---|---|---|---|
| 接穗高度 Scion height SH-1 | 7.03 | CH04g09 | C12371 | 25.5 |
| 接穗高度 Scion height SH-2 | 7.57 | C21057 | CH03a09 | 19.3 |
| 接穗横截面积 Trunk cross-sectional area TCA-1 | 33.28 | CH04g09 | C12371 | 55.1 |
| 接穗横截面积 Trunk cross-sectional area TCA-2 | 24.02 | C21057 | CH03a09 | 51.9 |
新窗口打开
2.3 SSR引物开发及精细定位
根据生长性状QTL初步定位区间的基因组序列,设计了新的SSR引物24对,其中在亲本间表现出多态的有10对(表3)。Table 3
表3
表3苹果5号染色体开发的SSR标记
Table 3SSR markers developed on LG5 in apple
| 引物名称 Primer name | 重复基元 Motif | 目的片段大小 Size (bp) | 上游引物(5’-3’) Forward primer | 下游引物(5’-3’) Reverse primer |
|---|---|---|---|---|
| L05001 | (AT)31 | 313 | GCCAGAGACTAAACCTAGCCCAATT | GGGTTCGGAAGTATGAATTTTAAGT |
| L05004 | (GA)12 | 133 | GCCGTCAAAGTAGCAACACAC | ACATACCGAAGAGAAAGCCCAT |
| L05005 | (GA)18 | 108 | CAGCATCAAGAGAAATGGTGG | GTGACCCCTCTTTCCTTCTCTTT |
| L05006 | (AG)13 | 91 | GAATAATAATGCGAAAAATAGGGA | CGTATTTTGTCCCTTCCTGC |
| L05009 | (AG)10 | 176 | TCAAACCCAAAACGTACACATCC | TCCTCTCACATTTGCCTTCCAT |
| L05012 | (TA)11 | 85 | ACAAATAACCACCCCGTCAA | GTGCTTGTTTTGGGTTGGC |
| L05018 | (GA)9 | 246 | GGTTGATAATGGGGCGGAG | TAGAGCAGTTCCACTCCTAAAAAAA |
| L05019 | (AT)23 | 91 | GTGTGAATGTGATGTATGATAGCCT | CGGGGTTTTGGAACTTTTG |
| L05020 | (TA)11 | 141 | TGGATACGATGATGGGAAAGAC | CGCAAAGAACTCGTTTACAGATAC |
| L05024 | (GA)21 | 152 | AGGGGATTTGGGTGATTAGGT | TCCCTATCCCTCTCCCCAA |
新窗口打开
利用该10对在亲本间表现出多态性的SSR引物,对5号连锁群重新分析。构建了包含21个SSR标记标记、总长为86.0 cM的遗传图谱,其平均遗传距离为7.52 cM。
其中,在遗传距离为3.68 cM范围内,与苹果生长性状连锁最紧密的SSR标记为L05018、L05024、C13449、Hi09b04。SSR标记在苹果5号连锁群的位置、LOD值、表型变异解释等信息见表4,QTL位点在染色体上的分布见图3。
Table 4
表4
表4生长相关性状的QTL位点
Table 4QTLs of growth traits
| 性状 Traits | 位置 Position | 引物名称 Locus | LOD | 表型变异率 PVE (%) |
|---|---|---|---|---|
| 接穗高度 Scion height SH | 11.249 | L05024 | 8.46 | 19.2 |
| 11.654 | L05018 | 7.08 | 16.5 | |
| 14.229 | C13449 | 7.70 | 17.4 | |
| 14.929 | Hi09b04 | 7.67 | 17.1 | |
| 接穗横截面积 Trunk cross-sectional area TCA | 11.249 | L05024 | 28.85 | 51.7 |
| 11.654 | L05018 | 25.65 | 47.8 | |
| 14.229 | C13449 | 24.36 | 45.1 | |
| 14.929 | Hi09b04 | 27.55 | 49.1 |
新窗口打开
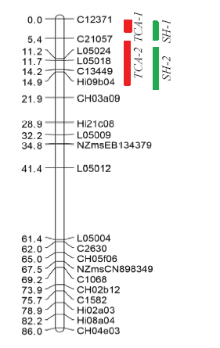 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3QTL位点在染色体上的分布
-->Fig. 3Distribution of QTLs in the chromosome 5 of G.41
-->
2.4 候选基因的预测
根据QTL显著性区间内的SSR标记所在参考基因组中的位置,利用苹果基因组计划提供的基因预测、注释结果,对生长基因定位区域进行了候选基因的预测。发现在与接穗高度、接穗横截面积性状紧密连锁的两个标记L05024和Hi09b04之间,其物理距离为4.048—4.591 Mb范围内,共包含95个基因,在染色体上的分布见图4。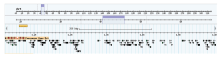 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4候选基因在5号染色体上的分布
-->Fig. 4Distribution of predicted genes on chromosome 5
-->
这些注释基因的功能主要有:锌指蛋白、DNA绑定、氧化还原酶、双链RNA结合蛋白等。其中筛选出16个已知功能注释的基因(表5),其中1个基因可能与植株生长性状相关。基因MDP0000323212来源于序列MDC019125.95,该基因序列长度855 bp,该基因家族基因是与植物休眠和生长素抑制蛋白相关基因,包括3部分编码序列。
Table 5
表5
表5生长候选基因的功能注释
Table 5Functional annotation of candidate genes for growth
| 编号 Number | 候选基因 Candidate genes | 注释信息 Function annotation |
|---|---|---|
| 1 | MDP0000487493 | 未整合的 Unintegrated |
| 2 | MDP0000216963 | 未知功能的DUF1077家族;未整合的Protein of unknown function DUF1077 family; unintegrated |
| 3 | MDP0000256637 | 铁离子结合分子功能;氧化还原酶;分子催化功能;脂氧合酶活性分子催化功能 Iron ion binding molecular function; oxidoreductase activity; Incorporati molecular function Catalysis; Lipoxygenase activity molecular function Catalysis |
| 4 | MDP0000147135 | 铁离子结合分子功能;氧化还原酶;分子催化功能;脂氧合酶活性分子催化功能 Iron ion binding molecular function; oxidoreductase activity; Incorporati molecular function Catalysis; Lipoxygenase activity molecular function Catalysis |
| 5 | MDP0000429215 | 锌指结构;Dof-type家族;未整合的;DNA分子结合功能 Zinc finger; Dof-type family; Unintegrated; DNA binding molecular function |
| 6 | MDP0000429216 | 未整合的 Unintegrated |
| 7 | MDP0000429218 | 未整合的 Unintegrated |
| 8 | MDP0000873874 | 未整合的 Unintegrated |
| 9 | MDP0000160647 | DNA分子结合功能;转录的调控;依赖DNA的生物过程;B3转录因子家族;未整合的 DNA binding molecular function; Regulation of transcription; DNA-dependent biological process; Transcriptional factor B3 family; Unintegrated |
| 10 | MDP0000266765 | 小核糖体亚基细胞成分;RNA分子结合功能;核糖体分子功能的结构组成;胞内成分;生物翻译过程;RNA和蛋白质的组成;核糖体蛋白S5家族;C末端结构域;Bacterial-type家族;N末端结构域;双链RNA结合相关域;未整合的 Small ribosomal subunit cellular component; RNA binding molecular function; Structural constituent of ribosome molecular function; Intracellular cellular component; Translation biological process; Consisting of RNA and protein; Ribosomal protein S5 family; C-terminal domain; Bacterial-type family; N-terminal domain; Double-stranded RNA-binding-like domain; Unintegrated |
| 11 | MDP0000429223 | DNA分子结合功能;核细胞组件;WD40重复;WD40重复相关域;WD40/YVTN重复相关域;未整合的 DNA binding molecular function;Nucleus cellular component; WD40 repeat repeat; WD40 repeat-like domain; WD40/YVTN repeat-like domain; Unintegrated |
| 12 | MDP0000323212 | 休眠生长相关家族;未整合的 Dormancyauxin associated family; Unintegrated |
| 13 | MDP0000249796 | 锌离子结合分子功能;核酸分子结合功能;锌指结构;CCCH类型域;未整合的 Zinc ion binding molecular function; Nucleic acid binding molecular function; Zinc finger; CCCH-type domain; Unintegrated |
| 14 | MDP0000253857 | 未整合的 Unintegrated |
| 15 | MDP0000253858 | 锌离子分子结合功能;核酸分子结合功能;锌指结构;CCCH类型域;未整合的 Zinc ion binding molecular function; Nucleic acid binding molecular function; Zinc finger; CCCH-type domain; Unintegrated |
| 16 | MDP0000933747 | Yippee相关蛋白家族;未整合的 Yippee-like protein family; Unintegrated |
新窗口打开
3 讨论
苹果砧木育种是实现苹果高产的重要方式之一,在前人对苹果砧木研究中发现,砧木对地上部接穗的生长具有重要的影响[4]。接穗高度、接穗横截面积已经被广泛看做砧木诱导接穗矮化的特征指标[15,16]。本研究以G.41×新疆野苹果杂交F1代为试材,在其上嫁接‘富士冠军’接穗,通过对接穗高度和接穗横截面积的调查,也发现苹果砧木对接穗生长具有重要影响。研究结果表明,生长性状是由苹果砧木诱导的微效多基因控制的数量性状。砧木诱导接穗矮化是一个极其复杂的性状,不仅受到砧木与接穗基因型的的影响,而且受到环境以及嫁接技术等因素的影响[15]。本研究通过选取长势一致的‘富士冠军’嫁接到杂交F1代植株上,并在相同环境条件下进行管理,以减小误差。相对于农作物和蔬菜作物,苹果砧木等多年生果树的遗传图谱通常以杂交F1代、杂交F2代和BC群体为研究对象,本研究以F1代为研究对象,通过对188株F1个体嫁接‘富士冠军’接穗进行表型鉴定,作图群体相对较大,定位结果更加准确[9]。
近年来,以DNA分子标记技术为基础,构建相应的遗传图谱,对目标性状进行QTL定位,成为果树遗传育种研究的热点。利用DNA分子标记进行分子标记辅助选择,可以提高育种效率,加速育种进程。本研究利用广泛分布于生物基因组中的遗传多态性高、重复稳定性好的共显性SSR标记[26,27,28],通过全基因组QTL定位分析,在母本‘G.41’的5号连锁群上定位到与接穗高度和接穗横截面积性状相关的QTL位点。在实验室前期研究和其他相关研究[13,14,15,16,17]基础上,本研究结合初步定位情况,在目标区域开发了一批SSR引物。相对于LIEBHARD等[29]在整个苹果基因组范围内对遗传图谱进行加密,本试验对5号染色体目标QTL区域进行SSR标记加密,提高了遗传图谱的分辨率,有利于候选基因预测,其效率更高,成本更低。
本研究将与接穗高度相关QTL定位到了苹果5号连锁群上,其峰值标记为L05024,LOD=8.46,解释的表型变异率为19.2%。KENIS等[30]同样定位到与植株高度相关的QTL位点,其解释的表型变异率为3.9%—7.9%,相对于KENIS等[30]的研究,本研究解释的表型变异率更高,结果更准确。PILCHER等[13]和FOSTER等[15]利用矮化砧木‘M.9’和乔化砧木‘Robusta 5’的后代群体为试材,定位到了位于‘M.9’的5号连锁群和11号连锁群以及‘R.5’的6号连锁群的一系列与一年生接穗高度相关QTL位点,且该位点成簇分布,并将该系列位点命名为苹果砧木诱导矮化主效Dw1、Dw2位点。本研究中在母本‘G.41’的5号连锁群上同样找到了与一年生接穗高度和接穗横
截面积相关QTL位点,且该位点在染色体成簇分布,与PILCHER等[8]结果一致。另外,接穗高度与接穗横截面积性状相关性极显著,可能存在一因多效现象。HARRISON等[16]研究认为根皮率与苹果砧木诱导矮化相关,并与接穗直径呈显著负相关,相对于HARRISON等[16]的研究,本研究进一步分析预测了与植株生长相关的基因,为今后基因克隆及分子辅助育种打下了一定基础。
4 结论
本研究以矮化苹果砧木‘G.41’和乔化苹果砧木新疆野苹果(Malus sieversii)为亲本构建的F1代分离群体为试材,基于SSR标记技术,构建了苹果5号染色体的遗传连锁图,最近的两侧翼SSR标记为L05024和Hi09b04,该区段物理距离为543 kb,并将苹果砧木诱导接穗生长候选基因确定到16个基因范围内,根据苹果基因组数据库对基因的分析及其注释信息,进一步确定了MDP0000323212可能参与植株生长的调控。The authors have declared that no competing interests exist.
