0 引言
【研究意义】玉米是世界上重要的粮食作物之一。近几年,玉米倒伏严重影响了作物的产量、降低了生产效率,减少了农民收入。玉米倒伏是一个受到多种因素影响的复杂性状,茎秆拉力、茎秆穿刺力、茎粗、株高和穗位都与倒伏性显著相关,并且与气生根层数也显著相关[1]。茎秆抗推力是衡量玉米抗倒伏的重要指标之一,对培育抗倒伏的玉米品种具有重要意义。【前人研究进展】评价玉米抗倒性的研究方法很多,有关研究表明,因茎秆强度与倒伏具有极显著相关性,可以作为衡量玉米抗倒性的重要指标[2,3]。丰光等[4]利用41个不同玉米杂交种,在不同生育时期和不同部位对其做茎秆穿刺力和拉力试验,结果表明,地上部第三茎节穿刺强度能较好地反映玉米品种的抗倒伏性。国外大部分研究者会选择在地上部第二或第三节间中部测定茎秆穿刺强度,乳熟期、蜡熟期至成熟期都会被选择为主要测定时期,也有些研究会选择开花期和抽雄前期测定[5,6]。国内研究大多数选择地上部第三节间中部或穗下节间,主要的测定时期乳熟期至腊熟期[7,8,9]。分子标记的发展让QTL作图成为解析相关性状遗传结构有效的方法[10]。近几年,关于倒伏相关性状的基因定位研究比较多,与倒伏相关性状的SNP在玉米10条染色体上均有分布,HU等[11]以B73和Ce03005组成的玉米重组自交系(RIL)群体为材料,结合分布在10条染色体的129个SSR标记构建的遗传连锁图谱,对茎秆抗弯曲强度等倒伏相关性状的QTL定位,发现7个与抗倒性显著相关位点被检测出,主要定位在第5、10染色体上,解释的表型变异区间在17.2%—26.1%。FLINT-GARCIA等[12]以3个F2:3群体作为试验材料,对抗穿刺阻力、穗位高以和经穗位矫正后的抗穿刺阻力3个性状进行QTL定位,共定位到27个相关QTL,分布在10条染色体。全基因关联分析是将表型数据与基因芯片相结合对相关基因进行的初步定位,相对于传统双亲QTL作图,全基因组关联分析具有更高分辨率、高通量等优势,成为鉴定现有种质资源中的有利等位基因的有效方法[13,14]。目前,全基因组关联分析大多数被应用于植物复杂的数量性状分析。KASHIWAGI等[15]对水稻茎粗和茎秆底部抗推力状状进行QTL定位,共检测到5个与茎秆底部抗推力显著相关的QTL,主要分布在第4、5、6、11和12染色体,发现6个控制茎粗的QTL,分布在第1、3、6、8和12染色体上。HUANG等[16]以950个水稻品种为材料,对其进行全基因组关联分析,发现32个与开花期显著相关位点,10个与产量性状关联的遗传位点。【本研究切入点】前人主要采用传统的QTL作图对相关基因进行定位,定位区间比较大,并且对茎秆推力定位研究比较少,关联分析比连锁分析具有更高的检测力,在全基因组范围筛检与目标性状相关联的遗传变异。【拟解决的关键问题】本研究以292份玉米自交系组成的自然群体,采用基因型对茎秆抗推力进行关联分析,在不同的生态环境中同时找出控制茎秆抗推力的相关SNP,进而挖掘出候选基因,为培育抗倒性玉米品种提供依据。1 材料与方法
1.1 试验材料及设计
292份自交系组成的自然群体(电子附表1),来自青岛农业大学玉米分子育种研究室,自交系选自国内骨干群体和国际主要的优势群。2015年夏,将其分别种植于山东省枣庄、青州、菏泽三地,2016年夏将其种植于山东省胶州、枣庄两地。田间试验采取单行区种植,小区行长3 m,行距0.6 m,每行15株,3个重复,田间生长进行统一管理。在玉米灌浆期,对其进行株高、穗位高、雄穗长、雄穗柄长、茎秆抗推力的测量。1.2 基因型的测定
每份自交系选取6株用打孔器在叶片中取样,采用CTAB法[17]提取DNA,由美国先峰良种国际有限公司利用MaizeSNP50基因芯片进行基因分型。该芯片包括55 126个SNP标记,均匀分布玉米自交系B73全基因组。SNP基因型检测参照Illumina公司提供的操作指南,采用Illumina Bead Station 500G SNP分型系统完成。参考WENG等[18]方法控制芯片基因型数据质量。
1.3 表型数据的测定方法
在玉米完成授粉后的灌浆期(约15 d),每行选取生长情况一致的6棵代表植株,用数显植物茎秆强度检测仪(浙江托普仪器有限公司,型号YYD-1B),在植株基部第三节中部(大约离地面20 cm处)以茎秆的垂直短轴方向推倒至45°角,瞬间读取并记录试验数据,单位是牛顿(N)。同时测定株高、穗位高、雄穗长和雄穗柄长。1.4 表型数据的统计分析
运用SPSS statistics 20.0数据分析软件和Excel 2007软件对表型数据进行统计分析和相关性分析。1.5 茎秆抗推力的全基因组关联分析
该芯片包括55 126个SNP,去除杂合率大于10%、缺失率大于20%和最小等位基因频率低于0.05的标记,剩余25 331个SNP(MAF≥0.05),利用farmCPU对茎秆抗推力表型数据进行全基因组关联分析。在P<0.0001水平上,判定SNP标记与茎秆抗推力关联的显著性。2 结果
2.1 玉米抗推力的统计分析
由表1可知,两年茎秆抗推力的均值为103.64、101.81、93.85、98.33和93.10,偏度和峰度都接近于零,说明大多数符合正态分布。Pearson相关分析发现2015菏泽、2015青州、2015枣庄、2016胶州和2016枣庄的茎秆抗推力差异极显著,适合进一步关联分析。2.2 茎秆抗推力的全基因组关联分析
采用分布于玉米基因组的55 126个SNP标记对292份玉米自交系茎秆抗推力进行关联分析(表2和图1)。共检测到16个与茎秆抗推力显著关联的SNP位点(P<0.0001),分别位于染色体框3.06、5.02、4.08、6.04、8.03和9.03,其中在染色体框3.06、5.02和6.04有2个显著的SNP,在染色体框4.08和9.03分别检测出3个显著的SNP。在两年的枣庄环境下检测出4个显著SNP位于8.03染色体框中。通过采用分子标记对茎秆抗推力进行全基因组关联分析获得QQ图(图1),表明关联群体的群体结构得到了较好控制。对标记位点进行扫描分析,共获5个候选基因(表2),分别是GRMZM2G504401编码几丁质酶A,GRMZM2G062974编码产物是bZIP转录因子超家族的蛋白,bZIP蛋白在生命活动中发挥重要作用。GRMZM2G053767编码核糖体40S亚基S4类蛋白,GRMZM2G348551和GRMZM2G024260编码一个可溶性淀粉合成酶2-3、腺嘌呤核苷酸水解酶超家族蛋白。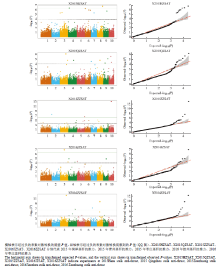 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1全基因组关联分析(manhattan)图(左)和全基因组关联分析QQ图(右)
-->Fig. 1(Left) Genome-wide association study of stalk anti-thrust (manhattan plot) (Right) Quantile-quantile plot for stalk anti-thrust (QQ plot)
-->
Table 1
表1
表1玉米茎秆抗推力的统计分析
Table 1Statistical analysis of maize stalk anti-thrust
| 环境 Environment | 均值 Mean | 标准差 SD | 偏度Kurtosis | 峰度Skewness | 相关系数 Correlation coefficient | |||
|---|---|---|---|---|---|---|---|---|
| 2015菏泽 Heze 2015 | 2015青州 Qingzhou 2015 | 2015枣庄 Zaozhuang 2015 | 2016胶州 Jiaozhou 2016 | |||||
| 2015菏泽 Heze 2015 | 103.67 | 38.75 | 0.653 | 0.370 | ||||
| 2015青州 Qingzhou 2015 | 101.67 | 31.75 | 0.122 | 0.549 | 0.514** | |||
| 2015枣庄 Zaozhuang 2015 | 93.80 | 31.35 | 0.355 | 0.143 | 0.643** | 0.579** | ||
| 2016胶州 Jiaozhou 2016 | 101.63 | 28.19 | 0.059 | 0.353 | 0.038** | 0.496** | 0.485** | |
| 2016枣庄 Zaozhuang 2016 | 92.28 | 28.92 | 0.316 | 0.224 | 0.174** | 0.376** | 0.216** | 0.339** |
新窗口打开
Table 2
表2
表2与茎秆抗推力相关的显著关联的SNP位点
Table 2SNPs identified to be associated with stalk anti-thrust (P<0.0001)
| 标记名称 SNP | 基因型 Genotype | 染色体位置 Bin | 物理位置 Physical position (bp) | P值P-value | 候选基因Candidate gene |
|---|---|---|---|---|---|
| PZE_103111295 | C/T | 3.06 | 171756718 | 7.69E-07 | |
| PZE_103125327 | A/T | 3.06 | 182769908 | 6.20E-13 | GRMZM2G504401 |
| PZE_104115471 | A/G | 4.08 | 191414929 | 8.26E-05 | |
| PZE_104111700 | C/T | 4.08 | 100995544 | 7.92E-05 | |
| PZE_104115494 | C/G | 4.08 | 191442456 | 1.99E-07 | |
| PZE_108026930 | C/T | 8.03 | 27500809 | 5.80E-06 | |
| SYN19532 | C/T | 8.03 | 62032801 | 2.81E-05 | |
| PZE_108048987 | G/T | 8.03 | 83263452 | 2.65E-06 | |
| PZE_108050769 | C/G | 8.03 | 88321076 | 1.76E-10 | GRMZM2G062974 |
| PZE_109038100 | G/T | 9.03 | 57135281 | 5.88E-05 | |
| PZE_109053667 | C/T | 9.03 | 93668797 | 1.32E-05 | |
| PZE_109030486 | T/C | 9.03 | 35717991 | 6.22E-05 | |
| SYN29805 | A/G | 6.04 | 114919673 | 1.02E-06 | GRMZM2G053767 |
| PZE_106062467 | G/A | 6.04 | 113417198 | 1.04E-05 | GRMZM2G348551 |
| PZE_105094195 | A/G | 5.02 | 137605204 | 6.06E-05 | GRMZM2G024260 |
| PZE_105024985 | T/C | 5.02 | 12545280 | 4.99E-05 |
新窗口打开
2.3 玉米茎秆抗推力与其他农艺性状的相关性分析
在菏泽、青州、枣庄、胶州4个环境中,利用SPSS20.0软件对茎秆抗推力与株高、穗位高、雄穗长和雄穗柄长4个性状进行相关性分析(表3)。在胶州、青州和菏泽,茎秆抗推力与株高、穗位高都有显著相关。除胶州外,其他环境茎秆抗推力与雄穗长、雄穗柄长都显著相关,枣庄的茎秆抗推力与株高、穗位高相关性不显著,可能与环境有关。研究发现,茎秆抗推力与4个农艺性状具有一定的相关性,但是相关性不是很稳定。Table 3
表3
表3茎秆抗推力与4个农艺性状的相关性
Table 3Correlation between stalk unti-thrust and four agronomic traits
| 环境 Environment | 株高 Plant height | 穗位高 Ear height | 雄穗长 Tassel length | 雄穗柄长 Male spike length |
|---|---|---|---|---|
| 胶州 Jiaozhou | 0.142* | 0.179** | 0.109* | 0.008 |
| 青州 Qingzhou | 0.334** | 0.404** | 0.337** | 0.215** |
| 菏泽 Heze | 0.934** | 0.185** | 0.482** | 0.210** |
| 枣庄 Zaozhuang | -0.028 | -0.006 | 0.422** | 0.249** |
新窗口打开
3 讨论
3.1 茎秆抗推力关联分析
关联分析(association analysis)又称关联作图(association mapping),是一种基于连锁不平衡的作图方法。该方法利用不同位点等位基因之间的连锁不平衡,对标记基因与性状结合的相关性分析,从而检测出与表型性状变异密切相关的功能性等位基因[19]。目前,关联分析已经应用于植物复杂数量性状的多方面研究,但是对于研究玉米倒伏方面的关联分析还相对较少。本研究对292份玉米自交系组成的自然群体进行全基因组关联分析,在两年数据中共检测出16个与茎秆抗推力显著关联的SNP(P<0.0001)。研究表明利用连锁定位和关联分析定位到的QTL在位置上大部分具有一致性[20,21,22]。LI等[23]以2个玉米重组自交系为材料并对其遗传分析,共获得7个与茎秆强度显著相关的QTL位点。染色体框9.03共有3个标记与茎秆抗推力显著关联,PZE_109038100、PZE_109053667和PZE_109030486均位于LI等定位的PZE-109058177—PZE-10907676区间,并且在染色体框9.03中检测的3个SNP,在枣庄、青州、胶州等地都被检测到相同的染色体位置,具有较高的可信度。染色体框4.08、8.03和6.04均都被检测到多个SNP,染色体框8.03中被检测到共有4个SNP,包括2015年枣庄环境中的检测到PZE_108026930、SYN19532和2016年枣庄PZE_108048987、PZE_108050769共4个SNP,并且与HU等[24]利用B73×Ce03005建立的重组自交系群体检测到phi100175-umc1562的染色体区段一致。染色框4.08有3个SNP,分别在2015年枣庄、2016年枣庄和2016年胶州被检测到,同时FLINT-GARCIA等[25]以4个F2:3玉米群体为研究材料,对茎秆穿刺强度进行了QTL分析,相关位点也定位到染色体框4.08。本研究在多个环境中定位区间存在重叠现象,表明这些染色体框有可在不同环境或不同群体稳定表达的QTL。HU等[24]利用B73与玉米自交系Ce03005创建RIL群体,进行茎秆穿刺强度测定,结果中检测到了相同的染色体框。因此,通过群体间基因定位结果相互验证,表明该QTL在不同环境和群体间表达的稳定性以及检测结果的可靠性。本研究共检测出5候选基因,为下一步研究抗倒伏奠定了基础。3.2 候选基因分析
共检测出5个候选基因,GRMZM2G504401编码几丁质酶A,几丁质是由1 000—3 000个N-乙酰葡萄糖胺残基通过β-1,4糖苷键构成的聚合物。在植物与病原物的相互作用中,几丁质的降解产物N-乙酰葡萄糖胺可通过相关的分子模式的作用激发植物对病原物的防卫反应[26]。拟南芥受体激At CERK1的胞外区3个赖氨酸基序与几丁质寡糖结合,激活植物免疫反应,并且几丁质诱导的At CERK1的二聚化可激发At CERK1介导的信号途径[27]。玉米茎秆中存在几丁质酶受体蛋白能有效的防御外来侵害。GRMZM2G062974编码产物是bZIP转录因子超家族的蛋白,b ZIP蛋白存在于真核生物中一类重要的TF,广泛存在于动物、植物等真核生物中[28]。GRMZM2G348551编码可溶性淀粉合成酶,参与糖类代谢途径,可能与茎秆强度有关。KEELING等[29]认为,可溶性淀粉酶是控制淀粉积累的关键酶。GRMZM2G024260编码腺嘌呤核苷酸水解酶超家族蛋白,植物核糖核酸酶作为RNA代谢关键酶,环境和自然衰老都可使得该酶活性增强,从而使玉米茎秆能够抵御不良胁迫。GRMZM2G053767编码核糖体40S亚基S4类蛋白,核糖体蛋白是核糖体的重要组成部分,主要参与细胞内的重要活动,并且对核糖体转录效率及稳定性有重要作用。3.3 茎秆抗推力与4个农艺性状的相关性分析
丰光等[1]以3个玉米品种为材料,研究茎秆性状与倒伏的关系,结果表明茎粗、株高和穗位高与倒伏有极显著相关,茎粗和穗位高对茎秆倒伏相关性比较大,可以作为衡量玉米倒伏的指标。本研究在4个环境下,对茎秆抗推力与株高、穗位高、雄穗长、雄穗柄长做了相关性研究。研究发现在胶州、青州、菏泽茎秆抗推力与株高、穗位高呈显著或极显著相关,跟前人的结果一致,菏泽相关性不显著,可能与环境有关。付志远等[30]研究认为穗位高/株高可以作为衡量倒伏的一个重要指标,通过减小穗位高系数成为一种提高植株抗倒伏性的可行途径。近几年,在倒伏方面研究株高、穗位高、茎粗、茎节长的比较多,但是研究雄穗长雄穗柄长的相对较少。通过表3可知在枣庄、菏泽、青州,茎秆抗推力与雄穗长、雄穗柄长都有显著的相关性,在以后倒伏方面的研究中,雄穗长、雄穗柄长也是影响因素。4 结论
共检测出16个与茎秆抗推力显著相关的SNP(P<0.0001),获得5个候选基因,分别是GRMZM2G504401、GRMZM2G062974、GRMZM2G053767、GRMZM2G348551和GRMZM2G024260。倒伏的发生可能会受到多种非生物胁迫的影响,GRMZM2G062974参与植物在逆境下的表达调控,GRMZM2G504401的表达会受到多种胁迫的诱导,响应多种生物或者非生物胁迫。倒伏的发生也会受到自身因素的影响,GRMZM2G348551参与糖代谢途径,调控支链淀粉的合成,与细胞壁的形成有关。The authors have declared that no competing interests exist.
