0 引言
【研究意义】桃[Prunus persica (L.)Batsch]是中国栽培面积较大的落叶果树之一。2014年中国桃栽培面积约799 500 hm2,居世界桃栽培第一位(FAO)。其中,矮化型桃因其节间短、成花好是观赏桃和砧木育种的发展方向之一。通过对桃矮化基因进行精细定位是建立目标性状分子辅助选种体系和品种改良的基础。【前人研究进展】中国是桃起源中心,具有丰富的遗传资源。作为重要的农艺性状之一,树型种类多样,包括普通生长型(Standard type)、柱型(Pillar type)、半矮生型(Semi-dwarf type)、垂枝型(Weeping type)、直立型(Upright type)、短枝型(Spur type)和矮化型(Dwarf type)等几类[1]。其中,矮化型桃的明显特征是节间长度极短、树冠密。LAMMERTS[2]最早报道了来源中国的矮化桃遗传资源,之后多位科研人员开始了一系列的研究[3],并育成了不少矮化桃品种,如‘Bonfire’‘Leprechaun’等[4]。国内育种家同步选育了矮化观赏型桃品种,宗学普等[5]开展了桃矮化品种的选育,选出的矮化油桃新品系‘矮丽红’。YAMAMOTO等[6]是最早开展桃矮化基因的定位工作,将控制桃矮化的dw定位在连锁群6的前端。HOLLENDER等[7]采用重测序的方法将来源日本观赏桃的dw进行定位,获得了编码赤霉素受体的候选基因(GID1c)。【本研究切入点】矮化型桃有多种类型,由于树体矮小,是盆栽观赏和砧木的重要育种资源。虽然,我国拥有重要的矮化性状遗传资源,但是桃矮化基因定位研究工作开展较少,相关的分子辅助选种体系还没有建立。【拟解决的关键问题】本研究以来源中国的矮化桃品种为材料,通过构建杂交群体对控制桃矮化性状基因进行精细定位,获得紧密连锁的SNP标记,为建立目标性状的分子鉴定体系,有目的选育矮化观赏桃和砧木品种奠定基础。1 材料与方法
田间试验于2014—2016年在中国农业科学院郑州果树研究所新乡试验基地(河南新乡)进行,室内试验在中国农业科学院郑州果树研究所农业部果树育种技术重点实验室(河南郑州)进行。1.1 研究材料
根据该矮化性状的遗传特征,以单株‘05-2-144’为亲本自交构建分离群体,其中‘05-2-144’来源于2005年‘97矮’(矮化型)ב鸳鸯垂枝’桃(非矮化型)杂交单株。于2014年和2015年分别以‘05-2-144’为亲本自交,共获得395个后代单株。用于分子鉴定的亲本为‘10-7’(普通型)ב96-5-1’(普通型),双亲均来源于97矮毛桃,共获得89个后代单株;2个组合的后代桃核经自然干燥后用锤子将外壳敲开,取种子进行包衣(包衣剂,先正达),晾干后,放于湿润的粗砂中进行层积处理,次年1月取出萌发的种子,种植于穴盘中用于表型观察和后续研究。1.2 基因组DNA的提取
基因定位亲本和自交后代单株叶片基因组DNA的提取采用CTAB法[8],略作修改。获得沉淀DNA后,加入200 μL体积的0.1×TE溶解沉淀DNA,同时加入0.5 μL的RNase,去除RNA污染(长期保存在-20℃冰箱,常用则存于4℃冰箱)。采用NanoDrop 1000 spectrophotometer(Themo)和1%的琼脂糖胶检测DNA的纯度浓度和完整度,稀释成工作液浓度(25 ng∙μL-1)备用。自交亲本提取DNA纯化后送样建库。1.3 基于Sanger测序SNP标记的开发
参考桃基因组序列信息(Genome Database for Rosaceae数据库Version 2.0)中桃基因组Pp 01—08,采用primer3Web Version 4.0(http://primer3.ut.ee/)设计引物,约每1—2 Mb设计1对引物,引物退火温度在59—63℃,引物长度20—23 bp,引物扩增片段长度为1 600 bp或750 bp左右。基于Sanger测序开发基因型和表型一致的SNP标记,用于目标基因的定位。1.4 基于二代测序SNP标记的开发
构建好文库后对亲本‘05-2-144’进行深度测序,在仪器HiSeq 4000(Illumina,CA)上进行,测序深度约为70X。在定位区域内采用Integrative Genomics Viewer 2.3(IGV)对Bam格式数据进行分析[9],确定目标区域内基因型为Dwdw的SNP,以设计候选标记,用于连锁SNP标记的开发。1.5 基于HRM技术的SNP基因分型
根据深度测序的结果设计引物。采用HRM master mix(Roche)试剂进行PCR扩增,反应总体积为15 μL。其中含15 ng模板DNA、0.17 μmol·L-1正/反引物和1μL HRM master荧光染料(Roche)。在罗氏LightCycler 480II定量PCR仪(Roche)进行PCR扩增和HRM分析。高分辨率熔解曲线分析采用Gene Scanning软件(version1.50),具体参考鲁振华等[10]。1.6 矮化性状的精细定位与验证
根据表型鉴定结果,在矮化型和普通型各两个子代和亲本中寻找连锁的SNP标记。具体基因型表现为,自交亲本单株‘05-2-144’基因型为Dwdw,矮化型子代基因型为dwdw,普通型子代基因型为DwDw和Dwdw,在4个子代中获得连锁的SNP标记后,扩大至分离后代各20个单株中,确定连锁后进一步扩大至全部样品,完成初步定位。同时结合亲本二代测序结果开发的SNP标记,在群体所有后代单株中进行SNP分析,确定交换单株并获得紧密连锁的SNP标记。遗传距离的计算采用JoinMap(3.0)[11],以F2群体方法进行遗传距离计算,参数设置为默认。2 结果
2.1 杂交群体表型鉴定和遗传特征分析
通过对‘05-2-144’自交后代实生苗观察,2014年得到102株杂种苗,其中普通型和矮化型分别为78株和24株,二者比例接近3﹕1(P值为0.732;χ2为0.118)。为进一步对目标基因进行精细定位,于2015年进行了自交,共得到293株自交后代单株,对其进行目测观察,区分普通型和矮化型幼苗分开定植。其中普通型222株,矮化型71株,二者比例接近3﹕1(P值为0.761;χ2为0.092)。用于分子鉴定的单株来源于‘10-7’(普通型)ב96-5-1’(普通型)杂交组合,共获得89个后代单株,其中普通型66株;矮化型23株(P值为0.854;χ2为0.034)。目标性状分离比例符合孟德尔遗传规律,矮化性状受隐性单基因控制。同时对定植2年后植株表型进行了再鉴定,幼苗与定植后2年植侏表型观察结果一致(图1)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1普通型桃(左)和矮化型桃(右)植株表型
-->Fig. 1Phenotype of standard type (left) and dwarf type (right) peach
-->
2.2 矮化基因的初步定位
参考桃基因组(https://www.rosaceae.org/gb/gbrowse/ prunus_persica_v2.0.a1/ Version 2.0)序列信息,设计了桃基因组Pp 01—08的引物,约每1—2 Mb设计1对引物,引物扩增片段长度为1 600 bp或750 bp左右(表1)。基于Sanger测序法获得SNP标记,并根据子代基因型和表型确定候选SNP标记,并在杂交群体后代单株中(普通型和矮化型各4株)进行序列测定获得多态性的SNP标记进行初步分析。在Pp06的25 230 425 bp和27 191 090 bp处找到了可能连锁的SNP标记。在获得可能的SNP标记后扩大了杂交群体,在普通型和矮化型各20个后代单株中进一步验证连锁关系,进而在全部样品中验证。通过扩大群体分析,在Pp06上25 230 425 bp(LG6-25.2)和27 191 090 bp(LG6-27)处获得了与dw连锁的SNP标记,根据重组单株以及重组单株数目的变化表明,目标基因位于获得的两个标记右侧。2.3 矮化基因的精细定位
在初步获得连锁的SNP分子标记后,对亲本进行深度测序。根据亲本‘05-2-144’基因型和表型开发更多Aa基因型的SNP标记。后扩大分离群体进行连锁关系验证,以获得紧密连锁的SNP标记。亲本‘05-2-144’测序共产生Raw data 17.956 Gb,过滤后的Clean data 17.098 Gb,测序质量较高,其中Q20≥94.9%,Q30≥88.57%,全基因组GC含量为39.04%。参考基因组大小为227 411 381 bp,所有样本的比对率为96.71%,对参考基因组(排除N区)的平均覆盖深度为66.89X,1X覆盖度(至少有一个碱基的覆盖)在98.77%以上,可用于后续的变异检测及相关分析。根据开发的SNP标记,在参考基因组(Version 2.0)30.003191 Mb处获得了另一侧连锁的SNP标记。基于重测序数据和定位区间,共设计了15对SNP引物,其中12对引物与重测序SNP基因型一致,3对引物与重测序SNP基因型不一致成功率为80.0%(表1)。利用其中SNP标记进行了目标性状的精细定位,在桃基因组Pp06的28 712 165 bp(JXSNP-5)和28 989 661 bp(JXHRM-SNP-3)之间,物理距离区间约为277 kb(图2)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2桃矮化基因的遗传距离分析
-->Fig. 2Linkage map of dwarfing gene for peach
-->
Table 1
表1
表1本研究所用引物信息
Table 1Primers information used in this study
| 引物名称 Primer code | 引物序列(5′-3′) Primer sequence(5′-3′) | SNP位置 SNP Position (bp) | SNP类型 SNP variants |
|---|---|---|---|
| LG6-25.2 | TCATGCCGAAGTGATGGTGC GCCAGACTGAAAGGGATCGG | Pp06_25230425 | C/A |
| LG6-27 | AGTAACTATTGAGGCGATGGTGC AGCCTTCCAAAGTTTCTGCTCA | Pp06_27191090 | T/A |
| JXSNP-1 | AAGCACGGTAGTTTGTAGTATGC TCAAACGTACTGCAAGGGAGT | Pp06_28004260 | A/C |
| JXSNP-3 | TGACATGAGCGAGGAGGTGT CACCAATGGAGCCTCAACCC | Pp06_28264485 | A/C |
| JXSNP-4 | ATGTAAAGGCTCCCGGGAAA ACTGCAATTCACGCCAGAAG | Pp06_28428373 | C/G |
| JXHRM-SNP-1 | AGGTCTAGGGAAGCAACTCCA ATGCCCCTTTCAACGAAGCC | Pp06_28646348 | T/C |
| JXSNP-5 | TACGTCAGACTTGGTGGCCA ACACCTAGGAATCCAGGCGG | Pp06_28712165 | T/G |
| JXHRM-SNP-2 | GGAAGTGTTTTGGGCCGACA TTGTTGCACTCTTGTCGGCC | Pp06_28978905 | C/T |
| JXHRM-SNP-3 | GCATTGGCGCAGAAACCCTA CTCGTCCCTGCAACCCAATC | Pp06_28989661 | A/T |
| JXHRM-SNP-5 | GGTGATAACTATGAGGGCTGGG TGTCACCTCAGCTGCTTGAA | Pp06_29046792 | C/G |
| JXHRM-SNP-7 | TGGGCTCGTACTTCTTTGCA AGGCTGAGCTGAAGGAGAAG | Pp06_29373633 | C/G |
| JXHRM-SNP-8 | TGGGAATGTGCTTTCGGTCT ACCTACCCAACACCACGTAG | Pp06_29646207 | T/C |
| JXHRM-SNP-9 | CCTCTCGTTATCTGAGTATGAGCT TCATTTGTTTGGGTTGTGTGCA | Pp06_29821407 | A/G |
| JXHRM-SNP-10 | TTTGATCGAGTGGTGGGCAT GGGTCATCCTCCTCTTTCAAGA | Pp06_30003191 | A/G |
| YZ-SNP260k1 | AGGGTTTCATGGCGTTAAAGC AAACTGAACTGCTCTTCCACGG | Pp06_28108436 | C/T |
| YZ-SNP260k2 | CTTTTCTCCGCCGCGTTAAT CCCGGGATGTGACAATTTGG | Pp06_29247763 | A/G |
新窗口打开
2.4 精细定位区域的候选基因
参考桃基因组数据库(Version 2.0)在精细定位区域277 kb内,包含已知转录本54个,具体如表2所示。精细定位区域内基因包括螺旋-环-螺旋(bHLH)、DHHC类锌指蛋白、GAG反转座子、信号肽酶、受体类激酶、赤霉素受体(GA不敏感矮化型)、未知功能蛋白等(表2)。其中,GIDC基因可能是候选基因,这与HOLLENDER等[7]报道一致。2.5 植株表型的分子鉴定
根据已经获得的紧密连锁的SNP标记的物理位置,并基于参考桃基因组数据在新的亲本(‘10-7’X‘96-5-1’)中开发基因型和表型一致的SNP标记用于分子鉴定,在精细定位目标基因两翼分别设计1对SNP引物,验证分子标记对杂交后代单株表型鉴定的准确性。其中引物YZ-SNP260k1位于Pp06的28 108 436 bp处,SNP类型为C/T;引物YZ-SNP260k2位于Pp06的29 247 763 bp处,SNP类型为G/A。通过标记基因型和表型比较,89株后代单株的验证符合率为100%(图3)。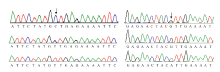 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3基于SNP标记桃矮化性状的分子鉴定
-->Fig. 3Dwarfing trait detection for peach based on SNP Marker
-->
Table 2
表2
表2精细定位区域内候选基因
Table 2Candidate genes in fine mapping region
| 基因ID Gene ID | 基因描述 Gene description |
|---|---|
| Prupe.6G327100 | 未知蛋白 Unknown protein |
| Prupe.6G327200 | 糖蛋白膜前体 Glycoprotein membrane precursor GPI-anchored |
| Prupe.6G327300 | 糖蛋白膜前体 Glycoprotein membrane precursor GPI-anchored |
| Prupe.6G327400 | NAD (P-)结合蛋白 NAD(P)-binding Rossmann-fold superfamily protein |
| Prupe.6G327500 | GAG反转座子 Retrotransposon gag protein (InterPro:IPR005162) |
| Prupe.6G327600 | 甲羟戊酸激酶 Mevalonate kinase |
| Prupe.6G327700 | 酰胺酶I Amidase 1 |
| Prupe.6G327800 | U-box超级家族蛋白U-box superfamily protein |
| Prupe.6G328100 | 向光响应蛋白 Phototropic-responsive NPH3 family protein |
| Prupe.6G328300 | 线粒体编辑因子 Mitochondrial editing factor 19 |
| Prupe.6G328400 | 热激蛋白类伴侣 HSP20-like chaperones superfamily protein |
| Prupe.6G328500 | 核Y因子 Nuclear factor Y, subunit C9 |
| Prupe.6G328600 | 阳离子交换Cation exchanger 11 |
| Prupe.6G328700 | 未知蛋白 Unknown protein |
| Prupe.6G328900 | 信号肽酶 Signal peptidase subunit |
| Prupe.6G329000 | 真核天冬氨酸蛋白酶 Eukaryotic aspartyl protease family protein |
| Prupe.6G329100 | 重金属转运蛋白 Heavy metal transport/detoxification superfamily protein |
| Prupe.6G329200 | 糖基转移酶Glycosyltransferase family protein 28 |
| Prupe.6G329300 | 蛋白激酶 Protein kinase superfamily protein |
| Prupe.6G329400 | 核苷酸修复蛋白 Nucleotide repair protein, putative |
| Prupe.6G329500 | U-box超级家族蛋白U-box superfamily protein |
| Prupe.6G329600 | bHLH DNA结合蛋白 (bHLH) DNA-binding superfamily protein |
| Prupe.6G329700 | 甲基转移酶 Methyltransferases |
| Prupe.6G329800 | RNA结合蛋白RNA-binding family protein with retrovirus zinc finger-like domain |
| Prupe.6G329900 | 液泡铁转运蛋白 Vacuolar iron transporter (VIT) family protein |
| Prupe.6G330000 | D-氨基酸转氨酶D-aminoacid aminotransferase-like |
| Prupe.6G330100 | S-腺苷-L-甲硫氨酸依赖性甲基转移酶S-adenosyl-L-methionine-dependent methyltransferases superfamily protein |
| Prupe.6G330200 | ATP结合微管马达蛋白ATP binding microtubule motor family protein |
| Prupe.6G330300 | GDSL类酯酶GDSL-like Lipase |
| Prupe.6G330400 | 类囊体腔 Thylakoid lumen 18.3 kDa protein |
| Prupe.6G330500 | 光系统II反应中心Psb蛋白Photosystem II reaction center PsbP family protein |
| Prupe.6G330600 | 钠氢交换体 Sodium hydrogen exchanger 2 |
| Prupe.6G330700 | 线粒体内膜转位酶 Mitochondrial inner membrane translocase complex |
| Prupe.6G331000 | 谷胱甘肽合成酶 Glutathione synthetase 2 |
| Prupe.6G331100 | 未知功能蛋白 Protein of unknown function (DUF1068) |
| Prupe.6G331200 | 磷酸甘油酸变位酶 Phosphoglycerate mutase family protein |
| Prupe.6G331300 | 磷酸甘油酸变位酶 Phosphoglycerate mutase family protein |
| Prupe.6G331500 | 未知功能蛋白 Protein of unknown function (DUF3049) |
| Prupe.6G331600 | 丙二烯氧化物酶Allene oxide cyclase 4 |
| Prupe.6G331700 | 未知功能蛋白 Protein of unknown function (DUF679) |
| 基因ID Gene ID | 基因描述 Gene description |
| Prupe.6G331800 | 协运超家族蛋白Facilitator superfamily protein |
| Prupe.6G331900 | Rab护卫蛋白 Rab escort protein |
| Prupe.6G332100 | 受体类激酶 Rreceptor-like kinase in in flowers 3 |
| Prupe.6G332200 | 受体类激酶 Receptor-like kinase in in flowers 3 |
| Prupe.6G332300 | ROP结合蛋白激酶ROP binding protein kinases 2 |
| Prupe.6G332400 | 酵母自噬同源蛋白Homolog of yeast autophagy 18 (ATG18) H |
| Prupe.6G332500 | 含组氨酸的磷酸转移酶Histidine-containing phosphotransmitter 1 |
| Prupe.6G332600 | 肌苷-尿苷优选核苷水解酶Inosine-uridine preferring nucleoside hydrolase |
| Prupe.6G332700 | 前折叠素分子伴侣蛋白Prefoldin chaperone subunit family protein |
| Prupe.6G332800 | GA受体 GID1C, alpha/beta-Hydrolases superfamily protein |
| Prupe.6G332900 | γ组蛋白变异体 Gamma histone variant H2AX |
| Prupe.6G333000 | 未知蛋白 Unknown protein |
| Prupe.6G333100 | DHHC类锌指蛋白 DHHC-type zinc finger family protein |
| Prupe.6G333200 | 富精氨酸细胞周期蛋白 Arginine-rich cyclin 1 |
新窗口打开
3 讨论
在遗传图谱构建方面,SNP标记优势突出[12-13]。近年来随着生物技术的进步,特别是基于二代测序的标记技术加速了作物基因的精细定位和分子辅助选种体系的建立[14-17]。建立桃树分子辅助选种体系是实现目标性状早期鉴定的关键环节,可加速育种进程,实现多个优良性状的聚合,提高果树的经济价值。如何准确鉴定目标性状是育种者关心的科学问题。精细定位和克隆目标性状基因是关键,在精细定位区域内开发与目标性状紧密连锁的分子标记,包括SSR、SNP和Indel等,而克隆到目标基因是实现对目标性状直接鉴定的重要途径。在桃中,已完成红肉性状[18]、黄/白肉[19-20]、果皮颜色亮度[21]、肉质类型[22]、分枝角度[23]和矮化性状[7]等基因的直接分子鉴定。以上研究为加快桃遗传改良进程奠定了基础。果树树型性状与果实性状不同,在苗期即可实现部分性状的表型鉴定,但对于隐性单基因和多基因控制的树体性状,很难实现早期的表型鉴定,仍需通过基因定位建立辅助选种体系[22]。果树作物中,苹果最先在生产中利用了柱型性状,实现了高产、高密栽培,兼顾省时和省力[23-27]。对桃树型研究主要集中在株高和节间长度方面[28-30]。YAMAMOTO等[6]将控制桃株高矮化基因定位在LG6上;张妤艳等[31]采用Fl杂交分离群体共123个单株,获得了与矮化性状连锁的SRAP标记,遗传距离3.2 cM。最近,HOLLENDER等[7]采用BSA-seq重测序的方法对来源于日本矮化品种‘Juseito’的BD(Brachytic dwarfism)性状的dw位点进行了定位,获得了候选基因赤霉素受体(GID1c),并验证了基因的功能。尽管功能验证表明GID1c与株高表型有关,但该矮化基因是从900 Kb的定位区间内筛选的候选基因,本研究则提供了进一步的数据支持。本研究定位的dw基因与日本的矮化品种‘Juseito’的dw位于同一区域,但二者是否存在不同的变异形式以及来源是否相同,仍需进一步研究。
4 结论
本研究对来源‘97矮’桃单株的矮化性状基因进行了精细定位,目标性状位点位于Pp06的28 712 165—28 899 661 bp,物理距离约为277 kb,包含54个已知转录本。在精细定位目标基因两翼分别设计1对SNP引物,89株后代单株的分子鉴定符合率为100%,研究结果为揭示桃矮化性状的遗传机制并建立目标性状的分子辅助选种体系奠定了基础。The authors have declared that no competing interests exist.
