0 引言
【研究意义】稻瘟病由子囊真菌Magnaporthe oryzae引起,是最严重的水稻病害之一,其危害面积和危害程度较大,已成为水稻高产稳产的严重阻碍。据统计,稻瘟病使全球每年损失稻谷产量占总产量的10%—15%,造成经济损失达数十亿美元[1-2]。目前,通过育种培育广谱持久抗病的水稻品种是控制稻瘟病的主要措施[3],而发掘稻瘟病抗性基因,深入研究抗性基因的作用机制则是有效育种的前提。由于稻瘟病菌变异性强,需要加强对水稻抗性基因各方面的研究,从而采取措施以避免抗性品种种植几年就丧失抗性[4-5]。【前人研究进展】Pi-ta是最早被克隆的稻瘟病抗性基因之一[6]。目前,水稻Pi-ta和稻瘟菌无毒基因AVR-Pita是研究最为详尽的一对互作关系,也是基因对基因学说的一个良好范例[7]。Pi-ta编码一个长度为928个氨基酸的细胞质膜受体蛋白,该蛋白含核苷酸结合位点结构域(NBS)和富亮氨酸结构域(LRD)。AVR-Pita编码一个长度为223个氨基酸的金属蛋白酶,该蛋白酶能与抗性基因Pi-ta产物直接作用,引发水稻对稻瘟病的抗性[8]。Pi-ta存在多态现象,其编码产物也分为多个单倍型,而其中只有一种单倍型是抗病的。一个关键位点氨基酸的不同决定了抗病单倍型和感病单倍型之间的差异,即第918位氨基酸(位于LRD区)由丙氨酸突变为丝氨酸。抗病型的Pita蛋白(918为丙氨酸)能与稻瘟菌无毒基因AVR-Pita编码产物相互作用,引起水稻防卫响应和细胞程序性死亡,从而免受稻瘟病菌侵染。而感病型的Pita蛋白(918为丝氨酸)则不能与AVR-Pita编码产物相互作用,导致感病[6, 8]。Pita蛋白918氨基酸的改变是由于Pi-ta的DNA序列第6 640位点(GenBank No. AF207842)的突变引起的,当该位点核苷酸为G时,所在密码子编码丙氨酸;当该位点为T时,所在密码子编码丝氨酸[9]。有研究报道,Pi-ta抗性单倍型连锁模块至少5.4 Mb,远远长于感病的Pi-ta单倍型连锁模块(2.2 Mb),这说明Pi-ta编码区以外的不翻译区与Pi-ta的抗性是有关联的[10]。真核细胞成熟mRNA通常包括一个5'非翻译区(5'-UTR)、编码区(coding region)和3'非翻译区(3'-UTR)。虽然UTR区域不编码氨基酸,但却在基因的表达调控方面起着重要的作用。在转录后调控中,mRNA的非编码区(包括5'-UTR和3'-UTR,尤其是3'-UTR)决定了mRNA的翻译速度、稳定性、亚细胞定位、降解等一系列特征[11-13]。先前研究表明,在Pi-ta所有抗性单倍型的5'-UTR序列中,位于起始密码子上游1 012 bp处皆有一个3 364 bp的插入片段,经克隆和测序发现该插入片段是一段转座子序列(GenBank accession No. GQ984160)[14],进一步说明UTR区可能具有对Pi-ta功能调控的作用。【本研究切入点】如上所述,Pi-ta编码区与抗性功能之间的关系已经有了很多的研究,在这些研究中虽然已有不少迹象表明Pi-ta的非编码区,尤其是3'-UTR,也可能参与了Pi-ta的抗性决定作用,但Pi-ta的3′-UTR与抗性功能之间的相关性研究却鲜见报道。在遗传多样性高的稻种资源中分析Pi-ta的3'-UTR的遗传多态,有助于发掘3'-UTR和Pi-ta抗性功能之间的关联。云南位于中国的西南部,具有地形复杂、气候变化大等特点,使得稻作物资源非常丰富。不同的民俗和文化在一定程度上也影响着农民对水稻品种栽培的选择和稻米品味的需求,因而造就了云南水稻地方品种具有丰富遗传多样性的特点[15]。【拟解决的关键问题】本研究拟在云南水稻地方品种中开展Pi-ta的3′-UTR遗传多态的研究,分析Pi-ta 3'-UTR区的遗传多态,通过单倍型分布以及和关键抗病位点的关联来探讨3′-UTR与Pi-ta抗性功能之间的相关性,研究结果将有利于详细了解Pi-ta的抗性决定机制,为培养更持久的抗性品种提供依据。1 材料与方法
1.1 材料选择与育苗
对云南地方水稻品种进行种子收集,为获取更全面的遗传多态信息,收集原则就是在遗传多样性丰富的地区尽可能多地囊括不同的品种。共收集到137个云南地方水稻品种。对种子进行消毒(消毒方法:1.5%次氯酸钠溶液消毒5 min后,用灭菌水洗涤种子10次以上),然后在培养箱内28℃水培发芽;随后移植到温室内的育苗盆中以土壤(水稻土、腐殖土、草木灰的混合)为基质进行种植约20 d(三叶一心期)。1.2 采样
采集三叶一心期的水稻幼苗植株置于15 mL灭菌的离心管中,液氮速冻后置于-80℃保存备用。1.3 DNA提取及质检
使用基因组DNA纯化试剂盒(Promega,A1120),根据操作说明,提取水稻幼苗DNA并纯化。2%琼脂糖凝胶电泳检测DNA的完整性(试验使用电泳仪电源和电泳槽厂家为北京六一,使用电压120 V,时间30 min),最后所得基因组DNA条带单一,无拖带,代表基因组DNA完整性良好。采用Nanodrop ND 1000分光光度计(厂家:美国NanoDrop)和2 μL DNA原液进行OD260/OD280比值和基因组DNA浓度的测量计算。OD260/OD280比值在1.7—2.0代表纯度良好,DNA浓度按计算值稀释到约20 ng·μL-1。1.4 Pi-ta的3'-UTR序列扩增区域选择及引物设计
基于Pi-ta(UniGene No. Os12g0281300)已发表的mRNA序列和3' EST序列(GenBank access No.见电子版附表1),确定了Pi-ta的3'-UTR长度。用3′-末端带有poly(A)的EST与全长cDNA序列进行比对,得出Pi-ta的3'-UTR的DNA编码区长度为1.1 kb。为了研究Pi-ta的3'-UTR区的遗传多态,根据终止密码子处第6 675—7 830这一段约1.2 kb的DNA序列设计引物,可以完整覆盖3'-UTR的长度。同时,为分析3'-UTR是否与Pi-ta关键功能位点6 640之间存在一定的联系,同时还扩增了第6 640处到终止密码子第 6 675处这一段的序列。所用引物信息如表1所示。
Table 1
表1
表1PCR扩增引物和测序引物
Table 1Primers for amplification and sequencing
| 引物名称 Primer name | 引物序列 Primer sequence (5′-3′) | 定位 Location | 产物长度 Product length (bp) |
|---|---|---|---|
| P918-1-R | CCACACCTTCTAGGTACCCG | 6861—6842 | |
| P918-2-F | CCGACTCTCGCTTCACTTCA | 5613—5632 | 1266 |
| P918-2-R | GGAGGTAAGACTAGGAACCACAC | 6878—6856 | |
| P6737-F | CCGGAATCCTCTTCACGTCT | 6737—6756 | 1116 |
| P7852-R | TGTTGCAAGGGTAATCAGAAGAA | 7852—7830 |
新窗口打开
1.5 PCR扩增及测序
PCR扩增采用的反应体系为10×PCR反应缓冲液2.8 µL、2.5 mmol·L-1 dNTP 1.0 µL、BSA 1.0 µL、前引物0.8 µL、后引物0.8 µL、r-taq酶0.25 µL、20 ng·µL-1、DNA为1.0 µL,加水至总体积为25 µL。将PCR反应体系瞬时离心,放入PCR仪(厂家:美国ABI,型号:veriti96)。PCR运行程序为94℃ 3 min;94℃ 30 s,60℃ 30 s,72℃ 1 min,循环33次;72℃ 10 min。扩增结束后,将PCR产物进行1.5%琼脂糖凝胶电泳检测,120 V恒定电压下电泳30 min,关闭电源,凝胶成像系统拍照并对带型进行分析。引物对P918-2-F/P918-2-R和引物对P6737-F/P7852R扩增出的产物均为条带单一的PCR产物。对PCR产物进行纯化、测序,共计测序137个品种(双向测序,共274条序列)。测序引物除PCR所用双向引物外,又分别增加了测序引物为P918-1-R和P6737-F(表1)。
所测样本采用DNASTAR Lasergene v7.1.0软件进行序列的拼接和比对,统计比对差异。以最早报道的Pi-ta序列(GenBank序列号:AF207842)为参考序列,输出变异位点。SNP位点的分析和单倍型分析采用软件DnaSP 4.9[16]。基于不同单倍型之间谱系关系的单倍型网络图构建采用TCS 1.21软件[17]。
2 结果
2.1 Pi-ta的3'-UTR区序列数据
以云南137个水稻地方品种为研究对象,测定了Pi-ta的3'末端编码区(覆盖6640抗性功能位点)和完整的不翻译区的DNA序列。序列结果已投递到GenBank(http://www.ncbi.nlm.nih.gov/Genbank/),序列号为KX752434-KX752570。Pi-ta的3'-UTR区较长,约为1120 bp,是植物基因3'-UTR平均长度(200 bp)的5倍多[18],这可能预示着Pi-ta的3'-UTR具有特定的功能。此外,Pi-ta的3'-UTR的G+C含量相对较低,为40.43%,这也符合3'-UTR的长度与其自身的G+C含量呈负相关的研究报道[18]。在137个水稻地方品种中,所有品种的Pi-ta的3'-UTR编码区长度一致,不存在插入或缺失导致的长度多态性。2.2 3'-UTR区序列特征分析
有研究表明水稻基因的3'-UTR通常不存在保守的polyA位点[19],在Pi-ta的3'-UTR也没有发现保守的polyA位点(AATAAA),但在Pi-ta的3'-UTR序列中却存在多个非保守的潜在polyA位点(图1)。此外,Pi-ta的3'-UTR区域还存在非常高频率的TTTT序列(总计12处,图1),有研究报道这种尿嘧啶原件是一种高度保守的序列结构,这些结构可能与转录终止或者与3′端添加多聚A顺序有关[20],这种高频率的TTTT序列从另一个侧面提示Pi-ta转录终止时具有复杂的调控机制。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1富含尿嘧啶原件的Pi-ta的3'-UTR序列
-->Fig. 1Diagram of Pi-ta gene 3'-UTR coding sequence which is rich in uracil elements
-->
2.3 3'-UTR区单倍型分布
获得137个云南水稻品种的3'-UTR区序列,所有序列均是双向测序,序列经过比对后,以GQ984160为参考序列检出所有品种的SNP变异位点。对Pi-ta的3'-UTR中存在的所有SNP进行了分析,结果表明,3'-UTR呈现出高度多态性,长为1.1 kb的3'-UTR区域共有12个SNP位点,由这些SNP可将137个品种分成7个单倍型。单倍型之间没有重组的信号。为了进一步分析3'-UTR与Pi-ta的抗性功能之间的关系,还测定了所有品种Pi-ta的3'末端第6 640处抗性功能位点所在的编码区序列。如前所述,第6 640处位点G与T的差别直接决定该品种对持有功能性Avr-Pita菌株的抗感性,通过对联合3'-UTR单倍型分布和关键功能位点第6 640处的状态进行分析(表2),表明3'-UTR的SNP高度多态性都出现在感病品种中,所有携带6640G的抗性品种都属于一种3'-UTR单倍型(图2中单倍型H-2)。之前的研究发现Pi-ta编码区目前也只有一种抗性单倍型[9],即携带6640G的抗性单倍型。如今所有6640G所在的基因序列其3'-UTR序列也只存在一种单倍型(参见序列:KX752434- KX752570),这表明3'-UTR与其编码区是紧密关联的,在功能上和所受到的选择压力方面是连续和一致的。抗性品种在Pi-ta的编码区和3'-UTR维持单一单倍型(单倍型H-2),而感病品种在这个区域出现了六种单倍型,呈现高度的多态性。造成这一现象可能的原因是,存在一种强烈的选择压力来维持抗性单倍型(包括编码区和3'-UTR)的唯一性,这对于抗性功能可能是必须的。Table 2
表2
表2云南地方水稻品种的抗性反应和6640位点鉴定
Table 2Disease reaction and nucleotide status in 6640 of 137 rice landraces from Yunnan
| 样本编号 Samples No. | 品种名 Landraces name | 6640位点 Nucleotide 6640 | 抗性鉴定 Reaction to O-137 | 抗病指数 RF (%) | 3'-UTR单倍型 3'-UTR haplotype |
|---|---|---|---|---|---|
| YN-1 | 毛香糯 Maoxiangnuo | T | R | 100.0 | Hap_1 |
| YN-2 | 细红谷 Xihonggu | G | R | 100.0 | Hap_2 |
| YN-3 | 红谷 Honggu | G | R | 84.6 | Hap_2 |
| YN-4 | 黑壳糯 Heikenuo | T | R | 100.0 | Hap_3 |
| YN-5 | 长红谷 Changhonggu | G | R | 75.0 | Hap_2 |
| YN-6 | 细黄壳糯 Xihuangkenuo | T | R | 92.9 | Hap_1 |
| YN-7 | 大勐卯 Damengmao | T | R | 92.3 | Hap_3 |
| YN-8 | 大六方谷 Daliufanggu | G | R | 100.0 | Hap_2 |
| YN-9 | 梁河糯谷 Lianghenuogu | T | S | 18.2 | Hap_3 |
| YN-10 | 毫木西变种 Haomuxi-variant | T | R | 84.6 | Hap_3 |
| YN-11 | 团紫糯 Tuanzinuo | T | S | 27.3 | Hap_4 |
| YN-12 | 紫糯谷 Zinuogu | T | S | 25.0 | Hap_3 |
| YN-13 | 凉粉谷 Liangfengu | T | R | 100.0 | Hap_5 |
| YN-14 | 德优 Deyou | T | S | 33.3 | Hap_3 |
| YN-15 | 红壳糯 Hongkenuo | T | R | 100.0 | Hap_5 |
| YN-16 | 大谷 Dagu | T | S | 38.5 | Hap_1 |
| YN-17 | 黄皮谷 Huangpigu | T | S | 9.1 | Hap_3 |
| YN-18 | 银丰糯 Yinfengnuo | T | R | 92.3 | Hap_3 |
| YN-19 | 团六方 Tuanliufang | T | S | 30.8 | Hap_1 |
| YN-20 | 杂交糯 Hybrid-nuo | T | R | 76.9 | Hap_3 |
| YN-21 | 502变种 502-variant | T | S | 27.3 | Hap_3 |
| YN-22 | 可然考兰 Kerankaolan | T | R | 100.0 | Hap_1 |
| YN-23 | 团力 Tuanli | T | R | 85.7 | Hap_5 |
| YN-24 | 凹录 Aolu | T | S | 36.4 | Hap_5 |
| YN-25 | 紫糯 Zinuo | T | R | 100.0 | Hap_4 |
| YN-26 | 香糯 Xiangnuo | T | R | 100.0 | Hap_3 |
| YN-27 | 版纳小软 Bannaxiaoruan | T | R | 100.0 | Hap_3 |
| YN-28 | 红米谷 Hongmigu | G | R | 100.0 | Hap_2 |
| YN-29 | 长白糯 Changbainuo | T | R | 92.3 | Hap_3 |
| YN-30 | 大白谷 Dabaigu | T | S | 15.4 | Hap_3 |
| YN-31 | 团糯谷 Tuannuogu | T | R | 91.7 | Hap_1 |
| YN-32 | 矮杆201 Aigan201 | T | R | 78.6 | Hap_3 |
| YN-33 | —五 Yiwu | T | S | 16.7 | Hap_3 |
| YN-34 | 高杆201 Gaogan201 | T | S | 36.4 | Hap_6 |
| YN-35 | 老品种201 Laopinzhong201 | T | R | 100.0 | Hap_3 |
| YN-36 | 陇川大白 Longchuandabai | T | R | 78.6 | Hap_3 |
| YN-37 | 王子树谷 Wangzishugu | T | S | 27.3 | Hap_1 |
| YN-38 | 起采谷 Qicaigu | G | R | 100.0 | Hap_2 |
| YN-39 | 矮脚糯 Aijiaonuo | T | R | 81.8 | Hap_1 |
| YN-40 | 句谷 Jugu | T | R | 80.0 | Hap_1 |
| YN-41 | 灰背谷 Huibeigu | T | S | 27.3 | Hap_4 |
| YN-42 | 梁河大白 Lianghedabai | T | R | 84.6 | Hap_3 |
| YN-43 | 耿马糯 Gengmanuo | T | R | 92.3 | Hap_4 |
| YN-44 | 德国谷 Deguogu | G | R | 100.0 | Hap_2 |
| YN-45 | 毛香糯 Maoxiangnuo | T | R | 100.0 | Hap_3 |
| YN-46 | 老凹糯 Laoaonuo | G | R | 100.0 | Hap_2 |
| YN-47 | 长灰背谷 Changhuibeigu | G | R | 100.0 | Hap_2 |
| YN-48 | 恩来 Enlai | T | S | 27.3 | Hap_1 |
| YN-49 | 黄壳糯 Huangkenuo | T | R | 100.0 | Hap_1 |
| YN-50 | 旱坝糯 Hanbanuo | T | R | 92.9 | Hap_1 |
| YN-51 | 瓦底糯 Wadinuo | T | R | 100.0 | Hap_3 |
| YN-52 | 大白谷 Dabaigu | T | R | 100.0 | Hap_1 |
| YN-53 | 大红谷 Dahonggu | T | S | 38.5 | Hap_1 |
| YN-54 | 矮中籼 Aizhongxian | G | R | 100.0 | Hap_2 |
| YN-55 | 单选糯 Danxuannuo | T | R | 93.3 | Hap_1 |
| YN-56 | 户撒糯 Husanuo | T | R | 92.9 | Hap_4 |
| YN-57 | 黄壳糯 Huangkenuo | T | R | 85.7 | Hap_1 |
| YN-58 | 小红谷 Xiaohonggu | T | S | 42.9 | Hap_7 |
| YN-59 | 灰谷 Huigu | T | R | 93.3 | Hap_1 |
| YN-60 | 白壳糯 Baikenuo | T | S | 31.3 | Hap_3 |
| YN-61 | 白糯 Bainuo | G | R | 100.0 | Hap_2 |
| YN-62 | 老鼠牙 Laoshuya | T | R | 100.0 | Hap_3 |
| YN-63 | 龙江糯 Longjiangnuo | T | R | 100.0 | Hap_4 |
| YN-64 | 长毛糯 Changmaonuo | T | R | 90.9 | Hap_4 |
| YN-65 | 晚镇安谷 Wanzhenangu | T | S | 25.0 | Hap_4 |
| YN-66 | 银粳 Yingeng | T | R | 100.0 | Hap_1 |
| YN-67 | 矮杆冬 Aaigandong | G | R | 85.7 | Hap_2 |
| YN-68 | 早镇安谷 Zaozhenangu | T | R | 81.8 | Hap_1 |
| YN-69 | 麻谷 Magu | T | R | 100.0 | Hap_7 |
| YN-70 | 金裹银 Jinguoyin | T | R | 100.0 | Hap_3 |
| YN-71 | 黑嘴谷 Heizhuigu | G | R | 100.0 | Hap_2 |
| YN-72 | 尖嘴白 Jianzhuibai | T | R | 87.5 | Hap_3 |
| YN-73 | 小红谷 Xiaohonggu | T | R | 84.6 | Hap_1 |
| YN-74 | 毫比相糯 Haobixiangnuo | G | R | 100.0 | Hap_2 |
| YN-75 | 盐霜糯 Yanshuangnuo | T | S | 30.8 | Hap_1 |
| YN-76 | 毫比相弄 Haobixiangnong | T | S | 36.4 | Hap_3 |
| YN-77 | 毫比相 Haobixiang | T | S | 33.3 | Hap_5 |
| YN-78 | 遮放红谷 Zhefanghonggu | T | R | 100.0 | Hap_5 |
| YN-79 | 傣珍 Daizhen | G | R | 93.3 | Hap_2 |
| YN-80 | 傣珍红谷 Daizhenhonggu | T | R | 100.0 | Hap_3 |
| YN-81 | 黄壳软谷 Huangkeruangu | G | R | 100.0 | Hap_2 |
| YN-82 | 毫木细矮杆 Haomuxiaigan | G | R | 92.9 | Hap_2 |
| YN-83 | 白壳糯 Baikenuo | T | R | 83.3 | Hap_3 |
| YN-84 | 花壳糯 Huakenuo | T | R | 100.0 | Hap_3 |
| YN-85 | 黄壳谷 Huangkegu | G | R | 100.0 | Hap_2 |
| YN-86 | 梁河黄板所 Lianghehuangbansuo | T | S | 42.9 | Hap_3 |
| YN-87 | 黄壳糯 Huangkenuo | T | R | 100.0 | Hap_5 |
| YN-88 | 毫木吕 Haomulv | T | S | 8.3 | Hap_3 |
| YN-89 | 黄瓜糯 Huangguanuo | T | S | 36.4 | Hap_3 |
| YN-90 | 长白谷 Changbaigu | T | R | 100.0 | Hap_3 |
| YN-91 | 白早谷 Baizaogu | G | R | 100.0 | Hap_2 |
| YN-92 | 大白谷 Dabaigu | T | R | 100.0 | Hap_5 |
| YN-93 | 黄长细谷 Huangchangxigu | G | R | 100.0 | Hap_2 |
| YN-94 | 大白软 Dabairuan | T | R | 84.6 | Hap_5 |
| YN-95 | 长白谷 Changbaigu | T | S | 33.3 | Hap_3 |
| YN-96 | 细长白谷 Xichangbaigu | T | S | 33.3 | Hap_3 |
| YN-97 | 高杆细长谷 Gaoganxichanggu | T | R | 100.0 | Hap_3 |
| YN-98 | 晚白早 Wanbaizao | T | S | 33.3 | Hap_3 |
| YN-99 | 红细长谷 Hongxichanggu | T | R | 100.0 | Hap_3 |
| YN-100 | 长白早谷 Changbaizaogu | T | S | 30.8 | Hap_3 |
| YN-101 | 白谷 Baigu | T | R | 84.6 | Hap_3 |
| YN-102 | 黄壳糯 Huangkenuo | T | R | 90.0 | Hap_3 |
| YN-103 | 红谷 Honggu | G | R | 100.0 | Hap_2 |
| YN-104 | 傣族谷 Daizugu | T | R | 73.3 | Hap_3 |
| YN-105 | 毫木吕变种 Haomulv-variant | T | S | 27.3 | Hap_3 |
| YN-106 | 毫木吕变种 Haomulv-variant | G | R | 100.0 | Hap_2 |
| YN-107 | 香软谷 Xiangruangu | G | R | 100.0 | Hap_2 |
| YN-108 | 小勐卯 Xiaomengmao | T | S | 33.3 | Hap_3 |
| YN-109 | 小黄糯 Xiaohuangnuo | T | S | 27.3 | Hap_3 |
| YN-110 | 紫格糯 Zigenuo | T | S | 38.5 | Hap_3 |
| YN-111 | 细红谷 Xihonggu | G | R | 100.0 | Hap_2 |
| YN-112 | 烂地糯 Landinuo | T | R | 71.4 | Hap_3 |
| YN-113 | 烂地糯 Landinuo | T | R | 93.3 | Hap_3 |
| YN-114 | 紫糯 Zinuo | T | S | 30.0 | Hap_4 |
| YN-115 | 文奥谷 Wenaogu | T | S | 30.8 | Hap_3 |
| YN-116 | 王八谷 Wangbagu | T | S | 35.7 | Hap_5 |
| YN-117 | 红米谷 Hongmigu | G | R | 100.0 | Hap_2 |
| YN-118 | 小白谷 Xiaobaigu | G | R | 100.0 | Hap_2 |
| YN-119 | 红心糯 Hongxinnuo | T | S | 18.2 | Hap_3 |
| YN-120 | 大白调 Dabaidiao | G | R | 100.0 | Hap_2 |
| YN-121 | 黄板所 Huangbansuo | T | R | 100.0 | Hap_3 |
| YN-122 | 白团棵 Baituanke | G | R | 100.0 | Hap_2 |
| YN-123 | 花谷 Huagu | T | R | 91.7 | Hap_5 |
| YN-124 | 细之谷 Xizhigu | G | R | 100.0 | Hap_2 |
| YN-125 | 紫杆糯 Zigannuo | G | R | 100.0 | Hap_2 |
| YN-126 | 长白调 Changbaidiao | T | S | 0.0 | Hap_3 |
| YN-127 | 红细牙谷 Hongxiyagu | G | R | 100.0 | Hap_2 |
| YN-128 | 大白皮糯 Dabaipinuo | T | S | 8.3 | Hap_5 |
| YN-129 | 南高谷 Nangaogu | G | R | 100.0 | Hap_2 |
| YN-130 | 冷水白谷 Lengshuibaigu | G | R | 100.0 | Hap_2 |
| YN-131 | 红长毛 Hongchangmao | T | R | 100.0 | Hap_5 |
| YN-132 | 早白谷 Zaobaigu | G | R | 100.0 | Hap_2 |
| YN-133 | 细谷 Xigu | G | R | 100.0 | Hap_2 |
| YN-134 | 昌宁大白 Changningdabai | G | R | 100.0 | Hap_2 |
| YN-135 | 满冬谷 Mandonggu | T | R | 91.7 | Hap_3 |
| YN-136 | 黄壳油粘 Huangkeyounian | T | R | 85.7 | Hap_3 |
| YN-137 | 小药谷 Xiaoyaogu | G | R | 100.0 | Hap_2 |
新窗口打开
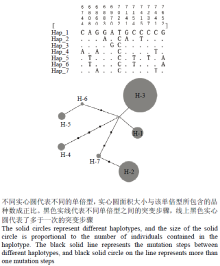 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2根据3'-UTR编码序列SNP(上)位点构建的单倍型网络图(下)
-->Fig. 2Haplotype network constructed based on 3'-UTR coding sequence SNPs
-->
3 讨论
近年来,Pi-ta编码区的多态与进化已有了较多的研究,但Pi-ta非编码区与抗性相关的转录和调控机制还有很多方面没有研究清楚。3'-UTR是microRNA的结合区域,对基因的转录水平调控具有重要的作用。如果突变位点位于mRNA 3'-UTR区,就会影响microRNA对其结合,导致mRNA降解受阻,mRNA表达上升。在云南水稻地方品种中的感病品种其Pi-ta的3'-UTR呈现出高度多态性,而抗病品种的Pi-ta的3'-UTR则只有一个单倍型。这说明3'-UTR的特定序列(单倍型H-2)对于Pi-ta的抗性功能是必需的,容不得突变的产生。此外,下载了世界其他地区的148条水稻Pi-ta序列,分析的结果与在云南水稻里的分析结果是一致的,也即是,携带6640G的抗性品种的3'-UTR也只有一个单倍型。PolyA位点是在mRNA 3'末端添加多聚腺苷酸的信号位点,Pi-ta的3'-UTR区不存在保守的polyA位点(AATAAA),却存在多个潜在polyA位点,这可能导致Pi-ta基因转录本(尤其是3'-UTR)的复杂多变。根据现有的mRNA和3′ EST序列数据分析表明,Pi-ta确实有许多多态性的转录本,COSTANZO和JIA利用已发表的基因组信息,预测Pi-ta至少有12种不同的转录本[21]。
对Pi-ta现已报道的不同mRNA转录本的分析表明,该基因转录终止的位置呈现多样化(图3),同时,该基因3'-UTR对应于编码区序列时可以是连续的,也可以是间断的,呈现选择性拼接的模式(图3和电子版附表1)。WHITHAM等[22]研究结果表明抗性基因不同的选择性拼接方式,可能影响着基因对基因学说中抗性基因的功能。Pi-ta的3'-UTR这种不同的选择性拼接方式很明显会影响microRNA与3'-UTR的结合,这说明3'-UTR区域可能以选择性拼接的方式直接参与了Pi-ta复杂的转录水平表达调控。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3Pi-ta全长(7 830 bp)及不同转录本3'-UTR区域在基因编码区的对应情况示意图
-->Fig. 3Diagram of the full length of Pi-ta gene (7 830 bp) and the corresponding mapping of different transcripts 3'-UTR in the gene coding region
-->
4 结论
感病水稻品种的Pi-ta的3'-UTR呈现高度多态,而携带6640G的抗性水稻品种的Pi-ta的3'-UTR没有检测到多态性,维持单一单倍型,这表明3'-UTR对于Pi-ta的抗性功能具有连锁的调控作用,受到纯净化选择压力。此外,选择性拼接可能也是3'-UTR参与Pi-ta转录调控的一种方式。3'-UTR的特定类型是抗性功能所不可或缺的一部分,这也提示在研制广谱抗性品种引入Pi-ta时需要保证其3'-UTR区也必须是抗性单倍型的。Table S1
附表 1
附表 1Pi-ta的mRNA序列和3’ EST序列信息表
Table S1 The information of mRNA sequences and 3’ EST sequences from Pi-ta gene
| GenBank登记号 GenBank access No. | 来源 Origin | 长度(bp) Length |
|---|---|---|
| AU085774 | mRNA sequence | len=668 |
| AU063103 | mRNA sequence | len=464 |
| AK071926 | cDNA clone | len=3448 |
| AK067534 | cDNA clone | len=2654 |
| AK066558 | cDNA clone | len=4038 |
| CI119026 | 3'EST | len=532 |
| CI123654 | 3'EST | len=425 |
| CI357159 | 3'EST | len=366 |
| CI338221 | 3'EST | len=463 |
| CI329103 | 3'EST | len=433 |
| CI323554 | 3'EST | len=485 |
| CI524574 | 3'EST | len=398 |
| CI418837 | 3'EST | len=395 |
| CI419326 | 3'EST | len=430 |
| CI429029 | 3'EST | len=449 |
| CI439303 | 3'EST | len=440 |
| CI439915 | 3'EST | len=413 |
| CI441199 | 3'EST | len=415 |
| CI445081 | 3'EST | len=427 |
| CI445124 | 3'EST | len=430 |
| CI445511 | 3'EST | len=427 |
| CI446863 | 3'EST | len=427 |
| CI448717 | 3'EST | len=422 |
| NM_001073106 | mRNA, complete cds | len=3447 |
| GQ463476 | mRNA, partial | len=566 |
| GQ463474 | mRNA, complete cds | len=2357 |
| GQ463472 | mRNA, partial | len=670 |
| GQ463471 | mRNA, partial | len=369 |
| GQ463470 | mRNA, partial | len=440 |
| GQ463469 | mRNA, partial | len=936 |
| GQ463468 | mRNA, complete cds | len=3395 |
| GQ463467 | mRNA, complete cds | len=3369 |
新窗口打开
The authors have declared that no competing interests exist.
