0 引言
【研究意义】光是作物生长发育所需的最重要生态因子之一,是作物产量形成的基础,同时光也可作为一种重要的环境信号,调节植物基因的表达、影响酶的活性以及植物的形态建成。在光信号途径中,光敏色素互作因子起着承上启下的作用,因此,利用CRISPR/Cas9基因编辑技术创制光敏色素互作因子OsPIL15的突变体,研究其在光信号调控途径中的作用,对挖掘水稻功能基因,丰富和完善水稻光信号调控分子机制具有重要意义。【前人研究进展】基因编辑技术指能够对目标基因进行“编辑”,实现对特定DNA片段的敲除、插入等。过去几年,以锌指核酸酶(zinc fnger nucleases,ZFNs)和类转录激活因子效应物核酸酶(transcription activator-like nucleases,TALENs)为代表的序列特异性核酸酶技术可以高效率的定点编辑基因组,在基础研究、基因治疗和遗传改良等方面发挥了重要作用[1-2]。近年来,一种名为CRISPR/Cas9(clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9,Cas9)的基因组定向编辑技术备受瞩目。2013年初,《Science》两篇文章首次报道了Cas9核酸酶用于人类和小鼠细胞基因组编辑[3-4],其作为第三代基因编辑技术受到人们广泛关注。CRISPR/Cas9技术是利用核酸酶Cas9蛋白与单导向RNA(single guide RNA,sgRNA)形成复合体,sgRNA通过碱基互补配对决定靶序列特异性,Cas9蛋白作为核酸酶切割与sgRNA上的间隔序列(spacers)互补的基因组DNA,造成双链DNA损伤,随后通过体内的NHEJ(non-homologous end joining)修复机制引入基因突变[5]。随着CRISPR/ Cas9系统在人类和动物细胞中建立与应用,CRISPR/ Cas9系统在拟南芥、烟草、水稻、小麦、玉米、高粱、番茄和甜橙等植物中均实现了基因组的定向编辑[6-7]。光敏色素互作因子PIF(phytochrome-interacting factor)或称PIL(phytochrome interacting factor-like)是bHLH转录因子家族中的一类转录因子,其主要特性是能与光敏色素互作直接或间接调控光响应基因。作为bHLH蛋白的一种,所有PIF家族的蛋白都包含在N端与光敏色素互作的APB(active phytochrome B-binding)或APA(active phytochrome A-binding)结构域和C端bHLH-DNA结合结构域及核定位结构域[8-9]。拟南芥PIF能够与靶基因启动子区域的G-box(5′-CACGTG-3′)或PBE-box(PIF- binding E-box, 5′-CACATG-3′)相结合[10],可调控种子萌发[11]、幼苗去黄化[12]、避阴反应[13]和昼夜节律[14]等植物生长发育的各个方面。NAKAMURA等[15]通过同源性分析在水稻基因组中鉴定了6个PIF转录因子(OsPIL11—OsPIL16),相对于拟南芥而言,PIFs在水稻生长发育过程中的作用研究相对较少。OsPIL11参与红光诱导的幼苗去黄化反应[16];OsPIL13在水稻中超表达后能够促进水稻节间的伸长,反之低表达则抑制其节间伸长[17];超表达OsPIL15的水稻种子在黑暗环境中的萌发受到抑制[18];OsPIL16能够负调控PGL1进而调控籽粒大小[19]。【本研究切入点】笔者前期通过高通量测序研究发现,在水稻籽粒抽穗后10、15、21、27和35 d这5个灌浆时期的强势粒和弱势粒中,6个水稻光敏色素互作因子有3个被鉴定出,其中OsPIL12和OsPIL13只在弱势粒某个时期表达,OsPIL15则在强弱势粒的5个灌浆时期均表达[20],而目前有关OsPIL15在水稻籽粒灌浆中的调控作用还不清楚。【拟解决的关键问题】本研究拟利用CRISPR/Cas9系统对水稻光敏色素互作因子OsPIL15进行编辑,转化粳稻品种日本晴,对其突变位点和潜在脱靶位点进行分析鉴定,通过对ospil15突变体表型进行观察,了解OsPIL15功能及其调控路径。1 材料与方法
1.1 试验材料
使用粳稻品种日本晴(Oryza sativa ssp. Japonica cv. Nipponbare)作为转基因受体材料,CRISPR/Cas9载体pBUN411由中国农业大学陈其军教授惠赠[21],试验所用引物(表1)和测序分析由苏州金唯智生物科技有限公司完成。Table 1
表1
表1本研究所用引物
Table 1Primers used in this study
| 引物名称 Primer name | 引物序列 Primer sequence (5′-3′) | 用途 Usage |
|---|---|---|
| sgRNA-F | ggcgGACTTCTTCTCCGAGCTCC | 靶序列合成 Construction of target site |
| sgRNA-R | aaacGGAGCTCGGAGAAGAAGTC | |
| pBUN411-VF | CCATGAAGCCTTTCAGGACATGTA | 菌落PCR验证 Colony PCR verification |
| pBUN411-VR | ACGCTGCAAACATGAGACGGAGAA | |
| Bar-F | AAGCACGGTCAACTTCCGTA | 抗除草剂基因验证 PCR detection of herbicide resistance gene |
| Bar-R | GAAGTCCAGCTGCCAGAAAC | |
| OsPIL15-F | TGTTTTGTGTGTGCAGGTCC | 靶点序列扩增 Amplification of target region |
| OsPIL15-R | CGGGAGAAGAGCGAGAAGTT | |
| Off-target1-F | AATCCAACGCGAGATCAAGC | 潜在脱靶位点扩增 Off target amplification |
| Off-target1-R | CCTTCCGCCCTAGAGAACAA | |
| Off-target2-F | TTGATGGTGATGTTGGTGGC | 潜在脱靶位点扩增 Off target amplification |
| Off-target2-R | GGTTGTTGTAGGCGAGGTTG | |
| Off-target3-F | TACATGGAGGACTACGAGGC | 潜在脱靶位点扩增 Off target amplification |
| Off-target3-R | CATGCTGAAGAACGCCGTG | |
| Off-target4-F | TCTGCACCATATCCCACCTC | 潜在脱靶位点扩增 Off target amplification |
| Off-target4-R | CCACGACCAAACCTATGCAG | |
| Off-target5-F | GATGCGTGAGAGGATGATGC | 潜在脱靶位点扩增 Off target amplification |
| Off-target5-R | CTGTGTTTAGTCCGTGTGCC |
新窗口打开
1.2 靶位点设计和CRISPR/Cas9表达载体构建
根据水稻OsPIL15(GenBank登录号AK102252.1)外显子序列设计sgRNA靶点序列。在尽量靠近外显子编码蛋白质的N端,选取PAM(protospacer-adjacent motif)序列(NGG)5′端的一段碱基序列作为靶位点,将该段序列在水稻基因组数据库中进行比对,以排除非特异性靶位点,同时尽可能使PAM 5′端存在常用酶切位点,以方便后期突变体鉴定。将选取的靶点序列作为正义链,其互补链作为反义链,分别在2条DNA单链5′端添加ggcg和aaac序列作为BsaⅠ限制性内切酶的黏性末端接头。使用限制性内切酶BsaⅠ酶切CRISPR/Cas9载体pBUN411质粒使其线性化,T4 DNA连接酶连接后用热激法转化大肠杆菌DH5α感受态细胞,使用引物pBUN411- VF和pBUN411-VR进行菌落PCR验证。CRISPR/Cas9表达载体转化农杆菌EHA105,用除草剂(Basta)筛选获得再生苗。
1.3 酶切和测序分析验证突变位点
为了检测靶位点的突变情况,在靶位点两侧设计引物OsPIL15-F和OsPIL15-R,以转基因阳性单株DNA为模板扩增靶位点序列。使用SacⅠ限制性内切酶酶切PCR产物,2%琼脂糖凝胶电泳检测是否发生突变。将突变单株PCR产物测序,对靶位点附近出现套峰的PCR产物进行TA克隆,随机选取单克隆测序,测序结果与野生型序列比对分析确定突变基因型。1.4 潜在脱靶位点分析
使用The Rice Annotation Project(http://rapdb.dna. affrc.go.jp/)在线BLAST工具,将OsPIL15靶序列在水稻全基因组中进行比对,选取与靶序列同源性较高、错配在4 bp以内且具有PAM序列NGG的位点作为潜在脱靶序列,通过酶切法评估其脱靶效应。2 结果
2.1 OsPIL15靶点设计和表达载体构建
根据CRISPR/Cas9技术原理,在OsPIL15外显子区域靠近起始密码ATG区域选择一段19 bp的序列(5′-GACTTCTTCTCCGAGCTCC-3′)作为靶位点。该序列3′端PAM序列为AGG,Cas9蛋白将在AGG序列上游第3—4 bp处剪切DNA形成平滑末端,该剪切处有一段限制性内切酶SacⅠ识别序列(5′-GAGCT▼C-3′),选择能被限制性内切酶识别并切割的位点进行突变,有利于后续对转基因阳性植株是否发生突变进行筛选。CRISPR/Cas9表达载体的靶序列由OsU3启动子驱动,编码Cas9蛋白基因由玉米泛素基因(Ubi)启动子驱动。2.2 OsPIL15 T0代突变体筛选鉴定
使用引物OsPIL15-F和OsPIL15-R扩增25株阳性转基因单株靶点序列(图1-A),野生型扩增片段大小为673 bp,单株1、2、23和24均扩增出片段明显小于野生型的条带,初步表明这4个单株可能发生较大片段缺失,其中单株1和24的PCR产物出现两条带,只有其中一条带明显小于野生型条带,因此二者可能只有一条同源染色体有较大片段缺失;单株2和23的PCR产物只有一条带且明显小于673 bp,可初步判断二者两条同源染色体都发生较大片段缺失。其余21个单株不能根据条带大小判断是否突变,因此,利用酶切法继续进行鉴定。使用SacⅠ酶切PCR产物,若PCR产物能被完全切开,得到400和273 bp的两条带,表明靶位点未发生突变,为未突变单株;若PCR产物被部分切开,得到400、273和约673 bp三条带,表明一条同源染色体突变,为杂合体;若PCR产物完全不能切开,表明两条同源染色体均发生突变,则可能为2个等位基因发生不同突变的双等位突变体或2个等位基因发生相同突变的纯合体。如图1-B所示,结果表明,单株4、5、6、7、10、12、15、16、18和25未发生突变;单株8、9、11和17的PCR产物被部分酶切,形成三条带,为一条同源染色体突变的杂合体;单株1、2、3、13、14、19、20、21、22、23和24的PCR产物完全不能切开,则可能为双等位突变或纯合突变体。
PCR产物片段大小和酶切PCR产物只能初步定性判断转基因植株靶点是否发生突变,而突变体靶点序列的改变还需测序进行鉴定。由于杂合突变或双等位突变的两条同源染色体突变情况不一致,用PCR产物测序时会在某一位点(一般在突变位点附近)之后出现重叠峰,后续序列无法准确得知,因此,对可能出现重叠峰的突变体进行TA克隆,随机选取单菌斑进行测序。同时对突变基因型一致的单株进行Southern blot分析(电子附图1),检测是否属同一株系,结果表明,1和24、2和23、3和14、19和21分属同一株系。综合分析测序结果和Southern blot结果得到各单株突变情况(图2),共获得5株纯合突变体(2、3、13、14和23)、6株双等位突变体(1、19、20、21、22和24)和4株杂合突变体(8、9、11和17),包含10种不同突变基因型和11个突变株系,多为单碱基的插入或缺失,但也得到了2种56和66 bp较大片段缺失的株系。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1酶切鉴定靶位点突变情况
-->Fig. 1Mutation identification at target site by enzyme digestion
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2靶位点突变序列分析
-->Fig. 2Analysis of mutation sequences of targets
-->
2.3 OsPIL15 T1代突变体分析鉴定
为验证T0代突变体产生的突变序列能否稳定遗传,选取部分纯合突变、双等位突变和杂合突变株系的下一代植株,对这些T1代植株突变情况进行测序分析(表2),纯合突变体(3和14)后代为纯合突变单株,但株系14的30株T1代植株中有29株纯合突变,发现1株未突变单株;3个双等位突变体(21、22和24)后代既获得了纯合突变体,也获得了双等位突变体;2个杂合突变体(9和17)的T1代单株中,既有纯合突变和杂合突变体,也有未突变的单株。上述结果表明,T0代突变体产生的突变基因型基本上能稳定遗传给下一代,纯合株系后代为纯合体,双等位株系后代产生纯合体和双等位突变体,而杂合株系后代会得到未突变、纯合和杂合3种类型。为了鉴定T1代单株是否会重新发生基因编辑,随机选取了62株T1代为阳性(含有Cas9基因)且在T0代未发生突变的转基因单株,使用限制性内切酶SacⅠ进行酶切鉴定(图3),62个阳性转基因单株PCR产物均被切开,得到400和273 bp的两条带,因此,判定这62株T1代转基因植株均未发生突变。表明T0代未突变株系,T1代依然未出现突变。
Table 2
表2
表2T1代植株分析鉴定
Table 2Analysis of T1 plants
| 单株 Lines | T0代T0 generation | T1代 T1 generation | ||||
|---|---|---|---|---|---|---|
| 突变类型 Mutation types | 测试株数 No. of plants examined | 未突变株数 No. of non- mutations plants | 纯合株数(M1/M2) No. of homozygous plants (M1/M2) | 杂合株数 No. of heterozygous plants | 双等位株数 No. of biallelic plants | |
| 3 | 纯合突变 Homozygous mutation | 17 | — | 17 | — | — |
| 14 | 纯合突变 Homozygous mutation | 30 | 1 | 29 | — | — |
| 21 | 双等位突变 Biallelic mutation | 23 | — | 4/10 | — | 9 |
| 22 | 双等位突变 Biallelic mutation | 13 | — | 3/3 | — | 7 |
| 24 | 双等位突变 Biallelic mutation | 31 | — | 12/13 | — | 6 |
| 9 | 杂合突变 Heterozygous mutation | 20 | 7 | 7 | 6 | — |
| 17 | 杂合突变 Heterozygous mutation | 13 | 4 | 4 | 5 | — |
| --- | 未突变 No mutation | 62 | 62 | — | — | — |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3酶切分析T0代未突变单株的T1代阳性转基因单株靶位点
-->Fig. 3PCR/RE assay to detect engineered target site of T1 positive transgenic plants which had no mutation in T0
-->
2.4 ospil15突变体脱靶效应评估
CRISPR/Cas9系统基因编辑的特异性主要取决于sgRNA的识别序列,但由于高等生物基因组较大且较为复杂,sgRNA可能会与非靶点DNA序列发生局部错配,造成CRISPR/Cas9系统基因编辑的脱靶。因此,需要对OsPIL15的突变体脱靶效应进行评估(表3),选取水稻基因组中与sgRNA序列错配碱基数在4 bp以内且具有PAM序列的5个位点作为潜在脱靶位点,每个潜在脱靶位点均随机选取10株T0代突变单株和10株T1代突变单株,通过酶切法进行检测(图4)。Off-target1位点若被SacⅠ酶切可形成444和179 bp的两条带,Off-target2位点酶切后产生359和173 bp的两条带,Off-target3位点酶切可得到268和152 bp的两条带,Off-target4位点可被酶切为411和153 bp的两条带,Off-target5位点若被切开会得到360和254 bp两条带。所选取的20个突变单株的5个潜在脱靶位点PCR产物均能被SacⅠ酶成功切割,因此,这5个潜在脱靶位点在T0代和T1代均未产生脱靶效应,表明所设计的sgRNA序列具有较高的特异性。Table 3
表3
表3CRISPR/Cas9系统潜在脱靶位点突变检测
Table 3Mutations detected in the putative CRISPR/Cas9 off-target sites
| 编号 Serial number | 潜在脱靶位置 Putative off-target locus | 潜在脱靶序列 Sequence of the putative off-target site (5′-3′) | 错配碱基数 No. of mismatching bases | 测试株数T0/T1 No. of plants tested | 突变株数T0/T1 No. of plants with mutations |
|---|---|---|---|---|---|
| Off-target1 | Chr06:5007999—5008020 | GTCTTCTTCTCCGAGCTCGTGG | 2 | 10/10 | 0/0 |
| Off-target2 | Chr03:10438029—10438050 | GGCTTCCTCCCCGAGCTCCGGG | 3 | 10/10 | 0/0 |
| Off-target3 | Chr11:22491660—22491681 | GACCTCTACACCGAGCTCCCGG | 3 | 10/10 | 0/0 |
| Off-target4 | Chr02:30705125—30705146 | GGCTTCCTGACCGAGCTCCTGG | 4 | 10/10 | 0/0 |
| Off-target5 | Chr12:18985586—18985565 | GGCCTCCTCTGCGAGCTCCTGG | 4 | 10/10 | 0/0 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图45个潜在脱靶位点脱靶效应评估
-->Fig. 4Evaluating off-target effects of 5 potential off-target sites
-->
2.5 OsPIL15功能初步分析
选择水稻中PIFs家族的6个基因OsPIL11(Os12g0610200)、OsPIL12(Os03g0639300)、OsPIL13(Os03g0782500)、OsPIL14(Os07g0143200)、OsPIL15(Os01g0286100)、OsPIL16(Os05g0139100)和拟南芥中PIFs家族的7个基因PIF1/PIL5(AT2G20180)、PIF3(AT1G09530)、PIF4(AT2G43010)、PIF5/PIL6(AT3G59060)、PIF6/PIL2(AT3G62090)、PIF7(AT5G61270)、PIF8(AT4G00050),根据它们的氨基酸序列进行系统进化树分析(图5-A),水稻中光敏色素互作因子OsPIL15和OsPIL16亲缘关系最近,二者蛋白同源性几乎达到了100%。有研究表明,超表达OsPIL16能使水稻籽粒减小[22],OsPIL15和OsPIL16十分相近的亲缘关系表明OsPIL15也有可能调控水稻籽粒大小。因此,选取T1代不同基因型的三组突变体,对其表型进行观察,与野生型相比,ospil15突变体生育期没有变化,株高极显著降低,平均降低12.49%;有效分蘖数没有明显差异;而籽粒粒长极显著增加,其中,突变体22增加最高,增幅达5.69%,增幅最小的突变体21也达3.88%(图5-B—图5-E)。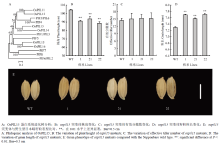 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5OsPIL15功能初步分析
-->Fig. 5Preliminary functional analysis of OsPIL15
-->
3 讨论
CRISPR/Cas9技术以其突变高效、操作简单、成本低廉等优势在动植物中得到了广泛应用。本研究从靶位点选择,表达载体构建,突变位点分析和脱靶效应评估等方面,详细阐述了利用CRISPR/Cas9系统对水稻OsPIL15的编辑。本研究针对OsPIL15设计了1个靶向位点,有研究认为单靶点在T0代形成的小片段突变,使得T1代植株中sgRNA仍有可能识别靶序列造成二次编辑,后代突变具有不稳定性[23]。本研究在纯合突变体14后代中发现了一株未突变单株,且突变体14仅为1个单碱基插入突变,是否是由于CRISPR/ Cas9系统在T1代再次对靶位点进行编辑形成新的碱基改变还需进一步验证。本试验获得突变体多为单碱基插入或缺失,但也得到了两种分别缺失66和56 bp的较大片段缺失突变体。针对基因编码序列设计多个靶位点有利于形成大片段缺失突变而获得稳定遗传,但靶点越多产生脱靶的可能性越大[24],需综合考虑目标基因靶点位置及数量,尽可能保证其特异性。本试验采用酶切和测序相结合的方法对突变位点进行分析鉴定,但由于杂合突变和双等位突变的PCR产物直接测序会出现重叠峰而无法准确读取,需将PCR产物进行TA克隆后挑选单菌斑测序,工作量较大,成本较高。为此华南农业大学刘耀光教授课题组基于DSD(degenerate sequence decoding)法[25]开发了在线工具DSDecode(http://dsdecode.scgene.com/),可对各突变类型测序文件自动解码[26],但限于不同公司和不同PCR产物测序质量,部分测序结果不能很好地解码,因此,此方法可为测序分析提供参考,具体仍需验证。CRISPR/Cas9系统成功对植物基因组编辑后,所获得突变基因序列能否稳定遗传给下一代决定了该突变体是否具有应用前景。由于拟南芥多采用蘸花法进行转基因,CRISPR/Cas9系统诱导的突变在T1代中主要发生在体细胞,从而形成嵌合体,较难稳定遗传给下一代,而发生在生殖细胞中的突变可稳定遗传[27]。水稻多采用农杆菌介导愈伤组织,转基因苗多来源于单个细胞或细胞团,因此本试验在T0代即获得了纯合突变体,对纯合突变、双等位突变和杂合突变的T1代植株分析显示这些突变绝大多数均能稳定遗传,有研究证明这些稳定遗传突变体自交后代分离均符合孟德尔遗传定律[28-29]。有报道认为CRISPR/Cas9系统在后代植株当中对野生型依然可行使功能[30],T0代转基因植株靶位点表现为野生型的植株后代仍有可能发生一定比例的突变[27]。本研究对T0代未突变植株的T1代(阳性)靶位点突变情况进行鉴定,未获得新的突变单株。ZHANG等[31]认为这可能与CRISPR/Cas9系统插入到基因组的位置有一定关系。本研究仅对部分T1代植株进行了鉴定,CRISPR/Cas9系统是否会在其他后继世代中重新发挥作用还需作进一步验证。
CRISPR/Cas9系统在植物基因编辑中的特异性对于其安全高效的利用具有重要意义,因此有必要对可能出现的脱靶效应进行全面准确地评估。有研究表明,针对人源CLTA基因的4个靶位点均存在脱靶现象,脱靶效率最高可达84%[32];针对鼠源Crygc基因的2个靶点存在40%的脱靶率[33]。而在植物中CRISPR/Cas9系统脱靶效率较低,朱健康教授实验室通过对拟南芥全基因组测序和潜在脱靶位点分析未检测到脱靶效应[27],对水稻中多个与靶位点高度同源序列进行检测,仅在一个碱基不匹配处发现脱靶效应[31],杜彦修等[34]对水稻OsbHLH116基因两个潜在脱靶位点进行检测并未发现脱靶。本研究选择了PAM序列5′端同源性较高的5个位点作为潜在脱靶位点,经检测这5个位点均未发生脱靶效应,因此设计的sgRNA序列具有较高特异性。有研究表明,sgRNA的特异性主要由靠近PAM序列的10—12 bp碱基所决定,而远离PAM区的8—10 bp碱基对sgRNA的特异性识别影响不大[3, 35]。本试验只是对最有可能出现脱靶效应的5个位点进行了检测,不排除其他位点出现脱靶的可能性,更有效的方法则是对获得的突变体进行全基因组深度测序分析,但其成本较高且费时费力。为此科学家开发了新的基于Cas9核酸酶消化基因组(Digenome- seq)技术和基于整合酶缺陷型慢病毒载体(IDLV)技术来精确检测CRISPR/Cas9系统脱靶效应[36-37]。通过优化sgRNA序列(长度和GC含量等)、应用双Cas9切口酶[38]和应用fCas9复合体[39]等方法可降低CRISPR/Cas9系统脱靶效应。
本试验利用CRISPR/Cas9技术创制了ospil15突变体,并对其T1代表型进行了初步观察,发现突变体株高极显著降低,籽粒粒长极显著增大,与OsPIL15蛋白系统进化分析结果是一致的。后续仍需进行多代鉴定,获得稳定纯合的突变体,同时对不同突变类型的株系进行观察,以确定不同突变类型造成的氨基酸改变而产生的表型是否一致。
4 结论
依据CRISPR/Cas9系统原理创制ospil15突变体,成功获得5株纯合突变体、6株双等位突变体和4株杂合突变体。对部分突变体后代突变序列进行分析,发现绝大多数突变均能稳定遗传。62株T0代表现为未突变的T1代阳性植株经酶切鉴定均未发现新的突变,表明CRISPR/Cas9系统在T1代转基因阳性植株中未重新发挥基因编辑作用。5个与靶序列同源性较高且错配在4 bp以内的潜在脱靶位点经评估均未出现脱靶效应,表明所选取sgRNA有较高的特异性。对获得的ospil15突变体表型进行初步观察发现,与野生型相比T1代突变体株高极显著降低,分蘖数未发生显著变化,籽粒粒长极显著增大。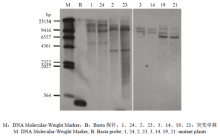 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT附图1Southern blot分析突变单株的T-DNA插入
-->Supplemental Fig. 1Analysis of T -DNA region by southern blot
-->
The authors have declared that no competing interests exist.
