0 引言
【研究意义】油菜(主要指甘蓝型油菜)是中国重要的油料作物。目前,菜籽油消费约占中国食用植物油总消费量的35%,但中国食用植物油60%以上却依赖进口[1],油菜产量的高低直接影响中国食用植物油的供给,因此,如何提高油菜产量一直是油菜育种和栽培最重要的目标。角果长度是油菜籽粒产量的重要决定因素和育种目标性状,明确其遗传基础对油菜产量提升具有重要意义。【前人研究进展】角果对油菜产量形成起重要决定作用,它不仅是油菜吸收和积累叶片制造的光合产物的库器官,而且还是为种子发育提供营养的源器官[2]。在种子发育后期,油菜功能叶面积快速减少,绿色角果的光合作用成为种子发育所需营养的主要来源[3]。另外,角果细胞壁还会传送信号来调节油菜籽粒的填充速率及营养物质的再分配[4]。一般来说,较长角果相对较短角果来说,会产生更多和更大的种子[5],通过特长角果油菜品种培育可以提高油菜的产量潜力[6]。在油菜高产育种中角果长度常被作为产量相关性状进行考察[7]。油菜的角果长度是受多基因控制的数量性状,前人采用双亲作图群体对角果长度性状进行了QTL定位研究,鉴定出多个控制角果长度性状变异的QTL,目前已在甘蓝型油菜15条染色体上探测到30个以上角果长度QTL,解释的表型贡献率从5.5%—53.4%不等[8-10]。但由于传统QTL定位方法需要构建专门的作图群体,检测到的等位基因位点仅来源于双亲材料,检测到的基因位点数目偏少,因此应用较为有限。【本研究切入点】全基因组关联分析(genome-wide association study,GWAS)是利用自然群体探测物种的遗传变异,进而挖掘与复杂农艺性状相关的遗传位点的研究方法,具有不需构建作图群体和一次性可同时检测多个等位基因位点的优势,近年来已在水稻、玉米、小麦、大麦、拟南芥等作物中均有广泛研究[11]。近几年许多****利用新开发的芸薹属60K SNP(single nucleotide polymorphism)芯片技术,对油菜籽粒产量及品质相关性状开展了全基因组关联分析研究,获得了较多新的控制油菜相关性状的基因位点[12-15]。而随着甘蓝型油菜基因组测序的完成[16],利用高通量数据对油菜复杂性状开展GWAS研究越来越便利,SLAF-seq(specific-locus amplified fragment sequencing)技术作为一种新发展的简化基因组测序技术,通过限制性内切酶消化基因组DNA,选取一定长度的片段进行测序,降低了基因组的复杂度,并且不依赖基因组序列,一次测序可以获得数以百万计的SNP[17],另外,与芯片相比,高通量测序可以检测基因组上未知变异位点中新的SNP,因而,采用SLAF-seq技术开发SNP并用于全基因组关联分析有望探测到较多SNP位点和候选基因。【拟解决的关键问题】本研究以来源广泛的300份甘蓝型油菜自交系为关联分析研究群体,并采用前期基于SLAF-seq技术开发的201 817个SNP标记对甘蓝型油菜角果长度性状进行全基因组关联分析,旨在挖掘与之显著相关的SNP位点及候选基因,为油菜产量分子标记辅助选择育种和油菜产量性状遗传调控提供理论依据。1 材料与方法
1.1 试验材料
以300份具有独立来源的甘蓝型油菜自交系为材料,所有材料均由江西农业大学作物生理生态与遗传育种教育部重点实验室提供。1.2 田间试验和数据统计
2014年9月28日,将300份甘蓝型油菜自交系分别同时播种于江西农业大学农学试验田(简称JXAU)和江西省红壤研究所油菜试验田(简称JXIRS),随机区组设计,每份材料种植一个小区,每个小区种植3行,行距40 cm,株距20 cm,条播,于5—7叶期定苗,两地各2个重复。田间管理同常规生产,确保同一地区所有材料的生长环境一致。待成熟时,每小区取十株长势一致的植株,每株选取主花序中部的20个角果进行角果长度测量[18]。利用Excel和DPS统计分析软件对所得性状的测定值进行统计分析,包括计算每个表型性状的平均值、标准差、变异系数等。1.3 基因型测序分型
利用简化基因组测序技术(specific locus amplified fragment sequencing,SLAF-seq)对300份甘蓝型油菜自交系基因组DNA进行测序并分析。具体做法是以公开的甘蓝型油菜品种“Darmor-Bzh”的基因组测序信息为参考[16],进行电子酶切预测,根据酶切方案选择原则确定以RsaⅠ+HaeⅢ进行酶切,筛选酶切片段长度在314—414 bp的序列并定义为SLAF标签,结合通过生物信息学分析,共获得529 080个SLAF标签,其中多态性的SLAF标签共有238 711个,由此得到1 197 282个群体SNP。再根据次等位基因频率(minor allele frequency,MAF)>0.05和完整度(即非N的SNP位点数占总SNP位点的比例)>0.8对SNP进行筛选,最终得到均匀分布于甘蓝型油菜19条染色体上的201 817个高质量且位置唯一的群体SNP用于后续分析(数据在另文中待发表)。1.4 连锁不平衡分析
通过在同一个染色体的SNP组合,分析SNP在所有样品中的连锁情况和LD衰退距离,其连锁强度以D′或r2表示,采用PLINK2软件(http://www.cog- genomics.org/plink2/)分析所有材料的连锁不平衡和LD衰退情况。依据PURCELL等[19]报道,参数设定为:MAF>0.05,r2,ld-window 999999,ld-window-r2 0。本研究选用r2等于0.1的数据作为连锁不平衡的衰减(LD decay)的数值[20]。1.5 角果长度性状全基因组关联分析
基于前期DNA测序开发的201 817个群体SNP,利用TASSEL软件[21]的GLM模型和MLM模型得到与2个性状的关联值,通过SPAGeDi[22]软件计算样品间亲缘关系K,通过admixture软件[23]计算样品群体结构Q,一般线性模型使用Q群体结构信息,而混合线性模型使用Q+K即群体结构和亲缘关系的信息。基于公式(Y=Xα+Qβ+Kµ+e)(X为基因型,Y为表型),计算最终每个SNP位点所对应的关联值P。利用GGplot2软件[24]做出Quantile-Quantile散点图(Q-Q plot),利用QQman绘制曼哈顿(Manhattan)图[25],用Manhattan图来显示关联分析后每个位点的显著性,其中,Manhattan图显著关联SNP阈值取为0.1/SNP总数目的负对数,本试验计算结果约等于6.2,极显著位点阈值为多重假设检验(FDR检验)的显著性阈值。1.6 候选基因预测
将与角果长度性状紧密连锁的SNP位点,根据序列位置将其定位到油菜基因组参考物理图谱上[16]。基于群体连锁不平衡分析结果确定搜寻每个SNP位点两侧100 kb范围内的基因,利用BLAST软件将关联区域内基因分别与NR、SwissProt、GO、COG、KEGG数据库比对,获得关联区域内基因的注释信息,根据注释信息找出与性状相关的候选基因。2 结果
2.1 甘蓝型油菜角果长度性状表型统计及方差分析
从表1中可以看出,农大试验地中所有材料的平均角果长度为70.72 mm,变异幅度在46.35—107.07 mm,变异系数为16.47%,正态性检验结果为W=0.946329,P=0.000001,所有材料的表型值在农大试验地表现出极显著差异。红壤所试验地中平均角果长度为67.60 mm,变异幅度在39.41—101.35 mm,变异系数为17.41%,正态性检验结果为W=0.987718,P=0.011974,材料表型值在红壤所试验地差异显著,频次分布图具有显著的正态分布特征(图1),表明角果长度性状为典型的数量性状,采用GWAS分析方法可有效进行该性状的基因定位。两地试验的方差分析结果显示(表2),角果长度性状在300份基因型材料中表现出极显著差异,基因型方差所占总表型方差比例为13.70%,角果长度性状在两地的平均遗传力为0.6057,材料间的遗传差异较大;角果长度性状在2个试验地环境下表现出极显著差异,环境效应方差占总表型方差比例为83.92%,说明环境因素对甘蓝型油菜角果长度性状具有重要影响。另外,该性状基因型与环境互作方差同样达到极显著差异,但角果长度性状在区组(重复)间差异未达到显著水平。
Table 1
表1
表1油菜角果长度性状统计分析
Table 1Statistical analysis of silique length traits of rapeseed
| 环境 Environment | 均值±标准差 Mean ± SD | 众数 Mode | 最小值 Minimum | 50%分位数 50% quantile | 最大值 Max | 变异系数 CV (%) | Shapiro-Wilk |
|---|---|---|---|---|---|---|---|
| JXAU | 70.72±11.65 | 67.88 | 46.35 | 68.28 | 107.07 | 16.47 | W=0.946329 P=0.000001 |
| JXIRS | 67.60±11.77 | 64.96 | 39.41 | 66.54 | 101.35 | 17.41 | W=0.987718 P=0.011974 |
新窗口打开
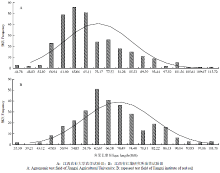 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1油菜角果长度性状频次分布
-->Fig. 1Frequency distribution of silique length of B. napus
-->
Table 2
表2
表2两地试验角果长度性状的方差分析
Table 2Variance analysis of silique length in two places
| 变异来源 Source of variation | DF | 方差及占总方差的比例 MS (SS/SST) (%) |
|---|---|---|
| 基因型 Genotype (G) | 299 | 478.55** (13.70) |
| 环境 Environment(E) | 1 | 2933.38** (83.92) |
| 区组 Block | 1 | 13.36 (0.38) |
| 基因型×环境 G×E | 299 | 69.83** (2.00) |
| 误差 Error | 599 | 49.76 |
新窗口打开
2.2 甘蓝型油菜角果长度性状全基因组关联分析
通过GLM模型对角果长度性状进行全基因组关联分析(图2、图3,表3和表4),在农大试验地中共探测到121个显著相关的SNP位点,其中,包括66个为极显著相关SNP位点(P<4.96E-08)和55个显著相关SNP位点(P<4.96E-07),在A04、A06、A08、A09、C02、C03、C06和C09等8条染色体均有分布,其中A09染色体上分布最多(83个)。在红壤所试验地共探测到22个显著相关的SNP位点,包括12个极显著和10个显著相关的SNP位点,其中,1个在C09染色体上,其余21个SNP均分布于A09染色体,通过GLM方法在两试验地探测到20个一致性SNP位点。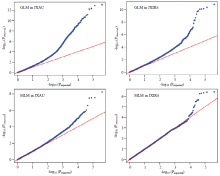 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2GLM和MLM两种模型下角果长度性状GWAS Q-Q Plot图
-->Fig. 2Quantile-quantile plots of estimated-lg (P) from association analysis of silique length using two models with GLM and MLM
-->
从表图3和5可以看出,通过MLM模型在农大试验地中共找到5个显著相关的SNP位点,其中包括4个极显著(P<4.96E-08)和1个显著(P<4.96E-07)相关的SNP位点,单个SNP解释的表型贡献率(R2)的变异为0.1153—0.1377,最小等位基因频率变异范围为0.2266—0.2992。红壤所试验地中共找到3个显著相关的SNP位点(P<4.96E-07),这三个位点与农大试验地试验结果一致,单个SNP解释的表型贡献率(R2)的变异为0.1020—0.1037。且所有位点均都位于A09染色体上。
通过GLM和MLM两种模型对甘蓝型油菜角果长度性状进行全基因组关联分析比较结果表明,GLM关联分析模型在多条染色体上探测到显著性SNP位点,且多数分布在A09染色体上;而MLM关联分析模型由于综合考虑了群体结构和亲缘关系2个方面,因此检测到的位点较少,且全部分布在A09染色体上,说明A09染色体上很有可能主要分布有控制油菜角果长度的等位基因位点及候选基因。
2.3 LD分析及候选基因挖掘
利用全基因组获得的201 817个SNP标记对300份甘蓝型油菜自交系品种的LD进行分析,得到LD-decay衰减曲线(图4),从A和C亚基因组各条染色体LD衰退曲线可以看出(图4-a和图4-b),在r2=0.1时,A05染色体衰退最快,衰退距离为1.5 kb,而A01、A04、A06衰退也较快,衰退距离均小于50 kb。在C亚基因组中,C09衰退最快,r2=0.1时衰退距离为4.5 kb,其余染色体衰退较慢。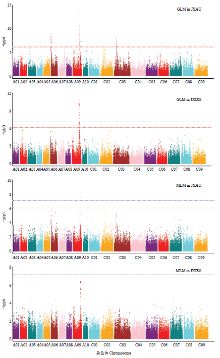 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3角果长度性状全基因组关联分析Manhattan图
-->Fig. 3Manhattan of GWAS for silique length of B. napus
-->
Table 3
表3
表3农大试验地角果长度显著关联SNP位点(GLM)
Table 3SNPs associated with silique length in JXAU (GLM)
| 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | |
|---|---|---|---|---|---|---|---|---|
| 1 | A04 | 10196815 | 3.06E-07 | 40 | A09 | 27454324 | 5.60E-08 | |
| 2 | A04 | 10197157 | 5.96E-08 | 41 | A09 | 27454353 | 3.17E-08 | |
| 3 | A06 | 1290726 | 1.39E-07 | 42 | A09 | 27454397 | 3.17E-08 | |
| 4 | A06 | 1298939 | 4.39E-08 | 43 | A09 | 27454757 | 9.40E-08 | |
| 5 | A06 | 1298955 | 1.84E-08 | 44 | A09 | 27454823 | 2.31E-11 | |
| 6 | A06 | 1298956 | 4.93E-08 | 45 | A09 | 27455018 | 1.78E-09 | |
| 7 | A06 | 1298958 | 2.28E-08 | 46 | A09 | 27455019 | 5.44E-09 | |
| 8 | A06 | 1298986 | 2.34E-08 | 47 | A09 | 27466747 | 3.21E-10 | |
| 9 | A06 | 1299000 | 1.06E-07 | 48 | A09 | 27528594 | 3.10E-08 | |
| 10 | A06 | 1299003 | 1.52E-07 | 49 | A09 | 27528616 | 2.13E-07 | |
| 11 | A06 | 1300019 | 3.18E-07 | 50 | A09 | 27573865 | 3.51E-08 | |
| 12 | A06 | 1300193 | 1.03E-08 | 51 | A09 | 27573876 | 1.50E-07 | |
| 13 | A06 | 1300209 | 1.54E-08 | 52 | A09 | 27574065 | 1.18E-07 | |
| 14 | A06 | 1302471 | 4.08E-09 | 53 | A09 | 27575614 | 9.42E-10 | |
| 15 | A06 | 1302541 | 9.26E-08 | 54 | A09 | 27575680 | 1.66E-09 | |
| 16 | A06 | 1317042 | 1.01E-09 | 55 | A09 | 27575683 | 3.10E-09 | |
| 17 | A06 | 6361553 | 9.27E-08 | 56 | A09 | 27575771 | 7.28E-10 | |
| 18 | A06 | 6361761 | 5.30E-09 | 57 | A09 | 27575942 | 1.66E-09 | |
| 19 | A08 | 9693836 | 4.74E-07 | 58 | A09 | 27575995 | 2.31E-09 | |
| 20 | A09 | 27262605 | 2.63E-07 | 59 | A09 | 27576016 | 1.92E-09 | |
| 21 | A09 | 27273147 | 6.83E-08 | 60 | A09 | 27576025 | 2.42E-09 | |
| 22 | A09 | 27273373 | 1.69E-07 | 61 | A09 | 27578045 | 1.12E-09 | |
| 23 | A09 | 27273382 | 9.70E-08 | 62 | A09 | 27638145 | 2.27E-08 | |
| 24 | A09 | 27325190 | 4.15E-07 | 63 | A09 | 27638182 | 3.03E-07 | |
| 25 | A09 | 27325200 | 2.23E-07 | 64 | A09 | 27638223 | 3.75E-07 | |
| 26 | A09 | 27325202 | 1.35E-07 | 65 | A09 | 27684007 | 8.70E-10 | |
| 27 | A09 | 27325248 | 3.30E-07 | 66 | A09 | 27693788 | 2.63E-07 | |
| 28 | A09 | 27325403 | 6.91E-08 | 67 | A09 | 27694067 | 2.12E-07 | |
| 29 | A09 | 27325468 | 4.85E-08 | 68 | A09 | 27694070 | 1.16E-08 | |
| 30 | A09 | 27415506 | 4.84E-08 | 69 | A09 | 27694097 | 8.06E-08 | |
| 31 | A09 | 27431963 | 5.12E-09 | 70 | A09 | 27694105 | 1.79E-09 | |
| 32 | A09 | 27433836 | 2.42E-10 | 71 | A09 | 27694122 | 4.36E-08 | |
| 33 | A09 | 27440938 | 5.23E-11 | 72 | A09 | 27694124 | 2.24E-07 | |
| 34 | A09 | 27441015 | 2.94E-10 | 73 | A09 | 27694125 | 2.21E-07 | |
| 35 | A09 | 27441252 | 2.11E-10 | 74 | A09 | 27694133 | 2.20E-07 | |
| 36 | A09 | 27450650 | 1.65E-07 | 75 | A09 | 27767046 | 1.29E-13 | |
| 37 | A09 | 27454051 | 1.89E-08 | 76 | A09 | 27767179 | 6.23E-13 | |
| 38 | A09 | 27454304 | 3.94E-08 | 77 | A09 | 27767188 | 8.74E-14 | |
| 39 | A09 | 27454314 | 3.17E-08 | 78 | A09 | 27767206 | 1.52E-09 | |
| 续表3 Continued table 3 | ||||||||
| 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | |
| 79 | A09 | 27768124 | 6.00E-09 | 101 | A09 | 28156822 | 4.89E-13 | |
| 80 | A09 | 27768172 | 5.47E-09 | 102 | A09 | 28267003 | 5.21E-08 | |
| 81 | A09 | 27788523 | 3.83E-07 | 103 | C02 | 9987290 | 3.79E-07 | |
| 82 | A09 | 27788524 | 3.91E-07 | 104 | C02 | 9990486 | 1.47E-07 | |
| 83 | A09 | 27788809 | 3.95E-11 | 105 | C03 | 9909951 | 6.34E-09 | |
| 84 | A09 | 27788849 | 9.60E-12 | 106 | C03 | 9909957 | 1.33E-08 | |
| 85 | A09 | 27788853 | 1.58E-07 | 107 | C03 | 9909973 | 6.12E-08 | |
| 86 | A09 | 27788865 | 2.78E-07 | 108 | C03 | 9909983 | 1.78E-07 | |
| 87 | A09 | 27788868 | 2.03E-11 | 109 | C03 | 9910205 | 3.63E-08 | |
| 88 | A09 | 27815044 | 1.17E-08 | 110 | C03 | 10308045 | 2.03E-07 | |
| 89 | A09 | 27827711 | 8.82E-08 | 111 | C03 | 10308070 | 3.54E-07 | |
| 90 | A09 | 27882634 | 7.56E-12 | 112 | C03 | 10308082 | 3.47E-07 | |
| 91 | A09 | 27882638 | 1.42E-08 | 113 | C03 | 10308094 | 3.96E-07 | |
| 92 | A09 | 27882689 | 1.42E-08 | 114 | C03 | 10350428 | 1.24E-07 | |
| 93 | A09 | 27900638 | 3.95E-07 | 115 | C03 | 10350451 | 4.43E-07 | |
| 94 | A09 | 27900841 | 1.68E-08 | 116 | C03 | 10406203 | 2.96E-07 | |
| 95 | A09 | 27930164 | 3.70E-11 | 117 | C03 | 10419432 | 4.57E-07 | |
| 96 | A09 | 27930301 | 9.81E-11 | 118 | C03 | 54672110 | 1.04E-07 | |
| 97 | A09 | 27948842 | 1.16E-09 | 119 | C06 | 5842471 | 2.80E-07 | |
| 98 | A09 | 27948857 | 1.09E-10 | 120 | C09 | 33738790 | 4.30E-07 | |
| 99 | A09 | 28050920 | 3.46E-10 | 121 | C09 | 33738791 | 2.53E-07 | |
| 100 | A09 | 28108404 | 4.75E-10 | |||||
新窗口打开
Table 4
表4
表4红壤所试验地角果长度显著关联SNP位点(MLM)
Table 4SNPs associated with silique length in JXIRS (MLM)
| 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | 序号 Code | 染色体 Chr. | SNP位置 SNP position | P值 P value | |
|---|---|---|---|---|---|---|---|---|
| 1 | A09 | 3899079 | 3.29E-07 | 12 | A09 | 27788849 | 7.37E-11 | |
| 2 | A09 | 27440938 | 6.69E-08 | 13 | A09 | 27788868 | 8.68E-11 | |
| 3 | A09 | 27441015 | 1.67E-07 | 14 | A09 | 27882634 | 4.25E-07 | |
| 4 | A09 | 27454823 | 1.49E-07 | 15 | A09 | 27900638 | 1.69E-08 | |
| 5 | A09 | 27578045 | 1.19E-07 | 16 | A09 | 27900841 | 2.12E-09 | |
| 6 | A09 | 27767046 | 1.52E-11 | 17 | A09 | 27930164 | 2.09E-07 | |
| 7 | A09 | 27767179 | 8.38E-11 | 18 | A09 | 27930301 | 2.73E-07 | |
| 8 | A09 | 27767188 | 4.71E-11 | 19 | A09 | 27948842 | 9.77E-08 | |
| 9 | A09 | 27768124 | 5.95E-09 | 20 | A09 | 27948857 | 5.81E-10 | |
| 10 | A09 | 27768172 | 5.16E-09 | 21 | A09 | 28156822 | 1.76E-10 | |
| 11 | A09 | 27788809 | 2.16E-10 | 22 | C09 | 23116992 | 1.55E-07 |
新窗口打开
Table 5
表5
表5角果长度显著关联SNP位点(MLM)
Table 5SNPs associated with silique length (MLM)
| 环境 Enviroment | 序号 Code | SNP位置 SNP position | 染色体 Chr. | P值 P value | 贡献率 R2 | 等位 基因Allele | 最小等位基因频率 MAF |
|---|---|---|---|---|---|---|---|
| JXAU | 1 | 27948857 | A09 | 2.55E-07 | 0.1153 | [C/T] | 0.2392 |
| 2 | 27767179 | A09 | 2.94E-08 | 0.1267 | [A/T] | 0.2266 | |
| 3 | 27767188 | A09 | 5.55E-09 | 0.1377 | [C/T] | 0.2283 | |
| 4 | 27767046 | A09 | 3.18E-08 | 0.1234 | [C/T] | 0.2295 | |
| 5 | 28156822 | A09 | 2.81E-08 | 0.1268 | [C/G] | 0.2299 | |
| JXIRS | 1 | 27767046 | A09 | 4.90E-07 | 0.1037 | [C/T] | 0.2295 |
| 2 | 27767179 | A09 | 4.16E-07 | 0.1042 | [A/T] | 0.2266 | |
| 3 | 27767188 | A09 | 3.86E-07 | 0.1020 | [C/T] | 0.2283 |
新窗口打开
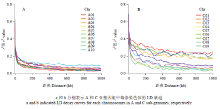 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4甘蓝型油菜A和C基因组不同染色体的LD衰减
-->Fig. 4LD decay of different chromosomes for A and C subgenomes in B. napus
-->
采用GLM和MLM两种方法均在A09染色体上找到与油菜角果长度显著关联的SNP位点,因此,对A09位点两侧进行角果长度候选基因预测较为可靠,通过LD分析表明,A09染色体的LD衰退距离为95.02 kb,所以本试验确定对显著关联SNP位点两侧100 kb进行基因搜寻并进行功能注释。
在甘蓝型油菜A09染色体的Bn-A09-p27767046、Bn-A09-p27948857和Bn-A09-p28156822 3个SNP标记位点附近共搜寻到125个基因,Bn-A09-p27767046位点附近有搜寻到41个基因,在Bn-A09-p27948857位点附近搜寻到28个基因,在Bn-A09-p28156822位点附近搜寻到56个候选基因(具体数据未列出)。对上述基因进行基于油菜候选基因序列及拟南芥同源基因的功能预测,结果表明,在上述候选基因中存在40个与碳水化合物的运输及合成、细胞伸长与分裂、花粉管伸长、花器官发育、种子发育、信号转导、胁迫响应等重要的生理过程相关的基因(部分结果见表6)。它们可能通过上述功能影响油菜花序或角果的发育,导致角果长度的差异。
3 讨论
油菜籽粒直接由种子数目、大小和重量构成,这些种子构成要素都和角果长度直接相关。甘蓝型油菜角果长度与每角果粒数和粒重显著相关[26-27],LIU等[28] 研究表明角果长度主要通过调控光合产物的积累来影响油菜粒重。随着DNA分子标记技术的发展,自20世纪90年代以来,已有众多研究人员陆续开展油菜角果长度的QTL定位研究,如CHEN等[29]利用甘蓝型油菜杂交组合Quantum×No.2717-17的DH和F2群体进行产量及其构成因子QTL定位,共在A01、A09、A10、C01、C02、C05和C07染色体上找到11个与角果长度性状显著关联的QTL。袁泽俊等[30]对油菜角果长度进行了QTL定位和遗传分析,结果在A09染色体上检测到4个与角果长度相关的QTL。ZHANG等[31]利用甘蓝型油菜品系Y106与HZ396为亲本构建了DH群体,通过QTL元分析共检测到6个QTL与角果长度有关,但在A09染色体上未发现与角果长度有关的QTL。而YANG等[9]通过两年的田间试验在186个油菜重组自交系(RIL)的作图群体中A09染色体上探测到10个角果长度QTL,其中一个主效QTL解释表型变异率为53.4%,另外在A01、C02、C03和C04染色体上也发现存在角果长度QTL。漆丽萍[10]检测到11个与角果长度性状相关的QTL,分别分布在A01、A07、A09和A10染色体上。本研究对300份甘蓝型油菜的角果长度性状进行全基因组关联分析,通过GLM模型在农大环境下挖掘了与油菜角果长度显著关联的SNP位点121个,在红壤所试验地环境下找到22个,在A04、A06、A08、A09、C02、C03、C06和C09等8条染色体均有分布,其中A09染色体上分布较为集中,与上述研究比较,本研究找到的角果长度性状关联的SNP位点数较多。而通过MLM模型在2个环境下都仅在A09染色体上找到与角果长度显著关联的SNP位点,以往研究也多在A09染色体上有类似发现,这表明A09染色体上确实存在控制油菜角果长度的基因位点。Table 6
表6
表6角果长度显著关联SNP标记区间候选基因
Table 6candidate genes related with silique length in the intervals of significant association SNP markers
| 基因名称 Gene name | SNP标记 SNP marker | 起点 Start (bp) | 终点 End (bp) | SNP位置 SNP position (bp) | 同源基因名称 Homologous gene name | 同源基因对应物种 Species |
|---|---|---|---|---|---|---|
| GSBRNA2T00092005001 | Bn-A09-p27767046 | 27801685 | 27803935 | 27767046 | SYP73 | 拟南芥 A. thaliana thaliana |
| GSBRNA2T00006767001 | Bn-A09-p27767046 | 27667675 | 27669854 | 27767046 | CYP76C3 | 拟南芥 A. thaliana |
| GSBRNA2T00092007001 | Bn-A09-p27767046 | 27787216 | 27788864 | 27767046 | PAQ1 | 萝卜 Raphanus sativus |
| GSBRNA2T00092005001 | Bn-A09-p27767046 | 27801685 | 27803935 | 27767046 | SYP73 | 拟南芥 A.thaliana |
| GSBRNA2T00049465001 | Bn-A09-p28156822 | 28090658 | 28091698 | 28156822 | IAA30 | 琴叶拟南芥 A. lyrata |
| GSBRNA2T00049463001 | Bn-A09-p28156822 | 28082650 | 28083966 | 28156822 | PIF6 | 拟南芥 A. thaliana |
| GSBRNA2T00049463001 | Bn-A09-p28156822 | 28082650 | 28083966 | 28156822 | PIF6 | 拟南芥 A. thaliana |
| GSBRNA2T00049503001 | Bn-A09-p28156822 | 28211644 | 28213300 | 28156822 | ARR20 | 拟南芥 A.thaliana |
| GSBRNA2T00049509001 | Bn-A09-p28156822 | 28240677 | 28241359 | 28156822 | XT1 | 拟南芥 A.thaliana |
| GSBRNA2T00049485001 | Bn-A09-p28156822 | 28152728 | 28153237 | 28156822 | CP12-2 | 拟南芥 A.thaliana |
| GSBRNA2T00049416001 | Bn-A09-p27948857 | 27875339 | 27878474 | 27948857 | HWS | 琴叶拟南芥 A.lyrata |
新窗口打开
关于调控角果长度的候选基因方面的研究较少,LIU等[28]在A09染色体的p33066395位点附近发现控制角果长度的Auxin-response factor 18(ARF18),并进一步通过转录组分析发现该基因可以在角果壁中通过调控植物激素合成来控制角果细胞壁的生长。本研究通过对A09染色体上角果长度显著关联的SNP位点两侧的基因进行搜寻,通过位置比较分析,检测到多个与ARF18不同的新的角果长度相关的候选基因。如候选基因GSBRNA2T00049485001,其拟南芥的同源基因为CP12-2,与碳水化合物合成代谢有关[32]。而GSBRNA2T00092005001候选基因与花粉管伸长有关,其拟南芥同源基因为SYP73(Syntaxin of plants 73)[33]。而候选基因GSBRNA2T00006774001通过与玉米基因组比对发现为一类伸展蛋白相关的基因,它是细胞壁中的主要结构蛋白,对植物的形态及正常生长发育都具有重要作用[34]。GSBRNA2T00049416001是与器官发育有关的基因,属于F-box蛋白家族,是植物泛素酶体的重要组成部分,通过泛素化途径参与到多种细胞通路,比如花器官发育、信号转导、胁迫响应等重要的生理过程[35]。GSBRNA2T00049503001候选基因的同源基因为ARR20,能够促进植物种子的发育[36]。上述基因是否对角果长度起作用还有待于进一步功能验证。
4 结论
采用GLM模型分析在南昌和进贤两环境下分别探测到121和22个与油菜角果长度显著关联的SNP位点,两地检测到的相同SNP位点为20个。采用MLM模型分析在两地分别探测到5个和3个SNP位点,其中3个SNP位点两地一致。所有两地一致性SNP位点均都位于A09染色体上,基于LD分析结果对A09染色体上显著性SNP位点两侧100 kb区域内基因进行搜寻并进行功能注释,发现多个候选基因参与调节碳水化合物的运输与合成、花器官和种子的发育、信号转导等过程, 它们可能通过上述功能影响油菜角果的生长,导致角果长度的差异。
The authors have declared that no competing interests exist.
