0 引言
【研究意义】甘薯(Ipomoea batatas(L.)Lam.)是世界上第七大主要作物,每年产量达到1.04×109 t,其中95%的产量来自于发展中国家,是生物质能源发展的优势作物[1]。因此,高淀粉含量成为甘薯育种和生物技术研究所关注的重要品质性状。对甘薯具有重要价值的性状进行数量性状位点(QTL)定位,利用与目标性状QTL紧密连锁的遗传标记,对其进行跟踪选择,并分析这些数量性状的遗传规律,将优良的基因逐步累积到栽培品种中,培育具有突破性的高淀粉甘薯新品种,是今后甘薯育种研究的必然趋势。【前人研究进展】甘薯是复杂的同源异源六倍体杂交种(2n=6x=90),染色体数目多且不稳定,杂合性很强,遗传背景复杂。自交不孕以及种内、种间存在广泛的杂交不亲和。近缘野生种利用困难、遗传资源匮乏、又以无性繁殖为主,遗传分析和改良非常困难,极大地制约了甘薯生产的发展[2]。随着分子生物学的发展,“双假测交”理论和分子标记技术的应用,推动了多倍体植物遗传作图的迅速发展。1994年HEMMAT等[3]提出了“双假测交”(double pseudo-testcross)的构想。1994年GRATTAPAGLIA等[4]采用“双假测交”作图方法,利用RAPD技术,分别构建了桉树双亲的遗传图谱。1995年LODHI等[5]采用“双假测交”理论,分别构建了葡萄双亲的分子遗传图谱。“双假测交”作图理论和joinMap软件开发[6]同样为遗传背景复杂、自交不亲和、亲本高度杂合的多倍体植物甘薯遗传图谱的构建提供了方法。多种分子标记被应用于甘薯遗传多样性的研究和遗传图谱构建。1995年ARTHUR等[7]用RAPD标记在甘薯品种“Jewel”无性系材料中,检测出变异在7.1%—35.7%。1996年ARTHUR等[8]对不同来源的芽产生的变异进行了RAPD分析,发现分生组织进行无性繁殖更能保持基因型的一致。1997年THOMPSON等[9]采用RAPD标记分析,构建了具有134个标记的甘薯遗传图谱。1997年UKOSKIT等[10]利用196个RAPD标记构建了一个低密度遗传图谱。2003年KRIEGNER等[11]利用甘薯品种“Tanzania×Bikilamaliya”的杂交F1作图群体,构建了AFLP标记的遗传连锁图,2个亲本分别产生90和80个连锁群,其中632个标记在Tanzania图谱上,435个标记在Bikilamaliya图谱上,连锁图谱长度分别为3 655.6 cM和3 011.5 cM,标记位点间平均图距为5.8 cM。2004年LANDER等[12]在前者的基础上将100—200个SSR共显性标记整合上去,这一作图群体目前大约有500株。2008年CERVANTES- FLORES等[13]利用AFLP技术,以甘薯品种Tanzania×Beauregard杂交后代240个分离群体为材料,构建了双亲分别为86个和90个连锁群的遗传图谱。其中947个标记构建在亲本Tanzania的连锁图谱上,总图距为5 792 cM,平均距离4.5 cM。726个标记构建在亲本Beauregard的连锁图谱上,总图距为5 276 cM,平均距离4.8 cM。2010年LI等[14]利用Luoxushu 8×Zhengshu 20的杂交F1群体,分别构建了含有81个(Luoxushu 8)连锁群(包含473个SRAP标记)的连锁图谱和66个(Zhengshu 20)连锁群(包含328个SRAP标记)的连锁图谱。2013年ZHAO等[15]利用AFLP和SSR技术,分别构建了双亲高密度遗传图谱。徐781连锁图谱包括90个连锁群,总图距为8 151.7 cM,平均距离为4.2 cM。徐薯18连锁图谱总图距为8 184.5 cM,平均距离为3.9 cM。随着国内外关于甘薯遗传图谱的成功构建,甘薯重要性状的QTL定位也开展了大量研究。2005年吴洁等[16]利用构建的甘薯SRAP连锁图。检测到1个淀粉含量QTL,红旗4号的加性效应增加淀粉6.37%,解释表型变异的20.1%。2010年蒲志刚等[17]利用AFLP构建甘薯连锁图,在绵粉1号遗传图的第2连锁群上检测到E1M722可作为淀粉的临近QTL。2010年李爱贤等[16]利用SRAP标记构建了2个亲本的高密度分子连锁图谱。同时检测到了1个主效位点,加性效应为-0.73,解释表型变异的7.70%。2010年唐茜等[18]用BB3-26和潮薯1号的SRAP遗传连锁图谱,检测到1个淀粉含量QTL,BB3-26的加性效应增加淀粉含量2.12%,解释表型变异的21.3%。2011年CERVANTES- FLORES等[19]利用AFLP标记技术,定位了13个干物质含量、12个淀粉含量和8个胡萝卜素含量的QTL。2014年YU等[20]定位了8个甘薯淀粉含量的QTL。【本研究切入点】目前,国内外甘薯遗传图谱大多以AFLP、SRAP、RAPD和少量SSR分子标记技术构建完成,尚未有完全采用EST-SSR标记构建甘薯遗传连锁图谱和淀粉含量QTL定位的研究报道。采用通用性高、易于检测、能直接定位功能基因的EST-SSR标记可以促进在甘薯新性状基因的发现和构建甘薯高密度遗传图谱中发挥重要作用。【拟解决的关键问题】本研究利用甘薯EST-SSR标记分别构建甘薯亲本连锁遗传图谱,并在2年的环境中对甘薯淀粉含量进行QTL定位,为重要农艺性状进行精确分析和分子标记辅助育种奠定基础。1 材料与方法
1.1 试验时间、地点
田间试验于2012—2013年在重庆市北碚区歇马基地(北纬29°45′28″、东经106°22′56″,海拔250 m)和重庆市槽上基地(北纬29°40′18″、东经106°20′30″,海拔750 m)进行,室内试验在西南大学南方山地农业教育部工程研究中心进行。1.2 试验材料
1.2.1 甘薯品种 万薯5号是重庆三峡农业科学院选育出的高产高淀粉品种,该品种红色薯皮、白色薯肉,淀粉率在24%—26%,干率为34.9%—37.2%。高产低淀粉品种商丘52-7系河南省商丘地区农业科学研究所选育,该品种红色薯皮、淡黄色薯肉,淀粉率在8%—12%,干率为17%—21%。1.2.2 群体构建 2012年以万薯5号为母本,商丘52-7为父本在海南陵水基地通过严格的人工杂交技术,共得到F1实生系189株;2012年6月将实生系材料繁殖分为两套,分别将189株材料及亲本在歇马基地和槽上基地种植、收获、留种并检测薯块淀粉含量。2013年6月将全部留种材料繁殖分为两套,再次在歇马基地和槽上基地种植、收获、留种并检测薯块淀粉含量。在2个基地选择全部株系建立标记作图群体。
1.3 试验设计
2012年全部F1实生系群体及亲本在歇马和槽上基地种植,每株系种植3株。2013年每株系采用5 m×0.8 m的单行小区设计。2次重复,收获时亲本及群体株系每重复随机取3个鲜薯,并将鲜薯去皮切丁,薯丁混匀后称取100 g装入培养皿,3次重复,之后摊开转入培养皿并在80℃烘箱中放置48 h,等薯干重量稳定后用天平称量并记录干率数据。分别以2012年、2013年歇马、槽上两点干率的平均值进行数据统计分析,干率与淀粉率的换算公式为淀粉率(%)=干率(%)×0.86945-6.34587。1.4 总DNA提取、引物选择和SSR分析
采用改良CTAB法[21]提取亲本及F1实生系幼嫩植株叶片的DNA,1%琼脂糖凝胶电泳检测所提DNA的纯度和完整性,-20℃保存备用。EST-SSR引物来源于广东农业科学院作物科学研究所和西南大学薯类作物研究所等开发设计的有关甘薯EST-SSR标记[22-26]。引物序列由上海英骏和上海生工公司合成。反应体系为10 μL,包括1.2 μL 10×buffer(含Mg2+ 15 mmol·L-1)、0.2 μL dNTP、1 μL引物、0.1 μL Taq酶、1 μL模板DNA(约50 ng·μL -1)、5.5 μL ddH2O。在ABI9700PCR仪上进行。反应程序为94℃ 5 min;94℃ 45 s,55℃ 45 s,72℃ 1 min,35个循环;72℃ 5 min;25℃ 5 min,最后4℃冰箱保存备用。SSR扩增产物经8%的非变性聚丙烯酰胺凝胶电泳分离,采用银染法检测。1.5 亲本间标记多态性与杂交群体(F1)标记基因型检测
利用不同的甘薯EST-SSR引物系列对亲本万薯5号、商丘52-7进行多态性筛选,在亲本中表现多态性的引物被用于检测杂交群体189个单株的多态性。根据位点分离类型,依照Joinmap4.0说明书中的标准基因型分析方法对杂交群体中189个单株分别记录其基因型代码。与亲本万薯5号具有相同带型记为lm,没有记为ll,与亲本商丘52-7具有相同带型记为np,没有记为nn;在亲本万薯5号和亲本商丘52-7中均有,但在杂交后代中发生分离的也按照以上标准记录。只有清晰的带型才被记录,缺失和模糊带型用“--”表示。卡方检测用于检测位点的偏分离情况。1.6 遗传连锁图谱的构建
遗传连锁图谱的构建采用作图软件JoinMap4.0[27],以LOD≥5.0,Kosambi作图函数进行连锁分析。2个亲本连锁群分别命名,万薯5号和商丘52-7连锁图谱分别用“W”和“S”表示,其小数点后的数字01—90表示所属连锁群的序号。1.7 标记位点命名
标记命名为引物名后加一大写字母,A/B/C/D表示同一EST-SSR引物扩增出的不同位点。如标记SIP272B表示标记SIP272的第二条多态性带;引物名后加一大写字母再加数字,表示双亲本共显但后代发生分离的四种标记命名,如SIP272D3表示共显性标记SIP272的第四条多态性带的第三种分离带型。1.8 QTL定位
采用MapQTL5.0软件对甘薯淀粉含量进行统计分析[28],确定不同水平淀粉含量在群体中的分布频率。利用QTL分析软件MapChart 2.2对SSR数据进行分析[29],采用区间作图法(interval mapping,IM)对全基因组进行QTL扫描,选择1.0 cM的步长,参数为1 000次回归,显著水平为0.01,以LOD≥3.0作为阈值对淀粉含量可能存在的QTL进行定位和效应估计。加性效应以万薯5号的等位基因为背景,即正效应表示万薯5号的等位基因增加性状表型值,负效应表示万薯5号的等位基因减少性状表型值,而商丘52-7的等位基因效应相反。2 结果
2.1 甘薯EST-SSR分子标记引物多态性分析
利用1 679对EST-SSR引物筛选亲本间的引物多态性,共获得1 045对多态性引物,利用1 045对多态性引物检测189个F1群体的标记基因型,每个标记检测到有1—5个位点,共获得了1 418个标记位点,含共显现标记位点166个,占11.7%。1 418个标记位点中属于母本的标记位点642个,属于父本的标记位点776个。2.2 甘薯EST-SSR分子标记遗传连锁图谱的构建
利用JoinMap4.0软件分别对上述获得的父母本多态性标记进行遗传连锁分析,在LOD≥5.0情况下,分别构建父母本的连锁遗传图谱。其中属于母本的642个标记位点构建了74个连锁群,共有215个标记位点构建在连锁图谱上,占标记总数的33.5%,每个连锁群上有2—11个标记位点,连锁群长度在0.6—129.4 cM,图谱总长为3 826.07 cM,标记间平均距离为17.80 cM。属于父本的776个标记位点构建了80个连锁群,共有252个标记位点构建在连锁图谱上,占标记总数的32.5%,每个连锁群上有2—24个标记位点,连锁群长度在2.0—156.8 cM,图谱总长为3 955.0 cM,标记间平均距离为15.7 cM(表1,图1—图2)。Table 1
表1
表1遗传连锁图谱中连锁群的图距和分布
Table 1The genetic distance and distribution of markers in the linkage groups
| 连锁群 Linkage group | 标记数 Number of markers | 长度 Distance (cM) | 平均图距 Average distance (cM) | 连锁群 Linkage group | 标记数 Number of markers | 长度 Distance (cM) | 平均图距 Average distance (cM) | |
|---|---|---|---|---|---|---|---|---|
| W.01 | 11 | 63.9 | 5.8 | S.01 | 24 | 156.8 | 6.5 | |
| W.02 | 2 | 2.2 | 1.1 | S.02 | 3 | 81.6 | 27.2 | |
| W.03 | 3 | 39.3 | 13.1 | S.04 | 5 | 91.1 | 18.2 | |
| W.04 | 3 | 36.8 | 12.3 | S.05 | 4 | 43.2 | 10.8 | |
| W.05 | 3 | 54.6 | 18.2 | S.06 | 2 | 16.6 | 8.3 | |
| W.06 | 2 | 31.5 | 15.8 | S.07 | 3 | 44.3 | 14.8 | |
| W.07 | 2 | 34.1 | 17.1 | S.08 | 3 | 52.6 | 17.5 | |
| W.08 | 3 | 64.3 | 21.4 | S.09 | 3 | 51.7 | 17.2 | |
| W.09 | 5 | 72.1 | 14.4 | S.10 | 2 | 29.1 | 14.5 | |
| W.10 | 3 | 9.1 | 3.0 | S.11 | 2 | 29.6 | 14.8 | |
| W.11 | 3 | 24.2 | 8.1 | S.12 | 2 | 16.8 | 8.4 | |
| W.12 | 3 | 62.6 | 20.9 | S.13 | 2 | 45.7 | 22.8 | |
| W.13 | 2 | 54.9 | 27.5 | S.14 | 4 | 98.1 | 24.5 | |
| W.14 | 3 | 62.8 | 20.9 | S.15 | 2 | 29.6 | 14.8 | |
| W.15 | 2 | 29.5 | 14.8 | S.16 | 2 | 36.9 | 18.4 | |
| W.16 | 2 | 22.6 | 11.3 | S.17 | 5 | 93.8 | 18.8 | |
| W.17 | 2 | 24.9 | 12.5 | S.18 | 5 | 107.3 | 21.5 | |
| W.18 | 2 | 7.9 | 4.0 | S.19 | 5 | 74.7 | 14.9 | |
| W.19 | 2 | 69.3 | 34.7 | S.20 | 2 | 54.4 | 27.2 | |
| W.20 | 7 | 129.4 | 18.5 | S.21 | 3 | 43.0 | 14.3 | |
| W.21 | 8 | 114.6 | 14.3 | S.22 | 5 | 111.2 | 22.2 | |
| W.22 | 4 | 81.9 | 20.5 | S.23 | 5 | 101.8 | 20.4 | |
| W.23 | 7 | 116.4 | 16.6 | S.24 | 4 | 116.6 | 29.1 | |
| W.24 | 5 | 47.2 | 9.4 | S.25 | 2 | 4.9 | 2.4 | |
| W.25 | 6 | 123.6 | 20.6 | S.26 | 2 | 50.4 | 25.2 | |
| W.26 | 5 | 108.4 | 21.7 | S.27 | 3 | 34.9 | 11.6 | |
| W.27 | 4 | 67.7 | 16.9 | S.28 | 3 | 62.1 | 20.7 | |
| W.28 | 3 | 66.7 | 22.2 | S.29 | 2 | 35.8 | 17.9 | |
| W.29 | 4 | 70.07 | 17.5 | S.30 | 2 | 18.0 | 9.0 | |
| W.30 | 2 | 59.5 | 29.8 | S.31 | 6 | 124.2 | 20.7 | |
| W.31 | 4 | 65.4 | 16.4 | S.32 | 2 | 39.6 | 19.8 | |
| W.32 | 4 | 94.8 | 23.7 | S.33 | 5 | 79.3 | 15.9 | |
| W.33 | 4 | 86.1 | 21.5 | S.34 | 4 | 87.3 | 21.8 | |
| W.34 | 2 | 42 | 21.0 | S.35 | 5 | 100.8 | 20.2 | |
| W.35 | 2 | 78 | 39.0 | S.36 | 2 | 47.0 | 23.5 | |
| W.36 | 3 | 45.8 | 15.3 | S.37 | 2 | 28.5 | 14.3 | |
| W.37 | 2 | 57.4 | 28.7 | S.38 | 3 | 74.3 | 24.8 | |
| W.38 | 2 | 11 | 5.5 | S.39 | 3 | 81.2 | 27.1 | |
| W.39 | 2 | 57.3 | 28.7 | S.40 | 3 | 67.4 | 22.5 | |
| W.40 | 2 | 57.2 | 28.6 | S.41 | 2 | 9.6 | 4.8 | |
| W.41 | 3 | 71.7 | 23.9 | S.42 | 2 | 8.7 | 4.3 | |
| W.42 | 3 | 23.4 | 7.8 | S.43 | 2 | 25.0 | 12.5 | |
| W.43 | 2 | 46 | 23.0 | S.44 | 2 | 10.6 | 5.3 | |
| W.44 | 2 | 53.8 | 26.9 | S.45 | 2 | 14.1 | 7.1 | |
| W.45 | 2 | 46.8 | 23.4 | S.46 | 2 | 28.2 | 14.1 | |
| W.46 | 2 | 41.1 | 20.6 | S.47 | 2 | 7.6 | 3.8 | |
| W.47 | 2 | 5.7 | 2.9 | S.48 | 2 | 32.6 | 16.3 | |
| W.48 | 2 | 80.5 | 40.3 | S.49 | 5 | 94.5 | 18.9 | |
| W.49 | 2 | 19.9 | 10.0 | S.50 | 4 | 88.8 | 22.2 | |
| W.50 | 2 | 38.3 | 19.2 | S.51 | 2 | 11.0 | 5.5 | |
| W.51 | 2 | 11.4 | 5.7 | S.52 | 3 | 21.8 | 7.3 | |
| W.52 | 2 | 42.4 | 21.2 | S.53 | 2 | 38.3 | 19.1 | |
| W.53 | 2 | 72.2 | 36.1 | S.54 | 8 | 70.9 | 8.9 | |
| W.54 | 2 | 7.1 | 3.6 | S.55 | 8 | 95.4 | 11.9 | |
| W.55 | 2 | 36.3 | 18.2 | S.56 | 3 | 46.0 | 15.3 | |
| W.56 | 3 | 43 | 14.3 | S.57 | 4 | 79.0 | 19.7 | |
| W.57 | 5 | 97 | 19.4 | S.58 | 2 | 57.8 | 28.9 | |
| W.58 | 2 | 75.4 | 37.7 | S.59 | 3 | 25.3 | 8.4 | |
| W.59 | 2 | 42.9 | 21.5 | S.60 | 2 | 29.8 | 14.9 | |
| W.60 | 2 | 38.1 | 19.1 | S.61 | 2 | 11.2 | 5.6 | |
| W.61 | 3 | 93.2 | 31.1 | S.62 | 4 | 63.2 | 15.8 | |
| W.62 | 2 | 48.5 | 24.3 | S.63 | 2 | 15.3 | 7.7 | |
| W.63 | 2 | 52.2 | 26.1 | S.64 | 2 | 40.7 | 20.3 | |
| W.64 | 2 | 63.1 | 31.6 | S.65 | 2 | 11.9 | 5.9 | |
| W.65 | 2 | 22.3 | 11.2 | S.66 | 3 | 54.8 | 18.3 | |
| W.66 | 2 | 39.1 | 19.6 | S.67 | 2 | 78.3 | 39.1 | |
| W.67 | 2 | 16.7 | 8.4 | S.68 | 2 | 66.1 | 33.0 | |
| W.68 | 2 | 33 | 16.5 | S.69 | 2 | 43.6 | 21.8 | |
| W.69 | 2 | 78 | 39.0 | S.70 | 2 | 55.5 | 27.7 | |
| W.70 | 2 | 64.4 | 32.2 | S.71 | 3 | 44.4 | 14.8 | |
| W.71 | 2 | 2 | 1.0 | S.72 | 2 | 29.3 | 14.6 | |
| W.72 | 2 | 27.4 | 13.7 | S.73 | 2 | 7.9 | 3.9 | |
| W.73 | 2 | 45.5 | 22.8 | S.74 | 2 | 14.4 | 7.2 | |
| W.74 | 2 | 0.6 | 0.3 | S.75 | 2 | 39.5 | 19.8 | |
| 合计Total | 215 | 3826.07 | 17.80 | S.76 | 2 | 42.0 | 21.0 | |
| S.77 | 2 | 16.7 | 8.4 | |||||
| S.78 | 2 | 5.7 | 2.9 | |||||
| S.79 | 2 | 55.5 | 27.8 | |||||
| S.80 | 2 | 2.0 | 1.0 | |||||
| 合计Total | 252 | 3955.0 | 15.7 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
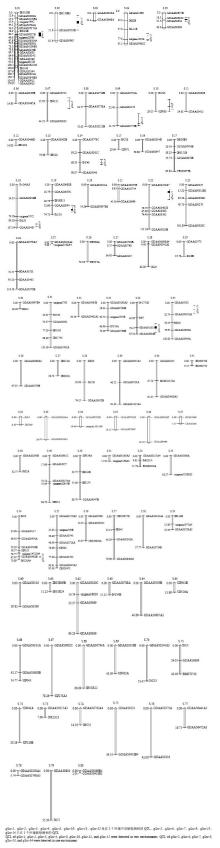
显示原图|下载原图ZIP|生成PPT图1万薯5号EST-SSR分子标记连锁图及淀粉含量QTL的分布
-->Fig. 1The EST-SSR linkage map of Wanshu 5 and distribution of QTLs for starch content
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2商丘52-7 EST-SSR分子标记连锁图及淀粉含量QTL的分布
-->Fig. 2The EST-SSR linkage map of Shangqiu52-7 and distribution of QTLs for starch content
-->
2.3 甘薯淀粉含量的表型分析
通过对亲本及后代群体甘薯淀粉含量的测定(表2),结果表明,淀粉含量在后代群体中差异较明显,亲本万薯5号的淀粉含量在2012年和2013年分别为24.52%和23.10%,商丘52-7分别为4.86%和4.35%,因此,万薯5号的淀粉含量明显高于商丘52-7。F1后代有超亲表现,且呈连续分布,说明淀粉含量性状是受多基因控制的数量性状,群体符合QTL定位的要求。通过对F1群体方差分析表明(表3)。淀粉含量不仅受基因型影响外,还受环境条件的影响。Table 2
表2
表2亲本及F1群体淀粉含量的表型分析
Table 2Phenotypic analysis of starch content of the parents and F1 families
| 性状 Trait | 年份 Year | 亲本Parent | F1群体 F1 Population | |||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 万薯5号(♀) Wanshu5 | 商丘52-7(♂) Shangqiu 52-7 | 最小值 Min | 最大值 Max | 均值 Mean | 标准差 SD | 变异系数 CV(%) | 峰值 Kurtosis | 偏度 Skewness | ||
| 淀粉含量 Starch content (%) | 2012 | 24.52 | 4.86 | 6.50 | 28.99 | 16.35 | 4.48 | 27.40 | -0.08 | 0.46 |
| 2013 | 23.10 | 4.35 | 7.87 | 28.62 | 15.85 | 4.11 | 25.93 | 0.26 | 0.68 | |
新窗口打开
Table 3
表3
表3F1群体淀粉含量的方差分析
Table 3Variance analysis of the starch content of F1 populations
| 性状Trait | 变异来源 Source of variation | 平方和 SS | 自由度 df | 均方 Variances | F |
|---|---|---|---|---|---|
| 淀粉含量 Starch content (%) | 年份Year | 22.1198 | 1.00 | 22.12 | 8.339** |
| 基因型Genotype | 6304.3922 | 188.00 | 33.53 | 12.643** | |
| 误差Error | 498.664 | 188 | 2.6525 | ||
| 总变异Total variation | 6825.1759 | 377 |
新窗口打开
由频率分布图(图3)可知,2012年和2013年(两点平均)甘薯的淀粉含量指标在群体中表现明显正态分布,在后代群体中分布的峰度和偏度均小于1,且均有一定数量的超亲类型存在,呈现为连续变异,变异系数分别为19.74%和18.51%,具有数量性状遗传的典型特点,适于进一步QTL分析。
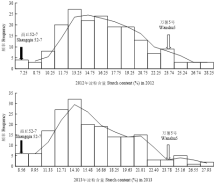 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3淀粉含量的频率分布图
-->Fig. 3The frequency distribution of starch content
-->
2.4 甘薯淀粉含量的QTL分析
利用区间作图法在2012年和2013年共检测到17个与淀粉含量性状相关的QTL。其中,3个位于母本万薯5号遗传图谱连锁群上,14个位于父本商丘52-7遗传图谱连锁群上(表4,图1—图2)。qWsc-1定位在母本的W.01连锁群上,且在2年中均被检测到,置信区间分别为21.70—26.77,与最近的标记GDAAS0603A平均距离为2.5 cM,其2年平均贡献率为31.9%。qWsc-1母本万薯5号的等位基因为负的加性效应,减少性状表型值,2年分别为-2.93和-3.04。
qWsc-2定位在母本的W.10连锁群上,且在2年中均被检测到,置信区间为3.00—8.74,与最近的标记GDAAS0163A2平均距离为1.0 cM,其2年平均贡献率为11.05%。qWsc-2母本万薯5号的等位基因为加性效应,增加性状表型值,2年分别为+3.43和+2.54。
qWsc-3定位在母本的W.28连锁群上,且在2年中均被检测到,置信区间为0.00—29.00,与最近的标记SIP010距离2年平均为5.0 cM,其2年平均贡献率为17.2%。qWsc-3母本万薯5号的等位基因为负的加性效应,减少性状表型值,2年分别为-2.01和-1.55。
qSsc-1定位在父本的S.01连锁群上,且在2年中均被检测到,置信区间为62.07—72.35,与最近的标记GDAAS0809A的距离2年平均为1.5 cM,其2年平均贡献率为33.5%。qSsc-1母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+3.74和+3.17。
qSsc-2定位在父本的S.02连锁群上,且在2年中均被检测到,置信区间为8.00—70.04,与最近的标记GDAAS0519B距离2年平均为24.0 cM,其2年平均贡献率为23.75%。qSsc-2母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+2.28和+1.96。
qSsc-3定位在父本的S.04连锁群上,且在2年中均被检测到,置信区间为60.89—81.6,在标记unigene12034的位置,其2年平均贡献率为10.10%。qSsc-3母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+1.49和+1.25。
qSsc-4定位在父本的S.05连锁群上,且在,2年中均被检测到,置信区间为14.00—26.68,在标记GDAAS0603C的的位置,其2年平均贡献率为10.15%。qSsc-4母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+1.46和+1.29。
qSsc-5定位在父本的S.07连锁群上,只在2013年中检测到,置信区间为8.00—24.92,在标记IBS121的位置,其2013年贡献率为8.4%。qSsc-5母本万薯5号的等位基因为加性效应,增加性状表型值为+1.21。
qSsc-6定位在父本的S.09连锁群上,只在2013年中检测到,置信区间为32.40—51.70,在标记GDAAS0776B的位置,其2013年贡献率为8.4%。qSsc-6母本万薯5号的等位基因为加性效应,增加性状表型值为+1.21。
qSsc-7定位在父本的S.10连锁群上,只在2012年中检测到,置信区间为10.00—29.051,与最近的标记SIP003的距离6.05 cM,其2012年贡献率为14.6%。qSsc-7母本万薯5号的等位基因为加性效应,增加性状表型值为+1.77。
qSsc-8定位在父本的S.14连锁群上,且在2年中均都检测到,置信区间为60.20—75.33,在标记IbY40的位置,其两年平均贡献率为13.10%。qSsc-8母本万薯5号的等位基因为加性效应,增加性状表型值,2年分别为+1.65和+1.57。
qSsc-9定位在父本的S.18连锁群上,只在2012年中检测到,置信区间为97.02—105.02,与最近的标记GDAAS0485的距离5.3 cM,其2012年贡献率为24.4%。qSsc-9母本万薯5号的等位基因为加性效应,增加性状表型值为+2.35。
qSsc-10定位在父本的S.19连锁群上,且在2年中均都检测到,置信区间为52.6—68.6,与最近的标记GDAAS0894的距离两年平均为10.0 cM,其2年平均贡献率为24.05%。qSsc-10母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+2.24和+1.17。
qSsc-11定位在父本的S.22连锁群上,且在2年中均都检测到,置信区间为0.00—38.00,与最近的标记GDAAS0479A的距离两年平均为18.5 cM,其2年平均贡献率为27.5%。qSsc-11母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+2.35和+2.20。
qSsc-12定位在父本的S.34连锁群上,且在2年中均都检测到,置信区间为60.83—85.17,与最近的标记GDAAS0102的距离两年平均为2.1 cM,其2年平均贡献率为14.15%。qSsc-12母本万薯5号的等位基因为加性效应,增加性状表型值2年分别为+1.74和+1.57。
qSsc-13定位在父本的S.35连锁群上,只在2012年中检测到,置信区间为10.00—32.74,与最近的标记GDAAS0035A3的距离12.74 cM,其2012年贡献率为19.2%。qSsc-13母本万薯5号的等位基因为加性效应,增加性状表型值为+1.96。
qSsc-14定位在父本的S.54连锁群上,只在2013年中检测到,置信区间为68.64—70.88,与最近的标记IbU20A4的距离1.44 cM,其2013年贡献率为25.1%。qSsc-14母本万薯5号的等位基因为加性效应,增加性状表型值为+2.52。
Table 4
表4
表4甘薯淀粉含量的QTL及其效应分析
Table 4QTL loci and effects on starch content of sweet potato
| QTL位点 QTL loci | 年份 Year | 连锁群 Group | 峰值位置 Peak position | 置信区间 Confidence interval | 最近标记 Nearest marker | LOD值 | 加性效应a1 Additive | 加性效应a2 Additive | 贡献率 Expl. (%) |
|---|---|---|---|---|---|---|---|---|---|
| qWsc-1 | 2012 | W.01 | 23.77 | 21.70—26.76 | GDAAS0603A | 10.57 | -2.93 | 0.00 | 28.6 |
| 2013 | W.01 | 24.77 | 21.77—26.77 | GDAAS0603A | 13.44 | -3.04 | 0.00 | 35.2 | |
| qWsc-2 | 2012 | W.10 | 6.75 | 4.74—8.74 | GDAAS0163A2 | 4.08 | +3.43 | 0.00 | 12.0 |
| 2013 | W.10 | 4.75 | 3.00—3.44 | GDAAS0163A2 | 4.08 | +2.54 | 0.00 | 10.1 | |
| qWsc-3 | 2012 | W.28 | 7.00 | 0.00—27.00 | SIP010 | 4.67 | -2.01 | 0.00 | 20.1 |
| 2013 | W.28 | 6.00 | 0.00—29.00 | SIP010 | 3.16 | -1.55 | 0.00 | 14.3 | |
| qSsc-1 | 2012 | S.01 | 71.35 | 70.35—72.35 | GDAAS0809A | 15.66 | 0.00 | +3.74 | 40.5 |
| 2013 | S.01 | 63.67 | 62.67—66.58 | GDAAS0809A | 14.92 | 0.00 | +3.17 | 36.5 | |
| qSsc-2 | 2012 | S.02 | 24.00 | 8.00—50.00 | GDAAS0519B | 5.42 | 0.00 | +2.28 | 25.0 |
| 2013 | S.02 | 31.00 | 9.00—70.04 | GDAAS0519B | 5.98 | 0.00 | +1.96 | 22.5 | |
| qSsc-3 | 2012 | S.04 | 77.60 | 64.9—81.6 | unigene12034 | 4.31 | 0.00 | +1.49 | 11.0 |
| 2013 | S.04 | 77.60 | 60.89—80.59 | unigene12034 | 3.62 | 0.00 | +1.25 | 9.2 | |
| qSsc-4 | 2012 | S.05 | 18.46 | 14—21.46 | GDAAS0603C | 4.33 | 0.00 | +1.46 | 10.5 |
| 2013 | S.05 | 19.46 | 14.00—26.68 | GDAAS0603C | 4.04 | 0.00 | +1.29 | 9.8 | |
| qSsc-5 | 2013 | S.07 | 12.92 | 8.00—24.92 | IBS121 | 3.51 | 0.00 | +1.21 | 8.4 |
| qSsc-6 | 2013 | S.09 | 48.40 | 32.40—51.70 | GDAAS0776B | 3.91 | 0.00 | +1.35 | 10.8 |
| qSsc-7 | 2012 | S.10 | 22.00 | 10.00—29.051 | SIP003 | 4.35 | 0.00 | +1.77 | 14.6 |
| qSsc-8 | 2012 | S.14 | 68.33 | 60.21—75.33 | IbY40 | 5.41 | 0.00 | +1.65 | 12.7 |
| 2013 | S.14 | 68.33 | 60.20—73.32 | IbY40 | 5.81 | 0.00 | +1.57 | 13.5 | |
| qSsc-9 | 2012 | S.18 | 101.02 | 97.02—105.02 | GDAAS0485 | 6.64 | 0.00 | +2.35 | 24.4 |
| qSsc-10 | 2012 | S.19 | 61.60 | 52.6—68.6 | GDAAS0894 | 5.99 | 0.00 | +2.24 | 25.1 |
| 2013 | S.19 | 63.60 | 52.6—68.6 | GDAAS0894 | 5.06 | 0.00 | +1.97 | 23.0 | |
| qSsc-11 | 2012 | S.22 | 17.00 | 0.00—35.00 | GDAAS0479A | 6.04 | 0.00 | +2.35 | 26.7 |
| 2013 | S.22 | 20.00 | 0.00—38.00 | GDAAS0479A | 5.91 | 0.00 | +2.20 | 28.0 | |
| qSsc-12 | 2012 | S.34 | 75.17 | 70.17—85.17 | GDAAS0102 | 5.07 | 0.00 | +1.74 | 14.5 |
| 2013 | S.34 | 70.17 | 60.83—80.17 | GDAAS0102 | 5.88 | 0.00 | +1.57 | 13.8 | |
| qSsc-13 | 2012 | S.35 | 21.00 | 10.00—32.74 | GDAAS0035A3 | 4.50 | 0.00 | +1.96 | 19.2 |
| qSsc-14 | 2013 | S.54 | 69.65 | 68.64—70.88 | IbU20A4 | 7.42 | 0.00 | +2.52 | 25.1 |
新窗口打开
3 讨论
3.1 甘薯作图标记的选用
分子标记是指基于生物个体之间DNA序列的差异而建立的遗传标记,是构建遗传连锁图谱、基因定位、目标性状分子标记筛选等研究的理想工具。衡量遗传图谱质量的一个重要标准是标记分布的均匀程度,而标记类型是影响连锁群上的标记均匀分布与否的重要因素之一。目前用于甘薯研究的分子标记主要包括AFLP、RAPD、RFLP、SSR、EST-SSR,其中EST-SSR标记成为构建遗传图谱的新资源。EST-SSR标记是采用高通量数据直接测序开发,cDNA又不含内含子,300—400 bp的EST-SSR就能反映出基因的性质,具有多、快、好、省的特点。同时EST-SSR标记来自转录基因,可有效揭示基因的转录功能,标记间的定位关系就是对基因组序列的定位,因而可直接用于甘薯遗传图谱构建和功能基因作图,加速了甘薯新性状基因的发现,所以EST-SSR标记技术将在构建甘薯遗传图谱中发挥重要的作用。
由于EST-SSR引物的特异性和开发的成本较高,目前,开发出的有关甘薯的EST-SSR引物较少,利用EST-SSR引物构建的甘薯遗传图谱更少。本研究利用开发的EST-SSR引物[22-26],首次开展了全部基于EST-SSR标记的甘薯遗传图谱构建研究,为以后甘薯图谱构建研究提供参考标记,为甘薯重要品质性状的QTL定位提供更多的研究方法和分子标记。
3.2 甘薯的遗传图谱
甘薯(2n=6x=90)是多倍体无性繁殖植物,因为同时具有其染色体数量多还不稳定;自交不亲和同一孕群内品种间杂交不亲和;材料间异交导致其遗传组成高度杂合,且F1就大量分离等特点,是公认的图谱构建、QTL定位等遗传学研究较困难的主要农作物[30]。THOMPSON等[9]采用RAPD标记分析了引物的多态性,并构建甘薯遗传图谱。JIE等[30]、KRIEGNER等[11]和CERVANTES-FLORES等[13]利用AFLP技术和“双假测交”策略分别构建了不同群体、连锁群数量不等的双亲的遗传图谱,蒲志刚等[17]利用AFLP技术构建了连锁群数量较少的遗传图谱。吴洁等[16]和唐茜等[18]利用SRAP标记构建了连锁群数量较少的遗传图谱,但李爱贤等[14]利用SRAP标记构建了2个亲本的、连锁群数量多的高密度分子连锁图谱。ZHAO等[15]和LI等[31]利用AFLP和SSR两种类型标记,构建2个亲本密度高、连锁群数量多的遗传图谱。以上研究只有吴洁等[14]、唐茜等[17]和蒲志刚等[15]利用杂交群体,构建的是群体的图谱,但均有连锁群数量少、图谱密度较小的不足。其他研究均采用了“双假测交”策略,构建的是双亲的遗传图谱。本研究运用EST-SSR标记,采用JoinMap4.0软件和“双假测交”策略分别构建父母本的连锁遗传图谱。有215个标记位点构建在母本连锁图谱上,252个标记位点构建在父本连锁图谱上,是目前构建的EST-SSR标记数量最多的甘薯遗传图谱。母本74个连锁群图谱总长为3 826.07 cM,标记间平均距离为17.80 cM,父本80个连锁群图谱总长为3 955.0 cM,标记间平均距离为15.7 cM,是连锁群数量多、密度中等的遗传图谱。
本研究构建的遗传图谱连锁群数量母本为74个,父本为80个,与实际甘薯90个连锁群数目有差异,主要是因为EST-SSR标记在染色体上分布是随机的,染色体上不同区域的交换值具有异质性,出现大量小片段的连锁群或连锁上有较大的空隙。同时,甘薯为区段异源多倍体(多数同源),遗传表现与正常的物种不同,在分子标记条带记录上未表现出来,导致很多标记不能与连锁群对应,也可能导致结论出现差异。增加群体数量、增加标记数量、增加标记种类有可能解决这些问题。
3.3 甘薯的QTL定位
淀粉含量是甘薯的主要品质性状,高淀粉含量的甘薯品种具有更高的利用价值,甘薯块根干物质含量又与淀粉含量显著正相关,控制淀粉含量和干物质含量的QTL定位是甘薯研究的重点。CERVANTES- FLORES等[19]的研究结果表明,淀粉含量受多基因控制的数量性状,表现型是基因型和环境共同作用的结果。吴洁等[16]检测到1个淀粉含量QTL,红旗4号的加性效应增加淀粉6.37%,解释表型变异的20.1%。蒲志刚等[17]在绵粉1号遗传图的第2连锁群上检测到E1M722可作为淀粉的临近QTL。李爱贤等[14]检测到了1个主效位点,位于父本郑薯20的Z31连锁群上,QTL的加性效应为-0.73,解释表型变异的7.70%。唐茜等[18]检测到1个淀粉含量QTL,BB3-26的加性效应增加淀粉含量2.12%,解释表型变异的21.3%。ZHAO等[15]检测到27个干物质含量QTL,供献率在9.0%—45%。LI等[31]定位了9个与块根产量相关的主效QTL。以上研究定位的与淀粉含量相关的QTL较少,并且多数只是在1个环境中被检测到,没有考虑多个环境的影响。本研究利用EST-SSR技术,在2012年和2013年共检测到17个与淀粉含量性状相关的QTL,贡献率在8.4%—40.5%。其中,3个位于母本万薯5号遗传图谱连锁群上,且在2年中均检测到相同的标记。14个位于父本商丘52-7遗传图谱连锁群上,且在2年中有8个均检测到相同的标记。由于采用的是不同标记类型和作图群体,定位的QTL无法进行相互比较和验证,这也是未来研究的方向。
3.4 QTL定位图谱分布及加性效应
采用不同的作图群体,检测到的QTL在亲本图谱上分布具有差异,其加性效应也有差异。与前人研究相比,CERVANTES-FLORES等[19]检测到13个干物质含量QTL。5个位于高干物质亲本连锁图谱上,其中,4个具有正加性效应,1个具有负加性效应;8个位于低干物质亲本连锁图谱上,其中4个具有正加性效应,4个具有负加性效应;同时还检测到12个淀粉含量的QTL,5个位于高淀粉亲本连锁图谱上,其中,3个具有正加性效应,2个具有负加性效应;7个位于低淀粉亲本连锁图谱上,其中,3个具有正加性效应,4个具有负加性效应。ZHAO等[15]检测到的27个干物质含量QTL,22个定位在高干率亲本徐781的遗传图谱上,其中20个具有加性效应,增加性状的表型值。5个定位在低干率亲本徐薯18图谱上,其中只有1个具有加性效应,增加性状的表型值。LI等[31]定位的9个与块根产量相关的主效QTL。4个定位在高产亲本徐薯18的遗传图谱上,其中2个具有加性效应,增加性状的表型值。5个定位在低产亲本徐781的遗传图谱上,其中只有1个具有加性效应,增加性状的表型值。本研究定位的17个QTL,对于高淀粉亲本万薯5号的等位基因,15个QTL的加性效应为正值,增加淀粉性状的表现值,其中1个位于高淀粉亲本万薯5号遗传图谱连锁群上,14个位于低淀粉亲本商丘52-7遗传图谱连锁群上。只有2个QTL加性效应为负值,减少淀粉性状的表型值,定位在高淀粉亲本万薯5号遗传图谱连锁群上。出现这些差异可能具有2个方面的原因:一是选用的材料不同,万薯5号是超高淀粉含量的甘薯品种(25%左右),而商丘52-7是超低淀粉含量的甘薯品种(5%左右),在一定概率范围内子代大多数超过低淀粉含量亲本,增加表型性状,表现为加性效应;二是采用“双假测交”的作图方法,双亲分别作图,控制高淀粉的等位基因在低淀粉性状亲本的遗传图谱上也能检测到。这也印证了CERVANTES- FLORES等[19]在高淀粉育种中亲本淀粉含量高有利于子代有利基因的积累的结论。
3.5 本研究需要说明的问题
由于采用的是不同分子标记和研究群体,分子标记间缺乏一致性,不同群体构建的连锁图谱覆盖的染色体区域存在差异,限制了遗传图谱和QTL间的相互比较。同时,由于公布的EST-SSR标记数量太少,且本研究尚有大量的标记不能构建在连锁遗传图谱上,造成2—3个标记的连锁群数量偏多。为了构建高密度的甘薯分子连锁图谱,可增加EST-SSR标记的数量和群体的数量,或者添加更多的标记种类,有利于甘薯QTL精细定位的后续研究。4 结论
以高淀粉含量品种万薯5号为母本、低淀粉含量品种商丘52-7为父本建立了189个杂交群体,分别构建了基于EST-SSR标记的双亲遗传连锁 图谱。其中母本构建了74个连锁群,图谱总长为3 826.07 cM,标记间平均距离为17.80 cM。父本构建了80个连锁群,图谱总长为3 955.0 cM,标记间平均距离为15.7 cM。检测到17个与淀粉含量性状相关的QTL,贡献率在8.4%—40.5%。其中3个位于母本万薯5号遗传图谱连锁群上,且在2年中均检测到相同的标记;14个位于父本商丘52-7遗传图谱连锁群上,且在2年中有8个均检测到相同的标记,标记GDAAS0603在双亲中和2个环境中同时检测到,这些环境稳定QTL可用于分子标记辅助选择。该研究丰富了构建甘薯遗传图谱的标记类型。(责任编辑 李莉)
The authors have declared that no competing interests exist.
