0 引言
【研究意义】桃(Prunus persica L.)作为中国重要的落叶果树树种之一,栽培历史悠久,因其果实营养丰富、风味鲜美,深受人们的喜爱。分子生物学和生物信息学技术的发展以及桃基因组草图的公布,为研究和揭示桃生长和发育的分子机理奠定了基础。研究‘鲁星’桃(Prunus persica var. nectarina ‘Luxing’)中MADS-box,对探索其在花及果实发育过程中的作用具有重要参考意义。【前人研究进展】在植物中,MADS-box转录因子是一个大家族转录调控子,是植物界中研究最为广泛的基因家族之一,在植物的花和果实发育及成熟等多个过程中具有重要的调控作用;其名称源于4个物种的MADS-box,即酿酒酵母的MCM1、拟南芥的AGAMOUS、金鱼草的DEFICIENS及人类SRF4的首字母,在其N末端含有一段约为60个氨基酸组成的高度保守结构域,称之为MADS-box结构域,负责识别和绑定靶基因中调控区域的CArG盒[CC(A/T)6GG][1-3]。根据结构域的不同,植物MADS-box基因家族可分为2个亚家族:Type I和Type II。Type I可继续分为Mα、Mβ、Mγ和Mδ组;Type II又称为MIKC类型,因其含有4个保守结构域:MADS(M)盒、Intervening(I)区域、Keratin(K)盒和C-terminal(C)末端[4]。此外,根据上述保守域特征,MIKC类型可进一步分为MIKCC和MIKC*,二者之间的差别是MIKC*缺少I区域和K盒[5]。MIKCC依据进化关系又可进一步分为AP1、SVP、AP3、SOC1、AG、SEP、AGL12、AGL17和FLC等亚组,同时MIKCC也是植物中研究最为广泛和深入的类型[3,6]。近年来随着MADS-box研究的不断深入,花形成的分子调控机理已经从最初的ABC模型发展为ABCDE模型。根据ABCDE模型的阐述,A类(APETALA(AP1))和E类(SEPALLATA(SEP1、SEP2、SEP3和SEP4))基因调控萼片的形成;A类、B类(PISTILATA(PI)和AP3)和E类基因调控花瓣的形成;B类、C类(AGAMOUS(AG))和E类基因调控雄蕊的形成;C类和E类基因调控心皮的形成;D类(SEEDSTICK/ AGAMOUS-LINE11(STK/AGL11))和E类基因调控胚珠的形成[4, 6-8]。此外,MADS-box还参与调控果实的发育[9-10]、成熟和开裂[11-13],调控光合作用和营养代谢[14]以及参与激素信号转导等途径[3,15]。【本研究切入点】根据前人研究结果[16-17],已经鉴定得到79个桃MADS-box。本研究以‘鲁星’桃为试材,去除GenBank数据库中已登录的基因,克隆不同亚组的桃MADS-box(PpMADS)。【拟解决的关键问题】克隆PpMADS,详细分析PpMADS的MADS-box保守结构域、进化关系、染色体分布、基因结构和保守元件,并研究其在不同组织以及花和果实发育过程中的表达情况,为桃MADS-box的深入研究提供参考。1 材料与方法
试验于2014—2015年在山东省果树生物技术育种重点实验室和山东农业大学园艺学院作物生物学国家重点实验室进行。1.1 植物材料
供试材料为2014年山东省果树研究所天平湖试验示范基地(山东泰安)3年生‘鲁星’桃(Prunus persica var. nectarina ‘Luxing’,证书编号:鲁农审2014047号,亲本:Yukon King/曙光),一年生的枝条(茎)(4月5日)、新叶(4月5日)、7个不同发育阶段的整花(花蕾期:1阶段,4月2日取样;露瓣期:2阶段,4月2日取样;初开期:3阶段(小),4月2日取样;初开期:4—5阶段(中—大),4月9日取样;盛花期:6阶段,4月9日取样;衰败期:7阶段,4月15日取样)[18-19]、雄蕊(4月5日)、萼片(4月5日)、子房(4月5日)、花瓣(4月5日)和5个不同发育阶段的果实(花后30、50、60、70和80 d)(以4月5日开始计算)。上述植物材料均从3棵及以上树上采集发育阶段和状态较一致的样品。于液氮中速冻,-80℃保存,以便提取RNA进行表达分析。1.2 基因克隆与序列分析
采用改良热硼酸法提取混和样品(包括1.1中的混合组织材料:茎、叶、7阶段整花和80 d果实)RNA,用反转录试剂盒PrimeScriptTM1st Strand cDNA Synthesis Kit(TaRaKa公司)合成cDNA。根据前人研究结果[20-21],鉴定得到79个PpMADS,通过序列比对去除GenBank数据库中存在的15条PpMADS,选择克隆分属不同亚组的10个PpMADS。设计特异性引物(表1)进行PCR扩增,PCR反应条件为:94℃预变性5 min;94℃ 45 s,56—62℃ 45 s,72℃ 90 s,35个循环;72℃延伸10 min。回收PCR产物并连接到pMD18-T载体上,构建重组质粒转化E. coli DH5α感受态细胞,筛选阳性克隆,送至北京六合华大基因科技股份有限公司测序。Table 1
表1
表1基因克隆引物序列
Table 1Gene cloning primers and sequences
| 用途 Use | 引物名称 Primer name | 上游引物序列 Forward primer sequence (5′- 3′) | Tm值 Tm value | 下游引物序列 Reverse primer sequence (5′- 3′) | Tm值 Tm value |
|---|---|---|---|---|---|
| ORF扩增 Complete ORF amplification | PpMADS11 | ATGGCGAGGGGTAAGATCCAGA | 61.0 | TTAAGGCACACTGCATACAAAT | 52.5 |
| PpMADS12 | ATGGGGAGAGGCAAGATTGTGAT | 59.1 | TCAATGACAAGTCAATCCAATCA | 52.4 | |
| PpMADS19 | ATGGGTCGTGTTAAGCTGGAGAT | 59.6 | TTAATGTCCATTAAGCTGAGGGA | 53.2 | |
| PpMADS20 | ATGGTGAAGAAGACTAGGGGTCG | 58.9 | TCAGAGAAAGCCTCCACCGAGGT | 62.7 | |
| PpMADS21 | ATGGTGATGAAGATCAAGAAACC | 53.0 | TCAAAAAAGACCATGGCCAGCAT | 57.8 | |
| PpMADS22 | ATGATGAAGAAGAAACCAGGCCA | 57.2 | TTACCATTCATCCGAAGAGTGGT | 56.3 | |
| PpMADS28 | ATGGCTAGGACAAAGGTGAAGCT | 59.7 | TCACTCCTCACCACCTCCATTAT | 58.9 | |
| PpMADS29 | ATGGGCAAAAATAAAATTAAGCA | 53.8 | TTAGCCTTGATCCCCCATCTCAT | 57.6 | |
| PpMADS30 | ATGACTAGAAAGAAGGTCAGCCT | 54.3 | TCAAGGAAAAAAAGGATTGTTAG | 52.1 | |
| PpMADS31 | ATGACAAGAAAGCAAATTGACCT | 53.9 | TCATGGTTTTTCCAGTGAGGTCA | 56.7 |
新窗口打开
在桃基因组数据库(https://www.rosaceae.org/gb/ gbrowse/prunus_persica/)中,通过序列比对查找PpMADS的启动子序列,由于桃基因组数据库测序质量较好,可直接进行启动序列分析。在线软件PlantCARE(http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)进行启动子序列分析;DNAMAN 6.0.3.99软件分析基因的开放阅读框(ORF)和编码氨基酸序列;在线软件WebLogo 3(http://weblogo. threeplusone. com/)分析PpMADS蛋白的MADS-box结构域;MEGA6.0软件构建PpMADS和其他物种MADS-box的系统进化树;LocTree3(https://rostlab. org/services/ loctree3/)和SoftBerry ProtComp 9.0(http://linux1. softberry.com/berry. phtml)进行亚细胞定位预测[22-23]。
1.3 PpMADS的表达分析
使用RNeasy Plant Mini Kit(QIAGEN公司)提取组织RNA,用反转录试剂盒PrimeScriptTM 1st Strand cDNA Synthesis Kit(TaRaKa公司)合成cDNA。采用PPERACTIN-2作为半定量PCR(RT-PCR)内参基因[21]。RT-PCR扩增体系为25 μL:1 μL cDNA;2.5 μL 10×Taq缓冲液;2.5 mmol dNTP 2 μL;上、下游引物(10 μmol·L-1)各1 μL;Taq DNA聚合酶(购自全式金公司)0.25 μL;剩余用ddH2O补齐。循环参数为:94℃ 5 min;94℃ 25 s;58℃ 25 s;72℃ 30 s;目的基因为36个循环,内参基因为28个循环。每一个基因的RT-PCR反应均重复3次。采用TEF2作为实时荧光定量PCR(qRT-PCR)的内参基因[21]。参照谷彦冰等[21]的方法,采用三步法进行qRT-PCR分析。所用仪器为BIO-RAD IQ5(USA),所有qRT-PCR反应都设3次重复。PCR反应体系为:SYBR Green Master I 10 μL,上、下游引物(5 μmol·L-1)各1 μL,模板1 μL,加去离子水至20 μL。PCR反应程序为:95℃预变性3 min;95℃ 10 s,58℃ 30 s,72℃ 15 s,40个循环;最后退火至55℃,每隔7 s上升0.5℃,至95℃,共81个循环。试验结果用2-ΔΔCT法对数据进行定量分析。引物设计均避开MADS-box保守域,所用引物具体序列详见表2。
Table 2
表2
表2RT-PCR和qRT-PCR引物序列
Table 2RT-PCR and qRT-PCR primers and sequences
| 用途 Use | 引物名称 Primer name | 上游引物序列 Forward primer sequence (5′- 3′) | Tm值 Tm value | 下游引物序列 Reverse primer sequence (5′- 3′) | Tm值 Tm value | 产物大小 Product size |
|---|---|---|---|---|---|---|
| RT-PCR | RT-PpMADS11 | CTCACTGTTCTCTGCGATGC | 59.0 | CCTTTGCCTGATCTGCTTCC | 58.9 | 222 |
| RT-PpMADS12 | ATGGGGAGAGGCAAGATTGT | 58.7 | TAATCGTAAAGCCTGCCGGT | 58.7 | 170 | |
| RT-PpMADS19 | GCCGAGGAGCAGATAAGGAT | 59.1 | ACCTGCAACCTCATTCTCGA | 59.1 | 207 | |
| RT-PpMADS20 | GGGCACTGAGACATTGTTGG | 58.8 | TCCCCTCTTTGTTGGCCTTC | 59.1 | 206 | |
| RT-PpMADS21 | CTAGCCAAGGTCGCCAAAAG | 59.2 | GTTGGCCGGAGAGAAGACTA | 59.4 | 161 | |
| RT-PpMADS22 | TGACCGCTTTCTTTGCACTC | 59.1 | ACCAACACCGTCTCGATGAT | 59.1 | 221 | |
| RT-PpMADS28 | GGACAAAGGTGAAGCTTGCA | 58.9 | TCTTGGACCGTTCTGCTTCT | 58.9 | 237 | |
| RT-PpMADS29 | GGCAAGTCTGTTCAGAAAGCT | 58.7 | TCCTCGTCTTTGTTCGGGTA | 59.6 | 166 | |
| RT-PpMADS30 | GGAGAAGGAGATGTCGAGGG | 58.9 | TGGGACTGGGACTGGATTTC | 59.0 | 178 | |
| RT-PpMADS31 | CTGATGGAGCCCTACGAACA | 59.1 | GCTCCCGTAAATGGCTTGAA | 58.5 | 182 | |
| PPERACTIN-2 | GTGGGGATGGGACAGAAAGATG | 61.3 | GAGGTCAAGCCGGAGGATGG | 62.0 | 402 | |
| qRT-PCR | qRT-PpMADS11 | TGATGAACTGCGTGGTGTTG | 59.1 | TCTGTTGCACTCCGTAGCTT | 59.3 | 123 |
| qRT-PpMADS12 | CCGGCAGGCTTTACGATTAT | 58.1 | AGCTGCAGTTCAATGACAAGT | 58.4 | 137 | |
| qRT-PpMADS19 | ATGCCAGTGATGGAGAGCTT | 59.0 | ATTTCTGGCATGTTCGAGCC | 58.9 | 133 | |
| qRT-PpMADS22 | AGAGCTTGACGAGATGAGCA | 58.8 | CCTCCTTCATTGCAGCCTTC | 58.9 | 119 | |
| qRT-PpMADS29 | CAGGCTGAATGGACAATTGGA | 58.5 | TGCTAACAAACTTCCGAGACG | 58.6 | 127 | |
| TEF2 | GGTGTGACGATGAAGAGTGATG | 59.6 | TGAAGGAGAGGGAAGGTGAAAG | 60.0 | 129 |
新窗口打开
2 结果
2.1 PpMADS克隆
经测序克隆得到分属不同亚组的10个PpMADS,基因命名、ORF、蛋白大小、分子量、等电点、所属类型和GenBank登录号详见表3。对10个PpMADS蛋白氨基酸序列进行同源性比对分析,结果显示均含有MADS-box结构域(图1)。Table 3
表3
表3PpMADS基因信息
Table 3The information of PpMADS genes
| 基因名称 Gene name | 定位名称 Locus name | 开放阅读框 ORF | 蛋白大小 Protein size (aa) | 分子量 MW (KDa) | 等电点 PI | 类型 Group | GenBank登录号 GenBank accession No. |
|---|---|---|---|---|---|---|---|
| PpMADS11 | ppa026083m | 522 | 173 | 20.275 | 10.051 | AP3 | KU559577 |
| PpMADS12 | ppa024736m | 279 | 92 | 10.331 | 10.037 | AGL17 | KU559578 |
| PpMADS19 | ppa007816m | 1065 | 354 | 39.781 | 5.451 | MIKC* | KU559585 |
| PpMADS20 | ppa026089m | 828 | 275 | 29.988 | 6.358 | Mα | KU559586 |
| PpMADS21 | ppa017246m | 723 | 240 | 26.891 | 9.115 | Mα | KU559587 |
| PpMADS22 | ppa023670m | 600 | 199 | 22.502 | 9.067 | Mα | KU559588 |
| PpMADS28 | ppa014932m | 636 | 211 | 23.872 | 7.322 | Mγ | KU559594 |
| PpMADS29 | ppa014627m | 534 | 177 | 20.38 | 7.945 | Mγ | KU559595 |
| PpMADS30 | ppa019749m | 750 | 249 | 28.499 | 9.616 | Mγ | KU559596 |
| PpMADS31 | ppa017207m | 480 | 159 | 18.255 | 9.162 | Mγ | KU559597 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1PpMADS蛋白氨基酸序列同源性比对和MADS-box结构域序列标签分析
-->Fig. 1Homology comparison of the deduced amino acid sequence alignment of PpMADS proteins and analysis of the MADS-box domain logo
-->
2.2 PpMADS进化分析
利用MEGA6.0软件对本研究克隆的10个PpMADS蛋白、GenBank数据库中有记载的15个PpMADS蛋白[17]、拟南芥MADS-box蛋白(AGL)[22]和水稻MADS-box蛋白(OsMADS)[23]进行MADS-box结构域聚类分析(图2)。结果表明,Type II MADS-box蛋白被分为MIKCC和MIKC*两组,其中MIKCC可继续分为9个亚组;Type I MADS-box蛋白被分为Mα、Mβ、Mγ和Mδ四组。PpMADS11属于AP3亚组;PpMADS12属于AGL17亚组;PpMADS19属于MIKC*组;PpMADS20、PpMADS21和PpMADS22属于Mα组;PpMADS28、PpMADS29、PpMADS30和 PpMADS31同属于Mγ组(图2)。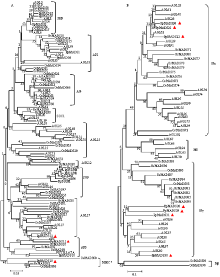 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2桃、拟南芥和水稻Type II(A)和Type I(B)MADS-box蛋白的MADS-box结构域进化关系和亚组分类分析
-->Fig. 2Phylogenetic relationships and subgroup designations of MADS-box domain from Type II (A) and Type I (B) MADS-box proteins in peach, Arabidopsis and rice
-->
2.3 PpMADS亚细胞定位预测
通过在线预测软件SoftBerry ProtComp 9.0对PpMADS蛋白进行亚细胞定位预测。结果显示(表4),PpMADS11、PpMADS12、PpMADS19、PpMADS20、PpMADS21、PpMADS22、PpMADS28、PpMADS、PpMADS30和PpMADS31定位在细胞核的相应预测数值均为最高(共10分:数值越高表示可信度越大)。进一步利用在线预测软件LOCTREE3对PpMADS蛋白进行亚细胞定位,结果显示PpMADS蛋白均定位在细胞核。由此判断,PpMADS蛋白可能定位于细胞核中。Table 4
表4
表4PpMADS亚细胞定位预测
Table 4The information in predicting PpMADSs subcellular localization
| 定位 Location | PpMADS11 | PpMADS12 | PpMADS19 | PpMADS20 | PpMADS21 | PpMADS22 | PpMADS28 | PpMADS29 | PpMADS30 | PpMADS31 |
|---|---|---|---|---|---|---|---|---|---|---|
| 细胞核 Nuclear | 9.36 | 9.38 | 8.09 | 9.09 | 8.09 | 7.77 | 8.87 | 8.83 | 9.12 | 8.98 |
| 质膜 Plasma membrane | 0.04 | 0 | 0.24 | 0.52 | 0.34 | 1.5 | 0.57 | 0.7 | 0.54 | 0.74 |
| 胞外 Extracellular | 0.5 | 0 | 1.1 | 0.17 | 0.96 | 0 | 0.06 | 0 | 0.1 | 0 |
| 细胞质 Cytoplasmic | 0.01 | 0 | 0.48 | 0.02 | 0.48 | 0.3 | 0 | 0 | 0.08 | 0.01 |
| 线粒体 Mitochondrial | 0 | 0 | 0 | 0 | 0 | 0 | 0.09 | 0.01 | 0 | 0 |
| 细胞质内层 Endoplasm Retic. | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 过氧化物酶体 Peroxisomal | 0.04 | 0.03 | 0.06 | 0.07 | 0.07 | 0.12 | 0.05 | 0.08 | 0.07 | 0.05 |
| 高尔基体 Golgi | 0.04 | 0.4 | 0.03 | 0 | 0.05 | 0 | 0 | 0 | 0 | 0 |
| 叶绿体 Chloroplast | 0 | 0 | 0 | 0 | 0 | 0 | 0.08 | 0.07 | 0 | 0 |
| 液泡 Vacuolar | 0 | 0.19 | 0 | 0.14 | 0 | 0.31 | 0.27 | 0.31 | 0.1 | 0.21 |
新窗口打开
2.4 PpMADS启动子序列分析
通过桃基因组数据库(https://www.rosaceae.org/ gb/gbrowse/prunus_persica/)下载基因组定位信息,获得10个PpMADS的启动子序列(1 500 bp)。对启动子序列上顺式作用元件进行分析,发现10个PpMADS启动子上含有多个光响应元件、防御及逆境响应元件、干旱诱导的MYB结合位点、热激响应元件、低温响应元件、真菌效应子响应元件、伤害响应元件、厌氧响应元件、GA响应元件、Auxin响应元件、MeJA响应元件、ABA响应元件、SA响应元件和乙烯响应元件。其中,PpMADS11启动子含有2个真菌效应子响应元件和2个厌氧响应元件;PpMADS12启动子含有7个厌氧响应元件和4个MeJA响应元件;PpMADS19启动子含有2个热激响应元件、2个SA响应元件和2个防御及逆境响应元件;PpMADS20启动子含有4个厌氧响应元件;PpMADS21启动子含有3个干旱诱导的MYB结合位点;PpMADS22启动子含有4个干旱诱导的MYB结合位点;PpMADS28启动子含有4个GA响应元件和4个防御及逆境响应元件;PpMADS29 和PpMADS30启动子分别含有5个厌氧响应元件;PpMADS31启动子含有2个SA响应元件和2个GA响应元件(图3)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3PpMADSs启动子序列顺式作用元件分析
-->Fig. 3Cis-acting elements potentially associated with PpMADSs promoter
-->
2.5 PpMADS的表达分析
利用半定量RT-PCR技术对10个PpMADS进行不同组织、花发育和果实发育阶段的表达分析。结果表明,PpMADS12、Mα组和Mγ组成员在茎、叶、萼片、子房、雄蕊和花瓣中均有表达;PpMADS11在茎、叶、萼片、子房、雄蕊和花瓣中均表达;而PpMADS19在萼片、雄蕊和花瓣中表达(图4)。在花不同发育阶段中,PpMADS12、Mα组和Mγ组成员持续高表达;PpMADS11在蕾期(1阶段)、露瓣期(2阶段)和初开期(3阶段)表达量逐渐上升,在初开期(4—5阶段)和盛开期(6阶段)表达量逐渐下降,在衰败期(7阶段)表达更加微弱;PpMADS19的表达模式与PpMADS11相似,不同的是在蕾期(1阶段)无表达(图5)。在果实不同发育阶段中,PpMADS12、PpMADS19、PpMADS20、PpMADS21、PpMADS28和PpMADS31均无表达;PpMADS11则持续在不同果实发育阶段表达;PpMADS22、PpMADS29和PpMADS30在30 d和50 d过程中表达逐渐上调,60 d表达下调,而后表达持续上升(图6)。为了验证上述基因表达的准确性,选择分属不同亚组的5个PpMADS在不同组织、花不同发育阶段和果实不同发育阶段进行qRT-PCR表达分析,结果与半定量RT-PCR结果一致(图7)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4PpMADS在桃不同组织中的半定量表达分析
-->Fig. 4RT-PCR expression analysis of PpMADS genes in different tissues of peach
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5PpMADS在花不同发育阶段中的半定量表达分析
-->Fig. 5RT-PCR expression analysis of PpMADS genes at different stages of flower development in peach
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6PpMADS在果实不同发育阶段中的半定量表达分析
-->Fig. 6RT-PCR expression analysis of PpMADS genes at different stages of fruit development in peach
-->
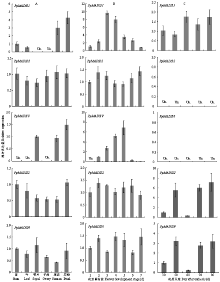 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7PpMADS在不同组织(A)、花不同发育阶段(B)和果实不同发育阶段(C)的实时荧光定量表达分析
-->Fig. 7qRT-PCR expression analysis of PpMADS genes in different tissues (A), at different stages of flower development (B) and at different stages of fruit development (C) in peach
-->
3 讨论
作为转录因子大家族,MADS-box广泛参与到植物多种生理和生殖过程的调控。2013年,VERDE等[24]公布了桃基因组草图,使得在基因组范围内鉴定和分析桃MADS-box家族成为可能。2015年,WELLS等[16]鉴定了桃基因组中存在79个MADS- box。已有研究表明,MADS-box受到非生物胁迫后,表现出不同的表达模式[23,25-27]。水稻多个MADS-box在盐、干旱、冷和ABA处理后出现不同响应[23,25];二穗短柄草幼苗在面对盐、干旱和冷胁迫处理后,MADS-box的相对表达呈现不同程度地上调和下 降[26];大白菜幼苗在面对赤霉素、水杨酸、脱落酸、冷和热胁迫处理后,多个MADS-box的相对表达也呈现不同程度地上调和下降[27]。本文中,顺式作用元件分析表明10个PpMADS的启动子含有多个与激素和逆境胁迫相关的作用元件,因此,暗示着这10个PpMADS的转录可能受到激素和逆境胁迫的诱导。
研究表明,B类MADS-box包括AP3和PI在花瓣和雄蕊中起到特异性的调控作用[4,6-8]。拟南芥AP3或者PI突变都会导致第二轮的花瓣转换为萼片,而第三轮雄蕊转换为心皮结构[28]。此外,在多个物种的突变体中也发现这种相似表型,例如,双子叶植物金鱼草、矮牵牛花、非洲菊、罂粟、欧洲耧斗菜、番茄、本氏烟以及单子叶植物玉米和水稻[29]。这些结果表明,AP3和PI的调控功能非常保守。尽管AP3在花瓣和雄蕊中起到特异性调控作用,但其在花组织中的表达却是多种多样的。例如,水稻AP3同源基因OsMADS16在花瓣和雄蕊中有表达[30];拟南芥AP3在花瓣、雄蕊和子房中有表达[31];玉米AP3同源基因SILKY1在萼片、花瓣、雄蕊和子房中均有表达[32];非洲菊AP3同源基因GDEF1在萼片、花瓣、雄蕊和子房以及非花器官中均有表达[31];蒺藜苜蓿AP3同源基因NMH7只在非花器官叶中表达[33];苹果AP3亚组成员MdMADS23在非花器官茎、叶中有表达,花中无表达,而且在果实发育过程中持续表达[6]。本研究克隆得到的AP3同源基因PpMADS11,在非花器官茎、叶和雄蕊以及花瓣中均表达,并且在花发育的过程中,表达模式是先升高后降低;在果实发育过程中,持续高表达。在拟南芥[22]、二穗短柄草[26]和黄瓜[34]中,AGL17亚组成员在根中特异表达;而在苹果[6]、水稻[23]和葡萄[35]中,AGL17亚组成员还在其他组织中表达。本研究中,比较不同的是,PpMADS12在茎、叶、萼片、花瓣、雄蕊和子房以及花发育的各个阶段中均有表达,在果实发育过程中则没有表达。前人研究表明,拟南芥包含6个MIKC*类型的MADS-box,分别是:AGAMOUS-LIKE30(AGL30)、AGL65、AGL66、AGL67、AGL94和AGL104。除了AGL67,其他成员均在花粉发育过程中特异表达,并且它们之间的功能高度冗余,其单突变体并没有缺陷表型,而双突变体agl66 agl104-2则表现为:花粉发育能力下降,延迟萌发,花粉管生长畸形[36]。水稻包含3个MIKC*类型成员,分别是OsMADS62、OsMADS63和OsMADS68,而且与拟南芥MIKC*类型的MADS-box具有相似功能,不同的是其单突变体就能表现为花粉成熟和萌发受到严重破坏[37]。在本文中,检测到PpMADS19在萼片、雄蕊和花瓣中有表达,在花发育的过程中,表达模式是先升高后降低。以上结果表明,AP3亚组、AGL17亚组和MIKC*类型成员在各个物种中调控花发育的相关功能高度保守,但在各个物种的进化过程中又赋予其新的功能。
相对于MIKC类型来说,Type I的相关研究较少。最近研究表明,拟南芥Type I成员在雌配子体的决定以及胚和胚乳的发育过程中具有重要作用[38-39]。本研究中,Mα组和Mγ组成员在茎、叶、萼片、花瓣、雄蕊和子房以及花发育的各个阶段中均有表达,在果实发育过程中则是先上升后下降再上升;相似的结果也出现在梅花Mα组和Mγ组成员中[40];拟南芥[22]、水稻[23]、黄瓜[34]和大豆[41]Mα组和Mγ组成员在叶、花和果实也均有表达;而苹果[6]、二穗短柄草[26]和大白菜[27]Mα组和Mγ组成员在多种组织中特异性表达。以上结果表明,不同物种Mα组和Mγ组成员在植物营养生长和生殖发育过程中扮演相似或者独特的功能。
此外,WELLS等[16]通过RNA-seq数据分析了桃(Prunus persica ‘Lovell’)中根、成熟叶、幼叶和果实等组织的表达模式。对于同一基因在不同桃品种相同组织的表达模式,有些类似,有些则差别较大。例如,PpMADS11、PpMADS19、PpMADS29和PpMADS30的RNA-seq表达模式与本研究中RT-PCR的表达模式相似,而PpMADS12、PpMADS20、PpMADS21、PpMADS22、PpMADS28和PpMADS31的表达模式则不同。这可能是由于品种特性、组织取样的时间和空间、栽培条件以及外界环境等多种因素造成的。因此,桃不同品种间MADS-box的表达模式及其相关生物学功能有待进一步探究。
4 结论
克隆获得了‘鲁星’桃10个PpMADS,均含有MADS-box结构域,分属5个亚组,可能都定位于细胞核中,含有多个顺式作用元件;PpMADS11在茎、叶、萼片、子房、雄蕊、花瓣、花发育和果实发育中均表达;PpMADS12在茎、叶、萼片、子房、雄蕊、花瓣和花发育中表达;PpMADS19在萼片、雄蕊、花瓣和花发育中(蕾期除外)表达;Mα组和Mγ组所有成员在茎、叶、萼片、子房、雄蕊、花瓣和花发育中均有表达,部分成员在果实发育中表达,暗示其在营养生长以及花和果实发育过程中可能具有重要的调控作用。(责任编辑 赵伶俐)
The authors have declared that no competing interests exist.
