0 引言
【研究意义】纤维素、半纤维素是地球上最丰富和重要的可再生资源。纤维素是由D-葡萄糖通过β-1,4糖苷键连接而成的一种线性葡聚糖,而半纤维素主要包含木聚糖、葡甘露聚糖和半乳葡甘露聚糖等,它们共同组成了细胞壁的主要骨架结构。深入研究纤维植物中纤维素的生物合成过程,挖掘控制纤维发育的重要基因,进而通过遗传操作改良植物的纤维品质和产量具有重要的理论和实践意义。【前人研究进展】纤维素、半纤维素作为细胞壁的重要组成部分,对其生物合成的研究具有重要意义。纤维素合酶超基因家族包括纤维素合酶(cellulose synthase,CesA)和纤维素合酶类蛋白(cellulose_synthase_like protein,Csl蛋白),是纤维形成的关键酶,分别参与纤维素和半纤维的合成[1-3]。目前已从40多种植物中克隆了1 400多个CesA的相关序列[4-5]。在拟南芥(Arabidopsis thaliana)及毛果杨(Populus trichocarpa)中分别鉴定出10个和18个CesA。在四倍体陆地棉(Gossypium barbadense)中共鉴定出37个CesA,其中,19个来自At亚基因组,18个来自Dt亚基因组,而在二倍体雷蒙德氏棉(Gossypium raimondii)中鉴定出15个CesA[6]。在拟南芥中,AtCesA1、AtCesA3、AtCesA6组成纤维素合酶复合体,参与初生细胞壁合成,AtCesA4、AtCesA7、AtCesA8则参与次生细胞壁合成[7-13]。AtCesA2、AtCesA5、AtCesA9被认为是AtCesA6的同源蛋白,这些蛋白彼此之间存在功能冗余。在棉花中,GhCesA3、GhCesA5、GhCesA6、GhCesA9、GhCesA10参与初生细胞壁合成,GhCesA1、GhCesA2、GhCesA7、GhCesA8参与次生细胞壁合成[14]。杨树木质部细胞膜上至少存在2种纤维素合酶复合体,复合体Ⅰ由PtCesA4、PtCesA7、PtCesA8、PtCesA17、PtCesA18组成,参与细胞次生壁的合成;复合体Ⅱ由PtCesA3、PtCesA10、PtCesA11、PtCesA13、PtCesA15、PtCesA16组成,参与细胞初生壁和次生壁的合成[15]。除CesA蛋白之外,植物体内还有Csl蛋白,该类酶则在高尔基体体腔中介导半纤维素的合成[16-17]。依据序列结构特征的不同,Csl蛋白被分为9个亚族,分别为CslA—CslH及CslJ,其中CslF、CslH和CslJ只存在于单子叶植物中[18-19]。CslA催化(1,4)-b-D-甘露聚糖的合成,CslC参与催化木葡聚糖骨架的形成,CslD则在多糖(木聚糖和半乳醛聚糖)的合成中起作用。但目前,CslB、CslE和CslG的生物学功能尚不清楚。亚麻(Linum usitatissimum L.)是一种重要的韧皮纤维作物。其韧皮纤维细胞的发育主要经历2个阶段:细胞伸长和次生细胞壁加厚,这两个时期互不重叠,分别决定了韧皮纤维的长度和强度,次生细胞壁加厚与韧皮纤维的品质密切相关。韧皮纤维细胞发育是个复杂的过程,受很多基因表达调控影响,如CesA、Susy、Kor等,其中,CesA是催化纤维素合成的关键酶之一,一直是植物纤维素合成研究领域中的一个热点。但亚麻纤维发育的分子生物学基础相对滞后。随着亚麻基因组测序计划的完成,生物信息学、生物化学、分子生物学和遗传学等研究方法的不断完善,亚麻纤维发育的分子生物学基础才得以不断推进。笔者前期研究发现,现蕾期喷施适当浓度的植物激素油菜素内酯(brassinosteroids,BR)及芸苔素吡咯(brassinazole,Brz)能够提高亚麻的纤维产量和耐盐性。为深入研究BR、Brz调控亚麻纤维发育和提高耐盐性的分子机制,利用Solexa技术,比较分析了BR、NaCl、Brz处理对亚麻现蕾期茎部组织基因转录表达谱的影响。结果发现,差异基因中存在大量细胞壁合成相关基因,这些基因涉及纤维素合成、木质素合成、细胞壁结构、细胞壁多糖合成代谢等。【本研究切入点】尽管对纤维素合酶超基因家族的研究有了较大进展,但大多局限于拟南芥、棉花、杨树等模式植物中,亚麻中大多CesA、Csl成员尚未得到分离和鉴定。随着亚麻基因组序列的公布,有必要对亚麻纤维素合酶超家族基因进行全基因组挖掘和全面、系统分析。【拟解决的关键问题】本研究采用生物信息学方法,从基因组水平对亚麻纤维素合酶超家族基因的理化特性、定位、基因结构、进化等进行阐释,并根据Solexa表达谱数据对CesA/Csl的表达特性进行分析,为亚麻纤维发育的机理研究奠定基础。1 材料与方法
1.1 植物材料
材料为高纤亚麻品种Diana,在不同时期播种于黑龙江省农业科学院民主园区试验田,以期在同一时期获得不同发育阶段的亚麻植株。选择苗期植株喷施清水;同时,选择现蕾期植株,分别喷施清水(对照CK)、BR(0.2 mg·L-1)和Brz(1 mg·L-1)及根灌NaCl(300 mmol·L-1)等4种处理。处理12 h后分别选择生长一致的植株,取其茎中部1/3,每个处理分别取3株,3次生物学重复。取材后将样品置于液氮中迅速冷冻,-80℃保存,用于RNA的提取及Solexa数字表达谱分析。1.2 亚麻CesA/Csl的鉴定
根据拟南芥AtCesA及AtCsl蛋白氨基酸序列,在Phytozome数据库中进行BLASTP比对,搜索亚麻同源序列。利用NCBI CDD(http://www.ncbi.nlm.nih. gov/Structure/cdd/)在线工具对获得的基因是否含有Cellulose-synt保守结构域进行确认。亚麻CesA/Csl基因家族蛋白序列下载自phytozome数据库(http:// phytozome.jgi.doe.gov/),拟南芥CesA/Csl蛋白序列下载自TAIR(http://www.arabiodpsis.org)。1.3 亚麻CesA/Csl的特性分析
通过phytozome数据库查询CesA/Csl的Scaffold位点信息,利用ExPASy Proteomics Server(http://web. expasy.org/protparam/)预测蛋白氨基酸序列的基本信息,包括蛋白质的长度、分子量、等电点。利用TMHMM Server V2.0(http://www.cbs.dtu.dk/services/ TMHMM/)进行蛋白跨膜螺旋预测。通过在线软件WoLF PSORT(http://wolfpsort.or)进行亚细胞定位分析。1.4 亚麻CesA/Csl系统发育树的构建
使用Clustal X软件对鉴定出的45个亚麻CesA/Csl蛋白与分别来自于杨树、棉花和拟南芥的CesA/Csl蛋白进行氨基酸序列比对,比对结果通过MEGA5软件生成进化树,采用邻接法(Neighbor-Joining,NJ)构建进化树,矫正参数Bootstrap重复1 000次。1.5 亚麻CesA/Csl基因家族的基因结构分析及蛋白基序分析
利用Gene Structure Display Server(http://gsds1.cbi. pku.edu.cn/)在线分析CesA/Csl基因家族的基因结构;通过MEME网站(http://meme-suite.org/tools/meme)在线分析CesA/Csl基因家族蛋白的保守基序,参数设置:基序宽度为6×50,鉴定最大基序数量为20。1.6 亚麻CesA/Csl表达分析
对高通量测序表达谱数据进行背景扣除、均一化,提取亚麻45个CesA/Csl在苗期、现蕾期以及BR、NaCl及Brz不同处理的表达量(RPKM值),利用Excel软件绘制柱形图。运用SPSS 16.0软件进行数据分析,一维方差分析(One-way ANOVA)进行组间比较,多重比较使用最小差数比较法(LSD),P<0.05代表差异显著。2 结果
2.1 亚麻CesA/Csl的鉴定
从亚麻全基因组中共筛选到51个拟南芥AtCesA/ Csl的同源蛋白,利用NCBI CDD(http://www.ncbi.nlm. nih.gov/Structure/cdd/)在线工具对其进一步鉴定,获得45个在CDD软件上预测含有CesA保守结构域的蛋白,其中,35个蛋白含有CESA_CelA_like(cd06421)结构域,10个含有CESA_CaSu_A2(cd06437)结构域。因此,该45个蛋白被确认为亚麻CesA/Csl家族蛋白。2.2 亚麻CesA/Csl的基因组定位及特性分析
CesA/Csl蛋白理化特性分析表明,氨基酸长度为409—1 167,分子量为47 401.1—130 578.3,等电点分布在5.43—9.08。根据WoLF PSORT预测显示(表1),35个CesA/Csl蛋白定位于质膜上,是亚麻CesA/Csl蛋白定位最多的细胞部位。4个CesA/Csl蛋白(Lus10023056、Lus10032415、Lus10025046、Lus10022982)属于叶绿体蛋白,1个CesA/Csl蛋白(Lus10030035)位于细胞核内,还有5个CesA/Csl蛋白(Lus10003525、Lus10013851、Lus10026568、Lus10020539、Lus10039475)暂不能确定其亚细胞位置。大多数CesA/Csl蛋白都含有跨膜结构域,跨膜区是β-1,4-葡萄糖苷链穿过质膜进入细胞壁的重要通道,在亚麻中,除Lus10003525蛋白可能是截短的不完整蛋白序列,未预测到跨膜结构域外,其余CesA/Csl蛋白均含有跨膜结构域,数目为2—8。Table 1
表1
表1亚麻CesA/Csl基因家族信息
Table 1The information of CesA/Csl gene family in flax
| 基因登录号 Gene accession | 保守结构域 Conserved domains | 大小 Size (aa) | 分子量 Molecular weight (D) | 等电点 Isoelectric point | 亚细胞定位 Sublocation | 跨膜螺旋数 Transmembrane helices number | 基因定位 Scaffold location |
|---|---|---|---|---|---|---|---|
| Lus10003525 | CESA_CelA_like(cd06421) | 687 | 77073.7 | 5.43 | — | 0 | 144 |
| Lus10006161 | CESA_CelA_like(cd06421) | 1074 | 121442.0 | 6.79 | 质膜Plas | 6 | 779 |
| Lus10041063 | CESA_CelA_like(cd06421) | 1096 | 123703.2 | 6.79 | 质膜Plas | 6 | 280 |
| Lus10002940 | CESA_CelA_like(cd06421) | 906 | 102055.5 | 7.26 | 质膜Plas | 5 | 1060 |
| Lus10002939 | CESA_CelA_like(cd06421) | 1097 | 123554.2 | 7.06 | 质膜Plas | 6 | 1060 |
| Lus10003526 | CESA_CelA_like(cd06421) | 1097 | 123633.1 | 6.78 | 质膜Plas | 6 | 144 |
| Lus10022449 | CESA_CelA_like(cd06421) | 1035 | 116929.4 | 6.09 | 质膜Plas | 6 | 38 |
| Lus10007538 | CESA_CelA_like(cd06421) | 1092 | 122311.1 | 7.46 | 质膜Plas | 8 | 259 |
| Lus10012198 | CESA_CelA_like(cd06421) | 1094 | 122623.5 | 7.03 | 质膜Plas | 8 | 1186 |
| Lus10039607 | CESA_CelA_like(cd06421) | 1069 | 120160.1 | 8.01 | 质膜Plas | 6 | 15 |
| Lus10018902 | CESA_CelA_like(cd06421) | 1079 | 121660.4 | 6.26 | 质膜Plas | 6 | 103 |
| Lus10028597 | CESA_CelA_like(cd06421) | 1079 | 121623.3 | 6.28 | 质膜Plas | 6 | 346 |
| Lus10007296 | CESA_CelA_like(cd06421) | 988 | 111446.2 | 6.39 | 质膜Plas | 8 | 859 |
| 基因登录号 Gene accession | 保守结构域 Conserved domains | 大小 Size (aa) | 分子量 Molecular weight (D) | 等电点 Isoelectric point | 亚细胞定位 Sublocation | 跨膜螺旋数 Transmembrane helices number | 基因定位 Scaffold location |
| Lus10029245 | CESA_CelA_like(cd06421) | 988 | 111276.1 | 6.73 | 质膜Plas | 8 | 360 |
| Lus10013851 | CESA_CelA_like(cd06421) | 1128 | 126664.0 | 6.43 | — | 8 | 618 |
| Lus10026568 | CESA_CelA_like(cd06421) | 1135 | 127121.5 | 6.62 | — | 8 | 617 |
| Lus10022982 | CESA_CelA_like(cd06421) | 1127 | 125504.9 | 5.88 | 叶绿体Chlo | 6 | 355 |
| Lus10000755 | CESA_CelA_like(cd06421) | 991 | 111050.4 | 8.56 | 质膜Plas | 8 | 1775 |
| Lus10011736 | CESA_CelA_like(cd06421) | 949 | 106646.4 | 8.92 | 质膜Plas | 8 | 568 |
| Lus10009248 | CESA_CelA_like(cd06421) | 1095 | 122791.1 | 6.70 | 质膜Plas | 6 | 267 |
| Lus10038008 | CESA_CelA_like(cd06421) | 1064 | 119477.7 | 6.36 | 质膜Plas | 3 | 475 |
| Lus10026610 | CESA_CelA_like(cd06421) | 1056 | 117718.6 | 6.58 | 质膜Plas | 4 | 617 |
| Lus10026609 | CESA_CelA_like(cd06421) | 1031 | 115654.6 | 7.90 | 质膜Plas | 6 | 617 |
| Lus10030455 | CESA_CelA_like(cd06421) | 953 | 106594.3 | 7.26 | 质膜Plas | 6 | 917 |
| Lus10030453 | CESA_CelA_like(cd06421) | 869 | 98148.4 | 7.79 | 质膜Plas | 8 | 917 |
| Lus10010024 | CESA_CelA_like(cd06421) | 1167 | 130578.3 | 8.35 | 质膜Plas | 6 | 1630 |
| Lus10025046 | CESA_CelA_like(cd06421) | 1073 | 120501.7 | 8.85 | 叶绿体Chlo | 4 | 294 |
| Lus10002134 | CESA_CelA_like(cd06421) | 1060 | 118537.2 | 5.58 | 质膜Plas | 6 | 1106 |
| Lus10012119 | CESA_CelA_like(cd06421) | 1065 | 118230.7 | 6.31 | 质膜Plas | 5 | 1186 |
| Lus10023056 | CESA_CelA_like(cd06421) | 753 | 84500.7 | 7.26 | 叶绿体Chlo | 8 | 325 |
| Lus10003196 | CESA_CelA_like(cd06421) | 769 | 86162.9 | 6.04 | 质膜Plas | 7 | 1056 |
| Lus10023057 | CESA_CelA_like(cd06421) | 743 | 82891.8 | 7.18 | 质膜Plas | 8 | 325 |
| Lus10032415 | CESA_CelA_like(cd06421) | 744 | 83157.7 | 6.40 | 叶绿体Chlo | 8 | 291 |
| Lus10016625 | CESA_CelA_like(cd06421) | 762 | 86303.4 | 5.59 | 质膜Plas | 8 | 915 |
| Lus10030035 | CESA_CelA_like(cd06421) | 838 | 93042.2 | 6.81 | 细胞核Nucl | 4 | 416 |
| Lus10009387 | CESA_CaSu_A2(cd06437) | 541 | 62521.2 | 9.05 | 质膜Plas | 5 | 137 |
| Lus10020539 | CESA_CaSu_A2(cd06437) | 409 | 47401.1 | 8.30 | — | 3 | 77 |
| Lus10025886 | CESA_CaSu_A2(cd06437) | 693 | 79117.1 | 9.08 | 质膜Plas | 3 | 605 |
| Lus10038217 | CESA_CaSu_A2(cd06437) | 495 | 56683.3 | 8.53 | 质膜Plas | 2 | 28 |
| Lus10007715 | CESA_CaSu_A2(cd06437) | 640 | 73270.5 | 8.68 | 质膜Plas | 6 | 993 |
| Lus10018651 | CESA_CaSu_A2(cd06437) | 615 | 70871.7 | 8.62 | 质膜Plas | 5 | 461 |
| Lus10020120 | CESA_CaSu_A2(cd06437) | 636 | 72771.3 | 9.06 | 质膜Plas | 5 | 454 |
| Lus10026923 | CESA_CaSu_A2(cd06437) | 705 | 80606.6 | 9.07 | 质膜Plas | 6 | 651 |
| Lus10039440 | CESA_CaSu_A2(cd06437) | 676 | 77143.6 | 8.77 | 质膜Plas | 6 | 33 |
| Lus10039475 | CESA_CaSu_A2(cd06437) | 676 | 77097.5 | 8.77 | — | 6 | 33 |
| Lus10001619 | 无None | 1069 | 119166.7 | 6.50 | — | 6 | 1025 |
| Lus10008225 | 无None | 508 | 58057.2 | 5.52 | — | 0 | 157 |
| Lus10008226 | 无None | 479 | 53298.3 | 8.92 | 质膜Plas | 6 | 157 |
| Lus10012198 | 无None | 1094 | 122623.5 | 7.03 | 质膜Plas | 8 | 1186 |
| Lus10032416 | 无None | 156 | 17570.6 | 6.69 | — | 2 | 291 |
| Lus10038009 | 无None | 100 | 10938.0 | 10.42 | — | 2 | 475 |
新窗口打开
尽管亚麻全基因测序已完成,但并未公布测序亚麻品种的染色体物理图谱,所以目前亚麻CesA/ Csl家族基因仅能定位于scaffolds上,尚无法定位到染色体上。亚麻CesA/Csl家族基因在scaffolds上是分散分布的,无明显的成簇现象。在Scaffold 617中含有3个CesA/Csl,在Scaffold 33、Scaffold144、Scaffold 325、Scaffold 917、Scaffold 1060、Scaffold 1186中均各含有2个CesA/Csl,其他Scaffold上仅含有1个CesA/Csl。
2.3 亚麻与杨树、棉花及拟南芥CesA/Csl的进化关系
对已鉴定含有纤维素合酶保守结构域的45个亚麻纤维素合酶CesA/Csl蛋白与杨树、棉花、拟南芥的CesA/Csl蛋白构建系统进化树(图1),主要聚为7个明显的分支。根据氨基酸序列比对和进化关系远近,将CesA/Csl蛋白分为CesA、Csl两类,而Csl蛋白被细分为6组,分别命名为CslA、CslB、CslC、CslD、CslE与CslG。不同组间的CesA/Csl之间直向相似度(ortholog)远大于平行(paralog)进化相似度,表明纤维素合酶在进化中产生分化的时期很早,在这4个物种分化之前,不同的纤维素合酶就已进化产生了不同的类型和组型。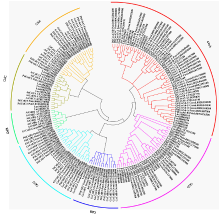 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1亚麻与杨树、棉花、拟南芥CesA/Csl基因家族的进化关系
-->Fig. 1Phylogenetic relationship of CesA/Csl gene family among Flax, Populus, Gossypium and Arabidopsis
-->
在基因进化过程中,突变积累的数量和时间成正比,从进化树可以看出,亚麻CesA/Csl和毛果杨的遗传距离相对较近,与棉花次之,而与拟南芥的遗传距离相对较远,CesA/Csl与物种间的生物进化关系趋于一致。进一步分析发现,CesA在这4个物种中数目分布基本一致,但Csl在不同物种中数量分布具有较大差异,如亚麻CslD组基因数目较多,CslB、CslE组基因数目较少;拟南芥CslA组基因数目较多,CslE组基因数目较少;棉花CslE、CslG组基因数目较多,CslB、CslC组基因数目较少;而杨树各组基因数目分布较为均匀。推测在这4个物种的祖先中可能只存在少量的CesA/Csl,在物种分离之后,CesA/Csl通过基因重复的方式进行了扩增,但不同物种在不同组基因中扩张程度并不一致。
进化树上,CesA组与CslD组位于相邻的2个分支,进化关系最近。亚麻CesA组有13个基因成员,其中11个基因与杨树、棉花、拟南芥的CesA1、CesA2、CesA3、CesA5、CesA6、CesA9、CesA10聚在一起,在功能上可能参与初生细胞壁的合成;2个基因与CesA8聚在一起,在功能上可能参与次生细胞壁的合成,但没有发现CesA4和CesA7的亚麻同源基因。亚麻CslD组有15个基因,为基因成员最多的一组。RICHMOND等[16]推测CslD为纤维素合酶超家族中最为古老的一个,可能在进化时间上早于CesA。4个基因与AtCslG1—AtCslG3聚为一组(CslG组),CslB与CslE组各有1个基因成员,2个基因与CslA聚为一组(CslA组),CslC组有8个基因成员。CslA、CslC组基因在进化树上位于最远的分支,与CesA组基因遗传距离最远,分化最大。
2.4 亚麻CesA/Csl家族的基因结构分析
拟南芥中已克隆了10个CesA,通过比较分析发现,拟南芥的10个CesA最大的差异在于某些区域中内含子的有无,内含子的位置是确定纤维素合成酶功能的关键因素之一[20]。基因结构分析显示,亚麻CesA/Csl的长度为2.1—6.8 kb,外显子数量为2—14。外显子数量在不同组间有较大变化(图2),CesA组成员外显子数目较多,在11—14,CslD组外显子数目较少(外显子数目为2—5)。不同组中,各组内部成员之间外显子数目相近,其中CesA组、CslC组及CslD组内部成员的基因结构保守性较高。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2亚麻CesA/Csl基因家族进化树和基因结构
-->Fig. 2The phylogenetic tree and gene structures of CesA/Csl gene family in Flax
-->
2.5 亚麻CesA/Csl家族的蛋白基序分析
利用MEME在线工具对亚麻CesA/Csl蛋白序列进行了保守基序分析,预测出20个Motif(图3)。在同一组内,大多数成员的Motif组成基本相同,不同组间Motif组成有一定的差异。在进化上,CesA组与CslD组亲缘关系最近,Motif组成相对保守,但也存在分化,Motif13、Motif15特异分布于CesA组中,Motif14、Motif 19则特异分布于CslD组中。Motif13是CesA蛋白N末端特有的锌指结构域,是维持纤维素合成酶复合体稳定结构的重要功能区。亚麻CesA和Csl之间存在的最大区别是Csl蛋白(除5个CslD蛋白)缺少该锌指结构域(Motif 13)。与CslD相比,CslE、CslB、CslG、CslA、CslC组蛋白序列相对较短,保守基序数目显著减少。在进化上,CslE、B、G三组蛋白亲缘关系较近,Motif组成相对保守;CslA、CslC组蛋白则分支较远,序列保守程度低,保守基序数目较少。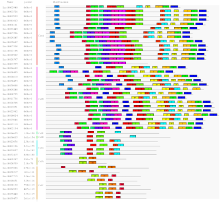 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3亚麻CesA/Csl基因家族蛋白的保守基序
-->Fig. 3The conserved Motif of CesA/Csl gene family in Flax
-->
CesA组及CslD、CslE、CslB、CslG组成员均具有CESA_CelA_like(cd06421)保守结构域,与具有CESA_CaSu_A2(cd06437)保守结构域的CslA、CslC组成员在Motif组成上具有明显的差异。Motif 12、Motif 4、Motif 1、Motif 3、Motif 2特异分布于CesA及CslD、CslE、CslB、CslG组蛋白中,这些Motif主要分布于酶的中央结构域中,酶的中央结构域通常包含2个保守区(A区和B区),Motif 12、Motif 4、Motif 1、Motif 3位于A区中,该区域可结合纤维素合成的底物;Motif 2位于B区中,含有保守序列QxxRW, 与纤维素合酶的催化活性有关。Motif 20、Motif 18特异分布于CslA、C组蛋白中,Motif 20与Motif 3、Motif 18与Motif 2在功能上具有相似性,但经过长期的进化,除保守结构域D、QxxRW外,序列已发生明显改变
2.6 亚麻CesA/Csl的表达分析
亚麻不同发育时期、不同处理的RNA-Seq数据进行预处理和统计分析后,获得45个CesA/Csl基因家族成员对应表达量的RPKM值(图4),其中6个基因RPKM值都小于1,说明这些基因在5个样品中的表达量极低或不表达。通过分析发现亚麻CesA组基因中大多数基因在亚麻的苗期和现蕾期中均有较强表达,并且存在明显的共表达,表明这些基因是植株生长发育所必需的基因。Lus10039607、Lus10002939、Lus10007296、Lus10029245则表现为苗期低丰度表达,现蕾期高丰度表达,差异达到显著水平(P<0.05)。其中,Lus10007296、Lus10029245是AtCesA8的同源基因。笔者通过荧光定量PCR分析不同发育阶段亚麻Lus10007296、Lus10029245基因的表达特性,发现这2个基因表达模式相似,在快速生长期,基因的表达量达到最高;进入花期和绿熟期时基因的表达量再次上调。快速生长期是亚麻纤维细胞启动次生细胞壁加厚阶段,至花期、绿熟期亚麻次生细胞壁进一步加厚,纤维素沉积,纤维逐渐成熟,由此推测Lus10007296、Lus10029245基因与亚麻次生细胞壁加厚密切相关。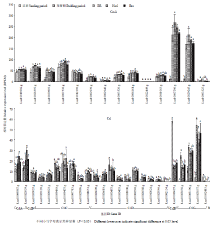 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4亚麻CesA/Csl的相对表达量
-->Fig. 4Relative expression of CesA/Csl gene family in Flax
-->
亚麻Csl蛋白被分为CslA、CslB、CslC、CslD、CslE、CslG共6个亚组。CslA催化(1,4)-b-D-甘露聚糖的合成,亚麻中2个CslA基因在苗期、现蕾期表现出相似的表达丰度,但它们均可被NaCl诱导上调表达,差异达到显著水平(P<0.05)。亚麻中有1个CslB(Lus10030035),该基因在不同发育时期、不同处理下均低丰度表达。CslC参与催化木葡聚糖骨架的形成,CslD则在多糖(木聚糖和半乳醛聚糖)的合成中起作用,亚麻中有8个CslC和15个CslD,其中Lus10039440、Lus10039475、Lus10007715、Lus10030453、Lus10009248的RPKM值为10—20,其他基因在不同发育时期、不同处理下均低丰度表达或无表达(RPKM<10)。其中,Lus10039475、Lus10007715 2个基因可被NaCl诱导上调表达,被Brz抑制表达,但差异未达到显著水平(P>0.05)。亚麻CslE组只有1个基因(Lus10016625),该基因在苗期高丰度表达,现蕾期低丰度表达,且可被Brz诱导上调表达。亚麻CslG组有4个基因,该组基因在不同发育时期均表现出明显的不同,其中3个基因Lus10023056、Lus10003196、Lus10023057 现蕾期与苗期相比较上调表达,1个基因(Lus10032415)现蕾期与苗期相比较下调表达。Lus10023056可被BR、NaCl、Brz诱导下调表达;Lus10003196可被NaCl诱导下调表达,差异均达到显著水平(P<0.05)。与其他Csl组比较,CslE、CslG组基因的表达量相对较高,而且在苗期和现蕾期,CslE、CslG组基因的表达丰度发生显著变化,由此推测CslE、CslG组基因可能在纤维发育中起重要作用。
3 讨论
本研究通过对亚麻全基因组生物信息学分析,共鉴定出2类,7组共45个亚麻CesA/Csl 蛋白超家族成员,该基因家族与拟南芥、杨树同源性较高,是一类植物进化中保守的家族。在亚麻中发现的CesA编码的蛋白质与杨树、拟南芥具有相似的结构,在N末端都有1个环形锌指结构或LIM结构域(Motif 13),此种结构域具有保守序列CxxC(半胱氨酸-xx-半胱氨酸)。KUREK等[21]通过酵母双杂交发现,GhCesA1和GhCesA2通过此锌指结构可以形成为同源或异源的二聚体。除草剂(CGA325’615)能够通过阻止各蛋白锌指结构域之间的相互作用从而破坏纤维素合成酶复合体的稳定结构[21]。由此推测,纤维素合成酶基因的锌指结构域与蛋白间的相互作用有关,是维持纤维素合成酶复合体稳定结构的重要功能区。CesA和Csl之间存在的最大区别是大多数Csl蛋白(除5个CslD蛋白)缺少锌指结构[22]。纤维素的合成必须在CesA蛋白复合体的作用下完成,相比而言Csl蛋白中大多数都没有锌指结构,因此,目前****认为类纤维素的合成很可能不需要Csl蛋白复合体结构,单一的Csl蛋白也具有催化类纤维素主链合成的活性[23]。RICHMOND和SOMERVILLE根据内含子与外显子排列组合的进化趋势,曾推测CslD基因家族为纤维素合酶超家族中最为古老的一个,可能在进化时间上早于CesA[16]。同时,本研究发现15个CslD蛋白中有5个成员与CesA相同,具有锌指结构,而且在进化上CslD蛋白与CesA蛋白亲缘关系最近,由此推测在进化上很可能由CslD蛋白分支出CesA和Csl类其他蛋白。大多数亚麻CesA蛋白含有8个跨膜结构域,N-端有2个预测的跨膜区在第250个氨基酸残基附近,C-末端有6个预测的跨膜区,跨膜区是β-1,4-葡萄糖苷链穿过质膜进入细胞壁的重要通道[24]。锌指结构域和N端跨膜结构域之间有一个蛋白序列的高变区Ⅰ(HVRI),富含酸性氨基酸,功能尚不清楚。在第2和第3跨膜结构域之间是酶的中央结构域,其间有一个蛋白序列高变区Ⅱ(HVRII)。通过高变区可以区分不同的CesA蛋白,在功能上,高变区可能参与调控不同发育时期纤维素的合成[25]。高变区Ⅱ两边各有一个保守基序A区和B区,A区含有保守基序Motif 12、Motif 4、Motif 8、Motif 15、Motif 1、Motif 17和Motif 3,该区域可结合纤维素合成的底物,B区除含有一个保守的D-天冬氨酸残基外(Motif 9),还有保守序列QxxRW(Motif 2),该保守区与纤维素合成酶的催化活性有关[25]。除了蛋白序列高变区Ⅱ外,不同CesA蛋白之间中央结构域高度保守。CesA、CslD组蛋白相比保守基序数目极多,序列更加保守,而CslA、CslB、CslC、CslE和CslG组蛋白相对序列较短,保守基序数目极少,序列保守程度低,由此产生的遗传多样性对于植物更好地调控自身纤维素合成具有重要意义。
不同的CesA/Csl基因家族成员在时空上具有不同的表达模式,预示不同的CesA/Csl基因家族成员存在着功能特异性[26-28]。SUZUKI等[17]对杨树中的48个CesA/Csl的表达特性进行分析,PtCesA通常在叶片组织中表达量较低,PtCesA13和PtCesA 18在发育的木质部中高丰度表达,PtCesA4、PtCesA 5、PtCesA 7、PtCesA 8和PtCesA 17在木质部中特异表达,但表达量相对较低。与拟南芥类似,PtCsl的表达量明显低于PtCesA,30个PtCsl中只有21个基因被检测出表达量。PtCslA1、PtCslA2、PtCslA5和PtCslD6在发育的木质部中高丰度表达,PtCslC1和PtCslC4在茎尖中组织特异性表达,其他15个PtCsl的表达不表现出组织特异性。LI等[29] 研究发现,在陆地棉和海岛棉中CesA8在纤维素大量积累时期发挥重要作用。CesA2在非纤维组织中大量表达,CesA1则类似持家基因,在不同组织中组成型表达。同时,他们发现果胶质的积累与木葡聚糖的减少促进细胞壁的转化。本文通过对RNA-seq分析发现,亚麻大部分Csl的表达丰度都低于CesA,如CslB、CslC及CslD中的部分成员在不同发育时期表达丰度都极低,这与杨树、拟南芥的表达特性相同,推测基因表达量可能与纤维素、半纤维素在次生木质部中所占组分相关。苗期为亚麻纤维细胞分化,初生细胞壁形成阶段;现蕾期亚麻纤维细胞数快速增加,次生细胞壁迅速加厚。亚麻CesA/Csl中,部分CesA及全部CslE、CslG组基因在苗期、现蕾期表达丰度表现出明显的不同,暗示这些基因可能在参与纤维发育及初生、次生细胞壁合成中担任不同的角色。目前,Csl蛋白的生物学功能尚不清楚,有待于深入研究。
对亚麻CesA/Csl上游启动子区域进行预测,发现多个逆境响应的顺式调控元件。RNA-seq结果表明,CesA/Csl家族基因中个别基因可被NaCl诱导上调或下调表达,推测这些基因参与逆境胁迫信号通路。BR是植物中的甾醇类生长促进激素,参与调控纤维发育。SUN等[30]研究发现,BR受体基因BRI1抑制表达的转基因棉花,棉纤维的伸长受影响较小,但次生细胞壁的发育受到明显抑制;相反,过量表达BRI1的转基因棉花纤维素沉积显著增加。笔者前期研究发现,在亚麻现蕾期喷施适当浓度的外源BR及BR特异性合成抑制剂Brz能够影响亚麻纤维发育,提高亚麻的纤维产量。本研究中,BR、Brz处理与对照相比,部分CesA/Csl被BR、Brz诱导上调或下调表达,这些基因可能是植物激素BR信号调控通路的下游基因。
植物纤维素生物合成机制的研究对纤维作物改良、木材定向培育以及纺织业、造纸等化工业都具有积极的科学意义,因此,通过了解植物纤维素的生物合成机制,进而改善植物纤维的品质与产量就显得十分必要。纤维素生物合成的调节涉及CesA的表达调控、CesA蛋白修饰、CesA蛋白在高尔基体中的分泌、胞质中纤维素合酶复合体的装配、纤维素的沉积等多个方面[31],但由于纤维素合酶的生化研究难度大,该领域研究一直停滞不前。近年来,随着基因组学研究的进展及植物细胞壁突变体的发现使植物CesA的功能研究成为可能[32],但是总体来说人们对Csl蛋白的功能尚不十分清楚,尤其CesA/Csl的表达调控及CesA/Csl的运输、复合体的装配等问题更有待于深入研究。
4 结论
鉴定出45个亚麻CesA/Csl家族基因成员,分属于2类、7组,分散分布于scaffolds上,基因结构和蛋白基序具有组间多样性和组内保守性。不同的基因在不同发育阶段具有一定的时空特异性,CesA/Csl中部分基因响应激素BR、Brz及NaCl胁迫。(责任编辑 李莉)
The authors have declared that no competing interests exist.
