 ,1,2, 宗亚奇1,2, 徐维华2, 韩蕾1, 孙浈育1,2, 陈朝晖3, 陈松利3, 张凯1, 程杰山1, 唐美玲
,1,2, 宗亚奇1,2, 徐维华2, 韩蕾1, 孙浈育1,2, 陈朝晖3, 陈松利3, 张凯1, 程杰山1, 唐美玲 ,1,2,3, 张洪霞1, 宋志忠
,1,2,3, 张洪霞1, 宋志忠 ,1
,1Identification, Cloning, and Expression Characteristics Analysis of Fe-S Cluster Assembly Genes in Grape
ZHANG Lu ,1,2, ZONG YaQi1,2, XU WeiHua2, HAN Lei1, SUN ZhenYu1,2, CHEN ZhaoHui3, CHEN SongLi3, ZHANG Kai1, CHENG JieShan1, TANG MeiLing
,1,2, ZONG YaQi1,2, XU WeiHua2, HAN Lei1, SUN ZhenYu1,2, CHEN ZhaoHui3, CHEN SongLi3, ZHANG Kai1, CHENG JieShan1, TANG MeiLing ,1,2,3, ZHANG HongXia1, SONG ZhiZhong
,1,2,3, ZHANG HongXia1, SONG ZhiZhong ,1
,1通讯作者:
责任编辑: 赵伶俐
收稿日期:2021-02-8接受日期:2021-05-5
| 基金资助: |
Received:2021-02-8Accepted:2021-05-5
作者简介 About authors
张璐,Tel:0535-6352051;E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1474KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张璐, 宗亚奇, 徐维华, 韩蕾, 孙浈育, 陈朝晖, 陈松利, 张凯, 程杰山, 唐美玲, 张洪霞, 宋志忠. 葡萄Fe-S簇装配基因的鉴定、克隆和表达特征分析. 中国农业科学, 2021, 54(23): 5068-5082 doi:10.3864/j.issn.0578-1752.2021.23.012
ZHANG Lu, ZONG YaQi, XU WeiHua, HAN Lei, SUN ZhenYu, CHEN ZhaoHui, CHEN SongLi, ZHANG Kai, CHENG JieShan, TANG MeiLing, ZHANG HongXia, SONG ZhiZhong.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】铁是植物正常生命活动所必需的微量矿质元素[1,2],是细胞色素、铁硫(Fe-S)蛋白的组成成分[3],与植物生长发育、花的形成和果实品质与产量密切相关,缺铁严重影响植物生长,降低果实产量和品质[4,5,6,7]。Fe-S簇是Fe-S蛋白的活性部位,虽然其组成元素和结构都较为简单,但是Fe-S簇的组装是需多种组装蛋白参与的有序进行的催化反应,是活细胞中一个高度复杂和协调的过程[8,9,10,11]。由此可见,Fe-S簇装配机制是植物铁素营养和铁代谢的核心环节,在植物的生命过程中具有至关重要的作用。鉴定和克隆果树Fe-S簇装配基因为研究果树Fe-S簇装配机制提供参考,并为解析果树铁素营养和代谢奠定理论基础。【前人研究进展】在植物中,Fe-S蛋白在呼吸、光合、硫和氮同化、氨基酸和嘌呤代谢、植物激素和辅酶合成,DNA修复和翻译等过程中发挥重要作用[8,9,10],大多数代谢途径和细胞过程发生在亚细胞部位,并依赖于Fe-S蛋白[3,12]。研究表明Fe-S蛋白存在于质体、线粒体、细胞质和细胞核中,对许多生理和代谢过程必不可少,例如,硝酸还原酶(nitrite reductase,NIR)在叶绿体中氮的同化过程至关重要,乌头酸酶(aconitase,ACO)和琥珀酸脱氢酶(succinate dehydrogenase,SDH)是参与线粒体糖代谢柠檬酸循环的关键酶[9]。特别地,Fe-S簇是Fe-S蛋白的辅因子,在光合作用、呼吸和DNA修复中起着不可或缺的作用[11,13-15]。植物中,一个高度保守的Fe-S簇装配过程包括在组装支架上形成Fe-S簇,并转移到目标载体蛋白上,涉及硫供体(NFS)、铁供体、支架(SUFB、SUFC、SUFD、NFU等)以及转运蛋白(ISA、GRX、HSCA)等40多个编码基因[9,12],其中,模式植物拟南芥中Fe-S簇装配机制的研究最为深入和透彻,目前已经确定了3种Fe-S簇装配机制,即质体中存在SUF(sulfur mobilization)装配机制[16],线粒体中采用ISC(iron-sulfur cluster)装配机制[16],这两种机制均以独立的方式运行,而细胞质中的Fe-S簇组装是新出现的一种依赖于线粒体的CIA(cytosolic iron-sulfur cluster assembly)装配机制[16]。在这3种机制中,Fe-S簇的装配过程均可分为两个阶段:第一阶段,即S和Fe结合在支架蛋白上,第二阶段,将Fe-S簇转移到靶蛋白。其中,第二阶段可能涉及具有特殊功能的不同的载体蛋白[9,10,11,12,13,14,15,16]。【本研究切入点】近年来,国内外对植物Fe-S簇装配分子机制的研究主要集中在一年生植物,包括拟南芥[12]、水稻[17]和大豆[18]等,果树学中研究较少,仅在桃[19,20]中有报道,葡萄中Fe-S簇装配机制及其分子基础依然未知。【拟解决的关键问题】本研究以‘马瑟兰’葡萄为材料,克隆葡萄Fe-S簇装配机制相关基因并鉴定其生物信息学特征,明确其在葡萄不同组织部位的表达特征,为研究葡萄Fe-S簇装配和果树铁素营养与代谢的分子机制提供基因资源和理论基础。1 材料与方法
1.1 试验时间、地点
试验于2019年6月至2020年12月在鲁东大学农林工程研究院和“十三五”山东省高等学校重点实验室——作物高产抗逆分子模块育种实验室进行。1.2 试验材料与处理
供试材料为烟台地区主栽酿酒葡萄品种‘马瑟兰’,由山东省烟台市农业科学研究院果树所提供,种植在招远市大户庄园农林专业合作社试验基地,树体健壮,南北行向,常规田间管理。分别于特定日期采集‘马瑟兰’的果实,包括幼果期(2019年6月30日)、硬核期(7月15日)、膨大期(8月3日)、转色期(8月17日)和成熟期(9月15日),以及葡萄幼叶(6月30日)和老叶(9月15日),样品采集后立即液氮冷冻并保存于-80℃低温冰箱中备用。胁迫试验供试材料为山东省烟台市农业科学研究院果树所提供的‘马瑟兰’组培脱毒幼苗。自2019年11月起,进行‘马瑟兰’组培脱毒苗的接种培养,所用培养基为1/2 MS固体培养基。组培室环境温度为25℃,湿度70%,光照3 000 lx。接种后的幼苗于一个月左右生根,继而长叶。培养近两个月即可将培养瓶放置于普通室内环境中,逐渐开盖进行3 d炼苗处理,早晚喷洒去离子水。幼苗生长状况良好的情况下,置于蛭石珍珠岩基质中,用正常MS培养液作为对照处理,用不含铁离子的MS培养液进行缺铁胁迫处理,早晚浇营养液,处理96 h后,分别采集处理幼苗的根部、茎部和叶部材料,立即液氮冷冻并保存于-80℃冰箱中备用。
1.3 葡萄Fe-S簇装配基因克隆
以拟南芥中43个Fe-S簇装配基因的氨基酸序列为参考序列,在Phytozome Grape Genome Database (Table 1
表1
表1葡萄Fe-S簇装配基因CDS扩增所需引物
Table 1
| 基因Gene | 上游引物 Forward primer (5′-3′) | 下游引物 Reverse primer (5′-3′) | 产物长度 Product length (bp) |
|---|---|---|---|
| ADX1 | ATGTTCGCTTCTAGACTTTCA | TTAGTGTGGTTTTGGAATAAA | 597 |
| ADXR | ATGGCGTTTTGTCGTGCGAAG | TCATTCCATGGTAACTTTCAG | 1461 |
| ATM3 | ATGGCGGGCGTTGCTTCTTGC | TCACGCCTCCAATTTAATGGT | 2181 |
| CIA1 | ATGGATTTCTCGGAAGAGGGC | TCAGGTTATAGAAGCCAGCTC | 1035 |
| CIA2 | ATGTTTTTCCCTGAGGTGGAC | TTAGAGTTCATTGGAGTAGAG | 360 |
| CIA3 | ATGTTCTTAAATCATATTAGA | TCAACCATATGATGGAGCCAG | 363 |
| DRE2 | ATGTTGCAAAATAGCACATTG | TTAAATGTCAGCCACAAGAA | 819 |
| ERV1 | ATGTCTGAAACCCCATTTCAT | TTAATGTGATCTCTCACCAAA | 597 |
| FH | ATGGCTTCTGCTTCTTCTTCC | TTAGGAAAGGCTGATGGGTGT | 594 |
| GRXS14 | ATGCTTATGCTTATATCAATA | TCAGGAGCACATCGCCTTCTC | 552 |
| GRXS15 | ATGGCAAGGTCACTATCTAAC | TCATTCGGACTTTTCCTGGGG | 513 |
| GRXS16 | ATGGCAACAATCAGTCTATCT | CTACTTCTTAAATAAACCAAC | 894 |
| HCF101 | ATGAGGCTAACAAACACTCTT | TCATGCCTGTGCAGGAGTTAG | 1671 |
| HSCA1 | ATGGCTGCCTCCGTTTTGCTC | CTACTTCTTCACCTCTTCATA | 1842 |
| HSCA2 | ATGGCCACTTCGGTGCTTCTT | CTACTTCTTGGCCTGCTCATA | 1716 |
| HSCA3 | ATGTCGGAGTGCCCAGCGATC | TCACATCACCATCCGGTCCCTT | 1503 |
| HSCA4 | ATGTACGGTGCGTGGGGTCTA | CTACAGCTCATCATGGGAGTC | 1947 |
| HSCA5 | ATGGACGAGGTGGGTGAGGA | TCACTTGCTCTCGGTGAAGTC | 1677 |
| HSCA6 | ATGGAAAGCTCGTGGAGAAGA | TCAAAGTTCATCATGGGAATC | 1821 |
| HSCA7 | ATGGCGGTGAAGAACAGGGCG | TCAGAACTGGAAGAAGCCCCA | 1731 |
| HSCB | ATGTGGAAGAAGAATCTATGG | TCAGAGTTTCTTCATGATTTC | 804 |
| IBA57 | ATGGCCTCGCTTCTCAAGTCC | CTAAGCGGACGCAGTATCTTG | 1113 |
| INDL | ATGGAGTCACCGACCCAGGGA | TCACAGTGAAATCTCTGGCCG | 1026 |
| ISA1 | ATGGCATCTTCATTTCTCGCG | CTACTTAGCAGCTCCAGGATT | 402 |
| ISA2 | ATGCCCCAGCCCTCCACTTCG | TCACATTTCAGCAGCAAAGGA | 453 |
| ISA3 | ATGTCAAGATCGGTGCTTCGG | CTATTCTTTCACCATGAAAGA | 465 |
| ISD11 | ATGGCATCAACACCTTCGAAA | TTATTCCAACTGCCTTCTGGC | 219 |
| ISU1 | ATGAAGCTTCAAATCAAGGTC | CTACTCAGTCTTTGCTCGCTT | 273 |
| MMS19 | ATGGCACAACTGAGTCAATTG | TCAGAAATGAAGACTTCTGGA | 3441 |
| NAR1 | ATGTCGGAGAAGTTCTCGGCA | TCACCAGTTGTGTAATTGAGA | 1437 |
| NBP35-1 | ATGGAGAACGGCGACAGCAAT | TCAAGTGGTAGACAGTGCCTG | 957 |
| NBP35-2 | ATGGAGTCACCGACCCAGGGA | TCACAGTGAAATCTCTGGCCG | 1026 |
| NFS1 | ATGGCTTCCAGGCTTCATTCT | TTAGGTTATTTGTTTTTGTAG | 3267 |
| NFS2 | ATGGCAAGTGTTCTGAAATT | CTACTTGAAAGAAGAGAAGA | 1392 |
| NFU1 | ATGGCGTCTCTCACAGCAACA | CTAGTCACTGAAGACAACATT | 642 |
| NFU2 | ATGCAAGGCGTGGTGGTGGCG | CTATAGAAGCTGAACTGCTGC | 684 |
| NFU3 | ATGGGCTTCACAGCATTCACA | TTATCCATCTATCAACTGGAC | 714 |
| NFU4 | ATGTTCATCCAAACTCAATCC | TTAATCCATCTGGCCAGATAG | 609 |
| SUFA | ATGCCCCAGCCCTCCACTTCG | TCACATTTCAGCAGCAAAGGA | 453 |
| SUFB | ATGGCTTCTCTACTGGCCAAC | CTAACCGACTGATCCTTCAAGC | 1536 |
| SUFC | ATGGCTCGATTCTGCTGCGCC | TTACAGTTGGGCCTCAGAAAT | 966 |
| SUFD | ATGGCTGCTACATTGCTTTCA | TCATGAGGAACCTTTAAGTGT | 1443 |
| SUFE1 | ATGTCTCTGTCCAATCTCCAA | TCAACATAGATTCCCAAGTTG | 1224 |
| SUFE2 | ATGGACTCTGCAACTTTGGG | TCATATCCTGAAAGGACTCTG | 855 |
| SUFE3 | ATGGGGTGTACGGCGCAGGTG | TCAACTTATTGAAGTTGATC | 1806 |
| TAH18 | ATGAATGGGAGGGAGAAGCAG | TCAAGACCATGCTTCCACATG | 1899 |
新窗口打开|下载CSV
1.4 葡萄Fe-S簇装配基因的生物信息学分析
参考LIANG等[17]和SONG等[20]的报道,在Phytozome葡萄基因组数据库中获得Fe-S簇装配基因的CDS编码区序列及基因组DNA序列和编码氨基酸序列,然后通过Gene Structure Display(1.5 总RNA提取与实时荧光定量PCR分析
采集田间葡萄树体不同发育时期的果实和叶片,通过RNAprep Pure多糖多酚植物总RNA提取试剂盒(DP441)(TIANGEN,北京)提取葡萄样品的总RNA,并利用Fast Quant cDNA第一链合成试剂盒(去基因组)100rxn(TIANGEN,北京)合成第一链cDNA作为模板,采用Perfect Start Green qPCR Super Mix(TransGen,北京)进行实时荧光定量PCR。利用NCBI/PrimerBLAST在线服务器,设计葡萄Fe-S簇装配基因的特异性表达引物(表2)。以葡萄内参基因Ubiquitin(GenBank No. MH114011)[21,22],通过BIO-RAD实时荧光定量PCR仪检测葡萄Fe-S簇装配基因在不同组织部位的表达特征。反应体系参照商品说明书的描述,反应程序为95℃ 30 s;95℃ 5 s,60℃ 34 s(40个循环);72℃ 10 s。每个样品进行3次生物学重复,不同样品在实时荧光定量PCR仪获得相应的Ct值,经内参基因Ubiquitin均一化处理后,采用2-ΔCT法计算基因的相对表达量[21,22]。根据DENG等[23]描述,通过Log2计算法分析缺铁胁迫前后表达倍数,通过HemI软件制作表达差异变化的热图。分别以对照植物根、茎或叶的表达值设定为1,若缺铁胁迫条件下的表达值<1,表示基因表达水平被下调;表达值>1,则表示基因表达水平被上调。Table 2
表2
表2本研究所用特异性表达引物
Table 2
| 基因 Gene | 上游引物 Forward primer (5′-3′) | 下游引物 Reverse primer (5′-3′) | 产物长度 Product length (bp) |
|---|---|---|---|
| ADX1 | AACCACAATCGGGGACCAAG | GTGGAACAGGCAAGTGAACC | 250 |
| ADXR | TGGGCATCCAGACTGCAAAA | TGGTCCACGTCTTCCTACCA | 211 |
| ATM3 | CACCGACACCCTAGTTGGAC | TAGCCCCAACCAAGAAACCC | 232 |
| CIA1 | GCAACGTTCTCCCTCCACTT | GACCCGGATGCATTCCAAGA | 231 |
| CIA2 | TGTTTTTCCCTGAGGTGGACAT | GCCAATTACTGTTGCCATGCT | 164 |
| CIA3 | AAACTTCTGCGCAGTTTGCC | GGAGCCAGACATTCATCAACC | 167 |
| DRE2 | ATAGCACATTGGCCCTCACA | TGTCCCACCAGGCTTCAAAA | 248 |
| ERV1 | TCGGCAGCAGAAGAAGGATG | CCCCACCTTGCATCAACTCT | 222 |
| FH | GGCCGCCATTGATTACAGTTC | GCTCACAGGAGAGGACAACC | 235 |
| GRXS14 | GGAACCAAGGAGTTCCCACA | CGCCTTCTCCAACTGTTCCT | 243 |
| GRXS15 | TGAAAGGGGTGCCTGATGTG | TTGTGGAAATGTGGGCCAGT | 158 |
| GRXS16 | AAACTGAGCCGATTCCCCTG | CTCTGGCACCGATTTCCTGT | 167 |
| HCF101 | GAGTGGGGAGAGCTGGACTA | AACAACAGCAACACAGGGGA | 192 |
| HSCA1 | CTCGCTTCCCCTTCTCTCTC | CCACTGATGGTGTAGTCCGAG | 235 |
| HSCA2 | CCATTCAGCTCAAAGCCTGC | GGGAGCTCTGAGTTTAGTTCCA | 243 |
| HSCA3 | AGAGAGGTTTGCAAAGGAGGA | GTCCCTTTCCTGATGACTTTCC | 244 |
| HSCA4 | ACCAATGACAAGGAGGAAGACA | CACTCGAGGGCCTCTTTCAC | 176 |
| HSCA5 | CAGAGACTGCCGAGAAAGCA | GCGTTGAACCACCAACAAGG | 246 |
| HSCA6 | GTCAAGGCTGAGGACAAGGG | GACAGCTTCCACCTCATCAATC | 165 |
| HSCA7 | TACACGAGTTTGCAGAAGAGG | AGCCCCACCACTCCTCATAAA | 177 |
| HSCB | TCCATGTTCCTCAACTGCCC | ACAAGAGTCGCAAGCCAGAA | 181 |
| IBA57 | GTGGATGCCACTGTCTTGGA | ATGTGCCAGCAGGATCAACA | 202 |
| INDL | AGTGGTTCTGCGGGGTTTAG | ATCAGCATCAAGCACACCCA | 168 |
| ISA1 | CCTAACCCTAACGGACGCTG | CCAATGACATGCATGAGAGCC | 213 |
| ISA2 | GCCCTCCACTTCGGTTTCTC | CCAGAGCATCCACCCTGTTT | 219 |
| ISA3 | TTGAGTGTCGAGACTGGTGG | TGCTCACCAGGAAAGCAGAA | 193 |
| ISD11 | ACGCGATTTCTCGGACTACAAT | TATTCCAACTGCCTTCTGGC | 156 |
| ISU1 | CCATCGCCTCTTCCTCTGTT | AGTCTTTGCTCGCTTGGCT | 188 |
| MMS19 | AGCTTGCTGTTGGGAGTTGT | GGGTTTGAGGACGAACTGCT | 231 |
| NAR1 | AAAGCTGAGGTTGTGACGGG | AAAGGAAGCCCTTGACTGGG | 204 |
| NBP35-1 | TTCCTAGTGGTTGATGCCCC | ACACCTCACTGAAATCTGTCAC | 232 |
| NBP35-2 | AGTGGTTCTGCGGGGTTTAG | ATCAGCATCAAGCACACCCA | 168 |
| NFS1 | GACAAATCAAAAAGCCATTTTCCAA | CACGTACTGCTCTCGGGTT | 150 |
| NFS2 | GTTCAGGTGGGGTTCCGTAG | AGGCTTCTGTGAGGTTGCTG | 183 |
| NFU1 | CAAAACTCAGCGTTTCGCCT | ATTTGTGGGCCGAGTAGAGC | 163 |
| NFU2 | CCTACTGCTGCAGAGCCAAA | GCAACAGCCTTGACAACTCG | 172 |
| NFU3 | AAACACGCCCTAGAGCCTTC | AGCCTTGACAACTCGGCTAC | 150 |
| NFU4 | GGGAAGCCACTGTTCCTTGA | CGATACTCGATGTCCCCACC | 152 |
| SUFA | GCCCTCCACTTCGGTTTCTC | CCAGAGCATCCACCCTGTTT | 219 |
| SUFB | ACAAAGTTTGCAGATGACAGGA | CTAGCCCCACTGCATAGACC | 221 |
| SUFC | CCATGCAGTCATGGGGAAGA | ACCTGGGATCTCTACTGGGG | 190 |
| SUFD | AATTGCCCACTGGCGTTTTC | ATATCCCCACCCTCAACGGA | 217 |
| SUFE1 | TGGACATGCAGGTGTTAGGG | TTCCCAAGTTGATTTCCCTGTCT | 219 |
| SUFE2 | TGATTCGGGTTTTGGACGGA | AGATATTGCAGTGCCAGAGACA | 227 |
| SUFE3 | TCCTGAGGTCCAAGGTGTCT | GCATCTGCCAAAGAGCAACC | 243 |
| TAH18 | GCGCTATTTCTTTGAGGCAAG | AATGGCATTTGCACTGAAGGG | 174 |
新窗口打开|下载CSV
2 结果
2.1 葡萄Fe-S簇装配基因检索与鉴定
以拟南芥Fe-S簇装配基因的氨基酸序列为参考序列[12],在葡萄基因组数据库中检索到46个Fe-S簇装配基因。其中,14个基因属于质体SUF机制,21个基因属于线粒体ISC机制,11个基因属于细胞质CIA机制(图1、表3)。与拟南芥相比,葡萄基因组中缺少了ISU2、ISU3、NFU5和ADX2,但增加了拟南芥中没有的HSCA3-7、NBP35-2和CIA3;与桃相比[19,20],葡萄基因组中缺少了ADX2,但增加了桃中没有的SUFC、HSCA6和HSCA7;与单子叶植物水稻相比[17],葡萄基因组中缺少了ISU2和ADX2,但检索到水稻中没有的SUFE2、HSCA3-7、NBP35-2和CIA3(表3)。图1
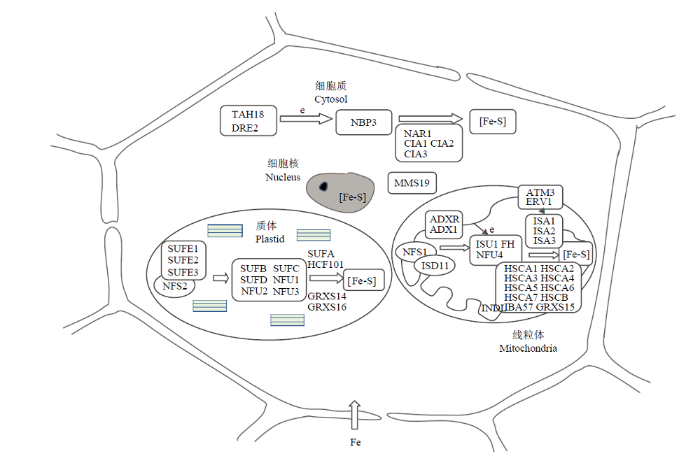 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1葡萄细胞中Fe-S簇装配基因功能模拟分布图
Fig. 1The proposed function and distribution model of Fe-S cluster assembly genes in grape
Table 3
表3
表3葡萄、拟南芥和桃Fe-S簇装配基因对比
Table 3
| 位置 Location | 基因 Gene | 登录号 Locus ID | |||
|---|---|---|---|---|---|
| 拟南芥 Arabidopsis | 葡萄Grape | 桃 Peach | 水稻 Rice | ||
| 质体 Plastid | NFS2 | At1g08490 | GSVIVT01037384001 | ppa005298m | Os12g18900 |
| SUFE1 | At4g26500 | GSVIVT01014835001 | ppa007330m | Os09g09790 | |
| SUFE2 | At1g67810 | GSVIVT01000550001 | ppa017530m | - | |
| SUFE3 | At5g50210 | GSVIVT01007621001 | ppa001921m | Os12g19304 | |
| SUFA | At1g10500 | GSVIVT01022247001 | ppa012351m | Os06g05400 | |
| SUFB | At4g04770 | GSVIVT01012742001 | ppa003788m | Os01g61400 | |
| SUFC | At3g10670 | GSVIVT01036588001 | - | Os03g21490 | |
| SUFD | At1g32500 | GSVIVT01013180001 | ppa004982m | Os01g03650 | |
| NFU1 | At4g01940 | GSVIVT01028158001 | ppa011214m | Os03g20010 | |
| NFU2 | At5g49940 | GSVIVT01023274001 | ppa011050m | Os11g07916 | |
| NFU3 | At4g25910 | GSVIVT01029242001 | ppa010743m | Os06g47940 | |
| HCF101 | At3g24430 | GSVIVT01015424001 | ppa006650m | Os01g52170 | |
| GRXS14 | At3g54900 | GSVIVT01032936001 | ppa012220m | Os03g63420 | |
| GRXS16 | At2g38270 | GSVIVT01011178001 | ppa009373m | Os12g07650 | |
| 线粒体 Mitochondria | NFS1 | At5g65720 | GSVIVT01003603001 | ppa005512m | Os09g16910 |
| ISD11 | At5g61220 | GSVIVT01007919001 | ppa013993m | Os08g14070 | |
| ISU1 | At4g22220 | GSVIVT01013764001 | ppa012356m | Os01g47340 | |
| ISU2 | At3g01020 | - | - | Os05g49300 | |
| ISU3 | At4g04080 | - | - | - | |
| ISA1 | At2g16710 | GSVIVT01033834001 | ppa013203m | Os12g30030 | |
| ISA2 | At2g36260 | GSVIVT01022247001 | ppa12351m | Os01g01610 | |
| ISA3 | At5g03905 | GSVIVT01033842001 | ppa012679m | Os08g28230 | |
| NFU4 | At3g20970 | GSVIVT01011272001 | ppa009781m | Os05g06330 | |
| NFU5 | At1g51390 | - | - | - | |
| ADX1 | At4g21090 | GSVIVT01015024001 | ppa012568m | Os09g26650 | |
| ADX2 | At4g05450 | - | ppa012238m | Os07g01930 | |
| ADXR | At4g32360 | GSVIVT01015352001 | ppa004960m | Os02g17700 | |
| FH | At4g03240 | GSVIVT01000441001 | ppa011940m | Os01g57460 | |
| HSCA1 | At4g37910 | GSVIVT01038517001 | ppa002402m | Os02g53420 | |
| HSCA2 | At5g09590 | GSVIVT01006769001 | ppa001973m | Os03g02260 | |
| HSCA3 | - | GSVIVT01008331001 | ppa002222m | Os09g31486 | |
| HSCA4 | - | GSVIVT01038580001 | ppa002489m | - | |
| HSCA5 | - | GSVIVT01026014001 | ppa002572m | - | |
| HSCA6 | - | GSVIVT01019607001 | - | - | |
| HSCA7 | - | GSVIVT01031125001 | - | - | |
| HSCB | At5g06410 | GSVIVT01022555001 | ppa016242m | Os12g27070 | |
| INDL | At4g19540 | GSVIVT01000762001 | ppa019981m | Os03g42880 | |
| IBA57 | At4g12130 | GSVIVT01004911001 | ppa006632m | Os06g04380 | |
| GRXS15 | At3g15660 | GSVIVT01017244001 | ppa012405m | Os01g07950 | |
| 细胞质 Cytosol | ATM3 | At5g58270 | GSVIVT01024527001 | ppa002114m | Os06g03770 |
| ERV1 | At1g49880 | GSVIVT01037859001 | ppa012227m | Os03g10850 | |
| NAR1 | At4g16440 | GSVIVT01007214001 | ppa005089m | Os03g53750 | |
| NBP35-1 | At5g50960 | GSVIVT01001845001 | ppa005998m | Os04g40880 | |
| NBP35-2 | - | GSVIVT01000762001 | ppa007759m | - | |
| TAH18 | At3g02280 | GSVIVT01031054001 | ppa002941m | Os01g53250 | |
| DRE2 | At5g18400 | GSVIVT01010181001 | ppa009994m | Os04g57810 | |
| CIA1 | At2g26060 | GSVIVT01033839001 | ppa007909m | Os07g14830 | |
| CIA2 | At1g68310 | GSVIVT01018962001 | ppa012624m | Os04g50864 | |
| CIA3 | - | GSVIVT01034035001 | ppa012667m | - | |
| MMS19 | At5g48120 | GSVIVT01016980001 | ppa023072m | Os07g08050 | |
新窗口打开|下载CSV
2.2 葡萄Fe-S簇装配基因及其编码蛋白特征
除NFS1、HSCA2和NAR1染色体定位情况未知外,其他43个葡萄Fe-S簇装配基因在除了5号染色体之外的16条染色体上均有分布;其中,8号染色体含有的基因数目最多,共6个(GRXS16、ISA1、ISA3、HSCB、CIA1和CIA3),7号染色体含有5个(SUFA、NFU1、ISA2、INDL、NBP35-2),其他染色体上至少含有一个基因;同一基因家族中的成员ISA1和ISA3、CIA1和CIA3均位于8号染色体(表3),然而,同属于HSCA基因家族的7个成员分布于截然不同的染色体上,但其CDS长度较为接近,均在1 500—2 000 bp(表4)。Table 4
表4
表4葡萄Fe-S簇装配基因信息
Table 4
| 位置 Location | 基因 Gene | GenBank登录号 GenBank No. | 染色体定位 Chromosome location | 编码区 CDS (bp) | 氨基酸数目 Amino acid No. | 内含子数目 Intron No. |
|---|---|---|---|---|---|---|
| 质体 Plastid | NFS2 | GSVIVT01037384001 | chr6:14614433..14627074 forward | 1392 | 463 | 9 |
| SUFE1 | GSVIVT01014835001 | chr19:10332463..10337676 reverse | 1224 | 407 | 5 | |
| SUFE2 | GSVIVT01000550001 | chr1:7199706..7203084 forward | 855 | 284 | 2 | |
| SUFE3 | GSVIVT01007621001 | chr17:11153636..11159809 reverse | 1806 | 601 | 6 | |
| SUFA | GSVIVT01022247001 | chr7:17815443..17819261 forward | 453 | 150 | 2 | |
| SUFB | GSVIVT01012742001 | chr10:1165899..1176476 reverse | 1536 | 511 | 3 | |
| SUFC | GSVIVT01036588001 | chr13:18723793..18744366 forward | 966 | 321 | 5 | |
| SUFD | GSVIVT01013180001 | chr2:7019468..7043516 forward | 1443 | 480 | 2 | |
| NFU1 | GSVIVT01028158001 | chr7:4298897..4301308 forward | 642 | 213 | 2 | |
| NFU2 | GSVIVT01023274001 | chr12:20249550..20254467 reverse | 684 | 227 | 3 | |
| NFU3 | GSVIVT01029242001 | chr11:18133595..18136869 forward | 714 | 237 | 3 | |
| HCF101 | GSVIVT01015424001 | chr11:3539907..3550291 forward | 1671 | 556 | 14 | |
| GRXS14 | GSVIVT01032936001 | chr14:24341805..24359981 reverse | 552 | 183 | 3 | |
| GRXS16 | GSVIVT01011178001 | chr8:7822419..7828262 reverse | 894 | 297 | 1 | |
| 线粒体 Mitochondria | NFS1 | GSVIVT01003603001 | chrUn:11176164..11189530 forward | 3267 | 1088 | 8 |
| ISD11 | GSVIVT01007919001 | chr17:7527950..7530932 reverse | 219 | 72 | 1 | |
| ISU1 | GSVIVT01013764001 | chr1:7813881..7823597 forward | 273 | 90 | 2 | |
| ISA1 | GSVIVT01033834001 | chr8:17357057..17361779 forward | 402 | 133 | 2 | |
| ISA2 | GSVIVT01022247001 | chr7:17815443..17819261 forward | 453 | 150 | 2 | |
| ISA3 | GSVIVT01033842001 | chr8:17302334..17306498 forward | 465 | 154 | 3 | |
| NFU4 | GSVIVT01011272001 | chr13:10306616..10320274 reverse | 609 | 202 | 4 | |
| ADX1 | GSVIVT01015024001 | chr11:532570..538242 forward | 597 | 198 | 6 | |
| ADXR | GSVIVT01015352001 | chr11:2891542..2897423 reverse | 1461 | 486 | 12 | |
| FH | GSVIVT01000441001 | chr12:8053062..8056733 reverse | 594 | 197 | 4 | |
| HSCA1 | GSVIVT01038517001 | chr3:10899748..10904643 forward | 1842 | 613 | 7 | |
| HSCA2 | GSVIVT01006769001 | chrUn:28290681..28295479 forward | 1716 | 571 | 8 | |
| HSCA3 | GSVIVT01008331001 | chr17:3189753..3194345 forward | 1503 | 500 | 9 | |
| HSCA4 | GSVIVT01038580001 | chr16:21738678..21742840 reverse | 1947 | 648 | 8 | |
| HSCA5 | GSVIVT01026014001 | chr18:25847096..25853527 forward | 1677 | 558 | 7 | |
| HSCA6 | GSVIVT01019607001 | chr2:1900757..1904873 forward | 1821 | 606 | 8 | |
| HSCA7 | GSVIVT01031125001 | chr14:1920620..1924241 forward | 1731 | 576 | 8 | |
| HSCB | GSVIVT01022555001 | chr8:4909017..4918207 forward | 804 | 267 | 5 | |
| INDL | GSVIVT01000762001 | chr7:498142..504281 reverse | 1026 | 341 | 8 | |
| IBA57 | GSVIVT01004911001 | chr2:4748585..4752156 forward | 1113 | 370 | 4 | |
| GRXS15 | GSVIVT01017244001 | chr9:5862839..5867495 reverse | 513 | 170 | 5 | |
| 细胞质 Cytosol | ATM3 | GSVIVT01024527001 | chr6:9155464..9213374 reverse | 2181 | 726 | 19 |
| ERV1 | GSVIVT01037859001 | chr3:7051150..7055710 forward | 597 | 198 | 7 | |
| NAR1 | GSVIVT01007214001 | chrUn:30906448..30911807 forward | 1437 | 478 | 10 | |
| NBP35-1 | GSVIVT01001845001 | chr14:26263495..26271378 reverse | 957 | 318 | 3 | |
| NBP35-2 | GSVIVT01000762001 | chr7:498142..504281 reverse | 1026 | 341 | 8 | |
| TAH18 | GSVIVT01031054001 | chr14:21477906..21501706 reverse | 1899 | 632 | 11 | |
| DRE2 | GSVIVT01010181001 | chr1:16897462..16906512 reverse | 819 | 272 | 6 | |
| CIA1 | GSVIVT01033839001 | chr8:17322989..17332968 forward | 1035 | 344 | 9 | |
| CIA2 | GSVIVT01018962001 | chr4:18301283..18304437 reverse | 360 | 119 | 5 | |
| CIA3 | GSVIVT01034035001 | chr8:15682406..15687298 forward | 363 | 120 | 3 | |
| MMS19 | GSVIVT01016980001 | chr9:3133979..3155012 forward | 3441 | 1146 | 21 |
新窗口打开|下载CSV
基因结构分析结果表明葡萄Fe-S簇装配基因均含有内含子,其中,GRXS16和ISD11仅含有1个内含子,而MMS19含有的内含子数目最多(21个),且长度不一(表4、图2)。但SUFD含有2个内含子,第2个内含子的长度超过20 kb。
图2
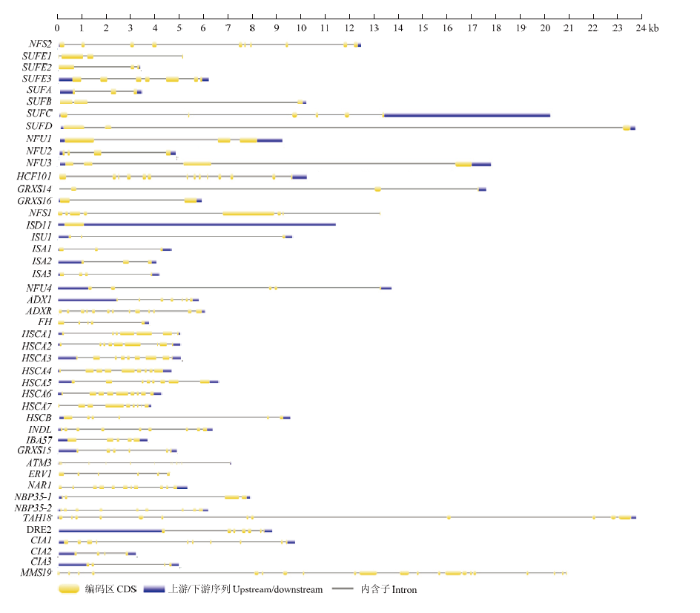 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2葡萄Fe-S簇装配基因的结构分析
Fig. 2Gene structure of Fe-S cluster assembly genes in grape
2.3 葡萄Fe-S簇装配基因亚细胞定位预测
由亚细胞定位预测结果可知,葡萄Fe-S簇装配机制相关蛋白在多种亚细胞结构中均有定位,且不同装配机制的蛋白亚细胞定位情况差异很大(表5)。其中,质体SUF装配机制的蛋白主要定位在叶绿体中,而SUFE1主要定位于细胞核,SUFE3主要定位于细胞质,此外部分蛋白成员在线粒体、内质网膜、液泡膜和细胞质膜也有不同比例的分布;线粒体ISC装配机制中,HSCA4和HSCA6是100%定位在内质网膜,其他成员中,10个主要定位于叶绿体,6个主要定位于线粒体,3个(HSAC3、HSAC5和INDL)主要定位于细胞质。此外,ISC装配机制的蛋白在细胞核、液泡膜、细胞质膜和高尔基体有不同比例的定位;细胞质CIA装配机制中,CIA3是100%定位在细胞质,其他成员中,6个主要定位于细胞核,3个(NAR1、NBP35-2和CIA2)主要定位于细胞质,ATM3主要在细胞质膜,NBP35-1主要在叶绿体;此外,CIA装配机制的蛋白在液泡膜、内质网膜、线粒体和高尔基体也有不同比例的定位(表5)。Table 5
表5
表5Fe-S簇装配基因亚细胞定位预测
Table 5
| 蛋白位置 Protein location | 蛋白 Protein | 亚细胞定位 Subcellular localization (%) | |||||||
|---|---|---|---|---|---|---|---|---|---|
| 叶绿体Chloroplast | 线粒体Mitochondria | 细胞质Cytosol | 细胞核Nucleus | 内质网膜 Endoplastic reticulum | 液泡膜 Vacular membrane | 细胞质膜 Plasma membrane | 高尔基体 Golgi | ||
| 质体 Plastid | NFS2 | 71.43 | 28.57 | - | - | - | - | - | - |
| SUFE1 | 7.14 | - | 21.43 | 57.15 | - | 7.14 | 7.14 | - | |
| SUFE2 | 92.86 | 7.14 | - | - | - | - | - | - | |
| SUFE3 | 7.14 | - | 64.29 | 14.29 | 7.14 | - | 7.14 | - | |
| SUFA | 85.72 | 7.14 | 7.14 | - | - | - | - | - | |
| NFU1 | 72 | 28 | - | - | - | - | - | - | |
| NFU2 | 85.72 | - | - | 7.14 | 7.14 | - | - | - | |
| NFU3 | 57.15 | - | 21.43 | 7.14 | - | - | 14.28 | - | |
| SUFB | 57.50 | 32.50 | - | 10.00 | - | - | - | - | |
| SUFC | 85.71 | 14.29 | - | - | - | - | - | - | |
| SUFD | 68.3 | 21.95 | - | 9.75 | - | - | - | - | |
| HCF101 | 57.14 | 14.29 | - | - | - | 21.43 | - | 7.14 | |
| GRXS14 | 92.86 | - | - | - | - | 7.14 | - | - | |
| GRXS16 | 83.72 | 16.28 | - | - | - | - | - | - | |
| 线粒体 Mitochondria | NFS1 | 35.71 | 35.71 | - | 28.58 | - | - | - | - |
| ISD11 | - | 77.42 | - | 16.13 | - | - | 6.45 | - | |
| ISU1 | 85.72 | - | 14.28 | - | - | - | - | - | |
| ISA1 | 92.85 | 7.15 | - | - | - | - | - | - | |
| ISA2 | 78.57 | 7.15 | 14.28 | - | - | - | - | - | |
| ISA3 | 21.63 | 45.94 | 27.03 | 5.40 | - | - | - | - | |
| NFU4 | 50 | 14.28 | - | 14.28 | - | 7.14 | - | - | |
| ADX1 | 14.28 | 78.58 | - | 7.14 | - | - | - | - | |
| ADXR | 50 | 35.72 | 7.14 | 7.14 | - | - | - | - | |
| FH | 50 | 35.72 | - | 14.28 | - | - | - | - | |
| HSCA1 | 14.28 | 85.72 | - | - | - | - | - | - | |
| HSCA2 | 14.28 | 85.72 | - | - | - | - | - | - | |
| HSCA3 | 21.42 | - | 78.58 | - | - | - | - | - | |
| HSCA4 | - | - | - | - | 100 | - | - | - | |
| HSCA5 | 7.14 | 7.14 | 78.58 | - | - | - | 7.14 | - | |
| HSCA6 | - | - | - | - | 100 | - | - | - | |
| HSCA7 | 43.76 | - | 15.62 | 15.62 | - | 12.5 | - | 12.5 | |
| HSCB | 71.44 | 14.28 | - | 14.28 | - | - | - | - | |
| INDL | 14.28 | - | 78.58 | 7.14 | - | - | - | - | |
| IBA57 | 64.30 | 14.28 | 7.14 | 7.14 | - | - | 7.14 | - | |
| GRXS15 | 42.86 | 57.14 | - | - | - | - | - | - | |
| 细胞质 Cytosol | ATM3 | 7.14 | 7.14 | - | - | 28.57 | - | 57.15 | - |
| ERV1 | 14.28 | - | 14.28 | 57.16 | - | 7.14 | 7.14 | - | |
| NAR1 | 21.42 | 7.14 | 42.88 | 21.42 | - | 7.14 | - | - | |
| NBP35-1 | 57.16 | - | 21.42 | 21.42 | - | - | - | - | |
| NBP35-2 | 14.28 | - | 78.58 | 7.14 | - | - | - | - | |
| TAH18 | 7.14 | - | 35.71 | 57.15 | - | - | - | - | |
| DRE2 | 21.42 | - | 21.42 | 57.16 | - | - | - | 7.14 | |
| CIA1 | 14.28 | - | 28.58 | 53 | - | 7.14 | - | - | |
| CIA2 | 7.14 | - | 64.30 | 21.42 | - | - | - | 7.14 | |
| CIA3 | - | - | 100 | - | - | - | - | - | |
| MMS19 | 14.28 | - | 28.57 | 35.73 | - | 14.28 | - | 7.14 | |
新窗口打开|下载CSV
2.4 葡萄Fe-S簇装配基因的表达特征分析
实时荧光定量PCR分析结果表明,葡萄Fe-S簇装配基因在成年‘马瑟兰’葡萄不同组织部位的表达水平差异较大:其中,ISU1在整体水平的表达量最高(特别是在成熟果实、老叶和幼叶中的表达量极高),其次是HSCA1、ISA2、NFU2、SUFA和SUFB等基因的整体表达水平较高,而SUFE2、NFS1、HSCA2、HSCA6、TAH18和CIA2均未检测到表达量,其他未提及的基因在本研究所有不同葡萄组织材料中有较低或极低的表达量(图3)。图3
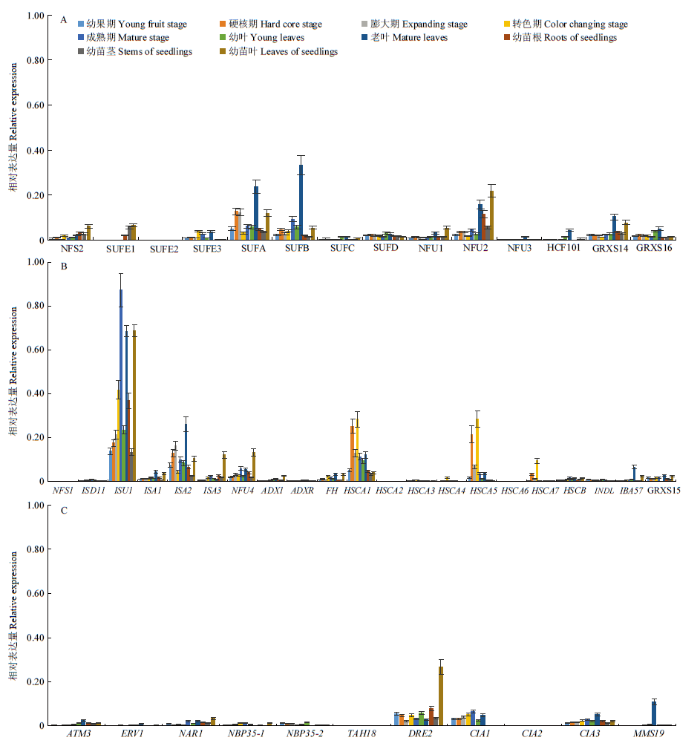 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3葡萄Fe-S簇装配基因组织特异性表达模式分析
A:质体SUF机制基因;B:线粒体ISC机制基因;C:细胞质CIA机制基因
Fig. 3Tissue specific expression pattern analysis of Fe-S cluster assembly genes in grape
A: Plastid SUF machinery genes; B: Mitochondria ISC machinery genes; C: Cytosol CIA machinery genes
在本研究所检测的不同葡萄组织材料中,13个葡萄Fe-S簇装配基因(SUFA、SUFB、NFU3、HCF101、GRXS14、GRXS16、ISA1、ISA2、FH、IBA57、GRXS15、ATM3和MMS19)在成熟叶片中的表达量最高,NFS2、SUFE1、NFU1、ISA3、NFU4、ADX1、NAR1和DRE2等8个基因在幼苗叶片的表达最高,而ISD11、ISU1、IBA57、HSCB、NBP35-1、CIA1和CIA3等6个基因在成熟期果实中的表达量最高,其表达水平随着果实的逐渐发育而递增,均在成熟期果实中达到最高;此外,除HSCA2和HSCA6没有检测到外,其他5个HSCA家族成员均在转色期果实中的表达量最高(图3)。
2.5 葡萄Fe-S簇装配基因对缺铁胁迫的响应差异分析
以‘马瑟兰’组培幼苗为材料,通过实时荧光定量PCR分析葡萄Fe-S簇装配基因在转录水平对缺铁胁迫的响应情况。如图4所示,葡萄Fe-S簇装配基因对缺铁处理较为敏感,所有46个基因至少在1个检测的组织部位对缺铁处理有响应,表达量发生显著变化。其中,22个基因(NFS2、SUFE1、SUFE3、SUFB、SUFC、NFU1、NFU2、NFU3、GRXS14、NFS1、ISA1-3、FH、HSCA4、HSCA6、HSCA7、AMT3、NAR1、TAH18、CIA1和CIA2)对缺铁胁迫最敏感,其表达水平在‘马瑟兰’幼苗所有检测组织中均受缺铁处理的影响而发生显著变化(图4)。根部中,24个葡萄Fe-S簇装配基因在转录水平的表达量受缺铁处理而降低,12个基因的表达量增强,10个基因的表达量没有显著变化;茎部中,14个葡萄Fe-S簇装配基因在转录水平的表达量受缺铁处理而降低,22个基因的表达量增强,10个基因的表达量没有显著变化;叶片中,9个葡萄Fe-S簇装配基因在转录水平的表达量受缺铁处理而降低,27个基因的表达量被增强,10个基因的表达量没有显著变化(图4)。图4
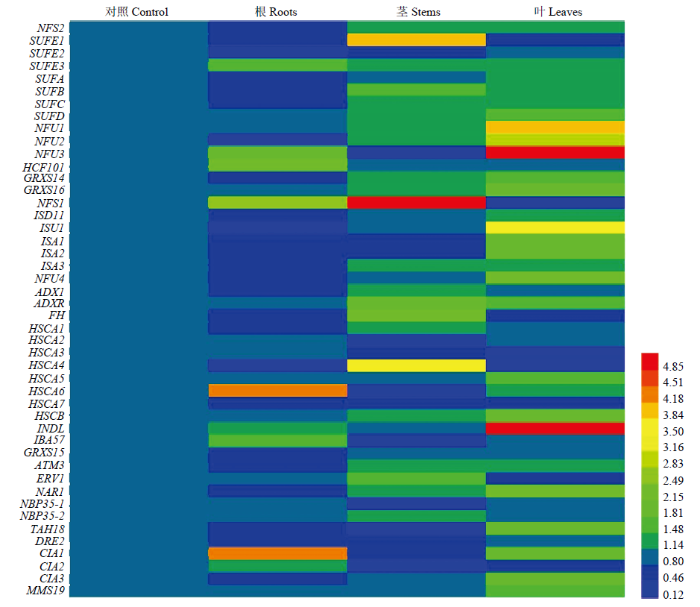 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4利用组培幼苗分析葡萄Fe-S簇装配基因对缺铁胁迫的响应差异
Fig. 4Analysis of differential responses of Fe-S cluster assembly genes under iron deficiency in grape tissue culture seedlings
3 讨论
不管是原核生物还是真核生物,Fe-S簇装配机制是极其复杂和高度保守的[9,11-12]。植物中,拟南芥Fe-S簇装配机制的研究最为透彻[9,12],果树学中Fe-S簇装配机制的研究较稀缺。葡萄中缺乏ADX2,仅含有ADX1,而ADX作为铁氧还蛋白起到电子转移体的作用,暗示葡萄ADX1可能功能独特或者能够独当一面;此外,葡萄中含有更多数目的HSCA,揭示葡萄Fe-S簇装配过程中需要更多的HSP70型伴侣蛋白[11,12]。但ISU和NFU家族基因均编码Scaffold支架蛋白进而参与植物Fe-S簇装配[9,11-14],然而,本研究意外发现多年生木本果树相较于一年生草本植物少了一些典型的支架蛋白,即ISU2、ISU3和NFU5丢失(表6),这些丢失的支架蛋白在本研究选定的木本果树作物中肯定不是Fe-S簇装配途径所必需的。其他支架蛋白,包括SUFB、SUFC、SUFD、NFU1-4、ISU1、NBP35-1和NBP35-2均存在,表明这10个基因在功能上对葡萄Fe-S簇装配及铁代谢途径是足够的。因此,推测高等植物Fe-S簇装配机制可能经历了复杂而长期的进化过程,特别是在线粒体ISC装配机制中,多年生木本植物更有可能进化出“非功能性”支架蛋白丢失的策略。Table 6
表6
表69种高等植物ISU和NFU家族同源基因分析
Table 6
| 基因 Gene | 拟南芥 Arabidopsis thaliana | 盐芥 Thellungiella halophila | 短柄草 Brachypodium sylvaticum | 水稻 Rice | 番茄 Tomato | 柑橘 Orange | 苹果 Apple | 桃 Peach | 葡萄 Grape |
|---|---|---|---|---|---|---|---|---|---|
| ISU1 | At4g22220 | Thhalv10026418m | Bra020855 | Os01g47340 | Solyc03g112900 | orange1.1g030644m | MDP0000778166 | ppa012356m | GSVIVT01013764001 |
| ISU2 | At3g01020 | Thhalv10028164m | Bra013601 | Os05g49300 | Solyc07g007450 | - | - | - | - |
| ISU3 | At4g04080 | Thhalv10029418m | Bra029483 | - | - | - | - | - | - |
| NFU1 | At4g01940 | Thhalv10028839m | Bra000905 | Os03g20010 | Solyc01g079220 | orange1.1g027469m | MDP0000245391 | ppa011214m | GSVIVT01028158001 |
| NFU2 | At5g49940 | Thhalv10014572m | Bra037947 | Os11g07916 | Solyc01g103710 | orange1.1g026830m | MDP0000285539 | ppa011050m | GSVIVT01023274001 |
| NFU3 | At4g25910 | Thhalv10026125m | Bra013933 | Os06g47940 | Solyc05g044630 | orange1.1g038446m | MDP0000952041 | ppa010743m | GSVIVT01029242001 |
| NFU4 | At3g20970 | Thhalv10021272m | Bra031245 | Os05g06330 | Solyc11g007120 | orange1.1g023823m | MDP0000150995 | ppa009781m | GSVIVT01011272001 |
| NFU5 | At1g51390 | Thhalv10011711m | Bra018906 | - | - | - | - | - | - |
新窗口打开|下载CSV
基因表达模式分析结果表明葡萄Fe-S簇装配基因在葡萄不同年龄、不同部位组织材料中的表达量差异很大(图3),且在幼苗不同部位对缺铁胁迫转录水平的响应差异明显,在幼苗根、茎、叶中均有36个基因的表达水平受缺铁处理调控:其中,根部Fe-S簇装配基因的表达水平易受缺铁胁迫诱导而上调,而地上部(茎和叶)Fe-S簇装配基因的表达水平易受缺铁胁迫抑制而下调(图4)。特别值得注意的是,ISU1在‘马瑟兰’葡萄组织中的表达量都是最丰富的,其次是HSCA1和ISA2,这一发现与水稻[17]和桃[19,20]中Fe-S簇装配基因的表达特征较为相似,而与大豆[18]略有差异,大豆HSCA1表达量最高,其次是HSCA2;值得一提的是,ISU1、HSCA1和ISA2都属于线粒体ISC机制的成员[9,10,11,12],暗示线粒体ISC机制需要更多功能性的支架蛋白、伴侣蛋白和电子转移体。
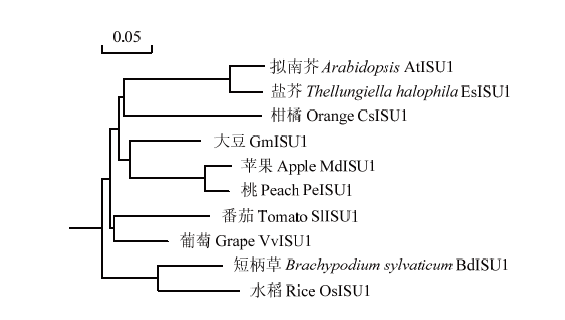
由于ISU1在‘马瑟兰’葡萄果实发育不同时期、叶片发育不同阶段和幼苗组织中的表达量均最高(图3),暗示ISU1是线粒体ISC装配也是葡萄Fe-S簇装配机制不可或缺的支架蛋白,在葡萄铁代谢方面可能发挥关键作用。本研究所分析10种植物ISU1蛋白序列的一致性高达77%,且具有多处高度保守的结构域区域(图5),这些发现暗示了遗传距离较近的不同物种之间的ISU1同源蛋白在长期的进化过程中可能具有相同或相近的功能。进一步的系统发育树分析表明葡萄ISU1和番茄ISU1紧密聚集在一起(图6),而番茄作为典型的模式作物,研究其ISU1蛋白功能可能为揭示葡萄ISU1功能提供理论依据。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5不同植物ISU1蛋白序列一致性分析
Fig. 5Identity analysis of ISU1 protein sequences from different species
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6不同植物ISU1蛋白系统进化树建立
Fig. 6Phylogenetic tree construction of ISU1 protein from different species
此外,本研究表明葡萄Fe-S簇装配基因在‘马瑟兰’成年树体与幼苗中的表达模式有所不同(图3)。但SUFE2编码叶绿体类SufE蛋白[9,12],其在‘马瑟兰’葡萄组织材料中均未检测到表达量,相比之下,SUFE1仅在‘马瑟兰’葡萄幼苗中检测到表达量,而SUFE3在葡萄组织中广泛表达,尽管表达水平相对较低(图3),并且SUFE不同基因成员在转录水平对缺铁胁迫的响应情况较为复杂,且差异明显(图4),这些结果表明同一基因家族不同成员之间的表达模式具有较强的组织特异性。已有研究表明,AtSUFE2表达具有花特异性,在拟南芥花粉中高量表达[20,24]。由此测,葡萄SUFE2可能也有类似特殊的功能,即参与葡萄花粉的发育,但需要进一步的后续功能验证。CIA基因家族编码WD40蛋白、DUF59功能域,在细胞质CIA装配机制中发挥重要功能[11,12,13,14],本研究中发现CIA1仅在‘马瑟兰’成年葡萄果实和叶片中检测到表达量,CIA2在所有检测组织中均没有表达,而CIA3在所有检测组织中的表达量较为均匀(图3),且仅有CIA1在幼苗根部受缺铁胁迫强烈诱导外,CIA家族基因在不同组织中易受缺铁胁迫抑制而降低(图4),再次表明同一基因家族不同成员之间的表达模式具有较强的组织特异性,也暗示葡萄CIA基因功能的发挥依赖于适量的铁素供应。此外,SUFB、SUFC、SUFD、NFU1-4、ISU1、NBP35-1和NBP35-2等支架蛋白编码基因在‘马瑟兰’葡萄不同组织中的表达量较为适中,且易受缺铁胁迫调控,再次暗示这10个基因直接参与葡萄Fe-S簇装配机制。
4 结论
从葡萄中克隆并鉴定了46个Fe-S簇装配基因,其在葡萄果实和叶片发育不同时期的表达水平差异很大,并在葡萄幼苗中对缺铁胁迫在转录水平的响应差异显著;ISU1在葡萄所有组织中的整体表达量较高,且葡萄ISU1和番茄ISU1之间的遗传进化距离最为接近,推测ISU1在葡萄铁代谢方面可能发挥关键作用。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
DOI:10.1080/01904160009382161URL [本文引用: 1]
DOI:10.1007/s11104-006-9166-4URL [本文引用: 1]
[本文引用: 1]
DOI:10.1093/treephys/25.6.761URL [本文引用: 1]
DOI:10.1016/j.tcb.2013.11.005URL [本文引用: 2]
DOI:10.1016/j.tplants.2005.05.002URL [本文引用: 10]
[本文引用: 4]
[本文引用: 4]
DOI:10.1038/nature08301URL [本文引用: 8]
DOI:10.1016/j.tplants.2010.12.006URL [本文引用: 11]
DOI:10.1146/biochem.2005.74.issue-1URL [本文引用: 3]
DOI:10.1146/cellbio.2006.22.issue-1URL [本文引用: 3]
DOI:10.1016/j.tig.2008.05.008URL [本文引用: 2]
[本文引用: 4]
[本文引用: 4]
DOI:10.1007/s00299-014-1718-0URL [本文引用: 2]
DOI:10.1016/j.scienta.2016.05.024URL [本文引用: 3]
[本文引用: 6]
[本文引用: 3]
[本文引用: 3]
[本文引用: 2]
[本文引用: 2]
DOI:10.1371/journal.pone.0111988URL [本文引用: 1]
DOI:10.1074/jbc.M701428200URL [本文引用: 1]
