 ,, 李宇涵, 谭敏, 汪露
,, 李宇涵, 谭敏, 汪露 ,, 黄志宏西南民族大学畜牧兽医学院,成都 610041
,, 黄志宏西南民族大学畜牧兽医学院,成都 610041Biofilm-Forming Phenotype, Antibacterial Resistance Genes, Integrase Genes and Virulence Genes Detection of Escherichia coli Isolated from Yaks and Tibetan Pigs in Northwest Sichuan Plateau
CHEN ChaoXi ,, LI YuHan, TAN Min, WANG Lu
,, LI YuHan, TAN Min, WANG Lu ,, HUANG ZhiHongCollege of Animal and Veterinary Sciences, Southwest Minzu University, Chengdu 610041
,, HUANG ZhiHongCollege of Animal and Veterinary Sciences, Southwest Minzu University, Chengdu 610041通讯作者:
责任编辑: 林鉴非
收稿日期:2020-10-22接受日期:2021-01-6
| 基金资助: |
Received:2020-10-22Accepted:2021-01-6
作者简介 About authors
陈朝喜,Tel:13980060375;E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1984KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
陈朝喜, 李宇涵, 谭敏, 汪露, 黄志宏. 川西北高原牦牛和藏猪源大肠杆菌生物被膜表型、耐药基因、整合酶基因和毒力基因检测. 中国农业科学, 2021, 54(23): 5144-5162 doi:10.3864/j.issn.0578-1752.2021.23.018
CHEN ChaoXi, LI YuHan, TAN Min, WANG Lu, HUANG ZhiHong.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】牦牛和藏猪对高寒、低氧等自然条件具有良好的适应能力而成为川西北高原独特的主要经济动物品种。大肠杆菌性腹泻引起牦牛和藏猪发病率和死亡率不断攀升,同时大量抗生素不合理地投入和使用,导致大肠杆菌在广泛持续的抗生素选择压力下产生抗性而引起腹泻迁延不愈。这在影响川西北高原养殖业健康快速发展的同时,给牧民健康也带来一定的潜在危害和威胁。对牦牛和藏猪源大肠杆菌进行药物敏感性试验、生物被膜形成能力、耐药基因、整合酶基因和毒力基因检测及遗传谱系分型分析,能够为牦牛和藏猪源以大肠杆菌为代表的细菌性疾病发病机制探讨和抗菌药物选择提供数据支持和理论依据。【前人研究进展】相关研究表明,牦牛和藏猪源大肠杆菌的耐药性呈现逐渐增长趋势[1,2,3,4],对氨苄西林、四环素等常用抗生素的耐药率甚至达到 90%以上[5,6,7]。携带的可移动元件如转座子、整合子-基因盒系统等是大肠杆菌多重耐药性传播的重要载体之一[8]。此外,为逃避抗菌药物的清除和各种外界不利因素的影响,大肠杆菌能够形成一种特殊的微生物群落结构——生物被膜[9,10,11],其中的纤维素合酶基因bcsA和bcsB、调节蛋白基因fimH和相变抗原基因agn43等均与其生物被膜的形成和耐药性密切相关[12,13]。此外,毒力基因也可以附着在噬菌体和质粒等可移动遗传元件或毒力岛而影响大肠杆菌的黏附能力和生物被膜形成[14,15]。尽管国内外对大肠杆菌耐药基因和毒力基因的相关性进行了大量研究[16],而川西北高原地区牦牛和藏猪源大肠杆菌耐药性、致病性和生物被膜之间相关性研究尚属空白。【本研究切入点】对2017—2018年无菌采自四川省川西北高原地区养殖场或屠宰场的牦牛、藏猪粪便和胃内容物样本进行大肠杆菌分离和鉴定,采用改良结晶紫半定量染色方法和微量肉汤稀释法对分离菌株分别进行生物被膜形成能力和药物敏感性试验;同时采用普通PCR或多重PCR方法对耐药基因、整合酶基因、毒力基因进行检测和遗传谱系分型分析,以期了解川西北高原牦牛和藏猪源大肠杆菌耐药现状、毒力特性及优势遗传谱系分布等相关生物学特征。【拟解决的关键问题】通过药物敏感性试验、生物被膜形成能力、耐药基因、整合酶基因、毒力基因检测和遗传谱系分析,为川西北高原牧区正确合理选择和使用抗菌药物防治牦牛和藏猪大肠杆菌性腹泻及探究大肠杆菌耐药机制提供参考依据。1 材料与方法
1.1 材料
1.1.1 样本采集和标准菌株 417份牦牛和藏猪新鲜粪便和胃内容物样本于2017年10月至2018年8月间无菌采自四川省川西北高原地区阿坝州和甘孜州部分养殖场或屠宰场(其中牦牛粪便和胃肠道内容物样本263份,藏猪粪便样本154份);大肠杆菌标准株ATCC25922由西南民族大学兽医药理学实验室保存并提供。1.1.2 培养基、抗菌药物和分子生物学试剂 麦康凯琼脂(MacConkey agar)、胰蛋白胨大豆肉汤(TSB,Trypticase soy broth)、LB肉汤(Luria-Bertani broth)购自青岛海博生物技术有限责任公司;MH肉汤(Mueller-Hinton broth)购自北京奥博星生物技术有限公司;15e肠杆菌科细菌生化编码鉴定管购自杭州天和微生物试剂有限公司;阿米卡星(AMK)、庆大霉素(GEN)、卡那霉素(KAN)、大观霉素(SPT)、链霉素(STR)、妥布霉素(TOB)、氨苄西林(AMP)、氯霉素(CHL)、氟苯尼考(FLR)、环丙沙星(CIP)、沙拉沙星(SAR)、达氟沙星(DAN)、恩诺沙星(ENR)、左氧氟沙星 (LVX)、萘啶酸(NAL)、土霉素(OXY)、多西环素(DOX)、多黏菌素B (POL)、利福平(RIF)、磺胺甲噁唑 (SMX)、磺胺二甲嘧啶(SM2)、头孢曲松(CRO)、头孢唑啉(CZO)和头孢噻呋(TIO)等抗菌药物购自上海源叶生物科技有限公司和阿拉丁试剂(上海)有限公司;2×Taq Master Mix购自南京诺唯赞生物科技有限公司;DL2000 DNA marker购自天根生化科技(北京)有限公司;PCR扩增引物由生工生物工程(上海)股份有限公司合成。
1.2 方法
1.2.1 牦牛和藏猪源大肠杆菌分离和鉴定 将无菌采集的新鲜粪便和胃肠道内容物样本置于提前准备好的LB肉汤中,37℃培养12—18 h,划线接种于麦康凯琼脂平板,37℃培养12—16 h,选取粉红色、表面光滑、大小适中的疑似菌落进行连续传代培养;对纯化三代的疑似大肠杆菌菌落进行革兰氏染色,并按照15e肠杆菌科细菌生化编码鉴定管说明书操作步骤进行生化鉴定。1.2.2 药物敏感性试验 以大肠杆菌标准株ATCC25922作为质控菌株,抗菌药物配制、药物敏感性试验操作步骤及其结果判定均参考CLSI(美国临床实验室标准化协会,Clinical and Laboratory Standards Institute)标准进行(2013版)。采用微量肉汤稀释法对分离鉴定的大肠杆菌进行24种抗菌药物敏感性试验,以抑制细菌生长的最低药物浓度作为测试药物的最小抑菌浓度(MIC,minimum inhibitory concentration),每种抗菌药物重复3次。
1.2.3 生物被膜形成能力试验 参照STEPANOVIĆ的改良半定量结晶紫染色法并略作改进[17],对分离鉴定的大肠杆菌进行生物被膜形成能力鉴定。生物被膜形成能力判定标准如下:OD570>4 ODc为强成膜能力,2 ODc<OD570≤ 4 ODc为中等成膜能力,ODc<OD570≤ 2 ODc为弱成膜能力,OD570≤ODc为无成膜能力。
1.2.4 耐药基因、整合酶基因和毒力基因检测 根据参考文献和NCBI数据库基因序列设计耐药基因、整合酶基因和毒力基因扩增引物序列(表1和表2),采用水煮法提取大肠杆菌总DNA。耐药基因、整合酶基因和毒力基因PCR扩增体系均为10 µL:2×Taq Master Mix 5 µL,上游引物(F)、下游引物(R)各0.5 µL,DNA模板1 µL,ddH2O 3 µL。PCR扩增程序为:94℃预变性5 min,94℃变性30 s,以相应退火温度退火30—50 s,72℃延伸30 s至1 min,共35个循环,后72℃再延伸10 min,最后4℃保存。各基因扩增过程中对引物用量和双蒸水用量进行调整,体系总体积不变,退火时间和延伸时间根据目的基因预期片段大小稍作调整。
Table 1
表1
表1耐药基因和整合酶基因扩增引物信息
Table 1
| 抗菌药物类别 Classes of antibacterial agents | 基因名称 Gene name | 引物序列(5′→3′) Primer sequence (5′→3′) | 扩增片段大小 Amplification size (bp) | 退火温度 Annealing temperature (℃) | 参考文献或序列号 Reference or sequence number |
|---|---|---|---|---|---|
| 四环素类 Tetracyclines | tetA | F-CACTATGGCATTCTGCTGGC | 948 | 60 | [18] |
| R-CATAGATCGCCGTGAAGAGG | |||||
| tetB | F-GCCCAGTGCTGTTGTTGTC | 553 | 60 | [18] | |
| R-AAGACCAAGACCCGCTAATG | |||||
| tetC | F-TCCTGCTCGCTTCGCTACT | 730 | 58 | [18] | |
| R-TGGTCGTCATCTACCTGC | |||||
| tetD | F-AAACCATTACGGCATTCTGC | 787 | 56 | [19] | |
| R-GACCGGATACACCATCCATC | |||||
| tetG | F-CGGTCTTATGGGTGCTCTA | 721 | 58 | [18] | |
| R-CCTTGCTTGTTACTGAC | |||||
| tetM | F-TTATCAACGGTTTATCAGG | 397 | 55 | [20] | |
| R-CGTATATATGCAAGACG | |||||
| tetX | F-CAATAATTGGTGGTGGACCC | 468 | 56 | [21] | |
| R-TTCTTACCTTGGACATCCCG | |||||
| 磺胺类 Sulfonamides | sul1 | F-TCAGACGTCGTGGATGTCG | 393 | 57 | [22] |
| R-CGAAGAACCGCACAATCTCG | |||||
| sul2 | F-CCTGTTTCGTCCGACACAGA | 435 | 59 | [23] | |
| R-GAAGCGCAGCCGCAATTCAT | |||||
| sul3 | F-AGATGTGATTGATTTGGGAGC | 443 | 53 | [24] | |
| R-TAGTTGTTTCTGGATTAGAGCCT | |||||
| 喹诺酮类 Quinolones | qnrA | F-TCAGCAAGAGGATTTCTCA | 627 | 55 | [25] |
| R-GGCAGCACTATTACTCCCA | |||||
| qnrB | F-ACGATGCCTGGTAGTTGTCC | 469 | 55 | [25] | |
| R-ACGACATTCGTCAACTGCAA | |||||
| 抗菌药物类别 Classes of antibacterial agents | 基因名称 Gene name | 引物序列(5′→3′) Primer sequence (5′→3′) | 扩增片段大小 Amplification size (bp) | 退火温度 Annealing temperature (℃) | 参考文献或序列号 Reference or sequence number |
| qnrS | F-ACGACATTCGTCAACTGCAA | 417 | 55 | [25] | |
| R-TAAATTGGCACCCTGTAGGC | |||||
| qepA | F-GCAGGTCCAGCAGCGGGTAG | 299 | 56 | [25] | |
| R-CTTCCTGCC CGAGTATCGTG | |||||
| β-内酰胺类 β-lactams | blaDHA | F-AACTTTCACAGGTGTGCTGGGT | 387 | 59 | [26] |
| R-CCGTACGCATACTGGCTTTGC | |||||
| blaTEM | F-ATAAAATTCTTGAAGACGAAA | 1080 | 52 | [26] | |
| R-GACAGTTACCAATGCTTAATC | |||||
| blaCMY-2 | F-ATGATGAAAAAATCGTTATGC | 1143 | 57 | [26] | |
| R-TTGCAGCTTTTCAAGAATGCG | |||||
| blaSHV | F-CACTCAAGGATGTATTGTG | 885 | 53 | [26] | |
| R-TTAGCGTTGCCAGTGCTCG | |||||
| 酰氨醇类 Amphenicols | floR | F-CTGAGGGTGTCGTCATCTAC | 673 | 54 | [23] |
| R-GCTCCGACAATGCTGACTAT | |||||
| cmlA | F-CGCCACGGTGTTGTTGTTAT | 394 | 59 | [23] | |
| R-GCGACCTGCGTAAATGTCAC | |||||
| cmlB | F-ACTCGGCATGGACATGTACT | 284 | 57 | [23] | |
| R-ACGGACTGCGGAATCCATAG | |||||
| 氨基糖苷类 Aminoglycosides | cat1 | F-CTTGTCGCCTTGCGTATAAT | 508 | 53 | [23] |
| R-ATCCCAATGGCATCGTAAAG | |||||
| cat2 | F-AACGGCATGATGAACCTGAA | 547 | 53 | [23] | |
| R-ATCCCAATGGCATCGTAAAG | |||||
| aac(3’)-Ⅳ | F-GGCCACTTGGACTGATCGAG | 409 | 58 | X01385 | |
| R-GCGGATGCAGGAAGATCAAC | |||||
| aadA2 | F-GGTGCTAAGCGTCATTGAGC | 470 | 57 | AB154408 | |
| R-GCTTCAAGGTTTCCCTCAGC | |||||
| rmtB | F-ACATCAACGATGCCCTCAC | 472 | 53 | AB103506 | |
| R-AAGTTCTGTTCCGATGGTC | |||||
| aac(6’)-Ib | F-TTGCGATGCTCTATGAGTGGCTA | 482 | 56 | [27] | |
| R-CTCGAATGCCTGGCGTGTTT | |||||
| aph (3’)-Ⅶ | F-TCCACAGGATGGCAAGATCC | 690 | 55 | AY260546 | |
| R-TTCAACGGGAAACGTCTTGC | |||||
| 整合酶基因 Integrase genes | intⅠ1 | F-CCTCCCGCACGATGATC | 280 | 57 | [28] |
| R-TCCACGCATCGTCAGGC | |||||
| intⅠ2 | F-TTATTGCTGGGATTAGGC | 233 | 52 | [28] | |
| R-ACGGCTACCCTCTGTTATC |
新窗口打开|下载CSV
Table 2
表2
表2毒力基因扩增引物信息
Table 2
| 毒力基因类别 Types of virluence genes | 基因名称 Gene name | 引物序列(5′→3′) Primer sequence(5′→3′) | 扩增片段大小 Amplification size(bp) | 退火温度 Annealing temperature (℃) | 参考文献或序列号 Reference or sequence number |
|---|---|---|---|---|---|
| 产志贺毒素毒力基因 Shiga toxin-producing virulence gene | stx1 | F-ACACTGGATGATCTCAGTGG | 614 | 58 | [29] |
| R-CTGAATCCCCCTCCATTATG | |||||
| stx2 | F-CCTGTCAACTGAGCACTTTG | 779 | 58 | [29] | |
| R-CCATGACAACGGACAGCAGTT | |||||
| 非菌毛黏附素 Non-fimbrial adhesin | eaeA | F-ATGCTTAGTGCTGGTTTAGG | 248 | 55 | EF079676 |
| R-GCCTTCATCATTTCGCTTTC | |||||
| EHEC溶血素 EHEC hemolysin | ehxA | F-CAATAATTGGTGGTGGACCC | 583 | 55 | EF088504 |
| R-TTCTTACCTTGGACATCCCG | |||||
| 相变抗原 Phase variation antigen | agn43 | F-GACTATGACCGGATTSTGGCAGGCT | 499 | 67 | [30] |
| R-GTGGCTCCAGCATCARTTGTCAG | |||||
| 铁结合蛋白 Iron transport periplasmic-binding protein | sitA | F-AGGGGGCACAACTGATTCTCG | 608 | 57 | [31] |
| R-TACCGGGCCGTTTTCTGTGC | |||||
| 溶血素 Hemolysin | hlyF | F-GGCGATTTAGGCATTCCGATACTC | 599 | 55 | [31] |
| R-ACGGGGTCGCTAGTTAAGGAG | |||||
| hlyA | F-AACAAGGATAAGCACTGTTCTGGCT | 1177 | 55 | [32] | |
| R-ACCATATAAGCGGTCATTCCCGTCA | |||||
| 耶尔森菌强毒力岛 Yersinia high-pathogenicity island | fyuA | F-TGATTAACCCCGCGACGGGAA | 880 | 55 | [32] |
| R-CGCAGTAGGCACGATGTTGTA | |||||
| 纤维素合成酶基因 Cellulose synthase operon genes | bcsA | F-GTATCGGTAGAAAGCAAACAGG | 185 | 55 | [33] |
| R-GAACGGTACACGAGAAGAGG | |||||
| bcsB | F-CTCGTGTACCGTTCAGGATTTCT | 388 | 55 | [33] | |
| R-CAGCCCAACTTCATTACCCAT | |||||
| Ⅰ型菌毛亚基 Type Ⅰ fimbriae | fimC | F-GGAAATAACATTCTGCTTGC | 288 | 56 | [34] |
| R-TTTGTTGCATCAAGAATACG | |||||
| 外膜蛋白基因 Outer membrane protein | ompT | F-ATCTAGCCGAAGAAGGAGGC | 559 | 55 | [34] |
| R-CCCGGGTCATAGTGTTCATC | |||||
| 铁离子摄取相关基因 Iron uptake-related gene | irp2 | F-AAGGATTCGCTGTTACCGGAC | 280 | 55 | [34] |
| R-TCGTCGGGCAGCGTTTCTTCT | |||||
| 热敏肠毒素 Heat-stable enterotoxin | LT | F-ATGAGTACTTCGATAGAGG | 279 | 55 | [34] |
| R-ATGGTATTCCACCTAACGC |
新窗口打开|下载CSV
1.2.5 遗传谱系分型 参照CLERMONT等[30]采用多重PCR方法进行chuA、yjaA和TspE4.C2扩增,以chuA、yjaA和TspE4.C2的不同组合作为大肠杆菌遗传谱系分型的判定依据。
1.2.6 数据处理 药物敏感性试验数据利用WHONET 5.6处理分析,相关性分析等利用SPSS 18.0处理分析。
2 结果
2.1 大肠杆菌分离鉴定
对417份样本中分离的疑似大肠杆菌进行革兰氏染色和生化鉴定,共分离鉴定大肠杆菌329株,分离率为78.9%(329/417),其中牦牛源和藏猪源大肠杆菌的分离率分别为78.33%(206/263)和79.87%(123/ 154)。2.2 生物被膜表型分析
329株大肠杆菌生物被膜形成能力表型分析结果如图1所示。大多数菌株表现出弱或无生物被膜形成能力表型,强、中、弱、无成膜能力大肠杆菌分别占0.61%(2/329)、4.56%(15/329)、54.7%(180/329) 和40.12%(132/329),牦牛和藏猪源大肠杆菌各1株为强生物被膜表型。206株牦牛源大肠杆菌中,强、中、弱、无成膜能力菌株分别占0.49%(1/206)、5.34%(11/206)、56.31%(116/206)和37.86%(78/206);123株藏猪源大肠杆菌中,强、中、弱、无成膜能力菌株分别占0.81%(1/123)、3.25%(4/123)、52.03%(64/123)和43.9%(54/123)。图1
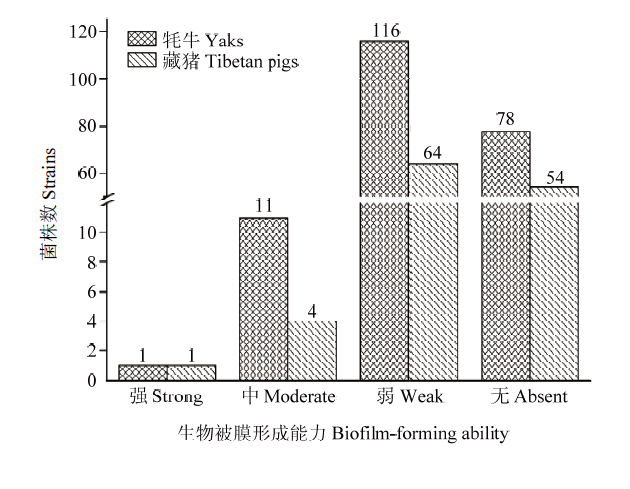 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1329株大肠杆菌生物被膜形成能力
Fig.1Biofilm-forming ability of 329 E.coli
2.3 耐药基因、整合酶基因和毒力基因检测与分析
329株大肠杆菌对卡那霉素、阿米卡星、壮观霉素以外的抗菌药物均表现出不同程度地耐药性,其中磺胺二甲嘧啶、磺胺甲噁唑 、链霉素、氯霉素、土霉素、利福平和氨苄西林等耐药较为严重,其耐药率分别为100.0%、100.0%、100.0%、97.0%、57.1%、56.5%和35.9%(表3);牦牛和藏猪源大肠杆菌对24种抗菌药物的耐药趋势基本一致,藏猪源大肠杆菌耐药水平略高于牦牛源大肠杆菌的原因可能与菌株数量有关。Table 3
表3
表3329株大肠杆菌对24种抗菌药物的药物敏感性试验
Table 3
| 抗菌药物 Antibacterial agents | 耐药率 Antibiotic resistance rate (%) | 中介率 Antibiotic mediate rate (%) | 敏感率 Antibiotic sensitivity rate (%) | ||||||
|---|---|---|---|---|---|---|---|---|---|
| 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | |
| 链霉素Streptomycin | 100.0 | 100.0 | 100.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 庆大霉素Gentamicin | 27.7 | 46.3 | 34.7 | 5.3 | 4.9 | 5.2 | 67.0 | 48.8 | 60.2 |
| 妥布霉素Tobramycin | 1.5 | 1.6 | 1.5 | 4.4 | 4.9 | 4.6 | 94.2 | 93.5 | 93.9 |
| 卡那霉素Kanamycin | 0.0 | 0.0 | 0.0 | 4.9 | 17.1 | 9.4 | 95.1 | 82.9 | 90.6 |
| 大观霉素Spectinomycin | 0.0 | 0.0 | 0.0 | 57.3 | 73.2 | 63.2 | 42.7 | 26.8 | 36.8 |
| 阿米卡星Amikacin | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 100.0 | 100.0 | 100.0 |
| 氯霉素Chloramphenicol | 96.6 | 97.6 | 97.0 | 1.5 | 1.6 | 1.5 | 1.9 | 0.8 | 1.5 |
| 氟苯尼考Florphenicol | 9.7 | 30.9 | 17.6 | 38.8 | 36.6 | 38.0 | 51.5 | 32.5 | 44.4 |
| 磺胺二甲嘧啶Sulfadimidine | 100.0 | 100.0 | 100.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 磺胺甲噁唑 Sulfamethoxazole | 100.0 | 100.0 | 100.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 利福平Rifampicin | 52.9 | 62.6 | 56.5 | 24.3 | 31.7 | 27.1 | 22.8 | 5.7 | 16.4 |
| 多黏菌素B Colistin B | 3.4 | 1.6 | 2.7 | 5.8 | 8.9 | 7.0 | 90.8 | 89.4 | 90.3 |
| 土霉素Oxytetracycline | 41.3 | 83.7 | 57.1 | 6.8 | 4.1 | 5.8 | 51.9 | 12.2 | 37.1 |
| 多西环素Doxycycline | 25.2 | 48.8 | 34.0 | 11.2 | 25.2 | 16.4 | 63.6 | 26.0 | 49.5 |
| 萘啶酸Nalidixic acid | 11.2 | 17.9 | 13.7 | 0.0 | 0.0 | 0.0 | 88.8 | 82.1 | 86.3 |
| 沙拉沙星Sarafloxacin | 7.8 | 3.2 | 6.1 | 9.2 | 9.8 | 9.4 | 83.0 | 87.0 | 84.5 |
| 恩诺沙星Enrofloxacin | 6.8 | 4.9 | 6.1 | 4.4 | 8.9 | 6.1 | 88.8 | 86.2 | 87.8 |
| 环丙沙星Ciprofloxacin | 5.8 | 4.9 | 5.5 | 0.0 | 0.8 | 0.3 | 94.2 | 94.3 | 94.2 |
| 达氟沙星Danofloxacin | 5.8 | 3.3 | 4.9 | 8.3 | 13.0 | 10.0 | 85.9 | 83.7 | 85.1 |
| 左氧氟沙星 Levofloxacin | 4.4 | 2.4 | 3.6 | 1.0 | 0.0 | 0.6 | 94.7 | 97.6 | 95.7 |
| 氨苄西林Ampicillin | 27.7 | 49.6 | 35.9 | 1.5 | 1.6 | 1.5 | 70.9 | 48.8 | 62.6 |
| 头孢噻呋Ceftiofur | 1.9 | 8.9 | 4.6 | 0.5 | 1.6 | 0.9 | 97.6 | 89.4 | 94.5 |
| 头孢曲松Ceftriaxone | 1.9 | 12.2 | 5.8 | 1.0 | 0.8 | 0.9 | 97.1 | 87.0 | 93.3 |
| 头孢唑啉Cefazolin | 0.5 | 0.0 | 0.3 | 0.0 | 0.0 | 0.0 | 99.5 | 100.0 | 99.7 |
新窗口打开|下载CSV
耐药谱型分析结果表明,329株大肠杆菌共存在117种不同的耐药谱型,且耐药谱型分布较为分散, 58.97%(194/329)的菌株表现为多重耐药,最多对18种抗菌药物耐药(表4)。其中,耐7种以上抗菌药物的菌株占80.55%(265/329),优势耐药谱型为CHL/DOX/OXY/RIF/SMX/SM2/STR,占3.34%(11/329);耐8种以上抗菌药物的菌株中,牦牛和藏猪源大肠杆菌分别占各自来源的36.89%(76/206)和60.98%(75/123),优势耐药谱型为AMP/CHL/GEN/OXY/RIF/ SMX/SM2/STR,占2.13%(7/329)。
Table 4
表4
表4329株大肠杆菌耐药谱型汇总
Table 4
| 耐药表型 Antibacterial susceptibility profiles | 菌株数 Strain numbers (%) | ||
|---|---|---|---|
| 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | |
| AMP/CHL/CIP/CRO/CTF/DAN/DOX/ENR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR/TOB | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CRO/CTF/DAN/ENR/FLR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR/TOB | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/DOX/ENR/FLR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
| AMP/CHL/CIP/CZO/DAN/DOX/ENR/FLR/GEN/LVX/NAL/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/DOX/ENR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/ENR/FLR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/DOX/ENR/GEN/LVX/NAL/OXY/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/ENR/GEN/LVX/NAL/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/ENR/GEN/LVX/NAL/OXY/SAR/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CIP/DAN/DOX/ENR/GEN/LVX/NAL/OXY/SAR/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DAN/ENR/GEN/NAL/OXY/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/DAN/DOX/ENR/GEN/NAL/OXY/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CRO/CTF/CZO/DOX/FLR/GEN/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/ENR/FLR/GEN/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CIP/CZO/DOX/FLR/GEN/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CIP/DAN/ENR/GEN/NAL/OXY/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CRO/CTF/CZO/DOX/NAL/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CTF/DOX/GEN/FLR/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/GEN/NAL/OXY/RIF/SMX/SM2/STR | 2 (0.97) | 0 (0.00) | 2 (0.61) |
| AMP/CHL/CRO/CTF/DOX/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CZO/DOX/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/DOX/FLR/GEN/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CTF/GEN/NAL/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CTF/DOX/GEN/OXY/POL/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/GEN/OXY/POL/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/GEN/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 2 (1.63) | 2 (0.61) |
| AMP/CHL/CZO/DOX/GEN/OXY/POL/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/CRO/CZO/DOX/GEN/NAL/OXY/POL/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/CZO/DOX/FLR/GEN/NAL/OXY/SMX/SM2/STR/TOB | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/DOX/ENR/FLR/GEN/NAL/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CTF/DOX/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/OXY/POL/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/GEN/OXY/RIF/SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
| AMP/CHL/CZO/FLR/GEN/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/DOX/GEN/FLR/OXY/RIF/SMX/SM2/STR | 3 (1.46) | 2 (1.63) | 5 (1.52) |
| AMP/CHL/DOX/GEN/OXY/RIF/STR/SMX/SM2/TOB | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| 耐药表型 Antibacterial susceptibility profiles | 菌株数 Strain numbers (%) | ||
| 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | |
| CHL/CZO/DOX/GEN/NAL/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 1 (0.80) | 2 (0.61) |
| AMP/CHL/CRO/CTF/DOX/GEN/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CTF/DOX/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CTF/DOX/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/DOX/FLR/OXY/SMX/SM2/STR | 1 (0.49) | 1 (0.80) | 2 (0.61) |
| AMP/CHL/CZO/DOX/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/FLR/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CZO/GEN/RIF/OXY/SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
| AMP/CHL/DOX/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 2 (1.63) | 2 (0.61) |
| AMP/CHL/DOX/FLR/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/DOX/GEN/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/FLR/GEN/OXY/RIF/SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
| AMP/CHL/GEN/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/CZO/DOX/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/CZO/DOX/GEN/NAL/OXY/SMX/SM2/STR | 1 (0.49) | 1 (0.80) | 2 (0.61) |
| CHL/CZO/DOX/GEN/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/DOX/GEN/FLR/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/CTF/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/CRO/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/DOX/ FLR/OXY/SMX/SM2/STR | 3 (1.46) | 0 (0.00) | 3 (0.91) |
| AMP/CHL/DOX/GEN/OXY/SMX/SM2/STR | 3 (1.46) | 3 (2.44) | 6 (1.82) |
| AMP/CHL/DOX/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 1 (0.80) | 2 (0.61) |
| AMP/CHL/GEN/FLR/OXY/SMX/SM2/STR | 0 (0.00) | 3 (2.44) | 3 (0.91) |
| AMP/CHL/GEN/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| AMP/CHL/GEN/OXY/RIF/SMX/SM2/STR | 6 (2.91) | 1 (0.80) | 7 (2.13) |
| AMP/CHL/NAL/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 2 (1.63) | 2 (0.61) |
| CHL/CRO/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/CTF/DOX/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/CZO/DOX/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/CZO/GEN/OXY/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/DOX/FLR/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/DOX/GEN/NAL/OXY/SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
| CHL/DOX/GEN/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/GEN/FLR/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CTF/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/CZO/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/DOX/OXY/SMX/SM2/STR | 0 (0.00) | 2 (1.63) | 2 (0.61) |
| AMP/CHL/OXY/RIF/SMX/SM2/STR | 4 (1.94) | 0 (0.00) | 4 (1.22) |
| AMP/CHL/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 8 (6.50) | 8 (2.43) |
| AMP/GEN/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/CIP/FLR/RIF/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/CZO/OXY/TOB/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| 耐药表型 Antibacterial susceptibility profiles | 菌株数 Strain numbers (%) | ||
| 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | |
| CHL/DOX/FLR/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/DOX/GEN/OXY/SMX/SM2/STR | 2 (0.97) | 2 (1.63) | 4 (1.22) |
| CHL/DOX/NAL/OXY/SMX/SM2/STR | 2 (0.97) | 0 (0.00) | 2 (0.61) |
| CHL/DOX/OXY/RIF/SMX/SM2/STR | 10 (4.85) | 1 (0.80) | 11 (3.34) |
| CHL/GEN/OXY/POL/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/FLR/GEN/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/GEN/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/GEN/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| GEN/OXY/RIF/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/GEN/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/OXY/SMX/SM2/STR | 0 (0.00) | 2 (1.63) | 2 (0.61) |
| AMP/CHL/RIF/SMX/SM2/STR | 3 (1.46) | 0 (0.00) | 3 (0.91) |
| AMP/DOX/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/CZO/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/DOX/OXY/SMX/SM2/STR | 4 (1.94) | 10 (8.13) | 14 (4.26) |
| CHL/ENR/RIF/SMX/SM2/STR | 0 (0.00)) | 1 (0.80) | 1 (0.30) |
| CHL/FLR/OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| CHL/FLR/RIF/SMX/SM2/STR | 2 (0.97) | 0 (0.00) | 2 (0.61) |
| CHL/GEN/OXY/SMX/SM2/STR | 1 (0.49) | 2 (1.63) | 3 (0.91) |
| CHL/NAL/OXY/SMX/SM2/STR | 0 (0.00) | 3 (2.44) | 3 (0.91) |
| CHL/POL/RIF/SMX/SM2/STR | 3 (1.46) | 0 (0.00) | 3 (0.91) |
| CHL/OXY/RIF/SMX/SM2/STR | 5 (2.43) | 1 (0.80) | 6 (1.82) |
| DOX/GEN/OXY/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| DOX/OXY/RIF/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| AMP/CHL/SMX/SM2/STR | 4 (1.94) | 2 (1.63) | 6 (1.82) |
| CHL/CRO/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/DOX/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/ENR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/FLR/SMX/SM2/STR | 2 (0.97) | 0 (0.00) | 2 (0.61) |
| CHL/GEN/SMX/SM2/STR | 1 (0.49) | 1 (0.80) | 2 (0.61) |
| CHL/OXY/SMX/SM2/STR | 1 (0.49) | 9 (7.32) | 10 (3.04) |
| CHL/SAR/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| CHL/RIF/SMX/SM2/STR | 36 (17.48) | 4 (3.25) | 40 (12.16) |
| CHL/SMX/SM2/STR | 57 (27.67) | 9 (7.32) | 66 (20.06) |
| GEN/SMX/SM2/STR | 1 (0.49) | 0 (0.00) | 1 (0.30) |
| OXY/SMX/SM2/STR | 0 (0.00) | 1 (0.80) | 1 (0.30) |
| SMX/SM2/STR | 2 (0.97) | 1 (0.80) | 3 (0.91) |
新窗口打开|下载CSV
除cat1、cat2、blaCMY-2、blaSHV、tetC、tetG、tetX外,329株大肠杆菌均检测到其它21个耐药基因,其中以aac(6')-Ib最为流行,其次为sul1和floR,其检出率分别为35.26%(116/329)、33.13%(109/329)和34.65%(114/329)。耐药表型与耐药基因型相关性分析表明,牦牛源大肠杆菌对酰胺醇类和氨基糖苷类耐药表型与携带的耐药基因型均相关(P<0.05),对β-内酰胺类耐药表型与blaTEM和blaDHA显著相关(P<0.01);藏猪源大肠杆菌对四环素类、酰胺醇类和氨基糖苷类耐药表型与携带的耐药基因型均相关(P<0.05),对喹诺酮类耐药表型与qnrA相关(P<0.05)。
整合酶基因检测结果表明,31.61%(104/329)的大肠杆菌携带至少一种整合酶基因,intⅠ1和 intⅠ2的检出率分别为30.09%(99/329)和4.56%(15/329),其中10株大肠杆菌(牦牛源2株,藏猪源8株)同时检测到intⅠ1和intⅠ2(图2)。牦牛源大肠杆菌intⅠ1和intⅠ2的检出率均低于藏猪源大肠杆菌,牦牛源大肠杆菌intⅠ1和intⅠ2的检出率分别为18.93%(39/206)和3.4%(7/206),藏猪源大肠杆菌intⅠ1和intⅠ2的检出率分别为48.78%(60/123)和6.5%(8/123)。
图2
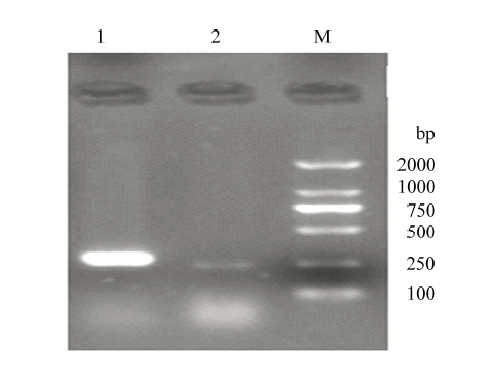 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2整合酶基因扩增产物电泳图
M:DL2000 Marker;1:intⅠ1;2:intⅠ2
Fig. 2Electrophoretogram of integrase genes amplified products
整合酶基因阳性菌株的耐药基因携带情况如表5所示。同时携带整合酶基因菌株的四环素类耐药基因tetA检出率高于未携带整合酶基因的大肠杆菌;sul1、aadA2、aph(3’)-Ⅶ、aac(3’)-Ⅳ和tetD等5个耐药基因与整合酶基因具有相关性(P<0.05),与rmtB、aac(6’)-Ib、tetB、tetM、cmlB、blaDHA 7个耐药基因显著相关(P<0.01),12个耐药基因属于磺胺类、氨基糖苷类、四环素类和β-内酰胺类。
Table 5
表5
表5整合酶基因阳性菌株的耐药基因携带情况信息表
Table 5
| 抗菌药物类别 Classes of antibacterial agents | 基因名称 Gene name | 动物来源 Animal origin | 耐药基因阳性菌株数 Strain number of ARGs positive | |
|---|---|---|---|---|
| 整合酶阳性 Integrase genes positive | 整合酶阴性 Integrase genes negative | |||
| 四环素类 Tetracyclines | tetA | 牦牛 Yaks | 8 | 7 |
| 藏猪Tibetan pigs | 24 | 15 | ||
| tetB | 牦牛 Yaks | 3 | 4 | |
| 藏猪Tibetan pigs | 10 | 13 | ||
| tetD | 牦牛 Yaks | 7 | 7 | |
| 藏猪Tibetan pigs | 12 | 11 | ||
| tetM | 牦牛 Yaks | 0 | 3 | |
| 藏猪Tibetan pigs | 1 | 1 | ||
| 磺胺类 Sulfonamides | Sul1 | 牦牛 Yaks | 13 | 79 |
| 藏猪Tibetan pigs | 7 | 15 | ||
| Sul2 | 牦牛 Yaks | 20 | 19 | |
| 藏猪Tibetan pig | 16 | 25 | ||
| Sul3 | 牦牛 Yak | 3 | 17 | |
| 藏猪Tibetan pig | 16 | 25 | ||
| 喹诺酮类 Quinolones | qnrA | 牦牛 Yak | 0 | 2 |
| 藏猪Tibetan pigs | 4 | 2 | ||
| qnrB | 牦牛 Yaks | 1 | 9 | |
| 藏猪Tibetan pigs | 10 | 14 | ||
| qnrS | 牦牛 Yaks | 8 | 18 | |
| 藏猪Tibetan pigs | 40 | 20 | ||
| qepA | 牦牛 Yaks | 0 | 2 | |
| 藏猪Tibetan pigs | 0 | 0 | ||
| β-内酰胺类 β-lactams | blaTEM | 牦牛 Yaks | 10 | 3 |
| 藏猪Tibetan pigs | 22 | 6 | ||
| blaDHA | 牦牛 Yaks | 28 | 16 | |
| 藏猪Tibetan pigs | 10 | 13 | ||
| 酰氨醇类 Amphenicols | floR | 牦牛 Yaks | 16 | 34 |
| 藏猪Tibetan pigs | 40 | 20 | ||
| cmlA | 牦牛 Yaks | 1 | 12 | |
| 藏猪Tibetan pigs | 21 | 5 | ||
| cmlB | 牦牛 Yaks | 1 | 3 | |
| 藏猪Tibetan pigs | 5 | 5 | ||
| 氨基糖苷类 Aminoglycosides | aac(3’)-Ⅳ | 牦牛 Yaks | 4 | 18 |
| 藏猪Tibetan pigs | 9 | 17 | ||
| rmtB | 牦牛 Yaks | 4 | 20 | |
| 藏猪Tibetan pigs | 10 | 16 | ||
| aadA2 | 牦牛 Yaks | 2 | 35 | |
| 藏猪Tibetan pigs | 18 | 13 | ||
| aac(6’)-Ib | 牦牛 Yaks | 16 | 64 | |
| 藏猪Tibetan pigs | 18 | 18 | ||
| aph(3’)-Ⅶ | 牦牛 Yaks | 14 | 31 | |
| 藏猪Tibetan pigs | 21 | 18 | ||
新窗口打开|下载CSV
毒力基因检测结果表明,除stx1、stx2、ehxA、bcsB、hlyA和hlyE外,其余9个毒力基因均有阳性检出(图3),其中以fimC阳性率最高,bcsA阳性率最低。agn43、bcsA、fyuA、irp2、sitA、fimC、LT、ompT、eaeA 的阳性率分别为53.5%(176/329)、4.26%(14/329)、6.08%(20/329)、7.29%(24/329)、45.59%(150/329)、75.68%(249/329)、12.16%(40/329)、35.26%(116/329)、32.52%(107/329)。牦牛源大肠杆菌agn43、bcsA、fyuA、irp2、sitA、fimC、LT、ompT、eaeA 的阳性率分别为66.99%(138/206)、2.91%(6/206)、7.28%(15/206)、7.28%(15/206)、41.26%(85/206)、85.44%(176/206)、8.25%(17/206)、43.2%(89/206)和30.58%(63/206);藏猪源大肠杆菌agn43、bcsA、fyuA、irp2、sitA、fimC、LT、ompT、eaeA 的阳性率分别为30.89%(38/123)、6.5%(8/123)、4.07%(5/123)、7.32%(9/123)、52.85%(65/123)、59.35%(73/123)、18.7%(23/123)、21.95 %(27/123)和35.77%(44/123)。
图3
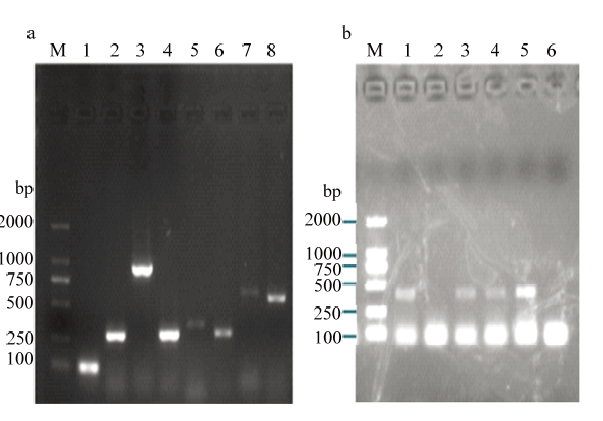 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图39个毒力基因扩增产物电泳
a:M:DL2000 Marker;1:bcsA;2:LT;3:fyuA;4:irp2;5:eaeA;6:fimC;7:sitA;8:ompT. b:M:DL2000 Marker;1-6为agn43扩增产物样本,其中1、3、4、5为阳性样本;2、6为阴性样本
Fig. 3Electrophoretogram of nine virulence genes amplified products
a: M: DL2000 Marker; 1: bcsA; 2: LT; 3: fyuA; 4: irp2; 5: eaeA; 6: fimC; 7: sitA; 8: ompT. b: M: DL2000 Marker; 1-6: PCR amplified products of agn43; 1, 3, 4, 5: positive samples; 2, 6: negative samples
329株大肠杆菌中,共有38种不同毒力谱型(表6,agn43与bcsA不具有直接致病性,毒力谱型和遗传谱系分型分析时不计算在内),285株大肠杆菌至少携带其余7个毒力基因中的1个,其中最多携带6个毒力基因;携带3个以上毒力基因的牦牛和藏猪源大肠杆菌占各自来源的30.1%(62/206)和50.0%(62/123),优势毒力谱型为fimC/ompT/sitA和eaeA/fimC/ompT/sitA,分别占8.25%(17/206)和8.94%(11/123)。
Table 6
表6
表6329株大肠杆菌毒力基因谱型
Table 6
| 毒力基因谱型 Virulence genotypes | 菌株数 Strain number (%) | ||
|---|---|---|---|
| 牦牛 Yaks | 藏猪 Tibetan pigs | 小计 Sub-total | |
| eaeA/fimC/fyuA/irp2/ompT/sitA | 2(0.97) | 7(5.69) | 9(2.74) |
| eaeA/fimC/fyuA/irp2/LT/sitA | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fimC/fyuA/irp2/ompT | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fyuA/irp2/ompT/sitA | 1(0.49) | 0(0.00) | 1(0.30) |
| fimC/fyuA/irp2/ompT/sitA | 1(0.49) | 0(0.00) | 1(0.30) |
| eaeA/fimC/fyuA/irp2 | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fimC/irp2/ompT | 1(0.49) | 1(0.80) | 2(0.61) |
| eaeA/fimC/LT/ompT | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fimC/LT/sitA | 1(0.49) | 1(0.80) | 2(0.61) |
| eaeA/fimC/ompT/sitA | 5(2.43) | 11(8.94) | 16(4.86) |
| fimC/fyuA/irp2/ompT | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fyuA/irp2/sitA | 1(0.49) | 0(0.00) | 1(0.30) |
| fimC/irp2/LT/sitA | 1(0.49) | 0(0.00) | 1(0.30) |
| fimC/irp2/ompT/sitA | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fimC/fyuA | 1(0.49) | 0(0.00) | 1(0.30) |
| eaeA/fimC/ompT | 5(2.43) | 6(4.88) | 11(3.34) |
| eaeA/fimC/sitA | 6(2.91) | 9(7.32) | 15(4.56) |
| eaeA/ompT/sitA | 0(0.00) | 2(1.63) | 2(0.61) |
| fimC/fyuA/irp2 | 1(0.49) | 1(0.80) | 2(0.61) |
| fimC/fyuA/ompT | 1(0.49) | 0(0.00) | 1(0.30) |
| fimC/irp2/LT | 3(1.46) | 3(2.44) | 6(1.82) |
| fimC/irp2/ompT | 0(0.00) | 1(0.80) | 1(0.30) |
| fimC/irp2/sitA | 0(0.00) | 1(0.80) | 1(0.30) |
| fimC/LT/ompT | 1(0.49) | 2(1.63) | 3(0.91) |
| fimC/LT/sitA | 14(6.79) | 3(2.44) | 17(5.17) |
| fimC/ompT/sitA | 17(8.25) | 7(5.69) | 24(7.29) |
| LT/ompT/sitA | 0(0.00) | 1(0.80) | 1(0.30) |
| eaeA/fimC | 14(6.79) | 13(10.57) | 27(8.21) |
| eaeA/sitA | 0(0.00) | 5(4.07) | 5(1.52) |
| fimC/LT | 4(1.94) | 2(1.63) | 6(1.82) |
| fimC/ompT | 25(12.14) | 10(8.13) | 35(10.64) |
| fimC/sitA | 28(13.59) | 5(4.07) | 33(10.03) |
| ompT/sitA | 2(0.97) | 0(0.00) | 2(0.61) |
| eaeA | 0(0.00) | 5(4.07) | 5(1.52) |
| fimC | 5(2.43) | 3(2.44) | 58(17.63) |
| LT | 2(0.97) | 0(0.00) | 2(0.61) |
| ompT | 2(0.97) | 0(0.00) | 2(0.61) |
| sitA | 10(4.85) | 7(5.69) | 17(5.17) |
| ND | 31(15.05) | 13(10.57) | 44(13.37) |
新窗口打开|下载CSV
2.4 耐药基因和毒力基因与遗传谱系分型相关性分析
遗传谱系分型结果如图4和表7所示:329株大肠杆菌遗传谱系以A型为主,共163株,占49.54%(163/329),其余依次为B1型、B2型和D型,分别占39.21%(129/329)、6.08%(20/329)和5.18%(17/329)。其中,牦牛源大肠杆菌遗传谱系以B1型为主,共103株,占50.0%(103/206),其余依次为A型、B2型和D型,分别占35.92% (74/206)、8.25%(17/206)和5.82%(12/206)。藏猪源大肠杆菌以A型为主,共89株,占72.36% (89/123),其余依次为B1型、D型和B2型,分别占21.14%(26/123)、4.07%(5/123)和2.44%(3/123)。图4
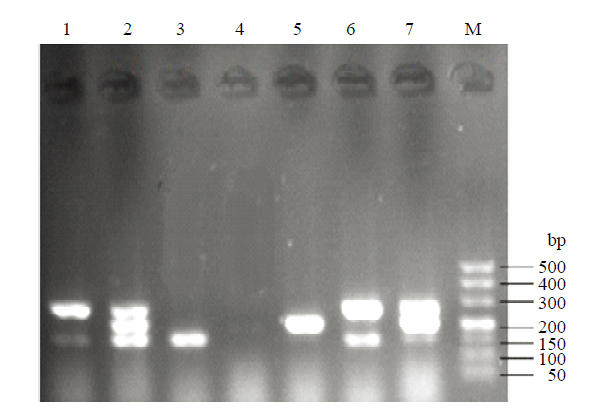 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4遗传谱系分型电泳图
M:DL500 Marker;1、6:遗传谱系D型;2、7:遗传谱系B2型;3:遗传谱系B1型;4、5:遗传谱系A型
Fig. 4Electrophoretogram of phylogenetic analyses
M: DL500 Marker; 1, 6: phylogroup D; 2, 7: phylogroup B2; 3: phylogroup B1; 4, 5: phylogroup A
Table 7
表7
表7329株大肠杆菌的遗传谱系分型分析
Table 7
| 菌株来源 Strain origin | 菌株数 Strain number | 遗传谱系百分比 Percentage of phylogroup (%) | |||
|---|---|---|---|---|---|
| A | B1 | B2 | D | ||
| 牦牛源Yak origin | 206 | 74(35.92) | 103(50.0) | 17(8.25) | 12(5.82) |
| 藏猪源Tibetan pig origin | 123 | 89(72.36) | 26(21.14) | 3(2.44) | 5(4.07) |
| 总计Total | 329 | 163(49.54) | 129(39.21) | 20(6.08) | 17(5.18) |
新窗口打开|下载CSV
329株大肠杆菌的耐药基因和毒力基因与遗传谱系分型相关性热图分析结果显示:A型和B1型分布的耐药基因种类较B2型和D型丰富;A型中sul3、qnrS、tetM耐药基因分布最广,而qnrA和tetD分布较少;B1型中sul1和aac(6')-Ib分布最广,不存在tetM和qnrA(图5-a)。毒力基因主要分布于A型和B1型,且B1型略高于A型,其中,fimC、sitA和ompT基因主要分布于A型和B1型,eaeA、fyuA和irp2的主要分布于B1型,LT基因主要分布于A型,仅1株分布于D型(图5-b)。
图5
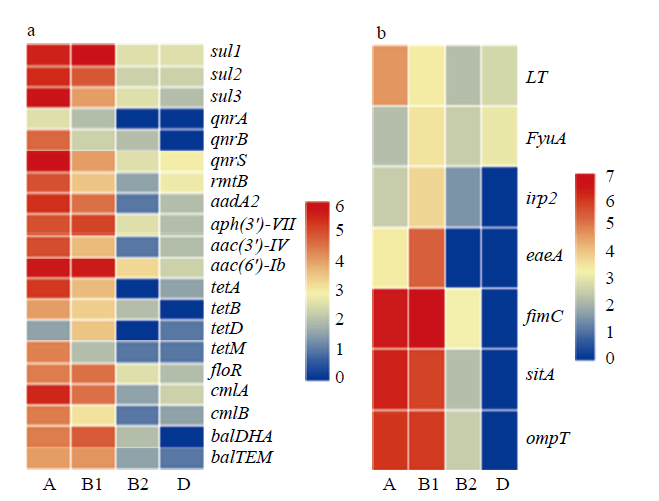 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5耐药基因和毒力基因在遗传谱系中的分布
(a)耐药基因 ARGs;(b)毒力基因 Virulence genes
Fig. 5Distribution of ARGs and virulence genes in phylogroups
3 讨论
3.1 牦牛和藏猪源大肠杆菌耐药现状及其用药情况分析
大肠杆菌血清型复杂和毒力多样的特点加上青藏高原地区牦牛和藏猪多采取自由放养方式等原因,由此引发的大肠杆菌病在影响动物健康的同时对牧民的生活和健康造成一定的影响。随着抗菌药物在牦牛和藏猪细菌性疾病治疗中的不断应用和推广,大肠杆菌在川西北高原已呈现一定的耐药性。药物敏感性试验结果表明,分离菌株均表现出不同程度的耐药性,藏猪源大肠杆菌的耐药率高于牦牛源,其中对氟苯尼考的耐药率达到30.9%,远高于牦牛源的9.7%,对四环素类抗生素(土霉素和多西环素)的耐药率均比牦牛源大肠杆菌高出1倍左右。本研究与李佛生[1]等报道的犊牦牛源大肠杆菌耐药率相近,但与彭青等[35]报道的结果相差较大,分析其原因可能与药敏试验方法和采样地点等因素存在一定的关系。与课题组前期研究相比[3],牦牛源大肠杆菌的耐药水平和整合子携带率较低,与2017—2018年样本采集地点较为分散和样本数量有关。值得注意的是,耐药谱型分析表明,58.97%(194/329)的菌株表现为多重耐药,最多对18种抗菌药物耐药,警示牦牛和藏猪源大肠杆菌耐药情况已较为严重,建议相关部门应及时关注牦牛和藏猪大肠杆菌的耐药现状及其动态变化,采用药物敏感性试验筛选较为敏感的抗菌药物进行治疗,尽量避免养殖人员使用耐药率高的抗菌药物,同时应严格控制抗菌药物用量和使用频率以避免耐药性的产生和广泛传播。在后续的研究中,必须从切断耐药性传播途径角度多方位探究川西北高原大肠杆菌耐药性产生的原因,从而阐明其耐药机制并指导抗菌药物的合理使用。3.2 牦牛和藏猪源大肠杆菌耐药基因、毒力基因、整合酶基因和生物被膜形成能力
抗生素耐药性被认为是全球性威胁之一,以大肠杆菌为代表的革兰氏阴性菌多重耐药菌株引起的持续性感染更加突显,在广泛持续的抗生素压力下,菌株也可以形成生物被膜逃避机体免疫系统的清除和外源性抗菌药物的杀灭,延长病程和增加医疗成本的同时造成疾病的迁延不愈。此外,在质粒、噬菌体、转座子、整合子-基因盒等可移动元件的介导下,耐药基因和毒力基因的广泛传播也给细菌性感染的治疗带来一定的困难[14]。尽管分离菌株大多表现出弱或无生物被膜形成能力,但是对选用的24种抗菌药物呈现不同程度的耐药性并呈现多重耐药现象,其中磺胺二甲嘧啶、磺胺甲噁唑和链霉素完全耐药。耐药表型和耐药基因型之间存在一定的相关性,其中检测到的21个耐药基因中,以aac(6')-Ib最为流行(35.2%),其次为sul1和floR,检出率均在30.0%以上,与SOUFI[36]等报道的耐药基因携带情况基本一致。此外,本研究中氯霉素的耐药率也表现出较高的耐药水平,考虑到氯霉素为食品动物禁用抗生素,其较高的耐药率是否与人与动物之间的直接接触或耐药性基因的水平传播有关需要在后续研究中进一步探究和阐明。
除stx1、stx2、ehxA、bcsB、hlyA和hlyE外,分离菌株中至少检测到agn43、sitA、ompT、eaeA,bcsA、fimC、LT、fyuA和irp2中的1个,fyuA和irp2的检出率均低于张艳芳等[37]的报道。
10株同时携带intⅠ1和 intⅠ2大肠杆菌的耐药基因表型、生物被膜表型和毒力基因型分析结果表明:5株对11种以上的抗菌药物表现出耐药性;70.0%(7/10)的菌株表现出弱生物被膜形成能力,其余3株为无生物被膜形成能力;10株大肠杆菌均携带eaeA,为后续深入研究以eaeA为代表的毒力基因在大肠杆菌生物被膜形成、耐药谱型和毒力谱型及整合子-基因盒系统之间的相关性及其相关性如何等提供一定的研究思路和数据支持,从而阐明毒力基因在大肠杆菌性腹泻致病过程及耐药机制中发挥的作用。
3.3 耐药基因和毒力基因与遗传谱系分型相关性
遗传谱系分型技术将大肠杆菌分为A、B1、B2和D等4个不同的型,其中A型和B1型多为肠道共生菌,而B2和D型多为肠外致病性菌群,该分型技术的优点是有助于了解大肠杆菌相关生物学特征和疾病之间的相关性[30,38]。本研究中,牦牛源大肠杆菌遗传谱系以B1型为主,占50.0%(103/206),其余依次为A型、B2型和D型,分别占35.92%(74/206)、8.25%(17/206)和5.82%(12/206),与王刚等[39]报道的结果基本一致;而藏猪源大肠杆菌遗传谱系以A型为主,占72.36%(89/123),其余依次为B1型、D型和B2型,分别占21.14%(26/123)、4.07%(5/123)和2.44%(3/123),与周陆红等[40]对猪源大肠杆菌遗传谱系分型结果基本一致。
329株大肠杆菌耐药基因和毒力基因与遗传谱系分型相关性分析结果表明,A型和B1型分布的耐药基因种类较B2型和D型多;A型中,sul3、qnrS、tetM耐药基因分布最广,B1型中sul1和aac(6')-Ib分布最广,不存在tetM和qnrA;毒力基因主要分布于A型和B1型,fimC、sitA和ompT主要分布于A型和B1型,eaeA、fyuA和irp2的主要分布于B1型,LT基因主要分布于A型,仅1株分布于D型。
本研究结果表明,尽管分离菌株大多为肠道共生菌群,与采样动物的健康状况有一定关系。然而,分离菌株携带大量的耐药基因和毒力基因的检测结果提示,健康动物可能作为耐药基因和毒力基因的储存库,因此,后续研究应结合细菌基因组重复序列PCR技术(enterobacterial repetitive intergenic consensus-PCR,ERIC-PCR)、多位点序列分型(multilocus sequence typing,MLST)和脉冲电场凝胶电泳(pulsed field gel electrophoresis,PFGE)等多种分型技术,从分子研究水平比较健康和患病动物耐药基因和毒力基因差异并探究耐药性和致病性之间的相关性。
4 结论
对无菌采自阿坝州和甘孜州部分养殖场或屠宰场牦牛、藏猪粪便和胃肠道内容物样本进行大肠杆菌分离和鉴定,同时对分离菌株(329株)进行生物被膜形成能力、药物敏感性试验、耐药基因、整合酶基因和毒力基因检测及遗传谱系分型相关性分析。329株大肠杆菌对24种抗菌药物呈现不同程度的耐药性并出现多重耐药现象,耐药表型和耐药基因之间存在一定的相关性;285株至少携带除agn43和bcsA外7个毒力基因中的1个,最多携带6个毒力基因。31.61%(104/329)的菌株携带至少一种整合酶基因,intⅠ1和 intⅠ2的检出率分别为30.09%和4.56%,其中10株大肠杆菌(牦牛源2株,藏猪源8株),同时检测到intⅠ1和intⅠ2。耐药基因和毒力基因与遗传谱系相关性分析结果表明,A型和B1型分布的耐药基因种类较B2型和D型丰富,毒力基因主要分布于A型和B1型。基于此,后续研究将从以下两方面入手:结合多种分型技术,以同时携带intⅠ1和 intⅠ2的10株大肠杆菌为对象,分析eaeA毒力基因与生物被膜表型、耐药谱型和毒力谱型及整合子-基因盒系统之间是否存在相关性及其相关性如何等?其次,基于牦牛和藏猪源大肠杆菌采样信息资料,对产ESBL/AmpC多重耐药菌株进行多位点序列分型和多因素逻辑回归分析,从而对β-内酰胺类抗菌药物耐药性流行情况、风险因子和ST分型进行深入探究探讨,从分子机制探讨其对β-内酰胺类抗菌药物耐药的分子机制。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
URL [本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1051/vetres/2010029URL [本文引用: 1]
DOI:10.1111/lam.2004.38.issue-5URL [本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1128/AEM.70.1.1-7.2004URL [本文引用: 6]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
DOI:10.1128/AEM.66.10.4555-4558.2000URL [本文引用: 3]
URL [本文引用: 2]
[本文引用: 2]
URL [本文引用: 2]
DOI:10.4142/jvs.2012.13.2.145URL [本文引用: 4]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
