 ,, 关利平, 贺亮亮, 宋银花, 章鹏, 刘三军
,, 关利平, 贺亮亮, 宋银花, 章鹏, 刘三军 ,中国农业科学院郑州果树研究所,郑州 450009
,中国农业科学院郑州果树研究所,郑州 450009Commonality Identification of Molecular Markers Linked to Seedless Genes in Grape
CHEN DouDou ,, GUAN LiPing, HE LiangLiang, SONG YinHua, ZHANG Peng, LIU SanJun
,, GUAN LiPing, HE LiangLiang, SONG YinHua, ZHANG Peng, LIU SanJun ,Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009
,Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009通讯作者:
责任编辑: 赵伶俐
收稿日期:2021-01-25接受日期:2021-04-8
| 基金资助: |
Received:2021-01-25Accepted:2021-04-8
作者简介 About authors
陈豆豆,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2721KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
陈豆豆, 关利平, 贺亮亮, 宋银花, 章鹏, 刘三军. 葡萄无核基因分子标记的通用性鉴定. 中国农业科学, 2021, 54(22): 4880-4893 doi:10.3864/j.issn.0578-1752.2021.22.014
CHEN DouDou, GUAN LiPing, HE LiangLiang, SONG YinHua, ZHANG Peng, LIU SanJun.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】无核葡萄是全世界葡萄育种的主要目标之一。由于葡萄童期较长,而无核表型只能在成年葡萄植株中筛选,且无核遗传机制尚不清楚,造成无核葡萄的育种进程很慢[1]。DNA标记技术为植物育种提供了广阔的应用前景,通过标记辅助选择(marker assisted-selection,MAS)可以提高植物育种的效率和精度[2],因此,筛选准确性高、通用性广的无核基因分子标记对于无核葡萄育种具有重要的意义。【前人研究进展】关于葡萄无核性状遗传规律,前人先后提出了多种假说[3,4,5,6,7]。目前普遍认为,种子败育无核性遗传主要由显性位点SDI(seed development inhibitor,种子发育抑制)调控,位于18号染色体,调控3个独立互补的隐性基因[7,8,9]。MEJÍA等[1]提出与SDI位点接近的基因AGAMOUS-like11(VvAGL11)是葡萄无核的主要功能候选基因,它属于D类MADS-box基因,参与胚珠的发育。这一点在ROYO等[10]的研究中得到证实,即种子形态调节基因VviAGL11中,导致Arg-197-Leu替代的点突变与种子败育完全相关。目前,基于葡萄无核主要位点SDI尤其是无核基因VvAGL11开发了一系列分子标记,主要包括序列特征扩增区(sequence characterized amplified region,SCAR)[9,11-12]及简单重复序列(simple sequence repeats,SSR)两种标记类型[1,8,13-16]。SCAR标记是用特异引物进行扩增,操作简单,成本较低,结果稳定性好。前人通过集群分离分析法(bulk segregant analysis,BSA)开发了与SDI位点连锁不平衡(linkage disequilibrium,LD)的SCAR标记SCC8-1080[9]、SCF27-2000[11]和GSLP1-569[12]。SCF27-2000与无核×无核(‘Ruby Seedless’בSultanina’)后代的无核性状高度相关(81%)[11],GSLP1-569标记也已被广泛用于分析无核性状[17,18,19]。但相关研究表明,GSLP1-569适合于对‘无核白’及其亲缘关系较近的无核品种的分子检测[17,18]。这些标记可以帮助预测无核后代,但也会因偶然出现的零等位基因或标记与SDI位点之间的不完全连锁影响其预测能力[20]。SSR标记是以特异引物PCR为基础的分子标记技术,是一类由几个核苷酸为重复单位组成的串联重复序列,因其具有高多态性、多等位基因、共显性遗传等优势而被广泛应用[21]。SSR标记VMC7f2和p3_VvAGL11被认为是无核性状早期选择的良好候选标记[1,22]。VMC7f2标记位于VvAGL11所预测的ORF上游463 bp[1],QTL(quantitative trait locus)关联分析表明VMC7f2标记的198-bp等位基因与无核性状显著关联,可作为选择无核基因型的潜在标记[13,22]。但在不同遗传背景下对VMC7f2进行验证,其有效性会发生显著变化[1,15,23-24]。p3_VvAGL11是位于基因VvAGL11启动子区的特异性标记[1]。对p3_VvAGL11在不同遗传背景下的测试结果表明,仅出现极低的假阳性率(1.68%)且没有出现假阴性[2]。BENNICI等[25]的研究也表明最有效的标记是VMC7f2和p3_VvAGL11。OCAREZ等[16]在简单重复序列p3_VvAGL11的同一区域开发出新的SSR标记5U_VviAGL11,信息更丰富,更可靠。VMC7f2、p3_VvAGL11和5U_VviAGL11与SDI位点的高度连锁不平衡,使它们成为无核标记辅助选择的主要候选标记,但也需要在其他遗传背景中进行验证。【本研究切入点】基于遗传图谱和BSA等方法开发的这5个无核基因分子标记,在不同遗传背景的鉴定效率尚不明晰,因此,需要对它们的通用性做进一步的鉴定。【拟解决的关键问题】本研究基于前人已开发的准确性较高的5个无核基因分子标记,通过在183份葡萄材料中进行通用性鉴定,旨在理清它们的适用范围及准确性,从中筛选出鉴定准确率高且检测方法简单的分子标记,为分子标记辅助无核葡萄育种提供技术支持。1 材料与方法
试验于2019—2020年在中国农业科学院郑州果树研究所进行。1.1 材料
试验材料共183份,包括96个葡萄品种(其中无核63个,有核33个,电子附表1),保存于中国农业科学院郑州果树研究所国家葡萄种质资源圃,87个F1(‘红地球’ב黎明无核’)杂交单株(表1)保存于中国农业科学院郑州果树研究所尉氏基地。Table 1
表1
表1‘红地球’ב黎明无核’F1代杂交单株的表型鉴定
Table 1
| 序号 No. | 亲本及F1单株 Parents and F1 individual | 种子性状 Seed trait | SCF27-2000 | 5U_Vvi AGL11 | p3_Vv AGL11 | 序号 No. | 亲本及F1单株 Parents and F1 individual | 种子性状 Seed trait | SCF27-2000 | 5U_Vvi AGL11 | p3_Vv AGL11 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ♀ | 红地球Red Globe | SD | 0 | 294:298 | 177:177 | 44 | 9-8-1 | RS | 1 | 298:306 | 177:187 | |
| ♂ | 黎明无核 Dawn Seedless | SL | 1 | 270:306 | 163:187 | 45 | 9-8-2 | SL | 1 | 294:306 | 177:187 | |
| 1 | 7-1-2 | RS | - | - | - | 46 | 9-10-4 | RS | 0 | 298:306 | 177:187 | |
| 2 | 7-1-4 | SD | 1 | 270:298:306 | 163:177:187 | 47 | 9-11-1 | SL | 1 | 298:306 | 177:187 | |
| 3 | 7-3-3 | SL | 1 | 298:306 | 177:187 | 48 | 9-11-4 | SL | 1 | 298:306 | 177:187 | |
| 4 | 7-4-2 | SL | 1 | 294:306 | 177:187 | 49 | 9-14-4 | SD | 1 | 270:294 | 163:177 | |
| 5 | 7-7-3 | RS | 1 | 298:306 | 177:187 | 50 | 9-14-3 | RS | 1 | 270:298 | 163:177 | |
| 6 | 7-7-5 | RS | 1 | 298:306 | 177:187 | 51 | 9-16-3 | SL | 1 | 294:306 | 177:187 | |
| 7 | 7-8-2 | RS | - | - | - | 52 | 9-18-1 | RS | 1 | 294:306 | 177:187 | |
| 8 | 7-9-2 | SL | 1 | 298:306 | 177:187 | 53 | 6-1-1 | SD | 1 | 294:306 | 177:187 | |
| 9 | 7-10-1 | RS | 1 | 294:306 | 177:187 | 54 | 6-2-1 | SD | 1 | 270:294 | 177:187 | |
| 10 | 7-10-3 | RS | 1 | 294:306 | 177:187 | 55 | 6-3-1 | SD | 0 | 270:294 | 163:177 | |
| 序号 No. | 亲本及F1单株 Parents and F1 individual | 种子性状 Seed trait | SCF27-2000 | 5U_Vvi AGL11 | p3_Vv AGL11 | 序号 No. | 亲本及F1单株 Parents and F1 individual | 种子性状 Seed trait | SCF27-2000 | 5U_Vvi AGL11 | p3_Vv AGL11 | |
| 11 | 7-11-1 | RS | 1 | 298:306 | 177:187 | 56 | 6-2-2 | RS | 1 | 294:306 | 177:187 | |
| 12 | 7-11-5 | RS | 1 | 294:306 | 177:187 | 57 | 6-2-3 | SL | 1 | 298:306 | 177:187 | |
| 13 | 7-12-3 | RS | 1 | 298:306 | 177:187 | 58 | 6-3-2 | RS | 1 | 298:306 | 177:187 | |
| 14 | 7-12-5 | RS | 1 | 298:306 | 177:187 | 59 | 6-4-1 | SD | 0 | 270:294 | 163:177 | |
| 15 | 7-13-4 | SL | - | - | - | 60 | 6-5-3 | RS | - | - | - | |
| 16 | 7-14-1 | RS | 1 | 298:306 | 177:187 | 61 | 6-7-2 | RS | 0 | 270:294 | 163:177 | |
| 17 | 7-14-3 | RS | 1 | 294:306 | 177:187 | 62 | 6-7-4 | RS | 0 | 270:294 | 163:177 | |
| 18 | 7-16-1 | SD | 1 | 270:294 | 163:177 | 63 | 6-8-2 | SL | 1 | 294:306 | 177:187 | |
| 19 | 7-17-1 | SD | 0 | 270:294 | 163:177 | 64 | 6-9-1 | SD | 1 | 270:294 | 163:177 | |
| 20 | 7-17-2 | SD | 0 | 270:294 | 163:177 | 65 | 6-9-2 | SD | 0 | 270:294 | 163:177 | |
| 21 | 7-17-3 | SD | 1 | 270:294 | 163:177 | 66 | 6-10-1 | SD | 0 | 270:294 | 163:177 | |
| 22 | 7-18-3 | RS | 1 | 294:306 | 177:187 | 67 | 6-10-2 | SD | 0 | 270:298 | 163:177 | |
| 23 | 8-1-2 | SL | 0 | 294:306 | 177:187 | 68 | 6-10-3 | SD | 1 | 298:306 | 177:187 | |
| 24 | 8-3-5 | SL | 0 | 294:306 | 177:187 | 69 | 6-11-1 | SD | 1 | 270:294 | 163:177 | |
| 25 | 8-4-4 | SL | 1 | 294:306 | 177:187 | 70 | 6-11-2 | SD | 0 | 270:298 | 163:177 | |
| 26 | 8-7-2 | SL | 1 | 270:294:306 | 163:177:187 | 71 | 6-11-3 | SD | 0 | 270:294 | 163:177 | |
| 27 | 8-9-1 | SL | 1 | 298:306 | 177:187 | 72 | 6-13-1 | RS | 1 | 298:306 | 177:187 | |
| 28 | 8-9-3 | SL | 1 | 298:306 | 177:187 | 73 | 6-13-2 | RS | 1 | 298:306 | 177:187 | |
| 29 | 8-10-1 | SL | 1 | 298:306 | 177:187 | 74 | 6-13-3 | RS | 1 | 294:306 | 177:187 | |
| 30 | 8-11-4 | SL | 1 | 298:306 | 177:187 | 75 | 6-14-1 | SD | 0 | 270:298 | 163:177 | |
| 31 | 8-11-5 | RS | 1 | 294:306 | 177:187 | 76 | 6-14-4 | SD | 1 | 270:294 | 163:177 | |
| 32 | 8-12-3 | RS | 1 | 298:306 | 177:187 | 77 | 6-14-2 | SD | - | - | - | |
| 33 | 8-13-3 | SL | 1 | 294:306 | 177:187 | 78 | 6-14-3 | RS | 1 | 298:306 | 177:187 | |
| 34 | 8-14-3 | SL | - | - | - | 79 | 6-15-2 | RS | 1 | 294:306 | 177:187 | |
| 35 | 8-15-2 | SL | 1 | 298:306 | 177:187 | 80 | 6-15-3 | RS | 1 | 298:306 | 177:187 | |
| 36 | 8-16-4 | SL | 1 | 270:294 | 163:177 | 81 | 6-15-4 | SD | 0 | 270:298 | 163:177 | |
| 37 | 8-17-1 | RS | 1 | 294:306 | 177:187 | 82 | 6-16-1 | SD | 1 | 270:294 | 163:177 | |
| 38 | 8-17-3 | RS | 1 | 298:306 | 177:187 | 83 | 6-17-1 | SD | 1 | 270:294 | 163:177 | |
| 39 | 8-13-2 | RS | 1 | 294:306 | 177:187 | 84 | 6-17-2 | RS | 1 | 298:306 | 177:187 | |
| 40 | 8-18-3 | RS | 1 | 294:306 | 177:187 | 85 | 6-18-1 | SD | 1 | 294:306 | 177:187 | |
| 41 | 9-1-3 | RS | 1 | 298:306 | 177:187 | 86 | 6-18-3 | SD | 1 | 270:294 | 163:177 | |
| 42 | 9-3-3 | RS | 1 | 294:306 | 177:187 | 87 | 6-18-2 | RS | 1 | 270:294 | 163:177 | |
| 43 | 9-4-2 | SL | 1 | 294:306 | 177:187 |
新窗口打开|下载CSV
1.2 葡萄DNA提取
取183份葡萄材料的健康幼嫩叶片,液氮研磨,采用CTAB植物基因组DNA快速提取试剂盒(北京艾德莱生物科技有限公司)提取叶片基因组DNA。利用1%琼脂糖凝胶电泳和NanoDrop 1000 spectrophotometer(Themo Scientific)紫外分光光度计检测DNA浓度和纯度,然后将DNA浓度稀释到工作液浓度(约10 ng·μL-1),保存于-20℃,用于后续的PCR扩增。1.3 选择的无核分子标记及PCR扩增条件
选用5个已报道的与葡萄无核性状相关的分子标记(表2),对183份试材进行标记验证。引物由生工生物工程(上海)股份有限公司合成,含有FAM和HEX两种荧光探针的SSR引物由北京阅微基因技术有限公司合成。Table 2
表2
表2分子标记对应引物名称及信息
Table 2
| 引物编号 Primer No. | 引物名称 Primer name | 引物序列(5′-3′) Primer sequences (5′-3′) | 退火温度 Annealing temperature (℃) | 产物预期大小 Fragment size (bp) | 参考文献 Reference |
|---|---|---|---|---|---|
| P1 | SCF27 | F: CAGGTGGGAGTAGTGGAATG R: CAGGTGGGAGTAAGATTTGT | 60 | 2000 | [11] |
| P2 | GSLP1 | CCAGTTCGCCCGTAAATG | 32 | 569 | [12] |
| P3 | VMC7f2 | F: AAGAAAGTTTGCAGTTTATGGTG R: AAGATGACAATAGCGAGAGAGAA | 61 | 198 | [13] |
| P4 | p3_VvAGL11 | F: CTCCCTTTCCCTCTCCCTCT R: AAACGCGTATCCCAATGAAG | 58 | 194 | [1] |
| P5 | 5U_VviAGL11 | F: CGCCCATTCTCTCTCGCTAT R: GTGCAAAAACGCGTATCCCA | 60 | 319 | [16] |
新窗口打开|下载CSV
本试验在参考文献的基础上,对PCR反应体系进行优化。SCAR标记SCF27-2000及GSLP1-569的PCR反应体系20 μL:2×Taq Master Mix 10 μL,正、反向引物各1 μL(10 μmol·L-1),2 μL DNA模板(10 ng·μL-1),ddH2O补至20 μL。反应程序:94℃预变性5 min;94℃变性30 s,适宜Tm值退火30 s,72℃延伸2 min,35个循环;72℃延伸5 min,扩增产物4℃保存。
SSR标记的PCR反应体系10 μL:2×Taq Master Mix for PAGE 5 μL,正、反向引物各0.5 μL(10 μmol·L-1),2 μL DNA模板(10 ng·μL-1),ddH2O补至10 μL。反应程序:94℃预变性3 min;94℃变性30 s,适宜Tm值退火30 s,72℃延伸40 s,29个循环;72℃延伸5 min,扩增产物4℃保存。具体退火温度根据引物设计进行筛选。
1.4 PCR产物检测
SCAR标记SCF27-2000及GSLP1-569的PCR产物采用1.5%琼脂糖凝胶电泳25—30 min(恒压150 V),使用UV凝胶成像仪拍照记录。SSR标记的扩增产物采用荧光毛细管电泳及8%聚丙烯酰胺凝胶(PAGE)电泳进行双验证。1.5 数据处理
根据VIHINEN[26]和BALDI等[27]计算5项指标:真阳性(true positive,TP)、真阴性(true negative,TN)、假阳性(false positive,FP)、假阴性(false negative,FN)、准确率(accuracy)。再根据马亚茹等[15]的方法计算无核检测率。假阳性即样本携带无核等位基因,不表达无核表型;而假阴性即不携带无核等位基因,但表达无核表型。准确率=(TP+TN)/(TP+FP+TN+FN),无核检测率=TP/(TP+FP)。为评估表型与分子数据之间的关联,利用软件SPSS 23.0对表型与SSR标记的等位基因及基因型之间进行卡方(χ2)独立性检验(P<0.05)。2 结果
2.1 SSR标记的PCR体系优化
利用4个无核葡萄品种(‘红宝石无核’‘黎明无核’‘瑞峰无核’‘中葡萄18号’)和4个有核葡萄品种(‘摩尔多瓦’‘红地球’‘玫瑰香’‘先锋’),对SSR标记(p3_VvAGL11、VMC7f2、5U_VviAGL11)进行PCR反应体系优化。如图1-A所示,标记p3_VvAGL11的最适退火温度在58℃时,不同品种的扩增产物表现多态性;如图1-B所示,标记5U_VviAGL11的退火温度在60℃时,得到清晰的多态性条带;如图1-C,标记VMC7f2的最适退火温度为61℃,扩增产物表现出清晰的多态性条带。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1SSR标记的PCR体系优化
A:p3_VvAGL11标记在退火温度58℃;B:5U_VviAGL11标记在退火温度60℃;C:VMC7f2标记在退火温度61℃
Fig. 1PCR system optimization for SSR marker
A: The performance of p3_VvAGL11 in grape at annealing temperature of 58℃; B: The performance of 5U_VviAGL11 in grape at annealing temperature of 60℃; C: The performance of VMC7f2 in grape at annealing temperature of 61℃
2.2 分子标记在96个葡萄品种中的通用性验证
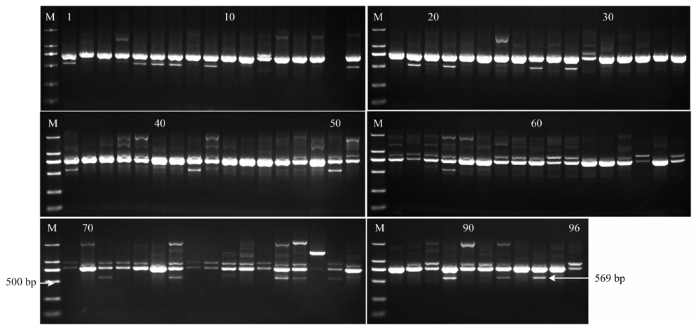
2.2.1 SCAR标记GSLP1-569和SCF27-2000的通用性 在96个葡萄品种(其中无核63个,有核33个)中验证无核标记GSLP1-569和SCF27-2000的通用性。结果显示,GSLP1-569分别在14个无核品种与8个有核品种中扩增出569 bp的无核特异片段,而SCF27-2000分别在62个无核品种与26个有核品种中扩增出2 000 bp的无核特异片段。经统计,分子标记GSLP1-569和SCF27-2000在96个葡萄品种间的鉴定准确率分别为40.6%和71.9%,无核检测率分别为63.6%和70.5%(图2、图3)。由GSLP1-569无核标记检测出的14个无核品种包括‘巩义无核白’‘白无核’‘布朗无核’‘长穗无核白’‘火焰无核’‘美丽无核’‘宁夏无核白’‘无核白-阿富汗’‘早无核白’‘无核白’‘夏日阳光’‘郑艳无核’‘黑爱默无核’‘波尔莱特’,其中11个由‘无核白’衍生而来(附表1、图2)。除体细胞变异品种‘瑞锋无核’外,SCF27-2000可以准确鉴定98.4%的无核品种,表明其在无核×无核的适用范围更广(图3)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2SCAR标记GSLP1-569对96个葡萄品种的验证
Fig. 2Validation of SCAR marker GSLP1-569 for 96 grape cultivars
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3SCAR标记SCF27-2000对96个葡萄品种的验证
Fig. 3Validation of SCAR marker SCF27-2000 for 96 grape cultivars
2.2.2 SSR标记的鉴定结果 在96个葡萄品种中验证3个SSR标记的通用性。结果表明,分子标记p3_VvAGL11检测到8个等位基因,分别为163、165、173、177、179、181、185、187;VMC7f2标记检测到7个等位基因,分别为187、189、191、193、195、197、201;5U_VviAGL11标记检测到17个等位基因,分别为260、262、266、270、272、278、280、282、288、294、296、298、302、304、306、308、312(表3)。在p3_VvAGL11标记检测到的等位基因中,187-bp(χ2=65.333,P=6.32E-16)和177-bp(χ2=8.637,P=0.003)与种子表型显著关联,在63个无核(-)品种中,存在187-bp且表现为无核表型的个体有93.7%,存在187-bp的个体中90.7%表现为无核表型,表明187-bp与无核性状最相关;在33个有核(+)品种中,存在177-bp且表现为有核的个体占87.9%,表明177-bp与有核性状关联(表3)。分子标记p3_VvAGL11检测到13种基因型,其中177/187(χ2=46.061,P=1.15E-11)、187/187(χ2=7.265,P=0.007)和177/177(χ2=9.872,P=0.002)与种子表型之间存在显著关联。基因型177/187在91.8%的无核和8.2%的有核个体中被检测到;187/187在100%的无核个体中被检测到,且在有核个体中缺失;177/177在84.2%有核和15.8%的无核植株中被检测到(表4)。
Table 3
表3
表3微卫星等位基因分布与所收集葡萄品种中无核的相关性
Table 3
| SSR标记 SSR marker | 等位基因 Allele | 种子 Seed | 相关性 Correlation | SSR标记 SSR marker | 等位基因 Allele | 种子 Seed | 相关性 Correlation | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| bp | 频率Freq. | - | + | χ2 | P | bp | 频率 Freq. | - | + | χ2 | P | |||
| p3_VvAGL11 | 163 | 0.03 | 2 | 1 | 0.339 | 0.561 | 5U_VviAGL11 | 260 | 0.03 | 2 | 1 | 0.339 | 0.561 | |
| 165 | 0.07 | 1 | 6 | 3.707 | 0.054 | 262 | 0.03 | 1 | 2 | 0.339 | 0.561 | |||
| 173 | 0.01 | 0 | 1 | 1.005 | 0.316 | 266 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 177 | 0.81 | 49 | 29 | 8.637 | 0.003** | 270 | 0.04 | 4 | 0 | 4.085 | 0.043* | |||
| 179 | 0.04 | 1 | 3 | 1.021 | 0.312 | 272 | 0.02 | 1 | 1 | 0.000 | 1 | |||
| 181 | 0.06 | 4 | 2 | 0.688 | 0.407 | 278 | 0.04 | 0 | 4 | 4.085 | 0.043* | |||
| 185 | 0.02 | 0 | 2 | 2.021 | 0.155 | 280 | 0.02 | 1 | 1 | 0.000 | 1 | |||
| 187 | 0.68 | 59 | 6 | 65.333 | 6.32E-16** | 282 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 288 | 0.04 | 1 | 3 | 1.021 | 0.312 | |||||||||
| 294 | 0.34 | 18 | 15 | 0.329 | 0.566 | |||||||||
| VMC7f2 | 187 | 0.10 | 6 | 4 | 0.422 | 0.516 | 296 | 0.06 | 1 | 5 | 2.753 | 0.097 | ||
| 189 | 0.72 | 59 | 10 | 54.317 | 1.71E-13** | 298 | 0.36 | 25 | 10 | 7.862 | 0.005** | |||
| 191 | 0.70 | 49 | 18 | 22.031 | 0.000003** | 302 | 0.09 | 5 | 4 | 0.117 | 0.733 | |||
| 193 | 0.05 | 3 | 2 | 0.205 | 0.65 | 304 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 195 | 0.07 | 2 | 5 | 1.334 | 0.248 | 306 | 0.67 | 58 | 6 | 63.375 | 1.71E-15** | |||
| 197 | 0.03 | 2 | 1 | 0.339 | 0.561 | 308 | 0.11 | 5 | 6 | 0.096 | 0.756 | |||
| 201 | 0.01 | 0 | 1 | 1.005 | 0.316 | 312 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
新窗口打开|下载CSV
Table 4
表4
表4微卫星基因型分布与所收集品种中无核的相关性
Table 4
| SSR标记 SSR marker | 基因型 Genotype | 种子Seed | 相关性Correlation | SSR标记 SSR marker | 基因型Genotype | 种子Seed | 相关性 Correlation | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Alleles | 频率Freq. | - | + | χ2 | P | Alleles | 频率Freq. | - | + | χ2 | P | |||
| p3_VvAGL11 | 163:177 | 0.01 | 0 | 1 | 1.005 | 0.316 | 5U_VviAGL11 | 260:298 | 0.02 | 1 | 1 | 0 | 1 | |
| 163:187 | 0.02 | 2 | 0 | 2.021 | 0.155 | 260:306 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 165:177 | 0.06 | 1 | 5 | 2.753 | 0.097 | 262:294 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 165:181 | 0.01 | 0 | 1 | 1.005 | 0.316 | 262:298 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 173:187 | 0.01 | 0 | 1 | 1.005 | 0.316 | 266:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 177:177 | 0.20 | 3 | 16 | 9.872 | 0.002* | 270:306 | 0.04 | 4 | 0 | 4.085 | 0.043* | |||
| 177:179 | 0.02 | 0 | 2 | 2.021 | 0.155 | 272:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 177:185 | 0.01 | 0 | 1 | 1.005 | 0.316 | 272:306 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 177:187 | 0.51 | 45 | 4 | 46.061 | 1.15E-11* | 278:296 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 179:185 | 0.01 | 0 | 1 | 1.005 | 0.316 | 278:298 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 179:187 | 0.01 | 1 | 0 | 1.005 | 0.316 | 278:308 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 181:187 | 0.05 | 4 | 1 | 1.848 | 0.174 | 280:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 187:187 | 0.07 | 7 | 0 | 7.265 | 0.007* | 280:298 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 282:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
| 282:306 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
| 288:302 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
| 288:306 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||||||||
| 288:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
| 288:312 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
| VMC7f2 | 187:187 | 0.01 | 0 | 1 | 1.005 | 0.316 | 294:294 | 0.01 | 0 | 1 | 1.005 | 0.316 | ||
| 187:189 | 0.04 | 4 | 0 | 4.085 | 0.043* | 294:298 | 0.03 | 0 | 3 | 3.048 | 0.081 | |||
| 187:191 | 0.05 | 2 | 3 | 0.205 | 0.65 | 294:302 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 189:189 | 0.10 | 4 | 6 | 0.422 | 0.516 | 294:304 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 189:191 | 0.49 | 45 | 2 | 52.092 | 5.30E-13* | 294:306 | 0.18 | 16 | 1 | 14.521 | 0.0001* | |||
| 189:193 | 0.04 | 3 | 1 | 1.021 | 0.312 | 294:308 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 189:195 | 0.01 | 1 | 0 | 1.005 | 0.316 | 294:312 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 189:197 | 0.02 | 2 | 0 | 2.021 | 0.155 | 296:306 | 0.03 | 1 | 2 | 0.339 | 0.561 | |||
| 189:201 | 0.01 | 0 | 1 | 1.005 | 0.316 | 296:308 | 0.02 | 0 | 2 | 2.021 | 0.155 | |||
| 191:191 | 0.14 | 1 | 12 | 9.984 | 0.0016* | 298:298 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||
| 191:195 | 0.02 | 1 | 1 | 0 | 1 | 298:306 | 0.25 | 23 | 1 | 23.048 | 0.000002* | |||
| 191:197 | 0.01 | 0 | 1 | 1.005 | 0.316 | 302:306 | 0.05 | 4 | 1 | 1.848 | 0.174 | |||
| 193:193 | 0.01 | 0 | 1 | 1.005 | 0.316 | 302:308 | 0.01 | 1 | 0 | 1.005 | 0.316 | |||
| 195:195 | 0.04 | 0 | 4 | 4.085 | 0.043* | 306:306 | 0.03 | 3 | 0 | 3.048 | 0.081 | |||
| 306:308 | 0.04 | 4 | 0 | 4.085 | 0.043* | |||||||||
| 308:308 | 0.01 | 0 | 1 | 1.005 | 0.316 | |||||||||
新窗口打开|下载CSV
由此可知,基因型177/187和187/187与无核性状显著相关,而177/177与有核性状显著相关。总体上,在具有分子标记p3_VvAGL11的强连锁不平衡中,等位基因187-bp的存在导致了无核表型,在96个葡萄品种中的鉴定准确率达89.6%,无核检测率为90.7%。
在VMC7f2标记的等位基因中,189-bp(χ2= 54.317,P=1.71E-13)和191-bp(χ2=22.031,P=0.0000)与种子表型显著关联,存在189-bp且表现为无核表型的个体占无核品种的93.7%,存在189-bp的个体中85.5%为无核品种,表明189-bp与无核性状关联最显著(表3)。分子标记VMC7f2检测到14种基因型,其中基因型187/189(χ2=4.085,P=0.043)、189/191(χ2=52.092,P=5.30E-13)、191/191(χ2=9.984,P=0.0016)和195/195(χ2=4.085,P=0.043)与种子表型显著关联(表4)。187/189在100%无核个体中被检测到,在有核品种中缺失;189/191在95.7%的无核个体和4.3%的有核个体中被检测到;191/191在92.3%的有核个体和7.7%的无核个体中被检测到;195/195在100%的有核个体中被检测到,但等位基因195-bp中与种子表型无相关性(表3、表4)。由此可知,基因型187/189和189/191与无核性状显著相关,而191/191与有核性状显著相关。总体上,在具有分子标记VMC7f2的强连锁不平衡中,等位基因189-bp的存在导致了无核表型,而191-bp可能与有核性状相关,在96个葡萄品种中其鉴定准确率为85.4%,无核检测率为85.5%。
在5U_VviAGL11标记检测的等位基因中,270-bp(χ2=4.085,P=0.043)、278-bp(χ2=4.085,P=0.043)、298-bp(χ2=7.005,P=0.008)和306-bp(χ2=63.375,P=1.71E-15)与种子表型显著相关(表3)。虽然等位基因270-bp和298-bp与无核性状关联,但由于只存在于少数无核品种中,不是最相关的无核等位基因;存在278-bp的个体100%为有核品种,表明其与有核性状关联;存在306-bp且表现为无核表型的个体占无核品种的92.1%,存在306-bp的个体中有90.6%为无核品种,表明等位基因306-bp与无核性状关联最显著。分子标记5U_VviAGL11检测到35种基因型,其中基因型294/306(χ2=14.521,P=0.0001)、298/306(χ2=23.048,P=0.000002)、270/306(χ2=4.085,P=0.043)和306/308(χ2=4.085,P=0.043)与种子表型显著关联。基因型294/306存在于94.1%的无核个体中;298/306存在于95.8%的无核个体中;270/306和306/308也存在于全部无核个体中(表4),由此可知,这4种基因型均与无核性状关联,其中基因型294/306和298/306与无核性状关联极显著。总体上,在具有分子标记5U_VviAGL11的强连锁不平衡中,等位基因306-bp的存在导致了无核表型,在96个葡萄品种中其鉴定准确率为88.5%,无核检测率为90.6%。
综上所述,分子标记p3_VvAGL11、VMC7f2和5U_VviAGL11可以分别利用其无核等位基因187-bp、189-bp和306-bp的存在与否对杂交群体进行分子标记辅助育种。
2.3 F1代的验证分析
通过分析上述5个分子标记在96个葡萄品种中的通用性和准确率,进一步利用SCAR标记SCF27-2000及SSR标记p3_VvAGL11、5U_VviAGL11对‘红地球’ב黎明无核’的F1杂交单株进行鉴定。F1代中有效的无核表型(SL)21个,残核表型(RS)35个,有核表型(SD)25个(表1)。SCF27-2000对F1代的鉴定结果(表5)显示,在90.48%的SL和91.43%的RS单株中检测到该标记,而在SD个体中扩增的相对频率(假阳性)下降到17.28%。存在2000-bp且表现为RS或SL表型的单株有78.5%(χ2=13.419,P=0.0012),在F1杂交单株中,该标记的鉴定准确率为76.54%,假阴性率为6.17%。
Table 5
表5
表5SCAR标记SCF27-2000对‘红地球’ב黎明无核’杂交后代的检测结果
Table 5
| 亲本基因型 Parental genotype | 无核性 Seedlessness | SCF27-2000 | SCF27-2000对杂交后代的检测结果 Detection results of SCF27-2000 for progenies | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2000-bp | 显著性 Significance | 携带2000 bp To keep 2000 bp | 无(残)核 SL, RS | 假阳性 False positives | 假阴性 False negatives | |||||||
| SCF27-2000 | 种子Seed N (%) | 0 | 2000-bp | + | - | χ2 | P | |||||
| 0×1 | SL | 21(26%) | 2 | 19 | 19 | 2 | 13.419 | 0.0012 (<0.01) | 65 (80.3%) | 51 (78.5%) | 14 (17.28%) | 5 (6.17%) |
| RS | 35(43%) | 3 | 32 | 32 | 3 | |||||||
| SD | 25(31%) | 11 | 14 | 14 | 11 | |||||||
| 总计Total | 81 | 16 | 65 | 65 | 16 | |||||||
新窗口打开|下载CSV
SSR标记p3_VvAGL11在F1代的鉴定结果(表6)表明,等位基因187-bp与SL性状显著关联(χ2= 41.188,P=1.14E-9),177-bp与SD性状有关,是从‘红地球’遗传的。无核等位基因187-bp在95.24%的SL和88.57%的RS植株中被检测到。存在187-bp且表现为RS或SL表型的杂交单株占91.1%。在F1杂交单株中,该标记的鉴定准确率为87.65%,假阳性、假阴性率分别为6.17%、6.17%。F1代中形成3种基因型,其中基因型163:177中有80%为SD性状,177:187和163:177:187中有91.1%为RS或SL性状,由此可知,无核等位基因187-bp为显性基因,符合SDI位点的显性遗传模型。
Table 6
表6
表6SSR标记p3_VvAGL11对‘红地球’ב黎明无核’杂交后代的检测结果
Table 6
| 亲本基因型 Parental genotype | 无核性 Seedlessness | p3_VvAGL11基因分型 p3_VvAGL11 genotyping | p3_VvAGL11对杂交后代的检测结果 Detection results of p3_VvAGL11 for progenies | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 187-bp | 显著性 Significance | 携带187 bp To keep 187 bp | 无(残)核 SL, RS | 假阳性 False positives | 假阴性 False negatives | ||||||||||||||||||||
| p3_VvAGL11 | 种子Seed N (%) | 163:177 | 177:187 | 163:177:187 | + | - | χ2 | P | |||||||||||||||||
| 177:177×163:187 | SL | 21(26%) | 1 | 19 | 1 | 20 | 1 | 41.188 | 1.14E-9 (<0.01) | 56 (69.1%) | 51 (91.0%) | 5 (6.17%) | 5 (6.17%) | ||||||||||||
| RS | 35(43%) | 4 | 31 | 0 | 31 | 4 | |||||||||||||||||||
| SD | 25(31%) | 20 | 4 | 1 | 5 | 20 | |||||||||||||||||||
| 总计Total | 81 | 25 | 54 | 2 | 56 | 25 | |||||||||||||||||||
新窗口打开|下载CSV
SSR标记5U_VviAGL11对F1代的鉴定结果(表7)表明,等位基因306-bp与无核性状显著关联(χ2=44.959,P=1.73E-10),306-bp分别存在于95.24%的SL和88.57%的RS植株中。存在306-bp且表现为RS或SL表型的杂交单株占92.7%。该标记的准确率为88.89%,假阳性、假阴性率分别为4.94%、6.17%。
Table 7
表7
表7SSR标记5U_VviAGL11对‘红地球’ב黎明无核’杂交后代的检测结果
Table 7
| 亲本基因型 Parental genotype | 无核性 Seedlessness | 5U_VviAGL11基因分型 5U_VviAGL11 genotyping | 5U_VviAGL11对杂交后代的检测结果 Detection results of 5U_VviAGL11 for progenies | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 306-bp | 显著性 Significance | 携带306 bp To keep 306 bp | 无(残)核 SL, RS | 假阳性 False positives | 假阴性 False negatives | ||||||||||||
| 5U_VviAGL11 | 种子Seed N (%) | 270:294 | 270:298 | 294:306 | 298:306 | 270:298:306 | 270:294:306 | + | - | χ2 | P | ||||||
| 294:298× 270:306 | SL | 21 (26%) | 1 | 0 | 9 | 10 | 0 | 1 | 20 | 1 | 44.959 | 1.73E-10 (<0.01) | 55 (67.9%) | 51 (92.7%) | 4 (4.94%) | 5 (6.17%) | |
| RS | 35 (43%) | 3 | 1 | 14 | 17 | 0 | 0 | 31 | 4 | ||||||||
| SD | 25 (31%) | 17 | 4 | 2 | 1 | 1 | 0 | 4 | 21 | ||||||||
| 总计Total | 81 | 21 | 5 | 25 | 28 | 1 | 1 | 55 | 26 | ||||||||
新窗口打开|下载CSV
此外,3个标记对No.61(RS)、No.62(RS)的鉴定结果一致,两个样品均不存在无核标记。综上,利用3个分子标记对‘红地球’ב黎明无核’的F1杂交单株鉴定时,5U_VviAGL11与p3_VvAGL11的准确率及无核检测率较高,且都有较低的假阳性和假阴性率,鉴定结果基本一致,因此均可用于无核葡萄育种的分子标记辅助育种。
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4SSR标记p3_VvAGL11对‘红地球’ב黎明无核’杂交后代的检测
Fig. 4Detection of SSR marker p3_VvAGL11 for the progenies of Red Globe × Dawn Seedless
3 讨论
葡萄是一种基因型高度杂合的多年生果树,童期较长,育种速度慢。尤其对于无核表型的筛选,通过传统手段只能等到植株成年才能进行选育,而利用分子标记对幼苗进行早期验证可以提高育种效率,加快育种进程。其中,选择适用且准确率高的分子标记是有效进行分子标记辅助育种的关键。前人利用BSA、基因组测序、QTL关联分析等方法先后开发了与无核主要位点SDI连锁的分子标记[1,8,9,11-16,22,28-29]。由于每个分子标记开发的遗传背景不同,其鉴定准确率及通用性无法进行比较,且可操作性有差异,因此本试验选择其中5个与SDI位点连锁紧密的分子标记在183份材料中进行鉴定。3.1 分子标记在葡萄品种中的鉴定
屈田田等[30]和LI等[17]研究发现,SCAR标记GSLP1-569在检测‘无核白’的后代时显示出更高的准确性。本研究中,GSLP1-569检测出的14个无核品种中有11个品种均由‘无核白’衍生而来,表明GSLP1-569更适合对‘无核白’及其亲缘关系较近的无核品种进行无核鉴定,这与前人研究结果类似。对于SCAR标记SCF27-2000,AKKURT等[24]研究发现该标记更适于无核×无核杂交后代中的无核性状鉴定,LI等[17]的研究证实了这一点。在本研究中,SCF27-2000在无核品种中的鉴定准确率高达98.4%,进一步印证了前人的观点。CABEZAS等[22]利用QTL定位到与葡萄无核性状紧密连锁的SSR标记VMC7f2,且检测到198-bp等位点与无核表型显著关联,与MEJIA等[31]的研究结果相似。BENNICI等[25]的研究也表明,VMC7f2的198-bp与无核性相关,200-bp等位点与有核表型相关。本研究中,用VMC7f2在96个品种中检测到189-bp与无核性状显著关联,191-bp与有核表型相关,但与前人扩增片段长度有所偏差,因此在后期结果比对时需要加上标准样品,以避免仪器误差。分子标记座位与目标基因座位之间的遗传距离决定了分子标记辅助选择的准确率。前人研究[8,22,31]表明SSR标记VMC7f2为最接近SDI位点的分子标记。本研究中,与SCAR标记GSLP1-569和SCF27-2000相比,VMC7f2在96个品种间鉴定的准确率(85.4%)及无核检测率(85.5%)均较高,说明VMC7f2更接近SDI位点。在基因VvAGL11的启动子区域开发了与SDI位点高度连锁不平衡的分子标记p3_VvAGL11,该标记鉴定的表型—基因型间有很高的一致性[2],使它成为无核分子标记辅助育种的主要候选标记之一。OCAREZ等[16]研究发现p3_VvAGL11的等位点194-bp与无核表型显著关联。本研究中,检测到187-bp与无核性状显著相关,且其鉴定的准确率(89.6%)及无核检测率(90.7%)较高。另有研究表明[25],p3_VvAGL11对于‘Melissa’‘Crimson Seedless’‘Sugraeighteen’‘Early Gold’‘Sugrathirteen’及‘Pink Muscat’均表现出较好的预测能力。本研究用p3_VvAGL11对葡萄材料的检测准确率为89.6%,这与前人的结果相一致,也表明了p3_VvAGL11的通用性强。与p3_VvAGL11相似,5U_VviAGL11标记也位于基因VvAGL11的启动子区域,其等位点306-bp与无核性状极显著相关,虽然本研究中也观察到270-bp和298-bp与无核相关,但这可能是假基因型效应,由于携带270-bp的4个无核品种,也都携带无核等位点306-bp;携带298-bp的25个无核品种有23个均携带306-bp。OCAREZ等[16]在209个葡萄品种中进行分子标记5U_VviAGL11的验证,发现其准确性为0.953,阳性预测值为0.836,阴性预测值为1.0,表现出较低的假阳性。同样,在本研究中,5U_VviAGL11在96个葡萄品种的无核鉴定中准确率为88.5%,无核检测率为90.6%,也表明该标记的通用性强。综上,标记p3_VvAGL11和5U_VviAGL11位于基因VvAGL11的启动子区域,推荐为首选的无核性鉴定标记。分子标记p3_VvAGL11和5U_VviAGL11在96个葡萄品种检测中具有有效性,证实了无核性状与VvAGL11之间密切关联,这2个标记在无核育种中有潜在的应用价值。另外,对于体细胞突变品种—‘瑞峰无核’,5个无核分子标记均不能鉴定出来,这与KARAAGAC等[23]关于体细胞突变无核品种‘chasselas apyrene’的研究结果一致,推测体细胞突变导致的无核与‘无核白’的无核性机理可能不同,那么开发分子标记区分体细胞突变体与亲本无性系也将是今后的一个研究方向。3.2 分子标记在杂交后代中的鉴定
由于分子标记为遗传标记,所以对杂交群体进行MAS时,要基于父母本的基因型,选择适用的分子标记。本研究中,杂交组合的无核亲本‘黎明无核’中不存在分子标记GSLP1-569,因此该标记不适于对供试的杂交组合F1代检测。SSR标记VMC7f2在‘黎明无核’(189:197)中虽然有无核等位基因189-bp,且与‘红地球’(191:191)间具有多态性,但等位基因189-bp和191-bp所对应的带型相距太近,采用聚丙烯酰胺凝胶电泳时不易区分。所以本试验利用分子标记SCF27-2000、p3_VvAGL11和5U_VviAGL11对F1杂交单株进行无核性状检测,重点评估其预测能力。BENNICI等[25]的研究表明分子标记SCF27-2000表现出较高的假阳性(17.4%),在本研究中,SCF27-2000也显示出较高的假阳性率(17.28%),与前人研究结果类似。有研究发现[21],SCF27-2000分子标记在无核性鉴定中表现出较低的准确率,是由于在供试样品中出现零等位基因,且该分子标记与SDI位点之间不完全连锁。另外,本研究利用F1杂交单株进一步证实p3_VvAGL11的187-bp和5U_VviAGL11的306-bp等位基因与无核性状显著关联。本研究中,p3_VvAGL11在F1杂交群体的无核性鉴定表现出较低的假阳性率(6.17%),这与OCAREZ等[16]在571个F1代和BERGAMINI等[2]在475个基因型进行无核性鉴定的结果一致。BENNICI等[25]在97个F1基因型的鉴定结果表明,分子标记p3_VvAGL11的无核性预测能力最强,是最有效的无核分子标记。本研究利用p3_VvAGL11进行无核性鉴定时,出现的假阳性率较低,鉴定的准确率及无核检测率高,说明p3_VvAGL11是良好的无核基因候选标记。此外,利用5U_VviAGL11对F1杂交单株进行无核性鉴定,该标记的准确率为88.89%,与p3_VvAGL11标记基本一致,前者的假阳性率略低于后者。总体而言,通过对杂交群体的无核性鉴定,发现分子标记p3_VvAGL11和5U_VviAGL11的无核鉴定准确率较高,且采用传统的电泳方法即可实现对这两个标记的检测,便于操作,成本低,是可靠的无核基因分子标记。
4 结论
SSR标记p3_VvAGL11的187-bp、5U_VviAGL11的306-bp以及VMC7f2的189-bp与无核性状显 著相关。以p3_VvAGL11和5U_VviAGL11的准确率和无核检测率高,假阳/假阴性率低,且应用成本低,是可靠的无核分子标记,可应用于无核葡萄育种。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1186/1471-2229-11-57URL [本文引用: 9]
DOI:10.1007/s12033-013-9654-8URL [本文引用: 4]
DOI:10.1093/oxfordjournals.jhered.a111419URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
DOI:10.1186/1471-2229-8-38URL [本文引用: 4]
DOI:10.1007/s001220050976URL [本文引用: 4]
DOI:10.1104/pp.18.00259URL [本文引用: 1]
[本文引用: 5]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
DOI:10.1007/s11032-004-7651-0URL
[本文引用: 2]
[本文引用: 2]
DOI:10.3390/genes11020151URL [本文引用: 7]
[本文引用: 4]
[本文引用: 2]
DOI:10.1007/s13205-019-1993-0URL [本文引用: 1]
[本文引用: 1]
DOI:10.4236/abb.2014.52016URL [本文引用: 2]
DOI:10.1139/g06-122URL [本文引用: 5]
[本文引用: 2]
DOI:10.4238/2012.August.13.2URL [本文引用: 2]
DOI:10.1016/j.scienta.2019.03.060URL [本文引用: 5]
[本文引用: 1]
DOI:10.1093/bioinformatics/16.5.412URL [本文引用: 1]
[本文引用: 1]
DOI:10.1038/s41438-018-0089-zURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
