 ,, 蔡禾, 李利, 王林杰, 仲涛, 张红平
,, 蔡禾, 李利, 王林杰, 仲涛, 张红平 ,四川农业大学畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130
,四川农业大学畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130Alternative Splicing of TNNT3 and Its Effect on the Differentiation of MuSCs in Goat
CHEN Yuan ,, CAI He, LI Li, WANG LinJie, ZHONG Tao, ZHANG HongPing
,, CAI He, LI Li, WANG LinJie, ZHONG Tao, ZHANG HongPing ,Farm Animal Genetic Resources Exploration and Innovation Key Laboratory of Sichuan Province, Sichuan Agricultural University, Chengdu 611130
,Farm Animal Genetic Resources Exploration and Innovation Key Laboratory of Sichuan Province, Sichuan Agricultural University, Chengdu 611130通讯作者:
责任编辑: 林鉴非
收稿日期:2020-09-9接受日期:2021-05-13
| 基金资助: |
Received:2020-09-9Accepted:2021-05-13
作者简介 About authors
陈媛,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (3340KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
陈媛, 蔡禾, 李利, 王林杰, 仲涛, 张红平. 山羊TNNT3基因可变剪切及其对骨骼肌细胞分化的作用. 中国农业科学, 2021, 54(20): 4466-4477 doi:10.3864/j.issn.0578-1752.2021.20.019
CHEN Yuan, CAI He, LI Li, WANG LinJie, ZHONG Tao, ZHANG HongPing.
开放科学(资源服务)标识码(OSID):

0 引言
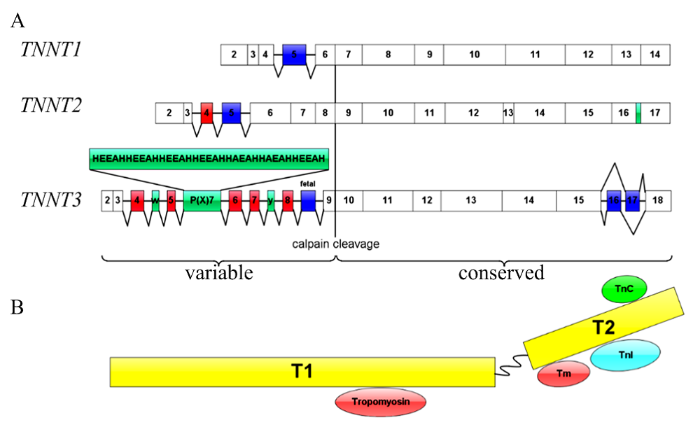
【研究意义】解析动物产肉性状形成的内在分子机制一直是肉用家畜领域研究的核心问题。肌钙蛋白(troponin, Tn)属于钙离子结合蛋白多基因家族的成员。肌钙蛋白复合物作为对钙敏感的分子开关,位于横纹肌的细丝上,能调节横纹肌收缩以响应细胞内钙浓度的变化[1]并影响家畜肉质性状[2,3]。简州大耳羊是我国培育的第二个肉用山羊新品种,具有体型大、生长速度快、耐粗饲、繁殖能力高、抗病能力强等特点。为了探究肌钙蛋白在山羊肌肉生长发育中的作用,为此研究了快速骨骼肌肌钙蛋白T(fast skeletal troponin T3,TNNT3)在简州大耳羊不同组织和不同发育阶段中的可变剪切及表达模式。【前人研究进展】Tn由EBASHI 于1963年首先发现[4],它是由3个亚基组成的复合物:分别是与钙结合的肌钙蛋白C(TNNC);原肌球蛋白Tm结合亚基肌钙蛋白T(TNNT);抑制肌肉收缩的肌钙蛋白I(TNNI)[5]。TNNT是肌钙蛋白复合物与Tm 之间的连接结构,是对Ca2+敏感及Ca2+介导的肌动球蛋白ATP酶活性活化所必要的成分[6],在向肌节发送Ca2+信号中起着关键作用。哺乳动物的TNNT是一种在动物体肌肉和器官组织熟化过程中容易降解的原纤维蛋白[7]。其分子量在31—32 kD之间,约含有223—305个氨基酸,由一个球形C端和一个杆状N端组成,呈不对称结构(图1-B)。TNNT具有3个亚型并在脊椎动物中呈现组织特异性:慢骨骼肌肌钙蛋白、心肌肌钙蛋白和快速骨骼肌肌钙蛋白,这3种亚型分别由基因TNNT1、TNNT2和TNNT3编码[8]。TNNT3与TNNT1、TNNT2最大的区别是可以发生复杂的可变剪切(图1-A)。在高脂饮食诱导的TNNT3选择性剪切中对饱和脂肪酸起到了修饰作用,如大鼠骨骼肌通过pre-mRNA选择性剪接修饰基因表达而对饮食中脂肪过量消耗作出反应。有趣的是,这种效应只存在于富含猪油的饮食中,因为富含单不饱和脂肪酸或多不饱和脂肪酸的饮食对TNNT3替代剪接没有影响[9]。然而,饱和脂肪酸是否直接作用于肌肉细胞以调节替代的pre-mRNA剪接未被评估,饱和脂肪酸促进选择性剪切改变的信号通路也尚不清楚。此外,TNNT3还参与骨骼肌的生长发育,是平滑肌生长和胎儿出生后生存所必需的[10]。例如在小鼠成肌细胞C2C12肌管分化过程中,TNNT3 mRNA、蛋白的表达显著升高,TNNT3剪接形式也由D0时的3种低丰度表达增加到D8时肌管中有15种未在成肌细胞中检测到的剪接形式[11]。【本研究切入点】选择性剪接是一种在转录后RNA水平上调控基因表达的重要机制,它涉及顺式和反式作用因子的动态相互作用以协调剪接体在前体mRNA (pre-mRNA)上的位置[12,13,14]。选择性剪接是pre-mRNA转化为成熟mRNA过程中的一个关键步骤,通过不同的剪切方式从一个基因中产生多个成熟的mRNA和结构功能差异的翻译产物[15],可以显著扩大转录组,同时增加蛋白质组的可塑性和功能性[16,17]。TNNT3基因已经在人、猪、斑马鱼、原鸡、牛和老鼠有一定研究[18,19],在牛的肌肉组织中也发现了不同类型的TNNT3剪切体,但目前关于山羊TNNT3可变剪切体的序列分析、表达规律以及具体功能尚未有报道。【拟解决的关键问题】本研究以简州大耳羊骨骼肌为研究材料,克隆山羊TNNT3基因可变剪切体的CDS序列,检测其在出生后3 d简州大耳羊(母羊3只)不同组织(心脏、肝脏、脾、肺、肾脏、背最长肌和半膜肌)以及不同发育阶段(胚胎期75、90、105 d以及出生后3、45、150和300 d)山羊肌肉组织中TNNT3 mRNA水平的表达差异,初探TNNT3基因在山羊肌肉生长发育过程中的表达模式,为揭示山羊TNNT3基因可变剪切体在哺乳动物中的作用及进一步拓展肉羊肌肉生长发育的分子调控机制提供科学依据。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1肌钙蛋白T的分子结构和互作蛋白(B)以及3种TnT亚型可变剪切外显子结构(A)示图(根据文献[20,21,22]改编)
A:3种TnT亚型可变剪切外显子结构示图: 红色表示可变剪切外显子;蓝色表示胚胎特异性外显子;绿色为鸟类特有的外显子;B:肌钙蛋白T的互作蛋白
Fig. 1Molecular structure and interaction proteins of Troponin T (B) and alternative spliced exons of three TnT isoforms (A), (Adapted from literature[20-22])
A: Alternative spliced exons of three TnT isoforms: Red box indicated alternatively spliced exons; Blue box indicated embryonic specific exons; Green box indicated avian exons; B: Proteins interact with TnT
1 材料与方法
本试验于2019年在四川农业大学畜禽遗传资源发掘与创新利用四川省重点实验室完成,试验涉及的所有过程均严格遵循动物福利进行。1.1 样品采集
大多数动物肌纤维发育在胚胎中后期,并在出生前就己确定肌纤维数目,出生后不再增加。动物出生后早期,肌纤维会随着骨骼肌对代谢与功能需求的改变而发生转变,运动、激素、神经刺激、不同生理状态等均可引起不同程度的骨骼肌肌纤维类型的转变[23]。所以本次试验动物样品选择了胚胎期75、90和105 d(E75、E90、E105)以及出生后3、45、150和300 d(B3、B45、B150、B300)共7个时间点的简州大耳羊(每个时期3只母羔)的背最长肌、半膜肌以及心、肝、脾、肺、肾组织样品,胚胎期羔羊通过剖腹产手术从妊娠母羊子宫中取出。整个采样操作在无菌条件下进行,半小时内采集清洗样品后,立刻冻入液氮中低温运回实验室后,置于-80℃超低温冰箱保存。1.2 RNA的抽提、检测与反转录
将样品在液氮不完全挥发条件下研磨成粉末状,取100 mg组织样采用Trizol(Invitrogen USA)法提取样品总RNA。利用凝胶成像分析仪(Bio-Bad)以及核酸蛋白仪测定RNA 18S与28S条带亮度和RNA在260nm和280nm下吸光度值及比值,初步判定质量并记录总RNA浓度与纯度。利用反转录试剂盒(PrimeScrip RT reagent Kit,TaKaRa,Japan)将RNA反转录为cDNA置于-20℃保存备用。1.3 山羊TNNT3基因的克隆
在NCBI数据库中选择山羊TNNT3(NM_ 001314210.1)基因和牛TNNT3(XM_010821200)基因保守区域,用Primer Premier 6.0设计山羊TNNT3基因CDS区分子克隆引物P1(表1)。以山羊不同时期肌肉组织cDNA混合样为模板,PCR扩增山羊TNNT3基因CDS区序列。PCR反应体系为50µL,包括2×Master Mix Taq酶20 µL,P1上、下游引物(10 µmol·L-1)各2 µL,cDNA 1.5 µL,ddH2O 24.5 µL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s, 58 ℃ 30 s, 72 ℃ 50 s, 36个循环;72 ℃延伸5 min,最后12 ℃保存。反应产物于1.5 %琼脂糖凝胶进行电泳,凝胶成像分析仪成像分析电泳条带,琼脂糖凝胶DNA纯化回收试剂盒(Omega,USA)进行产物回收。根据TaKaRa pMD®19-T Vector(Japan)使用说明,向1.5 mL离心管中依次加入pMD®19-T Vector 0.5 µL,PCR回收产物3.0 µL,分子生物学用水1.5 µL,Solution I 5.0 µL,连接后的载体转化至E.coli DH5α感受态细胞,将初步鉴定出的阳性单克隆菌液利用DL2000 DNA marker比对筛选出和目的条带有差异的条带,取500 μL菌液样品送至成都擎科梓熙生物技术有限公司进行测序。Table 1
表1
表1TNNT3基因引物序列信息
Table 1
| 引物名称 Primer name | 引物序列(5′— 3′) Primer sequence (5′— 3′) | 产物长度 Product length (bp) | 退火温度 Annealing temperature (℃) | 用途 Purpose | |
|---|---|---|---|---|---|
| P1 | TNNT3 | F: ACCATGTCGGACGAGGAAGT R: CACTCTACTTCCAGCACCC | 835 | 58.0 | 克隆CDS区全长 Cloning the full length of CDS |
| P2 | q-TNNT3 | F:AGGAGGGCTGAGGACGAT R:CGGTCTCCAGCTTGTAC | 253 | 52.7 | 定量PCR Quantitative PCR |
| P3 | TNNT3_1-5 | F: ACCATGTCGGACGAGGAAGT R:CTCCTTGAGGGCGACCAGC | 253 | 60.0 | 半定量PCR Semi-quantitative PCR |
| P4 | TNT- TNNT3_3 | F:CCCAAGCTTATGTCGGACGAGGAAGTCGA R:CCGCTCGAGCTTCCAGCGCCCGCCAACTT | 957 | 58.9 | TNT体外转录载体构建 Construction of TNT transcriptional vector in vitro |
| P5 | pTNNT3_3 | F:CCGCTCGAGATGTCGGACGAGGAAGTCGA R:CCCAAGCTTCTTCCAGCGCCCGCCAACTT | 957 | 59.2 | 过表达载体构建 Construction of overexpression vectors |
| P6 | β-actin | F:CCTGCGGCATTCACGAAACTAC R:ACAGCACCGTGTTGGCGTAGAG | 87 | 59.7 | 定量PCR Quantitative PCR |
新窗口打开|下载CSV
1.4 TNNT3生物信息学分析
将测序所得序列利用BLAST在线进行序列比对确认基因克隆准确性,使用ORF Finder(1.5 实时荧光定量及半定量
根据克隆出的山羊TNNT3新转录本序列,选用β-actin(NM_001009784)作为内参基因,采用Primer Premier 6.0在保守区域设计定量引物P2和P6(表1)。所用real-time PCR系统为Bio-Rad CFX96实时荧光定量PCR仪。扩增体系10 μL包括TB Green 5 μL,上、下游引物(10 µmol·L-1)各0.4 μL,cDNA 0.8 μL,灭菌蒸馏水3.4 μL。反应条件:95℃预变性30 s;95℃ 5 s;引物特异退火温度Tm 30 s,39个循环;以0.5℃/s的速度从65 ℃缓慢递增到95℃。设立3个生物学重复,每个样品3次技术重复。在各转录本差异序列的两端设计半定量引物P3(表1),对各转录本进行半定量检测PCR反应条件:95℃ 5 min;95℃ 30 s,Tm ℃ 30 s,72℃ 50 s,26个循环;72℃延伸5 min,最后12℃保存。1.6 TNT体外转录翻译
综合考虑TNNT3_3 转录本CDS区自带的酶切位点以及pCMV-Flag载体所自带的酶切位点,选定限制性内切酶Hind III和Xho I。结合酶切保护碱基以及CDS区序列信息设计TNT引物P4(表1)。利用设计引物进行PCR扩增,切胶回收产物送样测序正确后与pCMV-Flag载体同时进行双酶切。酶切反应体系:2×buffer 5 µL,胶回收产物/空载5 µL,限制性内切酶Hind III和Xho I各1 µL,ddH2O 38 µL。利用T4 DNA连接酶构建载体,反应体系为10 μL(目的片段的酶切胶回收产物7.2 μL,酶切后的空载回收产物0.8 μL,T4及T4 buffer各1 μL)。克隆获得的菌液利用E.Z.N.A.Endo-free Plasmid Mini kit II Spin Protocol 质粒提取试剂盒完成质粒提取。体外转录翻译使用试剂盒进行。1.7 Western Blot
翻译蛋白的定量参照BCA蛋白浓度测定试剂盒(P0010S)检测浓度后计算含10—50 μg蛋白的溶液体积上样。SDS-PAGE 电泳是采用10 %分离胶和4 %浓缩胶,95 °C变性5 min。电泳时设置电压40 V,电泳25 min,后用100 V,电泳80 min。蛋白质转移到PVDF膜按Bio-Rad蛋白转移装置说明进行(组装滤纸凝胶纤维素夹层,恒压选25 V,电泳30 min);转膜完毕后,将膜正面朝上完全浸入封闭液中,依据封闭液说明书,37 °C放置1.5—2 h。一抗4°C孵育过夜;用TBST漂洗4次,每次10 min,二抗37 °C孵育1.5—2 h,再用TBST漂洗3—5次,每次10 min。按照显色试剂盒进行操作显色后用成像仪照相得到蛋白图片。1.8 TNNT3_3过表达检测
利用过表达引物P5(表1)构建过表达质粒(载体构建及质粒提取操作与TNT体外转录翻译相同)。将MuSCs(畜禽遗传资源挖掘与创新利用四川省重点实验室分离冻存)以2.5×104个/孔的密度铺至6孔板中,汇合度为80 %进行瞬时转染,每孔用250 µL Opti-DMEM (无血清)分别室温孵育3 μg质粒DNA和2 µL lip2000 5 min,后混合孵育20 min;将孵育好的转染液逐滴加入到已更换为无血清培养基的细胞中,轻轻晃动6孔板;转染4—6 h后更换为10 % FBS的生长培养基;转染24 h改成分化培养基,通过显微镜观察绿色荧光蛋白判断转染效率;转染48 h收取对照组和处理组细胞进行RNA抽提。对标志基因进行RT-qPCR定量检测。1.9 免疫荧光
将山羊MuSCs以2.5×104个/孔的密度铺至35 mm单孔板,汇合度为80%进行转染,转染24 h后诱导分化。分化7d后用PBS轻轻晃动清洗3次,每次5 min;加入4%多聚甲醛,室温固定15 min;PBS晃动清洗3次,每次5 min,加入0.5 %曲拉通trion-X-100 4℃处理10 min;PBS晃动清洗3次,每次5 min,用2% BSA 37℃封闭2 h;弃去封闭液,加入稀释好的一抗,4℃孵育过夜;PBS晃动清洗3次,每次5 min,避光加入稀释好的二抗,37 ℃孵育2 h;避光条件下PBS晃动清洗3次,每次5 min,加入0.05%的DAPI染液,37℃避光孵育10 min;避光条件下PBS晃动清洗3次,每次5 min,在倒置荧光显微镜下成像拍照。1.10 数据分析
对扩增效率达到95%—105%的产物,根据Bio-Rad CFX96自带分析软件计算各基因各时间点的拷贝数,以β-actin作为内参基因,采用2-ΔΔCt法[24]进行目的基因相对表达量的计算。运用软件SAS 9.1的PROC GLM程序进行最小二乘分析,结果以最小二乘均数±标准误表示,并利用Duncan法进行均数间的多重比较。2 结果
2.1 山羊TNNT3基因的克隆及序列分析
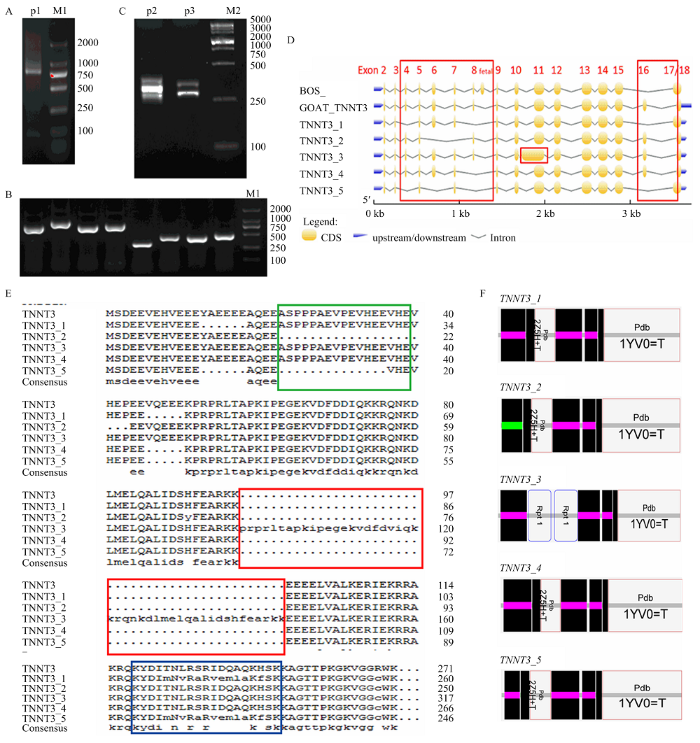
测序结果表明,利用引物P1以山羊背最长肌及半膜肌cDNA混合样作为模板进行PCR扩增得到产物长度为835 bp(图2-A),TA克隆后随机挑选单克隆菌落接种菌液并PCR扩增测序(图2-B),根据 NCBI在线BLAST比对获得了山羊TNNT3基因CDS区5种基因可变剪切体(图2-C):TNNT3_1、TNNT3_2、TNNT3_3、TNNT3_4和TNNT3_5,5种不同可变剪切体的开放阅读框编码起始位点(ATG)与编码终止位点(TAG)均一致,且特异性地表达了互斥外显子Exon 16/17。另外,TNNT3_3的外显子Exon 9—11出现了138 bp的重复(图2-D),这一外显子的重复插入使得TNNT3_3的开放阅读框加长,增加了46个氨基酸残基(图2-E),其余4个转录本均不同程度地缺失了个别外显子,但结构域均未发生改变(图2-F)。NCBI工具ORF Finder预测开放阅读框的长度分别为741、753、783、801和954 bp,其编码的氨基酸序列长度从246 AA 到317 AA,蛋白质分子量(kD)从29.17到37.40,蛋白质等电点(pI)范围从6.72到9.15(表2)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2山羊TNNT3基因PCR扩增、克隆及不同可变剪切体序列与山羊全基因组数据库比对
A:TNNT3 CDS区全长PCR产物(P1:835bp);B:TNNT3可变剪切体单克隆菌液PCR产物;C:TNNT3可变剪切体差异片段(exon4-8:193-253 bp)qPCR产物;M1:DNA marker DL 2000 bp;M2: DNA marker DL 5000 bp;D:TNNT3不同可变剪切体序列与山羊全基因组数据库比对结果,红色方框是发生可变剪切的外显子位点;E:山羊TNNT3不同可变剪切体的氨基酸序列对比;绿色方框:骨骼肌中最先被降解的产物[18];红色方框:TNNT3_3的外显子Exon 9—11重复,增加了46个氨基酸残基;蓝色方框:互斥表达的外显子16/17编码的氨基酸[25];F:TNNT3可变剪切体结构域预测
Fig. 2PCR amplification, cloning results of TNNT3 gene in goats and comparison results of different alternative transcripts sequences with the goat genome database
A: PCR products of TNNT3 full-length CDS region (835bp); B: PCR products of TNNT3 isoforms from monoclonal bacteria solution; C: qPCR products with TNNT3 transcripts at alternative fragment (Exon4-8: 193bp-253bp ; M1: DNA Marker DL 2000 bp; M2: DNA Marker DL 5000 bp; D: Comparison results of different alternative transcripts sequences with the Goat genome database; The red box is an exon site that can be alternative splicing; E: Amino acid sequences of different alternative transcripts of TNNT3 in goats; Green box: The first degraded product in skeletal muscle[18]; Red box: the Exon 9-11 of TNNT3_3 is repeated with 46 amino acid; Blue box: Exon 16 and 17 coding amino acid are alternatively spliced in a mutually exclusive manner[25]; F: TNNT3 alternative transcripts domain prediction
Table 2
表2
表2山羊TNNT3不同可变剪切体预测的氨基酸序列信息
Table 2
| 序列名称 Sequence name | CDS区长度 Length of CDS (nt) | 外显子数 Exon count | 氨基酸长度 Amino acid length (AA) | 蛋白质分子量 Protein molecular weight (kD) | 等电点 Isoelectric point |
|---|---|---|---|---|---|
| TNNT3_1 | 783 | 15 | 260 | 30.64 | 8.61 |
| TNNT3_2 | 753 | 15 | 250 | 29.73 | 8.30 |
| TNNT3_3 | 954 | 20 | 317 | 37.40 | 6.77 |
| TNNT3_4 | 801 | 16 | 266 | 31.39 | 6.72 |
| TNNT3_5 | 741 | 14 | 246 | 29.17 | 9.15 |
新窗口打开|下载CSV
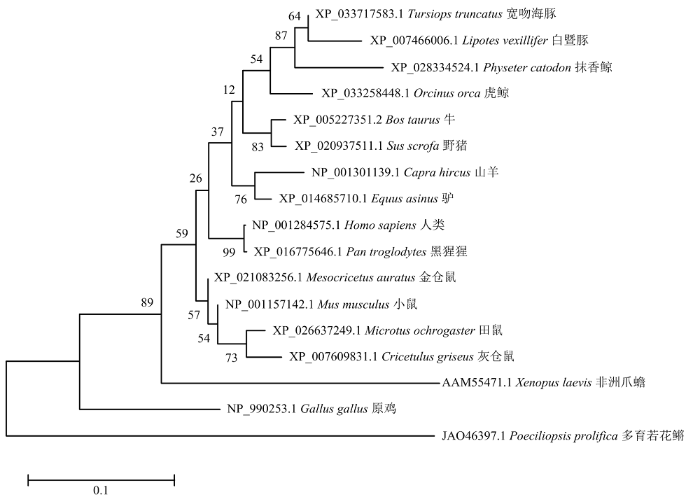
从NCBI上下载其他物种TNNT3氨基酸序列并利用MEGE 10.1.8构建进化树(图3),发现山羊TNNT3氨基酸序列系统发育进化关系最近的有哺乳动物牛(Bos taurus)、驴(Equus asinus)、猪(Sus scrofa)和人(Homo sapiens),而与爬行类(Xenopus laevis)和鸟类(Gallus gallus)的同源关系较远。此外,山羊TNNT3基因核苷酸序列在哺乳动物中高度保守,例如,山羊TNNT3_1和TNNT3_4转录本序列与绵羊、藏羚羊、牛、猪、狒狒、小鼠和兔子的基因序列相似性分别为99%、98%、98%、97%、95%、95%及91%;TNNT3_2和TNNT3_5转录本与绵羊、牛、猪、人和小鼠的相似性也较高性(分别为99%、99%、95%、96% 及91%,但TNNT3_3转录本序列与绵羊(94%)、藏羚羊(93%)、牛(94%)、猪(92%)、狒狒(94%)、小鼠(94%)的相似性则相对较低。
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3TNNT3氨基酸序列的系统发育进化分析
Fig. 3Phylogenetic tree and evolutionary analysis of the TNNT3 amino acid sequence
2.2 山羊TNNT3基因的组织表达分析
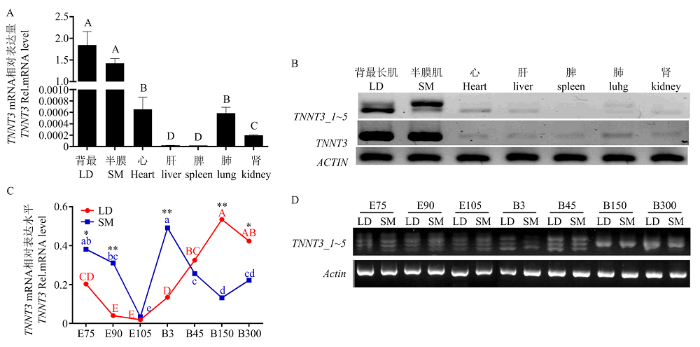
利用荧光定量及半定量PCR检测出生后3d的羔羊心、肝、脾、肺、肾以及背最长肌和半膜肌组织中TNNT3 mRNA水平表达,发现荧光定量(图4-A)及半定量(图4-B)结果一致:TNNT3 mRNA在检测的7个组织中都有表达,其中在背最长肌及半膜肌中的表达最高(P<0.01),心脏及肺次之(P<0.01),其余组织中的表达量相对较低(P>0.05)。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4山羊TNNT3不同可变剪切体组织分布比及不同时期可变剪切体的表达差异
A: TNNT3在不同组织中的mRNA水平;B: TNNT3可变剪切体在不同组织中的半定量电泳结果;C: TNNT3在背最长肌和半膜肌不同发育阶段中的mRNA水平;D: TNNT3可变剪切体在背最长肌(LD)和半膜肌(SM)不同发育阶段中的半定量电泳结果;图中所标不同字母表示差异极显著(P<0.01);同一时期两组织间的表达差异显著性用*(P<0.05)**(P<0.01)表示
Fig. 4The different alternative transcripts of goat TNNT3 distribution and expression levels at different stages
A: The mRNA level of TNNT3 in different tissues; B: The semi-quantitative result of goat TNNT3 alternative transcripts in different tissues; C: The mRNA level of TNNT3 in different development stages of LD and SM; D: The semi-quantitative result of TNNT3 alternative transcripts in different development stages of LD and SM; The different capital letter/ lowercase letter indicated extremely significant difference (P<0.01);During the same period, the significant of expression between the two tissues was expressed as * (P<0.05) ** (P<0.01)
2.3 山羊TNNT3不同转录本在胚胎期及出生后不同发育阶段的表达分析
根据上述TNNT3的组织表达结果,选择简州大耳羊在胚胎期(E75、E90和E105)和出生后(B3、B45、B150和B300)两个时间段共7个时间点的背最长肌(LD)和半膜肌(SM)检测TNNT3 mRNA基因表达(图4-C)。背最长肌和半膜肌组织中TNNT3 mRNA均呈现“下降-上升-下降”的表达趋势。在胚胎期LD和SM中TNNT3均是E75相对表达量高于其余两个胚胎时间点(E90和E105),且在半膜肌的表达量高于背最长肌;出生后45 dTNNT3在背最长肌的表达量高于半膜肌,B150达到峰值,显著高于B3和B45的表达量(P<0.01)。此外,随着山羊日龄增长TNNT3转录本数量减少,在B150和B300山羊LD和SM中不能明显区分多种转录本(图4-D),说明TNNT3各转录本可能在山羊胚胎期肌肉生长发育中起着重要的调控作用。2.4 TNNT3_3的TNT检测及过表达
为了初步探究TNNT3对肌细胞分化的影响,笔者在鉴定出的5种转录本中选取与其他物种序列相似性最低的TNNT3_3, 首先成功构建TNNT3_3体外转录翻译载体(图5-A),确定了TNNT3_3具有转录翻译蛋白的能力且蛋白大小与预期基本相符(图5-B)。进一步在山羊MuSCs中将转录本TNNT3_3过表达,分别检测增殖期(GM)和分化期(DM)基因的表达水平,发现与对照组相比,虽然过表达使增殖期细胞中TNNT3_3表达水平提高上万倍,但Pax7、Myomaker和MyoG表达量差异不显著,而MyH4 mRNA水平约上升1.5倍(P<0.05,图5-C GM);而在分化条件下,肌分化标志基因Myomaker、MyoG和MyH4相较于对照组均极显著升高(P<0.01),增殖相关基因Pax7表达仍未改变(图5-C DM)。与此相对应,过表达TNNT3_3后诱导分化7 d免疫荧光检测MyHC信号,发现TNNT3_3可以明显促进MyHC表达和肌管形成(图5-D)。图5
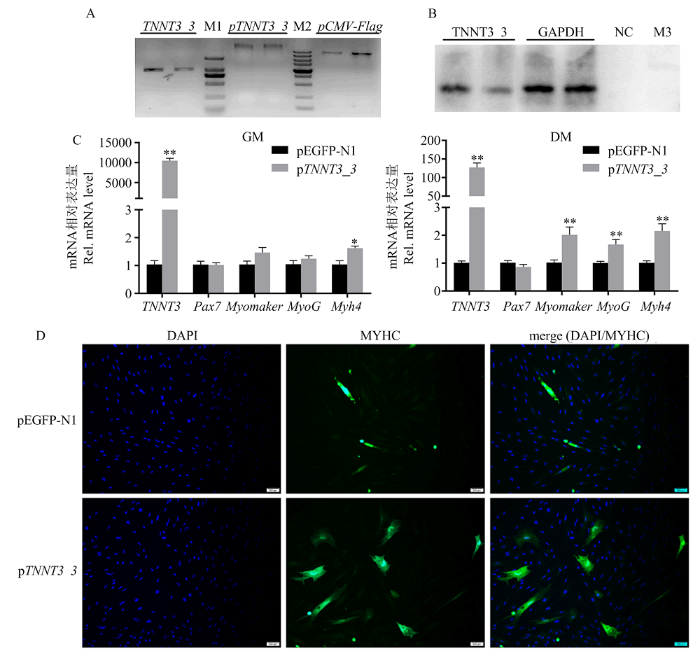 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5山羊TNNT3_3体外转录及对细胞分化的作用
A:TNNT3_3体外转录载体构建;B:TNNT3_3体外转录蛋白免疫印迹结果;C:TNNT3_3过表达后增殖GM/分化DM时期标志基因mRNA水平;D:分化7 d后MyHC免疫荧光结果;M1:DNA marker DL 2000 bp;M2:DNA marker DL 5000 bp;M3:蛋白marker
Fig. 5Transcription in vitro and overexpression of TNNT3_3 and its effect on the differentiation in goat MuSCs
A: Construction of TNNT3_3 transcriptional vector in vitro; B: Western blot results of TNNT3_3 transcriptional protein in vitro; C: mRNA levels of marker genes in the proliferative/ differentiation stage of MuSCs treated with TNNT3_3 vector; D: MyHC immunofluorescence results after differentiation for 7 days; M1:DNA marker DL 2000 bp;M2:DNA marker DL 5000 bp;M3:Protein marker
3 讨论
利用并整合其他哺乳动物已报道TNNT3基因序列,成功地克隆分离了山羊肌肉组织TNNT3基因的5个可变剪切体。同时,根据氨基酸序列构建的系统进化树表明,山羊TNNT3与绵羊和牛的氨基酸序列高度相似,归属于哺乳动物。值得注意的是,比对结果显示虽然TNNT3_1、TNNT3_2、TNNT3_4、TNNT3_5外显子均有不同程度的缺失,但开放阅读框的起始位点及结束位点均一致,这也证明了TNNT3基因在哺乳动物中序列高度保守的结论。在牛肌肉组织的免疫鉴定中发现一个TNNT的片段(31—32kD的多肽链)ASPPPAEVPEVHEEVH是骨骼肌中最先被降解的产物,伴随这个降解发生的是肉质嫩化的过程[18]。在克隆获得的山羊序列中也发现了这个片段,但TNNT3_5和TNNT3_2部分或完全缺失该片段,该片段的缺失对肌肉生长发育及肌肉降解的作用以及是否会影响肉质产品的风味还需进一步探索。在与山羊TNNT3基因的其他可变剪切体进行差异比对和分析时笔者发现TNNT3基因核酸序列在外显子缺失位点的两端,A、G含量高且多为重复序列,推测这可能是造成不同剪切形式的原因。这个推测与现代内含子的剪切模式研究观点相同[26],并且基因序列中出现了较多类似的回文结构,这也可能是造成了剪切识别位点错位的原因。肌钙蛋白复合物的3个亚基中的每一个都有多种亚型,这些亚型以不同的组织特异性和发育调控模式表达。在人和小鼠中TNNT3基因由19个外显子构成,包含编码271个氨基酸的CDS区。N末端编码的外显子4、5、6、7和8可发生约几十种选择性剪接[27,28],外显子8和9之间的胎儿外显子也可以剪切并在胚胎快速骨骼肌中独特表达[29],C末端与中间区域高度保守。C端的外显子16和外显子17是在生长发育过程中的剪切调控中相互排斥的[25],这两个外显子共同编码TnC与TnI结合位点的14个氨基酸残基的多肽片段(图1)[28]。通过cDNA序列比较,外显子16主要在成年的快速骨骼肌肌钙蛋白内表达。而外显子17所编码的氨基酸序列与慢骨骼肌肌钙蛋白(ssTnT)和心肌肌钙蛋白(cTnT)中的氨基酸序列具有较高的相似性,并主要在胚胎期以及新生儿的快速骨骼肌肌钙蛋白(TNNT3)中表达。并且,蛋白质相互作用研究表明,外显子17的掺入减弱了快速骨骼肌肌钙蛋白中TNNC与原肌球蛋白的亲和力[30]。
TNNT3基因已知可以调节快速骨骼肌的收缩,但其在肌肉中的具体作用仍存在争议。为确定其在体内的功能,有研究构建了TNNT3+/flox/lacZ小鼠,结果发现,TNNT3lacZ/+小鼠体型在整个发育过程中都比野生型小,TNNT3lacZ/lacZ胚胎比杂合子小,并在出生后不久死亡。总的来说,这个研究表明TNNT3在肌肉中表达是肌肉生长发育所必需的,并且是正常生长和新生小鼠产后呼吸生存所必需的[10]。饮食特异性对TNNT3 pre-mRNA选择性剪接的影响是由高脂猪油饮食中特定类型的脂肪酸或神经酰胺在肌肉内的积累引起的[9]。如PP1对于神经酰胺诱导的Bcl-xL和caspase-9 pre-mRNAs[31]替代剪接的改变是必要的。其他研究也报道了饮食或肥胖导致的pre-mRNA选择性剪接的变化[32,33,34],但控制代谢的激素反馈使体内调节选择性剪接的机制复杂化,并且对于特定脂肪酸在这一过程中的作用目前知之甚少。此外,因为承重肌肉会收到关于体重的机械反馈,并消耗ATP以产生足以抵消重力的能量。有研究利用大鼠研究了体重的变化如何影响TNNT3的选择性剪接,但结果发现其剪接形式与夜间能量消耗显著相关,而与自身体重无关[34],这一结果也进一步证实TNNT3通过协调肌丝的钙离子浓度依赖性ATP酶活性,从而在肌细胞收缩和舒张过程中发挥作用。
本研究以简州大耳羊为研究对象,实时荧光定量检测其组织表达结果显示,TNNT3 mRNA在肌肉组织中表达最高,且在出生前后均处于较高的表达水平。肌纤维的发育可分为4个阶段:中胚层干细胞分化为成肌细胞,成肌细胞融合为肌管细胞,肌管细胞转变为肌纤维,最后肌纤维生长至成熟。大多数动物肌纤维发育的前三个过程在动物胚胎期就己完成,肌纤维数目也在出生前就已确定[35,36,37]。动物骨骼肌在生长发育过程中,其肌纤维组成类型并不是完全固定的,肌纤维类型经过一个成熟过程以达到成年的分布模式,特别是在动物出生早期,肌纤维的收缩和代谢特性会随着骨骼肌对代谢与功能需求的改变而发生转变。本试验结果中,TNNT3 mRNA在背最长肌和半膜肌中的表达始终处于一个动态变化中。半膜肌属于I型肌纤维又称慢收缩型肌纤维,具有收缩缓慢的特性;这种类型肌纤维含有丰富的线粒体,表现出更强的氧化代谢能力,在耐力型工作中发挥主导作用。背最长肌主要是II型纤维又称快收缩型纤维,具有收缩速度快、易疲劳的特性;这种类型的肌纤维主要在强度要求高、速度要求快的运动中发挥作用。定量结果显示出生后半膜肌中TNNT3 mRNA的表达持续下降,背最长肌中TNNT3 mRNA的表达持续上升,并在快速发育以及成年阶段背最长肌中的表达都极显著高于半膜肌,且在150 d达到了峰值,这个结果符合TNNT3基因目前已知的生物学功能及表达特性。目前许多的资料表明,运动、激素、神经刺激、不同生理状态等均可引起不同程度的骨骼肌肌纤维类型的转变[23]。小鼠中的研究发现,TNNT3是产后及生长的肌肉中所必需[10],在C2C12中的表达量和可变剪切体的形式随着肌管分化逐渐增加[11],表明TNNT3在肌肉生长发育中发挥着重要的生物学功能。在发育过程中,TNNT3基因各转录本的表达在骨骼肌中经历了高分子量至低分子量的剪接形式转换[38],该转换也代表了低等电点结合形式向高等电点形式的转变,本研究也有类似的结果(图4)。目前对于TNNT3可变剪切体的研究集中在人和小鼠,不同的可变剪切体通过编码出不同肌钙蛋白亚型发挥生理作用。如TNNT3可能与远侧关节的挛缩有关,但其具体的生物学效应目前还不明确[1, 39-40]。在人的肌肉萎缩研究中发现,TNNT1的缺乏不影响TNNT3剪接形式的发育转换,这表明TNNT3 pre-mRNA的选择性剪接与骨骼肌纤维类型无关[38]。此外,已有研究证明,虽然TNNT3的N末端可变区不与细丝调节系统中的任何已知蛋白质结合,但TNNT3的N末端片段中的选择性剪接已经显示出对TNNT3蛋白的总体构象以及对TNNI和原肌球蛋白的结合亲和力的影响[41]。
4 结论
在山羊上成功获得TNNT3五种可变剪切转录本的CDS序列,山羊TNNT3基因与绵羊、牛、猪等哺乳动物具有很高的相似性,与鱼类爬行类动物一致性较低,证明TNNT3基因序列在哺乳动物中高度保守。TNNT3主要在肌肉组织(背最长肌和半膜肌)中高表达,并且过表达TNNT3_3后,可以促进山羊MuSCs分化,表明TNNT3在肌肉生长发育中具有潜在的重要生物学功能。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 2]
URL [本文引用: 1]
DOI:10.1016/j.gene.2015.07.038URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
DOI:10.1016/0022-2836(82)90540-XURL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 2]
[本文引用: 3]
[本文引用: 2]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 4]
URL [本文引用: 1]
URL [本文引用: 1]
DOI:10.1093/nar/gkw703URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 2]
DOI:10.1006/meth.2001.1262URL [本文引用: 1]
[本文引用: 3]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
DOI:10.1242/jeb.023903URL [本文引用: 1]
URL [本文引用: 2]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 2]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
